Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Bitkilerdeki MikroRNA Transkripsiyonlarını Doğru ve Verimli Bir Şekilde Analiz Eden Bir Biyoinformatik Boru Hattı
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bir biyoinformatik boru hattı, yani miRDeep-P2 (kısaca miRDP2), güncellenmiş bitki miRNA kriterleri ve elden algoritması ile, doğru ve verimli bitkilerde mikroRNA transkripsiyon analiz edebilir, özellikle karmaşık ve büyük genomları olan türler için.
Özet
MikroRNA'lar (miRNA'lar) 20-24 nükleotit (nt) endojen küçük RNA'lar (sRNA'lar) gen ekspresyonunun transkripsiyon sonrası düzeyde düzenlenmesinde güçlü rol oynayan bitki ve hayvanlarda yaygın olarak mevcuttur. Son on yılda miRNA transkripsiyonlarını tanımlamak ve analiz etmek için sRNA kitaplıklarının Yeni Nesil Sıralama (NGS) yöntemlerine göre sıralanması yaygın olarak kullanılmıştır ve bu da miRNA keşfinde hızlı bir artışa yol açmıştır. Bununla birlikte, sıralı sRNA kütüphanelerinin derinliğinin artmasının yanı sıra bitki genomlarının büyüklüğü ve karmaşıklığı nedeniyle bitki miRNA ek açıklamalarında iki büyük zorluk ortaya çıkar. İlk olarak, sRNA'ların diğer birçok türü, özellikle, sRNA kütüphanelerinden kısa müdahale rna'lar (siRNA'lar) hatalı bir şekilde birçok hesaplama aracı tarafından miRNA olarak eklenmiştir. İkinci olarak, büyük ve karmaşık genomlara sahip bitki türlerinde miRNA transkripsiyonlarının analizi için son derece zaman alan bir süreç haline gelir. Bu zorluklarıaşmak için miRDeep-P'yi (miRNA transkriptom analizleri için popüler bir araç) miRDeep-P2'ye (kısaca miRDP2) yeni bir filtreleme stratejisi uygulayarak, puanlama algoritmasını elden geçirerek ve yeni güncellenen bitki miRNA'sını birleştirerek yükselttik. ek açıklama kriterleri. MiRDP2'yi Arabidopsis, pirinç, domates, mısır ve buğday dahil olmak üzere genomik karmaşıklığı artan beş temsili bitkide sıralı sRNA popülasyonlarına karşı test ettik. Sonuçlar, miRDP2'nin bu görevleri çok yüksek verimlilikle işledi. Buna ek olarak, miRDP2 duyarlılık ve doğruluk ile ilgili diğer tahmin araçları geride. Birlikte ele alındığında, sonuçlarımız miRDP2 bitki miRNA transkriptomları analiz etmek için hızlı ve doğru bir araç olarak göstermek, bu nedenle toplumun daha iyi bitkilerde miRNA'lar açıklama yardımcı yararlı bir araçtır.
Giriş
Biyolojide son yirmi yılda yapılan en heyecan verici keşiflerden biri, sRNA türlerinin genom1'inçeşitli işlevlerini düzenlemedeki çoğalan rolüdür. Özellikle miRNA'lar ökaryotlarda 20 ila 24-nt sRNA'ların önemli bir sınıfını oluşturur ve esas olarak transkripsiyon sonrası düzeyde yaşam döngüsü gelişim aşamalarında belirgin gen düzenleyicileri olarak işlev görürler, ayrıca uyarıcı ve stres yanıtlarında2,3. Bitkilerde miRNA'lar pri-miRNA adı verilen birincil transkriptlerden kaynaklanmaktadır, bunlar genellikle RNA polimeraz II tarafından bireysel transkripsiyon üniteleri4,5olarak yazılır. Evrimsel olarak korunmuş hücresel makineler (hayvanlarda Drosha RNase III, bitkilerde DICER benzeri) tarafından işlenen pri-miRNA'lar, moleküler kök-döngü yapılarını oluşturan dizileri içeren miRNA öncüleri, pre-miRNA'lariçineboşaltılır. Pre-miRNA'lar daha sonra çift iplikli ara lara, yani miRNA dublekslere, fonksiyonel iplikçik, olgun miRNA ve daha az sık fonksiyonel ortak olan miRNA*2,8'denoluşan olarak işlenir. RNA kaynaklı susturma kompleksine (RISC) yüklendikten sonra, olgun miRNA'lar dizi tamamlayıcılığına dayalı olarak mRNA hedeflerini tanıyabilir ve bu da olumsuz bir düzenleyici fonksiyon2,8ile sonuçlanır. miRNA'lar ya hedef transkriptlerini bozabilir ya da hedef çevirisini engelleyebilir ler ama eski şekilde bitkilerde hakimdir8,9.
Nematod Caenorhabditis elegans10ilk miRNA tesadüfi keşfinden bu yana,11, çok araştırma miRNA tanımlama ve fonksiyonel analizi, özellikle NGS yönteminin kullanılabilirliğinden sonra taahhüt edilmiştir. NGS yönteminin geniş uygulaması, miRNA'ların benzersiz özelliğini yakalamak için tasarlanmış hesaplama araçlarının kullanımını büyük ölçüde teşvik etmiştir, örneğin öncüllerin kök-döngü yapısı ve olgun miRNA ve miRNA*'da okunan sıraların tercihli birikimi gibi. Sonuç olarak, araştırmacılar çeşitli türlerde miRNA'ları belirlemede kayda değer bir başarı elde etmişler. Daha önce açıklanan olasılık modeli12dayanarak , biz miRDeep-P13geliştirdi , HANGI NGS verilerinden bitki miRNA'ları keşfetmek için ilk hesaplama aracı oldu. miRDeep-P özellikle daha değişken öncüuzunluğu ve büyük paralogous aileler13,14,15içeren bitki miRNA'ları çözme zorlukları fethetmek amaçlandı . Yayımlanmasından sonra, bu program binlerce kez indirildi ve 40'tan fazla bitki türleri16miRNA transkriptomes açıklama için kullanılır. miRDeep-P gibi NGS tabanlı araçlar tarafından tahrik, kamu miRNA depo miRBase17kayıtlı miRNA sayısında dramatik bir artış olmuştur , 38.000 miRNA öğeleri şu anda barındırılan nerede (sürüm 22.1) sadece ~ 500 miRNA öğeleri (sürüm 2.0) ile karşılaştırıldığında 200818.
Ancak, bitki miRNA ek açıklama iki yeni sorunlar ortaya çıkmıştır. İlk olarak, yanlış pozitif lerin yüksek oranları aşağıdaki nedenlerle bitki miRNA ek açıklamaları16,19 kalitesini büyük ölçüde etkilemıştır: 1) NGS sRNA kütüphanelerinden endojen kısa müdahale RNA'ların (siRNA'lar) bir tufanı, katı bir miRNA açıklama kriterinin olmaması nedeniyle hatalı bir şekilde miRNA olarak anons edildi; 2) priori miRNA bilgisi olmayan türler için NGS verilerine dayalı olarak öngörülen yanlış pozitifleri ortadan kaldırmak zordur. Örnek olarak miRBase'i kullanan Taylor ve ark.20, 21numaralı kamu deposundaki bitki miRNA girişlerinin üçte birinin ikna edici destekleyici kanıtlardan yoksun olduğunu ve hatta bitki miRNA ailelerinin dörtte üçünün bile şüpheli olduğunu buldular. İkinci olarak, büyük ve karmaşık genomları16ile bitki miRNA'ları tahmin etmek için son derece zaman alıcı bir süreç haline gelir. Bu zorluklarıaşmak için miRDeep-P'yi yeni bir filtreleme stratejisi ekleyerek, puanlama algoritmasını elden geçirerek ve tesis miRNA ek açıklamaiçin yeni kriterleri entegre ederek güncelledik ve yeni miRDP2 sürümünü yayınladık. Buna ek olarak, miRDP2'yi NGS sRNA veri setlerini kullanarak test ettik ve giderek artan genom boyutları na sahipolduk: Arabidopsis, pirinç, domates, mısır ve buğday. Diğer beş yaygın olarak kullanılan araçlar ve eski sürümü ile karşılaştırıldığında, miRDP2 bu sRNA verileri ayrıştırılmış ve geliştirilmiş doğruluk ve hassasiyet ile daha hızlı miRNA transkripsiyonu analiz.
miRDP2 paketinin içeriği
MiRDP2 paketi, hazırlanan bash komut dosyası tarafından sırayla çalıştırılması gereken altı belgelenmiş Perl komut dosyasından oluşur. Altı komut dosyasından üçü(convert_bowtie_to_blast.pl, filter_alignments.pl, ve excise_candidate.pl) miRDeep-P'den miras kalır. Diğer komut dosyaları orijinal sürümden değiştirilir. Altı komut dosyasının işlevleri aşağıdaki şekilde açıklanmıştır:
preprocess_reads.pl filtreler giriş çok uzun veya çok kısa (<19 nt veya >25 nt) okumaları da dahil olmak üzere okur ve Rfam ncRNA dizileri ile ilişkili okur, yanı sıra RPM ile okur (Milyon Başına Okur) az 5. Komut dosyası daha sonra bilinen miRNA olgun dizileri ile ilişkili okur alır. Giriş dosyaları FASTA/FASTQ biçiminde orijinal okumalar ve miRNA ve ncRNA dizilerine eşleme okumanın bowtie2 çıktısI.
RPM hesaplama formülü aşağıdaki gibidir:
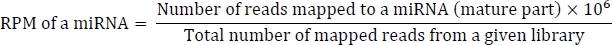
convert_bowtie_to_blast.pl bowtie biçimini BLAST-ayrıştı biçimine dönüştürür. BLAST-parsed biçimi standart NCBI BLASToutput biçiminden türetilen özel bir tabular ayrılmış biçimidir.
filter_alignments.pl derin sıralama okumalarının hizalamalarını bir genoma filtreler. Kısmi hizalamaları ve çok hizalı okumaları (kullanıcı tarafından belirtilen frekans kesme) filtreler. Temel giriş BLAST-parsed biçiminde bir dosyadır.
excise_candidate.pl, hizalanmış okumaları yönergeler olarak kullanarak bir başvuru dizisinden potansiyel öncül dizilerini keser. Temel giriş BLAST-parsed biçiminde bir dosya ve FASTA dosyasıdır. Çıktı FASTA formatında tüm potansiyel öncü dizileridir.
mod-miRDP.pl iki giriş dosyası, imza dosyası ve çekirdek miRDeep-P algoritmasından bitki özel parametreleri ile puanlama sistemi değiştirerek değiştirilir yapı dosyası gerekir. Giriş dosyaları nokta-köşeli öncül yapı dosyasıdır ve dağıtım imza dosyasını okur.
mod-rm_redundant_meet_plant.pl üç giriş dosyaları gerekir: chromosome_length, öncüleri ve original_prediction mod-miRDP.pl tarafından oluşturulan. Yeni güncellenen tesis miRNA ölçütlerine göre filtrelenmiş, gereksiz tahmin edilen ve öngörülen dosya olmak üzere iki çıktı dosyası oluşturur. Çıktı dosyasının biçimiyle ilgili ayrıntılar bölüm 1.4'te açıklanmıştır.
Protokol
1. Kurulum ve test
- İndir gerekli bağımlılıkları: Bowtie222 ve RNAfold23. Derlenmiş paketler önerilir.
- Download Bowtie2, bir okuma haritalama aracı, kendi ev sitesinden(http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- RNAfold, Viyana paketinin bir araç rnaikincil yapısı tahmin etmek için kullanılan bir araç, http://www.tbi.univie.ac.at/~ivo/RNA/ .
- MiRDP2'yi yüklemeden önce, bu iki bağımlılığın doğru şekilde yüklendiğinden emin olun ve bu iki bağımlılık için doğru bir YOL ayarlamak için bash ortamı dosyasını (örn. .bashrc) özelleştirin.
NOT: Bowtie24 gibi diğer haritalama araçları da miRDP2 için uygundur; bowtie veya Bowtie2 sürüm 1.1.3'ten sonra kullanılabilir.
- MiRDP2 paketini indirmek için https://sourceforge.net/projects/mirdp2/files/latest_version/ gidin ve tarball dosyalarını getirin.
- miRDP2'yi yüklemeden önce Perl'in PATH'de olduğundan emin olun. miRDP2'yi yüklemek için, indirilen tarball dosyasının tüm içeriğini tek bir klasöre ayıklayın (komut satırları 1.4.2'deki gibi) ve ardından klasör yolunu PATH'e ayarlayın.
NOT: MiRDP2 çalıştırmak için en az 8 GB RAM ve 100 GB depolama alanına sahip bir bilgisayar veya bilgisayar düğümü önerilir. - MiRDP2 boru hattını test edin.
- miRDP2'nin doğru yüklenip yüklenmediğini test etmek için test verilerini ve https://sourceforge.net/projects/mirdp2/files/TestData/bulunan beklenen çıktıyı kullanın. Test verileri bir biçimlendirilmiş GSM sıralama dosyası ve bir Arabidopsis thaliana genom dosyası içerir.
- İndirilen tüm dosyaları geçerli çalışma dizinine taşıyın:
mv miRDP2-v*.tar.gz TestData.tar.gz ncRNA_rfam.tar.gz
cd - Sıkıştırılmış tarball dosyalarını ayıklayın:
katran –xvzf miRDP2-v*.tar.gz
katran –xvzf TestData.tar.gz
katran –xvzf ncRNA_rfam.tar.gz - Arabidopsis genom referans indeksi oluşturun:
bowtie2-build -f ./TestData/TAIR10_genome.fa ./TestData/TAIR10_genome - NCRNA başvuru dizini oluşturun:
bowtie2-build -f ./ncRNA_rfam.fa ./1.1.3/script/index/rfam_index - MiRDP2 ardışık hattını çalıştırın:
bash ./1.1.3/miRDP2-v1.1.3_pipeline.bash –g ./TestData/TAIR10_genome.fa -i ./ TestData/TAIR 10_genome –f ./TestData/GSM2094927.fa –o .
NOT: Kullanılan Linux komutları kalın ve italik yazı tiplerinde, italik komut satırı seçenekleri yle birliktekullanılır. *miRDP2 sürümünü gösterir (geçerli sürüm 1.1.3'tür). Bowtie2-build komutu yaklaşık 10 dakika sürer ve miRDP2 boru hattı birkaç dakika içinde bitirmelidir
- Test çıktılarını kontrol edin.
- 'GSM2094927-15-0-10' adlı bir klasörün otomatik olarak içinde , tüm ara dosyaları ve sonuçları içeren oluşturulduğunu unutmayın.
- Sekme-delimited çıkış dosyası GSM2094927-15-0-10_filter_P_prediction, öngörülen miRNA'ların son çıktısı, kromozom id, iplikyönü, temsilci id okur, öncül id, olgun miRNA konumu, öncül gösteren sütunlar içerir konumu, olgun sırası ve öncül sırası. Daha fazla analiz kolaylaştırmak için bu dosyadan türetilen ek yatak dosyasına dikkat edin.
- Tamamlanan adımlar hakkında bilgi sağlayan "progress_log" dosyasını ve program çıktısı ve uyarıları içeren "script_log" ve "script_err" dosyalarını denetleyin.
NOT: Şu anda, bir küme sunucusunda CentOS sürümü 6.5 ve PC Windows sisteminde Cygwin 2.6.0 dahil olmak üzere iki Linux platformunda miRDP2 test ettik ve miRDP2 Perl destekleyen benzer sistemler üzerinde çalışması gerekir.
2. Roman miRNA'ları tanımlama
- Ardışık işlemi çalıştırmadan önce, giriş okumalarının uygun biçimde önceden işlendiğinden emin olun.
NOT: MiRDP2'nin yeni sürümü 1.1.3 orijinal FASTQ formatında dosyaları giriş olarak kabul edebilir, ancak okuma biçimlendirme işlemi önceki sürümlerde olduğu gibi gerçekleştirilir.- İlk olarak, derin sıralama okumalarının 5' ve 3' ucundaki bağdaştırıcıları çıkarın (varsa).
- İkinci olarak, derin sıralama fasta biçimine okur ayrıştın.
- Üçüncü olarak, aynı sırayla okur gibi artıklık kaldırmak tek ve benzersiz FASTA girişi ile temsil edilir.
- Son olarak, tüm FASTA tanımlayıcılarının benzersiz olduğundan emin olun. Her dizi tanımlayıcısı, derin sıralama veri kümelerinde alınan tam sıranın kopya numarasını gösteren bir '_x' ve bir tamsayı ile bitmelidir. Benzersiz FASTA tanımlayıcısını sağlamanın bir yolu, çalışan bir numarayı kimliknumarasına eklemektir. Başvuru için, test verilerinde(https://sourceforge.net/projects/mirdp2/files/TestData/)GSM2094927.fa dosyasına bakın.
- Doğru biçimlendirilmiş okuma örnekleri için aşağıdakileri görün:
>read0_x29909
TTTGGATTGAAGGGAGCTCTA
>read1_x36974
TTCCACAGCTTTTTTGAACTG
>read2_x32635
TTCCACAGCTTTTTTGAACTT
- Başvuru endeksleri oluşturun.
- Genom referansı için, zaman kazanmak için, ilgi türlerinin genom dizileri indekslenmişse, iGenomes web sitesinden Bowtie2 indeks dosyalarını indirin(https://support.illumina.com/sequencing/sequencing_software/igenome.html). Aksi takdirde, kullanıcılar başvuru dizilerini diziye diziler ve genom dizisinin yeniden dizilişi gerekebileceğinden proje bitene kadar dizin dosyasını bir süre saklar. Bir genom referansInin nasıl indekslendirilene ilişkin ayrıntılar bowtie2 kılavuzunda(http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml)yer almaktadır.
- Diğer kodlamayan RNA parçalarından gürültülü dizileri filtrelemek için miRNA olmayan başka bir ncRNA dizin ihtir. Dosya rRNA, tRNA, snRNA ve snoRNA dahil olmak üzere Rfam ana ncRNA dizileri, bir koleksiyondur. Bu dizini oluşturmak için, dizin doğru yerleştirilmeli ve adlandırılması gerektiği için lütfen bölüm 1.4'e bakın, yani /script/index/rfam_index.
- MiRDP2 çalıştırın.
- Derin sıralama verilerinden yeni miRNA'ları algılamak için miRDP2'yi kullanmak için, çözümleme ardışık hattını başlatmak için paketteki bash komut dosyasını çalıştırın (Bir örnek adım 1.4'te bulunabilir):
>path_to_miRDP2_folder>/miRDP2-v*.*_pipeline.bash –g -i -f -o
nerede * boru hattı bash komut dosyasının sürümünü gösterir. Değiştirilebilir üç parametre vardır: 1) bir okuma farklı konumların sayısı eşlenebilir, 2) bowtie2 çalıştırmak için uyumsuzluk numarası, ve 3) RPM eşiği (Milyon Başına Okur). Bunları sırasıyla -L, -M ve –R seçeneklerini kullanarak değiştirin. Ayrıntılı bir açıklama bölüm 3.1 bulunmaktadır.
- Derin sıralama verilerinden yeni miRNA'ları algılamak için miRDP2'yi kullanmak için, çözümleme ardışık hattını başlatmak için paketteki bash komut dosyasını çalıştırın (Bir örnek adım 1.4'te bulunabilir):
- MiRDP2 çıktılarını kontrol edin.
- Çıktı klasörünotomatik olarak , altında oluşturulacağını ve '-15-0-10' adlı; son 3 sayı, sırasıyla 1, 2 ve 3 parametreleri için değerleri (bu durumda varsayılan) gösterir. _filter_P_prediction dosyası, yeni güncellenen tesis miRNA ek açıklama kriterlerini karşılayan son öngörülen miRNA'ların bilgilerini içerir. Çıktı dosyasının biçimiyle ilgili ayrıntılar bölüm 1.4'te açıklanmıştır.
3. MiRDP2 kullanarak değişiklikler ve dikkat
- Değiştirilebilen parametreler
- Bir okumanın eşlenebileceği konum sınırını ayarlamak için '-L' seçeneğini kullanın (parametre 1). Okuma eşleme çok fazla site büyük olasılıkla yineleme dizileri ile ilişkilidir ve miRNA'lar olası değildir. Varsayılan ayar 15'tir. Belirli türler için, çok sayıda üyesi olan miRNA aileleri varsa, ilk parametre genom ortamına uyum sağlamak için el ile artırılabilir.
- Papyon için izin verilen uyuşmazlıkları ayarlamak için '-M' seçeneğini kullanın (parametre 2). Varsayılan ayar 0'dır.
- Olgun miRNA'lara (parametre 3) karşılık gelen okumaların eşiğini ayarlamak için '-R' seçeneğini kullanın. Zaman tüketimini ve yanlış pozitifleri azaltmak için RPM tarafından filtre okunur. Yalnızca belirli bir RPM eşiğini aşan okumalar arka plan gürültüsü yerine olgun miRNA dizilerini temsil edebilir ve daha fazla analiz için saklanır. Varsayılan ayar 10 RPM'dir.
- Bu parametreleri değiştirmenin performans ve zaman tüketimini etkileyebileceğini unutmayın. Genel olarak, parametre 1 ve 2'nin artması ve parametre 3'ün azalması daha az sıkı bir sonuç ve daha uzun çalışma süresi ve tam tersi oluşturur.
- Artıklık ve miRNA*
- MiRDP2'den çıkan çıkış miRNA'ların bilinen miRNA'lardan farklı olabileceğini unutmayın. Bunun başlıca iki nedenden biri olduğunu bulduk: olgun miRNA'ların heterojenliği veya miRNA ve miRNA*'nın göreceli bolluğu. Bunun öncüllerin optimal uzunluk seçimini ve bilinen miRNA genlerinin profilini etkilemediğini bulduk.
Sonuçlar
MiRNA ek açıklama boru hattı, miRDP2, burada açıklanan Arabidopsis thaliana, Oryza sativa (pirinç), Solanum lycopersicum (domates), Zea mays (mısır) ve Triticum aestivum (buğday)(Şekil 1A)dahil olmak üzere, giderek artan genom uzunluğu ile 5 bitki türünden 10 kamu sRNA-seq kütüphaneleri uygulanır. Genel olarak, her tür için, farklı dokulardan 2 temsilci sRNA kütüphaneleri (benzersiz okumalar halinde çökmü?...
Tartışmalar
NGS gelişiyle, miRNA loci çok sayıda çeşitli türler de sRNA sıralama veri giderek artan miktarda tespit edilmiştir29,30. Merkezi topluluk veritabanı miRBase21,yatırılan miRNA öğeleri son on yılda neredeyse 100 kat artmıştır. Ancak, hayvanlarda miRNA'lara kıyasla, bitki miRNA'ları tanımlama/ek açıklamayı daha karmaşık hale getiren birçok benzersiz özelliğe sahiptir13,
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma Pekin Tarım ve Orman Bilimleri Akademisi (KJCX201917, KJCX20180425 ve KJCX20180204) tarafından XY ve Çin Ulusal Doğa Bilimleri Vakfı (31621001) ll'ye destek verilmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
Referanslar
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır