Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un pipeline de bioinformatique pour analyser avec précision et efficacité les transcriptomes microARN dans les plantes
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un pipeline bioinformatique, à savoir miRDeep-P2 (miRDP2 pour faire court), avec des critères de miARN végétal mis à jour et un algorithme révisé, pourrait analyser avec précision et efficacité les transcriptomes de microARN chez les plantes, en particulier pour les espèces ayant des génomes complexes et volumineux.
Résumé
Les microARN (miARN) sont de 20 à 24 nucléotides (nt) endogènes petits ARN (sRNAs) largement existant dans les plantes et les animaux qui jouent un rôle puissant dans la régulation de l'expression des gènes au niveau post-transcriptionnel. Le séquençage des bibliothèques de sRNA par les méthodes de séquençage de la prochaine génération (NGS) a été largement utilisé pour identifier et analyser les transcriptomes de miRNA au cours de la dernière décennie, ce qui a entraîné une augmentation rapide de la découverte de miRNA. Cependant, deux défis majeurs se posent dans l'annotation de miRNA de plante due à la profondeur croissante des bibliothèques séquentielles de sRNA aussi bien que la taille et la complexité des génomes de plante. Tout d'abord, de nombreux autres types d'ARNr, en particulier, les ARN à court interférant (siRNAs) des bibliothèques d'ARNde, sont annotés à tort comme miRNAs par de nombreux outils informatiques. Deuxièmement, il devient un processus extrêmement long pour analyser les transcriptomes miRNA chez les espèces végétales avec des génomes grands et complexes. Pour surmonter ces défis, nous avons récemment mis à niveau miRDeep-P (un outil populaire pour les analyses de transcriptome miRNA) à miRDeep-P2 (miRDP2 pour faire court) en employant une nouvelle stratégie de filtrage, en remaniant l'algorithme de notation et en incorporant miRNA usine nouvellement mis à jour critères d'annotation. Nous avons testé miRDP2 contre des populations d'ARNs séquentielles dans cinq plantes représentatives présentant une complexité génomique croissante, y compris l'arabidopsis, le riz, la tomate, le maïs et le blé. Les résultats indiquent que miRDP2 a traité ces tâches avec une très grande efficacité. En outre, miRDP2 a surpassé les autres outils de prédiction en ce qui concerne la sensibilité et la précision. Pris ensemble, nos résultats démontrent miRDP2 comme un outil rapide et précis pour analyser les transcriptomes miRNA des plantes, donc un outil utile pour aider la communauté à mieux annoter miRNAs dans les plantes.
Introduction
L'une des découvertes les plus passionnantes des deux dernières décennies en biologie est le rôle proliférant des espèces d'ARNs dans la régulation des diverses fonctions du génome1. En particulier, les miARN constituent une classe importante de 20 à 24 sRNAs dans les eucaryotes, et fonctionnent principalement au niveau post-transcriptionnel en tant que régulateurs génétiques de premier plan tout au long des étapes de développement du cycle de vie ainsi que dans les réponses de stimulus et de stress2,3. Chez les plantes, les miARN proviennent de transcriptions primaires appelées pri-miRNAs, qui sont généralement transcrites par l'ARN polymérase II comme unités de transcription individuelles4,5. Traitées par des machines cellulaires évolutivement conservées (Drosha RNase III chez les animaux, comme dicER chez les plantes), les pri-miRNAs sont excisées dans les précurseurs immédiats de miRNA, les pré-miRNAs, qui contiennent des séquences formant des structures intramoléculaires de boucle de tige6,7. Les pré-miRNAs sont ensuite transformés en intermédiaires à double brin, à savoir les duplex miRNA, constitués du brin fonctionnel, du miRNA mature et du partenaire moins fréquemment fonctionnel,miRNAMD 2,8. Après avoir été chargés dans le complexe de silençage induit par l'ARN (RISC), les miARN matures pouvaient reconnaître leurs cibles d'ARNm en fonction de la complémentarité de la séquence, ce qui a entraîné une fonction réglementaire négative2,8. miRNAs pourrait soit déstabiliser leurs transcriptions cibles ou empêcher la traduction cible, mais l'ancienne manière est dominée dans les plantes8,9.
Depuis la découverte fortuite du premier miRNA dans le nématode Caenorhabditis elegans10,11, beaucoup de recherche a été engagée à l'identification miRNA et son analyse fonctionnelle, en particulier après la disponibilité de la méthode NGS. La large application de la méthode NGS a grandement favorisé l'utilisation d'outils informatiques conçus pour saisir la caractéristique unique des miARN, tels que la structure en boucle de tige des précurseurs et leur accumulation préférentielle de lectures de séquence séquencés sur le miRNA mature et le miRNAMD. En conséquence, les chercheurs ont obtenu un succès remarquable dans l'identification des miARN chez diverses espèces. Basé sur un modèle de probabilité précédemment décrit12,nous avons développé miRDeep-P13, qui a été le premier outil de calcul pour découvrir des miARN végétaux à partir de données NGS. miRDeep-P visait spécifiquement à surmonter les défis du décodage des miARN végétaux avec une longueur de précurseur plus variable et de grandes familles paralogues13,14,15. Après sa sortie, ce programme a été téléchargé des milliers de fois et utilisé pour annoter les transcriptomes miRNA dans plus de 40 espèces végétales16. Propulsé par des outils basés sur NGS comme miRDeep-P, il ya eu une augmentation spectaculaire du nombre de miRNAs enregistrés dans le dépôt de miRNA public miRBase17, où plus de 38.000 articles miRNA sont actuellement hébergés (libération 22.1) par rapport à seulement 500 articles miRNA (libération 2.0) en 200818.
Cependant, deux nouveaux défis ont surgi de l'annotation de miRNA de plante. Tout d'abord, des ratios élevés de faux positifs ont eu un impact important sur la qualité des annotations miRNA végétales16,19 pour les raisons suivantes : 1) un déluge d'ARN endogènes brefs interférants (siRNAs) des bibliothèques ngS sRNA ont été annotés à tort comme miRNAs en raison de l'absence d'un miRNA rigoureux et de critères; 2) pour les espèces sans information a priori miRNA, les faux positifs prédits sur la base des données NGS sont difficiles à éliminer. En utilisant miRBase à titre d'exemple, Taylor et coll.20 ont constaté qu'un tiers des entrées de miRNA des plantes dans le dépôt public21 (libération 21) n'avaient pas de preuves convaincantes à l'appui et même les trois quarts des familles de miRNA végétaux étaient discutables. Deuxièmement, il devient un processus extrêmement long pour prédire les miARN végétaux avec des génomes grands et complexes16. Pour surmonter ces défis, nous avons mis à jour miRDeep-P en ajoutant une nouvelle stratégie de filtrage, en remaniant l'algorithme de notation et en intégrant de nouveaux critères pour l'annotation miRNA végétale, et avons publié la nouvelle version miRDP2. En outre, nous avons testé miRDP2 à l'aide d'ensembles de données NGS sRNA avec des tailles de génome s'agrandit progressivement : Arabidopsis, riz, tomate, maïs et blé. Comparé à cinq autres outils largement utilisés et à son ancienne version, miRDP2 a analysé ces données d'ARNs et analysé les transcriptomes de miRNA plus rapidement avec une précision et une sensibilité améliorées.
Contenu du paquet miRDP2
Le paquet miRDP2 se compose de six scripts Perl documentés qui doivent être exécutés de façon séquentielle par le script bash préparé. Des six scripts, trois (convert_bowtie_to_blast.pl, filter_alignments.pl, et excise_candidate.pl) sont hérités de miRDeep-P. Les autres scripts sont modifiés à partir de la version originale. Les fonctions des six scripts sont décrites dans les éléments suivants :
preprocess_reads.pl filtre les lectures d'entrée, y compris les lectures qui sont trop longues ou trop courtes (lt;19 nt ou 'gt;25 nt), et se lit en corrélation avec les séquences Rfam ncRNA, ainsi que des lectures avec RPM (Reads Per Million) moins de 5. Le script récupère ensuite des lectures corrélées à des séquences matures connues de miRNA. Les fichiers d'entrée sont des lectures originales dans le format FASTA/FASTQ et la sortie bowtie2 de lectures cartographiques vers des séquences miRNA et ncRNA.
La formule de calcul du RPM est la suivante :
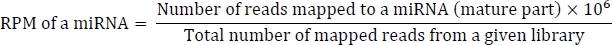
convert_bowtie_to_blast.pl change le format noeud papillon en format BLAST-parsed. Le format blast-parsed est un format tabulaire séparé personnalisé dérivé du format standard NCBI BLASToutput.
filter_alignments.pl filtre les alignements de séquençage profond se lit à un génome. Il filtre les alignements partiels ainsi que les lectures multi-alignées (coupure de fréquence spécifiée par l'utilisateur). L'entrée de base est un fichier en format BLAST-parsed.
excise_candidate.pl élimine les séquences précurseurs potentielles d'une séquence de référence à l'aide de lectures alignées comme lignes directrices. L'entrée de base est un fichier en format BLAST-parsed et un fichier FASTA. La sortie est toutes les séquences précurseurs potentiels en format FASTA.
mod-miRDP.pl a besoin de deux fichiers d'entrée, fichier de signature et fichier de structure, qui est modifié à partir de l'algorithme de base miRDeep-P en modifiant le système de notation avec des paramètres spécifiques à l'usine. Les fichiers d'entrée sont des fichiers de structure précurseur point-bracket et lit le fichier de signature de distribution.
mod-rm_redundant_meet_plant.pl a besoin de trois fichiers d'entrée : chromosome_length, précurseurs et original_prediction générés par mod-miRDP.pl. Il génère deux fichiers de sortie, fichier prévu non redondant et fichier prévu filtré par les critères de miRNA végétale récemment mis à jour. Les détails sur le format du fichier de sortie sont décrits à la section 1.4.
Protocole
1. Installation et essais
- Télécharger les dépendances requises: Bowtie222 et RNAfold23. Les paquets compilés sont recommandés.
- Téléchargez Bowtie2, un outil de cartographie de lecture, à partir de son site d'origine (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- Télécharger RNAfold, un outil du paquet de Vienne utilisé pour prédire la structure secondaire de l'ARN, à partir de http://www.tbi.univie.ac.at/~ivo/RNA/.
- Avant d'installer miRDP2, assurez-vous que ces deux dépendances sont correctement installées, et personnalisez le fichier d'environnement bash (par exemple, .bashrc) pour définir un PATH correct pour ces deux dépendances.
REMARQUE : D'autres outils de cartographie tels que Bowtie24 conviennent également au miRDP2; Bowtie ou Bowtie2 peuvent être utilisés après la version 1.1.3.
- Pour télécharger le paquet miRDP2, rendez-vous dans https://sourceforge.net/projects/mirdp2/files/latest_version/ et allez chercher les fichiers tarball.
- Avant d'installer miRDP2, assurez-vous que Perl est dans le PATH. Pour installer miRDP2, extraire tout le contenu du fichier tarball téléchargé dans un dossier (lignes de commande comme dans 1.4.2), puis définir le chemin du dossier dans le PATH.
REMARQUE : Un nœud informatique ou informatique avec au moins 8 Go de RAM et 100 Go de stockage sont recommandés pour exécuter miRDP2. - Testez le pipeline MiRDP2.
- Pour vérifier si miRDP2 a été correctement installé, utilisez les données de test et la sortie attendue trouvée dans https://sourceforge.net/projects/mirdp2/files/TestData/. Les données de test contiennent un fichier de séquençage GSM formaté et un fichier du génome Arabidopsis thaliana.
- Déplacez tous les fichiers téléchargés vers le répertoire de travail actuel :
mv miRDP2-v.tar.gz TestData.tar.gz ncRNA_rfam.tar.gz -lt;user_selected_folder-gt;
cd lt;user_selected_folder-gt; - Extraire les fichiers de tarball comprimé:
goudron 'xvzf miRDP2-v'.tar.gz
goudron 'xvzf TestData.tar.gz
goudron 'xvzf ncRNA_rfam.tar.gz - Construire l'indice de référence du génome d'Arabidopsis :
bowtie2-build -f ./TestData/TAIR10_genome.fa ./TestData/TAIR10_genome - Construire l'indice de référence ncRNA:
bowtie2-build -f ./ncRNA_rfam.fa ./1.1.3/script/index/rfam_index - Exécuter le pipeline miRDP2 :
bash ./1.1.3/miRDP2-v1.1.3_pipeline.bash ./TestData/TAIR10_genome.fa -i ./ TestData/TAIR 10_genome ./TestData/GSM2094927.fa .o .
REMARQUE : Les commandes Linux utilisées sont en caractères gras et italiques, avec des options de ligne de commande en italique. indique la version de miRDP2 (la version actuelle est 1.1.3). La commande de construction bowtie2 devrait prendre environ 10 minutes, et le pipeline miRDP2 devrait se terminer en quelques minutes
- Vérifiez les sorties de test.
- Notez qu'un dossier nommé 'GSM2094927-15-0-10' est automatiquement généré dans 'lt;user_selected_folder'gt;, contenant tous les fichiers intermédiaires et les résultats.
- Vérifiez que le fichier de sortie délimité par l'onglet GSM2094927-15-0-10_filter_P_prediction, la sortie finale des miARN prédits, contient des colonnes qui indiquent l'id chromosomique, la direction du brin, le représentant lit id, précurseur id, emplacement miRNA mature, précurseur l'emplacement, la séquence mature et la séquence précurseur. Notez le fichier de lit supplémentaire dérivé de ce fichier pour faciliter une analyse plus approfondie.
- Vérifiez le fichier "progress_log", qui fournit des informations sur les étapes terminées, et les fichiers "script_log" et "script_err", qui contiennent la sortie du programme et les avertissements.
REMARQUE: Actuellement, nous avons testé miRDP2 sur deux plates-formes Linux, y compris CentOS version 6.5 sur un serveur de cluster, et Cygwin 2.6.0 sur le système PC Windows, et miRDP2 devrait travailler sur des systèmes similaires qui prennent en charge Perl.
2. Identifier les nouveaux miRNAs
- Avant d'exécuter le pipeline, assurez-vous que les lectures d'entrée sont prétraitées en format approprié.
REMARQUE : La nouvelle version 1.1.3 de miRDP2 peut accepter les fichiers de format FASTQ originaux comme entrées, bien que le processus de mise en forme des lectures soit effectué comme dans les versions précédentes.- Tout d'abord, retirer les adaptateurs des extrémités de 5' et 3' du séquençage profond se lit (si présent).
- Deuxièmement, l'anèsre du séquençage profond se lit dans le format FASTA.
- Troisièmement, supprimer la redondance de telle sorte que les lectures avec séquence identique sont représentés avec une entrée EXPRES unique et unique.
- Enfin, assurez-vous que tous les identificateurs FASTA sont uniques. Chaque identificateur de séquence doit se terminer par un « _x » et un entier, indiquant le numéro de copie de la séquence exacte qui a été récupérée dans les ensembles de données de séquençage profond. Une façon d'assurer l'identifiant FASTA unique est d'inclure un numéro d'exécution dans l'ID. Pour référence, voir le fichier GSM2094927.fa dans les données de test (https://sourceforge.net/projects/mirdp2/files/TestData/).
- Voir ce qui suit pour des exemples de lectures correctement formatées :
read0_x29909
TTTGGATTGAAGGGAGCTCTTA TTTG
read1_x36974
TTCCACAGCTTTTTTTGAACTG
read2_x32635
TTCCACAGCTTTTTTTGAACTT
- Construire des indices de référence.
- Pour la référence du génome, pour gagner du temps, téléchargez les fichiers d'index Bowtie2 sur le site iGenomes(https://support.illumina.com/sequencing/sequencing_software/igenome.html) si les séquences génomiques des espèces d'intérêt ont été indexées. Dans le cas contraire, les utilisateurs indexent les séquences de référence et conservent le fichier d'index pendant un certain temps jusqu'à ce que le projet soit terminé puisque la séquence du génome pourrait devoir être réindexée. Les détails sur la façon d'indexer une référence génomique sont inclus dans le manuel bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml).
- Un autre indice ncRNA non-miRNA est également nécessaire pour filtrer les séquences bruyantes d'autres fragments d'ARN non codants. Le fichier est une collection de séquences principales ncRNA de Rfam, y compris l'ARNr, l'ARTT, le snRNA et le snoRNA. Pour établir cet indice, veuillez vous référer à la partie 1.4, car l'indice doit être placé et nommé correctement, c'est-à-dire le site de l'miRDP2_version./script/index/rfam_index.
- Exécuter miRDP2.
- Pour utiliser miRDP2 pour détecter de nouvelles miRNAs à partir de données de séquençage profond, exécutez le script bash dans le paquet pour démarrer le pipeline d'analyse (Un exemple peut être trouvé à l'étape 1.4):
lt;path_to_miRDP2_folder'/miRDP2-v._pipeline.bash 'g 'lt;genome_file 'gt; -i 'lt;path_to_index/index_prefix 'gt; -f 'lt;seq_file 'gt; 'o 'lt;output_folder 'gt;
où - indique la version du script de bash pipeline. Il y a trois paramètres qui peuvent être modifiés : 1) le nombre d'emplacements différents à laquelle une lecture peut être cartographiée, 2) le nombre d'inadéquation pour l'exécution du noeud papillon2, et 3) le seuil de RPM (Reads Per Million). Modifiez-les en utilisant les options L, M et R, respectivement. Une explication détaillée se trouve à la section 3.1.
- Pour utiliser miRDP2 pour détecter de nouvelles miRNAs à partir de données de séquençage profond, exécutez le script bash dans le paquet pour démarrer le pipeline d'analyse (Un exemple peut être trouvé à l'étape 1.4):
- Vérifiez les sorties miRDP2.
- Notez que le dossier de sortie sera automatiquement généré sous l'output_folder et le nom de «lt;seq_file_name'à 15-0-10'; les 3 derniers numéros indiquent les valeurs (par défaut dans ce cas) pour les paramètres 1, 2 et 3, respectivement. Le fichier 'lt;seq_file_name'_filter_P_prediction contient des informations sur les miRNAs prédits finaux satisfaisant les critères d'annotation de miRNA de plante nouvellement mis à jour. Les détails sur le format du fichier de sortie sont décrits dans la partie 1.4.
3. Modifications et prudence à l'aide de miRDP2
- Paramètres qui peuvent être modifiés
- Utilisez l'option '-L'pour définir la limite du nombre d'emplacements auxquels une lecture peut être cartographiée (paramètre 1). Lire la cartographie à trop de sites sont éventuellement associés à des séquences répétées, et ne sont pas susceptibles de miRNAs. Le paramètre par défaut est de 15. Pour des espèces spécifiques, s'il y a des familles de miRNA avec beaucoup de membres, le premier paramètre peut être augmenté manuellement pour s'adapter au paysage génomique.
- Utilisez l'option '-M'pour définir les décalages autorisés pour le noeud papillon (paramètre 2). Le paramètre par défaut est 0.
- Utilisez l'option '-R' pour définir le seuil pour les lectures potentiellement correspondant aux miARN matures (paramètre 3). Pour réduire la consommation de temps et les faux positifs, filtre lit par RPM. Seules les lectures dépassant un certain seuil de RPM peuvent représenter des séquences matures de miARN plutôt que du bruit de fond, et seraient conservées pour une analyse plus approfondie. Le paramètre par défaut est de 10 tr/min.
- Notez que la modification de ces paramètres peut potentiellement affecter les performances et la consommation de temps. En général, une augmentation des paramètres 1 et 2 et une diminution du paramètre 3 généreraient un résultat moins rigoureux et un temps de fonctionnement plus long et vice versa.
- Redondance et miRNA
- Notez que les miARN de sortie de miRDP2 peuvent différer des miRNAs connus. Nous avons constaté que cela est principalement dû à l'une des deux raisons : l'hétérogénéité des miARN matures ou l'abondance relative de miRNA et de miRNA. Nous avons constaté que cela n'a pas d'incidence sur la sélection optimale des précurseurs et sur le profilage des gènes miARN connus.
Résultats
Le pipeline d'annotation miRNA, miRDP2, décrit ci-dessus est appliqué à 10 bibliothèques publiques de sRNA-seq de 5 espèces végétales dont la longueur du génome a augmenté progressivement, y compris Arabidopsis thaliana, Oryza sativa (riz), Solanum lycopersicum (tomate), Zea mays (maïs) et Triticum aestivum (blé) (Figure 1A). Dans l'ensemble, pour chaque espèce, 2 bibliothèques représentatives de l'ARNM provenant de ...
Discussion
Avec l'avènement de NGS, un grand nombre de loci miRNA ont été identifiés à partir d'une quantité sans cesse croissante de données de séquençage de l'ARNde chez diverses espèces29,30. Dans la base de données communautaire centralisée miRBase21, les éléments miRNA déposés ont augmenté de près de 100 fois au cours de la dernière décennie. Cependant, par rapport aux miRNAs chez les animaux, les miARN végétaux ont de nomb...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par beijing Academy of Agriculture and Forestry Sciences (KJCX201917, KJCX20180425, et KJCX20180204) à XY et National Natural Science Foundation of China (31621001) à LL.
matériels
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
Références
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon