Method Article
Extracción y cuantificación de polifosfatos de inositol solubles y radiomarcados de diferentes especies vegetales utilizando SAX-HPLC
En este artículo
Resumen
Aquí describimos fuerte anión intercambio de cromatografía líquida de alto rendimiento de [3H]-myo-inositol-etiquetado plántulas que es un método altamente sensible para detectar y cuantificar polifosfatos de inositol en las plantas.
Resumen
Los ésteres de fosfato de myo-inositol, también llamados fosfatos de inositol (InsPs), son una clase de reguladores celulares que juegan un papel importante en la fisiología de la planta. Debido a su carga negativa, baja abundancia y susceptibilidad a las actividades hidrolíticas, la detección y cuantificación de estas moléculas es un reto. Este es particularmente el caso de las formas altamente fosforiladas que contienen enlaces difosfos de 'alta energía', también llamados pirofosfatos de inositol (PP-InsPs). Debido a su alta sensibilidad, fuerte anión intercambio de cromatografía líquida de alto rendimiento (SAX-HPLC) de plantas etiquetadas con [3H]-myo-inositol es actualmente el método de elección para analizar estas moléculas. Mediante el uso de [3H]-myo-inositol para la planta de radioetiqueta plant seedlings, varias especies de InsP incluyendo varios isómeros no enantioméricos pueden ser detectados y discriminados con alta sensibilidad. Aquí se describe la configuración de un sistema SAX-HPLC adecuado, así como el flujo de trabajo completo desde el cultivo de plantas, radioetiquetado y extracción InsP hasta la ejecución SAX-HPLC y el posterior análisis de datos. El protocolo presentado aquí permite la discriminación y cuantificación de varias especies de InsP, incluyendo varios isómeros no enantioméricos y de los PP-InsPs, InsP7 e InsP8,y se puede adaptar fácilmente a otras especies vegetales. Como ejemplos, se realizan análisis SAX-HPLC de las plántulas de Arabidopsis thaliana y Lotus japonicus y se presentan y discuten perfiles completos de InsP. El método descrito aquí representa una herramienta prometedora para comprender mejor las funciones biológicas de InsPs en las plantas.
Introducción
Hace casi cuatro décadas, los fosfatos de inositol (InsPs) surgieron como moléculas de señalización, después de Que Ins(1,4,5)P3 (InsP3) fue identificado como un segundo mensajero que activa la liberación mediada por el receptor de Ca2+ en células animales1,2. Hasta la fecha, no se ha identificado ningún receptor InsP3 (IP3-R) en las plantas, lo que cuestiona un papel de señalización directa para InsP3 en las células vegetales3. A pesar de todo, InsP3 sirve como precursor para otros InsPs involucrados en varios procesos de desarrollo de plantas, incluyendo la regulación de vías de señalización específicas3,4,5,6,7,8. Por ejemplo, InsP3 puede ser más fosforilado a InsP6, también conocido como "ácido fítico", que representa una fuente importante de fosfato, myo-inositol y cationes, y se demostró que juegan un papel clave en la defensa de la planta contra patógenos, exportación de ARNm y homeostasis fosfato5,9,10,11,12.
Los pirofosfatos de inositol (PP-InsPs) son una clase de InsPs que contienen al menos un enlace di-fosfo de alta energía, identificado inicialmente en células animales, ameba y levadura, donde desempeñan un papel crítico en varios procesos celulares13,,14,,15. A pesar del trabajo seminal sobre los PP-InsPs en las plantas16,17,18,19,20,21,22,23,24,25,26, las funciones biológicas y la identidad isómero de estas moléculas siguen siendo en gran medida enigmáticas. En la planta modelo Arabidopsis thaliana, se propuso el celular InsP8 para regular las defensas contra los herbívoros de insectos y hongos necrotróficos a través de la detección de coincidencias de InsP8 y jasmonato activo por el complejo receptor ASK1-COI1-JAZ17. Además, se han propuesto funciones de InsP8 y otros PP-InsPs en la homeostasis energética y la detección de nutrientes, así como la homeostasis de fosfato17,,23,,24,,25,,26.
Independientemente del sistema biológico empleado, uno de los principales desafíos metodológicos al estudiar InsPs ha sido la detección fiable y la cuantificación precisa de estas moléculas. Se han utilizado métodos basados en espectrometría de masas para detectar InsPs, incluidos los PP-InsP, a partir de extractos de celdas. Sin embargo, estos estudios no lograron diferenciar distintos isómeros26,,27. Otro enfoque para analizar InsPs emplea la extracción de InsPs de los lysates celulares usando cuentas TiO2, seguido de electroforesis de gel de poliacrilamida (PAGE) del InsPs eluido. Los InsPs pueden ser manchados por toluidine azul o DAPI24,28,29. Sin embargo, hasta ahora no es posible detectar de forma fiable InsPs inferior a InsP5 de extractos vegetales utilizando este método. Recientemente, un método que utiliza [13C]-myo-inositol para el análisis de resonancia magnética nuclear (NMR) de InsPs fue publicado como una alternativa a la cromatografía líquida fuerte de aniones de alto rendimiento (SAX-HPLC)30. Esta técnica se ha divulgado para lograr una sensibilidad similar en comparación con SAX-HPLC y para permitir la detección de 5-InsP7, así como la discriminación de diferentes isómeros No enantioméricos InsP5 de extractos celulares. Sin embargo, la implementación del método basado en RMN requiere sintetizado químicamente y comercialmente no disponible [13C]-myo-inositol. Por lo tanto, el método empleado en la mayoría de los casos es el radioetiquetado de muestras con [3H]-myo-inositol, seguido de SAX-HPLC31,32,33. Esta técnica se basa en la absorción de myo-inositolradiactivo en la planta y su conversión en diferentes InsPs por la actividad combinada de quinasas celulares dedicadas y fosfatasas.
Los InsP con etiqueta de [3H] se extraen y fraccionan con ácido mediante SAX-HPLC. Debido a su carga negativa, los InsPs interactúan fuertemente con la fase estacionaria cargada positivamente de la columna SAX-HPLC y se pueden eluerar con un gradiente tampón que contenga concentraciones de fosfato crecientes para ecompetear a Los InsP de la columna. Por lo tanto, los tiempos de elución dependen de la carga y la geometría de las especies de InsP que se van a separar. En ausencia de columnas quirales, sólo los isómeros no enantioméricos pueden separarse por este protocolo. Sin embargo, las normas radiomarcadas se pueden utilizar para asignar la naturaleza isomérica de un pico InsP específico. Múltiples esfuerzos en el pasado por parte de diversos laboratorios para generar normas etiquetadas y no etiquetadas con métodos (bio)químicos o para purificarlos de diversas células y organismos han ayudado a asignar picos a ciertas especies de InsP, y también para acotar la identidad isomérica de las especies individuales de InsP5,7,21,34,35,36,37,38,39,40,41,42,43. Asimismo, el reciente esclarecimiento de vías enzimáticas que conducen a la formación de PP-InsPs en las plantas, así como el descubrimiento de un efector bacteriano tipo III con una actividad específica de 1-phytasa, proporcionan información sobre cómo generar normas útiles para estos análisis10,,17,18,22,23.
Las fracciones resultantes se pueden medir en un contador de centelleo líquido debido a la β-decaimiento del tritio (3H). Con el aumento del tiempo de etiquetado, se alcanza un equilibrio isotópico de estado estacionario, después de lo cual los perfiles InsP obtenidos deben representar el estado InsP de la planta31. La principal ventaja de este protocolo en comparación con otras técnicas disponibles es la alta sensibilidad lograda por el uso del precursor directo para los InsPs y la medición de una señal radiactiva.
SAX-HPLC de muestras extraídas de [3H]-myo-plantas etiquetadas con inositol u otros organismos se utiliza comúnmente para la detección y cuantificación de InsPs que van desde especies insP más bajas a PP-InsPs, que representan una herramienta valiosa para comprender mejor el metabolismo, la función y los modos de acción de los InsPs. Hasta ahora, este método es también la opción más adecuada para los investigadores con especial interés en especies inferiores de InsP. Si bien los fundamentos de este procedimiento, sobre el que se basa el protocolo aquí descrito, se han descrito previamente7,21,31,34, todavía falta un protocolo detallado adaptado al análisis de los InsPs derivados de plantas y especialmente de los PP-InsPs. Publicaciones anteriores informaron de dificultades para detectar de manera fiable los especialmente InsP8, debido a uno o más de los siguientes factores: cantidades relativamente bajas de material vegetal, [3H]-myo-inositol con baja actividad específica (> 20 Ci/mmol), uso de tampones de extracción que no se basan en ácido perclórico o están menos concentrados que 1 M, diferentes buffers neutralizantes, así como gradientes subópticos o detección de [3H con un detector. En comparación con estos estudios, el protocolo presentado aquí está diseñado para la detección fiable de PP-InsPs7,21,34.
Aquí presentamos un flujo de trabajo detallado, desde la configuración del equipo hasta el cultivo y etiquetado de plantas, la extracción InsP y la propia ejecución SAX-HPLC. Aunque el método fue optimizado para la planta modelo A. thaliana,se puede modificar fácilmente para estudiar otras especies de plantas, como se muestra aquí con el primer perfil InsP reportado del modelo leguminosa Lotus japonicus. Aunque el uso de una especie vegetal diferente podría requerir cierta optimización, prevemos que serán menores, haciendo de este protocolo un buen punto de partida para la investigación posterior en insp de plantas. Con el fin de facilitar posibles optimizaciones, indicamos cada paso dentro del protocolo en el que las modificaciones son posibles, así como todos los pasos críticos que pueden ser difíciles al establecer el método por primera vez. Además, informamos de cómo se pueden utilizar los datos obtenidos por este método para la cuantificación de InsP específicos y cómo se pueden analizar y comparar diferentes muestras.
Protocolo
1. Configuración del sistema HPLC
- Configurar un sistema que consta de dos bombas HPLC independientes (bomba binaria), una para cada búfer. Ambas bombas deben ser controladas juntas a través de un ordenador con el software respectivo o teniendo una bomba maestra. Implemente un lavado de sello de pistón para ambas bombas, ya sea a través de la fuerza gravitacional o a través de una tercera bomba de baja presión. Designe una bomba para el buffer A (bomba denominada A) y una para el buffer B (llamado bomba B).
NOTA: Ambos tienen que ser capaces de generar presiones de hasta 60 bar (6 MPa) y caudales de al menos 0,5 ml/min. - Conecte ambas bombas a un mezclador dinámico.
- Conecte el mezclador a una válvula de inyección con un bucle de muestra de al menos 1 ml de capacidad.
- Conecte la válvula de inyección a la columna con un capilar a través de los accesorios finales correspondientes.
- Conecte la columna al colector de fracciones utilizando un capilar con una longitud adecuada.
NOTA: Esta descripción se basa en nuestro sistema HPLC (consulte la Tabla de Materiales),que requiere más pasos manuales que los sistemas más nuevos y sofisticados. Nuestro sistema permite un fácil acceso y modificación de todos los componentes. Las bombas cuaternarias (con el gradiente binario descrito aquí) también se pueden utilizar y darán lugar a perfiles de elución y calidad general de los análisis similares a los logrados con bombas binarias.
2. Preparación de tampones, columnas y sistema HPLC
- Preparar los buffers para la extracción de InsPs solubles: buffer de extracción (1 M HClO4) y buffer de neutralización (1 M K2CO3). Prepare ambos amortiguadores con agua desionizada ultrapura. Son estables a temperatura ambiente durante varios meses. Inmediatamente antes de la extracción, añada EDTA a ambas soluciones a una concentración final de 3 mM (por ejemplo, a partir de una solución filtrada de EDTA de 250 mM).
ADVERTENCIA: HClO4 (ácido perclórico) es fuertemente corrosivo. - Prepare los buffers para el tramo SAX-HPLC: buffer A (1 mM EDTA) y buffer B (1 mM EDTA, 1.3 M (NH4)2HPO4; pH 3.8 con H3PO4). Preparar ambos utilizando agua desionizada ultrapura seguida de filtración al vacío con filtros de membrana del tamaño de un poro de 0,2 m. Estos son estables a temperatura ambiente durante varios meses.
NOTA: EDTA debe incluirse en todos los búferes para evitar interacciones de cationes con InsPs, lo que podría dar lugar a una carga InsP alterada o incluso complejos de sal Insolubles InsP. - Programe el gradiente de la siguiente manera: 0-u20122 min, 0% buffer B; 2-u20127 min, hasta 10% buffer B; 7-u201268 min, hasta 84% buffer B; 68-u201282 min, hasta 100% buffer B; 82-u2012100 min, 100% buffer B, 100-u2012101 min, hasta 0% buffer B; 101-u2012125 min, 0% buffer B. El caudal óptimo para este gradiente es de 0,5 ml/min.
- Durante la carrera, recoger fracciones cada minuto, a partir del minuto 1 al minuto 96. Los 30 minutos restantes del gradiente sirven para lavar la columna y el sistema, y no tienen que ser recogidos para el conteo de centelleo.
- Si es posible, ajuste la presión máxima alcanzable antes del apagado de emergencia de las bombas HPLC a 80 bar (8 MPa). Esto evita daños críticos a la resina de la columna.
- Cuando utilice una nueva columna SAX HPLC, lávela a fondo (>50 ml) con agua desionizada ultrapura filtrada antes del primer uso.
NOTA: Esto asegurará la eliminación del metanol contenido, evitando así la precipitación de sal en pasos posteriores. Si es posible, utilice una bomba HPLC separada. Si esto no está disponible, asegúrese de que el HPLC se haya lavado con agua antes de lavar la columna. El caudal no debe exceder 2 ml/min. Después del lavado, la columna está lista para el análisis y, cuando se maneja correctamente, se puede utilizar para 20 u201240 ejecuciones. Después de eso, la resolución disminuirá sucesivamente. El lavado prolongado con el búfer A (>1 h) y la realización del paso 2.6 pueden ayudar a aumentar la vida útil de la columna. Si la disminución de la resolución persiste, es necesario intercambiar la columna. El gradiente se puede ajustar para aumentar la separación entre especies específicas de polifosfato de inositol o para disminuir el tiempo de ejecución general. El uso de diferentes sistemas HPLC (con diferente volumen vacío o diferente volumen de los capilares) afectará fuertemente los tiempos de retención. Además, los cambios de columna tienen efectos menores en los tiempos de retención. - Realice una "ejecución de burla". En lugar de una muestra extraída, inyecte agua desionizada ultrapura filtrada en el sistema HPLC y ejecute el gradiente estándar. Las fracciones no tienen que ser recogidas.
NOTA: El paso 2.6 es opcional. Sin embargo, se debe realizar si se aplica una de las siguientes situaciones: se instala una nueva columna; El sistema HPLC se ha utilizado para un método diferente de antemano; El sistema HPLC no se ha utilizado durante más de 3 días; Se ha detectado un problema con la ejecución anterior.
3. Cultivo y etiquetado de plantas con [3H]-myo-inositol
NOTA: Los siguientes pasos deben realizarse con componentes estériles y en condiciones estériles,mientras se usan guantes para proteger las manos de la contaminación con la radioetiqueta. Los medios vegetales, especialmente cuando contienen sacarosa, son propensos a la contaminación microbiana.
- Esterilice A. thaliana semillas con 1 ml de 1,2% de hipoclorito sódico durante 3 min seguido de 1 ml de etanol al 70% durante 3 min. A continuación, añadir 1 ml de etanol 100%, pipetear las semillas con el etanol en un papel de filtro circular y dejar que se sequen al aire bajo un flujo laminar en un banco limpio.
- Cuando utilice semillas de L. japonicus, colóquelas en un mortero y frote las semillas con papel de lija antes de la esterilización para asegurar una tasa de germinación suficiente.
- Siembre las semillas de Arabidopsis en 1-2 hileras en platos sólidos de Petri llenos de medios de crecimiento sólido que consisten en la solución de sal Murashige y Skoog (MS) de media fuerza, 1% sacarosa, 0.7% goma de gellan en agua desionizada ajustada a pH 5.7 con KOH y les permite estratificar durante al menos 1 día a 4 oC en la oscuridad.
- Para las semillas de Lotus, sembrarlas en 1 hilera sobre platos sólidos de Petri llenos de medios de crecimiento sólidos que consisten en un 0,8% de agar bacteriológico en agua desionizada y permitir que se estratiquen durante al menos 3 días a 4oC en la oscuridad.
- Colocar las placas verticalmente en una incubadora de crecimiento o cámara climática y permitir que crezcan durante 10-12 días en condiciones de día corto (8 h de luz a 22 oC, 16 h de oscuridad a 20 oC).
- Transfiera de 10 a 20 plántulas a un pozo de una placa de cultivo celular de fondo plano de 12 pozos llena de 2 ml de solución de sal MS de media resistencia complementada con 1% de sacarosa y ajustada a pH 5.7.
- Añadir 45 éCi de [3H]-myo-inositol (30–80 Ci/mmol, disuelto en 90% etanol) y mezclar por remolino suave. Cubra la placa con la tapa correspondiente y ciérrela con cinta quirúrgica microporosa (por ejemplo, cinta de microporos o leucopore), colocándola de nuevo en la incubadora de crecimiento.
ADVERTENCIA: [3H] es un emisor beta de baja energía que puede ser un peligro de radiación perjudicial cuando se inhala, ingiere o absorbe a través de la piel desnuda. Use siempre guantes cuando manipule material o equipo radiactivo que tenga contacto directo o indirecto con el material radiactivo. Siga también las normas locales para el manejo seguro de los radioquímicos (por ejemplo, usar ropa protectora adicional, el uso de un dosímetro y estudios de superficies para detectar contaminaciones de forma regular). - Después de 5 días de etiquetado, retire las plántulas de los medios y lávelas brevemente con agua desionizada. Secarlos con toallas de papel y transferirlos a un tubo de microcentrífuga de 1,5 ml. No sobrellene el tubo y no coloque más de 100 mg de FW/tubo, que corresponde a aproximadamente 10 u201220 plántulas de 17 días de edad.
NOTA: Un exceso de material vegetal diluirá el ácido durante el proceso de extracción y disminuirá fuertemente la eficiencia de extracción.- Congele el tubo en nitrógeno líquido y guárdelo a -80 oC hasta la extracción.
NOTA: Las muestras se pueden conservar a -80 oC durante varias semanas sin comprometer la calidad de la muestra. Las condiciones de crecimiento (medios, luz, temperatura, tiempo) pueden ser modificadas de acuerdo a las necesidades de un experimento específico o especies vegetales. Sin embargo, se debe tener cuidado al diluir el [3H]-myo-inositol, con el fin de garantizar funcionamientos cuantificables SAX-HPLC de buena calidad. Por lo tanto, se recomienda comenzar con el [3H]-concentraciones de myo-inositol indicado aquí y reducirlo paso a paso si se desea. Durante el tiempo de etiquetado, las plantas pueden someterse a diferentes tratamientos (por ejemplo, tensiones ambientales o agentes químicos) para evaluar el impacto de esas condiciones en los InsP globales. Para alcanzar el etiquetado en estado estacionario, recomendamos etiquetar las plantas durante al menos 5 días.
- Congele el tubo en nitrógeno líquido y guárdelo a -80 oC hasta la extracción.
4. Extracción de InsP soluble
NOTA: Mantenga las muestras y los reactivos sobre hielo durante todo el proceso de extracción. Use siempre guantes y gafas protectoras debido al alto riesgo de contacto con material radiactivo, especialmente durante la molienda. Todo lo que entra en contacto con muestras se considera como residuo radiactivo y debe eliminarse de acuerdo con las normas locales para la eliminación segura de material radiactivo.
- Prepare las soluciones de trabajo para el búfer de extracción y neutralización como en el paso 2.1. Cada muestra requerirá 600 l de tampón de extracción y 400 l de búfer de neutralización. Almacene los tampones en hielo.
- Tome las muestras del congelador de -80 oC y manténgalas en nitrógeno líquido hasta su posterior procesamiento. Moler las muestras con un pestle tubo de microcentrífuga hasta que comiencen a descongelar y añadir 500 l de tampón de extracción de hielo frío. Continuar moliendo hasta que la muestra esté completamente homogeneizada y la solución tenga un color verde profundo (si las hojas están presentes en la muestra).
- Centrifugar las muestras durante 10 min a 4oC a ≥ 18000 x g. Transfiera el sobrenadante a un tubo fresco de 1,5 ml. Tenga en cuenta que los tubos utilizados para la extracción se consideran residuos radiactivos sólidos y deben eliminarse en consecuencia.
-
Agregue con cuidado 300 l de tampón de neutralización al extracto. La precipitación de proteínas y el burbujeo comenzarán inmediatamente. Mezclar remolinos con una punta de pipeta después de un minuto y esperar unos segundos antes de pipetear una pequeña cantidad (5 l) en papel de pH (idealmente rango de pH 6–9). El pH debe estar entre pH 7 y 8 al final.
- Si es necesario, añada pequeñas cantidades (normalmente de 10 a 20 oL) de búfer de neutralización o tampón de extracción hasta que se alcance el pH deseado. Deje reposar las muestras sobre hielo durante al menos 1 h con una tapa abierta.
- Centrifugar las muestras durante 10 min a 4oC a ≥ 18.000 x g. Transfiera el sobrenadante a un tubo fresco de 1,5 ml.
NOTA: Las muestras pueden utilizarse directamente en una carrera SAX-HPLC o mantenerse en hielo (si se usan más tarde el mismo día) o congelarse en nitrógeno líquido y almacenarse a -80 oC durante 2 o 20124 semanas. Para garantizar una alta reproducibilidad y comparabilidad, se recomienda congelar siempre las muestras en nitrógeno líquido durante 5 min, incluso si se utilizarán directamente después. El almacenamiento a más largo plazo de las muestras extraídas a -80 oC es posible siempre y cuando las muestras solo se descongelen una vez. Si se utilizan muestras congeladas para el análisis, asegúrese de que no haya partículas visibles después de la descongelación. De lo contrario, centrifugar de nuevo durante 10 minutos a 4 oC a ≥ 18.000 x g y transfiera el sobrenadante a un tubo fresco de 1,5 ml.
5. Realizar la ejecución de HPLC
- Equipar el colector de fracción con 96 viales de centelleo pequeño (capacidad de 6 ml) y llenar cada vial con 2 ml de un cóctel de centelleo adecuado (por ejemplo, cóctel de centelleo líquido Ultima-Flo AP) compatible con tampones con pH bajo y alta concentración de fosfato de amonio (ver Tabla de materiales).
NOTA: El número de viales y el tamaño de los viales dependen del colector de fracciones y del contador de centelleo utilizado. Es importante al menos recoger las primeras 90 fracciones,si se utiliza el gradiente descrito aquí, para obtener un perfil completo de polifosfato de inositol. Asegúrese también de etiquetar correctamente cada vial y su tapa respectiva, para evitar la acumulación de fracciones o muestras. - Encienda el sistema/bombas HPLC y prepárelo para funcionar. Active el lavado del sello del pistón y manténgalo activado durante todo el tramo. Cargue la muestra inyectando manualmente el sobrenadante completo del paso 4.5 (aproximadamente 750 l) utilizando una jeringa adecuada (ver Tabla de materiales). Si es posible la inyección automática, transfiera la muestra al vial de muestra correspondiente. Gire la válvula de "carga" a "inyectar" e inicie el gradiente y el colector de fracciones.
NOTA: Dependiendo del sistema HPLC utilizado, el procedimiento de inicio puede diferir, especialmente cuando se comparan sistemas más antiguos (como se describe aquí) con un modelo más reciente totalmente controlado por software. Es muy importante asegurarse de que el gradiente, la inyección de muestra y la colección de fracciones comiencen simultáneamente. - Mientras la ejecución de HPLC está en curso, compruebe la presión regularmente. La presión inicial debe ser de alrededor de 18–24 bar (1.8–2.4 MPa) y debe elevarse lentamente a 50–60 bar (5-6 MPa) una vez que se alcanza el 100% de buffer B.
ADVERTENCIA: La disminución de la presión puede indicar una fuga en el sistema, mientras que el aumento de la presión indica un bloqueo. Las fluctuaciones de presión (≥ 3 bar en pocos segundos) pueden indicar la presencia de aire en el sistema. Tenga en cuenta que todo lo que sale de la columna, así como cada fuga que se produce en el inyector o después es radiactivo.
NOTA: La presión también depende del sistema HPLC y puede ser menor o mayor de lo que se indica aquí. Aumentará lentamente después de aproximadamente 15-20 carreras. Sin embargo, esto no influye necesariamente en la calidad de las corridas obtenidas. - Después de la carrera, cierre los viales firmemente y mezcle las fracciones con el cóctel de centelleo agitando vigorosamente. Proceda directamente con la medición o mantenga los viales en posición vertical, idealmente en la oscuridad.
NOTA: Las fracciones mezcladas con el cóctel de centelleo son estables durante semanas y se pueden medir más tarde. Dado que la vida media del tritio es de 12,32 años, la pérdida de señal es insignificante. - Una vez finalizada la ejecución de la última muestra del día, detenga ambas bombas HPLC.
- (Opcional) Para aumentar la longevidad del sistema, especialmente cuando no se utiliza regularmente, la bomba de lavado B y los capilares colocando el capilar del tampón B en una botella con tampón A y deje que la bomba funcione durante 10-15 min. Antes del siguiente uso, recuerde reemplazar el capilar en el tampón B y desacoplar la bomba B del mezclador para vaciarla con el búfer B. Una vez que la bomba y los capilares se llenen de nuevo con el tampón B, vuelva a conectarlo con el mezclador y el sistema esté listo para usar.
6. Medición de las fracciones
- Inserte los viales en bastidores de centelleo y mida cada vial durante 5 minutos en un contador de centelleo líquido.
- Idealmente, utilice bastidores que se ajusten directamente a viales pequeños y evite colgar en los viales en viales más grandes (por ejemplo, 20 ml) para reducir los errores de conteo. La configuración de software utilizada en este protocolo se muestra en la figura complementaria 1.
NOTA: Realice regularmente un protocolo SNC (autonorcuación y calibración) utilizando estándares sin forzar [3H]. Los tiempos de conteo más cortos (1-5 min) son posibles para reducir el tiempo de espera. Sin embargo, para garantizar una reproducibilidad y precisión de recuento alto, se recomiendan 5 minutos.
7. Análisis de datos
- Exporte las mediciones desde el contador de centelleo como un archivo de hoja de cálculo o un formato de archivo compatible/convertible. Evalúe los datos con un ordenador equipado con Excel o software similar, y un software de análisis adecuado como Origin.
- Prepare un gráfico de líneas 2D donde los recuentos medidos por minutos (cpm) se tracen con el tiempo de retención (consulte la Figura 1, Figura 2).
- Para comparar muestras entre sí, normalice los datos sumando la cpm de cada fracción eluida del minuto 25 a 96 para cada muestra individual.
NOTA: El minuto 25 se utiliza como corte para excluir el análisis no incorporado [3H]-myo-inositol, InsP1 e InsP2, ya que tienden a fluctuar fuertemente y no pueden estar bien separados (al menos con el gradiente propuesto en este protocolo) y por lo tanto cambian fuertemente el factor de normalización debido a su alta actividad. - Normalice todos los datos a la muestra con el cpm total más bajo (en fracciones 25-u201296) dividiendo el cpm total de la muestra con el cpm más bajo (en fracciones 25–96) por el cpm total (en fracciones 25–96) de las otras muestras. El factor resultante se puede utilizar para normalizar la cpm de cada fracción multiplicando la cpm de cada fracción con el factor.
NOTA: Al final, la suma de los valores cpm desde el minuto 25 hasta el final debe ser igual para todas las muestras en comparación entre sí. Solo las ejecuciones normalizadas deben presentarse en el mismo gráfico/figura (cuando se presentan como perfiles reales). La Figura suplementaria 2 muestra un ejemplo de cómo se realizan estos pasos de cálculo (utilizando sólo las fracciones 25–35 de dos muestras para simplificar). Sin embargo, en algunos casos no es necesario normalizar los datos. Por ejemplo, cuando los picos se cuantifican según el paso 7.4 y se presentan como porcentajes del Total de InsP (como se muestra en la Figura 3D). Como se ha indicado anteriormente, al presentar múltiples análisis uno al lado del otro como perfiles, o cuando la actividad medida real se utiliza para conclusiones (por ejemplo, el tratamiento a) aumenta InsP7 en x% en comparación con el control, refiriéndose a los valores cpm de InsP7 de ambas muestras y no a su porcentaje de insP total) se necesita la normalización. Para analizar el efecto del genotipo o las diferencias de tratamiento en la eficiencia del etiquetado, es importante no normalizar,ya que esto invalidaría estas diferencias. Sin embargo, la cuantificación absoluta con este método es difícil porque la eficiencia de extracción con este protocolo puede ser variable por varias razones y a veces incluso se observan cuando se analiza la réplica del mismo genotipo y tratamiento. Tenga en cuenta que, dependiendo del sistema HPLC, columna y gradiente utilizados para los análisis, es posible que sea necesario cambiar el corte. - Para realizar cuantificaciones relativas de ciertos picos de polifosfato de inositol y posteriormente crear gráficos de barras que contengan datos de replicaciones para análisis estadísticos, continúe el análisis con un software especializado que pueda calcular áreas pico de cromatogramas (por ejemplo, Origen). Vea la figura complementaria 3.
NOTA: La mayoría de los sistemas HPLC controlados por software se suministran con un software respectivo capaz de esta tarea. Los picos se determinan como las fracciones con valores cpm por encima del fondo (que varía en cierto grado entre las ejecuciones) y los tiempos de retención que son similares a los datos publicados anteriormente. El tiempo de retención de un pico específico se determina en el software de hoja de cálculo (por ejemplo, Excel) y se utiliza para asignar picos para el cálculo de integrales definidas (por ejemplo, en Origen). La Figura Complementaria 3 ilustra este proceso de determinación de picos, resta de fondo e integración de picos.
Resultados
Los resultados que se muestran aquí tienen como objetivo ilustrar los posibles resultados obtenidos de acuerdo con las variaciones a nivel técnico y biológico. El primero se ejemplifica mediante análisis utilizando columnas nuevas frente a envejecidas (Figura 1) y muestras frescas frente a almacenadas (Figura 3), y la segunda mediante la evaluación de extractos de dos sistemas vegetales diferentes, A. thaliana (Figura 1, Figura 3) y L. japonicus (Figura 2).
Una ejecución óptima SAX-HPLC se representa en la Figura 1A-u2012C, que muestra un espectro completo de polifosfato de inositol obtenido de extractos de A. thaliana después del conteo de centelleo. Tenga en cuenta que los picos están muy bien separados y se pueden asignar a diferentes isómeros (o pares de enantiómeros) basados en las movilidades cromatográficas descritas anteriormente5,7.
La Figura 2 muestra el resultado representativo de un análisis SAX-HPLC de las plántulas de L. japonicus que fueron cultivadas y etiquetadas en las mismas condiciones que las plántulas de Arabidopsis. Aunque presumiblemente se pueden observar todas las especies y picos de InsP que se conocen a partir de Arabidopsis, existen diferencias sustanciales con respecto a la cantidad relativa (por ejemplo, las relaciones entre los isómeros) de isómeros InsP específicos, al comparar los perfiles de ambas especies. Por ejemplo, los extractos de Lotus mostraron un aumento de InsP3c, InsP4b, InsP5b y reducido InsP3a, InsP4a, InsP5a e InsP5c en comparación con Arabidopsis que deja espacio para investigaciones posteriores. La Figura 2D ilustra las diferentes relaciones entre los isómeros InsP entre Arabidopsis y Lotus.
La figura 3 muestra dos perfiles InsP de una muestra que se dividió después de la extracción. La primera mitad se analizó inmediatamente y la segunda mitad un día después, después del almacenamiento a -80 oC. Tenga en cuenta que sólo se observan diferencias menores entre las diferentes muestras (es decir, líneas negras y rojas en la Figura 3Ade u2012Cy la Figura 3D). Esto ilustra que un ciclo de congelación-descongelación no daña la muestra y que el propio método genera resultados reproducibles.
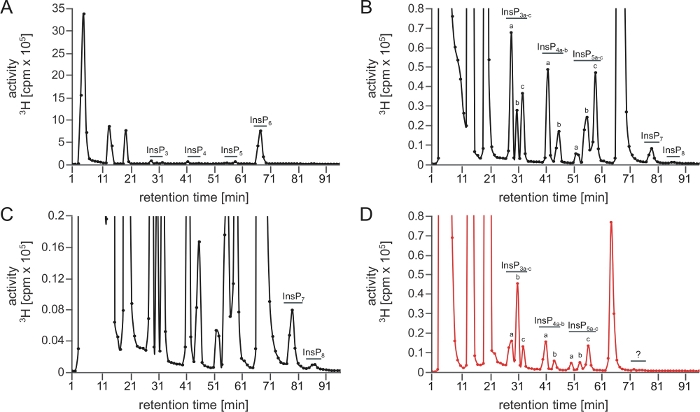
Figura 1: Perfil InsP típico de un análisis SAX-HPLC exitoso y de un análisis SAX-HPLC fallido realizado con este protocolo. (A.u2012C) PERFIL SAX-HPLC de 17 días de edad tipo salvaje (Col-0) Plántulas Arabidopsis radiomarcadas con [3H]-myo-inositol. La extracción Global InsP y la ejecución SAX-HPLC se realizaron el mismo día. (A) Espectros completos; (B, C) Zoom-ins del perfil que se muestra en A. Todos los picos visibles se resaltan y se asignan a las especies InsP correspondientes. Sobre la base de las movilizaciones cromatográficas publicadas5,7, InsP4a probablemente representa Ins(1,4,5,6)P4 o Ins(3,4,5,6)P4, InsP5a representa InsP5 [2-OH], InsP5b representa InsP5 [4-OH] o su forma enantiomérica InsP5 [6-OH], e InsP5c representa InsP5 [1-OH] o su forma enantiomérica InsP5 [3-OH]. Las naturalezas isoméricas de InsP3a-c, InsP4b, InsP7e InsP8 aún son desconocidas. Panel (D) muestra un perfil SAX-HPLC de plantas cultivadas de forma idéntica pero utilizando una columna envejecida (>40 corridas). Una clara reducción de InsP6 en comparación con otras especies de InsP y la ausencia de PP-InsPs es visible. Haga clic aquí para ver una versión más grande de esta figura.
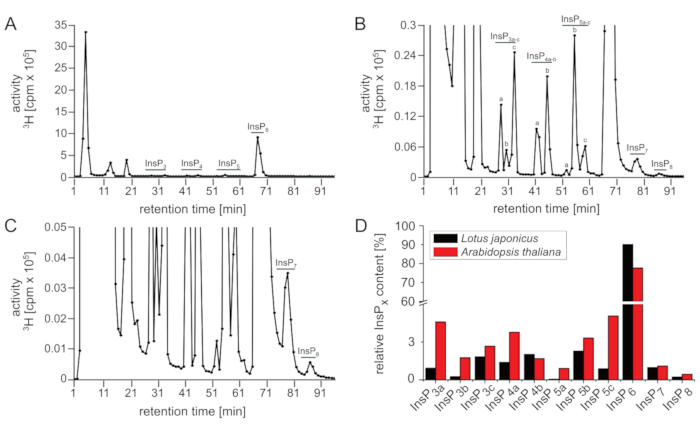
Figura 2: Perfil insp representativo de las plantas de L. japonicus. Perfil SAX-HPLC (A.u2012C) de tipo salvaje de 17 días de edad (Gifu) L. japonicus plántulas radiomarcadas con [3H]-myo-inositol. (A) Espectros completos; (B, C) Zoom-ins del perfil que se muestra en A. Todos los picos visibles se resaltan y se asignan a las especies InsP correspondientes. Basado en las movilizaciones cromatográficas publicadas5,7, InsP4a probablemente representa Ins(1,4,5,6)P4 o Ins(3,4,5,6)P4, InsP5b probablemente representa InsP5 [4-OH] o su forma enantiomérica InsP5 [6-OH], e InsP5c probablemente representa InsP5 [1-OH] o su forma enantiomérica InsP5 [3-OH]. Se desconocen las naturalezas isoméricas de InsP3a-c, InsP4b, InsP7e InsP8. (D) Comparación entre las especies individuales de InsP (en % de la actividad total a partir de la elución 25-u201296) de A. thaliana (datos de la Figura 1A-u2012C)y L. japonicus (datos de la Figura 2A–C). Haga clic aquí para ver una versión más grande de esta figura.
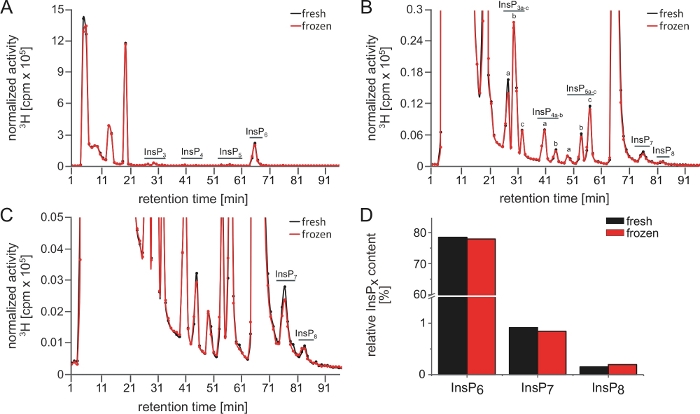
Figura 3: Perfiles insP de una muestra dividida que ilustra la reproducibilidad de los análisis SAX-HPLC. (A.u2012C) Perfiles SAX-HPLC de tipo salvaje de 17 días de edad (Col-0) Plántulas Arabidopsis radiomarcadas con [3H]-myo-inositol. Antes de la ejecución, la muestra se dividió y la mitad se ejecutó inmediatamente y la otra mitad un día después del almacenamiento a -80 oC. (A) Espectros completos; (B, C) Zoom-ins del perfil que se muestra en A. Todos los picos visibles se resaltan y se asignan a las especies InsP correspondientes. Sobre la base de las movilizaciones cromatográficas publicadas5,7, InsP4a probablemente representa Ins(1,4,5,6)P4 o Ins(3,4,5,6)P4, InsP5a representa InsP5 [2-OH], InsP5a representa InsP5 [2-OH], InsP5b representa InsP5 [4-OH] o su forma enantiomérica InsP5 [6-OH], e InsP5c representa InsP5 [1-OH] o su forma enantiomérica InsP5 [3-OH]. Las naturalezas isoméricas de InsP3a-c, InsP4b, InsP7e InsP8 aún son desconocidas. El Panel D muestra la cuantificación del InsP6 y los PP-InsPs InsP7 e InsP8 de ambas ejecuciones. Los valores representan la cantidad (en %) de las respectivas especies de InsP en relación con todas las InsP (actividad total de la elución 25–96). Haga clic aquí para ver una versión más grande de esta figura.
Figura suplementaria 1: Configuración del software para el recuento de centelleo líquido utilizando un contador de centelleo ligero. Se representan las capturas de pantalla que muestran la versión del software, así como los ajustes utilizados para el recuento de centelleo de muestras de[3H] realizadas con este protocolo. Haga clic aquí para descargar esta figura.
Figura complementaria 2: Ejemplo representativo de la normalización de datos. Una captura de pantalla de una hoja de trabajo muestra todos los pasos y fórmulas utilizados para normalizar las ejecuciones SAX-HPLC entre sí. Para simplificar, solo se muestran las fracciones 25–35 de las muestras. Haga clic aquí para descargar esta figura.
Figura suplementaria 3: Determinación de picos, resta de fondo e integración mediante software de análisis. (A) Los datos del análisis SAX-HPLC se cargan en el software (minutos 28–96) y se selecciona la herramienta de analizador de picos. (Bá52012E) La línea base se define manualmente estableciendo puntos entre picos individuales y se resta el fondo. (F) Los picos se determinan manualmente en función de la apariencia y las movilidades cromatográficas publicadas5,7. LosGrangos de picos se definen manualmente mediante valores cpm. (H) Los picos se integran y calculan como % de todos los picos. Haga clic aquí para descargar esta figura.
Discusión
Aquí presentamos un método versátil y sensible para cuantificar los InsPs incluyendo PP-InsPs en extractos vegetales y proporcionamos consejos prácticos sobre cómo establecer este método. Aunque el protocolo es generalmente robusto, pueden producirse corridas y análisis subóptimos. En la mayoría de los casos, esas corridas pueden identificarse mediante una fuerte reducción o incluso una pérdida completa de InsPs altamente fosforilados, especialmente las especies PP-InsP InsP7 e InsP8. Las posibles razones pueden ser la contaminación microbiana del material vegetal y la desactivación insuficiente de las hidrolasas PP-InsP de la planta endógena durante la extracción debido a la molienda y descongelación insuficientes de material vegetal que no entrará en contacto inmediato con el tampón de extracción. Otras razones incluyen un ajuste inexacto del pH por adición insuficiente o excesiva de tampón de neutralización, o simplemente material de muestra insuficiente. Esto último puede dificultar la detección de PP-InsPs, ya que a menudo están presentes en cantidades muy bajas en las células. Un exceso de material de la muestra o un secado ineficiente durante el paso 3.5 puede causar dilución del ácido perclorico, lo que también conduce a una desactivación insuficiente de la enzima y a una pérdida específica de InsP6 y PP-InsPs. La cantidad de material vegetal, así como la etiqueta radioeléctrico utilizada en este protocolo se optimizaron en función de los costos y el rendimiento, y por lo tanto está cerca de la cantidad más baja que todavía es suficiente para proporcionar resultados óptimos. Además, la resina de columna perderá gradualmente su capacidad de resolución. El primer signo de este proceso es (por razones no del todo claras para los autores) una pérdida específica de especies de InsP más fosforiladas como las PP-insPs en el espectro HPLC. Con el envejecimiento adicional, incluso InsP6 no se resolverá correctamente por la columna (Figura 1D). Por lo tanto, el uso de una columna adecuada, así como el manejo meticuloso de la muestra y el mantenimiento adecuado de los componentes HPLC es crucial para garantizar resultados precisos.
Al comparar muestras y corridas, especialmente cuando se generan con diferentes equipos (por ejemplo, sistemas y columnas HPLC) o en días diferentes, es crucial normalizar las muestras entre sí (como se describe en el paso 7.3) y analizarlas de la misma manera. Sólo a través de la normalización es posible y preciso mostrar varias muestras en el mismo gráfico (Figura 3). Para la cuantificación de InsPs individuales en relación con el total de InsPs, o a otra especie específica de InsP, no es necesario normalizar, siempre y cuando solo se muestren valores relativos y no valores absolutos. Idealmente, se muestran tanto los perfiles InsP como las cuantificaciones. Sin embargo, en algunos casos no es posible mostrar adecuadamente dos o más corridas en el mismo gráfico. Diferentes tiempos de retención o diferentes niveles de actividad en segundo plano pueden dificultar la comparación de perfiles SAX-HPLC no fortificados solos. Lo mismo ocurre cuando hay que comparar muchas muestras. En tales casos, es necesaria una evaluación adicional utilizando un software adicional (por ejemplo, Origen) para la cuantificación de picos individuales.
Los autores son conscientes de que el protocolo descrito aquí puede optimizarse y debe adaptarse a cada pregunta de investigación individual. Aunque está optimizado para extractos de Arabidopsis 7,,17 en este protocolo, este método es versátil y puede ayudar a determinar los perfiles InsP de otras especies de plantas también. Aquí ejemplificamos esta posibilidad presentando por primera vez un perfil InsP para L. japonicus,que no requirió modificaciones de las condiciones de etiquetado, extracción InsP o ejecución SAX-HPLC (Figura 2). En particular, aunque en general se observan diferencias entre los perfiles L. japonicus y Arabidopsis InsP. Por ejemplo, en L. japonicus InsP5 [4-OH] o su forma enantiomérica InsP5 [6-OH] son más abundantes que InsP5 [1-OH] o su forma enantiomérica InsP5 [3-OH] en comparación con Arabidopsis, donde InsP5 [1-OH] o su forma enantiomérica InsP5 [3-OH] son las especies dominantes de InsP5. Del mismo modo, prevemos que las alteraciones en la composición de los medios,[3H]- concentraciónde myo-inositol, edad de las plantas, condiciones ambientales (por ejemplo, luz y temperatura), adición de compuestos químicos o análisis de interacciones entre plantas y microbianos entre otros factores, podrían necesitar ser probadas y adaptadas.
Un inconveniente importante de este método que debe tenerse en cuenta es que el etiquetado se realiza en un cultivo líquido (estéril), que no representa un entorno fisiológico para la mayoría de las plantas terrestres. Además, debido a los altos costos de [3H]-myo-inositol, el volumen de la solución de etiquetado y el tamaño del recipiente de cultivo es generalmente limitado, lo que también restringe el tamaño de las plantas que se pueden utilizar. El cultivo en cultivo líquido se puede evitar infiltrando directamente, por ejemplo, hojas de plantas cultivadas en el suelo con [3H]-myo-inositol y posteriormente siguiendo el protocolo descrito aquí, como se informó previamente10.
Hay varios inconvenientes de este protocolo en comparación con métodos alternativos, como tiO2 pull-down seguido de PAGE o técnicas basadas en espectrometría de masas. Debido al[3H]-etiquetado myo-inositol, sólo las especies de InsP que se originan directamente de mio-inositol radiomarcado se detectarán al final. El método descrito aquí es ciego a otros isómeros Ins como scyllo-inositol y otros isómeros algunos de los cuales han sido identificados en ciertas plantas44. Además, myo-InsPs derivados de otras vías serán excluidos, incluyendo los sintetizados por síntesis de novo de myo-inositol y myo-inositol-3-fosfato a través de la isomerización de glucosa-6-fosfato, catalizado por myo-inositol-3-fosfato sintasa (MIPS) proteínas45. Aunque [32P] o [33P]-ortofoto-fosfato se puede utilizar como etiquetas alternativas, su uso plantea una desventaja importante, ya que cada molécula que contiene fosfato, incluyendo los nucleótidos abundantes y sus derivados, será etiquetado. Esas moléculas también se pueden extraer con este protocolo y unirse a la columna SAX, lo que dará lugar a un alto nivel de actividad en segundo plano que interferirá con la identificación de los picos individuales de InsP5. Además, la cuantificación de [32P]- o [33P] -etiquetada InsPs y PP-InsPs puede estar fuertemente influenciada por la rotación de la mitad del fosfato y pirofosfato y podría no informar de una lectura masiva para las especies de inositol.
Por otro lado, [3H]-myo-inositol etiqueta específicamente moléculas quecontienen myo -inositol. InsPs, lípidos que contienen inositol, como fosfoinositides, y galactinol son en este caso etiquetados. Sin embargo, sólo insPs serán analizados con este protocolo, ya que los lípidos son insolubles en el búfer de extracción y galactinol no se une a la columna SAX.
Hasta ahora, las diferencias con un perfil de InsP de la planta generado por [3H]-etiquetado myo-inositol en comparación con uno determinado por TiO2 pulldown/PAGE sigue siendo desconocido, ya que tales comparaciones no se han realizado en plantas. Un estudio reciente realizado en células animales abordó esta pregunta46. En ese trabajo, se identificó un conjunto de InsP6 que es invisible por [3H]-myo-inositol etiquetado, que por lo tanto debe derivarse directamente de glucosa-6-fosfato, se identificó comparando perfiles SAX-HPLC con geles PAGE de líneas celulares de mamíferos. 24 h de inanición de fosfato resultaron en un aumento del 150% de InsP6 al cuantificar geles PAGE de InsPs purificados usando TiO2 pulldown. Los análisis SAX-HPLC de [3H]- células etiquetadaspor io-inositol-etiquetadas que fueron tratadas de forma idéntica sólo mostraron un aumento en un 15% de [3H]-InsP6. Como se mencionó anteriormente, insPs inferiores a InsP5 son indetectables con el análisis PAGE en la mayoría de los casos. El radioetiquetado seguido de SAX-HPLC parece ser el método de elección, siempre y cuando los protocolos espectrométricos de masa no estén optimizados para detectar este grupo de moléculas altamente cargadas negativamente.
Otro reto restante es distinguir los enantiómeros en los análisis SAX-HPLC (o en cualquier otro método para el análisis InsP)10,,17. Este desafío puede abordarse mediante la adición de selectores quirales, es decir, compuestos enantiopur como la amida de L-arginina que interactúan con las moléculas enantioméricas respectivas para formar complejos diaesteroméricos que se pueden separar10. Hasta donde sabemos, este enfoque sólo se ha implementado para discriminar los isómeros InsP5 enantioméricos InsP5 [1-OH] e InsP5 [3-OH] mediante los análisis de RMN10. La discriminación de otros pares enantioméricos o la discriminación exitosa de enantiómeros por análisis CHIral SAX-HPLC o métodos quirales basados en PAGE aún no se han divulgado y deben desarrollarse más. Teniendo en cuenta la síntesis conservada y la regulación conservada de los PP-InsP por disponibilidad de fósforo, concebimos que especialmente los métodos no radiactivos como los métodos basados en PAGE o MS, junto con los análisis de nutrientes, ayudarán a los esfuerzos de verdad del suelo para calibrar los datos de teledetección diseñados para diagnosticar deficiencias de nutrientes en los cultivos17,,18,,24,,25. Sin embargo, el método presentado aquí todavía puede ser considerado el estándar de oro para los análisis de InsP y será instrumental para descubrir nuevas funciones de estos mensajeros intrigantes en las plantas.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por la Deutsche Forschungsgemeinschaft (DFG, Fundación Alemana de Investigación) bajo la Estrategia de Excelencia de Alemania - EXC-2070 – 390732324 (PhenoRob), el Grupo de Formación en Investigación GRK2064 y las becas de investigación individuales SCHA1274/4-1 y SCHA1274/5-1 a G.S.. Agradecemos también a Li Schláter y Brigitte Ueberbach por su asistencia técnica.
Materiales
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf AG | model: 5430 R | |
| Diammonium hydrogen phosphate, ≥97 % | Carl Roth GmbH + Co. KG | 0268.1 | |
| Ethylenediamine tetraacetic acid disodium salt dihydrate, ≥99 %, p.a., ACS | Carl Roth GmbH + Co. KG | 8043.2 | |
| Falcon 12-well clear flat bottom TC-treated multiwell cell culture plate, with lid, individually wrapped, sterile | Corning Inc. | 353043 | |
| Fraction collector | LAMBDA Instruments GmbH | model: OMNICOLL single channel collector | |
| Growth incubator | poly klima GmbH | model: PK 520-LED | |
| HPLC pumps | Kontron Instruments | model: 420 | |
| HPLC syringe for Rheodyne valves, 1 mL | Hamilton Company | 81365 | |
| Injector for HPLC | Supelco | model: Rheodyne 9725 | |
| Inositol, myo-[1,2-3H(N)] | American Radiolabeled Chemicals Inc. | ART 0261 | |
| Liquid nitrogen | University, Chemistry Department | ||
| Liquid scintillation counter | PerkinElmer Inc | model: TRI-CARB 2900TR | |
| Micro pestle | Carl Roth GmbH + Co. KG | CXH7.1 | |
| Mixed cellulose Eester filter, ME range (ME 24), plain, 0.2 µm pore size, 47 mm circle | GE Healthcare Life Sciences | 10401770 | |
| Mixer for HPLC | Kontron Instruments | model: M 800 | |
| Murashige & Skoog medium, salt mixture | Duchefa Biochemie | M0221 | |
| OriginPro software | OriginLab Corp. | ||
| Orthophosphoric acid, ≥85 %, p.a., ISO | Carl Roth GmbH + Co. KG | 6366.1 | |
| Partisphere 5 µm SAX cartridge column, 125 x 4.6 mm | Hichrom Limited | 4621-0505 | |
| Perchloric acid, 70 %, 99.999 % trace metals basis | Sigma-Aldrich | 311421 | |
| Petri dish, square, PS, clear, 120/120/17 mm, sterile | Greiner Bio One International GmbH | 688161 | |
| pH-indicator paper pH 5.5 - 9.0, Neutralit | Merck KGaA | 109564 | |
| Phytagel | Sigma-Aldrich | P8169 | |
| Potassium carbonate | Carl Roth GmbH + Co. KG | P743.2 | |
| Safe-Lock tubes, 1.5 mL | Eppendorf AG | 30120086 | |
| Sample loop for 9725 injectors, volume 2 mL, PEEK | Supelco | 57648 | |
| SNAPTWIST scintillation vial, 6.5 mL | Simport Scientific Inc. | S207-5 | |
| Sterile bench | LaboGene | model: ScanLaf MARS 900 | |
| Sucrose, ≥99,5 %, p.a. | Carl Roth GmbH + Co. KG | 4621.1 | |
| Ultima-Flo AP liquid scintillation cocktail | PerkinElmer Inc | 6013599 | |
| Ultra-pure deionized water | Milli-Q | ||
| Wrenchless WVS End Fitting Kit | Hichrom Limited | 4631-1001 |
Referencias
- Berridge, M. J., Irvine, R. F. Inositol Phosphates and Cell Signaling. Nature. 341 (6239), 197-205 (1989).
- Streb, H., Irvine, R. F., Berridge, M. J., Schulz, I. Release of Ca-2+ from a Nonmitochondrial Intracellular Store in Pancreatic Acinar-Cells by Inositol-1,4,5-Trisphosphate. Nature. 306 (5938), 67-69 (1983).
- Krinke, O., Novotna, Z., Valentova, O., Martinec, J. Inositol trisphosphate receptor in higher plants: is it real. Journal of Experimental Botany. 58 (3), 361-376 (2007).
- Kuo, H. F., et al. Arabidopsis inositol pentakisphosphate 2-kinase, AtIPK1, is required for growth and modulates phosphate homeostasis at the transcriptional level. Plant Journal. 80 (3), 503-515 (2014).
- Kuo, H. F., et al. Arabidopsis inositol phosphate kinases IPK1 and ITPK1 constitute a metabolic pathway in maintaining phosphate homeostasis. Plant Journal. 95 (4), 613-630 (2018).
- Gillaspy, G. E., Capelluto, D. G. S. . Lipid-mediated Protein Signaling. , 141-157 (2013).
- Stevenson-Paulik, J., Bastidas, R. J., Chiou, S. T., Frye, R. A., York, J. D. Generation of phytate-free seeds in Arabidopsis through disruption of inositol polyphosphate kinases. Proceedings of the National Academy of Sciences of the United States of America. 102 (35), 12612-12617 (2005).
- Lemtiri-Chlieh, F., MacRobbie, E. A. C., Brearley, C. A. Inositol hexakisphosphate is a physiological signal regulating the K+-inward rectifying conductance in guard cells. Proceedings of the National Academy of Sciences. 97 (15), 8687-8692 (2000).
- Lee, H. S., et al. InsP6-sensitive variants of the Gle1 mRNA export factor rescue growth and fertility defects of the ipk1 low-phytic-acid mutation in Arabidopsis. Plant Cell. 27 (2), 417-431 (2015).
- Blüher, D., et al. A 1-phytase type III effector interferes with plant hormone signaling. Nature Communications. 8, (2017).
- Murphy, A. M., Otto, B., Brearley, C. A., Carr, J. P., Hanke, D. E. A role for inositol hexakisphosphate in the maintenance of basal resistance to plant pathogens. Plant Journal. 56 (4), 638-652 (2008).
- Poon, J. S. Y., Le Fevre, R. E., Carr, J. P., Hanke, D. E., Murphy, A. M. Inositol hexakisphosphate biosynthesis underpins PAMP-triggered immunity to Pseudomonas syringae pv. tomato in Arabidopsis thaliana but is dispensable for establishment of systemic acquired resistance. Molecular Plant Pathology. 21 (3), 376-387 (2020).
- Wild, R., et al. Control of eukaryotic phosphate homeostasis by inositol polyphosphate sensor domains. Science. 352 (6288), 986-990 (2016).
- Shears, S. B. Inositol pyrophosphates: Why so many phosphates. Advances in Biological Regulation. 57, 203-216 (2015).
- Shears, S. B. Intimate connections: Inositol pyrophosphates at the interface of metabolic regulation and cell signaling. Journal of Cellular Physiology. 233 (3), 1897-1912 (2018).
- Desai, M., et al. Two inositol hexakisphosphate kinases drive inositol pyrophosphate synthesis in plants. Plant Journal. 80 (4), 642-653 (2014).
- Laha, D., et al. VIH2 Regulates the Synthesis of Inositol Pyrophosphate InsP8 and Jasmonate-Dependent Defenses in Arabidopsis. Plant Cell. 27 (4), 1082-1097 (2015).
- Laha, D., et al. Arabidopsis ITPK1 and ITPK2 Have an Evolutionarily Conserved Phytic Acid Kinase Activity. Acs Chemical Biology. 14 (10), 2127-2133 (2019).
- Dorsch, J. A., et al. Seed phosphorus and inositol phosphate phenotype of barley low phytic acid genotypes. Phytochemistry. 62 (5), 691-706 (2003).
- Flores, S., Smart, C. C. Abscisic acid-induced changes in inositol metabolism in Spirodela polyrrhiza. Planta. 211 (6), 823-832 (2000).
- Brearley, C. A., Hanke, D. E. Inositol phosphates in barley (Hordeum vulgare L) aleurone tissue are stereochemically similar to the products of breakdown of InsP(6) in vitro by wheat-bran phytase. Biochemical Journal. 318, 279-286 (1996).
- Laha, N. P., et al. ITPK1-Dependent Inositol Polyphosphates Regulate Auxin Responses in Arabidopsis thaliana. bioRxiv. , (2020).
- Laha, D., et al. Inositol Polyphosphate Binding Specificity of the Jasmonate Receptor Complex. Plant Physiology. 171 (4), 2364-2370 (2016).
- Dong, J. S., et al. Inositol Pyrophosphate InsP(8) Acts as an Intracellular Phosphate Signal in Arabidopsis. Molecular Plant. 12 (11), 1463-1473 (2019).
- Zhu, J., et al. Two bifunctional inositol pyrophosphate kinases/phosphatases control plant phosphate homeostasis. Elife. 8, (2019).
- Couso, I., et al. Synergism between Inositol Polyphosphates and TOR Kinase Signaling in Nutrient Sensing, Growth Control, and Lipid Metabolism in Chlamydomonas. Plant Cell. 28 (9), 2026-2042 (2016).
- Ito, M., et al. Hydrophilic interaction liquid chromatography-tandem mass spectrometry for the quantitative analysis of mammalian-derived inositol poly/pyrophosphates. Journal of Chromatography A. 1573, 87-97 (2018).
- Wilson, M. S. C., Saiardi, A. Inositol Phosphates Purification Using Titanium Dioxide Beads. Bio-Protocol. 8 (15), (2018).
- Loss, O., Azevedo, C., Szijgyarto, Z., Bosch, D., Saiardi, A. Preparation of Quality Inositol Pyrophosphates. Jove-Journal of Visualized Experiments. (55), e3027 (2011).
- Harmel, R. K., et al. Harnessing C-13-labeled myo-inositol to interrogate inositol phosphate messengers by NMR Electronic supplementary information (ESI) available. Chemical Science. 10 (20), 5267-5274 (2019).
- Azevedo, C., Saiardi, A. Extraction and analysis of soluble inositol polyphosphates from yeast. Nature Protocols. 1 (5), 2416-2422 (2006).
- Shears, S. B., Miller, G. J. . Inositol Phosphates: Methods and Protocols. , 1-28 (2020).
- Wilson, M. S. C., Saiardi, A. Importance of Radioactive Labelling to Elucidate Inositol Polyphosphate Signalling. Topics in Current Chemistry. 375 (1), (2017).
- Stevenson-Paulik, J., et al. Inositol phosphate metabolomics: Merging genetic perturbation with modernized radiolabeling methods. Methods. 39 (2), 112-121 (2006).
- Liu, C., Riley, A. M., Yang, X., Shears, S. B., Potter, B. V. L. Synthesis and Biological Activity of d- and l-chiro-Inositol 2,3,4,5-Tetrakisphosphate: Design of a Novel and Potent Inhibitor of Ins(3,4,5,6)P4 1-Kinase/Ins(1,3,4)P3 5/6-Kinase. Journal of Medicinal Chemistry. 44 (18), 2984-2989 (2001).
- Hughes, P. J., Hughes, A. R., Putney, J. W., Shears, S. B. The regulation of the phosphorylation of inositol 1,3,4-trisphosphate in cell-free preparations and its relevance to the formation of inositol 1,3,4,6-tetrakisphosphate in agonist-stimulated rat parotid acinar cells. Journal of Biological Chemistry. 264 (33), 19871-19878 (1989).
- Shears, S. B., Kirk, C. J., Michell, R. H. The pathway of myo-inositol 1,3,4-trisphosphate dephosphorylation in liver. The Biochemical journal. 248 (3), 977-980 (1987).
- Stevenson-Paulik, J., Odom, A. R., York, J. D. Molecular and Biochemical Characterization of Two Plant Inositol Polyphosphate 6-/3-/5-Kinases. Journal of Biological Chemistry. 277 (45), 42711-42718 (2002).
- Stephens, L. R., Hawkins, P. T., Downes, C. P. An analysis of myo-[3H]inositol trisphosphates found in myo-[3H]inositol prelabelled avian erythrocytes. The Biochemical journal. 262 (3), 727-737 (1989).
- Saiardi, A., et al. Mammalian inositol polyphosphate multikinase synthesizes inositol 1,4,5-trisphosphate and an inositol pyrophosphate. Proceedings of the National Academy of Sciences. 98 (5), 2306 (2001).
- Azevedo, C., Burton, A., Bennett, M., Onnebo, S. M. N., Saiardi, A., Barker, C. J. . Inositol Phosphates and Lipids: Methods and Protocols. , 73-85 (2010).
- Saiardi, A., Caffrey, J. J., Snyder, S. H., Shears, S. B. The Inositol Hexakisphosphate Kinase Family: CATALYTIC FLEXIBILITY AND FUNCTION IN YEAST VACUOLE BIOGENESIS. Journal of Biological Chemistry. 275 (32), 24686-24692 (2000).
- Brearley, C. A., Hanke, D. E. Inositol phosphates in the duckweed Spirodela polyrhiza L. Biochemical Journal. 314, 215-225 (1996).
- Pollard, J. K., Steward, F. C., Shantz, E. M. Hexitols in Coconut Milk - Their Role in Nurture of Dividing Cells. Plant Physiology. 36 (4), 492 (1961).
- Donahue, J. L., et al. The Arabidopsis thaliana Myo-inositol 1-phosphate synthase1 gene is required for Myo-inositol synthesis and suppression of cell death. Plant Cell. 22 (3), 888-903 (2010).
- Desfougeres, Y., Wilson, M. S. C., Laha, D., Miller, G. J., Saiardi, A. ITPK1 mediates the lipid-independent synthesis of inositol phosphates controlled by metabolism. Proceedings of the National Academy of Sciences of the United States of America. 116 (49), 24551-24561 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados