Method Article
Extraction et quantification de polyphosphates solubles et radioalétiques d’inositol provenant de différentes espèces végétales à l’aide de SAX-HPLC
Dans cet article
Résumé
Ici, nous décrivons l’échange d’anion forte chromatographie liquide haute performance de [3H]-myo-inositol-étiqueté semis qui est une méthode très sensible pour détecter et quantifier les polyphosphates d’inositol dans les plantes.
Résumé
Les esters myode phosphate du myo-inositol, également appelé phosphates d’inositol (InsPs), sont une classe de régulateurs cellulaires jouant un rôle important dans la physiologie des plantes. En raison de leur charge négative, de leur faible abondance et de leur faible sensibilité aux activités hydrolytiques, la détection et la quantification de ces molécules est difficile. C’est particulièrement le cas pour les formes fortement phosphorylées contenant des liaisons diphospho « à haute énergie », également appelées pyrophosphates d’inositol (PP-InsP). En raison de sa sensibilité élevée, la chromatographie liquide haute performance (SAX-HPLC) des plantes étiquetées avec[3H]-myo-inositol est actuellement la méthode de choix pour analyser ces molécules. En utilisant[3H]-myo-inositol aux semis de plantes radiolabel, diverses espèces de l’InsP, y compris plusieurs isomères non enantiomères peuvent être détectés et discriminés avec une sensibilité élevée. Ici, la configuration d’un système SAX-HPLC approprié est décrite, ainsi que le flux de travail complet de la culture des plantes, l’étiquetage radio et l’extraction InsP à l’exécution SAX-HPLC et l’analyse des données ultérieure. Le protocole présenté ici permet la discrimination et la quantification de diverses espèces de l’InsP, y compris plusieurs isomères non-enantiomeric et des PP-Insp, InsP7 et InsP8, et peut être facilement adapté à d’autres espèces végétales. À titre d’exemple, les analyses SAX-HPLC des semis Arabidopsis thaliana et Lotus japonicus sont effectuées et des profils InsP complets sont présentés et discutés. La méthode décrite ici représente un outil prometteur pour mieux comprendre les rôles biologiques des InsP dans les plantes.
Introduction
Il y a près de quatre décennies, les phosphates d’inositol (InsP) sont apparus comme des molécules de signalisation, après que l’Ins(1,4,5)P3 (InsP3) a été identifié comme un deuxième messager qui active la libération de Ca2+ par récepteur dans les cellules animales1,2. À ce jour, aucun récepteur InsP3 (IP3-R) n’a été identifié dans les plantes, ce qui remet en question un rôle de signalisation directe pour l’InsP3 dans les cellules végétales3. Quoi qu’il en soit, l’InsP3 sert de précurseur pour d’autres Insp impliqués dans plusieurs processus de développement des plantes, y compris la régulation de voies de signalisation spécifiques3,4,5,6,7,8. Par exemple, InsP3 peut être encore phosphorylé à l’InsP6, également connu sous le nom « acide phytique », qui représente une source majeure de phosphate, myo-inositol et cations, et a été montré pour jouer des rôles clés dans la défense des plantes contre les agents pathogènes, l’exportation d’ARNm et l’homéostasie du phosphate5,9,10,11,12.
Les pyrophosphates d’inositol (PP-Insp) sont une classe d’Insp qui contiennent au moins un lien di-phospho à haute énergie, initialement identifié dans les cellules animales, les amibes et la levure, où ils jouent un rôle critique dans divers processus cellulaires13,14,15. Malgré les travaux séminaux sur les PP-InsP dans les plantes16,17,18,19,20,21,22,23,24,25,26, les fonctions biologiques et l’identité isomère de ces molécules restent encore largement énigmatique. Dans la plante modèle Arabidopsis thaliana, cellulaire InsP8 a été proposé pour réglementer les défenses contre les herbivores d’insectes et les champignons nécrotrophiques par coïncidence-détection de l’InsP8 et jasmonate actif par le complexe récepteur ASK1-COI1-JAZ17. En outre, les rôles de l’InsP8 et d’autres PP-InsP dans l’homéostasie énergétique et la détection des nutriments, ainsi que l’homéostasie du phosphate ont été proposés17,23,24,25,26.
Indépendamment du système biologique utilisé, un défi méthodologique majeur lors de l’étude des Insp a été la détection fiable et la quantification précise de ces molécules. Des méthodes basées sur la spectrométrie de masse ont été utilisées pour détecter les Insp, y compris les PP-Insp, à partir d’extraits cellulaires. Cependant, ces études n’ont pas réussi à différencier les isomères distincts26,27. Une autre approche pour analyser les Insp utilise le retrait des Insp des lysates cellulaires à l’aide de perles TiO2, suivie de l’électrophorèse gel polyacrylamide (PAGE) des Insps. Les Insp peuvent alors être tachés par le bleu toluidine ou DAPI24,28,29. Cependant, il n’est pas encore possible de détecter de manière fiable insPs inférieur à l’InsP5 à partir d’extraits de plantes en utilisant cette méthode. Récemment, une méthode utilisant[13C]-myo-inositol pour la résonance magnétique nucléaire (RMN) analyse des Insp a été publié comme une alternative à l’échange d’anion forte haute performance chromatographie liquide (SAX-HPLC)30. Cette technique a été rapportée pour atteindre une sensibilité semblable par rapport à SAX-HPLC et pour permettre la détection de 5-InsP7, aussi bien que la discrimination de différents insp 5 non-enantiomeric5 isomers des extraits cellulaires. Toutefois, la mise en œuvre de la méthode basée sur la RMN nécessite une synthèse chimique et non disponible dans le commerce [13C]-myo-inositol. Par conséquent, la méthode utilisée dans la plupart des cas est l’étiquetage radio avec [3H]-myo-inositol, suivi par SAX-HPLC31,32,33. Cette technique est basée sur myol’absorption de myo-inositol radioactif dans la plante et sa conversion en différents Insp par l’activité combinée des kinases cellulaires dédiées et des phosphatases.
Les InsP étiquetés[3H] sont ensuite extraits à l’acide et fractionnés à l’aide de SAX-HPLC. En raison de leur charge négative, les Insp interagissent fortement avec la phase stationnaire chargée positivement de la colonne SAX-HPLC et peuvent être élutés avec un gradient tampon contenant des concentrations croissantes de phosphate pour surpasser les InsP de la colonne. Les temps d’élution dépendent donc de la charge et de la géométrie des espèces InsP à séparer. En l’absence de colonnes chirales, seuls les isomères non enantiomeriques peuvent par séparés par ce protocole. Toutefois, des normes radio-étiques peuvent être utilisées pour attribuer la nature isomérique d’un pic InsP spécifique. Les multiples efforts déployés dans le passé par divers laboratoires pour générer des normes étiquetées et non étiquetées avec des méthodes (bio)chimiques ou pour les purifier à partir de diverses cellules et organismes ont aidé à attribuer des pics à certaines espèces de l’InsP, et aussi de réduire l’identité isomérique des espèces individuelles InsP5,7,21,34,35,36,37,38,39,40,41,42,43. En outre, l’élucidation récente des voies enzymatiques menant à la formation de PP-Insp dans les plantes, ainsi que la découverte d’un effecteur bactérien de type III avec une activité spécifique 1-phytase, fournissent des informations sur la façon de générer des normes utiles pour ces analyses10,17,18,22,23.
Les fractions résultantes peuvent être mesurées dans un compteur de scintillation liquide en raison de la β-décomposition du tritium (3H). Avec l’augmentation du temps d’étiquetage, un équilibre isotopique à état stable est atteint, après quoi les profils InsP obtenus devraient représenter le statut InsP de la plante31. Le principal avantage de ce protocole par rapport à d’autres techniques disponibles est la haute sensibilité obtenue par l’utilisation du précurseur direct pour les Insp et la mesure d’un signal radioactif.
SAX-HPLC des échantillons extraits de [3H]-myo-inositol-étiqueté plantes ou d’autres organismes est couramment utilisé pour la détection et la quantification des Insp allant de l’espèce InsP inférieure aux PP-InsP, représentant un outil précieux pour mieux comprendre le métabolisme, la fonction et les modes d’action des Insp. Jusqu’à présent, cette méthode est également le choix le plus approprié pour les chercheurs ayant un intérêt particulier pour les espèces insP inférieures. Alors que les bases de cette procédure, sur laquelle le protocole décrit ici s’appuie, ont déjà été décrits7,21,31,34, un protocole détaillé adapté à l’analyse des InsP dérivés de plantes et en particulier des PP-Insp est toujours manquant. Les publications précédentes ont signalé des difficultés à détecter de manière fiable les PP-Insp peu abondants, en particulier l’InsP8, en raison d’un ou plusieurs des facteurs suivants : des quantités relativement faibles de matières végétales, [3H]-myo-inositol avec une faible activité spécifique (> 20 Ci/mmol), l’utilisation de tampons d’extraction qui ne sont pas basés sur l’acide perchlorique ou sont moins concentrés que 1 M, différents tampons de neutralisation, ainsi que des gradients sous-optimaux ou la détection de [3H] avec un détecteur en ligne. Par rapport à ces études, le protocole présenté ici est conçu pour la détection fiable des PP-Insp7,21,34.
Nous présentons ici un flux de travail détaillé, à partir de la configuration de l’équipement pour la culture et l’étiquetage des plantes, l’extraction InsP et le SAX-HPLC s’exécutent. Bien que la méthode a été optimisée à la plante modèle A. thaliana, il peut être facilement modifié pour étudier d’autres espèces végétales, comme indiqué ici avec le premier profil InsP rapporté du modèle de légumineuse Lotus japonicus. Bien que l’utilisation d’une espèce végétale différente pourrait nécessiter une certaine optimisation, nous prévoyons que ceux-ci seront mineurs, ce qui rend ce protocole un bon point de départ pour la poursuite de la recherche dans les insPs végétaux. Afin de faciliter les optimisations possibles, nous indiquons chaque étape dans le protocole dans lequel des modifications sont possibles, ainsi que toutes les étapes critiques qui peuvent être difficiles lors de l’établissement de la méthode pour la première fois. En outre, nous rapportons comment les données obtenues par cette méthode peuvent être utilisées pour la quantification d’Insp spécifiques et comment différents échantillons peuvent être analysés et comparés.
Protocole
1. Configuration du système HPLC
- Conse le jour d’un système composé de deux pompes HPLC indépendantes (pompe binaire), une pour chaque tampon. Les deux pompes doivent être contrôlées ensemble via un ordinateur avec un logiciel respectif ou en ayant une pompe principale. Implémentez un lave-linge à piston pour les deux pompes, soit par la force gravitationnelle, soit par une troisième pompe basse pression. Désigner une pompe pour la mémoire tampon A (appelée pompe A) et une pour la mémoire tampon B (appelée pompe B).
REMARQUE : Les deux doivent être en mesure de générer des pressions allant jusqu’à 60 bar (6 MPa) et des débits d’au moins 0,5 mL/min. - Connectez les deux pompes à un mélangeur dynamique.
- Connectez le mélangeur à une valve d’injection avec une boucle d’échantillon d’une capacité d’au moins 1 mL.
- Connectez la vanne d’injection à la colonne avec un capillaire par l’intermédiaire des raccords d’extrémité correspondants.
- Connectez la colonne au collecteur de fractions à l’aide d’un capillaire avec une longueur appropriée.
REMARQUE : Cette description est basée sur notre système HPLC (voir le Tableau des matériaux),qui nécessite plus d’étapes manuelles que les systèmes plus récents et plus sophistiqués. Notre système permet un accès facile et la modification de tous les composants. Les pompes quaternaires (avec le gradient binaire décrit ici) peuvent également être utilisées et mèneront aux profils d’élution et à la qualité globale des analyses semblables à celles réalisées avec des pompes binaires.
2. Préparation des tampons, des colonnes et du système HPLC
- Préparer les tampons pour l’extraction des InsP solubles : tampon d’extraction (1 M HClO4) et tampon de neutralisation (1 M K2CO3). Préparer les deux tampons avec de l’eau déionisée ultra-pure. Ils sont stables à température ambiante pendant plusieurs mois. Immédiatement avant l’extraction, ajoutez EDTA aux deux solutions à une concentration finale de 3 mM (p. ex., à partir d’une solution de stock EDTA de 250 mM filtrée).
ATTENTION : Le HClo4 (acide perchlorique) est fortement corrosif. - Préparer les tampons pour l’exécution SAX-HPLC : tampon A (1 mM EDTA) et tampon B (1 mM EDTA, 1,3 M (NH4)2HPO4; pH 3.8 avec H3PO4). Préparer les deux à l’aide d’eau déionisée ultra-pure suivie d’une filtration sous vide avec des filtres à membrane de la taille d’un pore de 0,2 μm. Ceux-ci sont stables à température ambiante pendant plusieurs mois.
REMARQUE : L’EDTA doit être inclus dans tous les tampons afin d’éviter les interactions de cations avec les Insp, ce qui pourrait entraîner une modification de la charge InsP ou même des complexes de sel insolubles InsP. - Programmez le gradient comme suit : 0\u20122 min, 0 % tampon B; 2\u20127 min, jusqu’à 10% tampon B; 7\u201268 min, jusqu’à 84 % tampon B; 68\u201282 min, jusqu’à 100% tampon B; 82\u2012100 min, tampon b 100%, 100\u2012101 min, jusqu’à 0% tampon B; 101\u2012125 min, 0% tampon B. Le débit optimal pour ce gradient est de 0,5 mL/min.
- Pendant la course, recueillir des fractions chaque minute, à partir de la minute 1 à la minute 96. Les 30 minutes restantes du gradient servent à laver la colonne et le système, et ne doivent pas être collectées pour le comptage de scintillation.
- Si possible, réglez la pression maximale accessible avant l’arrêt d’urgence des pompes HPLC à 80 bar (8 MPa). Cela empêche les dommages critiques à la résine de la colonne.
- Lorsque vous utilisez une nouvelle colonne SAX HPLC, lavez-la soigneusement (>50 mL) avec de l’eau déionisée ultra-pure filtrée avant la première utilisation.
REMARQUE : Cela permettra d’éliminer le méthanol contenu, empêchant ainsi les précipitations de sel par étapes ultérieures. Si possible, utilisez une pompe HPLC distincte. Si cela n’est pas disponible, assurez-vous que le HPLC a rincé avec de l’eau avant de laver la colonne. Le débit ne doit pas dépasser 2 mL/min. Après le lavage, la colonne est prête pour l’analyse et, lorsqu’elle est correctement manipulée, peut être utilisée pour les courses 20\u201240. Après cela, la résolution diminuera successivement. Le lavage prolongé avec la mémoire tampon A (>1 h) et l’étape 2.6 peuvent aider à augmenter la durée de vie de la colonne. Si la diminution de la résolution persiste, la colonne doit être échangée. Le gradient peut être ajusté pour augmenter la séparation entre des espèces spécifiques de polyphosphate d’inositol ou pour diminuer le temps de course global. L’utilisation de différents systèmes HPLC (avec un volume de vide différent ou un volume différent des capillaires) affectera fortement les temps de rétention. En outre, les modifications de colonne ont des effets mineurs sur les temps de rétention. - Effectuez une « course simulée ». Au lieu d’un échantillon extrait, injectez de l’eau déionisée ultra-pure filtrée dans le système HPLC et exécutez le gradient standard. Les fractions n’ont pas besoin d’être collectées.
REMARQUE : L’étape 2.6 est facultative. Toutefois, il doit être effectué si l’une des situations suivantes s’applique : une nouvelle colonne est installée ; Le système HPLC a été utilisé pour une méthode différente à l’avance; Le système HPLC n’a pas été utilisé depuis plus de 3 jours; Il y avait un problème avec la course précédente.
3. Culture et étiquetage des plantes avec [3H]-myo-inositol
REMARQUE : Les étapes suivantes doivent être effectuées avec des composants stériles et dans des conditions stériles,tout en portant des gants pour protéger les mains contre la contamination par le radiolabel. Les milieux végétaux, en particulier lorsqu’ils contiennent du saccharose, sont sujets à la contamination microbienne.
- Stériliser les graines de A. thaliana avec 1 mL d’hypochlorite de sodium de 1,2 % pendant 3 min, suivie de 1 mL d’éthanol à 70 % pendant 3 min. Ajouter ensuite 1 mL d’éthanol à 100 %, pipette les graines avec l’éthanol sur un papier filtre circulaire et les laisser sécher à l’air sous un débit laminaire sur un banc propre.
- Lorsque vous utilisez des graines de L. japonicus, placez-les dans un mortier et frottez les graines avec du papier de verre avant la stérilisation afin d’assurer un taux de germination suffisant.
- Sèmez les graines d’Arabidopsis en 1–2 rangées sur des plats carrés de Petri remplis de supports de croissance solides composés de la solution de sel de Murashige et de Skoog (MS) à demi-force, 1% de saccharose, 0,7% de gomme gellan dans de l’eau déionisée ajustée au pH 5.7 avec KOH et leur permettent de stratifier pendant au moins 1 jour à 4 °C dans l’obscurité.
- Pour les graines de Lotus, semez-les en 1 rangée sur des plats carrés Petri remplis de milieux de croissance solide composé de 0,8% d’agar bactériologique dans de l’eau déionisée et leur permettent de stratifier pendant au moins 3 jours à 4 °C dans l’obscurité.
- Placez les plaques verticalement dans un incubateur de croissance ou une chambre climatique et laissez-les pousser pendant 10 à 12 jours dans des conditions de courte journée (8 h de lumière à 22 °C, 16 h d’obscurité à 20 °C).
- Transférer 10 à 20 semis dans un puits d’une plaque de culture cellulaire à fond plat de 12 puits, remplie de 2 mL de solution de sel de MS à demi-résistance, complétée par un saccharose de 1 % et ajustée au pH 5,7.
- Ajouter 45 μCi de[3H]-myo-inositol (30–80 Ci/mmol, dissous dans 90% d’éthanol) et mélanger par doux tourbillonnement. Couvrez la plaque avec le couvercle correspondant et scellez-la avec du ruban chirurgical microporeux (p. ex., micropore ou ruban adhésif leucopore), en la replaçant dans l’incubateur de croissance.
ATTENTION : [3H] est un émetteur bêta à faible énergie qui peut être un risque de rayonnement nocif lorsqu’il est inhalé, ingéré ou absorbé par la peau nue. Portez toujours des gants lorsque vous manipulez des matières radioactives ou de l’équipement qui a un contact direct ou indirect avec des matières radioactives. Suivez également les règles locales pour la manipulation sécuritaire des produits radiochimiques (p. ex., port de vêtements de protection supplémentaires, utilisation régulière d’un dosimètre et relevés des surfaces pour les contaminations). - Après 5 jours d’étiquetage, retirer les semis du milieu de support et les laver brièvement avec de l’eau déionisée. Séchez-les avec des serviettes en papier et transférez-les dans un tube de microcentrifuge de 1,5 m L. Ne remplissez pas trop le tube et ne placez pas plus de 100 mg fw/tube, ce qui correspond à environ 10\u201220 semis de 17 jours.
REMARQUE : Un excès de matériel végétal diluera l’acide pendant le processus d’extraction et diminuera fortement l’efficacité d’extraction.- Congeler le tube dans de l’azote liquide et le conserver à -80 °C jusqu’à l’extraction.
REMARQUE : Les échantillons peuvent être conservés à -80 °C pendant plusieurs semaines sans compromettre la qualité de l’échantillon. Les conditions de croissance (milieux, lumière, température, temps) peuvent être modifiées en fonction des besoins d’une expérience ou d’une espèce végétale spécifique. Toutefois, il faut faire attention lors de la dilution de l’inositol[3H]- myo-inositol, afin d’assurer des fonctionnements quantifiables SAX-HPLC de bonne qualité.myo Par conséquent, il est recommandé de commencer par les concentrations demyo-inositol [3H] énoncées ici et de la réduire en toute étapes si désiré. Pendant la période d’étiquetage, les plantes peuvent être soumises à différents traitements (p. ex., stress environnementaux ou agents chimiques) afin d’évaluer l’impact de ces conditions sur les Insp mondiaux. Pour atteindre l’étiquetage à l’état stable, nous vous recommandons d’étiqueter les plantes pendant au moins 5 jours.
- Congeler le tube dans de l’azote liquide et le conserver à -80 °C jusqu’à l’extraction.
4. Extraction d’insps solubles
REMARQUE : Conservez les échantillons et les réactifs sur la glace pendant tout le processus d’extraction. Portez toujours des gants et des lunettes de protection en raison du risque élevé de contact avec des matières radioactives, en particulier pendant le broyage. Tout ce qui entre en contact avec les échantillons est considéré comme des déchets radioactifs et doit être éliminé conformément aux règles locales pour l’élimination sécuritaire des matières radioactives.
- Préparer les solutions de travail pour la mémoire tampon d’extraction et de neutralisation comme à l’étape 2.1. Chaque échantillon nécessitera 600 μL de tampon d’extraction et 400 μL de tampon de neutralisation. Rangez les tampons sur la glace.
- Prenez les échantillons du congélateur de -80 °C et conservez-les dans de l’azote liquide jusqu’à ce qu’ils soient traités davantage. Broyer les échantillons à l’aide d’un pilon à tubes microcentrifuges jusqu’à ce qu’ils commencent à décongeler et ajouter 500 μL de tampon d’extraction de glace froide. Continuer le broyage jusqu’à ce que l’échantillon soit complètement homogénéisé et que la solution ait une couleur vert foncé (si des feuilles sont présentes dans l’échantillon).
- Centrifugez les échantillons pendant 10 min à 4 °C à ≥ 18000 x g. Transférer le supernatant dans un tube frais de 1,5 mL. Gardez à l’esprit que les tubes utilisés pour l’extraction sont considérés comme des déchets radioactifs solides et doivent être éliminés en conséquence.
-
Ajoutez soigneusement 300 μL de tampon de neutralisation à l’extrait. Les précipitations de protéines et de bouillonnement commenceront immédiatement. Mélanger en tourbillonnant avec une pointe de pipette après une minute et attendre quelques secondes avant de faire tourner une petite quantité (5 μL) sur du papier pH (idéalement une plage de pH 6–9). Le pH devrait être entre le pH 7 et 8 à la fin.
- Si nécessaire, ajoutez de petites quantités (généralement de 10 à 20 μL) de tampon de neutralisation ou de tampon d’extraction jusqu’à ce que le pH souhaité soit atteint. Laisser reposer les échantillons sur la glace pendant au moins 1 h avec un couvercle ouvert.
- Centrifugez les échantillons pendant 10 min à 4 °C à ≥ 18 000 x g. Transférer le supernatant dans un tube frais de 1,5 mL.
REMARQUE : Les échantillons peuvent être utilisés directement dans une course SAX-HPLC ou conservés sur la glace (s’ils sont utilisés plus tard le même jour) ou congelés dans de l’azote liquide et stockés à -80 °C pendant 2\u20124 semaines. Pour assurer une reproductibilité et une comparabilité élevées, il est recommandé de toujours congeler les échantillons dans de l’azote liquide pendant 5 min, même s’ils seront directement utilisés par la suite. Le stockage à plus long terme des échantillons extraits à -80 °C est possible tant que les échantillons ne sont décongelés qu’une seule fois. Si des échantillons congelés sont utilisés pour l’analyse, assurez-vous qu’aucune particule n’est visible après la décongélation. Sinon, centrifugez à nouveau pendant 10 min à 4 °C à ≥ 18 000 x g et transférez le supernatant dans un tube frais de 1,5 mL.
5. Exécution de l’exécution de HPLC
- Équipez le collecteur de fractions de 96 petites fioles de scintillation (capacité d’environ 6 mL) et remplissez chaque flacon de 2 mL d’un cocktail de scintillation approprié (p. ex., cocktail de scintillation liquide Ultima-Flo AP) compatible avec des tampons à faible pH et à haute concentration de phosphate d’ammonium (voir tableau des matériaux).
REMARQUE : Le nombre de flacons et la taille des flacons dépendent du collecteur de fractions et du compteur de scintillation utilisé. Il est important de recueillir au moins les 90 premières fractions,si le gradient décrit ici est utilisé, pour obtenir un profil polyphosphate d’inositol complet. Assurez-vous également d’étiqueter correctement chaque flacon et son couvercle respectif, afin d’éviter la confusion des fractions ou des échantillons. - Démarrez le système/pompe HPLC et préparez-le à fonctionner. Activez le lavage du joint à piston et maintenez-le activé pendant toute la course. Chargez l’échantillon en injectant manuellement le supernatant complet à partir de l’étape 4.5 (environ 750 μL) à l’aide d’uneseringue appropriée (voir tableau des matériaux). Si l’injection automatique est possible, transférer l’échantillon dans le flacon d’échantillon correspondant. Tournez la vanne de la position « charge » à la position « injecter » et démarrez le gradient et le collecteur de fractions.
REMARQUE : Selon le système HPLC utilisé, la procédure de démarrage peut différer, en particulier lorsqu’on compare les anciens systèmes (comme décrit ici) à un nouveau modèle entièrement contrôlé par logiciel. Il est très important de s’assurer que le gradient, l’injection d’échantillon et la collecte des fractions commencent simultanément. - Pendant que la course HPLC est en cours, vérifiez la pression régulièrement. La pression de départ devrait être d’environ 18 à 24 bar (1,8–2,4 MPa) et devrait augmenter lentement à 50–60 bar (5–6 MPa) une fois que le tampon B à 100 % est atteint.
ATTENTION : Une diminution de la pression peut indiquer une fuite dans le système tandis que l’augmentation de la pression indique un blocage. Les fluctuations de pression (≥ 3 bar en quelques secondes) peuvent indiquer la présence d’air dans le système. Gardez à l’esprit que tout ce qui quitte la colonne, ainsi que chaque fuite qui se produit à l’injecteur ou après est radioactif.
REMARQUE : La pression dépend également du système HPLC et peut être inférieure ou supérieure à celle indiquée ici. Il augmentera lentement après environ 15 à 20 manches. Toutefois, cela n’influence pas nécessairement la qualité des pistes obtenues. - Après la course, fermer les flacons hermétiquement et mélanger les fractions avec le cocktail de scintillation en secouant vigoureusement. Procédez directement à la mesure ou maintenez les flacons en position verticale, idéalement dans l’obscurité.
REMARQUE : Les fractions mélangées au cocktail de scintillation sont stables pendant des semaines et peuvent être mesurées plus tard. Puisque la demi-vie du tritium est de 12,32 ans, la perte de signal est négligeable. - Une fois la course du dernier échantillon de la journée terminée, arrêtez les deux pompes HPLC.
- (Facultatif) Pour augmenter la longévité du système, surtout lorsqu’il n’est pas utilisé régulièrement, lavez la pompe B et les capillaires en plaçant le capillaire du tampon B dans une bouteille avec tampon A et laissez la pompe fonctionner pendant 10 à 15 min. Avant l’utilisation suivante, n’oubliez pas de remplacer le capillaire en tampon B et de découpler la pompe B du mélangeur pour la rincer avec le tampon B. Une fois que la pompe et les capillaires sont remplis à nouveau avec la mémoire tampon B, reconnectez-la avec le mélangeur et le système est prêt à l’emploi.
6. Mesurer les fractions
- Insérez les flacons dans des supports de compteur de scintillation et mesurez chaque flacon pendant 5 min dans un compteur de scintillation liquide.
- Idéalement, utilisez des supports qui s’adaptent directement aux petites fioles et évitez de les accrocher dans les flacons dans des flacons plus grands (p. ex., 20 mL) pour réduire les erreurs de comptage. Les paramètres logiciels utilisés dans ce protocole sont affichés dans la figure supplémentaire 1.
REMARQUE : Effectuez régulièrement un protocole SNC (auto-normalisation et étalonnage) à l’aide de normes non incinées[3H]. Des temps de comptage plus courts (1–5 min) sont possibles pour réduire le temps d’attente. Toutefois, pour assurer une reproductibilité et une précision de comptage élevées, 5 min sont recommandés.
7. Analyse des données
- Exportez les mesures à partir du compteur de scintillation sous forme de fichier feuille de calcul ou de format de fichier compatible/convertible. Évaluez les données à l’aide d’un ordinateur équipé d’Excel ou d’un logiciel similaire, et d’un logiciel d’analyse approprié comme Origin.
- Préparer un graphique linéaire 2D où le nombre mesuré par minute (cpm) est tracé en fonction du temps de rétention (voir la figure 1, figure 2).
- Pour comparer les échantillons les uns avec les autres, normaliser les données en résumant le cpm de chaque fraction éluted de la minute 25 à 96 pour chaque échantillon individuel.
REMARQUE : La minute 25 sert de seuil pour exclure les personnes non constituées en société [3H]-myo-inositol, InsP1 et InsP2 de l’analyse, car celles-ci ont tendance à fluctuer fortement et ne peuvent pas être bien séparées (du moins avec le gradient proposé dans ce protocole) et modifient ainsi fortement le facteur de normalisation en raison de leur activité élevée. - Normaliser toutes les données de l’échantillon avec le cpm total le plus faible (en fractions 25\u201296) en divisant le cpm total de l’échantillon avec le cpm le plus bas (en fractions 25–96) par le nombre total de cpm (en fractions 25–96) des autres échantillons. Le facteur résultant peut alors être utilisé pour normaliser le cpm de chaque fraction en multipliant le cpm de chaque fraction avec le facteur.
REMARQUE : En fin de compte, la somme des valeurs de cpm de la minute 25 à la fin doit être égale pour tous les échantillons par rapport aux autres. Seules les séries normalisées doivent être présentées dans le même graphique/chiffre (lorsqu’elles sont présentées comme des profils réels). La figure supplémentaire 2 montre un exemple de la façon dont ces étapes de calcul sont effectuées (en utilisant seulement les fractions 25–35 de deux échantillons pour la simplification). Toutefois, dans certains cas, il n’est pas nécessaire de normaliser les données. Par exemple, lorsque les pics sont quantifiés selon l’étape 7.4 et présentés en pourcentages du total des Insp (comme indiqué à la figure 3D). Comme indiqué précédemment, lors de la présentation de multiples analyses côte à côte comme profils, ou lorsque l’activité mesurée réelle est utilisée pour les conclusions (p. ex., le traitement a) augmente InsP7 de x% par rapport au contrôle, se référant aux valeurs cpm de l’InsP7 des deux échantillons et non à leur pourcentage de l’InsP total) normalisation est nécessaire. Pour analyser l’effet des différences de génotype ou de traitement sur l’efficacité de l’étiquetage, il est important de ne pas normaliser,car cela invaliderait ces différences. Cependant, la quantification absolue avec cette méthode est difficile parce que l’efficacité d’extraction avec ce protocole peut être variable pour diverses raisons et sont parfois même observées lorsque la réplique du même génotype et le traitement sont analysés. Gardez à l’esprit que selon le système HPLC, la colonne et le dégradé utilisés pour les analyses, le seuil peut devoir être modifié. - Pour effectuer des quantifications relatives de certains pics de polyphosphate d’inositol et pour créer par la suite des graphiques à barres qui contiennent des données de réplications pour les analyses statistiques, poursuivez l’analyse avec un logiciel spécialisé qui peut calculer les zones de pointe des chromatogrammes (p. ex., Origin). Voir Figure supplémentaire 3.
REMARQUE : La plupart des systèmes HPLC contrôlés par logiciel sont fournis avec un logiciel respectif capable de cette tâche. Les pics sont déterminés comme les fractions avec des valeurs cpm au-dessus de l’arrière-plan (qui varie dans une certaine mesure entre les pistes) et les temps de rétention qui sont similaires aux données publiées précédemment. Le temps de rétention d’un pic spécifique est déterminé dans un logiciel de feuille de calcul (p. ex., Excel) et utilisé pour attribuer des pics pour le calcul d’intégrales définies (p. ex., dans Origin). La figure supplémentaire 3 illustre ce processus de détermination maximale, de soustraction de fond et d’intégration des pics.
Résultats
Les résultats présentés ici visent à illustrer les résultats possibles obtenus en fonction des variations aux niveaux technique et biologique. La première est illustrée par des analyses utilisant de nouvelles colonnes par rapport aux colonnes âgées (figure 1) et des échantillons frais par rapport aux échantillons stockés (figure 3), et la seconde par l’évaluation d’extraits de deux systèmes végétaux différents, A. thaliana (Figure 1, Figure 3) et L. japonicus (Figure 2).
Une course optimale de SAX-HPLC est représentée à la figure 1A\u2012C, qui montre un spectre polyphosphate d’inositol complet obtenu à partir d’extraits de A. thaliana après comptage de scintillation. Notez que les pics sont bien séparés et peuvent être attribués à différents isomères (ou enantiomère-paires) basés sur les mobilités chromatographiques décrites plus tôt5,7.
La figure 2 montre le résultat représentatif d’une analyse SAX-HPLC des semis de L. japonicus qui ont été cultivés et étiquetés dans les mêmes conditions que les semis d’Arabidopsis. Bien que l’on puisse vraisemblablement voir toutes les espèces et les pics de l’InsP connus à partir d’Arabidopsis, il existe des différences importantes en ce qui concerne la quantité relative (p. ex., les ratios entre isomères) de isomères InsP spécifiques, lorsqu’on compare les profils des deux espèces. Par exemple, les extraits de Lotus ont montré augmenté InsP3c, InsP4b, InsP5b et réduit InsP3a, InsP4a, InsP5a et InsP5c par rapport à Arabidopsis qui laisse place à d’autres enquêtes. La figure 2D illustre les différents ratios entre les isomères d’InsP entre Arabidopsis et Lotus.
La figure 3 montre deux profils InsP d’un échantillon qui a été divisé après l’extraction. La première moitié a été immédiatement analysée et la seconde moitié un jour plus tard, après stockage à -80 °C. Notez que seules des différences mineures sont observées entre les différents échantillons (c.-à-d. lignes noires et rouges à la figure 3A\u2012C) et figure 3D). Cela montre qu’un cycle gel-dégel ne nuit pas à l’échantillon et que la méthode elle-même génère des résultats reproductibles.
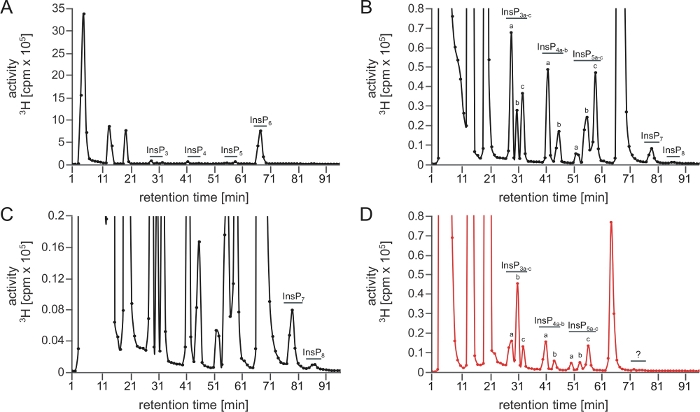
Figure 1 : Profil insP typique d’une analyse RÉUSSIE et d’une analyse SAX-HPLC infructueuse effectuée avec ce protocole. (A\u2012C) Profil SAX-HPLC de 17 jours de type sauvage (Col-0) Arabidopsis semis radiolabeled avec [3H]-myo-inositol. L’extraction globale d’InsP et l’exécution de SAX-HPLC ont été exécutées le même jour. (A) Spectres complets; (B, C) Zoom-ins du profil indiqué dans A. Tous les pics visibles sont mis en évidence et attribués aux espèces InsP correspondantes. Basé sur les mobilités chromatographiques publiées5,7, InsP4a représente probablement Ins(1,4,5,6)P4 ou Ins(3,4,5,6)P4, InsP5a représente InsP5 [2-OH], InsP5b représente l’InsP5 [4-OH] ou sa forme enantiomère InsP5 [6-OH], et InsP5c représente InsP5 [1-OH] ou sa forme enantiomérique InsP5 [3-OH]. Les natures isomériiques d’InsP3a-c, InsP4b, InsP7et InsP8 sont encore inconnues. Panel (D) montre un profil SAX-HPLC des plantes cultivées de façon identique, mais à l’aide d’une colonne vieillie (>40 fonctionne). Une réduction nette de l’InsP6 par rapport à d’autres espèces de l’InsP et l’absence de PP-InsPs est visible. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
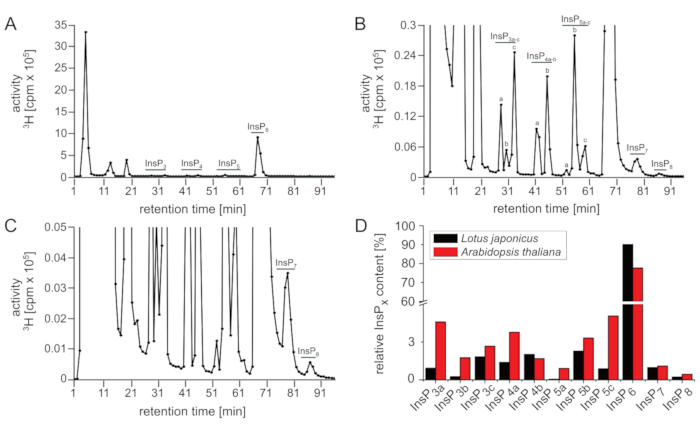
Figure 2 : Profil insP représentatif des plantes L. japonicus. Profil SAX-HPLC (A\u2012C) de 17 jours de type sauvage (Gifu) L. japonicus semis radiolabeled avec [3H]-myo-inositol. (A) Spectres complets; (B, C) Zoom-ins du profil indiqué dans A. Tous les pics visibles sont mis en évidence et attribués aux espèces InsP correspondantes. D’après les mobilités chromatographiques publiées5,7, InsP4a représente probablement Ins(1,4,5,6)P4 ou Ins(3,4,5,6)P4, InsP5b représente probablement InsP5 [4-OH] ou sa forme enantiomérique InsP5 [6-OH], et InsP5c représente probablement InsP5 [1-OH] ou sa forme enantiomère InsP5 [3-OH]. Les natures isomériiques d’InsP3a-c, InsP4b, InsP7et InsP8 sont inconnues. (D) Comparaison entre les espèces insP individuelles (en % de l’activité totale de l’élution 25\u201296) de A. thaliana (données de la figure 1A\u2012C) et L. japonicus (données de la figure 2A–C). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
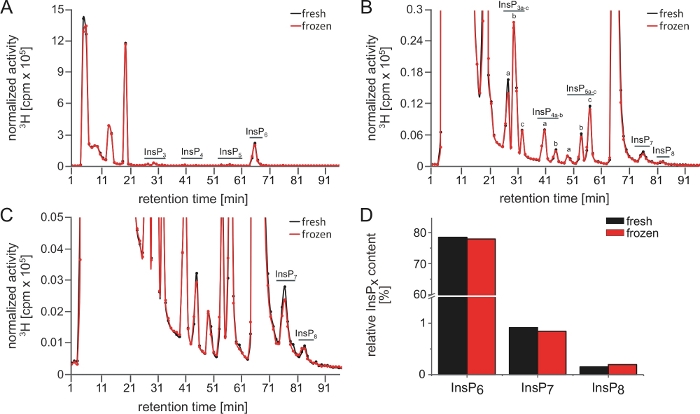
Figure 3 : Profils Insp d’un échantillon fractionné illustrant la reproductibilité des analyses SAX-HPLC. (A\u2012C) Profils SAX-HPLC de 17 jours de type sauvage (Col-0) Arabidopsis semis radiolabeled avec [3H]-myo-inositol. Avant la course, l’échantillon a été divisé et une demi-exécution immédiatement et l’autre moitié un jour plus tard après le stockage à -80 °C. (A) Spectres complets; (B, C) Zoom-ins du profil indiqué dans A. Tous les pics visibles sont mis en évidence et attribués aux espèces InsP correspondantes. Basé sur les mobilités chromatographiques publiées5,7, InsP4a représente probablement Ins(1,4,5,6)P4 ou Ins(3,4,5,6)P4, InsP5a représente InsP5 [2-OH], InsP5a représente InsP5 [2-OH], InsP5b représente InsP5 [4-OH] ou sa forme enantiomérique InsP5 [6-OH], et InsP5c représente InsP5 [1-OH] ou sa forme enantiomérique InsP5 [3-OH]. Les natures isomériiques d’InsP3a-c, InsP4b, InsP7et InsP8 sont encore inconnues. Le panneau D montre la quantification de l’InsP6 et des PP-InsP InsP7 et InsP8 des deux pistes. Les valeurs représentent le montant (en %) des espèces respectives de l’InsP par rapport à toutes les InsP (activité totale de l’élution 25–96). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Figure supplémentaire 1 : Paramètres logiciels pour le comptage de la scintillation liquide à l’aide d’un compteur de scintillation légère. Des captures d’écran montrant la version logicielle, ainsi que les paramètres utilisés pour le comptage de scintillation des échantillons de[3H] effectués avec ce protocole sont représentés. Veuillez cliquer ici pour télécharger ce chiffre.
Figure supplémentaire 2 : Exemple représentatif de normalisation des données. Une capture d’écran d’une feuille de calcul montre toutes les étapes et formules utilisées pour normaliser les courses SAX-HPLC les unes aux autres. Pour la simplification, seules les fractions 25 à 35 des échantillons sont indiquées. Veuillez cliquer ici pour télécharger ce chiffre.
Figure supplémentaire 3 : Détermination maximale, soustraction de fond et intégration à l’aide d’un logiciel d’analyse. (A) Les données de l’analyse SAX-HPLC sont chargées dans le logiciel (minutes 28–96) et l’outil d’analyse de pointe est sélectionné. (B\u2012E) La ligne de base est définie manuellement par le réglage des points entre les pics individuels et l’arrière-plan est soustrait. (F) Les pics sont déterminés manuellement en fonction de l’apparence et des mobilités chromatographiques publiées5,7. (G) Les plages de pointe sont définies manuellement par des valeurs cpm. (H) Les pics sont intégrés et calculés en % de tous les pics. Veuillez cliquer ici pour télécharger ce chiffre.
Discussion
Ici, nous présentons une méthode polyvalente et sensible pour quantifier les Insp, y compris les PPP-InsPs dans les extraits de plantes et de fournir des conseils pratiques sur la façon d’obtenir cette méthode établie. Même si le protocole est généralement robuste, des pistes et des analyses sous-optimales peuvent se produire. Dans la plupart des cas, ces pistes peuvent être identifiées par une forte réduction ou même la perte complète d’Insps hautement phosphorylés, en particulier les espèces PP-InsP InsP7 et InsP8. Les raisons possibles peuvent être la contamination microbienne du matériel végétal et la désactivation insuffisante des hydrolases PP-InsP de la plante endogène pendant l’extraction en raison d’un broyage et d’un dégel insuffisants du matériel végétal qui ne sera pas en contact immédiat avec le tampon d’extraction. D’autres raisons incluent un ajustement inexact du pH par ajout insuffisant ou excessif de tampon de neutralisation, ou tout simplement un matériel d’échantillonnage insuffisant. Ce dernier peut rendre difficile la détection des PP-InsP, car ceux-ci sont souvent présents en très faibles quantités dans les cellules. Un excès de matériel d’échantillon ou un séchage inefficace au cours de l’étape 3.5 peut provoquer la dilution de l’acide perchlorique, ce qui entraîne également une désactivation insuffisante des enzymes et une perte spécifique des Insp6 et PP-InsP. La quantité de matériel végétal, ainsi que le radiolabel utilisé dans ce protocole ont été optimisées en fonction des coûts et des performances, et est donc proche de la quantité la plus basse qui est encore suffisante pour fournir des résultats optimaux. De plus, la résine de colonne perdra progressivement sa capacité de résolution. Le premier signe de ce processus est (pour des raisons qui ne sont pas tout à fait claires pour les auteurs) une perte spécifique d’espèces insp plus phosphorylées comme les PP-Insp dans le spectre hplc. Avec le vieillissement supplémentaire, même InsP6 ne sera pas résolu correctement par la colonne (Figure 1D). Par conséquent, l’utilisation d’une colonne adéquate, ainsi que la manipulation méticuleuse de l’échantillon et la maintenance appropriée des composants HPLC est cruciale pour assurer des résultats précis.
Lorsqu’on compare des échantillons et des pistes, en particulier lorsqu’ils sont générés avec différents équipements (p. ex., les systèmes et colonnes hplc) ou à des jours différents, il est crucial de normaliser les échantillons les uns aux autres (comme décrit à l’étape 7.3) et de les analyser de la même manière. Ce n’est que par la normalisation qu’il est possible et précis d’afficher plusieurs échantillons dans le même graphique (figure 3). Pour la quantification des Insp individuels par rapport aux Insp totaux, ou à une autre espèce spécifique de l’InsP, il n’est pas nécessaire de normaliser, tant que seules les valeurs relatives et non les valeurs absolues sont affichées. Idéalement, les profils InsP et les quantifications sont indiqués. Toutefois, dans certains cas, il n’est pas possible d’afficher adéquatement deux ou plusieurs séries dans le même graphique. Des temps de rétention différents ou différents niveaux d’activité en arrière-plan peuvent rendre difficile la comparaison des profils SAX-HPLC non quantifiés seuls. Il en va de même lorsque de nombreux échantillons doivent être comparés. Dans de tels cas, une évaluation plus poussée à l’aide d’un logiciel supplémentaire (p. ex., Origin) pour la quantification individuelle des pics est nécessaire.
Les auteurs sont conscients que le protocole décrit ici peut être optimisé et doit être adapté à chaque question de recherche individuelle. Bien qu’elle soit optimisée pour les extraits d’Arabidopsis 7,17 dans ce protocole, cette méthode est polyvalente et peut aider à déterminer les profils InsP d’autres espèces végétales ainsi. Ici, nous exemplifions cette possibilité en présentant pour la première fois un profil InsP pour L. japonicus, qui n’a exigé aucune modification des conditions d’étiquetage, extraction InsP ou sax-HPLC exécuter (Figure 2). Notamment, bien que dans l’ensemble similaire, des différences sont observées entre les profils L. japonicus et Arabidopsis InsP. Par exemple, dans L. japonicus Insp5 [4-OH] ou sa forme enantiomérique InsP5 [6-OH] sont plus abondants que l’InsP5 [1-OH] ou sa forme enantioérique InsP5 [3-OH] par rapport à Arabidopsis, où l’InsP5 [1-OH] ou sa forme enantiomérique InsP5 [3-OH] sont les espèces dominantes de l’InsP5. De même, nous prévoyons que des altérations de la composition des milieux,[3H]- concentration demyo-inositol, âge des plantes, conditions environnementales (par exemple, lumière et température), ajout de composés chimiques ou analyses d’interactions végétales-microbiennes entre autres facteurs, pourraient devoir être testées et adaptées.
Un inconvénient important de cette méthode qui doit être pris en considération est que l’étiquetage se fait dans une culture liquide (stérile), qui ne représente pas un environnement physiologique pour la plupart des plantes terrestres. En outre, en raison des coûts élevés de [³H]-myo-inositol, le volume de la solution d’étiquetage et la taille du navire de culture est généralement limité, ce qui limite également la taille des plantes qui peuvent être utilisées. La culture liquide peut être évitée en infiltrant directement par exemple les feuilles de plantes cultivées dans le sol avec [³H]-myo-inositol et en suivant par la suite le protocole décrit ici, comme précédemment rapporté10.
Il existe plusieurs inconvénients de ce protocole par rapport aux méthodes alternatives, telles que TiO2 pull-down suivie par PAGE ou des techniques basées sur la spectrométrie de masse. En raison de l’étiquetage demyo-inositol de[3H], seules les espèces d’InsP qui proviennent directement du myoradio-inositol seront détectées à la fin. La méthode décrite ici est aveugle à d’autres isomères Ins tels que le scyllo-inositol et d’autres isomères dont certains ont été identifiés dans certaines plantes44. En outre, le myo-InsP dérivé d’autres voies sera exclu, y compris ceux synthétisés par synthèse de novo de myo-inositol et myo-inositol-3-phosphate via isomérisation du glucose-6-phosphate, catalysé par myo-inositol-3-phosphate synthase (MIPS) protéines45. myo Bien que[32P] ou [33P]-ortho-phosphatepuisse être utilisé comme étiquettes alternatives, leur utilisation pose un inconvénient majeur, puisque chaque molécule contenant du phosphate, y compris les nucléotides abondants et ses dérivés, sera étiquetée. Ces molécules peuvent également être extraites avec ce protocole et se lier à la colonne SAX, ce qui se traduira par un niveau élevé d’activité de fond qui interférera avec l’identification des pics individuels InsP5. En outre, la quantification de[32P]- ou [33P] -étiquetés Insp et PP-Insp peut être fortement influencée par le chiffre d’affaires de phosphate et de pyrophosphate moiety et pourrait ne pas signaler une lecture de masse pour les espèces d’inositol.
D’autre part, [3H]-myo-inositol étiquette spécifiquement myo-molécules contenant de l’inositol. Les insP, les lipides contenant de l’inositol, tels que les phosphoinositides, et le galactinol sont dans ce cas étiquetés. Cependant, seuls les Insp seront analysés avec ce protocole, puisque les lipides sont insolubles dans le tampon d’extraction et le galactinol ne se lie pas à la colonne SAX.
Jusqu’à présent, les différences par rapport au profil InsP d’une usine généré par [3H]-étiquetage myo-inositolpar rapport à celui déterminé par TiO2 pulldown/PAGE reste inconnue, puisque de telles comparaisons n’ont pas été effectuées dans les plantes. Une étude récente sur les cellules animales a abordé cette question46. Dans ce travail, un pool d’InsP6 qui est invisible par[3H]- étiquetage myo-inositol, qui devrait ainsi être directement dérivé du glucose-6-phosphate, a été identifié en comparant les profils SAX-HPLC avec des gels PAGE des lignées cellulaires des mammifères.myo 24 h de famine de phosphate a eu comme conséquence une augmentation de 150% de l’InsP6 en quantifiant des gels de PAGE des InsP purifiés utilisant le pulldown de TiO2. Les analyses SAX-HPLC de [3H]-cellules étiquetées myo-inositolqui ont été traitées de façon identique seulement ont montré une augmentation de 15% de [3H]-InsP6. Comme nous l’avons mentionné précédemment, les Insp inférieurs à l’InsP5 sont indétectables avec l’analyse PAGE dans la plupart des cas. L’étiquetage radio suivi par SAX-HPLC semble être la méthode de choix, tant que les protocoles spectrométriques de masse ne sont pas optimisés pour détecter ce groupe de molécules fortement chargées négativement.
Un autre défi restant consiste à distinguer les enantiomères dans les analyses SAX-HPLC (ou dans toute autre méthode d’analyse InsP)10,17. Ce défi peut être relevé par l’ajout de sélecteurs chiraux, c’est-à-dire des composés enantiopures comme l’amide L-arginine qui interagissent avec les molécules enantiomérites respectives pour former des complexes diastéréomérisaires qui peuvent être séparés10. À notre connaissance, cette approche n’a été mise en œuvre que pour discriminer les insp5 isomers insP5 [1-OH] et InsP5 [3-OH] par les analyses RMN10. La discrimination d’autres paires enantioméries ou la discrimination réussie des enantiomères par l’analyse chirale SAX-HPLC ou les méthodes chirales basées sur la PAGE n’ont pas encore été signalées et devraient être développées davantage. Compte tenu de la synthèse conservée et de la réglementation conservée des PP-InsP par disponibilité au phosphore, nous envisageons que des méthodes particulièrement non radioactives telles que les méthodes à base de PAGE ou de MS, ainsi que les analyses de nutriments, aideront à mettre au sol les efforts de vérité pour calibrer les données de télédétection conçues pour diagnostiquer les carences en nutriments dans les cultures17,18,24,25. Cependant, la méthode présentée ici peut actuellement encore être considérée comme l’étalon-or pour les analyses InsP et sera déterminante pour découvrir de nouvelles fonctions de ces messagers intrigants dans les plantes.
Déclarations de divulgation
Les auteurs n’ont rien à révéler.
Remerciements
Ces travaux ont été financés par la Deutsche Forschungsgemeinschaft (DFG, Fondation allemande de recherche) dans le cadre de la stratégie d’excellence de l’Allemagne - EXC-2070 – 390732324 (PhenoRob), le Groupe de formation à la recherche GRK2064 et des subventions individuelles de recherche SCHA1274/4-1 et SCHA1274/5-1 à G.S. Nous remercions également Li Schlüter et Brigitte Ueberbach pour leur assistance technique.
matériels
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf AG | model: 5430 R | |
| Diammonium hydrogen phosphate, ≥97 % | Carl Roth GmbH + Co. KG | 0268.1 | |
| Ethylenediamine tetraacetic acid disodium salt dihydrate, ≥99 %, p.a., ACS | Carl Roth GmbH + Co. KG | 8043.2 | |
| Falcon 12-well clear flat bottom TC-treated multiwell cell culture plate, with lid, individually wrapped, sterile | Corning Inc. | 353043 | |
| Fraction collector | LAMBDA Instruments GmbH | model: OMNICOLL single channel collector | |
| Growth incubator | poly klima GmbH | model: PK 520-LED | |
| HPLC pumps | Kontron Instruments | model: 420 | |
| HPLC syringe for Rheodyne valves, 1 mL | Hamilton Company | 81365 | |
| Injector for HPLC | Supelco | model: Rheodyne 9725 | |
| Inositol, myo-[1,2-3H(N)] | American Radiolabeled Chemicals Inc. | ART 0261 | |
| Liquid nitrogen | University, Chemistry Department | ||
| Liquid scintillation counter | PerkinElmer Inc | model: TRI-CARB 2900TR | |
| Micro pestle | Carl Roth GmbH + Co. KG | CXH7.1 | |
| Mixed cellulose Eester filter, ME range (ME 24), plain, 0.2 µm pore size, 47 mm circle | GE Healthcare Life Sciences | 10401770 | |
| Mixer for HPLC | Kontron Instruments | model: M 800 | |
| Murashige & Skoog medium, salt mixture | Duchefa Biochemie | M0221 | |
| OriginPro software | OriginLab Corp. | ||
| Orthophosphoric acid, ≥85 %, p.a., ISO | Carl Roth GmbH + Co. KG | 6366.1 | |
| Partisphere 5 µm SAX cartridge column, 125 x 4.6 mm | Hichrom Limited | 4621-0505 | |
| Perchloric acid, 70 %, 99.999 % trace metals basis | Sigma-Aldrich | 311421 | |
| Petri dish, square, PS, clear, 120/120/17 mm, sterile | Greiner Bio One International GmbH | 688161 | |
| pH-indicator paper pH 5.5 - 9.0, Neutralit | Merck KGaA | 109564 | |
| Phytagel | Sigma-Aldrich | P8169 | |
| Potassium carbonate | Carl Roth GmbH + Co. KG | P743.2 | |
| Safe-Lock tubes, 1.5 mL | Eppendorf AG | 30120086 | |
| Sample loop for 9725 injectors, volume 2 mL, PEEK | Supelco | 57648 | |
| SNAPTWIST scintillation vial, 6.5 mL | Simport Scientific Inc. | S207-5 | |
| Sterile bench | LaboGene | model: ScanLaf MARS 900 | |
| Sucrose, ≥99,5 %, p.a. | Carl Roth GmbH + Co. KG | 4621.1 | |
| Ultima-Flo AP liquid scintillation cocktail | PerkinElmer Inc | 6013599 | |
| Ultra-pure deionized water | Milli-Q | ||
| Wrenchless WVS End Fitting Kit | Hichrom Limited | 4631-1001 |
Références
- Berridge, M. J., Irvine, R. F. Inositol Phosphates and Cell Signaling. Nature. 341 (6239), 197-205 (1989).
- Streb, H., Irvine, R. F., Berridge, M. J., Schulz, I. Release of Ca-2+ from a Nonmitochondrial Intracellular Store in Pancreatic Acinar-Cells by Inositol-1,4,5-Trisphosphate. Nature. 306 (5938), 67-69 (1983).
- Krinke, O., Novotna, Z., Valentova, O., Martinec, J. Inositol trisphosphate receptor in higher plants: is it real. Journal of Experimental Botany. 58 (3), 361-376 (2007).
- Kuo, H. F., et al. Arabidopsis inositol pentakisphosphate 2-kinase, AtIPK1, is required for growth and modulates phosphate homeostasis at the transcriptional level. Plant Journal. 80 (3), 503-515 (2014).
- Kuo, H. F., et al. Arabidopsis inositol phosphate kinases IPK1 and ITPK1 constitute a metabolic pathway in maintaining phosphate homeostasis. Plant Journal. 95 (4), 613-630 (2018).
- Gillaspy, G. E., Capelluto, D. G. S. . Lipid-mediated Protein Signaling. , 141-157 (2013).
- Stevenson-Paulik, J., Bastidas, R. J., Chiou, S. T., Frye, R. A., York, J. D. Generation of phytate-free seeds in Arabidopsis through disruption of inositol polyphosphate kinases. Proceedings of the National Academy of Sciences of the United States of America. 102 (35), 12612-12617 (2005).
- Lemtiri-Chlieh, F., MacRobbie, E. A. C., Brearley, C. A. Inositol hexakisphosphate is a physiological signal regulating the K+-inward rectifying conductance in guard cells. Proceedings of the National Academy of Sciences. 97 (15), 8687-8692 (2000).
- Lee, H. S., et al. InsP6-sensitive variants of the Gle1 mRNA export factor rescue growth and fertility defects of the ipk1 low-phytic-acid mutation in Arabidopsis. Plant Cell. 27 (2), 417-431 (2015).
- Blüher, D., et al. A 1-phytase type III effector interferes with plant hormone signaling. Nature Communications. 8, (2017).
- Murphy, A. M., Otto, B., Brearley, C. A., Carr, J. P., Hanke, D. E. A role for inositol hexakisphosphate in the maintenance of basal resistance to plant pathogens. Plant Journal. 56 (4), 638-652 (2008).
- Poon, J. S. Y., Le Fevre, R. E., Carr, J. P., Hanke, D. E., Murphy, A. M. Inositol hexakisphosphate biosynthesis underpins PAMP-triggered immunity to Pseudomonas syringae pv. tomato in Arabidopsis thaliana but is dispensable for establishment of systemic acquired resistance. Molecular Plant Pathology. 21 (3), 376-387 (2020).
- Wild, R., et al. Control of eukaryotic phosphate homeostasis by inositol polyphosphate sensor domains. Science. 352 (6288), 986-990 (2016).
- Shears, S. B. Inositol pyrophosphates: Why so many phosphates. Advances in Biological Regulation. 57, 203-216 (2015).
- Shears, S. B. Intimate connections: Inositol pyrophosphates at the interface of metabolic regulation and cell signaling. Journal of Cellular Physiology. 233 (3), 1897-1912 (2018).
- Desai, M., et al. Two inositol hexakisphosphate kinases drive inositol pyrophosphate synthesis in plants. Plant Journal. 80 (4), 642-653 (2014).
- Laha, D., et al. VIH2 Regulates the Synthesis of Inositol Pyrophosphate InsP8 and Jasmonate-Dependent Defenses in Arabidopsis. Plant Cell. 27 (4), 1082-1097 (2015).
- Laha, D., et al. Arabidopsis ITPK1 and ITPK2 Have an Evolutionarily Conserved Phytic Acid Kinase Activity. Acs Chemical Biology. 14 (10), 2127-2133 (2019).
- Dorsch, J. A., et al. Seed phosphorus and inositol phosphate phenotype of barley low phytic acid genotypes. Phytochemistry. 62 (5), 691-706 (2003).
- Flores, S., Smart, C. C. Abscisic acid-induced changes in inositol metabolism in Spirodela polyrrhiza. Planta. 211 (6), 823-832 (2000).
- Brearley, C. A., Hanke, D. E. Inositol phosphates in barley (Hordeum vulgare L) aleurone tissue are stereochemically similar to the products of breakdown of InsP(6) in vitro by wheat-bran phytase. Biochemical Journal. 318, 279-286 (1996).
- Laha, N. P., et al. ITPK1-Dependent Inositol Polyphosphates Regulate Auxin Responses in Arabidopsis thaliana. bioRxiv. , (2020).
- Laha, D., et al. Inositol Polyphosphate Binding Specificity of the Jasmonate Receptor Complex. Plant Physiology. 171 (4), 2364-2370 (2016).
- Dong, J. S., et al. Inositol Pyrophosphate InsP(8) Acts as an Intracellular Phosphate Signal in Arabidopsis. Molecular Plant. 12 (11), 1463-1473 (2019).
- Zhu, J., et al. Two bifunctional inositol pyrophosphate kinases/phosphatases control plant phosphate homeostasis. Elife. 8, (2019).
- Couso, I., et al. Synergism between Inositol Polyphosphates and TOR Kinase Signaling in Nutrient Sensing, Growth Control, and Lipid Metabolism in Chlamydomonas. Plant Cell. 28 (9), 2026-2042 (2016).
- Ito, M., et al. Hydrophilic interaction liquid chromatography-tandem mass spectrometry for the quantitative analysis of mammalian-derived inositol poly/pyrophosphates. Journal of Chromatography A. 1573, 87-97 (2018).
- Wilson, M. S. C., Saiardi, A. Inositol Phosphates Purification Using Titanium Dioxide Beads. Bio-Protocol. 8 (15), (2018).
- Loss, O., Azevedo, C., Szijgyarto, Z., Bosch, D., Saiardi, A. Preparation of Quality Inositol Pyrophosphates. Jove-Journal of Visualized Experiments. (55), e3027 (2011).
- Harmel, R. K., et al. Harnessing C-13-labeled myo-inositol to interrogate inositol phosphate messengers by NMR Electronic supplementary information (ESI) available. Chemical Science. 10 (20), 5267-5274 (2019).
- Azevedo, C., Saiardi, A. Extraction and analysis of soluble inositol polyphosphates from yeast. Nature Protocols. 1 (5), 2416-2422 (2006).
- Shears, S. B., Miller, G. J. . Inositol Phosphates: Methods and Protocols. , 1-28 (2020).
- Wilson, M. S. C., Saiardi, A. Importance of Radioactive Labelling to Elucidate Inositol Polyphosphate Signalling. Topics in Current Chemistry. 375 (1), (2017).
- Stevenson-Paulik, J., et al. Inositol phosphate metabolomics: Merging genetic perturbation with modernized radiolabeling methods. Methods. 39 (2), 112-121 (2006).
- Liu, C., Riley, A. M., Yang, X., Shears, S. B., Potter, B. V. L. Synthesis and Biological Activity of d- and l-chiro-Inositol 2,3,4,5-Tetrakisphosphate: Design of a Novel and Potent Inhibitor of Ins(3,4,5,6)P4 1-Kinase/Ins(1,3,4)P3 5/6-Kinase. Journal of Medicinal Chemistry. 44 (18), 2984-2989 (2001).
- Hughes, P. J., Hughes, A. R., Putney, J. W., Shears, S. B. The regulation of the phosphorylation of inositol 1,3,4-trisphosphate in cell-free preparations and its relevance to the formation of inositol 1,3,4,6-tetrakisphosphate in agonist-stimulated rat parotid acinar cells. Journal of Biological Chemistry. 264 (33), 19871-19878 (1989).
- Shears, S. B., Kirk, C. J., Michell, R. H. The pathway of myo-inositol 1,3,4-trisphosphate dephosphorylation in liver. The Biochemical journal. 248 (3), 977-980 (1987).
- Stevenson-Paulik, J., Odom, A. R., York, J. D. Molecular and Biochemical Characterization of Two Plant Inositol Polyphosphate 6-/3-/5-Kinases. Journal of Biological Chemistry. 277 (45), 42711-42718 (2002).
- Stephens, L. R., Hawkins, P. T., Downes, C. P. An analysis of myo-[3H]inositol trisphosphates found in myo-[3H]inositol prelabelled avian erythrocytes. The Biochemical journal. 262 (3), 727-737 (1989).
- Saiardi, A., et al. Mammalian inositol polyphosphate multikinase synthesizes inositol 1,4,5-trisphosphate and an inositol pyrophosphate. Proceedings of the National Academy of Sciences. 98 (5), 2306 (2001).
- Azevedo, C., Burton, A., Bennett, M., Onnebo, S. M. N., Saiardi, A., Barker, C. J. . Inositol Phosphates and Lipids: Methods and Protocols. , 73-85 (2010).
- Saiardi, A., Caffrey, J. J., Snyder, S. H., Shears, S. B. The Inositol Hexakisphosphate Kinase Family: CATALYTIC FLEXIBILITY AND FUNCTION IN YEAST VACUOLE BIOGENESIS. Journal of Biological Chemistry. 275 (32), 24686-24692 (2000).
- Brearley, C. A., Hanke, D. E. Inositol phosphates in the duckweed Spirodela polyrhiza L. Biochemical Journal. 314, 215-225 (1996).
- Pollard, J. K., Steward, F. C., Shantz, E. M. Hexitols in Coconut Milk - Their Role in Nurture of Dividing Cells. Plant Physiology. 36 (4), 492 (1961).
- Donahue, J. L., et al. The Arabidopsis thaliana Myo-inositol 1-phosphate synthase1 gene is required for Myo-inositol synthesis and suppression of cell death. Plant Cell. 22 (3), 888-903 (2010).
- Desfougeres, Y., Wilson, M. S. C., Laha, D., Miller, G. J., Saiardi, A. ITPK1 mediates the lipid-independent synthesis of inositol phosphates controlled by metabolism. Proceedings of the National Academy of Sciences of the United States of America. 116 (49), 24551-24561 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon