Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis de tiempo de permanencia de una sola molécula de la escisión de ADN mediada por endonucleasas de restricción
En este artículo
Resumen
Utilizando ADN marcado con puntos cuánticos y microscopía de fluorescencia de reflexión interna total, podemos investigar el mecanismo de reacción de las endonucleasas de restricción mientras usamos proteínas no marcadas. Esta técnica de una sola molécula permite la observación masiva y multiplexada de las interacciones individuales proteína-ADN, y los datos se pueden agrupar para generar distribuciones de tiempo de permanencia bien pobladas.
Resumen
Este novedoso ensayo basado en microscopía de fluorescencia de reflexión interna total facilita la medición simultánea de la longitud del ciclo catalítico para cientos de moléculas individuales de endonucleasa de restricción (REasa) en un experimento. Este ensayo no requiere marcaje de proteínas y se puede llevar a cabo con un solo canal de imagen. Además, los resultados de varios experimentos individuales se pueden agrupar para generar distribuciones de tiempo de permanencia bien pobladas. El análisis de las distribuciones de tiempo de permanencia resultantes puede ayudar a dilucidar el mecanismo de escisión del ADN al revelar la presencia de pasos cinéticos que no se pueden observar directamente. Los datos de ejemplo recopilados utilizando este ensayo con la bien estudiada REase, EcoRV, una endonucleasa dimérica de restricción de tipo IIP que escinde la secuencia palindrómica GAT↓ATC (donde ↓ es el sitio de corte), concuerdan con estudios previos. Estos resultados sugieren que hay al menos tres pasos en el camino hacia la escisión del ADN que se inicia mediante la introducción de magnesio después de que EcoRV se une al ADN en su ausencia, con una tasa promedio de 0,17 s-1 para cada paso.
Introducción
Las endonucleasas de restricción (REasas) son enzimas que producen roturas de doble cadena específicas de secuencia en el ADN. El descubrimiento de las REasas en la década de 1970 condujo al desarrollo de la tecnología del ADN recombinante, y estas enzimas son ahora herramientas de laboratorio indispensables para la modificación y manipulación genética. Las REasas de tipo II son las enzimas más utilizadas en esta clase, ya que escinden el ADN en una ubicación fija, ya sea dentro o cerca de su secuencia de reconocimiento. Sin embargo, existe una gran variación entre las REasas de Tipo II, y se dividen en varios subtipos basados en propiedades enzimáticas particulares en lugar de clasificarse de acuerdo con sus relaciones evolutivas. Entre cada subtipo, hay excepciones frecuentes al esquema de clasificación, y muchas enzimas pertenecen a múltiples subtipos2. Se han identificado miles de RAases de tipo II, y cientos de ellas están disponibles comercialmente.
Sin embargo, a pesar de la diversidad entre las REasas de Tipo II, muy pocas REases han sido estudiadas en detalle. De acuerdo con REBASE, la base de datos de enzimas de restricción establecida por Sir Richard Roberts en 19753, los datos cinéticos publicados están disponibles para menos de 20 de estas enzimas. Además, mientras que algunas REasas se han observado directamente a nivel de una sola molécula mientras se difunden a lo largo del ADN antes de encontrarse y unirse a su secuencia de reconocimiento 4,5,6,7, hay muy pocos estudios de una sola molécula de su cinética de reacción de escisión. Los estudios existentes no reportan estadísticas adecuadas para llevar a cabo un análisis detallado de la variación en los momentos en que ocurren los eventos de escisión única 8,9,10 o no son capaces de capturar la distribución completa de los tiempos de escisión11. Este tipo de análisis puede revelar la presencia de intermediarios cinéticos de vida relativamente larga y podría conducir a una mejor comprensión de los mecanismos de la escisión del ADN mediada por REasa.
A nivel de una sola molécula, los procesos bioquímicos son estocásticos: el tiempo de espera para que ocurra una sola instancia del proceso, τ, es variable. Sin embargo, se puede esperar que muchas mediciones de τ obedezcan a una distribución de probabilidad, p(τ), que es indicativa del tipo de proceso que tiene lugar. Por ejemplo, un proceso de un solo paso, como la liberación de una molécula de producto a partir de una enzima, obedecerá a las estadísticas de Poisson, y p(τ) tomará la forma de una distribución exponencial negativa:
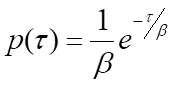
donde β es el tiempo medio de espera. Tenga en cuenta que la velocidad del proceso, k, será igual a 1/β, el inverso del tiempo medio de espera. Para los procesos que requieren más de un paso, p(τ) será la convolución de las distribuciones exponenciales simples para cada uno de los pasos individuales. Una solución general para la convolución de N funciones de decaimiento exponencial simple con tiempos de espera medios idénticos, β, es la distribución de probabilidad gamma:
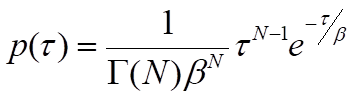
donde Γ(N) es la función gamma, que describe la interpolación del factorial de N-1 a valores no enteros de N. Aunque esta solución general se puede utilizar como una aproximación cuando los tiempos de espera medios de los pasos individuales son similares, debe entenderse que la presencia de pasos relativamente rápidos quedará enmascarada por pasos con tiempos de espera significativamente más largos. En otras palabras, el valor de N representa un límite inferior en el número de pasos12. Con un número adecuado de mediciones de tiempo de espera, los parámetros β y N se pueden estimar agrupando los eventos y ajustando la distribución gamma al histograma resultante o utilizando un enfoque de estimación de máxima verosimilitud. Por lo tanto, este tipo de análisis puede revelar la presencia de pasos cinéticos que no pueden resolverse fácilmente en ensayos de conjunto y requieren un gran número de observaciones para estimar los parámetros con precisión12,13.
Este artículo describe un método para utilizar el ADN marcado con puntos cuánticos y la microscopía de fluorescencia de reflexión interna total (TIRF) para observar cientos de eventos individuales de escisión de ADN mediados por REasa en paralelo. El diseño del ensayo permite agrupar los resultados de varios experimentos y puede crear distribuciones de tiempo de permanencia que contienen miles de eventos. La alta fotoestabilidad y el brillo de los puntos cuánticos permiten una resolución de tiempo de 10 Hz sin sacrificar la capacidad de observar los eventos de escisión que ocurren incluso muchos minutos después del inicio del experimento. La buena resolución temporal y un amplio rango dinámico, combinados con la capacidad de recopilar un gran conjunto de datos, permiten una caracterización precisa de las distribuciones de tiempo de permanencia para descubrir la presencia de múltiples pasos cinéticos en las vías de escisión de las REasas, que tienen tasas de recambio en el rango de 1 min-1 . En el caso de EcoRV, se pueden resolver tres pasos cinéticos, todos los cuales han sido identificados a través de otros medios, lo que confirma que el ensayo es sensible a la presencia de dichos pasos.
Los sustratos de ADN dúplex que contienen la secuencia de reconocimiento de interés se producen mediante el recocido de un oligonucleótido biotinilado en una hebra complementaria marcada con un único punto cuántico de nanocristal semiconductor unido covalentemente. Estos sustratos se introducen en un canal de flujo construido sobre un cubreobjetos de vidrio con un césped de moléculas de polietilenglicol (PEG) de alto peso molecular unidas covalentemente a su superficie. Los sustratos de ADN son capturados a través de un enlace biotina-estreptavidina-biotina por una fracción de las moléculas de PEG que tienen una biotina en su extremo libre. En la microscopía TIRF, una onda evanescente que decae exponencialmente con la distancia de la interfaz vidrio-líquido proporciona iluminación; La profundidad de penetración es del orden de la longitud de onda de la luz utilizada. En estas condiciones, solo se excitarán los puntos cuánticos que están atados a la superficie por una molécula de ADN que ha sido capturada en la superficie de vidrio funcionalizada. Los puntos cuánticos que están libres en solución no estarán restringidos dentro de la región iluminada y, por lo tanto, no luminiscentes. Si el ADN que ata un punto cuántico a la superficie se escinde, ese punto cuántico será libre de difundirse lejos de la superficie y desaparecerá de la imagen de fluorescencia.
Aunque se sabe que muchas REasas de tipo II se unen al ADN en ausencia de magnesio14, todas requieren magnesio para mediar la escisión del ADN15. Estas resinas pueden unirse al ADN inmovilizado en la superficie en ausencia de magnesio. Cuando el tampón que contiene magnesio fluye a través de un canal con REasa preunida al ADN, la escisión comienza inmediatamente, como lo indica la desaparición de los puntos cuánticos. La sincronización lograda mediante la preunión de las moléculas de REasa, y luego iniciando la escisión del ADN mediante la introducción de magnesio, facilita la medición del tiempo de retraso hasta la finalización de la escisión del ADN de forma independiente para cada molécula en la población de enzimas observadas en un experimento. La fluoresceína se incluye como colorante trazador en el tampón que contiene magnesio para indicar la llegada de magnesio al campo de visión. Como no se incluye ninguna enzima en el tampón que contiene magnesio, el tiempo de retraso desde la llegada del tampón que contiene magnesio hasta la desaparición de cada punto cuántico indica el tiempo que tarda una REasa que ya está unida al ADN en escindir el ADN y liberar el punto cuántico de la superficie del vidrio. La desaparición de los puntos cuánticos ocurre rápidamente y da lugar a una fuerte disminución de la trayectoria de intensidad, lo que proporciona una indicación clara del momento en el que se escinde una determinada molécula de ADN. La determinación de los tiempos de los eventos se logra mediante el análisis matemático de las trayectorias de intensidad, y un experimento típico da como resultado cientos de eventos de escisión identificables. Los resultados de múltiples experimentos se pueden agrupar para proporcionar estadísticas adecuadas que permitan la estimación de los parámetros, N y β, mediante análisis de mínimos cuadrados no lineales o de máxima verosimilitud.
Protocolo
1. Información general
- Diseño de oligonucleótidos
NOTA: El sustrato de ADN de 60 pares de bases (pb) de largo se forma a partir de un par de oligonucleótidos complementarios con una temperatura de fusión dúplex de 75 °C en 100 mM de NaCl.- Pida un oligonucleótido sintetizado con una sola modificación de biotina 5' y el otro con una modificación de tiol 5' (con un espaciador de seis carbonos). Coloque el sitio de reconocimiento en el centro de la región dúplex.
NOTA: Las secuencias de oligonucleótidos para su uso con EcoRV se muestran a continuación (sitio de reconocimiento en negrita).
5' biotina - AAA ACC GAC ATG TTG ATT TCC TGA AAC GGG ATA TCA TCA AAG CCA TGA ACA AAG CAG CCG - 3'
5' thiol - CGG CTG CTT TGT TCA TGG CTT TGA TGA TAT CCC GTT TCA GGA AAT CAA CAT GTC GGT TTT - 3'
- Pida un oligonucleótido sintetizado con una sola modificación de biotina 5' y el otro con una modificación de tiol 5' (con un espaciador de seis carbonos). Coloque el sitio de reconocimiento en el centro de la región dúplex.
- Utilice agua ultrapura con una resistividad de 18 MOhm para todos los pasos.
- Proteja todas las soluciones que contengan puntos cuánticos de la luz para evitar el fotoblanqueo.
- Utilice una fuente de aire comprimido para completar este protocolo.
2. Preparación de materiales de sustrato de ADN marcados con puntos cuánticos
NOTA: Además de los oligonucleótidos descritos anteriormente, consulte la Tabla de Materiales para otros materiales y la Tabla 1 para conocer los tampones necesarios para la preparación de sustratos de ADN marcados con puntos cuánticos.
- Reducir los grupos tiol 5' en el oligonucleótido para acoplarlos a puntos cuánticos.
- Resuspender cada oligonucleótido tiolado en agua a una concentración de 100 μM.
- Pipetear 650 μL de agua en una columna de espín de exclusión de tamaño para cada muestra de oligonucleótidos y vórtice durante ~15 s. Deje que la columna se empaque durante 30 min.
- Prepare una nueva solución de 100 mM de ditiotritol (DTT) inmediatamente antes de cada uso, ya que el DTT se degrada rápidamente en la solución. Abra cuidadosamente un vial que contenga 7,7 mg de DTT y pipetee 500 μL de tampón de fosfato de sodio en el vial; vórtice para mezclar bien.
- Para cada oligonucleótido resuspendido, prepare un tubo nuevo y agregue 50 μL del oligonucleótido y 50 μL de solución de DTT al tubo. Pipetear hacia arriba y hacia abajo para mezclar. Incubar durante 30 minutos a temperatura ambiente para reducir los enlaces disulfuro entre los grupos tiol oxidados en el extremo 5' de los oligonucleótidos.
- Retire las tapas de las columnas de centrifugado y coloque cada columna en el tubo de recolección. Centrifugar las columnas de centrifugación a 750 × g durante 2 min y desechar el eluido.
- Transfiera las columnas de centrifugación a tubos de centrífuga nuevos. Pipetear suavemente todo el volumen (100 μL) de una muestra de mezcla de ADN/DTT en cada columna preparada. Centrifugar a 750 × g durante 2 min y medir la absorbancia del eluido a 260 nm para confirmar que la concentración es de aproximadamente 40 μM.
- Almacene las muestras que no se vayan a utilizar inmediatamente a -20 °C para evitar la oxidación de los grupos tiol y la formación de enlaces disulfuro.
- Acoplamiento del ADN a los puntos cuánticos
- Pipetear una alícuota de 50 μL del material de puntos cuánticos en un dispositivo de diálisis para cada construcción que se vaya a fabricar, asegurándose de no tocar la membrana con la punta de la pipeta. Diálisar contra un volumen de tampón de ácido N-ciclohexil-2-aminoetanosulfónico (CHES) que sea al menos 1000 veces el volumen de la muestra agitando a ~ 100 rpm durante 15 min.
- Prepare una solución fresca de 6 mM de sulfosuccinimidil-4-(N-maleimidometil) ciclohexano-1-carboxilato (sulfo-SMCC) en tampón CHES inmediatamente antes de cada uso. Abra cuidadosamente un vial que contenga 2 mg de sulfo-SMCC y pipetee 800 μL de tampón CHES en el vial. Vórtice para mezclar bien.
- Con una pipeta, recupere cuidadosamente los puntos cuánticos suspendidos del dispositivo de diálisis y transfiéralos a un tubo nuevo que contenga un volumen igual de la solución de sulfo-SMCC; pipetear hacia arriba y hacia abajo para mezclar. Incubar durante 1 h a temperatura ambiente con agitación a 1000 rpm para permitir que el sulfo-SMCC reaccione con las aminas primarias en los puntos cuánticos.
- Con una pipeta, transfiera cuidadosamente cada muestra a un dispositivo de diálisis nuevo. Para eliminar el exceso de sulfo-SMCC, dialice contra un volumen de tampón CHES que sea al menos 1000 veces el volumen contenido en los dispositivos de diálisis, con agitación, durante 15 min. Cambie el tampón 2 veces y realice la diálisis con tampón CHES nuevo 3 veces en total, lo que permite que la diálisis continúe 15 minutos después de cada cambio de tampón.
- Para intercambiar el tampón en preparación para la segunda reacción, transfiera los dispositivos de diálisis a un vaso de precipitados que contenga un volumen de solución salina tamponada con fosfato (PBS) que sea al menos 1000 veces el volumen contenido en los dispositivos. Diálisis con agitación durante 15 minutos, cambie el tampón 2 veces y realice la diálisis con PBS fresco 3 veces en total, permitiendo que la diálisis continúe durante 15 minutos después de cada cambio de tampón.
- Con una pipeta, recupere cuidadosamente la solución que contiene los puntos cuánticos de los dispositivos de diálisis y transfiera cada muestra a un tubo nuevo que contenga una cantidad equimolar de oligonucleótido tiolado diluido en PBS. Combine un volumen igual de PBS y 1/10del volumen de la muestra de oligonucleótidos reducida y purificada (aproximadamente 40 μM de concentración) ya que la concentración de puntos cuánticos en este punto es de ~4 μM. Incubar durante 2 h a temperatura ambiente, con agitación a 1000 rpm.
- Añadir albúmina sérica bovina (BSA, 10 mg/mL) a cada muestra para obtener una concentración final de BSA de 0,5 mg/mL. Con una pipeta, transfiera cuidadosamente cada muestra a un dispositivo de diálisis nuevo y dialice contra un volumen de tampón de almacenamiento que sea al menos 1000 veces el volumen contenido en los dispositivos de diálisis. Cambie el tampón 2 veces y realice la diálisis con un tampón de almacenamiento fresco 3 veces en total, lo que permite que la diálisis continúe durante 15 minutos después de cada cambio de tampón.
- Con una pipeta, recupere cuidadosamente cada muestra, colóquela en un tubo nuevo y guárdela a 4 °C.
NOTA: No lo almacene a -20 °C, ya que los puntos cuánticos se dañan por la congelación.
- Recocido de oligonucleótido marcado con puntos cuánticos a oligonucleótido biotinilado
- Vuelva a suspender cada oligonucleótido biotinilado en un tampón de almacenamiento a una concentración de 100 μM. Almacene las muestras no utilizadas a -20 °C.
- Combine una muestra de oligonucleótido marcado con puntos cuánticos con un exceso molar de 10 veces de oligonucleótido biotinilado. Como la concentración estimada de ADN en la muestra marcada con puntos cuánticos es de 2 μM, agregue 0,2 μL de oligonucleótido biotinilado por cada 10 μL de esta muestra.
- Calentar la mezcla en un bloque de calor a 75 °C (la temperatura de fusión para la región complementaria). Mantenga a esa temperatura durante 5 minutos. Apague el bloque de calor y deje que se enfríe lentamente. Almacene la construcción terminada a 4 °C; No congelar.
3. Funcionalización de la superficie de los cubreobjetos
NOTA: Este proceso ha sido descrito previamente en otros protocolos de video JoVE 16,17. Este protocolo describe una versión adaptada del procedimiento con cambios menores para acomodar un portaobjetos de vidrio más pequeño. Consulte la Tabla de materiales para conocer otros materiales necesarios para la funcionalización de la superficie de los cubreobjetos.
- Coloque 5 cubreobjetos en cada soporte. Asegúrate de saltarte un espacio entre cada par de cubreobjetos para que no se peguen entre sí. Coloque los cubreobjetos en su soporte en un frasco con tapa, agregue etanol al frasco hasta que los cubreobjetos estén cubiertos y atornille la tapa de forma segura. Coloque todo el frasco en el agua en el sonicador de baño, sin sumergirlo por completo, y sonique durante 30 min.
- Llene un vaso o frasco limpio con agua ultrapura. Use pinzas de metal para quitar los cubreobjetos de su soporte del etanol y sumérjalos en agua para enjuagar. Luego, transfiera los cubreobjetos y el soporte a un frasco que contenga una solución de hidróxido de potasio (KOH) de 1 M. Enrosque bien la tapa, coloque el frasco en el baño del sonicador y sonique durante 30 minutos.
NOTA: Tenga cuidado al manipular la solución de KOH, ya que es corrosiva e irritante. - Repita la sonicación en una solución de etanol y KOH, enjuagando los cubreobjetos con agua como se ha descrito anteriormente entre cada paso.
- Transfiera los cubreobjetos en su soporte a un frasco limpio lleno de acetona pura. Termine la limpieza sonicando el frasco como se ha descrito anteriormente durante 30 minutos. No enjuague con agua después de este paso.
- Con unas pinzas metálicas, transfiera los cubreobjetos de su soporte a un vaso de precipitados limpio de 100 ml que contenga 80 ml de acetona fresca y una barra agitadora a microescala. Coloque el vaso de precipitados en una placa de agitación magnética ajustada a al menos 1.000 rpm. Mientras se agita la acetona vigorosamente, pipetee 1,6 ml de 3-aminopropiltrietoxisilano (APTES) en el vaso de precipitados para hacer una solución al 2% v/v.
NOTA: Tenga cuidado al pipetear APTES, ya que es corrosivo. - Deje que los cubreobjetos se incuben en la solución durante 2 minutos, luego use pinzas de metal para transferir los cubreobjetos en su soporte a un vaso de precipitados con agua para apagar la reacción. Enjuague los cubreobjetos dos veces más, reemplazando el agua en el vaso de precipitados.
- Cura el vidrio silanizado en el horno a 120 °C durante 75 min. Si no continúa con el siguiente paso inmediatamente, guarde los cubreobjetos al vacío durante un máximo de unos días.
- Disuelva los polietilenglicoles (PEG) derivados del éster de N-hidroxisuccinimida (NHS) en 100 mM de bicarbonato de sodio (pH 8,2). Utilice una proporción de 10:1 de PEG terminado en metoxi y PEG terminado en biotina, con ~100 mg/mL de PEG terminado en metoxi.
- Pipetear 100 μL de la solución de PEG en la mitad de los cubreobjetos silanizados secos y cubrir cada uno con un segundo cubreobjetos. Use pequeños pedazos de parafilm colocados en cada esquina como espaciadores para evitar que los cubreobjetos se peguen entre sí.
- Incubar los cubreobjetos en un ambiente húmedo durante 3,5 h. Separe los sándwiches de cubreobjetos. Use una botella rociadora para lavar cada cubreobjetos con abundante agua y secar con aire comprimido.
- Guarde los cubreobjetos funcionalizados al vacío. Asegúrese de mantener el lado tratado con PEG hacia arriba porque el otro lado no capturará el sustrato de ADN.
4. Construcción de dispositivos microfluídicos
NOTA: Consulte la Tabla de materiales para conocer otros materiales necesarios para la construcción del dispositivo microfluídico.
- Usando una multiherramienta rotativa de mano equipada con una broca de rueda cónica con punta de diamante, taladre dos orificios en las esquinas opuestas del portaobjetos de cuarzo para que sirvan como entrada y salida. Asegúrese de asegurar la corredera en su lugar, lubricar la broca y deslizarla constantemente con agua mientras perfora. Después de cada experimento, recupere el portaobjetos de cuarzo preparado para su reutilización empapando un dispositivo usado en acetona para disolver el adhesivo y el epoxi; Deseche los componentes restantes.
NOTA: La perforación se puede realizar a mano, pero el uso de un soporte tipo prensa para la multiherramienta facilita el proceso. - Combine 25 μL de solución de estreptavidina con 80 μL de PBS y 20 μL de tampón de bloqueo. Cubra un cubreobjetos tratado con PEG con esta mezcla e incube en un ambiente húmedo durante 30 minutos.
- Durante la incubación del cubreobjetos, prepare el espaciador de imágenes para crear un canal de flujo. Corte una pieza cuadrada de 1 pulgada de material espaciador de imágenes, luego marque un canal de 2 mm de ancho alineado con los extremos de los orificios perforados en el portaobjetos de cuarzo (Figura 2). Corta el canal del material espaciador con un bisturí.
- Despegue un lado del respaldo del espaciador de imagen y colóquelo con cuidado en la corredera de cuarzo, teniendo cuidado de no cubrir los orificios de entrada y salida. Asegúrese de limpiar bien el portaobjetos de cuarzo con acetona para eliminar cualquier resto de adhesivo de experimentos anteriores.
- Lave el cubreobjetos con agua y séquelo con aire comprimido. Retire el otro lado del respaldo del espaciador de imágenes y colóquelo entre el portaobjetos de cuarzo y el cubreobjetos funcionalizado recubierto de estreptavidina, utilizando las pinzas de plástico para presionar el conjunto y eliminar las burbujas de aire del adhesivo.
- Inserte un tubo de polietileno de 30 cm de largo en cada orificio del portaobjetos de cuarzo. Asegúrese de cortar los extremos del tubo en ángulo para garantizar el libre flujo de la solución. Use una rejilla para tubos u otro soporte para mantener los tubos en su lugar y selle los tubos en su lugar y los bordes del dispositivo ensamblado con epoxi.
NOTA: Funciona bien construir el dispositivo sobre un trozo de parapelícula, que se puede despegar fácilmente de la parte inferior cuando el epoxi se ha endurecido. - Una vez que el epoxi se haya endurecido, inserte la aguja roma en la jeringa vacía en el tubo de salida del dispositivo y sumerja el extremo del tubo de entrada en un recipiente lleno de tampón de bloqueo. Tire hacia atrás del émbolo de la jeringa para llenar el dispositivo con tampón de bloqueo. Deje incubar el dispositivo durante al menos 30 minutos antes de usarlo.
5. Anclaje superficial de sustrato de ADN marcado con puntos cuánticos
NOTA: Además del dispositivo microfluídico, el sustrato de ADN y el tampón de bloqueo descritos anteriormente, consulte la Tabla de materiales para otros materiales y la Tabla 1 para conocer los tampones necesarios para el anclaje de superficie de sustratos de ADN marcados con puntos cuánticos.
- Fije el dispositivo microfluídico a la placa de la platina del microscopio con cinta adhesiva, ponga el objetivo en contacto con la parte inferior del dispositivo y coloque el objetivo de modo que el campo de visión esté dentro del canal microfluídico.
- Enjuague el canal microfluídico con un tampón de bloqueo nuevo tirando hacia atrás del émbolo de la jeringa después de conectar el tubo de salida a la bomba de jeringa. Verifique que no haya burbujas atrapadas en el tubo o en el canal.
NOTA: A partir de este punto, asegúrese de que el tubo de entrada esté en líquido para que no se introduzcan burbujas de aire en el dispositivo. - Diluir 1 μL del sustrato de ADN preparado en 1 mL de tampón de bloqueo. Coloque el tubo de entrada en el sustrato de ADN diluido y haga fluir 800 μL de la solución de sustrato a través del canal a una velocidad de 200 μL/min. Deje que la solución de ADN se incube en el canal sin ser molestada durante 15 minutos después de que se detenga el flujo.
- Fluya al menos 800 μL de tampón de bloqueo a través del canal a una velocidad de 200 μL/min para enjuagar el ADN no unido fuera del canal.
- Ajuste la potencia del láser, el enfoque del microscopio y el ángulo TIRF para que los puntos cuánticos atados a la superficie sean claramente visibles.
NOTA: Aunque los puntos cuánticos no se blanquean rápidamente, es mejor mantener la potencia lo más baja posible para minimizar el parpadeo.
6. Escisión de ADN mediada por REasa
NOTA: Consulte la Tabla de Materiales para los materiales y la Tabla 1 para conocer los tampones necesarios para la escisión del ADN mediada por REasa.
- Asegúrese de que la cámara esté refrigerada a la temperatura óptima de funcionamiento y configurada para la transmisión a alta velocidad con un tiempo de exposición de 0,10 s. Esté preparado para recopilar datos durante ~ 4 minutos.
- Enjuague el dispositivo microfluídico con 800 μL de tampón experimental sin magnesio a un caudal de 200 μL/min.
- Añadir la REase a una alícuota (1 mL) de tampón experimental sin magnesio y mezclar suavemente mediante pipeteo. Utilice 4 μL de 100.000 unidades/mL de stock, lo que corresponde a 400 unidades/mL de EcoRV. Caudal de 800 μL de la enzima diluida a través del canal a un caudal de 200 μL/min.
- Comience el experimento haciendo fluir un tampón experimental que contenga magnesio y fluoresceína a un caudal de 200 μL/min. Comience a capturar datos inmediatamente después de poner en marcha la bomba de jeringa. Después de fluir 800 μL de búfer, detenga la adquisición de datos.
7. Análisis de datos
NOTA: Consulte la Tabla de materiales para conocer el software de análisis de datos utilizado para este protocolo y realice ajustes si utiliza una plataforma de análisis diferente.
- Determinación de punto de tiempo cero
- Reste la fluorescencia de fondo de cada fotograma de la película experimental utilizando la función imtophat , una función de filtrado morfológico integrada en Image Processing Toolbox. Seleccione un disco con un radio de tres píxeles como elemento estructurante. Obtenga la intensidad del fondo restando la imagen filtrada de la imagen original.
NOTA: El filtro solo conserva las características de la imagen más brillantes que el fondo y más pequeñas que el elemento estructurador. - Calcula el promedio del fondo restado en cada fotograma de película. Utilice una trayectoria de este valor para determinar el punto de tiempo cero para el experimento (Figura 3A). Determine la hora de inicio encontrando el primer fotograma en el que la tasa de aumento supera 3 veces la desviación estándar de la tasa de cambio durante el flujo de volumen muerto.
NOTA: Un aumento brusco en la tasa de cambio del valor promedio del fondo sustraído para cada fotograma de película indica el momento en que el tampón experimental final que contiene el colorante trazador ingresó a la celda de flujo (Figura 3B).
- Reste la fluorescencia de fondo de cada fotograma de la película experimental utilizando la función imtophat , una función de filtrado morfológico integrada en Image Processing Toolbox. Seleccione un disco con un radio de tres píxeles como elemento estructurante. Obtenga la intensidad del fondo restando la imagen filtrada de la imagen original.
- Cálculo y análisis de la trayectoria de la intensidad de los puntos cuánticos
- Calcule una proyección de intensidad máxima para el conjunto de fotogramas de película corregidos en el fondo grabados antes de la llegada del colorante trazador. Determine las ubicaciones de los puntos cuánticos con una precisión de un píxel en esta imagen de proyección utilizando una función de búsqueda de picos como pkfnd.
- Genere trayectorias de intensidad para puntos cuánticos individuales calculando la intensidad media en cada fotograma de película de una región cuadrada de tres píxeles por lado que rodea la ubicación devuelta por la función de búsqueda de picos.
NOTA: La corrección de fondo descrita anteriormente elimina los artefactos introducidos por el tinte trazador. Las trayectorias de intensidad resultantes deben ser relativamente planas, pero aun así incluir fluctuaciones de intensidad que ocurren naturalmente (Figura 4). - Identifique los eventos de desaparición de puntos cuánticos mediante el análisis estadístico de las trayectorias de intensidad. Para cada trayectoria de intensidad de punto cuántico, calcule una intensidad umbral que esté por encima del valor mínimo en una fracción apropiada de la diferencia entre los valores máximo y mínimo para esa trayectoria, dependiendo del ruido observado en las trayectorias.
NOTA: La fluctuación de intensidad del punto cuántico suele ser mucho mayor que la fluctuación de fondo. Un valor apropiado se puede determinar comparando estos valores: el umbral seleccionado debe ser mayor que el valor de fondo más alto, pero idealmente debe ser más bajo que el valor más bajo para la fluorescencia de puntos cuánticos. Para los datos presentados, se calculó que el umbral utilizado estaba por encima del valor mínimo para cada trayectoria más un tercio de la diferencia entre los valores máximo y mínimo para esa trayectoria. Se puede considerar que un punto cuántico desaparece en el último punto de tiempo en que la intensidad observada en su ubicación estaba por encima de este umbral. - Confirmar eventos de desaparición putativos. Solo incluya los eventos en el análisis final si la intensidad observada está por encima del punto medio entre los valores de intensidad mínima y máxima para la trayectoria de más de la mitad de los fotogramas de la película antes del momento de la desaparición y la desviación estándar de la trayectoria de intensidad disminuyó después del evento.
NOTA: Se pueden emplear otras pruebas estadísticas para garantizar que solo se registren los eventos de desaparición válidos.
Resultados
La celda de flujo está acoplada directamente a un objetivo de aumento de 60x de inmersión en aceite de alta apertura numérica en un microscopio invertido equipado con iluminación láser para imágenes TIRF a través del objetivo (Figura 5A). Después de introducir el sustrato de ADN y lavar el exceso de ADN y puntos cuánticos, normalmente hay miles de puntos cuánticos individuales en un campo de visión (Figura 5B). Estos puntos cuánticos están unidos de...
Discusión
El sustrato de ADN para este ensayo se marca con un punto cuántico utilizando un esquema de reacción de dos pasos utilizando sulfo-SMCC. Este reticulante bifuncional consta de una fracción de éster del NHS que puede reaccionar con una amina primaria y una fracción de maleimida que puede reaccionar con un grupo sulfhidrilo20. Los oligonucleótidos tiolados utilizados para preparar el sustrato se envían en su forma oxidada. Es importante reducirlos y purificarlos, como se describe, antes de co...
Divulgaciones
Los autores no tienen intereses financieros contrapuestos ni otros conflictos de intereses
Agradecimientos
Este trabajo fue apoyado por el Premio Número K12GM074869 a CME del Instituto Nacional de Ciencias Médicas Generales. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente los puntos de vista oficiales del Instituto Nacional de Ciencias Médicas Generales o los Institutos Nacionales de Salud.
Materiales
| Name | Company | Catalog Number | Comments |
| 3-aminopropyltriethoxysilane (APTS) | Sigma Aldrich | 440140-100ML | Store in dessicator box |
| 5 Minute Epoxy | Devcon | 20845 | use for sealing the microfluidic device |
| acetone | Pharmco | 329000ACS | use for cleaning coverslips |
| bath sonicator | Fisher Scientific | CPXH Model 2800 | catalog number 15-337-410 |
| Beaker, glass, 100 mL | |||
| Benchtop centrifuge | |||
| Biotin-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | BIO-SVA-5K | Succinimidyl valerate has a longer half-life than succinimidyl carbonate |
| biotinylated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Biotin modification and HPLC purification |
| Bovine Serum Albumin (BSA) | VWR | 0903-5G | prepare a 10 mg/mL solution (aq) and heat to 95 °C before using |
| Centri-Spin-10 Size Exclusion Spin Columns | Princeton Separations | CS-100 or CS-101 | used to purify thiolated oligonucleotides after reducing the disulfide bond |
| Centrifuge tubes 1.5. mL | |||
| Coverslips, 1-inch square glass | |||
| coverslip holders | |||
| diamond point wheel | Dremel | 7134 | use for drilling holes in quartz flow cell topper |
| dithiothreitol (DTT) | Thermo Scientific | A39255 | No-Weigh Format, 7.7 mg/vial |
| drill press rotary tool workstation stand | Dremel | 220-01 | facilitates quartz drilling |
| EcoRV (REase used to generate example data) | New England Biolabs | R0195T or R0195M | Use 100,000 units/mL stock to avoid adding excess glycerol Check REBASE for suppliers of other REases |
| ethanol | various | CAS 64-17-5 | denatured or 95% are acceptable, use for cleaning coverslips |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | EDS | BioUltra, anhydrous, store in dessicator box |
| Flea Micro Spinbar | Fisherbrand | 14-513-65 | 3 mm x 10 mm size to fit beneath coverslip rack |
| fluorescein | Acros Organics | 17324 | use to make experimental buffers |
| gravity convection oven | Binder | 9010-0131 | |
| handheld rotary multitool | Dremel | 8220 | use for drilling holes in quartz flow cell topper |
| ImagEM X2 EM-CCD Camera | Hamamatsu | C9100-23B | air cooling is adequate for this experiment, use HCImage software or similar to control |
| Imaging spacer, double-sided, adhesive | |||
| Jar, glass with screw cap, (approximately 50 mm diameter by 50 mm high) | |||
| magnesium chloride hexahydrate | Fisher Bioreagents | BP214-500 | use to make experimental buffer with magnesium |
| MATLAB software | Data analysis | ||
| metal tweezers | Fisher Brand | 16-100-110 | |
| methoxy-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | M-SVA-5K | Both PEGs should have the same NHS ester so that the rate of reaction is consistent |
| microcentrifuge | Eppendorf | 5424 | |
| multiposition magnetic stirrer | VWR | 12621-022 | |
| N-cyclohexyl-2-aminoethanesulfonic acid (CHES) | Acros Organics | AC20818 | CAS 103-47-9, use to make CHES buffer |
| orbital shaker and heater for microcentrifuge tubes | Q Instruments | 1808-0506 | with 1808-1061 adaptor for 24 x 2.0 mL or 15 x 0.5 mL tubes |
| Parafilm | |||
| PE60 Polyethylene tubing (inner diameter 0.76 mm, outer diameter 1.22 mm) | Intramedic | 6258917 | 22 G blunt needles are a good fit for this tubing size |
| Phosphate-Buffered Saline (PBS) 10x | Sigma Aldrich | P7059 | Use at 1x strength |
| potassium hydroxide | VWR Chemicals BDH | BDH9262 | use a 1 M solution to clean coverslips |
| Qdot 655 ITK Amino (PEG) Quantum Dots | Invitrogen | Q21521MP | |
| Quartz Slide, 1 inch square, 1 mm thick | Electron Microscopy Sciences | 72250-10 | holes must be drilled in the corners for inlet and outlet tubing insertion |
| reinforced plastic tweezers | Rubis | K35a | use for handling coverslips and building microfluidic device |
| SecureSeal Adhesive Sheets | Grace Biolabs | SA-S-1L | cut to form spacer for microfluidic device |
| Single channel syringe pump for microfluidics | New Era Pump Systems | NE-1002X-US | fitted with a 50 mL syringe and a 22 G blunt needle |
| Slide-a-Lyzer MINI Dialysis Devices, 10 kDa MWCO, 0.1 mL | Thermo Scientific | 69570 or 69572 | used for buffer exchange during quantum dot coupling to DNA |
| sodium bicarbonate | EMD Millipore | SX0320 | use to make buffer for surface functionalization; 100 mM, pH 8 |
| sodium chloride | Macron | 7581-12 | use to make experimental buffers |
| Sodium phosphate dibasic solution (BioUltra, 0.5 M in water) | Sigma Aldrich | 94046 | use to make 100 mM sodium phosphate buffer |
| Sodium phosphate monobasic solution (BioUltra, 5M in water) | Sigma Aldrich | 74092 | use to adjust pH of 100 mM sodium phosphate buffer |
| Streptavidin from Streptomyces avidinii | Sigma Aldrich | S4762 | dissolve at 1 mg/mL and store 25 mL aliqouts at -20 ? |
| Sulfosuccinimidyl-4-(N-maleimidomethyl) cyclohexane-1-carboxylate (sulfo-SMCC) | Thermo Scientific | A39268 | No-Weigh Format, 2 mg/vial |
| Syringe fitted with blunt 21 G needle | |||
| Syringe pump | |||
| thiolated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Thiol Modifier C6 S-S and HPLC purificaiton |
| TIRF imaging system with 488 nm laser illumination | various | custom built | |
| Tris -HCl | Research Products International | T60050 | use to make experimental buffers |
| Tris base | JT Baker | 4101 | use to make experimental buffers |
| Tween-20 | Sigma | P7949 | use to make blocking buffer |
| Ultrapure water | |||
| vortex mixer | VWR | 10153-842 | |
| Wash-N-Dry Coverslip Rack | Electron Microscopy Sciences | 70366-16 | used for surface functionalization of coverslips |
Referencias
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5905-5908 (2005).
- Loenen, W. A., Dryden, D. T., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Research. 42 (1), 3-19 (2014).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE--a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Research. 43, 298-299 (2015).
- Bonnet, I., et al. Sliding and jumping of single EcoRV restriction enzymes on non-cognate DNA. Nucleic Acids Research. 36 (12), 4118-4127 (2008).
- Biebricher, A., Wende, W., Escude, C., Pingoud, A., Desbiolles, P. Tracking of single quantum dot labeled EcoRV sliding along DNA manipulated by double optical tweezers. Biophysical Journal. 96 (8), 50-52 (2009).
- Blainey, P. C., et al. Nonspecifically bound proteins spin while diffusing along DNA. Nature Structural & Molecular Biology. 16 (12), 1224-1229 (2009).
- Huang, C. -. F., et al. Direct visualization of DNA recognition by restriction endonuclease EcoRI. Journal of Experimental & Clinical Medicine. 5 (1), 25-29 (2013).
- Palma, M., et al. Selective biomolecular nanoarrays for parallel single-molecule investigations. Journal of American Chemical Society. 133 (20), 7656-7659 (2011).
- Reinhard, B. M., Sheikholeslami, S., Mastroianni, A., Alivisatos, A. P., Liphardt, J. Use of plasmon coupling to reveal the dynamics of DNA bending and cleavage by single EcoRV restriction enzymes. Proceedings of the National Academy of Sciences of the United States of America. 104 (8), 2667-2672 (2007).
- van den Broek, B., Noom, M. C., Wuite, G. J. DNA-tension dependence of restriction enzyme activity reveals mechanochemical properties of the reaction pathway. Nucleic Acids Research. 33 (8), 2676-2684 (2005).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Loparo, J. J., van Oijen, A., Hinterdorfer, P., Oijen, A. Single-molecule enzymology. Handbook of Single-Molecule Biophysics. , 165-182 (2009).
- Taylor, J. D., Badcoe, I. G., Clarke, A. R., Halford, S. E. EcoRV restriction endonuclease binds all DNA sequences with equal affinity. Biochemistry. 30 (36), 8743-8753 (1991).
- Pingoud, A., Jeltsch, A. Structure and function of type II restriction endonucleases. Nucleic Acids Research. 29 (18), 3705-3727 (2001).
- Tanner, N. A., Loparo, J. J., van Oijen, A. M. Visualizing single-molecule DNA replication with fluorescence microscopy. Journal of Visualized Experiments: JoVE. (32), e1529 (2009).
- Kulczyk, A. W., Tanner, N. A., Loparo, J. J., Richardson, C. C., van Oijen, A. M. Direct observation of enzymes replicating DNA using a single-molecule DNA stretching assay. Journal of Visualized Experiments: JoVE. (37), e1689 (2010).
- Baldwin, G. S., Vipond, I. B., Halford, S. E. Rapid reaction analysis of the catalytic cycle of the EcoRV restriction endonuclease. Biochemistry. 34 (2), 705-714 (1995).
- Winkler, F. K., et al. The crystal-structure of Ecorv endonuclease and of Its complexes with cognate and non-cognate DNA fragments. EMBO Journal. 12 (5), 1781-1795 (1993).
- Sioss, J. A., Stoermer, R. L., Sha, M. Y., Keating, C. D. Silica-coated, Au/Ag striped nanowires for bioanalysis. Langmuir. 23 (22), 11334-11341 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados