É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise de tempo de permanência de molécula única de clivagem de DNA mediada por endonuclease de restrição
Neste Artigo
Resumo
Usando DNA marcado com ponto quântico e microscopia de fluorescência de reflexão interna total, podemos investigar o mecanismo de reação das endonucleases de restrição ao usar proteína não marcada. Essa técnica de molécula única permite a observação massivamente multiplexada de interações proteína-DNA individuais, e os dados podem ser agrupados para gerar distribuições de tempo de permanência bem povoadas.
Resumo
Este novo ensaio baseado em microscopia de fluorescência de reflexão interna total facilita a medição simultânea da duração do ciclo catalítico para centenas de moléculas individuais de endonuclease de restrição (REase) em um experimento. Este ensaio não requer marcação de proteínas e pode ser realizado com um único canal de imagem. Além disso, os resultados de vários experimentos individuais podem ser agrupados para gerar distribuições de tempo de permanência bem preenchidas. A análise das distribuições de tempo de permanência resultantes pode ajudar a elucidar o mecanismo de clivagem do DNA, revelando a presença de etapas cinéticas que não podem ser observadas diretamente. Exemplos de dados coletados usando este ensaio com a bem estudada REase, EcoRV - uma endonuclease de restrição dimérica do Tipo IIP que cliva a sequência palindrômica GAT↓ATC (onde ↓ é o local do corte) - estão de acordo com estudos anteriores. Esses resultados sugerem que existem pelo menos três etapas no caminho para a clivagem do DNA que é iniciada pela introdução de magnésio após o EcoRV se ligar ao DNA em sua ausência, com uma taxa média de 0,17 s-1 para cada etapa.
Introdução
As endonucleases de restrição (REases) são enzimas que afetam quebras de fita dupla específicas da sequência no DNA. A descoberta das REases na década de 1970 levou ao desenvolvimento da tecnologia de DNA recombinante, e essas enzimas são agora ferramentas laboratoriais indispensáveis para modificação e manipulação genética1. As REases do tipo II são as enzimas mais utilizadas nesta classe, pois clivam o DNA em um local fixo dentro ou perto de sua sequência de reconhecimento. No entanto, há uma grande variação entre as REases do Tipo II, e elas são divididas em vários subtipos com base em propriedades enzimáticas particulares, em vez de serem classificadas de acordo com suas relações evolutivas. Entre cada subtipo, há exceções frequentes ao esquema de classificação, e muitas enzimas pertencem a vários subtipos2. Milhares de REases do Tipo II foram identificadas, e centenas delas estão disponíveis comercialmente.
No entanto, apesar da diversidade entre as REases do tipo II, muito poucas REases foram estudadas em detalhes. De acordo com o REBASE, o banco de dados de enzimas de restrição estabelecido por Sir Richard Roberts em 19753, os dados cinéticos publicados estão disponíveis para menos de 20 dessas enzimas. Além disso, embora algumas REases tenham sido observadas diretamente no nível de molécula única enquanto se difundiam ao longo do DNA antes de encontrar e se ligar à sua sequência de reconhecimento 4,5,6,7, existem muito poucos estudos de molécula única de sua cinética de reação de clivagem. Os estudos existentes não relatam estatísticas adequadas para realizar uma análise detalhada da variação nos tempos em que ocorrem eventos de clivagem única 8,9,10 ou não são capazes de capturar a distribuição completa dos tempos de clivagem11. Esse tipo de análise pode revelar a presença de intermediários cinéticos de vida relativamente longa e pode levar a uma melhor compreensão dos mecanismos de clivagem do DNA mediada por REase.
No nível de uma única molécula, os processos bioquímicos são estocásticos - o tempo de espera para que uma única instância do processo ocorra, τ, é variável. No entanto, pode-se esperar que muitas medições de τ obedeçam a uma distribuição de probabilidade, p(τ), que é indicativa do tipo de processo que está ocorrendo. Por exemplo, um processo de etapa única, como a liberação de uma molécula de produto de uma enzima, obedecerá à estatística de Poisson, e p(τ) assumirá a forma de uma distribuição exponencial negativa:
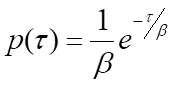
onde β é o tempo médio de espera. Observe que a taxa do processo, k, será igual a 1/β, o inverso do tempo médio de espera. Para processos que requerem mais de uma etapa, p(τ) será a convolução das distribuições exponenciais únicas para cada uma das etapas individuais. Uma solução geral para a convolução de N funções de decaimento exponencial único com tempos de espera médios idênticos, β, é a distribuição de probabilidade gama:
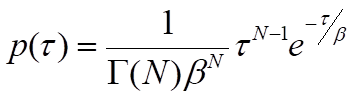
onde Γ(N) é a função gama, que descreve a interpolação do fatorial de N-1 para valores não inteiros de N. Embora esta solução geral possa ser usada como uma aproximação quando os tempos médios de espera de etapas individuais são semelhantes, deve-se entender que a presença de etapas relativamente rápidas será mascarada por etapas com tempos de espera significativamente mais longos. Em outras palavras, o valor de N representa um limite inferior no número de etapas12. Com um número adequado de medições de tempo de espera, os parâmetros β e N podem ser estimados agrupando os eventos e ajustando a distribuição gama ao histograma resultante ou usando uma abordagem de estimativa de máxima verossimilhança. Esse tipo de análise pode, portanto, revelar a presença de etapas cinéticas que não podem ser facilmente resolvidas em ensaios de conjunto e requer um grande número de observações para estimar os parâmetros com precisão12,13.
Este artigo descreve um método para usar DNA marcado com pontos quânticos e microscopia de fluorescência de reflexão interna total (TIRF) para observar centenas de eventos individuais de clivagem de DNA mediados por REase em paralelo. O design do ensaio torna possível agrupar os resultados de vários experimentos e pode criar distribuições de tempo de permanência contendo milhares de eventos. A alta fotoestabilidade e brilho dos pontos quânticos permitem uma resolução de tempo de 10 Hz sem sacrificar a capacidade de observar eventos de clivagem que ocorrem mesmo muitos minutos após o início do experimento. Boa resolução temporal e uma ampla faixa dinâmica, combinadas com a capacidade de coletar um grande conjunto de dados, permitem uma caracterização precisa das distribuições de tempo de permanência para descobrir a presença de múltiplas etapas cinéticas nas vias de clivagem das REases, que têm taxas de rotatividade na faixa de 1 min-1 . No caso do EcoRV, três etapas cinéticas podem ser resolvidas, todas identificadas por outros meios, confirmando que o ensaio é sensível à presença de tais etapas.
Os substratos de DNA duplex contendo a sequência de reconhecimento de interesse são produzidos pelo recozimento de um oligonucleotídeo biotinilado em uma fita complementar marcada com um único ponto quântico de nanocristal semicondutor covalentemente anexado. Esses substratos são introduzidos em um canal de fluxo construído sobre uma lamínula de vidro com um gramado de moléculas de polietilenoglicol (PEG) de alto peso molecular covalentemente ligadas à sua superfície. Os substratos de DNA são capturados por meio de uma ligação biotina-estreptavidina-biotina por uma fração das moléculas de PEG que possuem uma biotina em sua extremidade livre. Na microscopia TIRF, uma onda evanescente que decai exponencialmente com a distância da interface vidro-líquido fornece iluminação; A profundidade de penetração é da ordem do comprimento de onda da luz usada. Nessas condições, apenas os pontos quânticos que estão presos à superfície por uma molécula de DNA que foi capturada na superfície do vidro funcionalizado serão excitados. Os pontos quânticos que estão livres em solução não serão restringidos dentro da região iluminada e, portanto, não serão luminescentes. Se o DNA que amarra um ponto quântico à superfície for clivado, esse ponto quântico estará livre para se difundir para longe da superfície e desaparecerá da imagem de fluorescência.
Embora muitas REases Tipo II sejam conhecidas por se ligarem ao DNA na ausência de magnésio14, todas requerem magnésio para mediar a clivagem do DNA15. Essas REases podem se ligar ao DNA imobilizado na superfície na ausência de magnésio. Quando o tampão contendo magnésio flui através de um canal com REase pré-ligada ao DNA, a clivagem começa imediatamente, conforme indicado pelo desaparecimento dos pontos quânticos. A sincronização alcançada pela pré-ligação das moléculas de REase e, em seguida, iniciando a clivagem do DNA pela introdução de magnésio, facilita a medição do tempo de atraso até a conclusão da clivagem do DNA independentemente para cada molécula na população de enzimas observadas em um experimento. A fluoresceína é incluída como um corante marcador no tampão contendo magnésio para indicar a chegada do magnésio ao campo de visão. Como nenhuma enzima está incluída no tampão contendo magnésio, o tempo de atraso desde a chegada do tampão contendo magnésio até o desaparecimento de cada ponto quântico indica o tempo que leva para uma REase que já está ligada ao DNA clivar o DNA e liberar o ponto quântico da superfície do vidro. O desaparecimento do ponto quântico acontece rapidamente e resulta em uma diminuição acentuada na trajetória de intensidade, fornecendo uma indicação clara do tempo em que uma determinada molécula de DNA é clivada. A determinação dos tempos dos eventos é realizada pela análise matemática das trajetórias de intensidade, e um experimento típico resulta em centenas de eventos de clivagem identificáveis. Os resultados de vários experimentos podem ser agrupados para fornecer estatísticas adequadas para permitir a estimativa dos parâmetros, N e β, por análise de mínimos quadrados não lineares ou de máxima verossimilhança.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Informações gerais
- Projeto de oligonucleotídeos
NOTA: O substrato de DNA de 60 pares de bases (pb) de comprimento é formado por um par de oligonucleotídeos complementares com uma temperatura de fusão duplex de 75 ° C em 100 mM NaCl.- Peça um oligonucleotídeo sintetizado com uma única modificação de biotina 5 'e o outro com uma modificação de tiol 5' (com um espaçador de seis carbonos). Coloque o local de reconhecimento no centro da região duplex.
NOTA: As sequências de oligonucleotídeos para uso com EcoRV são mostradas abaixo (local de reconhecimento em negrito).
5' biotina - AAA ACC GAC ATG TTG ATT TCC TGA AAC GGG ATA TCA TCA AAG CCA TGA ACA AAG CAG CCG - 3'
5' thiol - CGG CTG CTT TGT TCA TGG CTT TGA TGA TAT CCC GTT TCA GGA AAT CAA CAT GTC GGT TTT - 3'
- Peça um oligonucleotídeo sintetizado com uma única modificação de biotina 5 'e o outro com uma modificação de tiol 5' (com um espaçador de seis carbonos). Coloque o local de reconhecimento no centro da região duplex.
- Use água ultrapura com resistividade de 18 MOhm para todas as etapas.
- Proteja todas as soluções que contenham pontos quânticos da luz para evitar o fotobranqueamento.
- Use uma fonte de ar comprimido para completar este protocolo.
2. Preparação de materiais de substrato de DNA marcados com pontos quânticos
NOTA: Além dos oligonucleotídeos descritos acima, consulte a Tabela de Materiais para outros materiais e a Tabela 1 para tampões necessários para a preparação de substratos de DNA marcados com pontos quânticos.
- Reduza os grupos tióis 5' no oligonucleotídeo a ser acoplado a pontos quânticos.
- Ressuspender cada oligonucleótido tiolado em água a uma concentração de 100 μM.
- Pipetar 650 μL de água em uma coluna de rotação de exclusão de tamanho para cada amostra de oligonucleotídeo e vórtice por ~ 15 s. Deixe a coluna embalar por 30 min.
- Preparar uma solução fresca de ditiotreitol (DTT) a 100 mM imediatamente antes de cada utilização, uma vez que a DTT se degrada rapidamente na solução. Abra cuidadosamente um frasco contendo 7,7 mg de TDT e pipete 500 μL de tampão fosfato de sódio no frasco; vórtice para misturar completamente.
- Para cada oligonucleotídeo ressuspenso, prepare um novo tubo e adicione 50 μL do oligonucleotídeo e 50 μL de solução de DTT ao tubo. Pipet para cima e para baixo para misturar. Incubar por 30 min à temperatura ambiente para reduzir as ligações dissulfeto entre os grupos tióis oxidados na extremidade 5' dos oligonucleotídeos.
- Remova as tampas da coluna giratória e coloque cada coluna no tubo de coleta. Centrifugar as colunas de centrifugação a 750 × g durante 2 min e rejeitar o eluído.
- Transfira as colunas de centrifugação para tubos de centrífuga novos. Pipetar suavemente todo o volume (100 μL) de uma amostra de mistura de DNA/TDT em cada coluna preparada. Centrifugar a 750 × g durante 2 min e medir a absorvância do eluído a 260 nm para confirmar que a concentração é de aproximadamente 40 μM.
- Armazene todas as amostras que não serão utilizadas imediatamente a -20 °C para evitar a oxidação dos grupos tióis e a formação de ligações dissulfeto.
- Acoplamento de DNA a pontos quânticos
- Pipetar uma alíquota de 50 μL do estoque de pontos quânticos em um dispositivo de diálise para cada construção a ser feita, certificando-se de não tocar na membrana com a ponta da pipeta. Dialisar contra um volume de tampão de ácido N-ciclohexil-2-aminoetanossulfônico (CHES) que seja pelo menos 1000x o volume da amostra com agitação a ~ 100 rpm por 15 min.
- Preparar uma solução nova de 6 mM de sulfosuccinimidil-4-(N-maleimidometil)ciclohexano-1-carboxilato (sulfo-SMCC) em tampão CHES imediatamente antes de cada utilização. Abra cuidadosamente um frasco contendo 2 mg de sulfo-SMCC e pipete 800 μL de tampão CHES no frasco para injetáveis. Vórtice para misturar bem.
- Usando um pipetador, recupere cuidadosamente os pontos quânticos suspensos do dispositivo de diálise e transfira para um novo tubo contendo um volume igual da solução sulfo-SMCC; pipet para cima e para baixo para misturar. Incubar por 1 h em temperatura ambiente com agitação a 1000 rpm para permitir que o sulfo-SMCC reaja com as aminas primárias nos pontos quânticos.
- Usando um pipetador, transfira cuidadosamente cada amostra para um novo dispositivo de diálise. Para remover o excesso de sulfo-SMCC, dialise contra um volume de tampão CHES que seja pelo menos 1000x o volume contido no(s) dispositivo(s) de diálise, com agitação, por 15 min. Troque o tampão 2x e realize a diálise com tampão CHES novo 3x no total, permitindo que a diálise prossiga 15 minutos após cada troca de tampão.
- Para trocar o tampão em preparação para a segunda reação, transfira os dispositivos de diálise para um béquer contendo um volume de solução salina tamponada com fosfato (PBS) que seja pelo menos 1000x o volume contido no(s) dispositivo(s). Dialise com agitação por 15 min, troque o tampão 2x e faça diálise com PBS fresco 3x no total, permitindo que a diálise prossiga por 15 min após cada troca de tampão.
- Usando um pipetador, recupere cuidadosamente a solução contendo os pontos quânticos do(s) dispositivo(s) de diálise e transfira cada amostra para um novo tubo contendo uma quantidade equimolar de oligonucleotídeo tiolado diluído em PBS. Combine um volume igual de PBS e 1/10do volume da amostra de oligonucleotídeo reduzida e purificada (concentração de aproximadamente 40 μM), pois a concentração de pontos quânticos neste ponto é de ~ 4 μM. Incubar por 2 h à temperatura ambiente, agitando a 1000 rpm.
- Adicione albumina de soro bovino (BSA, 10 mg / mL) a cada amostra para obter 0,5 mg / mL de concentração final de BSA. Usando um pipetador, transfira cuidadosamente cada amostra para um novo dispositivo de diálise e dialise contra um volume de tampão de armazenamento que seja pelo menos 1000x o volume contido no(s) dispositivo(s) de diálise. Troque o tampão 2x e realize a diálise com tampão de armazenamento fresco 3x no total, permitindo que a diálise prossiga por 15 minutos após cada troca de tampão.
- Recuperando cuidadosamente cada amostra, utilizando um pipetador, colocá-la num novo tubo e conservá-la a 4 °C.
NOTA: Não armazene a -20 °C, pois os pontos quânticos são danificados pelo congelamento.
- Recozimento de oligonucleotídeo marcado com pontos quânticos em oligonucleotídeo biotinilado
- Ressuspender cada oligonucleótido biotinilado em tampão de armazenamento a uma concentração de 100 μM. Armazenar as amostras não utilizadas a -20 °C.
- Combine uma amostra de oligonucleotídeo marcado com pontos quânticos com um excesso molar de 10 vezes de oligonucleotídeo biotinilado. Como a concentração estimada de DNA na amostra marcada com pontos quânticos é de 2 μM, adicione 0,2 μL de oligonucleotídeo biotinilado para cada 10 μL desta amostra.
- Aquecer a mistura num bloco de calor a 75 °C (temperatura de fusão para a região complementar). Mantenha nessa temperatura por 5 min. Desligue o bloco de calor e deixe esfriar lentamente. Armazenar a construção completa a 4 °C; Não congele.
3. Funcionalização superficial de lamínulas
NOTA: Este processo foi descrito anteriormente em outros protocolos de vídeo JoVE 16,17. Este protocolo descreve uma versão adaptada do procedimento com pequenas alterações para acomodar uma lâmina de vidro menor. Consulte a Tabela de Materiais para outros materiais necessários para a funcionalização da superfície de lamínulas.
- Coloque 5 lamínulas em cada porta-lamínulas. Certifique-se de pular um espaço entre cada par de lamínulas para que elas não grudem umas nas outras. Coloque as lamínulas em seu suporte em uma jarra com tampa, adicione etanol à jarra até que as lamínulas estejam cobertas e aperte bem a tampa. Coloque o frasco inteiro na água do sonicador do banho, sem submergi-lo completamente, e sonice por 30 min.
- Encha um copo ou jarra limpa com água ultrapura. Use uma pinça de metal para remover as lamínulas do suporte do etanol e mergulhe-as em água para enxaguar. Em seguida, transfira as lamínulas e o suporte para um frasco contendo uma solução de hidróxido de potássio (KOH) 1 M. Aparafuse bem a tampa, coloque o frasco no banho do sonicador e sonice por 30 min.
NOTA: Tenha cuidado ao manusear a solução de KOH, pois é corrosiva e irritante. - Repetir a sonicação em etanol e solução de KOH, lavando as lamínulas em água como acima descrito entre cada etapa.
- Transfira as lamínulas em seu suporte para um frasco limpo cheio de acetona pura. Termine a limpeza sonicando o frasco conforme descrito acima por 30 min. Não enxágue com água após esta etapa.
- Usando uma pinça de metal, transfira as lamínulas em seu suporte para um béquer limpo de 100 mL contendo 80 mL de acetona fresca e uma barra de agitação em microescala. Coloque o copo em uma placa de agitação magnética regulada para pelo menos 1.000 rpm. Enquanto a acetona está sendo agitada vigorosamente, pipete 1,6 mL de 3-aminopropiltrietoxissilano (APTES) no béquer para fazer uma solução a 2% v / v.
NOTA: Tenha cuidado ao pipetar APTES, pois é corrosivo. - Deixe as lamínulas incubarem na solução por 2 min e, em seguida, use uma pinça de metal para transferir as lamínulas em seu suporte para um copo de água para extinguir a reação. Enxágue as lamínulas mais duas vezes, recolocando a água no copo.
- Catalise o vidro silanizado no forno a 120 °C durante 75 min. Se não continuar para a próxima etapa imediatamente, armazene as lamínulas sob vácuo por no máximo alguns dias.
- Dissolver os polietilenoglicóis (PEGs) derivados de éster de N-hidroxisuccinimida (NHS) em bicarbonato de sódio a 100 mM (pH 8,2). Use uma proporção de 10:1 de PEG terminado em metoxi para PEG terminado em biotina, com ~ 100 mg / mL de PEG terminado em metoxi.
- Pipetar 100 μL da solução de PEG em metade das lamínulas silanizadas secas e cobrir cada uma com uma segunda lamínula. Use pequenos pedaços de parafilme colocados em cada canto como espaçadores para evitar que as lamínulas grudem umas nas outras.
- Incube as lamínulas em um ambiente úmido por 3,5 h. Separe os sanduíches de lamínula. Use uma garrafa de esguicho para lavar cada lamínula com água abundante e seque com ar comprimido.
- Armazene lamínulas funcionalizadas sob vácuo. Certifique-se de manter o lado tratado com PEG para cima, pois o outro lado não capturará o substrato de DNA.
4. Construção de dispositivos microfluídicos
NOTA: Consulte a Tabela de Materiais para outros materiais necessários para a construção do dispositivo microfluídico.
- Usando uma multiferramenta rotativa portátil equipada com uma broca de roda de ponta de diamante cônica, faça dois furos em cantos opostos da corrediça de quartzo para servir como entrada e saída. Certifique-se de prender o slide no lugar, lubrificar a broca e deslizar constantemente com água durante a perfuração. Após cada experimento, recupere a lâmina de quartzo preparada para reutilização, embebendo um dispositivo usado em acetona para dissolver o adesivo e o epóxi; descarte os componentes restantes.
NOTA: A perfuração pode ser realizada manualmente, mas o uso de um suporte tipo prensa para a multiferramenta facilita o processo. - Combine 25 μL de solução de estreptavidina com 80 μL de PBS e 20 μL de tampão de bloqueio. Cubra uma lamínula tratada com PEG com esta mistura e incube em um ambiente úmido por 30 min.
- Durante a incubação da lamínula, prepare o espaçador de imagem para criar um canal de fluxo. Corte um pedaço quadrado de 1 polegada de material espaçador de imagem e, em seguida, marque um canal de 2 mm de largura alinhado às extremidades dos orifícios perfurados na lâmina de quartzo (Figura 2). Corte o canal do material espaçador com um bisturi.
- Retire um lado do suporte do espaçador de imagem e coloque-o cuidadosamente na lâmina de quartzo, tomando cuidado para não cobrir os orifícios de entrada e saída. Certifique-se de limpar bem a lâmina de quartzo com acetona para remover quaisquer restos de adesivo de experimentos anteriores.
- Lave a lamínula com água e seque com ar comprimido. Remova o outro lado do suporte do espaçador de imagem e coloque-o entre a lâmina de quartzo e a lamínula funcionalizada revestida com estreptavidina, usando a pinça de plástico para pressionar o conjunto e remover as bolhas de ar do adesivo.
- Insira um tubo de polietileno de 30 cm de comprimento em cada orifício da lâmina de quartzo. Certifique-se de cortar as extremidades do tubo em um ângulo para garantir o fluxo livre da solução. Use um suporte para tubos ou outro suporte para manter os tubos no lugar e vede os tubos no lugar e as bordas do dispositivo montado com epóxi.
NOTA: Funciona bem construir o dispositivo em um pedaço de parafilme, que pode ser facilmente retirado da parte inferior quando o epóxi estiver endurecido. - Assim que o epóxi endurecer, insira a agulha romba da seringa vazia no tubo de saída do dispositivo e mergulhe a extremidade do tubo de entrada em um recipiente cheio de tampão de bloqueio. Puxe o êmbolo da seringa para trás para encher o dispositivo com tampão de bloqueio. Deixe o dispositivo incubar por pelo menos 30 minutos antes de usar.
5. Amarração de superfície de substrato de DNA marcado com pontos quânticos
NOTA: Além do dispositivo microfluídico, substrato de DNA e tampão de bloqueio descritos acima, consulte a Tabela de Materiais para outros materiais e a Tabela 1 para tethering de superfície de substratos de DNA marcados com pontos quânticos.
- Prenda o dispositivo microfluídico à placa de platina do microscópio com fita adesiva, coloque a objetiva em contato com a parte inferior do dispositivo e posicione a objetiva de forma que o campo de visão esteja dentro do canal microfluídico.
- Lave o canal microfluídico com tampão de bloqueio fresco puxando para trás o êmbolo da seringa depois de conectar o tubo de saída à bomba da seringa. Verifique se não há bolhas presas na tubulação ou no canal.
NOTA: A partir deste ponto, certifique-se de que o tubo de entrada esteja em líquido para que nenhuma bolha de ar seja introduzida no dispositivo. - Diluir 1 μL do substrato de ADN preparado em 1 ml de tampão de bloqueio. Coloque o tubo de entrada no substrato de DNA diluído e flua 800 μL da solução de substrato através do canal a uma taxa de 200 μL / min. Deixe a solução de DNA incubar no canal sem perturbação por 15 minutos após a interrupção do fluxo.
- Fluxo de pelo menos 800 μL de tampão de bloqueio através do canal a uma taxa de 200 μL/min para enxaguar o DNA não ligado para fora do canal.
- Ajuste a potência do laser, o foco do microscópio e o ângulo TIRF para que os pontos quânticos conectados à superfície fiquem claramente visíveis.
NOTA: Embora os pontos quânticos não branqueiem rapidamente, é melhor manter a potência o mais baixa possível para minimizar o piscar.
6. Clivagem de DNA mediada por REase
NOTA: Consulte a Tabela de Materiais para materiais e a Tabela 1 para tampões necessários para clivagem de DNA mediada por REase.
- Certifique-se de que a câmera esteja resfriada até a temperatura operacional ideal e configurada para streaming de alta velocidade com um tempo de exposição de 0,10 seg. Esteja preparado para coletar dados por ~4 min.
- Lave o dispositivo microfluídico com 800 μL de tampão experimental sem magnésio a uma taxa de fluxo de 200 μL / min.
- Adicionar a REase a uma alíquota (1 ml) de tampão experimental sem magnésio e misturar delicadamente por pipetagem. Use 4 μL de estoque de 100.000 unidades/mL, o que corresponde a 400 unidades/mL de EcoRV. Fluxo de 800 μL da enzima diluída através do canal a uma taxa de fluxo de 200 μL / min.
- Iniciar a experiência com um tampão experimental contendo magnésio e fluoresceína a um caudal de 200 μl/min. Comece a capturar dados imediatamente após iniciar a bomba de seringa. Depois de fluir 800 μL de buffer, pare a aquisição de dados.
7. Análise dos dados
NOTA: Consulte a Tabela de Materiais para o software de análise de dados usado para este protocolo e faça ajustes se estiver usando uma plataforma de análise diferente.
- Determinação de ponto de tempo zero
- Subtraia a fluorescência de fundo de cada quadro do filme experimental usando a função imtophat , uma função de filtragem morfológica integrada na caixa de ferramentas de processamento de imagem. Selecione um disco com um raio de três pixels como elemento estruturante. Obtenha a intensidade do plano de fundo subtraindo a imagem filtrada da imagem original.
NOTA: O filtro retém apenas os recursos da imagem mais brilhantes que o plano de fundo e menores que o elemento estruturante. - Calcule a média do plano de fundo subtraído em cada quadro do filme. Use uma trajetória desse valor para determinar o ponto de tempo zero para o experimento (Figura 3A). Determine a hora de início encontrando o primeiro quadro no qual a taxa de aumento excede 3x o desvio padrão da taxa de mudança durante o fluxo de volume morto.
NOTA: Um aumento acentuado na taxa de variação do valor médio do fundo subtraído para cada quadro de filme indica o momento em que o tampão experimental final contendo o corante traçador entrou na célula de fluxo (Figura 3B).
- Subtraia a fluorescência de fundo de cada quadro do filme experimental usando a função imtophat , uma função de filtragem morfológica integrada na caixa de ferramentas de processamento de imagem. Selecione um disco com um raio de três pixels como elemento estruturante. Obtenha a intensidade do plano de fundo subtraindo a imagem filtrada da imagem original.
- Cálculo e análise da trajetória de intensidade de pontos quânticos
- Calcule uma projeção de intensidade máxima para o conjunto de quadros de filme corrigidos pelo fundo gravados antes da chegada do corante traçador. Determine as localizações dos pontos quânticos com precisão de um pixel nesta imagem de projeção usando uma função de localização de pico, como pkfnd.
- Gere trajetórias de intensidade para pontos quânticos individuais calculando a intensidade média em cada quadro de filme de uma região quadrada de três pixels por lado ao redor do local retornado pela função de localização de pico.
NOTA: A correção de fundo descrita acima remove os artefatos introduzidos pelo corante traçador. As trajetórias de intensidade resultantes devem ser relativamente planas, mas ainda incluir flutuações de intensidade que ocorrem naturalmente (Figura 4). - Identifique eventos de desaparecimento de pontos quânticos por meio de análise estatística das trajetórias de intensidade. Para cada trajetória de intensidade de ponto quântico, calcule uma intensidade limite que esteja acima do valor mínimo por uma fração apropriada da diferença entre os valores máximo e mínimo para essa trajetória, dependendo do ruído observado nas trajetórias.
NOTA: A flutuação de intensidade do ponto quântico é normalmente muito maior do que a flutuação de fundo. Um valor apropriado pode ser determinado comparando esses valores: o limite selecionado deve ser maior que o valor de fundo mais alto, mas idealmente menor que o valor mais baixo para fluorescência de pontos quânticos. Para os dados apresentados, o limiar utilizado foi calculado acima do valor mínimo para cada trajetória mais um terço da diferença entre os valores máximo e mínimo para essa trajetória. Um ponto quântico pode ser considerado como desaparecendo no último ponto de tempo em que a intensidade observada em sua localização estava acima desse limite. - Confirme eventos de desaparecimento putativos. Incluir eventos na análise final apenas se a intensidade observada estiver acima do ponto médio entre os valores de intensidade mínima e máxima para a trajetória para mais da metade dos quadros do filme antes do momento do desaparecimento e o desvio-padrão da trajetória de intensidade diminuir após o evento.
NOTA: Outros testes estatísticos podem ser empregados para garantir que apenas eventos de desaparecimento válidos sejam registrados.
Access restricted. Please log in or start a trial to view this content.
Resultados
A célula de fluxo é acoplada diretamente a uma objetiva de ampliação de 60x de imersão em óleo de alta abertura numérica em um microscópio invertido equipado com iluminação a laser para imagens TIRF através da objetiva (Figura 5A). Depois de introduzir o substrato de DNA e lavar o excesso de DNA e pontos quânticos, normalmente existem milhares de pontos quânticos individuais em um campo de visão (Figura 5B). Esses pontos quânticos são fixados de ...
Access restricted. Please log in or start a trial to view this content.
Discussão
O substrato de DNA para este ensaio é marcado com um ponto quântico usando um esquema de reação em duas etapas usando sulfo-SMCC. Este reticulador bifuncional consiste em uma porção éster NHS que pode reagir com uma amina primária e uma porção maleimida que pode reagir com um grupo sulfidrila20. Os oligonucleotídeos tiolados usados para preparar o substrato são enviados em sua forma oxidada. É importante reduzi-los e purificá-los, conforme descrito, antes de prosseguir com o procedim...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm interesses financeiros concorrentes ou outros conflitos de interesse
Agradecimentos
Este trabalho foi apoiado pelo Prêmio Número K12GM074869 para CME do Instituto Nacional de Ciências Médicas Gerais. O conteúdo é de responsabilidade exclusiva dos autores e não representa necessariamente as opiniões oficiais do Instituto Nacional de Ciências Médicas Gerais ou dos Institutos Nacionais de Saúde.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 3-aminopropyltriethoxysilane (APTS) | Sigma Aldrich | 440140-100ML | Store in dessicator box |
| 5 Minute Epoxy | Devcon | 20845 | use for sealing the microfluidic device |
| acetone | Pharmco | 329000ACS | use for cleaning coverslips |
| bath sonicator | Fisher Scientific | CPXH Model 2800 | catalog number 15-337-410 |
| Beaker, glass, 100 mL | |||
| Benchtop centrifuge | |||
| Biotin-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | BIO-SVA-5K | Succinimidyl valerate has a longer half-life than succinimidyl carbonate |
| biotinylated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Biotin modification and HPLC purification |
| Bovine Serum Albumin (BSA) | VWR | 0903-5G | prepare a 10 mg/mL solution (aq) and heat to 95 °C before using |
| Centri-Spin-10 Size Exclusion Spin Columns | Princeton Separations | CS-100 or CS-101 | used to purify thiolated oligonucleotides after reducing the disulfide bond |
| Centrifuge tubes 1.5. mL | |||
| Coverslips, 1-inch square glass | |||
| coverslip holders | |||
| diamond point wheel | Dremel | 7134 | use for drilling holes in quartz flow cell topper |
| dithiothreitol (DTT) | Thermo Scientific | A39255 | No-Weigh Format, 7.7 mg/vial |
| drill press rotary tool workstation stand | Dremel | 220-01 | facilitates quartz drilling |
| EcoRV (REase used to generate example data) | New England Biolabs | R0195T or R0195M | Use 100,000 units/mL stock to avoid adding excess glycerol Check REBASE for suppliers of other REases |
| ethanol | various | CAS 64-17-5 | denatured or 95% are acceptable, use for cleaning coverslips |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | EDS | BioUltra, anhydrous, store in dessicator box |
| Flea Micro Spinbar | Fisherbrand | 14-513-65 | 3 mm x 10 mm size to fit beneath coverslip rack |
| fluorescein | Acros Organics | 17324 | use to make experimental buffers |
| gravity convection oven | Binder | 9010-0131 | |
| handheld rotary multitool | Dremel | 8220 | use for drilling holes in quartz flow cell topper |
| ImagEM X2 EM-CCD Camera | Hamamatsu | C9100-23B | air cooling is adequate for this experiment, use HCImage software or similar to control |
| Imaging spacer, double-sided, adhesive | |||
| Jar, glass with screw cap, (approximately 50 mm diameter by 50 mm high) | |||
| magnesium chloride hexahydrate | Fisher Bioreagents | BP214-500 | use to make experimental buffer with magnesium |
| MATLAB software | Data analysis | ||
| metal tweezers | Fisher Brand | 16-100-110 | |
| methoxy-PEG-Succinimidyl Valerate (MW 5,000) | Laysan Bio | M-SVA-5K | Both PEGs should have the same NHS ester so that the rate of reaction is consistent |
| microcentrifuge | Eppendorf | 5424 | |
| multiposition magnetic stirrer | VWR | 12621-022 | |
| N-cyclohexyl-2-aminoethanesulfonic acid (CHES) | Acros Organics | AC20818 | CAS 103-47-9, use to make CHES buffer |
| orbital shaker and heater for microcentrifuge tubes | Q Instruments | 1808-0506 | with 1808-1061 adaptor for 24 x 2.0 mL or 15 x 0.5 mL tubes |
| Parafilm | |||
| PE60 Polyethylene tubing (inner diameter 0.76 mm, outer diameter 1.22 mm) | Intramedic | 6258917 | 22 G blunt needles are a good fit for this tubing size |
| Phosphate-Buffered Saline (PBS) 10x | Sigma Aldrich | P7059 | Use at 1x strength |
| potassium hydroxide | VWR Chemicals BDH | BDH9262 | use a 1 M solution to clean coverslips |
| Qdot 655 ITK Amino (PEG) Quantum Dots | Invitrogen | Q21521MP | |
| Quartz Slide, 1 inch square, 1 mm thick | Electron Microscopy Sciences | 72250-10 | holes must be drilled in the corners for inlet and outlet tubing insertion |
| reinforced plastic tweezers | Rubis | K35a | use for handling coverslips and building microfluidic device |
| SecureSeal Adhesive Sheets | Grace Biolabs | SA-S-1L | cut to form spacer for microfluidic device |
| Single channel syringe pump for microfluidics | New Era Pump Systems | NE-1002X-US | fitted with a 50 mL syringe and a 22 G blunt needle |
| Slide-a-Lyzer MINI Dialysis Devices, 10 kDa MWCO, 0.1 mL | Thermo Scientific | 69570 or 69572 | used for buffer exchange during quantum dot coupling to DNA |
| sodium bicarbonate | EMD Millipore | SX0320 | use to make buffer for surface functionalization; 100 mM, pH 8 |
| sodium chloride | Macron | 7581-12 | use to make experimental buffers |
| Sodium phosphate dibasic solution (BioUltra, 0.5 M in water) | Sigma Aldrich | 94046 | use to make 100 mM sodium phosphate buffer |
| Sodium phosphate monobasic solution (BioUltra, 5M in water) | Sigma Aldrich | 74092 | use to adjust pH of 100 mM sodium phosphate buffer |
| Streptavidin from Streptomyces avidinii | Sigma Aldrich | S4762 | dissolve at 1 mg/mL and store 25 mL aliqouts at -20 ? |
| Sulfosuccinimidyl-4-(N-maleimidomethyl) cyclohexane-1-carboxylate (sulfo-SMCC) | Thermo Scientific | A39268 | No-Weigh Format, 2 mg/vial |
| Syringe fitted with blunt 21 G needle | |||
| Syringe pump | |||
| thiolated oligonucleotide | Integrated DNA Technologies | custom - see protocol for design considerations | Request 5' Thiol Modifier C6 S-S and HPLC purificaiton |
| TIRF imaging system with 488 nm laser illumination | various | custom built | |
| Tris -HCl | Research Products International | T60050 | use to make experimental buffers |
| Tris base | JT Baker | 4101 | use to make experimental buffers |
| Tween-20 | Sigma | P7949 | use to make blocking buffer |
| Ultrapure water | |||
| vortex mixer | VWR | 10153-842 | |
| Wash-N-Dry Coverslip Rack | Electron Microscopy Sciences | 70366-16 | used for surface functionalization of coverslips |
Referências
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5905-5908 (2005).
- Loenen, W. A., Dryden, D. T., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Research. 42 (1), 3-19 (2014).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE--a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Research. 43, Database issue 298-299 (2015).
- Bonnet, I., et al. Sliding and jumping of single EcoRV restriction enzymes on non-cognate DNA. Nucleic Acids Research. 36 (12), 4118-4127 (2008).
- Biebricher, A., Wende, W., Escude, C., Pingoud, A., Desbiolles, P. Tracking of single quantum dot labeled EcoRV sliding along DNA manipulated by double optical tweezers. Biophysical Journal. 96 (8), 50-52 (2009).
- Blainey, P. C., et al. Nonspecifically bound proteins spin while diffusing along DNA. Nature Structural & Molecular Biology. 16 (12), 1224-1229 (2009).
- Huang, C. -F., et al. Direct visualization of DNA recognition by restriction endonuclease EcoRI. Journal of Experimental & Clinical Medicine. 5 (1), 25-29 (2013).
- Palma, M., et al. Selective biomolecular nanoarrays for parallel single-molecule investigations. Journal of American Chemical Society. 133 (20), 7656-7659 (2011).
- Reinhard, B. M., Sheikholeslami, S., Mastroianni, A., Alivisatos, A. P., Liphardt, J. Use of plasmon coupling to reveal the dynamics of DNA bending and cleavage by single EcoRV restriction enzymes. Proceedings of the National Academy of Sciences of the United States of America. 104 (8), 2667-2672 (2007).
- van den Broek, B., Noom, M. C., Wuite, G. J. DNA-tension dependence of restriction enzyme activity reveals mechanochemical properties of the reaction pathway. Nucleic Acids Research. 33 (8), 2676-2684 (2005).
- Gambino, S., et al. A single molecule assay for measuring site-specific DNA cleavage. Analytical Biochemistry. 495, 3-5 (2016).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Loparo, J. J., van Oijen, A. Single-molecule enzymology. Handbook of Single-Molecule Biophysics. Hinterdorfer, P., Oijen, A. , Springer. New York, NY. 165-182 (2009).
- Taylor, J. D., Badcoe, I. G., Clarke, A. R., Halford, S. E. EcoRV restriction endonuclease binds all DNA sequences with equal affinity. Biochemistry. 30 (36), 8743-8753 (1991).
- Pingoud, A., Jeltsch, A. Structure and function of type II restriction endonucleases. Nucleic Acids Research. 29 (18), 3705-3727 (2001).
- Tanner, N. A., Loparo, J. J., van Oijen, A. M. Visualizing single-molecule DNA replication with fluorescence microscopy. Journal of Visualized Experiments: JoVE. (32), e1529(2009).
- Kulczyk, A. W., Tanner, N. A., Loparo, J. J., Richardson, C. C., van Oijen, A. M. Direct observation of enzymes replicating DNA using a single-molecule DNA stretching assay. Journal of Visualized Experiments: JoVE. (37), e1689(2010).
- Baldwin, G. S., Vipond, I. B., Halford, S. E. Rapid reaction analysis of the catalytic cycle of the EcoRV restriction endonuclease. Biochemistry. 34 (2), 705-714 (1995).
- Winkler, F. K., et al. The crystal-structure of Ecorv endonuclease and of Its complexes with cognate and non-cognate DNA fragments. EMBO Journal. 12 (5), 1781-1795 (1993).
- Sioss, J. A., Stoermer, R. L., Sha, M. Y., Keating, C. D. Silica-coated, Au/Ag striped nanowires for bioanalysis. Langmuir. 23 (22), 11334-11341 (2007).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados