Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Uso de la secuenciación de próxima generación para identificar mutaciones asociadas con la reparación de una rotura de doble cadena inducida por CAS9 cerca del promotor CD4
En este artículo
Resumen
Aquí se presenta la endonucleasa sgRNA / CAS9 y el protocolo de secuenciación de próxima generación que se puede utilizar para identificar las mutaciones asociadas con la reparación de rotura de doble cadena cerca del promotor CD4.
Resumen
Las roturas de doble cadena (DSB) en el ADN son el tipo más citotóxico de daño en el ADN. Debido a que una miríada de insultos pueden resultar en estas lesiones (por ejemplo, estrés de replicación, radiación ionizante, daño UV no reparado), los DSB ocurren en la mayoría de las células cada día. Además de la muerte celular, los DSB no reparados reducen la integridad del genoma y las mutaciones resultantes pueden impulsar la tumorigénesis. Estos riesgos y la prevalencia de los DSB motivan las investigaciones sobre los mecanismos por los cuales las células reparan estas lesiones. La secuenciación de próxima generación se puede combinar con la inducción de DSB mediante radiación ionizante para proporcionar una herramienta poderosa para definir con precisión las mutaciones asociadas con los defectos de reparación de DSB. Sin embargo, este enfoque requiere una secuenciación del genoma completo computacionalmente desafiante y prohibitiva para detectar la reparación de los DSB que ocurren al azar asociados con la radiación ionizante. Las endonucleasas de corte raras, como I-Sce1, proporcionan la capacidad de generar un solo DSB, pero sus sitios de reconocimiento deben insertarse en el genoma de interés. Como resultado, el sitio de reparación es inherentemente artificial. Los avances recientes permiten que el ARN guía (sgRNA) dirija una endonucleasa Cas9 a cualquier locus del genoma de interés. Esto podría aplicarse al estudio de la reparación del DSB, lo que hace que la secuenciación de próxima generación sea más rentable al permitir que se centre en el ADN que flanquea el DSB inducido por Cas9. El objetivo del manuscrito es demostrar la viabilidad de este enfoque mediante la presentación de un protocolo que puede definir mutaciones que se derivan de la reparación de un DSB aguas arriba del gen CD4. El protocolo se puede adaptar para determinar los cambios en el potencial mutagénico del DSB asociados con factores exógenos, como inhibidores de la reparación, expresión de proteínas virales, mutaciones y exposiciones ambientales con requisitos de cálculo relativamente limitados. Una vez que el genoma de un organismo ha sido secuenciado, este método puede ser empleado teóricamente en cualquier locus genómico y en cualquier modelo de cultivo celular de ese organismo que pueda ser transfectado. Adaptaciones similares del enfoque podrían permitir comparaciones de la fidelidad de reparación entre diferentes loci en el mismo fondo genético.
Introducción
Mantener la estabilidad genómica es fundamental para todos los organismos vivos. La replicación precisa del ADN y una respuesta robusta al daño del ADN (DDR) son necesarias para propagar fielmente el material genético 1,2. Los daños en el ADN ocurren regularmente en la mayoría de las células 2,3. Cuando se detectan estos daños, se detiene la progresión del ciclo celular y se activan los mecanismos de reparación del ADN. Las roturas de doble cadena en el ADN o DSB son el tipo de daño al ADN más tóxico y mutagénico 3,4.
Si bien varias vías de señalización DDR pueden reparar estas lesiones, las vías de reparación de DSB más estudiadas son la recombinación homóloga (HR) y la unión final no homóloga (NHEJ). HR es una vía en gran parte libre de errores que repara un DSB utilizando una cromátida hermana como plantilla homóloga. Esto tiende a suceder en la fase S y la fase G2 de un ciclo celular 5,6,7. NHEJ es más propenso a errores, pero puede ocurrir a lo largo del ciclo celular 8,9. Se han desarrollado varios ensayos de reporteros para medir la eficiencia de mecanismos de reparación específicos 10,11,12. Estos ensayos tienden a basarse en la citometría de flujo para una medición de alto rendimiento de la actividad de la vía de reparación de DSB utilizando GFP o mCherry como lectura11,13. Si bien son altamente eficientes, se basan en la reparación canónica que ocurre en un DSB introducido artificialmente.
Hay una variedad de otros métodos utilizados para estudiar la reparación de DSB. Muchos de estos se basan en la microscopía de inmunofluorescencia (IF) 1,14. La microscopía IF detecta focos nucleares discretos representativos de complejos de reparación después de que los DSB son inducidos por la exposición a productos químicos genotóxicos o radiaciones ionizantes15,16. El seguimiento de la formación y resolución de estos focos proporciona una indicación del inicio y la finalización de la reparación, respectivamente14,17. Sin embargo, estos métodos de inducción de DSB (es decir, productos químicos o radiación ionizante) no causan DSB en lugares definidos en el genoma. También es funcionalmente imposible usarlos para inducir consistentemente solo un pequeño número (por ejemplo, 2-4) de DSB. Como resultado, los métodos más utilizados para inducir DSB causan una multitud de lesiones distribuidas aleatoriamente por todo el genoma18. Se puede introducir un pequeño número de DSB insertando el sitio de reconocimiento para una endonucleasa de corte raro y expresando la endonucleasa pertinente, como I-Sce119. Desafortunadamente, la integración requerida de un sitio objetivo impide el examen de DSB en loci genómicos endógenos.
Este manuscrito describe un método para detectar mutaciones asociadas con la reparación de un DSB generado en un locus definido por el usuario. Proporcionamos un ejemplo representativo del enfoque aplicado para evaluar la capacidad de una proteína viral para aumentar el número de mutaciones asociadas con un DSB. Específicamente, este manuscrito describe el uso de un ARN guía único (sgRNA) para dirigir una endonucleasa CAS9 para inducir un DSB en el marco de lectura abierto CD4 humano en queratinocitos del prepucio humano que expresan control vectorial (HFK LXSN) y HFK que expresa la proteína E6 del virus del papiloma humano tipo 8 (HFK 8E6). La secuenciación dirigida de próxima generación (NGS) de la región que rodea la ruptura permite definir rigurosamente las mutaciones asociadas con la reparación de la lesión. Estos datos demuestran que la proteína viral causa un aumento de aproximadamente 20 veces en las mutaciones durante la reparación del DSB. También proporciona una caracterización imparcial de las consecuencias mutagénicas de los DSB en un solo locus sin la necesidad de secuenciar el genoma completo. En principio, el protocolo podría adaptarse fácilmente para comparar el riesgo relativo de mutaciones entre loci del genoma o líneas celulares.
Protocolo
1. Revestimiento celular
- Cultive células HFK LXSN y HFK 8E6 en placas de 10 cm en medios de cultivo de queratinocitos (10 ml / placa) con suplemento de crecimiento de queratinocitos humanos (HKGS) y penicilina / estreptomicina al 1%. Cultive las células a aproximadamente el 80% de confluencia a 37 °C en una incubadora de camisa con un 5% de CO2.
- Reemplace los medios de cultivo con 3 ml de tripsina-EDTA (0.05%, ácido tetraacético etilendiamina). Incubar a 37 °C durante 3 min. Neutralizar la tripsina con el mismo volumen de suero fetal bovino (FBS) suplementado con medios y transferir células a un tubo centrífugo de 15 ml. Centrifugadora a 300 x g durante 5 min.
- Células resuspend con 10 ml de medios de cultivo de queratinocitos con HKGS. Determine la concentración de células con un hemocitómetro.
- Placa 4 x 105 células/placas de 6 cm (semilla dos placas para HFK LXSN y dos placas para HFK 8E6) en 4mL de medios de cultivo de queratinocitos con HKGS y penicilina y estreptomicina al 1%. Cultivar a 37 °C en una incubadora de chaqueta con 5% de CO2.
NOTA: El análisis de células HFK fue elegido para este protocolo por dos razones. En primer lugar, HFK es una línea celular difícil de transfectar. Por lo tanto, al demostrar que el protocolo funciona en esta línea celular, se proporciona evidencia de que probablemente funcionará en células más comúnmente utilizadas y más fácilmente transfectadas. En segundo lugar, datos publicados anteriormente demuestran que una proteína viral (8E6) dificulta la reparación de roturas de doble cadena en el ADN 20,21,22. Así, comparar HFK LXSN y HFK 8E6 nos permite demostrar la capacidad del ensayo para detectar aumentos en las mutaciones asociadas con una reducción de la capacidad de reparación celular.
2. Transfección
- El día de la transfección (24 h después del recubrimiento), reemplace los medios con 3 ml de medios suplementados sin antibióticos. Incubar durante 2 h a 37 °C en una incubadora de chaqueta.
- Transfectar células con reactivos de transfección adecuados a base de lípidos de acuerdo con las instrucciones del fabricante.
- Caliente los reactivos de transfección a temperatura ambiente y pipete suavemente antes de usar.
- Para cada línea celular (HFK LXSN y HFK 8E6), coloque la cantidad adecuada de tampón de transfección (según las indicaciones del fabricante) en un tubo de centrífuga estéril de 1,5 ml (tubo 1). Incluya otro tubo con la misma cantidad de tampón de transfección (transfección simulada o tubo 2).
- Añadir 2 μg de ADN plásmido que exprese CAS9/sgRNA dirigido a CD4 humano (px330-CD4, 5'- GGCGTATCTGTGTGAGGACT) al tubo 1 del paso 2.2.2. Pipete suavemente para mezclar completamente. Agregue el mismo volumen de agua estéril al tubo 2.
- Incluya una placa de control con reactivos de transfección solos (sin plásmido) para cada línea celular.
NOTA: La segunda placa sirve como control negativo en el experimento, permitiendo al usuario confirmar que la transfección con el CAS9/sgRNA no es responsable de ninguna mutación. - Añadir la cantidad adecuada del reactivo de transfección (según las indicaciones del fabricante) al tubo con la mezcla de ADN (tubo 1) del paso 2.2.3 y la transfección simulada (tubo 2) del paso 2.2.2. Pipete suavemente para mezclar completamente. Incubar a temperatura ambiente durante 15-30 min para dar tiempo suficiente a la formación de complejos.
- Agregue la mezcla de transfección gota a gota a la placa. Sacude suavemente el cultivo durante 1 minuto para distribuir uniformemente la mezcla de transfección.
- Incubar durante 48 h después de la transfección para permitir la expresión de CAS9.
- Cosechar células por tripsinización.
- Reemplace los medios de cultivo con 1 ml de tripsina-EDTA (0.05%, ácido tetraacético etilendiamina). Incubar a 37 °C durante 3 min. Neutralizar la tripsina con el mismo volumen de medios suplementados con FBS.
- Para cada placa de células, transfiera la suspensión celular a dos tubos de microcentrífuga con alícuotas iguales. Centrifugadora a 300 x g durante 5 min.
- Resuspend el pellet celular de un tubo en el paso 2.4.2 en 1 ml de solución salina tamponada con fosfato (PBS) para la secuenciación. Vuelva a suspender el otro tubo del paso 2.4.2 con PBS helado para immunoblot.
- Cosecha los lisados de células enteras para el inmunoblot.
- Centrifugar el tubo a 300 x g durante 5 min. Deseche el sobrenadante.
- Añadir un tampón de ensayo de radioinmunoprecipitación de 100 μL (tampón de lisis RIPA) mezclado con un inhibidor de la proteasa al 1 % y un inhibidor de la fosfatasa al 1 % en el tubo, mezclar bien con una pipeta e incubar durante 10 minutos en hielo.
NOTA: El tampón de lisis RIPA contiene 10 mM Tris-HCl, pH 8.0; 1 mM EDTA; 0,5 mM EGTA; 1% Tritón X-100; 0.1% Desoxicolato de Sodio; 0,1% SDS; 140 mM NaCl, y agua desionizada. - La centrífuga lisa a 13.000 x g durante 10 min. Recolectar sobrenadantes para immunoblot.
3. Medición de la expresión de CAS9 a través de immunoblot
- Determine la concentración de proteínas con un ensayo de ácido bicinconínico (BCA) de acuerdo con las instrucciones del fabricante.
- Ejecute 20 μg de proteína de cada muestra en los pocillos de un gel de trisacetato al 3%-8% durante 150 minutos y transferencia semidrica (10 V durante 30 minutos y luego 25 V durante 12 min) a la membrana de difluoruro de polivinilideno.
- Después de bloquear la membrana en leche seca sin grasa al 5% en PBS con interpolación al 0,1% (PBST) durante 1 h a temperatura ambiente, agregue anticuerpos anti-CAS9 (1:1000) y anti-GAPDH (1:1000). Incubar a 4 °C durante la noche.
- Después de lavar la membrana con PBST, incube la membrana con anticuerpos secundarios en leche seca sin grasa al 5% en PBST durante 1 h a temperatura ambiente.
- Imagine la mancha y determine el nivel CAS9 mediante densitometría23. Consulte la Figura 1 para ver una mancha representativa.
NOTA: La detección de la formación de focos H2AX fosforilados (S139) mediante microscopía de inmunofluorescencia se puede utilizar para validar la actividad de CAS914. Se espera un bajo número de focos distintos (típicamente 1-4 focos) dependiendo de la posición del ciclo celular, si las mutaciones en el sitio objetivo CAS9 impiden un mayor corte y cuántas copias del sitio de corte CAS9 existen en el genoma de interés. En la figura 2 se muestra una imagen representativa.
4. Extracción de ácidos nucleicos y generación de amplicones
- Extraiga ADN de muestras celulares del paso 2.4.3 utilizando un kit de extracción de ADN de alto peso molecular, según lo especificado por el fabricante.
- Imprimaciones resuspend con disolvente indicado según la ficha técnica. Diluir con el mismo reactivo a 20 μM y agrupar los cebadores de 20 μM en las piscinas indicadas.
NOTA: La piscina de imprimación aparece en la Tabla suplementaria 1. - Cree una mezcla maestra de PCR utilizando una polimerasa Taq de amplificación larga para cada grupo de imprimación de 20 μM como se especifica en la Tabla 1.
- Añadir 21 μL de las mastermixes para separar los tubos de PCR.
- Añadir 4 μL de la muestra objetivo (100 ng/μL) del paso 4.1 a los tubos de ensayo de PCR que contengan tubos de ensayo mastermix y cap. Asegure reacciones separadas para cada piscina de imprimación.
- Vórtice para mezclar tubos de ensayo de PCR y centrífuga (giro rápido) para eliminar las gotas de las tapas de los tubos.
- Coloque los tubos de PCR en una máquina termocicladora convencional.
- Programa PCR máquina como se especifica en la Tabla 2.
- Ejecute el programa en un termociclador.
5. Limpieza por PCR
- Retire las imprimaciones de las reacciones de PCR utilizando un sistema de limpieza de PCR basado en cuentas.
- Retire las cuentas de limpieza del refrigerador 30 minutos antes de su uso.
- Las cuentas de vórtice se encuentran bien antes de su uso y asegúrese de que todas las cuentas se resuspendan.
- Agregue 30 μL (1.2x) de perlas resuspendidas a cada pozo de una placa de pozo profundo de 96 pocillos.
- Añadir 25 μL de reacción de PCR a los pocillos que contengan perlas.
- Coloque la placa en un agitador de placas a 2000 rpm durante 2 min.
- Deje que la placa permanezca a temperatura ambiente durante 5 minutos después de la agitación.
- Coloque la placa de pozo profundo en un imán de placa de 96 pocillos e incube durante 2 minutos.
- Retire y deseche el sobrenadante sin las perlas perturbadoras.
- Mientras la placa permanezca en el imán, agregue 180 μL de etanol al 80% e incube durante 30 s. Retire y deseche el sobrenadante.
- Repita el paso 5.1.9.
- Usando una pipeta de 10 μL, retire y deseche cualquier líquido restante de los pozos.
- Deje que las perlas se sequen a temperatura ambiente durante 10 min.
- Agregue 20 μL de agua libre de nucleasa a los pozos que contienen perlas y retire la placa del imán.
- Agite la placa a 2000 rpm durante 2 minutos a temperatura ambiente.
- Incubar la placa a temperatura ambiente durante 5 min.
- Coloque la placa en un soporte magnético e incube durante 2 minutos a temperatura ambiente.
- Retire el sobrenadante en una segunda placa de PCR etiquetada. Esto contiene el ADN limpio.
- Mida la concentración de cada reacción con un fluorómetro.
- Asegúrese de que los reactivos del fluorómetro dsDNA estén a temperatura ambiente.
- Configure tubos de ensayo fluorométricos más dos tubos adicionales para estándares.
- Agregue 199 μL de solución de trabajo 1x dsDNA a todos los tubos excepto a dos. Agregue 190 μL de la solución de trabajo a los dos últimos tubos.
- Agregue 10 μL de los dos estándares (incluidos en la Tabla de Materiales) para separar los tubos de ensayo.
- Agregue 1 μL de cada reacción de PCR a los tubos de mezcla maestra del fluorómetro.
- Tubos de vórtice para mezclar e incubar a temperatura ambiente durante 2 min.
- En la pantalla de inicio del fluorómetro, seleccione el botón con el kitin de ensayo use (1x dsDNA) y luego seleccione Leer estándares y Ejecutar muestras.
- Inserte el tubo estándar 1, seleccione el botón Leer y luego repita para el estándar 2.
- Siguiendo el paso 5.2.6, repita para una muestra, seleccione un volumen de muestra de 1 μL y se proporcionará la concentración resultante.
- Repita el paso 5.2.7 para las muestras restantes.
- Calcule la molaridad proyectada de todas las reacciones y acumule concentraciones iguales de reacciones de cada muestra individual por separado (una piscina final por muestra) utilizando la ecuación a continuación.
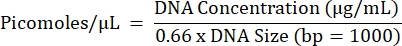
- Repita los pasos 5.2.1 a 5.2.7 para obtener la concentración final de la piscina.
- Compruebe la piscina de amplicón en una máquina de electroforesis capilar / gel de agarosa según lo especificado por el fabricante.
- Preparar tubos de electroforesis capilar para el número adecuado de muestras. Añadir 7 μL del tampón de ADN según lo especificado por el fabricante.
- Agregue 4 μL de la piscina de amplicón al tubo que contiene el tampón de ADN.
- Coloque tubos en la máquina de electroforesis y haga funcionar la máquina según lo especificado por el fabricante para dsDNA.
- Vea las imágenes del gel de electroforesis asegurando que las bandas se localicen a ~ 5 kb (tamaño de los amplicones).
- Calcule la molaridad proyectada de todas las reacciones y acumule concentraciones iguales de reacciones de cada muestra individual por separado (una piscina final por muestra) utilizando la ecuación a continuación.
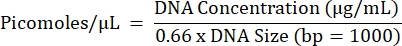
6. Preparación de la biblioteca
- Diluya los grupos de muestras del paso 5.3.1 a 0,2 ng/μL para la preparación de la biblioteca.
- Mediante un kit de preparación de bibliotecas de baja entrada compatible con secuencias cortas (300 pb), prepare bibliotecas utilizando combinaciones de índices únicas para cada grupo de muestras creado en el paso 5.3 siguiendo las instrucciones del fabricante.
NOTA: Siga las instrucciones del fabricante para seleccionar secuencias de índice. Todos los índices susceptibles al kit de preparación de la biblioteca funcionarán para las muestras. - Después de la preparación de la biblioteca, agrupe todas las muestras de acuerdo con las instrucciones del fabricante.
NOTA: Antes de crear el grupo de bibliotecas, calcule el número de lecturas necesarias para una cobertura de 250 veces de la secuencia de destino y asegúrese de que el cartucho de secuenciación seleccionado pueda proporcionar una cobertura adecuada para cada muestra incluida. Para un total de 0.5Mb, esto equivaldrá a 1M de lecturas.
- Mediante un kit de preparación de bibliotecas de baja entrada compatible con secuencias cortas (300 pb), prepare bibliotecas utilizando combinaciones de índices únicas para cada grupo de muestras creado en el paso 5.3 siguiendo las instrucciones del fabricante.
- Prepare el grupo de bibliotecas para la secuenciación.
- Descongele y prepare un cartucho de 300 ciclos y reactivos de secuenciación.
- Desnaturalizar y diluir el grupo de secuenciación creado en el paso 5.1.1 de acuerdo con las instrucciones del fabricante del secuenciador.
- Agregue el grupo de bibliotecas desnaturalizadas y diluidas a los reactivos de secuenciación y ejecute la máquina de secuenciación según lo especificado por el fabricante.
NOTA: Consulte la Tabla 3 adjunta para la resolución de problemas.
7. Análisis de datos
NOTA: Todos los pasos de datos se realizan en el software de análisis de datos genómicos. Los paréntesis indican la entrada del usuario. El signo mayor que indica el orden de los clics del mouse para cualquier paso dado (por ejemplo,1er clic del mouse>2do clic del mouse)
- Importe las lecturas haciendo clic en Abrir software > Importar > Illumina > Seleccionar archivos > Siguiente > Seleccionar ubicación para guardar > finalizar. Las lecturas aparecerán ahora en el software.
- Recortar y filtrar las lecturas.
- En el software de análisis de datos de secuencia profunda, recorte los parámetros predeterminados de lecturas sin procesar.
- Resalte las lecturas y haga clic en Cuadro de herramientas > Preparar datos de secuenciación > Recortar lecturas > siguiente > siguiente > siguiente > siguiente > Guardar > siguiente > (Seleccionar ubicación para guardar) > Finalizar
- Asigne las lecturas recortadas a la referencia.
- El mapa recortado se lee a la secuencia de referencia utilizada en el paso 4.1 utilizando una puntuación de coincidencia de 2, un costo de desajuste de 3 y costos de inserción/eliminación de 2. Asegúrese de que la fracción de longitud esté por encima de 0,7 y que la fracción de similitud esté en o por encima de 0,8.
- Resalte el archivo de lectura recortado y haga clic en Caja de herramientas > Análisis de resecuenciación > lecturas de mapa para hacer referencia > siguiente > (Seleccionar secuencia de referencia) > siguiente > (Asegúrese de que los parámetros se indican como se indica anteriormente) > Siguiente > Guardar > (Seleccionar ubicación para guardar) > Finalizar.
- Extraer variantes e indels.
- Con un llamador indel apropiado, extraiga indels usando un umbral de valor p de 0.005 o inferior y un número máximo de desajustes de 3.
- Resalte el archivo de lectura asignado y haga clic en Caja de herramientas > análisis de resecuenciación > detección de variantes > Indels y variantes estructurales > siguiente > (Asegúrese de que la importancia requerida sea la entrada > Siguiente > Guardar > (Seleccionar ubicación para guardar) > Finalizar.
- Usando una variante apropiada para llamar, llame a variantes de la asignación de lectura usando una significación del 5%.
- Resalte el archivo de lectura asignado y haga clic en Caja de herramientas > Análisis de resecuenciación > Detección de variantes > Detección de variantes de baja frecuencia > siguiente > (Asegúrese de que la importancia requerida sea la entrada > Siguiente > Siguiente > Guardar > (Seleccionar ubicación para guardar) > Finalizar.
NOTA: Asegúrese de tener en cuenta la ploidía del genoma del huésped en las personas que llaman indel y variante. No extraiga mutantes por debajo del 5%. Este umbral tiene en cuenta los errores de PCR y secuenciación asociados con el ensayo. La normalización (basada en la detección de inmunoblot de CAS9) debe realizarse ajustando la cobertura de secuenciación. Por ejemplo, si la muestra A tiene el doble de la eficiencia de transfección de la muestra B, entonces el 50% de las lecturas de la muestra A deben usarse para el análisis. Esto debe hacerse mediante muestreo aleatorio y no reducir la cobertura para ninguna muestra por debajo de 100x.
Resultados
Se presentan tres resultados representativos para este protocolo. La Figura 1 es una expresión inmunoblot que confirma la expresión de CAS9 en el control de HFK (LXSN) y HFK que expresa beta-HPV 8E6 (8E6). 48 h después de la transfección, se recolectaron lisados de células enteras y posteriormente se sondearon con un anticuerpo anti-CAS9 (o GAPDH como control de carga). El resultado muestra que HFK LXSN y HFK 8E6 están expresando una cantidad similar de...
Discusión
Además de la profundidad de la información proporcionada, hay varias ventajas de este método. En primer lugar, la reparación de DSB, en teoría, se puede evaluar en cualquier loci genómico sin modificar el genoma de la célula de interés. En segundo lugar, el acceso al análisis ngsano de la reparación se ve incrementado por la reducción del costo y el esfuerzo computacional que ofrece la creación y el análisis de un único OSD dirigido a una zona definida. Finalmente, con los genomas de organismos adicionales ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
La investigación reportada en este manuscrito fue apoyada por el Instituto Nacional de Ciencias Médicas Generales de los Institutos Nacionales de Salud (P20GM130448) (NAW y RP); Instituto Nacional del Cáncer de los Institutos Nacionales de Salud (NCI R15 CA242057 01A1); Centro de Investigación del Cáncer Johnson en la Universidad Estatal de Kansas; y el Departamento de Defensa de los Estados Unidos (CMDRP PRCRP CA160224 (NAW)). Agradecemos a KSU-CVM Confocal Core y a Joel Sanneman por nuestra microscopía de inmunofluorescencia. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente las opiniones oficiales de estas agencias de financiación.
Materiales
| Name | Company | Catalog Number | Comments |
| 6 Well Tissue Culture Plate | Celltreat | 229106 | Cell culture plate |
| BCA Kit | VWR | 89167-794 | BCA assay kit |
| Centrifuge 5910 R | Eppendorf | 2231000772 | Tabletop Centrifuge |
| CLC Genomic Workbench | Qiagen | 832001 | deep sequence data analysis software/indel caller/variant caller |
| Digital Microplate Genie pulse | Scientific industries | SI-400A | Plate shaker |
| DYKDDDDK Tag Monoclonal Antibody (FG4R) | ThermoFisher Scientific | MA191878 | Anti-FLAG antibody |
| Epilife CF Kit | ThermoFisher Scientific | MEPICF500 | Cell cultrue media and supplements |
| Fetal Bovine Serum (FBS) | VWR | 89510-194 | Cell culture supplement |
| Goat anti-Rabbit IgG | ThermoFisher Scientific | A-11012 | Secondary antibody |
| HighPrep PCR Clean-up system | MagBio | AC-60005 | Bead-based PCR cleanup kit |
| KAPA HiFi HotStart ReadyMix PCR Kit | KAPA Biosystems | KK2600 | PCR mastermix/PCR assay |
| MagAttract HMW DNA kit | Qiagen | 67563 | High Molecular Weight DNA extraction kit |
| Magnetic Stand-96 | Thermo Fisher Scientific | AM10027 | 96-Well Magnetic Rack |
| MiniAmp Thermal Cycler | Applied Biosystems | A37834 | Thermal Cycler |
| Miseq | Illumina | SY-410-1003 | Sequencer |
| Miseq v2 300 cycle reagent kit | Illumina | MS-102-2002 | 300-cycle cartridge/sequencing reagents |
| Nextera XT DNA Library Prep kit | Illumina | FC-131-1024 | Library preparation kit |
| Nextera XT Kit v2 Set A | Illumina | 20027215 | Indexes |
| Nunc 96-well polypropylene DeepWell Stroage plates | Thermo Fisher Scientific | 260251 | deep well 96-well plates |
| Penicillin-Streptomycin Solution (100X) | Calsson Labs | PSL02-6X100ML | Antibiotics for cell culture |
| Phosphate Buffered Saline (PBS) | Bio Basic | PD8117 | PBS |
| px330-CD4 | Addgen | 136938 | SgRNA/CAS9 plasmids targeting 5’- GGCGTATCTGTGTGAGGACT |
| QIAxcel Advanced System | Qiagen | 9001941 | capillary electrophersis machine |
| QIAxcel DNA screening kit | Qiagen | 929004 | DNA buffer/ capillary electrophersis tubes |
| Qubit 1x ds HS Assay Kit | ThermoFisher Scientific | Q23851 | Fluorometer reagents/1x dsDNA solution |
| Qubit 4 Fluorometer | ThermoFisher Scientific | Q33238 | Fluorometer |
| Qubit Assay Tubes | Thermo Fisher Scientific | Q32856 | Fluorometer assay tubes |
| RIPA Lysis Buffer | VWR | VWRVN653-100ML | Lysis buffer for protein extraction |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | Trypsin |
| Vortex-Genie 2 | Scientific industries | SI-0236 | Vortex |
| Xfect Transfection Reagent | Takara Bio | 631318 | Transfection reagent |
| genomic data analysis software | QIAGEN | CLC Workbench v21.0. | Data analysis software |
Referencias
- Vítor, A. C., Huertas, P., Legube, G., de Almeida, S. F. Studying DNA double-strand break repair: an ever-growing toolbox. Frontiers in Molecular Biosciences. 7, (2020).
- Giglia-Mari, G., Zotter, A., Vermeulen, W. DNA damage response. Cold Spring Harbor Perspectives in Biology. 3 (1), 000745 (2011).
- Khanna, K. K., Jackson, S. P. DNA double-strand breaks: Signaling, repair and the cancer connection. Nature Genetics. 27 (3), 247-254 (2001).
- vanden Berg, J. G., et al. A limited number of double-strand DNA breaks is sufficient to delay cell cycle progression. Nucleic Acids Research. 46 (19), 10132-10144 (2018).
- Chang, H. H. Y., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nature Reviews Molecular Cell Biology. 18 (8), 495-506 (2017).
- Daley, J. M., Sung, P. 5. 3. B. P. 1. BRCA1, and the choice between recombination and end joining at DNA double-strand breaks. Molecular and Cellular Biology. 34 (8), 1380-1388 (2014).
- Godin, S. K., Sullivan, M. R., Bernstein, K. A. Novel insights into RAD51 activity and regulation during homologous recombination and DNA replication. Biochemistry and Cell Biology = Biochimie et Biologie Cellulaire. 94 (5), 407-418 (2016).
- Jette, N., Lees-Miller, S. P. The DNA-dependent protein kinase: a multifunctional protein kinase with roles in DNA double strand break repair and mitosis. Progress in Biophysics and Molecular Biology. 117, 194-205 (2015).
- Weterings, E., van Gent, D. C. The mechanism of non-homologous end-joining: A synopsis of synapsis. DNA Repair. 3 (11), 1425-1435 (2004).
- Bhargava, R., Lopezcolorado, F. W., Tsai, L. J., Stark, J. M. The canonical non-homologous end joining factor XLF promotes chromosomal deletion rearrangements in human cells. Journal of Biological Chemistry. 295 (1), 125-137 (2020).
- Gunn, A., Bennardo, N., Cheng, A., Stark, J. M. Correct end use during end joining of multiple chromosomal double strand breaks is influenced by repair protein RAD50, DNA-dependent protein kinase DNA-PKcs, and transcription context. Journal of Biological Chemistry. 286 (49), 42470-42482 (2011).
- Simsek, D., Jasin, M. Alternative end-joining is suppressed by the canonical NHEJ component Xrcc4-ligase IV during chromosomal translocation formation. Nature Structural & Molecular Biology. 17 (4), 410-416 (2010).
- Certo, M. T., et al. Tracking genome engineering outcome at individual DNA breakpoints. Nature Methods. 8 (8), 671-676 (2011).
- Murthy, V., et al. Characterizing DNA repair processes at transient and long-lasting double-strand DNA breaks by immunofluorescence microscopy. JoVE Journal of Visualized Experiments. (136), e57653 (2018).
- Wang, J. L., et al. Dissection of DNA double-strand-break repair using novel single-molecule forceps. Nature Structural & Molecular Biology. , (2018).
- Azzam, E. I., Jay-Gerin, J. -. P., Pain, D. Ionizing radiation-induced metabolic oxidative stress and prolonged cell injury. Cancer Letters. 327, 48-60 (2012).
- Kuo, L. J., Yang, L. -. X. γ-H2AX - A novel biomarker for DNA double-strand breaks. In Vivo. 5, (2008).
- Sanders, J. T., et al. Radiation-induced DNA damage and repair effects on 3D genome organization. Nature Communications. 11 (1), 6178 (2020).
- Bellaiche, Y., Mogila, V., Perrimon, N. I-SceI endonuclease, a new tool for studying DNA double-strand break repair mechanisms in Drosophila. Genetics. 152 (3), 1037-1044 (1999).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. β-HPV 5 and 8 E6 disrupt homology dependent double strand break repair by attenuating BRCA1 and BRCA2 expression and foci formation. PLOS Pathogens. 11 (3), 1004687 (2015).
- Hu, C., Bugbee, T., Gamez, M., Wallace, N. A. Beta human papillomavirus 8E6 attenuates non-homologous end joining by hindering DNA-PKcs activity. Cancers. 12 (9), 2356 (2020).
- Hu, C., Bugbee, T., Dacus, D., Palinski, R., Wallace, N. A. Beta human papillomavirus 8 E6 allows colocalization of non-homologous end joining and homologous recombination repair factors. PLOS Pathogens. 18 (3), 1010275 (2022).
- Butler, T. A. J., Paul, J. W., Chan, E. -. C., Smith, R., Tolosa, J. M. Misleading westerns: Common quantification mistakes in western blot densitometry and proposed corrective measures. BioMed Research International. 2019, 5214821 (2019).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX Phosphorylation on serine 139. Journal of Biological Chemistry. 273 (10), 5858-5868 (1998).
- Taning, C. N. T., Van Eynde, B., Yu, N., Ma, S., Smagghe, G. CRISPR/Cas9 in insects: Applications, best practices and biosafety concerns. Journal of Insect Physiology. 98, 245-257 (2017).
- Ghezraoui, H., et al. Chromosomal translocations in human cells are generated by canonical nonhomologous end-joining. Molecular Cell. 55 (6), 829-842 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados