このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
次世代シーケンシングを使用して、CD4プロモーター近傍のCAS9誘導二本鎖切断の修復に関連する変異を同定
要約
ここでは、CD4プロモーター近傍の二本鎖切断修復に関連する変異を同定するために使用できるsgRNA/CAS9エンドヌクレアーゼおよび次世代シーケンシングプロトコルを紹介します。
要約
DNA中の二本鎖切断(DSB)は、DNA損傷の最も細胞傷害性のあるタイプである。無数の侮辱がこれらの病変(例えば、複製ストレス、電離放射線、修復されていないUV損傷)をもたらす可能性があるため、DSBは毎日ほとんどの細胞で発生します。細胞死に加えて、修復されていないDSBはゲノムの完全性を低下させ、結果として生じる変異は腫瘍形成を促進する可能性がある。これらのリスクとDSBの有病率は、細胞がこれらの病変を修復するメカニズムの調査を動機付ける。次世代シーケンシングは、電離放射線によるDSBの誘導と組み合わせることができ、DSB修復欠陥に関連する変異を正確に定義するための強力なツールを提供します。しかし、このアプローチでは、電離放射線に関連するランダムに発生するDSBの修復を検出するために、計算上困難でコストのかかる全ゲノムシーケンシングが必要です。I-Sce1などの希少切断エンドヌクレアーゼは、単一のDSBを生成する能力を提供するが、それらの認識部位は目的のゲノムに挿入されなければならない。その結果、修復部位は本質的に人工的である。最近の進歩により、ガイドRNA(sgRNA)はCas9エンドヌクレアーゼを目的の任意のゲノム遺伝子座に誘導することができます。これは、Cas9誘導DSBに隣接するDNAに焦点を合わせることを可能にすることによって、次世代シーケンシングをより費用対効果の高いものにするDSB修復の研究に適用できる可能性がある。原稿の目標は、CD4遺伝子の上流のDSBの修復に由来する変異を定義できるプロトコルを提示することによって、このアプローチの実現可能性を実証することである。このプロトコルは、比較的限られた計算要件で、修復阻害剤、ウイルスタンパク質発現、突然変異、および環境曝露などの外因性因子に関連するDSBの変異原性電位の変化を決定するために適合させることができる。生物のゲノムが配列決定されると、この方法は理論的には、任意のゲノム遺伝子座およびトランスフェクト可能なその生物の任意の細胞培養モデルにおいて採用され得る。このアプローチの同様の適応は、同じ遺伝的背景にある異なる遺伝子座間の修復忠実度の比較を可能にする可能性がある。
概要
ゲノムの安定性を維持することは、すべての生物にとって非常に重要です。正確なDNA複製と堅牢なDNA損傷応答(DDR)は、遺伝物質を忠実に伝播するために必要である1,2。DNA損傷は、ほとんどの細胞で定期的に起こる2,3。これらの損傷が感知されると、細胞周期の進行が止まり、DNA修復機構が活性化される。DNAまたはDSBの二本鎖切断は、DNA損傷の中で最も有毒で変異原性のあるタイプである3,4。
いくつかのDDRシグナル伝達経路がこれらの病変を修復することができるが、最も徹底的に研究されたDSB修復経路は相同組換え(HR)および非相同末端結合(NHEJ)である。HRは、姉妹染色分体を相同テンプレートとして使用してDSBを修復する、ほとんどエラーのない経路です。これは、細胞周期5、6、7のS期およびG2期において起こる傾向がある。NHEJはよりエラーが発生しやすいが、細胞周期全体を通して起こり得る8,9。特異的修復機構の効率を測定するために様々なレポーターアッセイが開発されている10、11、12。これらのアッセイは、GFPまたはmCherryを読み出しとして用いたDSB修復経路活性のハイスループット測定のためにフローサイトメトリーに依存する傾向がある11、13。非常に効率的ですが、人工的に導入されたDSBで発生する標準的な修復に依存しています。
DSB修復を研究するために使用される他の様々な方法があります。これらの多くは、免疫蛍光(IF)顕微鏡1,14に依存している。IF顕微鏡は、DSBが遺伝毒性化学物質または電離放射線への曝露によって誘導された後の修復複合体を代表する離散核病巣を検出する15,16。これらの病巣の形成および解決を追跡することは、それぞれ修復の開始および完了の指標を提供する14、17。しかしながら、DSB誘導のこれらの方法(すなわち、化学物質または電離放射線)は、ゲノム中の定義された位置でDSBを引き起こさない。また、それらを使用して、少数のDSBのみ(例えば、2〜4個)のみを一貫して誘導することも機能的に不可能である。その結果、DSBを誘導する最も一般的に使用される方法は、ゲノム全体にランダムに分布する多数の病変を引き起こす18。希少切断エンドヌクレアーゼの認識部位を挿入し、I-Sce119などの適切なエンドヌクレアーゼを発現させることにより、少数のDSBを導入することができる。残念なことに、標的部位の必要な統合は、内因性ゲノム遺伝子座におけるDSBの検査を妨げる。
本稿では、ユーザ定義遺伝子座で発生したDSBの修復に関連する変異を検出する方法を記載する。我々は、DSBに関連する変異の数を増加させるウイルスタンパク質の能力を評価するために適用されるアプローチの代表例を提供する。具体的には、この原稿は、ヒト包皮ケラチノサイトにおけるヒトCD4オープンリーディングフレームでDSBを誘導するためにCAS9エンドヌクレアーゼを指示するための単一のガイドRNA(sgRNA)の使用を記載するベクターコントロール(HFK LXSN)およびヒトパピローマウイルス8型(HFK 8E6)のE6タンパク質を発現するHFKを発現する。ブレークを取り囲む領域の標的次世代シーケンシング(NGS)は、病変の修復に関連する変異を厳密に定義することを可能にする。これらのデータは、ウイルスタンパク質がDSB修復中に変異の約20倍の増加を引き起こすことを実証している。また、全ゲノムシーケンシングを必要とせずに、単一の遺伝子座におけるDSBの変異原性の影響を偏りなく特徴付けます。原則として、このプロトコルは、ゲノム遺伝子座または細胞株間の突然変異の相対リスクを比較するために容易に適合させることができる。
プロトコル
1. セルプレーティング
- ヒトケラチノサイト増殖サプリメント(HKGS)および1%ペニシリン/ストレプトマイシンを含むケラチノサイト培養培地(10mL/プレート)中の10cmプレートでHFK LXSNおよびHFK 8E6細胞を増殖させる。5%CO2を有するジャケットインキュベーター中で37°Cで約80%コンフルエントまで細胞を増殖させる。
- 培養培地を3mLのトリプシン-EDTA(0.05%、エチレンジアミン四酢酸)と交換する。37°Cで3分間インキュベートする。等量のウシ胎児血清(FBS)添加培地でトリプシンを中和し、細胞を15mL遠沈管に移す。300 x g で5分間遠心分離する。
- 細胞をHKGSで10mLのケラチノサイト培養培地で再懸濁する。血球計数器で細胞の濃度を決定する。
- HKGSおよび1%ペニシリンおよびストレプトマイシンを含む4mLのケラチノサイト培養培地中のプレート4 x 105細胞/ 6cmプレート(HFK LXSN用のシード2プレートおよびHFK 8E6用の2つのプレート)をプレート4 x105 細胞/ 6cmプレート。5%CO2を有するジャケットインキュベーター中で37°Cで増殖させる。
注:HFK細胞の分析は、2つの理由でこのプロトコルのために選択された。第1に、HFKはトランスフェクトが困難な細胞株である。したがって、このプロトコールがこの細胞株において機能することを実証することによって、より一般的に使用され、より容易にトランスフェクトされた細胞において作用する可能性が高いという証拠が提供される。第二に、以前に発表されたデータは、ウイルスタンパク質(8E6)がDNA20、21、22の二本鎖切断の修復を妨げることを示している。したがって、HFK LXSNとHFK 8E6を比較することで、細胞修復能力の低下に関連する変異の増加を検出するアッセイの能力を実証することができます。
2. トランスフェクション
- トランスフェクション当日(めっき後24時間)に、培地を抗生物質を含まない3 mLの補充培地と交換してください。ジャケットインキュベーター内で37°Cで2時間インキュベートする。
- 適切な脂質ベースのトランスフェクション試薬で細胞をトランスフェクトし、製造元の指示に従ってください。
- トランスフェクション試薬を室温に温め、使用前に穏やかにピペットを挽回します。
- 各細胞株(HFK LXSNおよびHFK 8E6)について、適量のトランスフェクションバッファー(製造業者の指示により)を滅菌1.5mL遠沈管(チューブ1)に入れます。同じ量のトランスフェクションバッファー(モックトランスフェクションまたはチューブ2)を含む別のチューブを含めます。
- ヒトCD4を標的とするCAS9/sgRNAを発現するプラスミドDNAを2 μg(px330-CD4, 5'-GGCGTATCTGTGTGAGGACT)をステップ2.2.2からチューブ1に加える。ピペットで優しく混ぜて完全に混ぜる。チューブ2に等量の滅菌水を加える。
- 各細胞株についてトランスフェクション試薬単独(プラスミドなし)を備えたコントロールプレートを含む。
注:2番目のプレートは実験においてネガティブコントロールとして機能し、CAS9/sgRNAによるトランスフェクションがいかなる変異にも責任を負わないことをユーザーが確認できるようにします。 - ステップ 2.2.3 の DNA 混合物 (チューブ 1) およびステップ 2.2.2 のモックトランスフェクション (チューブ 2) を含むチューブに、適量のトランスフェクション試薬 (メーカーの指示に従って) を追加します。ピペットで優しく混ぜて完全に混ぜる。複合体が形成されるのに十分な時間を可能にするために、室温で15〜30分間インキュベートする。
- トランスフェクション混合物をプレートに滴下して加える。培養液を 1 分間穏やかに揺らして、トランスフェクション混合物を均等に分配します。
- トランスフェクション後48時間インキュベートして、CAS9発現を可能にする。
- トリプシン処理によって細胞を回収する。
- 培養培地を1mLのトリプシン-EDTA(0.05%、エチレンジアミン四酢酸)と交換する。37°Cで3分間インキュベートする。等量のFBS添加培地でトリプシンを中和する。
- 細胞の各プレートについて、細胞懸濁液を等しいアリコートを有する2つの微量遠心チューブに移す。300 x g で5分間遠心分離する。
- ステップ2.4.2で細胞ペレットを1本のチューブから1mLのリン酸緩衝生理食塩水(PBS)に再懸濁し、シークエンシングを行う。ステップ2.4.2からもう一方のチューブをイムノブロット用の氷冷PBSで再懸濁する。
- イムノブロットのために全細胞溶解物を回収する。
- チューブを300 x g で5分間遠心分離します。上清を捨てる。
- 1%プロテアーゼ阻害剤および1%ホスファターゼ阻害剤と混合した100μLの放射免疫沈降アッセイバッファー(RIPA溶解バッファー)をチューブに加え、ピペットで十分に混合し、氷上で10分間インキュベートする。
注:RIPA溶解緩衝液は、10mMトリス塩酸、pH8.0を含有する。1 mM EDTA;0.5 ミリグラムのエグタ;1% トリトン X-100;0.1%デオキシコール酸ナトリウム;0.1% SDS;140mM NaCl、及び脱イオン水。 - 遠心分離機溶解物を13,000 x g で10分間遠心分離する。イムノブロットのために上清を集める。
3. イムノブロットによるCAS9発現の測定
- 製造元の指示に従ってビシンコニン酸(BCA)アッセイでタンパク質濃度を決定します。
- 各サンプルのタンパク質20μgを3%-8%トリス酢酸ゲルのウェルで150分間実行し、ポリフッ化ビニリデン膜に半乾式移送(10Vで30分間、次に25Vで12分間)します。
- PBS中の5%脱脂粉乳中の膜を0.1%トゥイーン(PBST)で室温で1時間ブロッキングした後、抗CAS9(1:1000)および抗GAPDH(1:1000)抗体を加える。4°Cで一晩インキュベートする。
- PBSTでメンブレンを洗浄した後、PBST中の5%脱脂粉乳中の二次抗体と共にメンブレンを室温で1時間インキュベートする。
- ブロットを画像化し、デンシトメトリー23によってCAS9レベルを決定する。代表的なブロットについては、 図1 を参照してください。
注:免疫蛍光顕微鏡によるリン酸化H2AX(S139)病巣形成の検出は、CAS9活性14を検証するために使用することができる。細胞周期の位置、CAS9標的部位の変異がさらなる切断を妨げるかどうか、およびCAS9切断部位のコピーが目的のゲノムに存在するかに応じて、少数の異なる病巣(典型的には1〜4個の病巣)が予想される。代表的な画像を 図2に示す。
4. 核酸抽出とアンプリコン生成
- 製造元が指定する高分子量 DNA 抽出キットを使用して、ステップ 2.4.3 の細胞サンプルから DNA を抽出します。
- データシートに従って、示された溶媒でプライマーを再懸濁する。同じ試薬で20 μMに希釈し、20 μMのプライマーを示されたプールにプールします。
注:プライマープールは 補足表1にリストされている。 - 表1に規定されているように、各20μMプライマープールに対して長増幅Taqポリメラーゼを使用してPCRマスターミックスを作成します。
- 21 μL のマスターミックスを別々の PCR チューブに加えます。
- ステップ 4.1 のターゲットサンプル 4 μL (100 ng/μL) を、マスターミックスおよびキャップアッセイチューブを含む PCR アッセイチューブに加えます。プライマープールごとに別々の反応を確認してください。
- ボルテックスは、PCRアッセイチューブと遠心分離機(クイックスピン)を混合して、チューブの蓋から液滴を除去します。
- PCRチューブを従来のサーマルサイクラーマシンの上に置きます。
- 表2に規定されているようにPCRマシンをプログラムする。
- サーマルサイクラーでプログラムを実行します。
5. PCR クリーンアップ
- ビーズベースのPCRクリーンアップシステムを用いてPCR反応からプライマーを除去します。
- 使用の30分前に冷蔵庫からクリーンアップビーズを取り出してください。
- 使用前にボルテックスビーズを十分に準備し、すべてのビーズが再懸濁されていることを確認してください。
- 30 μL (1.2x) の再懸濁ビーズをディープウェル 96 ウェルプレートの各ウェルに加えます。
- ビーズを含むウェルに25 μLのPCR反応を加える。
- プレートをプレートシェーカー上に2000rpmで2分間置く。
- 振とう後、プレートを5分間室温にとどめます。
- ディープウェルプレートを96ウェルプレート磁石の上に置き、2分間インキュベートする。
- 邪魔なビーズなしで上清を取り除き、捨てる。
- プレートを磁石上に残したまま、180μLの80%エタノールを加え、30秒間インキュベートする。上清を取り除いて捨てる。
- 手順 5.1.9 を繰り返します。
- 10 μL のピペットを使用して、ウェルから残りの液体を取り出して廃棄します。
- ビーズを室温で10分間乾燥させます。
- ビーズを含むウェルにヌクレアーゼ自由水20 μLを加え、プレートを磁石から取り出します。
- プレートを室温で2000rpmで2分間振る。
- プレートを室温で5分間インキュベートする。
- プレートをマグネットスタンドに置き、室温で2分間インキュベートする。
- 上清を第2の標識PCRプレートに除去する。これには、クリーンアップされたDNAが含まれています。
- 各反応の濃度を蛍光光度計で測定する。
- dsDNA蛍光光度計試薬が室温であることを確認します。
- 蛍光測定アッセイチューブと、標準用に 2 本のチューブを追加でセットアップします。
- 199 μL の 1x dsDNA 作業溶液を 2 本を除くすべてのチューブに加えます。最後の2本のチューブに190μLの作業溶液を加える。
- 2つの標準物質( 材料表に含まれる)を10 μL加えて、アッセイチューブを分離します。
- 各PCR反応の1 μLを蛍光光度計マスターミックスチューブに加えます。
- ボルテックスチューブを混合し、室温で2分間インキュベートする。
- 蛍光光度計のホーム画面で、アッセイキットの使用(1x dsDNA)のボタンを選択し、[ 標準の読み取り]および[サンプルの実行]を選択します。
- 標準 1 本のチューブを挿入し、[ 読み取り ] ボタンを選択してから、標準 2 に対して繰り返します。
- ステップ5.2.6に続いて、1つのサンプルに対して繰り返し、1μLのサンプル容量を選択すると、結果として得られる濃度が提供されます。
- 残りのサンプルについて、手順 5.2.7 を繰り返します。
- 以下の式を使用して、個々のサンプルからのすべての反応およびプールの等しい濃度の反応の投影モル濃度を別々に計算します(サンプルごとに1つの最終プール)。
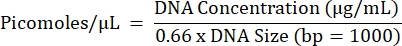
- 手順 5.2.1 ~ 5.2.7 を繰り返して、最終プール濃度を取得します。
- 製造元の指定に従って、キャピラリー電気泳動機/アガロースゲル上のアンプリコンプールを確認してください。
- 適切なサンプル数用のキャピラリー電気泳動チューブを用意します。製造元の指定に従って 7 μL の DNA バッファーを追加します。
- 4 μLのアンプリコンプールをDNAバッファーを含むチューブに加える。
- 電気泳動機にチューブを置き、dsDNAの製造元が指定したとおりに機械を稼働させます。
- 電気泳動ゲルの写真を見て、バンドが約5 kb(アンプリコンのサイズ)に局在することを確認します。
- 以下の式を使用して、個々のサンプルからのすべての反応およびプールの等しい濃度の反応の投影モル濃度を別々に計算します(サンプルごとに1つの最終プール)。
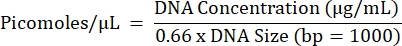
6. 図書館の準備
- ライブラリー調製のために、サンプルプールをステップ 5.3.1 から 0.2 ng/μL に希釈します。
- 短いシーケンス(300bp)と互換性のある低入力ライブラリ準備キットを使用して、製造元の指示に従って、ステップ5.3で作成した各サンプルプールに対して一意のインデックスの組み合わせを使用してライブラリを準備します。
メモ: 製造元の指示に従って、インデックスシーケンスを選択します。ライブラリ準備キットに適合するすべてのインデックスは、サンプルに対して機能します。 - ライブラリの準備に続いて、製造元の指示に従ってすべてのサンプルをプールします。
メモ: ライブラリプールを作成する前に、ターゲットシーケンスの 250 倍のカバレッジに必要な読み取り数を計算し、選択したシーケンシングカートリッジが付属サンプルごとに十分なカバレッジを提供できることを確認してください。合計が 0.5 Mb の場合、これは 1M の読み取りに相当します。
- 短いシーケンス(300bp)と互換性のある低入力ライブラリ準備キットを使用して、製造元の指示に従って、ステップ5.3で作成した各サンプルプールに対して一意のインデックスの組み合わせを使用してライブラリを準備します。
- シーケンス処理のためにライブラリー・プールを準備します。
- 解凍し、300サイクルカートリッジとシーケンシング試薬を準備します。
- 手順 5.1.1 で作成したシーケンシングプールを、シーケンサーの製造元の指示に従って変性および希釈します。
- 変性および希釈されたライブラリープールをシーケンシング試薬に追加し、製造元の指定に従ってシーケンシングマシン を実行します 。
メモ:トラブルシューティングについては、添付 の表3 を参照してください。
7. データ解析
注:すべてのデータステップは、ゲノムデータ解析ソフトウェアで実行されます。括弧はユーザー入力を示します。より大きい記号は、任意のステップ(例えば、1回目の マウスクリック>2回目の マウスクリック)のマウスクリックの順序を示す。
- [ソフトウェアを開く]をクリックして読み取りをインポートし、> Illumina>インポート>、次にファイルを選択し、>>保存する場所を選択>ます。読み取りがソフトウェアに表示されます。
- 読み取りをトリミングしてフィルター処理します。
- ディープシーケンスデータ解析ソフトウェアで、生の読み取りデフォルトパラメータをトリミングします。
- 読み取りを強調表示し、[ツールボックス] をクリックして [シーケンス データの準備] > [読み取り>トリム] > [次へ] > [次へ] > [次へ] > [次へ] > [保存] > [次の> (保存する場所を選択) > 終了] をクリックします。
- トリミングされた読み取りを参照にマップします。
- マップトリミングされた読み取りは、一致スコア 2、不一致コスト 3、挿入/削除コスト 2 を使用して、ステップ 4.1 で使用した参照シーケンスにマップします。長さ分数が0.7を超え、類似度分数が0.8以上であることを確認します。
- トリミングされた読み取りファイルを強調表示し、[ツールボックス] > [ 再順序付け解析] > [読み取りを参照>にマップ] [次> > (参照シーケンスの選択)] > [次へ] (パラメーターが上記のように表示されているこ>を確認) > [次へ] > [保存先を選択] > [完了] をクリックします。
- バリアントとインデルを抽出します。
- 適切な indel 呼び出し元を使用して、p 値のしきい値が 0.005 以下で、不一致の最大数が 3 である indels を抽出します。
- マップされた読み取りファイルを強調表示し、[ ツールボックス] をクリックして [再配列決定解析] > [インデルス>構造バリアントの検出> 次の>] をクリックします (必要な有意性が入力されていることを確認する > [次へ] > [保存する場所を選択] > [完了] >>します。
- 適切なバリアント呼び出し元を使用して、5% の有意性を使用して読み取りマッピングからバリアントを呼び出します。
- マップされた読み取りファイルを強調表示し、[ ツールボックス] をクリックして [再配列決定解析] > [バリアント検出> 低頻度バリアント検出> 次の>] をクリックします (必要な有意性が入力されているこ>を確認 > [次へ] > [次へ] > [>を保存] (保存する場所を選択) > [完了] をクリックします。
注:インデルおよびバリアントの呼び出し元における宿主ゲノムの倍数性を考慮するようにしてください。5%未満の変異体を抽出しないでください。この閾値は、アッセイに関連するPCRおよびシーケンシングエラーを説明する。正規化(CAS9のイムノブロット検出に基づく)は、シーケンシングカバレッジを調整することによって行う必要があります。たとえば、サンプル A のトランスフェクション効率がサンプル B の 2 倍の場合、サンプル A からの読み取り値の 50% を分析に使用する必要があります。これはランダムサンプリングによって行うべきであり、100x未満のサンプルのカバレッジを低下させないでください。
結果
このプロトコルの 3 つの代表的な結果を示します。図1は、β-HPV 8E6(8E6)を発現するHFKコントロール(LXSN)およびHFKにおけるCAS9の発現を確認したイムノブロットである。トランスフェクションの48時間後、全細胞ライセートを回収し、続いて抗CAS9抗体(またはローディングコントロールとしてGAPDH)でプローブした。結果は、HFK LXSNおよびHFK 8E6が同量のC...
ディスカッション
提供される情報の深さに加えて、この方法にはいくつかの利点があります。第1に、DSB修復は、理論的には、目的の細胞のゲノムを改変することなく任意のゲノム遺伝子座で評価することができる。第二に、修理のNGS分析へのアクセスは、定義された領域をターゲットにした単一のDSBを作成して分析することによって得られるコストと計算労力の削減によって増加します。最後に、追加の生物...
開示事項
著者らは開示するものは何もありません。
謝辞
この原稿で報告された研究は、国立衛生研究所(P20GM130448)(NAWおよびRP)の国立一般医学研究所によって支援されました。国立衛生研究所の国立がん研究所 (NCI R15 CA242057 01A1);カンザス州立大学のジョンソンがん研究センター;米国国防総省(CMDRP PRCRP CA160224(NAW))です。KSU-CVM Confocal CoreとJoel Sannemanの免疫蛍光顕微鏡検査に感謝します。コンテンツは著者の責任であり、必ずしもこれらの資金提供機関の公式見解を表すものではありません。
資料
| Name | Company | Catalog Number | Comments |
| 6 Well Tissue Culture Plate | Celltreat | 229106 | Cell culture plate |
| BCA Kit | VWR | 89167-794 | BCA assay kit |
| Centrifuge 5910 R | Eppendorf | 2231000772 | Tabletop Centrifuge |
| CLC Genomic Workbench | Qiagen | 832001 | deep sequence data analysis software/indel caller/variant caller |
| Digital Microplate Genie pulse | Scientific industries | SI-400A | Plate shaker |
| DYKDDDDK Tag Monoclonal Antibody (FG4R) | ThermoFisher Scientific | MA191878 | Anti-FLAG antibody |
| Epilife CF Kit | ThermoFisher Scientific | MEPICF500 | Cell cultrue media and supplements |
| Fetal Bovine Serum (FBS) | VWR | 89510-194 | Cell culture supplement |
| Goat anti-Rabbit IgG | ThermoFisher Scientific | A-11012 | Secondary antibody |
| HighPrep PCR Clean-up system | MagBio | AC-60005 | Bead-based PCR cleanup kit |
| KAPA HiFi HotStart ReadyMix PCR Kit | KAPA Biosystems | KK2600 | PCR mastermix/PCR assay |
| MagAttract HMW DNA kit | Qiagen | 67563 | High Molecular Weight DNA extraction kit |
| Magnetic Stand-96 | Thermo Fisher Scientific | AM10027 | 96-Well Magnetic Rack |
| MiniAmp Thermal Cycler | Applied Biosystems | A37834 | Thermal Cycler |
| Miseq | Illumina | SY-410-1003 | Sequencer |
| Miseq v2 300 cycle reagent kit | Illumina | MS-102-2002 | 300-cycle cartridge/sequencing reagents |
| Nextera XT DNA Library Prep kit | Illumina | FC-131-1024 | Library preparation kit |
| Nextera XT Kit v2 Set A | Illumina | 20027215 | Indexes |
| Nunc 96-well polypropylene DeepWell Stroage plates | Thermo Fisher Scientific | 260251 | deep well 96-well plates |
| Penicillin-Streptomycin Solution (100X) | Calsson Labs | PSL02-6X100ML | Antibiotics for cell culture |
| Phosphate Buffered Saline (PBS) | Bio Basic | PD8117 | PBS |
| px330-CD4 | Addgen | 136938 | SgRNA/CAS9 plasmids targeting 5’- GGCGTATCTGTGTGAGGACT |
| QIAxcel Advanced System | Qiagen | 9001941 | capillary electrophersis machine |
| QIAxcel DNA screening kit | Qiagen | 929004 | DNA buffer/ capillary electrophersis tubes |
| Qubit 1x ds HS Assay Kit | ThermoFisher Scientific | Q23851 | Fluorometer reagents/1x dsDNA solution |
| Qubit 4 Fluorometer | ThermoFisher Scientific | Q33238 | Fluorometer |
| Qubit Assay Tubes | Thermo Fisher Scientific | Q32856 | Fluorometer assay tubes |
| RIPA Lysis Buffer | VWR | VWRVN653-100ML | Lysis buffer for protein extraction |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | Trypsin |
| Vortex-Genie 2 | Scientific industries | SI-0236 | Vortex |
| Xfect Transfection Reagent | Takara Bio | 631318 | Transfection reagent |
| genomic data analysis software | QIAGEN | CLC Workbench v21.0. | Data analysis software |
参考文献
- Vítor, A. C., Huertas, P., Legube, G., de Almeida, S. F. Studying DNA double-strand break repair: an ever-growing toolbox. Frontiers in Molecular Biosciences. 7, (2020).
- Giglia-Mari, G., Zotter, A., Vermeulen, W. DNA damage response. Cold Spring Harbor Perspectives in Biology. 3 (1), 000745 (2011).
- Khanna, K. K., Jackson, S. P. DNA double-strand breaks: Signaling, repair and the cancer connection. Nature Genetics. 27 (3), 247-254 (2001).
- vanden Berg, J. G., et al. A limited number of double-strand DNA breaks is sufficient to delay cell cycle progression. Nucleic Acids Research. 46 (19), 10132-10144 (2018).
- Chang, H. H. Y., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nature Reviews Molecular Cell Biology. 18 (8), 495-506 (2017).
- Daley, J. M., Sung, P. 5. 3. B. P. 1. BRCA1, and the choice between recombination and end joining at DNA double-strand breaks. Molecular and Cellular Biology. 34 (8), 1380-1388 (2014).
- Godin, S. K., Sullivan, M. R., Bernstein, K. A. Novel insights into RAD51 activity and regulation during homologous recombination and DNA replication. Biochemistry and Cell Biology = Biochimie et Biologie Cellulaire. 94 (5), 407-418 (2016).
- Jette, N., Lees-Miller, S. P. The DNA-dependent protein kinase: a multifunctional protein kinase with roles in DNA double strand break repair and mitosis. Progress in Biophysics and Molecular Biology. 117, 194-205 (2015).
- Weterings, E., van Gent, D. C. The mechanism of non-homologous end-joining: A synopsis of synapsis. DNA Repair. 3 (11), 1425-1435 (2004).
- Bhargava, R., Lopezcolorado, F. W., Tsai, L. J., Stark, J. M. The canonical non-homologous end joining factor XLF promotes chromosomal deletion rearrangements in human cells. Journal of Biological Chemistry. 295 (1), 125-137 (2020).
- Gunn, A., Bennardo, N., Cheng, A., Stark, J. M. Correct end use during end joining of multiple chromosomal double strand breaks is influenced by repair protein RAD50, DNA-dependent protein kinase DNA-PKcs, and transcription context. Journal of Biological Chemistry. 286 (49), 42470-42482 (2011).
- Simsek, D., Jasin, M. Alternative end-joining is suppressed by the canonical NHEJ component Xrcc4-ligase IV during chromosomal translocation formation. Nature Structural & Molecular Biology. 17 (4), 410-416 (2010).
- Certo, M. T., et al. Tracking genome engineering outcome at individual DNA breakpoints. Nature Methods. 8 (8), 671-676 (2011).
- Murthy, V., et al. Characterizing DNA repair processes at transient and long-lasting double-strand DNA breaks by immunofluorescence microscopy. JoVE Journal of Visualized Experiments. (136), e57653 (2018).
- Wang, J. L., et al. Dissection of DNA double-strand-break repair using novel single-molecule forceps. Nature Structural & Molecular Biology. , (2018).
- Azzam, E. I., Jay-Gerin, J. -. P., Pain, D. Ionizing radiation-induced metabolic oxidative stress and prolonged cell injury. Cancer Letters. 327, 48-60 (2012).
- Kuo, L. J., Yang, L. -. X. γ-H2AX - A novel biomarker for DNA double-strand breaks. In Vivo. 5, (2008).
- Sanders, J. T., et al. Radiation-induced DNA damage and repair effects on 3D genome organization. Nature Communications. 11 (1), 6178 (2020).
- Bellaiche, Y., Mogila, V., Perrimon, N. I-SceI endonuclease, a new tool for studying DNA double-strand break repair mechanisms in Drosophila. Genetics. 152 (3), 1037-1044 (1999).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. β-HPV 5 and 8 E6 disrupt homology dependent double strand break repair by attenuating BRCA1 and BRCA2 expression and foci formation. PLOS Pathogens. 11 (3), 1004687 (2015).
- Hu, C., Bugbee, T., Gamez, M., Wallace, N. A. Beta human papillomavirus 8E6 attenuates non-homologous end joining by hindering DNA-PKcs activity. Cancers. 12 (9), 2356 (2020).
- Hu, C., Bugbee, T., Dacus, D., Palinski, R., Wallace, N. A. Beta human papillomavirus 8 E6 allows colocalization of non-homologous end joining and homologous recombination repair factors. PLOS Pathogens. 18 (3), 1010275 (2022).
- Butler, T. A. J., Paul, J. W., Chan, E. -. C., Smith, R., Tolosa, J. M. Misleading westerns: Common quantification mistakes in western blot densitometry and proposed corrective measures. BioMed Research International. 2019, 5214821 (2019).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX Phosphorylation on serine 139. Journal of Biological Chemistry. 273 (10), 5858-5868 (1998).
- Taning, C. N. T., Van Eynde, B., Yu, N., Ma, S., Smagghe, G. CRISPR/Cas9 in insects: Applications, best practices and biosafety concerns. Journal of Insect Physiology. 98, 245-257 (2017).
- Ghezraoui, H., et al. Chromosomal translocations in human cells are generated by canonical nonhomologous end-joining. Molecular Cell. 55 (6), 829-842 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved