Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Visualización y cuantificación de compuestos farmacéuticos dentro de la piel utilizando imágenes de dispersión Raman coherente
En este artículo
Resumen
Se describe una metodología coherente de imágenes de dispersión Raman para visualizar y cuantificar compuestos farmacéuticos dentro de la piel. Este artículo describe la preparación del tejido de la piel (humano y ratón) y la aplicación de formulación tópica, la adquisición de imágenes para cuantificar los perfiles de concentración espaciotemporal y el análisis farmacocinético preliminar para evaluar la administración tópica de fármacos.
Resumen
La farmacocinética cutánea (cPK) después de la aplicación de formulación tópica ha sido un área de investigación de particular interés para que los científicos reguladores y de desarrollo de fármacos comprendan mecánicamente la biodisponibilidad tópica (BA). Las técnicas semiinvasivas, como el pelado con cinta, la microdiálisis dérmica o la microperfusión dérmica de flujo abierto, cuantifican la cPK a macroescala. Si bien estas técnicas han proporcionado un vasto conocimiento de cPK, la comunidad carece de una comprensión mecanicista de la penetración y permeación de ingredientes farmacéuticos activos (API) a nivel celular.
Un enfoque no invasivo para abordar la cPK a microescala es la imagen de dispersión Raman (CRI) coherente, que se dirige selectivamente a las vibraciones moleculares intrínsecas sin la necesidad de etiquetas extrínsecas o modificación química. CRI tiene dos métodos principales: la dispersión Raman anti-Stokes (CARS) coherente y la dispersión Raman estimulada (SRS), que permiten la cuantificación sensible y selectiva de API o ingredientes inactivos. CARS se utiliza típicamente para derivar información estructural de la piel o visualizar el contraste químico. Por el contrario, la señal SRS, que es lineal con la concentración molecular, se utiliza para cuantificar las API o los ingredientes inactivos dentro de las estratificaciones de la piel.
Aunque el tejido de ratón se ha utilizado comúnmente para cPK con CRI, la BA tópica y la bioequivalencia (BE) deben evaluarse en última instancia en el tejido humano antes de la aprobación regulatoria. Este trabajo presenta una metodología para preparar e visualizar la piel ex vivo para ser utilizada en estudios cuantitativos farmacocinéticos de CRI en la evaluación de BA y BE tópicos. Esta metodología permite una cuantificación de API confiable y reproducible dentro de la piel humana y de ratón a lo largo del tiempo. Se cuantifican las concentraciones dentro de los compartimentos ricos en lípidos y pobres en lípidos, así como la concentración total de API a lo largo del tiempo; estos se utilizan para estimaciones de BA a micro y macroescala y, potencialmente, BE.
Introducción
Las metodologías para evaluar la cPK después de la aplicación de medicamentos tópicos se han expandido de los estudios clásicos de pruebas de permeación in vitro (IVPT) 1,2,3,4,5 y la extracción con cinta 6,7,8 a metodologías adicionales como la microperfusión de flujo abierto o la microdiálisis dérmica 9,10,11, 12,13,14. Existen potencialmente varios sitios locales de acción terapéutica dependiendo de la enfermedad de interés. Por lo tanto, puede haber un número correspondiente de metodologías para evaluar la velocidad y el grado en que una API llega al sitio de acción local previsto. Si bien cada una de las metodologías mencionadas anteriormente tiene sus ventajas, la principal desventaja es la falta de información cPK a microescala (es decir, la incapacidad de visualizar a dónde va la API y cómo impregna).
Una metodología no invasiva de interés para estimar ba y BE tópicos es CRI, que se puede dividir en dos modalidades de imagen: CARS y microscopía SRS. Estos métodos Raman coherentes permiten obtener imágenes químicamente específicas de moléculas a través de efectos Raman no lineales. En CRI, dos trenes de pulso láser se enfocan y escanean dentro de una muestra; la diferencia de energía entre las frecuencias láser se establece para apuntar a modos vibratorios específicos de las estructuras químicas de interés. Como los procesos CRI no son lineales, solo se genera una señal en el foco del microscopio, lo que permite obtener imágenes tomográficas farmacocinéticas tridimensionales del tejido. En el contexto de cPK, CARS se ha utilizado para obtener información estructural de tejidos, como la ubicación de estructuras cutáneas ricas en lípidos15. Por el contrario, SRS se ha utilizado para cuantificar la concentración molecular, ya que su señal es lineal con la concentración. Para especímenes de piel ex vivo , es ventajoso llevar a cabo CARS en la dirección epi16 y SRS en el modo de transmisión17. Por lo tanto, las muestras de tejido que son delgadas permitirán la detección y cuantificación de la señal SRS.
Como tejido modelo, la oreja de ratón desnuda presenta varias ventajas con inconvenientes menores. Una ventaja es que el tejido ya tiene ~ 200-300 μm de espesor y no requiere más preparación de muestras. Además, se observan varias estratificaciones de la piel al enfocar axialmente a través de un campo de visión (por ejemplo, estrato córneo, glándulas sebáceas (SG), adipocitos y grasa subcutánea)16,18. Esto permite una estimación preclínica preliminar de las vías de permeación cutánea y estimaciones tópicas de BA antes de pasar a muestras de piel humana. Sin embargo, el modelo de ratón desnudo presenta limitaciones como la dificultad en la extrapolación a escenarios in vivo debido a las diferencias en la estructura de la piel19. Mientras que la oreja de ratón desnuda es un excelente modelo para obtener resultados preliminares, el modelo de piel humana es el estándar de oro. Aunque ha habido varios comentarios sobre la idoneidad y aplicabilidad de la piel humana congelada para recapitular con precisión la cinética de permeación in vivo 20,21,22, el uso de piel humana congelada es un método aceptado para la evaluación de la cinética de permeación API in vitro 23,24,25 . Este protocolo visualiza varias capas de piel en la piel de ratón y humano mientras cuantifica las concentraciones de API dentro de estructuras ricas en lípidos y pobres en lípidos.
Si bien CRI se ha utilizado en numerosos campos para visualizar específicamente compuestos dentro de los tejidos, ha habido esfuerzos limitados para investigar el cPK de los productos farmacéuticos aplicados tópicamente. Para evaluar el BA/BE tópico de los productos tópicos que utilizan CRI, es necesario tener primero un protocolo estandarizado para hacer comparaciones precisas. Los esfuerzos anteriores que utilizan CRI para la administración de medicamentos a la piel han demostrado variabilidad dentro de los datos. Como se trata de una aplicación relativamente nueva de CRI, establecer un protocolo es fundamental para obtener resultados fiables 18,26,27. Este enfoque solo se dirige a un número de onda específico en la región biológica silenciosa del espectro Raman. Sin embargo, la mayoría de las API e ingredientes inactivos tienen cambios Raman dentro de la región de huellas dactilares. Esto ha planteado desafíos anteriormente debido a la señal inherente que surge del tejido en la región de la huella digital. Los recientes avances láser y computacionales han eliminado esta barrera, que también se puede utilizar en combinación con el enfoque presentado aquí28. Este enfoque presentado aquí permite la cuantificación de una API, que tiene un cambio Raman en la región silenciosa (2.000-2.300 cm-1). Esto no se limita a las propiedades fisicoquímicas del fármaco, lo que podría ser el caso de algunas metodologías de monitoreo cPK mencionadas anteriormente29.
El protocolo debe reducir la variabilidad de muestra a muestra en el grosor de la piel para varias preparaciones, ya que las muestras gruesas de piel humana producirán una señal mínima después de la aplicación del producto farmacéutico debido a la dispersión de la luz por la muestra gruesa. Un objetivo de este manuscrito es presentar una metodología de preparación de tejidos que asegure estándares de imágenes reproducibles. Además, el sistema CRI está configurado como se describe para reducir las posibles fuentes de error y minimizar la señal a ruido. Sin embargo, este documento no discutirá los principios rectores y los méritos técnicos del microscopio CRI, ya que esto se ha cubierto anteriormente30. Finalmente, se explora el extenso procedimiento de análisis de datos para permitir la interpretación de los resultados para determinar el éxito o el fracaso de un experimento.
Access restricted. Please log in or start a trial to view this content.
Protocolo
El uso de tejido de oreja de ratón desnudo fue aprobado por el Comité de Uso y Cuidado de Animales Institucionales del Hospital General de Massachusetts (IACUC), mientras que el uso de tejido de piel humana fue aprobado por la Junta de Revisión Institucional del Hospital General de Massachusetts (IRB). De acuerdo con los protocolos de la IACUC, se obtuvieron ratones recién sacrificados de colaboradores con colonias de ratones desnudos. El tejido humano se obtuvo de procedimientos electivos de abdominoplastia en el Hospital General de Massachusetts a través de un protocolo aprobado. Además, los tipos específicos de tejido distintos de la piel abdominal se adquirieron a través de una autoridad de donación de cuerpos, también a través de un protocolo aprobado por el IRB.
1. Preparación del tejido
- Preparación del tejido de la piel de la oreja de ratón desnudo
- Después de adquirir cuerpos de ratón desnudo recién cosechados, retire las orejas con fórceps y tijeras microquirúrgicas. Coloque una oreja en una placa de Petri pequeña (es decir, 35 mm x 10 mm). Coloque el cuerpo del ratón desnudo en una bolsa de riesgo biológico para desecharlo de acuerdo con los protocolos locales de la IACUC.
- Enjuague cada oreja de ratón con solución salina tamponada con fosfato (PBS) y séquela suavemente con un limpiaparabrisas de tareas. Repita dos veces para eliminar cualquier suciedad residual o residuos en el oído que puedan afectar la calidad de la imagen (consulte la Figura 1).
- Si la oreja se va a utilizar dentro de las 24 h, colóquela en una placa de Petri pequeña (es decir, 35 mm x 10 mm) con PBS fresco en un refrigerador (2-8 ° C). Si se va a utilizar la oreja después de 24 h de cosecha, colóquela en una placa de Petri (35 mm x 10 mm) sin PBS, cubra la placa con parafilm y colóquela en un congelador de -20 °C.
- Preparación de tejido de la piel humana
- Después de la obtención de tejido humano, colóquelo en una placa de Petri grande (es decir, 60 mm x 15 mm) en una campana biológica para permitir suficiente espacio para la preparación de la muestra.
- Coloque el estrato córneo hacia abajo de modo que la grasa subcutánea sea accesible.
- Usando fórceps y tijeras microquirúrgicas, comience a eliminar cuidadosamente la grasa subcutánea. Una vez que la grasa subcutánea ya no se pueda eliminar con las tijeras, cambie a un bisturí desechable de 10 cuchillas (o equivalente) para eliminar la grasa subcutánea restante. Use el bisturí en un ángulo de 45° con respecto a la piel mientras mantiene la piel quieta con fórceps (ver Figura 1).
NOTA: Para tener imágenes SRS de transmisión de alta calidad, las muestras deben ser lo más delgadas posible sin ser perforadas.
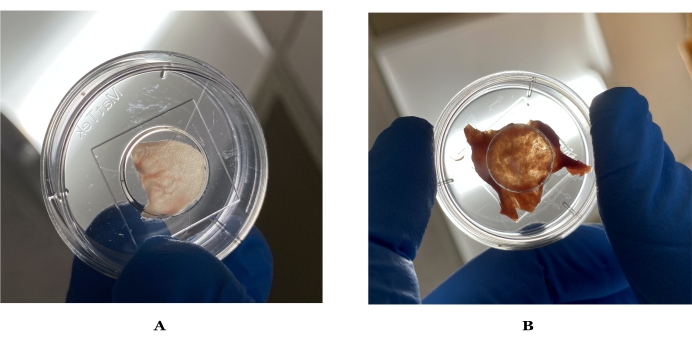
Figura 1: Imágenes de grosor ideal para obtener imágenes de la piel de ratón y humana. (A) Piel de oreja de ratón sostenida a la luz, que puede dejar pasar visiblemente la luz. (B) Piel humana ideal sostenida a la luz después de la preparación. Haga clic aquí para ver una versión más grande de esta figura.
- Secciona la piel humana en piezas de 1 cm x 1 cm.
NOTA: La piel fresca, se puede utilizar hasta por 24 h sin el uso de una cama de gel de agarosa, como se describió anteriormente31. Sin embargo, la piel fresca se puede utilizar durante más tiempo si se mantiene en una cama de gel de agarosa. Si la piel se va a utilizar más tarde, la piel se coloca en una bolsa de transporte de muestras y luego se coloca en un congelador de -20 °C. La piel congelada debe descongelarse mediante el procedimiento siguiente para obtener resultados óptimos (consulte el paso 3.1.2).
2. Configuración del láser y el microscopio
- Aproximadamente 30 minutos antes de la toma de imágenes, encienda el láser ultrarrápido ampliamente sintonizable (en adelante, el láser) y permita que se caliente. Habilite el sistema de señalización/bloqueo de advertencia láser para notificar al personal fuera del peligro potencial al ingresar.
NOTA: Siempre se deben usar gafas adecuadas cuando se trabaja con láseres de Clase IV. Para el láser específico utilizado aquí, las gafas adecuadas recomendadas son OD ≥ 6 para el rango de trabajo del láser 800-1,300 nm. - Mientras el láser se calienta, habilite el hardware restante para el control del microscopio, la detección de CARS y la detección de SRS.
- Alinee el microscopio correctamente para garantizar una imagen óptima. En la ventana Control de adquisición de imágenes en el software de control del microscopio (en adelante, el software MC), haga clic en la lámpara de transmisión para permitir que la luz provenga de la lámpara de transiluminación del microscopio.
- Asegure la iluminación correcta de Köhler para alinear el microscopio a lo largo del eje vertical: cierre el iris hacia abajo para que se vea una cantidad mínima de luz a través del ocular32.
- Mientras mira a través del ocular, abra el iris para ver si el polígono toca todos los lados simultáneamente. Ajuste la altura del condensador si no se puede ver una forma de polígono antes de abrir el iris.
- Si la forma del polígono no toca todos los lados al mismo tiempo, ajuste la posición de alineación de la apertura utilizando las perillas de ajuste.
NOTA: Ver Sanderson et al.33 para una configuración de microscopio en profundidad. - Coloque un portaobjetos de microscopio con un espaciador adhesivo de doble cara y una cubierta que contenga una muestra de aceite (por ejemplo, aceite de oliva, ya que hay muchos enlaces -CH2- dentro de los aceites) en el soporte de la etapa del microscopio.
- En la ventana Configuración de adquisición del software MC, busque el menú desplegable del microscopio y establezca el objetivo del microscopio en 20x.
- Verifique que el filtro de detección CARS (645 nm/50 nm) esté en posición de visualizar y medir la señal epiCARS a lo largo del tubo fotomultiplicador del puerto lateral del microscopio.
NOTA: Este filtro específico se selecciona para obtener imágenes de lípidos como la señal CARS anti-Stokes generada a 652 nm para una longitud de onda de bomba de 803 nm y una longitud de onda stokes de 1.040 nm (ver Eq. (1)).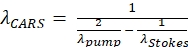 (1)
(1)
Donde las variables λ tienen unidades de nm; Labomba λ es la longitud de onda del haz de la bomba; y λStokes es la longitud de onda del haz de Stokes. - Mire a través del ocular para encontrar el borde de la muestra de aceite, que se utilizará para verificar la alineación del sistema.
- Asegúrese de que el borde esté en el enfoque Z utilizando las perillas de enfoque en el microscopio y ajuste el controlador de etapa para obtener el enfoque XY.
- Después de que el láser se haya calentado, ajuste el haz de la bomba a 803 nm con el ajuste fino del motor establecido en 50.0 en la interfaz gráfica de usuario del láser. El ajuste fino exacto del motor utilizado puede diferir de una configuración a otra.
NOTA: El haz de la bomba es el único haz con una longitud de onda ajustable en este láser, ya que la longitud de onda del haz de Stokes se fija en 1.040 nm. Esta configuración se dirige a la vibración CH a 2850 cm-1 (consulte Eq. (2) para calcular la longitud de onda de la bomba para la orientación del número de onda).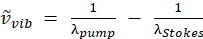 (2)
(2)
Donde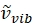 tiene unidades de número de onda relativo (cm−1), y las variables λ tienen unidades en cm.
tiene unidades de número de onda relativo (cm−1), y las variables λ tienen unidades en cm. - Verifique la alineación del láser en el microscopio antes de tomar imágenes; consulte la Figura 2 para la representación de la trayectoria del láser. Durante la configuración inicial del microscopio, instale dos iris en la trayectoria del haz como guías para una alineación correcta en el microscopio. Como la trayectoria óptica del láser puede desviarse con el tiempo, asegúrese de que las trayectorias del rayo láser atraviesen el centro de los iris para que entren correctamente en el microscopio.
- Usando el visor IR, cierre todos los iris por completo y asegúrese de que el haz esté centrado en ambos: primero en el iris más cercano al láser y finalmente en el puerto de entrada del microscopio.
- Primero, verifique el haz de la bomba para asegurarse de que el haz vaya directamente al microscopio. Una vez que la bomba esté alineada, verifique que el haz de Stokes también entre correctamente en el microscopio.
- Si los haces no están alineados a través de los iris, camine iterativamente los haces a través de los iris utilizando las perillas de ajuste x e y de los dos soportes de espejo para la bomba y las trayectorias de haz stokes.
- Una vez que los puntos del haz se superponen antes de ingresar al microscopio, verifique que estén atravesando el microscopio correctamente.
- En la ventana Control de adquisición de imágenes del software MC, haga clic en el canal TD para la transmisión, ALG1 (canal analógico 1) para el canal Raman anti-Stokes coherente y ALG2 (canal analógico 2) para el canal de dispersión Raman estimulado. Utilice la siguiente configuración (como en este protocolo): ganancia de 1 y un desplazamiento de -1 para ALG1, ganancia de 1,25 y desplazamiento de -2 para ALG2.
NOTA: Dependiendo de la configuración del sistema, los canales analógicos pueden tener una numeración diferente para los canales de imagen individuales. Si el detector SRS está en su lugar, no habrá señal de transmisión ya que no puede pasar la luz (es decir, no se pueden visualizar imágenes con TD y ALG2 simultáneamente en esta configuración).
- En la ventana Control de adquisición de imágenes del software MC, haga clic en el canal TD para la transmisión, ALG1 (canal analógico 1) para el canal Raman anti-Stokes coherente y ALG2 (canal analógico 2) para el canal de dispersión Raman estimulado. Utilice la siguiente configuración (como en este protocolo): ganancia de 1 y un desplazamiento de -1 para ALG1, ganancia de 1,25 y desplazamiento de -2 para ALG2.
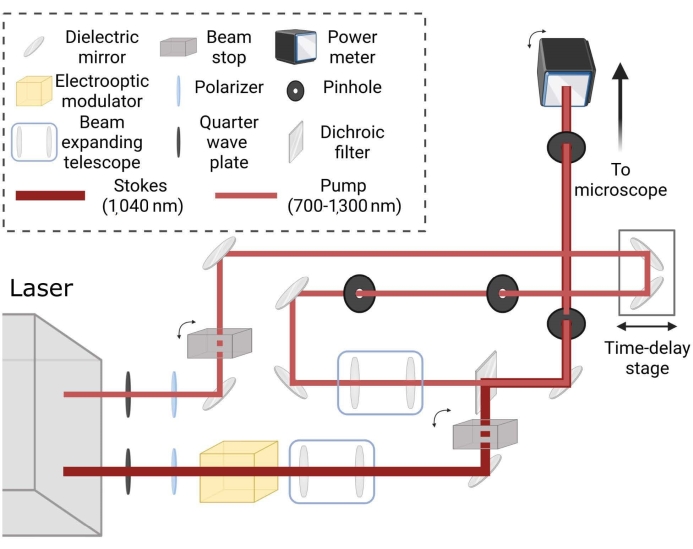
Figura 2: Diseño esquemático para una ruta coherente de imágenes láser Raman. Los haces se acondicionan de forma independiente para el tamaño del punto y se combinan a través de la etapa de retardo de tiempo para generar una dispersión Raman coherente en las muestras para la frecuencia de sintonización deseada. Haga clic aquí para ver una versión más grande de esta figura.
- Encienda el medidor de potencia y, utilizando un sensor de potencia térmica de alta potencia, mida la potencia de la bomba y los haces stokes individualmente para el experimento.
NOTA: En este ejemplo específico, la potencia del haz de la bomba era de 80 mW, mientras que la potencia del haz de Stokes era de 180 mW antes de la entrada al microscopio. - En la ventana Control de adquisición de imágenes, haga clic en el botón Focus x2 para ver la imagen en el software MC.
- En la ventana Configuración de adquisición , asegúrese de que la relación de píxeles y el tiempo de permanencia estén ajustados a los parámetros deseados para el experimento.
NOTA: En este protocolo se utilizó una relación de 1.024 x 1.024 píxeles y un tiempo de permanencia de 2 μs/píxel. - Confirme la alineación del láser con respecto al microscopio desbloqueando el haz de la bomba y mirando el canal TD .
NOTA: El láser se alinea correctamente si el haz se ve centrado en la imagen con la configuración correcta del detector. - De lo contrario, use perillas de ajuste X e Y en el periscopio para reposicionar el haz en el centro de la imagen.
- Confirme la alineación del láser y el microscopio viendo la misma imagen en los canales CARS y SRS.
- Para adquirir la imagen de alineación, haga clic en el botón de escaneo XY en la ventana Control de adquisición de imágenes con el modo de filtro establecido (por ejemplo, Línea Kalman 3).
- Guarde este conjunto de imágenes con un nombre de archivo descriptivo para comparar a lo largo del tiempo y confirmar el rendimiento/alineación del sistema.
3. Imágenes lipídicas
- Oreja de ratón y tejido humano
- Si usa tejido fresco, omita el paso 3.1.2.
- Retire la piel de la oreja de ratón del congelador de -20 °C y colóquela en una cámara de incubación (32 °C) durante 10 min. Retire la oreja de ratón de la cámara de incubación.
NOTA: Consulte el paso 1.1.2. para la preparación de la piel de la oreja de ratón. El manejo brusco o raspado del tejido puede resultar en degradación mecánica, destrucción o interrupción del tejido, especialmente el estrato córneo. - Si usa una oreja de ratón desnuda, coloque la parte anterior de la oreja mirando hacia el fondo de vidrio de un plato No. 0 de 35 mm. Si usa piel humana, colóquela con el estrato córneo boca abajo, ya que esto permitirá la cuantificación del fármaco desde las capas superficiales hasta las capas más profundas (Figura 1).
NOTA: La parte posterior de la oreja del ratón desnudo es más propensa a las imperfecciones de la carcasa. Si la piel humana no se coloca con el lado del estrato córneo mirando hacia abajo en el microscopio invertido, uno no podrá ver más allá de la dermis, ya que hay una buena cantidad de luz dispersada, y el medicamento que penetra en el estrato córneo no se puede ver. - Una vez que el tejido se haya centrado en el fondo de vidrio, use un aplicador con punta de algodón para asegurarse de que la piel sea plana y tenga contacto completo con la superficie de la cubierta del plato con fondo de vidrio.
NOTA: Este es un paso que puede causar dificultad al tomar imágenes de la piel si no está completamente plana. - Coloque una lavadora en la parte superior de la piel para evitar cualquier movimiento durante la toma de imágenes. Asegúrese de que el tejido sea visible a través del orificio central de la lavadora para la detección de transmisión de SRS.
- Retire el inserto de la etapa deslizante y reemplácelo con la cámara de incubación, que tiene el inserto de plato único.
- Coloque el plato con fondo de vidrio con el tejido de la piel en el accesorio de plato único de la cámara de incubación.
NOTA: Alternativamente, use una placa de 6 pocillos para obtener imágenes de múltiples muestras de piel y formulaciones a la vez. - Disminuya la relación de píxeles en la ventana Configuración de adquisición del software MC (por ejemplo, de 1.024 x 1.024 a 512 x 512) para una velocidad de escaneo galvo más rápida mientras se cambia la profundidad Z para encontrar el estrato córneo (consulte la Figura 3A para el ratón o la Figura 3E para humanos).
- Después de encontrar el estrato córneo, registre esa posición axial como la posición cero en la ventana Configuración de adquisición y cambie la relación de píxeles para cada experimento específico (por ejemplo, 1,024 x 1,024).
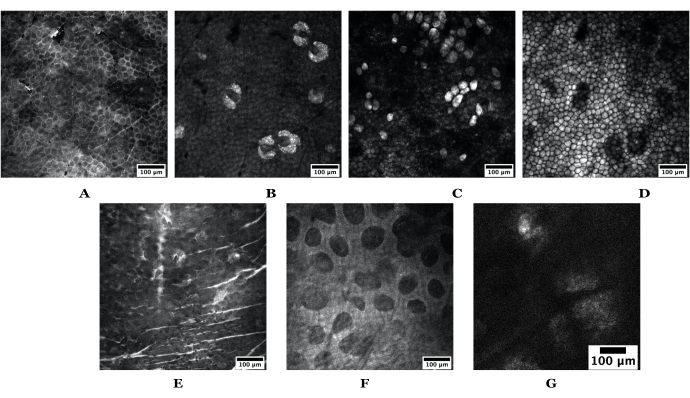
Figura 3: Ejemplo de profundidades de piel obtenidas usando SRS. El conjunto superior de imágenes son de piel de oreja de ratón desnuda que representa lo siguiente: (A) estrato córneo, (B) glándulas sebáceas, (C) adipocitos, (D) grasa subcutánea. El conjunto inferior de imágenes se obtiene de la piel humana que representa lo siguiente: (E) estrato córneo, (F) dermis papilar y (G) una glándula sebácea. Barras de escala = 100 μm. Tanto las imágenes del ratón como de la piel humana se adquirieron utilizando un objetivo de 20x a 1024 píxeles x 1024 píxeles; el SG humano fue tomado a 512 x 512 píxeles. Abreviaturas: SRS = dispersión Raman estimulada; SG = glándula sebácea. Haga clic aquí para ver una versión más grande de esta figura.
4. Aplicación de formulación tópica
- Pipetear la dosis predeterminada de la formulación sobre la piel (por ejemplo, 10 μL/cm2).
NOTA: La viscosidad de la formulación jugará un papel en la elección de la pipeta. Se recomienda el uso de una pipeta de desplazamiento positivo para formulaciones viscosas, como cremas o geles, y una pipeta de desplazamiento de aire para soluciones. En este experimento, ruxolitinib fue el compuesto modelo en una solución simple de propilenglicol (propano-1,2-diol). - Usando la punta del émbolo de una jeringa, o un dedo enguantado, frote la formulación en el sentido de las agujas del reloj durante 30 s. Tenga en cuenta el momento en que se aplica la formulación para el análisis posterior de cPK (consulte los pasos 6.15-6.17 a continuación).
NOTA: La duración de la aplicación depende del experimento; cada uno puede ser diferente. - Una vez transcurrido el tiempo asignado para que la formulación penetre, retire el exceso de formulación y coloque la piel con el lado de la formulación mirando hacia el plato con fondo de vidrio.
NOTA: La formulación se elimina utilizando un delicado limpiaparabrisas de tareas o una pequeña escobilla impresa en 3D (~ 1 pulgada) en una sola dirección (por ejemplo, de norte a sur).
5. Configuración experimental para la cuantificación de fármacos
- Ajuste el haz de la bomba a 803 nm. Compruebe la potencia de la bomba y del haz stokes con el fotodiodo para asegurarse de que son las potencias deseadas para el experimento. Desbloquee cada haz individualmente para medir la potencia y vuelva a bloquear los haces.
NOTA: En este ejemplo específico, la potencia del haz de la bomba era de 100 mW, mientras que la potencia del haz de Stokes era de 180 mW. - Coloque el plato con fondo de vidrio en una cámara de incubación con un inserto para un plato con fondo de vidrio. Asegure el plato con clips para evitar el movimiento durante la toma de imágenes.
- Encienda la luz de transmisión en el software MC. Mirando a través del ocular, ajuste el enfoque axial con la perilla de ajuste para asegurarse de que el tejido esté enfocado.
- Desbloquee tanto la bomba como las vigas Stokes. Asegúrese de que los canales ALG1 y ALG2 estén habilitados y, a continuación, haga clic en Focus x2 en el software MC para visualizar el aspecto en los canales CARS y SRS. Asegúrese de que el fotodiodo SRS esté en posición sobre el condensador.
- En el menú desplegable Dispositivo, haga clic en Lapso de tiempo de área múltiple (MATL). Busque una advertencia de etapa XY para que aparezca; cuando el escenario se mueva para encontrar su origen mecánico, haga clic en Aceptar.
- En el módulo MATL , vaya a Ver y, a continuación, haga clic en Lista de puntos registrados dentro del software MC. Comience a agregar 1) profundidades específicas dentro de la piel (es decir, estrato córneo, SG, adipocitos, grasa subcutánea según lo determinado durante las imágenes en vivo con contraste sintonizado con lípidos) o 2) posiciones XY si se van a tomar pilas de profundidad completas. Consulte la Figura 3 para ver ejemplos.
- Una vez que se identifica el estrato córneo, desplácese a través del foco axial (o enfoque z) para identificar estratificaciones tisulares específicas mencionadas anteriormente. Consulte la Figura 3 para ver ejemplos dentro de la piel humana y de ratón.
- En el caso de obtener imágenes de profundidades específicas en lugar de pilas de profundidad Z completas, para cada estratificación de piel, haga clic en Registrar punto para agregarlo a la cola MATL. Para pilas de profundidad completa, haga clic en Registrar punto para cada posición XY con selección de profundidad en la ventana parámetros de adquisición .
- Una vez que se hayan registrado todas las posiciones XY (pilas completas de profundidad Z) o XYZ (estratificaciones específicas de la piel) deseadas dentro del software MATL, cambie el directorio de archivos y el nombre de una manera que sea consistente a lo largo de los experimentos para los análisis de imágenes y cPK (consulte los pasos 6.15 a 6.17).
- Establezca el número de repeticiones en 1 en el módulo MATL y haga clic en Listo. Espere a que Play cambie de una flecha gris a una flecha negra, lo que indica que el software está listo. Pulse Reproducir para comenzar a obtener imágenes de la pila de lípidos preliminar (en adelante , Imágenes de lípidos).
NOTA: Esto se utilizará para separar las regiones ricas en lípidos y pobres en lípidos de las estratificaciones de tejidos individuales durante el análisis. El ciclo y el tiempo total se indican en la parte inferior derecha de la lista de puntos registrados. - Una vez que se haya completado el ciclo, bloquee tanto la bomba como las vigas stokes. Cambie la longitud de onda en la interfaz gráfica de usuario láser a la longitud de onda deseada en función del número de onda objetivo o la vibración Raman.
NOTA: Por ejemplo, se utilizan 843 nm para el haz de la bomba para apuntar a 2.250 cm-1 utilizando Eq. (2), y el ajuste fino del motor se cambia a 50,1. El fármaco de ejemplo presentado aquí, ruxolitinib, contiene un nitrilo que puede dirigirse a 2.250 cm-1. El número de onda objetivo para la estructura de la piel siempre será el mismo (2.850 cm-1); sin embargo, el número de onda para una API puede ser cualquier número de onda, pero debe conocerse o calcularse a priori. - Ajuste la etapa de retardo de tiempo manual (Figura 2) para garantizar la superposición en el tiempo para la nueva longitud de onda y la potencia del haz de la bomba. Asegúrese de que se utilizan las mismas potencias tanto para las imágenes lipídicas como para las API, que se establecen a priori.
- Utilice la duración por ciclo para calcular el número total de repeticiones requeridas por experimento. Simplemente divida el curso de tiempo total deseado del experimento por el de la duración del ciclo.
NOTA: La duración del ciclo es una función del tamaño de la imagen, el tiempo de permanencia de píxeles, el promedio de Kalman y el número de imágenes por ciclo. La optimización de estos parámetros acortará el tiempo de ciclo, aumentando así la resolución temporal. - Una vez que se haya elegido el número total de repeticiones de ciclo, desbloquee el haz de la bomba, verifique la potencia utilizando el fotodiodo y asegúrese de que coincida con la de la potencia deseada. Finalmente, desbloquee el haz de Stokes para permitir la obtención de imágenes.
- Pulse Reproducir para comenzar a crear imágenes automatizadas de los puntos de ajuste.
- Una vez completada la imagen MATL, vuelva a cambiar el haz de la bomba a 803 nm y ajuste la potencia de nuevo a la potencia original de imágenes lipídicas utilizada en el paso 5.1.
- Al igual que en los pasos anteriores, cambie el nombre del archivo para que sea coherente con las imágenes posteriores al experimento a lo largo del estudio.
- Establezca el número de repeticiones en 1.
- Haga clic en Listo | Botón de reproducción para adquirir una pila de lípidos posterior al curso de tiempo y asegurarse de que no ha habido movimiento del tejido durante la toma de imágenes (Figura 4).

Figura 4: Movimiento tisular en la piel desnuda de la oreja de ratón demostrada mediante la visualización de glándulas sebáceas. El ejemplo de movimiento limitado del tejido se representa en A y B, mientras que el movimiento sustancial del tejido se representa en C y D. (A) muestra las glándulas sebáceas en el momento de la aplicación de la formulación y (B) la misma profundidad a los 120 minutos después de la aplicación. (C) Glándulas sebáceas de ratón en el momento de la aplicación de la formulación y (D) 120 minutos después de la aplicación de la formulación; las glándulas sebáceas son apenas visibles, lo que es una indicación de que este experimento no midió la absorción en las glándulas sebáceas durante toda la duración experimental. Barras de escala = 100 μm. Las imágenes son de 1024 píxeles x 1024 píxeles. Haga clic aquí para ver una versión más grande de esta figura.
6. Análisis de datos
- Adquirir imágenes como . OIB (o . OIR dependiendo del microscopio y el software MC) tipos de archivos con cada posición XYZ que tiene una subcarpeta separada.
- Compile imágenes de lípidos con imágenes de canal API cambiando el nombre de las imágenes de lípidos con la siguiente terminación _lipid.oib.
- Realice los siguientes pasos con cada estratificación de la piel (que se muestra aquí utilizando solo la estratificación SG por simplicidad; consulte la Figura 3B).
- Importe una imagen lipídica SG en ImageJ (o Fiji)34 y marque la casilla con la etiqueta Canales divididos para dividir el archivo en los canales CARS y SRS.
NOTA: Fiji dividirá el archivo en el número de imágenes adquiridas durante el experimento en el número de canales. - Abra el administrador de regiones de interés (ROI) haciendo clic en Analizar | Herramientas | Gestor de ROI.
- Usando el canal SRS (por ejemplo, C = 1), demarque un SG en la imagen.
NOTA: Los SG son las ubicaciones brillantes debido a la vibración -CH2- dirigida. - Agregue esto al administrador de ROI haciendo clic en Agregar [t] en el administrador de ROI o presionando t en el teclado. Repita este proceso para cada SG dentro de la imagen.
- Para enmascarar las regiones ricas en lípidos, seleccione cada ROI y haga clic en la pestaña Más | | OR (Combinar) Añadir al gestor de ROI.
- Para enmascarar las regiones pobres en lípidos, utilice la herramienta de rectángulo en el menú FIJI y dibuje un cuadrado alrededor de toda la imagen. Agregue esto al administrador de ROI.
- Haga clic en el ROI cuadrado recién agregado, además del ROI que selecciona todas las regiones ricas en lípidos en el administrador de ROI. En Más, seleccione XOR para generar una máscara de las regiones pobres en lípidos y agréguela al administrador de ROI.
- Importe una imagen lipídica SG en ImageJ (o Fiji)34 y marque la casilla con la etiqueta Canales divididos para dividir el archivo en los canales CARS y SRS.
- Cargue las imágenes de la API en Fiji.
- Concatenar las imágenes en orden numérico (es decir, Image0001, Image0002, Image0003, etc.) utilizando la siguiente secuencia de menús: Imagen | Pilas | Herramientas | Concatenar.
- Alternativamente, importe estas imágenes cargando una de las imágenes en Fiji y luego seleccionando una opción llamada Agrupar archivos con nombres similares en la página de configuración .
NOTA: Esto proporciona la capacidad de importar todas las imágenes con un nombre de archivo similar y concatenarlas automáticamente.
- Alternativamente, importe estas imágenes cargando una de las imágenes en Fiji y luego seleccionando una opción llamada Agrupar archivos con nombres similares en la página de configuración .
- Mientras tenga activa la imagen concatenada, vaya al administrador de ROI, seleccione las regiones ricas en lípidos (es decir, el SG), haga clic en Más y seleccione Multimedida. Espere a que aparezca la ventana Resultados .
- Busque Área, Media, Mín., Máx. y Mediana en la configuración de medición predeterminada. Si se desean otras métricas para el análisis, habilite estas opciones marcando la casilla de verificación correspondiente en la ventana Establecer mediciones (Analizar | Establecer medidas...).
- Exporte los datos de la ventana Resultados a una hoja de cálculo y agregue una columna titulada Región.
- Agregue ricos en lípidos a cada fila de datos para las regiones ricas en lípidos. Agregue pobre en lípidos a las regiones que estaban fuera de los lípidos.
- Agregue una columna titulada capa y agregue la capa respectiva que se analizó (Tabla suplementaria S1).
- Repita los pasos 6.5 a 6.7 para las regiones pobres en lípidos mientras se selecciona el ROI apropiado.
- Para visualizar los datos, guarde la hoja de cálculo e impórtela en JupyterLab (paquete matplotlib)35 o en R (paquete ggplot2)36. Trazar los datos en función del número de imagen frente a la intensidad media para estimar los datos de tiempo de concentración. (Figura 5).
- Importe la hoja de cálculo en RStudio para realizar análisis no compartimentales (NCA) para el análisis farmacocinético de los datos de CRI.
- Agregue una columna titulada hora.
- Para Image0001, calcule la duración entre la aplicación de formulación y la primera imagen.
NOTA: Este es el primer punto de tiempo. La duración del ciclo se utiliza para calcular las imágenes restantes (y, por lo tanto, los puntos de tiempo) que aumentan con el tiempo. Por ejemplo, si el tiempo transcurrido desde la aplicación es de 30 min, Image0001 tendrá un punto de tiempo de 30 min y con una duración de ciclo de 8 min, Image0002 tendrá un punto de tiempo de 38 min, Image0003 tendrá un punto de tiempo de 46 min y así sucesivamente. - Ejecute NCA en RStudio (mediante el paquete NonCompart)37 en los datos de intensidad-tiempo importados de la hoja de cálculo, con la siguiente llamada para una capa/región:
sNCA(x = tiempo, y = media, dosis = 1, timeUnit = "s", doseUnit = "mg")
Donde x se refiere a puntos de tiempo, y se refiere a la intensidad, y la dosis se puede dejar como una, calculada como la dosis mM del medicamento en la formulación o la dosis del producto.
NOTA: La salida NCA proporcionará parámetros como Cmax y AUCtodos. Sin embargo, como se trata de piel ex vivo , estos parámetros son, de hecho, Jmax y AUCflux-all. - Compare visualmente las métricas Jmax y AUCflux-all graficándolas (Figura 6) además de las comparaciones estadísticas entre las condiciones experimentales. Ver Figura 6 para un ejemplo del análisis de estudios de CRI ex vivo .
NOTA: Las pruebas estadísticas apropiadas dependen de cada conjunto de datos específico. También es importante tener en cuenta que todos los parámetros farmacocinéticos se distribuyen log-normalmente, y cualquier comparación debe utilizar los datos log-transformados (log natural o log10).
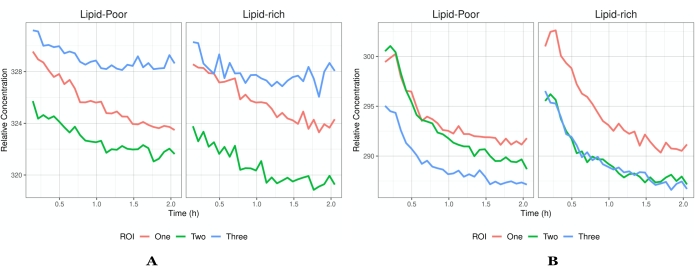
Figura 5: Intensidad vs. perfiles de tiempo. (A) Un ejemplo de perfiles de flujo que han alcanzado la saturación y, por lo tanto, solo se observa una disminución en la intensidad. Cada ROI tiene un perfil de flujo diferente para demostrar la heterogeneidad en los datos que uno podría adquirir. (B) Un ejemplo de concentraciones que aumentan después de que las imágenes han comenzado. Cada ROI es un campo de visión diferente (indicado por los diferentes rastros de color) dentro del mismo tejido del mismo experimento. Además de las concentraciones globales, existe la capacidad de dilucidar qué entorno local prefiere una API / formulación según lo indicado por las regiones ricas en lípidos y pobres en lípidos. Los perfiles presentados en A indican que no hay absorción de fármaco en el tejido, ya que el API ya ha permeado y ha comenzado a abandonar el tejido una vez que se han iniciado las imágenes. Sin embargo, en B, el tejido no ha alcanzado la saturación, y todavía hay absorción del API seguida de eliminación. La segmentación de imágenes en lípidos ricos y pobres en lípidos ayudará a dilucidar la localización del API (o inactivos) y las vías de permeación en la piel (es decir, estrato córneo). Una concentración más alta dentro de las regiones ricas en lípidos indica que el API se localiza dentro de la estructura lipídica de la capa bajo investigación, lo que ayuda en la información de administración dirigida de medicamentos. Abreviaturas: ROI = región de interés; API = ingrediente farmacéutico activo. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Las imágenes se consideran exitosas si el tejido no se ha movido significativamente en dirección axial (<10 μm) o lateral al finalizar el experimento (Figura 4). Esta es una indicación inmediata si la medición de SRS para la API de interés no es representativa de la profundidad inicial, para la cual la cuantificación es específica de la capa. Esto se mitiga mediante la obtención de imágenes de pilas z para cada posición XY de interés, con la compensación de la resolución tempor...
Access restricted. Please log in or start a trial to view this content.
Discusión
La evaluación de BA/BE tópico es un área de investigación que requiere un enfoque multifacético, ya que ningún método único puede caracterizar completamente la cPK in vivo . Este protocolo presenta una metodología para la evaluación del BA/BE de un fármaco tópico basada en imágenes Raman coherentes. Uno de los primeros puntos que podrían pasarse por alto es qué tan delgadas deben ser las muestras de piel, especialmente para las imágenes SRS de transmisión cuantitativa. Si la piel es demasiado gr...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
CLE es un inventor de patentes para microscopía CARS que han sido licenciadas a múltiples fabricantes de microscopios. Todos los demás autores no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores desean agradecer al Dr. Fotis Iliopoulos y Daniel Greenfield del Grupo Evans por su discusión y revisión de este manuscrito. Además, los autores desean agradecer el apoyo de LEO Pharma. La figura 2 se creó con BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Tissue Preparation | |||
| Autoclavable Biohazard Bags | FisherBrand | 22-044562 | As refered to in text: biohazard bags https://www.fishersci.com/shop/products/fisherbrand-polyethylene-biohazard-autoclave-bags-without-sterilization-indicator-8/22044562?searchHijack=true&searchTerm= 22044562&searchType=RAPID& matchedCatNo=22044562 |
| Cell Culture Buffers: Dulbecco's Phosphate-Buffered Salt Solution 1x | Corning | MT21030CV | As refered to in text: PBS https://www.fishersci.com/shop/products/corning-cellgro-cell-culture-buffers-dulbecco-s-phosphate-buffered-salt-solution-1x-8/MT21030CV?searchHijack=true&searchTerm= 21-030-cv&searchType= RAPID&matchedCatNo=21-030-cv |
| Disposable Scalpels | Exel International | 14-840-00 | As refered to in text: scalpel https://www.fishersci.com/shop/products/exel-international-disposable-scalpels-3/1484000?keyword=true |
| High Precision 45° Angle Broad Point Tweezers/Forceps | Fisherbrand | 12-000-132 | As refered to in text: forceps https://www.fishersci.com/shop/products/high-precision-45-angle-broad-point-tweezers-forceps/12000132#?keyword= |
| Kimwipes Delicate Task Wipers, 1-Ply | Kimberly-Clark Professional Kimtech Science | 06-666 | As refered to in text: task wiper https://www.fishersci.com/shop/products/kimberly-clark-kimtech-science-kimwipes-delicate-task-wipers-7/06666 |
| Parafilm M Laboratory Wrapping Film | Bemis | 13-374-12 | As refered to in text: parafilm https://www.fishersci.com/shop/products/curwood-parafilm-m-laboratory-wrapping-film-4/1337412 |
| Petri Dish (35 mm x 10 mm) | Fisherbrand | FB0875711YZ | As refered to in text: small petri dish https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-specialty-6/FB0875711YZ?keyword=true |
| Petri Dish (60 mm x 15 mm) | Fisherbrand | FB0875713A | As refered to in text: large petri dish https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-12/FB0875713A?keyword=true |
| Surgical Scissors | Roboz | NC9411473 | As refered to in text: scissors https://www.fishersci.com/shop/products/scissors-327/NC9411473?searchHijack=true&searchTerm= RS-5915SC&searchType=RAPID& matchedCatNo=RS-5915SC |
| Laser/microscope | |||
| 650/60 nm BrightLine single-band bandpass filter | Semrock | As refered to in text: CARS filter - CH2 vibrations (645nm/60nm filter) | |
| Control box IX2-UCB | Olympus | As refered to in text: Control Box | |
| D700/30m | Chroma | As refered to in text: CARS filter - deuterated band https://www.chroma.com/products/parts/d700-30m | |
| DeepSee Insight | Spectra-Physics | As refered to in text: Laser https://www.spectra-physics.com/f/insight-x3-tunable-laser | |
| Digital Handheld Optical Power and Energy Meter Console | ThorLabs | PM100D | As refered to in text: power meter https://www.thorlabs.com/newgrouppage9.cfm?objectgroup_id=3341 |
| Fluoview Software | Olympus | As refered to in text: Microscope Control software | |
| Frosted Microscope Slides | FisherBrand | As refered to in text: microscope slides https://www.fishersci.com/shop/products/fisherbrand-frosted-microscope-slides-4/22265446 | |
| FV1000 | Olympus | As refered to in text: Microscope | |
| Incubation Chamber | Tokai Hit | GM-800 | As refered to in text: incubation chamber |
| Integrating Sphere Photodiode Power Sensor | ThorLabs | S142C | As refered to in text: photodiode https://www.thorlabs.com/newgrouppage9.cfm?objectgroup_id=3341 |
| Power supply FV31-PSU | Olympus | As refered to in text: Power Supply | |
| Precision 4063, 80MHz Dual Channel Function Generator | BK Precision | As refered to in text: function generator | |
| ProScan – Precision Microscope Automation | Prior Scientific Instruments | As refered to in text: stage controller https://www.prior.com/microscope-automation/inverted-microscope-systems/proscan-linear-stage-highest-precision-microscope-automation | |
| SecureSeal Imaging Spacers | Grace Biolabs | 654004 | As refered to in text: spacer https://gracebio.com/product/secureseal-imaging-spacers-654004/ |
| SRS Detection Kit | APE | As refered to in text: SRS detector | |
| UPLSAPO 20X NA:0.75 | Olympus | As refered to in text: 20X Objective https://www.olympus-lifescience.com/en/objectives/uplsapo/ | |
| Lipid/Drug Imaging | |||
| 35 mm Dish, No. 0 Uncoated Coverslip, 14 mm Glass Diameter | MatTek Corporation | NC9711297 | As refered to in text: Glass bottom dish https://www.fishersci.com/shop/products/glass-bottom-mircrowell-dish/nc9711297 |
| Cotton-tipped applicators | FisherBrand | As refered to in text: Cotton-tipped applicator | |
| Distriman Postive Displacement Pipette | Gilson | As refered to in text: Postive Displacement Pipette https://www.fishersci.com/shop/products/gilson-distriman-positive-displacement-repetitive-pipette/F164001G#?keyword= | |
| Distriman Postive Displacement Pipette Tips | Gilson | As refered to in text: Tips for pipette https://www.fishersci.com/shop/products/gilson-distritip-syringes-6/f164100g?keyword=true | |
| Data Analysis | |||
| FIJI | Open-source | As refered to in text: FIJI/ImageJ https://imagej.net/software/fiji/ | |
| Jupyter-Lab | open-source | As refered to in text: JupyterLab https://jupyter.org/ | |
| Rstudio | Open-source | As refered to in text: Rstudio https://www.rstudio.com/ |
Referencias
- Finnin, B., Walters, K. A., Franz, T. J. In vitro skin permeation methodology. In Transdermal and topical drug delivery: principles and methodology. Transdermal and topical drug delivery: principles and practice. Benson, H. E., Watkinson, A. C. , John Wiley & Sons. Hoboken NJ USA. 85-108 (2012).
- Shin, S. H., et al. On the road to development of an in vitro permeation test (IVPT) model to compare heat effects on transdermal delivery systems: exploratory studies with nicotine and fentanyl. Pharmaceutical Research. 34 (9), 1817-1830 (2017).
- Hossain, A., et al. Preparation, characterisation, and topical delivery of terbinafine. Pharmaceutics. 11 (10), 548(2019).
- Santos, L. L., Swofford, N. J., Santiago, B. G. In vitro permeation test (IVPT) for pharmacokinetic assessment of topical dermatological formulations. Current Protocols in Pharmacology. 91 (1), 79(2020).
- Iliopoulos, F., Caspers, P. J., Puppels, G. J., Lane, M. E. Franz cell diffusion testing andquantitative confocal Raman spectroscopy: In vitro-in vivo correlation. Pharmaceutics. 12 (9), 887(2020).
- Cordery, S., et al. Topical bioavailability of diclofenac from locally-acting, dermatological formulations. International Journal of Pharmaceutics. 529 (1-2), 55-64 (2017).
- Pensado, A., et al. Stratum corneum sampling to assess bioequivalence between topicalacyclovir products. Pharmaceutical Research. 36 (12), 1-16 (2019).
- Zhang, Y., et al. Dermal delivery of niacinamide-in vivo studies. Pharmaceutics. 13 (5), 726(2021).
- Bodenlenz, M., et al. Open flow microperfusion as a dermal pharmacokinetic approach to evaluate topical bioequivalence. Clinical Pharmacokinetics. 56 (1), 91-98 (2017).
- Eirefelt, S., et al. Evaluating dermal pharmacokinetics and pharmacodymanic effect of soft topical PDE4 inhibitors:Open flow microperfusion and skin biopsies. Pharmaceutical Research. 37 (12), 1-12 (2020).
- Stagni, G., O'Donnell, D., Liu, Y. J., Kellogg, J. D. L., Shepherd, A. M. Iontophoretic current and intradermal microdialysis recovery in humans. Journal of Pharmacological and Toxicological Methods. 41 (1), 49-54 (1999).
- Garcia Ortiz, P., Hansen, S. H., Shah, V. P., Menne, T., Benfeldt, E. Impact of adultatopic dermatitis on topical drug penetration: assessment by cutaneous microdialysis and tape stripping. Acta Dermato-Venereologica. 89 (1), 33-38 (2009).
- Joshi, A., Patel, H., Joshi, A., Stagni, G. Pharmacokinetic applications of cutaneous microdialysis: Continuous+intermittent vs continuous-only sampling. Journal of Pharmacological and Toxicological Methods. 83, 16-20 (2017).
- Kuzma, B. A., et al. Evaluation of local bioavailability of metronidazole from topical formulations using dermal microdialysis: Preliminary study in a Yucatan mini-pig model. European Journal of Pharmaceutical Sciences. 159, 105741(2021).
- Begley, R., Harvey, A., Byer, R. L.Coherent anti-Stokes Raman spectroscopy. Applied Physics Letters. 25 (7), 387-390 (1974).
- Evans, C. L., et al. Chemical imaging of tissue in vivo with video-rate coherent anti-Stokes Raman scattering microscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (46), 16807-16812 (2005).
- Hill, A. H., Manifold, B., Fu, D. Tissue imaging depth limit of stimulated Raman scattering microscopy. Biomedical Optics Express. 11 (2), 762-774 (2020).
- Feizpour, A., Marstrand, T., Bastholm, L., Eirefelt, S., Evans, C. L. Label-free quantification of pharmacokinetics in skin with stimulated Raman scattering microscopy and deep learning. Journal of Investigative Dermatology. 141 (2), 395-403 (2021).
- Ghosh, B., Reddy, L. H., Kulkarni, R. V., Khanam, J. Comparison of skin permeability of drugs in mice and human cadaver skin. Indian Journal of Experimental Biology. 38 (1), 42-45 (2000).
- Nielsen, J. B., Plasencia, I., Sørensen, J. A., Bagatolli, L. Storage conditions of skin affect tissue structure and subsequent in vitro percutaneous penetration. Skin Pharmacology and Physiology. 24 (2), 93-102 (2011).
- Barbero, A. M., Frasch, H. F. Effect of frozen human epidermis storage duration and cryoprotectant on barrier function using two model compounds. Skin Pharmacology and Physiology. 29 (1), 31-40 (2016).
- Babu, R., et al. The influence of various methods of cold storage of skin on the permeation of melatonin and nimesulide. Journal of Controlled Release. 86 (1), 49-57 (2003).
- Skelly, J. P., et al. FDA and AAPS report of the workshop on principles and practices of in vitro percutaneous penetration studies: relevance to bioavailability and bioequivalence. Pharmaceutical Research. 4 (3), 265-267 (1987).
- OECD. Guidance document for the conduct of skin absorption studies. OECD. , (2004).
- OECD. Test no. 428: Skin absorption: In vitro method. OECD. , (2004).
- Saar, B. G., et al. Video-rate molecular imaging in vivo with stimulated Raman scattering. Science. 330 (6009), 1368-1370 (2010).
- Saar, B. G., Contreras-Rojas, L. R., Xie, X. S., Guy, R. H. Imaging drug delivery to skin with stimulated Raman scattering microscopy. Molecular Pharmaceutics. 8 (3), 969-975 (2011).
- Pence, I. J., Kuzma, B. A., Brinkmann, M., Hellwig, T., Evans, C. L. Multi-windowsparse spectral sampling stimulated Raman scattering microscopy. Biomedical Optics Express. 12 (10), 6095-6114 (2021).
- Herkenne, C., et al. In vivo methods for the assessment of topical drug bioavailability. Pharmaceutical Research. 25 (1), 87-103 (2008).
- Alfonso-Garcıa, A., Mittal, R., Lee, E. S., Potma, E. O. Biological imaging with coherent Raman scattering microscopy: a tutorial. Journal of Biomedical Optics. 19 (7), 071407(2014).
- Osseiran, S., et al. Longitudinal monitoring of cancer cell subpopulations in monolayers, 3D spheroids, and xenografts using the photoconvertible dye DiR. Scientific Reports. 9 (1), 1-10 (2019).
- Evennett, P. Kohler illumination: a simple interpretation. Proceedings of the Royal Microscopical Society. 28 (4), 189-192 (1983).
- Sanderson, J. Fundamentals of microscopy. Current Protocols in Mouse Biology. 10 (2), 76(2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Hunter, J. D. Matplotlib: A 2D graphics environment. Computing in Science & Engineering. 9 (3), 90-95 (2007).
- Wickham, H. ggplot2: Elegant Graphics for Data Analysis. , Springer-Verlag. New York. (2016).
- Kim, H., Han, S., Cho, Y. S., Yoon, S. K., Bae, K. Development of R packages:'Non-Compart' and 'ncar' for noncompartmental analysis (NCA). Translational and Clinical Pharmacology. 26 (1), 10-15 (2018).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados