Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Visualisation et quantification de composés pharmaceutiques dans la peau à l’aide de l’imagerie par diffusion Raman cohérente
Dans cet article
Résumé
Une méthodologie cohérente d’imagerie par diffusion Raman pour visualiser et quantifier les composés pharmaceutiques dans la peau est décrite. Cet article décrit la préparation de tissus cutanés (humains et souris) et l’application de formulation topique, l’acquisition d’images pour quantifier les profils de concentration spatio-temporelle et l’analyse pharmacocinétique préliminaire pour évaluer l’administration de médicaments topiques.
Résumé
La pharmacocinétique cutanée (cPK) après application de formulation topique a été un domaine de recherche d’un intérêt particulier pour les scientifiques de la réglementation et du développement de médicaments afin de comprendre mécaniquement la biodisponibilité topique (BA). Les techniques semi-invasives, telles que le décapage de bandes, la microdialyse cutanée ou la microperfusion cutanée à flux ouvert, quantifient toutes la cPK à l’échelle macro. Bien que ces techniques aient fourni de vastes connaissances en cPK, la communauté manque d’une compréhension mécaniste de la pénétration et de la perméation des ingrédients pharmaceutiques actifs (API) au niveau cellulaire.
Une approche non invasive pour traiter la cPK à l’échelle microscopique est l’imagerie par diffusion Raman cohérente (IRC), qui cible sélectivement les vibrations moléculaires intrinsèques sans avoir besoin d’étiquettes extrinsèques ou de modifications chimiques. L’IRC dispose de deux méthodes principales - la diffusion Raman anti-Stokes cohérente (CARS) et la diffusion Raman stimulée (SRS) - qui permettent une quantification sensible et sélective des API ou des ingrédients inactifs. CARS est généralement utilisé pour dériver des informations structurelles sur la peau ou visualiser le contraste chimique. En revanche, le signal SRS, qui est linéaire avec la concentration moléculaire, est utilisé pour quantifier les API ou les ingrédients inactifs dans les stratifications cutanées.
Bien que le tissu de souris ait été couramment utilisé pour la CPC avec IRC, le BA topique et la bioéquivalence (BE) doivent finalement être évalués dans les tissus humains avant l’approbation réglementaire. Cet article présente une méthodologie pour préparer et imager la peau ex vivo à utiliser dans les études quantitatives pharmacocinétiques CRI dans l’évaluation des BA et BE topiques. Cette méthodologie permet une quantification fiable et reproductible des API dans la peau de l’homme et de la souris au fil du temps. Les concentrations dans les compartiments riches en lipides et pauvres en lipides, ainsi que la concentration totale d’API au fil du temps sont quantifiées; ceux-ci sont utilisés pour les estimations de l’échelle micro et macro BA et, potentiellement, BE.
Introduction
Les méthodologies d’évaluation de la cPK après l’application d’un produit pharmaceutique topique sont passées des études classiques de test de perméation in vitro (IVPT) 1,2,3,4,5 et du décapage de bande 6,7,8 à des méthodologies supplémentaires telles que la microperfusion à flux ouvert ou la microdialyse cutanée 9,10,11, 12,13,14. Il existe potentiellement divers sites locaux d’action thérapeutique en fonction de la maladie d’intérêt. Par conséquent, il peut y avoir un nombre correspondant de méthodologies pour évaluer le taux et la mesure dans laquelle une API arrive au site d’action local prévu. Bien que chacune des méthodologies susmentionnées ait ses avantages, le principal inconvénient est le manque d’informations cPK à micro-échelle (c’est-à-dire l’incapacité de visualiser où va l’API et comment elle pénètre).
Une méthodologie non invasive d’intérêt pour estimer le BA et le BE topiques est l’IRC, qui peut être décomposé en deux modalités d’imagerie: CARS et SRS microscopie. Ces méthodes Raman cohérentes permettent une imagerie chimiquement spécifique des molécules via des effets Raman non linéaires. Dans l’IRC, deux trains d’impulsions laser sont focalisés et scannés dans un échantillon; la différence d’énergie entre les fréquences laser est réglée pour cibler les modes vibratoires spécifiques aux structures chimiques d’intérêt. Comme les processus CRI sont non linéaires, un signal n’est généré qu’au foyer du microscope, ce qui permet une imagerie tomographique pharmacocinétique tridimensionnelle du tissu. Dans le contexte de la cPK, CARS a été utilisé pour obtenir des informations structurelles tissulaires, telles que l’emplacement des structures cutanées riches en lipides15. En revanche, le SRS a été utilisé pour quantifier la concentration moléculaire car son signal est linéaire avec la concentration. Pour les échantillons de peau ex vivo , il est avantageux d’effectuer des CARS dans l’épi-direction16 et SRS en mode de transmission17. Par conséquent, les échantillons de tissus minces permettront la détection et la quantification du signal SRS.
En tant que tissu modèle, l’oreille de souris nue présente plusieurs avantages avec des inconvénients mineurs. Un avantage est que le tissu a déjà une épaisseur d’environ 200 à 300 μm et ne nécessite pas de préparation supplémentaire de l’échantillon. De plus, plusieurs stratifications cutanées sont observées en se concentrant axialement à travers un champ de vision (p. ex., strate cornée, glandes sébacées (SG), adipocytes et graisse sous-cutanée)16,18. Cela permet une estimation préclinique préliminaire des voies de perméation cutanée et des estimations topiques de BA avant de passer à des échantillons de peau humaine. Cependant, le modèle de souris nue présente des limites telles que la difficulté d’extrapolation à des scénarios in vivo en raison de différences dans la structure de la peau19. Alors que l’oreille de souris nue est un excellent modèle pour obtenir des résultats préliminaires, le modèle de peau humaine est la norme d’or. Bien qu’il y ait eu divers commentaires sur la pertinence et l’applicabilité de la peau humaine congelée pour récapituler avec précision la cinétique de perméation in vivo 20,21,22, l’utilisation de la peau humaine congelée est une méthode acceptée pour l’évaluation de la cinétique de perméation API in vitro 23,24,25 . Ce protocole visualise diverses couches cutanées de la peau de la souris et de l’homme tout en quantifiant les concentrations d’API dans les structures riches en lipides et pauvres en lipides.
Bien que l’IRC ait été utilisé dans de nombreux domaines pour visualiser spécifiquement les composés dans les tissus, les efforts ont été limités pour étudier la CPC des produits pharmaceutiques appliqués par voie topique. Pour évaluer le BA/BE topique des produits topiques utilisant l’IRC, il est nécessaire d’avoir d’abord un protocole normalisé en place pour faire des comparaisons précises. Les efforts antérieurs utilisant l’IRC pour l’administration de médicaments à la peau ont démontré une variabilité dans les données. Comme il s’agit d’une application relativement nouvelle de l’IRC, l’établissement d’un protocole est essentiel pour obtenir des résultats fiables 18,26,27. Cette approche ne cible qu’un nombre d’onde spécifique dans la région biologique silencieuse du spectre Raman. Cependant, la plupart des API et des ingrédients inactifs ont des décalages Raman dans la région des empreintes digitales. Cela a déjà posé des défis en raison du signal inhérent provenant du tissu dans la région des empreintes digitales. Les progrès récents du laser et du calcul ont éliminé cette barrière, qui peut également être utilisée en combinaison avec l’approche présentée ici28. Cette approche présentée ici permet de quantifier une API, qui a un décalage Raman dans la région silencieuse (2 000-2 300 cm-1). Cela ne se limite pas aux propriétés physico-chimiques du médicament, ce qui pourrait être le cas pour certaines méthodes de surveillance cPK mentionnées précédemment29.
Le protocole doit réduire la variabilité d’échantillon à échantillon de l’épaisseur de la peau pour diverses préparations, car les échantillons de peau humaine épaisse produiront un signal minimal après l’application du produit pharmaceutique en raison de la diffusion de la lumière par l’échantillon épais. L’un des objectifs de ce manuscrit est de présenter une méthodologie de préparation des tissus qui assure des normes d’imagerie reproductibles. En outre, le système CRI est configuré comme décrit pour réduire les sources potentielles d’erreur et minimiser le rapport signal-bruit. Cependant, cet article ne discutera pas des principes directeurs et des mérites techniques du microscope CRI car cela a déjà été couvert30. Enfin, la procédure d’analyse approfondie des données est explorée pour permettre l’interprétation des résultats afin de déterminer le succès ou l’échec d’une expérience.
Access restricted. Please log in or start a trial to view this content.
Protocole
L’utilisation de tissu auriculaire de souris nue a été approuvée par le Massachusetts General Hospital Institutional Animal Care and Use Committee (IACUC), tandis que l’utilisation de tissu cutané humain a été approuvée par le Massachusetts General Hospital Institutional Review Board (IRB). Selon les protocoles de l’IACUC, des souris fraîchement euthanasiées ont été obtenues auprès de collaborateurs avec des colonies de souris nues. Les tissus humains ont été obtenus à partir de procédures électives d’abdominoplastie au Massachusetts General Hospital via un protocole approuvé. En outre, des types de tissus spécifiques autres que la peau abdominale ont été acquis par l’intermédiaire d’une autorité de don de corps, également par le biais d’un protocole approuvé par la CISR.
1. Préparation des tissus
- Préparation de tissu cutané d’oreille de souris nue
- Après avoir acquis des corps de souris nues fraîchement récoltés, retirez les oreilles à l’aide de pinces et de ciseaux microchirurgicaux. Placez une oreille dans une petite boîte de Petri (c.-à-d. 35 mm x 10 mm). Placez le corps de souris nue dans un sac à risque biologique à éliminer conformément aux protocoles locaux de l’IACUC.
- Rincez chaque oreille de souris avec une solution saline tamponnée au phosphate (PBS) et séchez-la doucement avec un essuie-glace de travail. Répétez deux fois l’opération pour éliminer toute saleté ou débris résiduel sur l’oreille qui pourrait affecter la qualité de l’image (voir la figure 1).
- Si l’oreille doit être utilisée dans les 24 heures, placez-la dans une petite boîte de Petri (c.- à-d. 35 mm x 10 mm) avec du PBS frais au réfrigérateur (2-8 °C). Si l’oreille doit être utilisée après 24 h de récolte, placez-la dans une boîte de Pétri (35 mm x 10 mm) sans PBS, couvrez la boîte avec un parafilm et placez-la dans un congélateur à -20 °C.
- Préparation du tissu cutané humain
- Après l’obtention de tissu humain, placez-les dans une grande boîte de Pétri (c.-à-d. 60 mm x 15 mm) dans une hotte biologique pour laisser suffisamment d’espace pour la préparation des échantillons.
- Placez la couche cornée vers le bas de manière à ce que la graisse sous-cutanée soit accessible.
- À l’aide de pinces et de ciseaux microchirurgicaux, commencez à retirer soigneusement la graisse sous-cutanée. Une fois que la graisse sous-cutanée ne peut plus être enlevée avec les ciseaux, passez à un scalpel jetable à 10 lames (ou équivalent) pour éliminer la graisse sous-cutanée restante. Utilisez le scalpel à un angle de 45° par rapport à la peau tout en maintenant la peau immobile avec une pince (voir La figure 1).
REMARQUE: Pour avoir des images SRS de transmission de haute qualité, les échantillons doivent être aussi minces que possible sans être perforés.
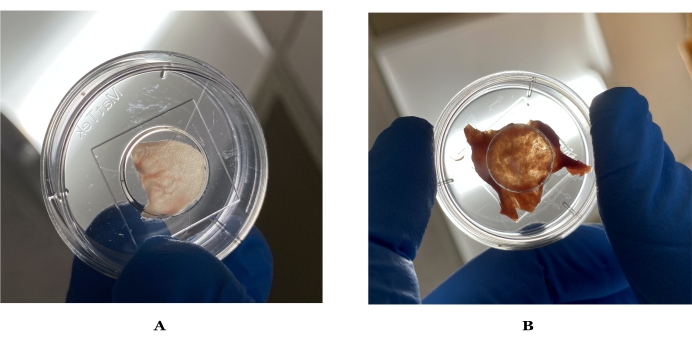
Figure 1: Images d’épaisseur idéale pour l’imagerie de la souris et de la peau humaine. (A) La peau de l’oreille de la souris est maintenue à la lumière, qui peut visiblement laisser passer la lumière. (B) Peau humaine idéale maintenue à la lumière après la préparation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Divisez la peau humaine en morceaux de 1 cm x 1 cm.
REMARQUE: Peau fraîche, peut être utilisé jusqu’à 24 heures sans l’utilisation d’un lit de gel d’agarose, comme décrit précédemment31. Cependant, la peau fraîche peut être utilisée plus longtemps si elle est conservée sur un lit de gel d’agarose. Si la peau doit être utilisée plus tard, la peau est placée dans un sac de transport d’échantillons, puis placée dans un congélateur à -20 °C. La peau congelée doit être décongelée en utilisant la procédure ci-dessous pour des résultats optimaux (voir étape 3.1.2).
2. Configuration du laser et du microscope
- Environ 30 minutes avant l’imagerie, allumez le laser ultrarapide largement accordable (ci-après dénommé laser) et laissez-le se réchauffer. Activez le panneau d’avertissement laser/ système de verrouillage pour informer le personnel à l’extérieur d’un danger potentiel à l’entrée.
REMARQUE: Des lunettes appropriées doivent toujours être portées lorsque vous travaillez avec des lasers de classe IV. Pour le laser spécifique utilisé ici, les lunettes appropriées recommandées sont OD ≥ 6 pour la plage de travail du laser 800-1 300 nm. - Pendant que le laser se réchauffe, activez le matériel restant pour le contrôle du microscope, la détection CARS et la détection SRS.
- Alignez correctement le microscope pour assurer une imagerie optimale. Dans la fenêtre Contrôle d’acquisition d’image du logiciel de contrôle du microscope (ci-après logiciel MC), cliquez sur la lampe de transmission pour permettre à la lumière de provenir de la lampe à transillumination du microscope.
- Assurez-vous d’un éclairage Köhler correct pour aligner le microscope le long de l’axe vertical : fermez l’iris pour qu’une quantité minimale de lumière soit vue à travers l’oculaire32.
- En regardant à travers l’oculaire, ouvrez l’iris pour voir si le polygone touche tous les côtés simultanément. Ajustez la hauteur du condenseur si une forme de polygone ne peut pas être vue avant d’ouvrir l’iris.
- Si la forme du polygone ne touche pas tous les côtés en même temps, ajustez la position d’alignement de l’ouverture à l’aide des boutons de réglage.
REMARQUE : Voir Sanderson et al.33 pour une configuration de microscope approfondie. - Placez une lame de microscope avec un couvercle et une entretoise adhésive double face contenant un échantillon d’huile (par exemple, de l’huile d’olive, car il existe de nombreuses liaisons -CH2- dans les huiles) sur le porte-étage du microscope.
- Dans la fenêtre Paramètres d’acquisition du logiciel MC, recherchez le menu déroulant du microscope et réglez l’objectif du microscope sur 20x.
- Vérifiez que le filtre de détection CARS (645 nm/50 nm) est en position de visualiser et de mesurer le signal epiCARS le long du tube photomultiplicateur du port latéral du microscope.
REMARQUE: Ce filtre spécifique est sélectionné pour l’imagerie des lipides comme le signal anti-Stokes CARS généré à 652 nm pour une longueur d’onde de pompe de 803 nm et une longueur d’onde de Stokes de 1 040 nm (voir Eq. (1)).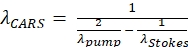 (1)
(1)
Où les variables λ ont des unités de nm; Lapompe λ est la longueur d’onde du faisceau de la pompe; et λStokes est la longueur d’onde du faisceau de Stokes. - Regardez à travers l’oculaire pour trouver le bord de l’échantillon d’huile, qui sera utilisé pour vérifier l’alignement du système.
- Assurez-vous que le bord est en mise au point Z à l’aide des boutons de mise au point du microscope et ajustez le contrôleur de scène pour obtenir la mise au point XY.
- Une fois le laser réchauffé, réglez le faisceau de la pompe à 803 nm avec le réglage fin du moteur réglé sur 50.0 dans l’interface utilisateur graphique du laser. La mise au point exacte du moteur utilisée peut différer d’une configuration à l’autre.
REMARQUE: Le faisceau de pompe est le seul faisceau avec une longueur d’onde réglable sur ce laser car la longueur d’onde du faisceau de Stokes est fixée à 1 040 nm. Cette configuration cible la vibration CH à 2850 cm-1 (voir Eq. (2) pour calculer la longueur d’onde de la pompe pour le ciblage du nombre d’ondes).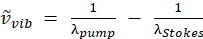 (2)
(2)
Où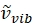 a des unités de nombre d’onde relatif (cm−1), et les variables λ ont des unités en cm.
a des unités de nombre d’onde relatif (cm−1), et les variables λ ont des unités en cm. - Vérifier l’alignement du laser dans le microscope avant l’imagerie; voir la figure 2 pour la représentation du trajet du laser. Lors de la configuration initiale du microscope, installez deux iris dans le trajet du faisceau comme guides pour un alignement correct dans le microscope. Comme le chemin optique du laser peut dériver au fil du temps, assurez-vous que les chemins du faisceau laser traversent le centre des iris afin qu’ils pénètrent correctement dans le microscope.
- À l’aide de la visionneuse IR, fermez complètement tous les iris et assurez-vous que le faisceau est centré sur les deux: d’abord sur l’iris le plus proche du laser et enfin sur le port d’entrée du microscope.
- Tout d’abord, vérifiez le faisceau de la pompe pour vous assurer que le faisceau va droit dans le microscope. Une fois la pompe alignée, vérifiez que le faisceau Stokes entre également dans le microscope correctement.
- Si les faisceaux ne sont pas alignés à travers les iris, parcourez de manière itérative les faisceaux à travers les iris à l’aide des boutons de réglage x et y des deux supports de miroir pour la pompe et les chemins de faisceau Stokes.
- Une fois que les points de faisceau se chevauchent avant d’entrer dans le microscope, vérifiez qu’ils traversent le microscope correctement.
- Dans la fenêtre Contrôle d’acquisition d’images du logiciel MC, cliquez sur le canal TD pour la transmission, ALG1 (canal analogique 1) pour le canal Raman anti-Stokes cohérent et ALG2 (canal analogique 2) pour le canal de diffusion Raman stimulé. Utilisez les paramètres suivants (comme dans ce protocole) : gain de 1 et décalage de -1 pour ALG1, gain de 1,25 et décalage de -2 pour ALG2.
REMARQUE: Selon la configuration du système, les canaux analogiques peuvent avoir une numérotation différente pour les canaux d’imagerie individuels. Si le détecteur SRS est en place, il n’y aura pas de signal de transmission car aucune lumière ne peut passer (c’est-à-dire qu’on ne peut pas visualiser les images avec TD et ALG2 simultanément dans cette configuration).
- Dans la fenêtre Contrôle d’acquisition d’images du logiciel MC, cliquez sur le canal TD pour la transmission, ALG1 (canal analogique 1) pour le canal Raman anti-Stokes cohérent et ALG2 (canal analogique 2) pour le canal de diffusion Raman stimulé. Utilisez les paramètres suivants (comme dans ce protocole) : gain de 1 et décalage de -1 pour ALG1, gain de 1,25 et décalage de -2 pour ALG2.
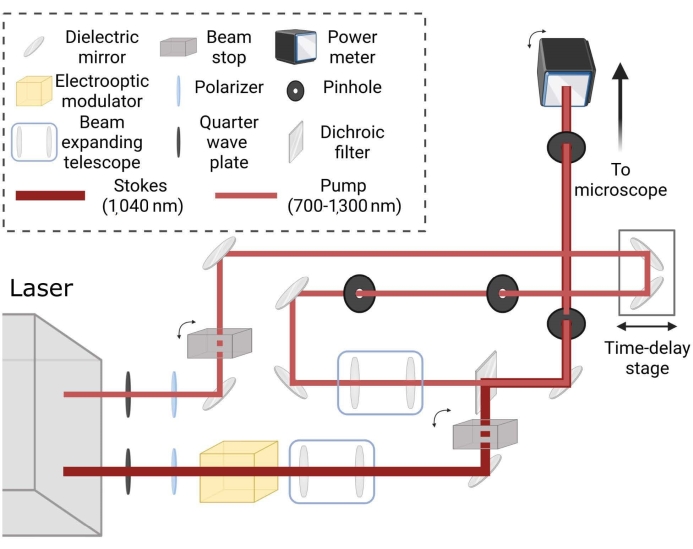
Figure 2 : Disposition schématique pour un chemin d’imagerie laser Raman cohérent. Les faisceaux sont conditionnés indépendamment pour la taille du spot et appariés via un étage de retard temporel pour générer une diffusion Raman cohérente dans les échantillons pour la fréquence de réglage souhaitée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Allumez le capteur de puissance et, à l’aide d’un capteur de puissance thermique haute puissance, mesurez la puissance de la pompe et des faisceaux stokes individuellement pour l’expérience.
REMARQUE: Dans cet exemple spécifique, la puissance du faisceau de la pompe était de 80 mW, tandis que la puissance du faisceau de Stokes était de 180 mW avant l’entrée du microscope. - Dans la fenêtre Contrôle d’acquisition d’image , cliquez sur le bouton Focus x2 pour afficher l’image dans le logiciel MC.
- Dans la fenêtre Paramètres d’acquisition , assurez-vous que le rapport de pixels et le temps de séjour sont définis sur les paramètres souhaités pour l’expérience.
REMARQUE: Un rapport de 1 024 x 1 024 pixels et un temps de séjour de 2 μs / pixel ont été utilisés dans ce protocole. - Confirmez l’alignement du laser par rapport au microscope en débloquant le faisceau de la pompe et en regardant le canal TD .
REMARQUE: Le laser est aligné correctement si le faisceau est vu centré dans l’image avec les paramètres de détecteur corrects. - Sinon, utilisez les boutons de réglage X et Y du périscope pour repositionner le faisceau au centre de l’image.
- Confirmez l’alignement du laser et du microscope en voyant la même image dans les canaux CARS et SRS.
- Pour acquérir l’image d’alignement, cliquez sur le bouton de numérisation XY dans la fenêtre Contrôle d’acquisition d’image avec le mode de filtre approprié défini (par exemple, Kalman Line 3).
- Enregistrez cet ensemble d’images avec un nom de fichier descriptif pour comparer au fil du temps et confirmer les performances/l’alignement du système.
3. Imagerie lipidique
- Oreille de souris et tissu humain
- Si vous utilisez des mouchoirs frais, sautez l’étape 3.1.2.
- Retirer la peau de l’oreille de souris du congélateur à -20 °C et la placer dans une chambre d’incubation (32 °C) pendant 10 min. Retirez l’oreille de la souris de la chambre d’incubation.
REMARQUE : Voir l’étape 1.1.2. pour la préparation de la peau de l’oreille de souris. Une manipulation ou un grattage brutal du tissu peut entraîner une dégradation mécanique, une destruction ou une perturbation du tissu, en particulier de la couche cornée. - Si vous utilisez une oreille de souris nue, placez la partie antérieure de l’oreille vers le fond en verre d’un plat n° 0 de 35 mm. Si vous utilisez de la peau humaine, placez-la avec la couche cornée face cachée, car cela permettra de quantifier les médicaments des couches superficielles aux couches plus profondes (Figure 1).
REMARQUE: La partie postérieure de l’oreille de souris nue est plus sujette aux imperfections du logement. Si la peau humaine n’est pas placée avec le côté de la couche cornée tourné vers le bas sur le microscope inversé, on ne pourra pas voir au-delà du derme car il y a une bonne quantité de lumière diffusée et le médicament pénétrant dans la couche cornée ne peut pas être vu. - Une fois que le tissu a été centré sur le fond en verre, utilisez un applicateur à pointe de coton pour vous assurer que la peau est plate et a un contact complet avec la surface de couverture du plat à fond de verre.
REMARQUE: Il s’agit d’une étape qui peut causer des difficultés lors de l’imagerie de la peau si elle n’est pas complètement plate. - Placez une rondelle sur le dessus de la peau pour éviter tout mouvement pendant l’imagerie. Assurez-vous que le tissu est visible à travers le trou central de la rondelle pour la détection de la transmission SRS.
- Retirez l’insert de l’étage coulissant et remplacez-le par la chambre d’incubation, qui a l’insert à plat unique.
- Placez le plat à fond de verre avec le tissu cutané dans la fixation à plat unique de la chambre d’incubation.
REMARQUE: Vous pouvez également utiliser une plaque à 6 puits pour imager plusieurs échantillons de peau et formulations à la fois. - Diminuez le rapport de pixels dans la fenêtre Paramètres d’acquisition du logiciel MC (par exemple, de 1 024 x 1 024 à 512 x 512) pour une vitesse de balayage galvo plus rapide pendant que la profondeur Z est modifiée pour trouver la couche cornée (voir la Figure 3A pour la souris ou la Figure 3E pour l’homme).
- Une fois que la couche cornée a été trouvée, enregistrez cette position axiale comme étant la position zéro dans la fenêtre Paramètres d’acquisition et modifiez le rapport de pixels pour chaque expérience spécifique (par exemple, 1 024 x 1 024).
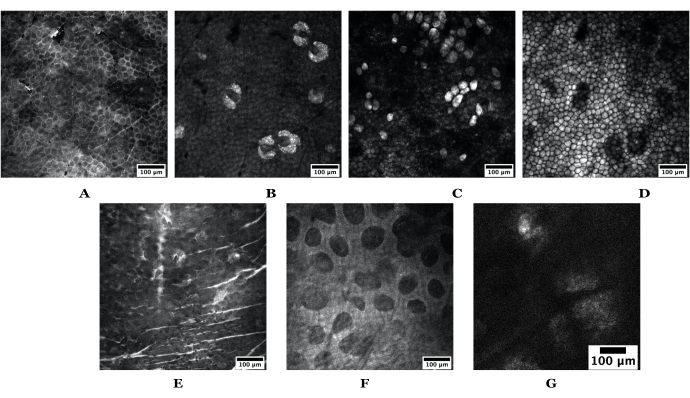
Figure 3 : Exemples de profondeurs de peau obtenues à l’aide de SRS. L’ensemble supérieur d’images provient de la peau d’oreille de souris nue représentant les éléments suivants: (A) stratum corneum, (B) glandes sébacées, (C) adipocytes, (D) graisse sous-cutanée. L’ensemble d’images du bas est obtenu à partir de la peau humaine représentant les éléments suivants: (E) stratum corneum, (F) derme papillaire et (G) une glande sébacée. Barres d’échelle = 100 μm. Les images de souris et de peau humaine ont été acquises à l’aide d’un objectif 20x à 1024 pixels x 1024 pixels; le SG humain a été pris à 512 x 512 pixels. Abréviations : SRS = diffusion Raman stimulée ; SG = glande sébacée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Application de la formulation topique
- Pipeter la dose de formulation prédéterminée sur la peau (p. ex., 10 μL/cm2).
REMARQUE: La viscosité de la formulation jouera un rôle dans le choix de la pipette. L’utilisation d’une pipette à déplacement positif pour les formulations visqueuses, telles que les crèmes ou les gels, et d’une pipette à déplacement d’air pour les solutions est recommandée. Dans cette expérience, le ruxolitinib était le composé modèle dans une solution simple de propylène glycol (propane-1,2-diol). - À l’aide de la pointe du piston d’une seringue ou d’un doigt ganté, frottez la formulation dans le sens des aiguilles d’une montre pendant 30 s. Notez le moment où la formulation est appliquée pour une analyse ultérieure de la CPC (voir les étapes 6.15 à 6.17 ci-dessous).
REMARQUE: La durée de l’application dépend de l’expérience; chacun peut être différent. - Une fois que le temps alloué pour que la formulation pénètre est écoulé, retirez l’excès de formulation et placez la peau avec le côté de la formulation tourné vers le plat à fond de verre.
REMARQUE: La formulation est retirée à l’aide d’un essuie-glace de tâche délicate ou d’une petite raclette imprimée en 3D (~ 1 pouce) dans une seule direction (par exemple, du nord au sud).
5. Configuration expérimentale pour la quantification des médicaments
- Réglez le faisceau de la pompe sur 803 nm. Vérifiez les puissances de la pompe et du faisceau de Stokes avec la photodiode pour vous assurer qu’il s’agit des puissances souhaitées pour l’expérience. Débloquez chaque faisceau individuellement pour mesurer la puissance et rebloquez les faisceaux.
REMARQUE: Dans cet exemple spécifique, la puissance du faisceau de la pompe était de 100 mW, tandis que la puissance du faisceau de Stokes était de 180 mW. - Placez le plat à fond de verre dans une chambre d’incubation avec un insert pour un plat à fond de verre. Fixez le plat avec des clips pour empêcher tout mouvement pendant l’imagerie.
- Allumez le voyant de transmission dans le logiciel MC. En regardant à travers l’oculaire, ajustez la mise au point axiale avec le bouton de réglage pour vous assurer que le tissu est au point.
- Débloquez à la fois la pompe et les poutres Stokes. Assurez-vous que les canaux ALG1 et ALG2 sont activés, puis cliquez sur Focus x2 sur le logiciel MC pour visualiser l’habillage dans les canaux CARS et SRS. Assurez-vous que la photodiode SRS est en position au-dessus du condenseur.
- Dans le menu déroulant Périphérique, cliquez sur Multi Area Time Lapse (MATL). Recherchez un avertissement d’étape XY à afficher; lorsque la scène se déplace pour trouver son origine mécanique, cliquez sur OK.
- Dans le module MATL , accédez à Affichage , puis cliquez sur Liste de points enregistrés dans le logiciel MC. Commencez à ajouter soit 1) des profondeurs spécifiques dans la peau (c.-à-d. la couche cornée, les SG, les adipocytes, la graisse sous-cutanée déterminée lors de l’imagerie en direct avec contraste accordé aux lipides) ou 2) des positions XY si des piles de profondeur entières doivent être prises. Voir la figure 3 pour des exemples.
- Une fois la couche cornée identifiée, faites défiler le foyer axial (ou z-focus) pour identifier les stratifications tissulaires spécifiques mentionnées ci-dessus. Voir la figure 3 pour des exemples dans la peau de l’homme et de la souris.
- Dans le cas de l’imagerie de profondeurs spécifiques par opposition à des piles de profondeur Z complètes, pour chaque stratification d’enveloppe, cliquez sur Enregistrer le point pour l’ajouter à la file d’attente MATL. Pour les piles pleine profondeur, cliquez sur Enregistrer le point pour chaque position XY avec sélection de profondeur dans la fenêtre des paramètres d’acquisition .
- Une fois que toutes les positions XY (piles de profondeur Z complètes) ou XYZ (stratifications de peau spécifiques) souhaitées ont été enregistrées dans le logiciel MATL, modifiez le répertoire de fichiers et le nom de manière cohérente tout au long des expériences pour les analyses d’images et de cPK (voir les étapes 6.15 à 6.17).
- Définissez le nombre de répétitions sur 1 dans le module MATL et cliquez sur Prêt. Attendez que Play passe d’une flèche grise à une flèche noire, indiquant que le logiciel est prêt. Appuyez sur Lecture pour commencer à imager la pile lipidique préliminaire (ci-après Lipid Images).
REMARQUE: Ceci sera utilisé pour séparer les régions riches en lipides et pauvres en lipides des stratifications tissulaires individuelles au cours de l’analyse. Le cycle et la durée totale sont indiqués en bas à droite de la liste des points enregistrés. - Une fois le cycle terminé, bloquez à la fois la pompe et les poutres stokes. Modifiez la longueur d’onde sur l’interface utilisateur graphique du laser à la longueur d’onde souhaitée en fonction du nombre d’onde ciblé ou de la vibration Raman.
REMARQUE: Par exemple, 843 nm pour le faisceau de la pompe est utilisé pour cibler 2 250 cm-1 en utilisant Eq. (2), et le réglage fin du moteur est changé à 50.1. L’exemple de médicament présenté ici, le ruxolitinib, contient un nitrile qui peut être ciblé à 2 250 cm-1. Le nombre d’onde ciblé pour la structure de la peau sera toujours le même (2 850 cm-1); cependant, le numéro d’onde d’une API peut être n’importe quel numéro d’onde mais doit être connu ou calculé a priori. - Ajustez l’étage de retard manuel (Figure 2) pour assurer le chevauchement dans le temps pour la nouvelle longueur d’onde et la puissance du faisceau de la pompe. Assurez-vous que les mêmes pouvoirs sont utilisés pour l’imagerie lipidique et API, qui sont établies a priori.
- Utilisez la durée par cycle pour calculer le nombre total de répétitions requises par expérience. Il suffit de diviser le temps total souhaité de l’expérience par celui de la durée du cycle.
REMARQUE: La durée du cycle est fonction de la taille de l’image, du temps de séjour en pixels, de la moyenne de Kalman et du nombre d’images par cycle. L’optimisation de ces paramètres raccourcira le temps de cycle, augmentant ainsi la résolution temporelle. - Une fois que le nombre total de répétitions de cycle a été choisi, débloquez le faisceau de la pompe, vérifiez la puissance à l’aide de la photodiode et assurez-vous qu’elle correspond à celle de la puissance souhaitée. Enfin, débloquez le faisceau de Stokes pour permettre l’imagerie.
- Appuyez sur Lecture pour lancer l’imagerie automatisée des points de consigne.
- Une fois l’imagerie MATL terminée, rétablissez le faisceau de la pompe à 803 nm et réglez la puissance à la puissance d’imagerie lipidique d’origine utilisée à l’étape 5.1.
- Comme dans les étapes précédentes, modifiez le nom du fichier pour qu’il soit cohérent pour les images post-expérience tout au long de l’étude.
- Définissez le nombre de répétitions sur 1.
- Cliquez sur Prêt | Bouton de lecture pour acquérir une pile lipidique post-cours temporel et s’assurer qu’il n’y a pas eu de mouvement tissulaire pendant l’imagerie (Figure 4).

Figure 4 : Mouvement tissulaire dans la peau d’oreille de souris nue démontré en visualisant les glandes sébacées. Un exemple de mouvement tissulaire limité est représenté en A et B, tandis que le mouvement tissulaire important est représenté en C et D. (A) montre les glandes sébacées au moment de l’application de la formulation et (B) la même profondeur à 120 min après l’application. (C) Glandes sébacées de souris au moment de l’application de la formulation et (D) 120 min après l’application de la formulation; les glandes sébacées sont à peine visibles, ce qui indique que cette expérience n’a pas mesuré l’absorption dans les glandes sébacées pendant toute la durée expérimentale. Barres d’échelle = 100 μm. Les images sont de 1024 pixels x 1024 pixels. Veuillez cliquer ici pour voir une version agrandie de cette figure.
6. Analyse des données
- Acquérir des images au format . OIB (ou . OIR en fonction du microscope et du logiciel MC) types de fichiers avec chaque position XYZ ayant un sous-dossier séparé.
- Compilez des images lipidiques avec des images de canal API en renommant les images lipidiques avec la fin suivante _lipid.oib.
- Effectuez les étapes suivantes avec chaque stratification de la peau (illustrée ici en utilisant uniquement la stratification SG par souci de simplicité ; voir la figure 3B).
- Importez une image lipidique SG dans ImageJ (ou Fiji)34 et cochez la case Split Channels pour diviser le fichier en canaux CARS et SRS.
REMARQUE: Fidji divisera le fichier en nombre d’images acquises au cours de l’expérience sur le nombre de canaux. - Ouvrez le gestionnaire de région d’intérêt (ROI) en cliquant sur Analyser | Outils | Gestionnaire de retour sur investissement.
- À l’aide du canal SRS (par exemple, C = 1), délimitez un SG dans l’image.
REMARQUE: Les SG sont les endroits lumineux en raison de la vibration ciblée -CH2-. - Ajoutez-le au gestionnaire de retour sur investissement en cliquant sur Ajouter [t] dans le gestionnaire de retour sur investissement ou en appuyant sur t sur le clavier. Répétez ce processus pour chaque SG de l’image.
- Pour masquer les régions riches en lipides, sélectionnez chaque retour sur investissement et cliquez sur l’onglet Plus | OU (Combiner) | Ajouter au gestionnaire de retour sur investissement.
- Pour masquer les régions pauvres en lipides, utilisez l’outil rectangle du menu FIDJI et dessinez un carré autour de l’image entière. Ajoutez ceci au gestionnaire de retour sur investissement.
- Cliquez sur le ROI carré nouvellement ajouté en plus du ROI qui sélectionne toutes les régions riches en lipides dans le gestionnaire de ROI. Sous Plus, sélectionnez XOR pour générer un masque des régions pauvres en lipides et l’ajouter au gestionnaire de retour sur investissement.
- Importez une image lipidique SG dans ImageJ (ou Fiji)34 et cochez la case Split Channels pour diviser le fichier en canaux CARS et SRS.
- Chargez les images de l’API dans Fidji.
- Concaténez les images dans l’ordre numérique (c.-à-d. Image0001, Image0002, Image0003, etc.) à l’aide de la séquence de menu suivante : Image | Piles | Outils | Concaténer.
- Vous pouvez également importer ces images en chargeant l’une des images dans Fidji, puis en sélectionnant une option appelée Fichiers de groupe avec des noms similaires sur la page de configuration .
REMARQUE: Cela permet d’importer toutes les images avec un nom de fichier similaire et de les concaténer automatiquement.
- Vous pouvez également importer ces images en chargeant l’une des images dans Fidji, puis en sélectionnant une option appelée Fichiers de groupe avec des noms similaires sur la page de configuration .
- Lorsque l’image concaténée est active, accédez au gestionnaire de retour sur investissement, sélectionnez les régions riches en lipides (c’est-à-dire le SG), cliquez sur Plus, puis sélectionnez Multi-mesure. Attendez que la fenêtre Résultats s’affiche.
- Recherchez Area, Mean, Min, Max et Median dans les paramètres de mesure par défaut. Si d’autres mesures sont souhaitées pour l’analyse, activez ces options en cochant la case correspondante dans la fenêtre Définir les mesures (Analyser | Définir les mesures...).
- Exportez les données de la fenêtre Résultats vers une feuille de calcul et ajoutez une colonne intitulée Région.
- Ajoutez des lipides riches à chaque ligne de données pour les régions riches en lipides. Ajoutez des lipides pauvres aux régions qui étaient en dehors des lipides.
- Ajoutez une colonne intitulée calque et ajoutez le calque correspondant qui a été analysé (tableau supplémentaire S1).
- Répétez les étapes 6.5 à 6.7 pour les régions pauvres en lipides pendant que le retour sur investissement approprié est sélectionné.
- Pour visualiser les données, enregistrez la feuille de calcul et importez-la dans JupyterLab (package matplotlib)35 ou dans R (package ggplot2)36. Tracez les données en fonction du nombre d’images par rapport à l’intensité moyenne pour estimer les données de temps de concentration. (Figure 5).
- Importez la feuille de calcul dans RStudio pour effectuer une analyse non ministérielle (NCA) pour l’analyse pharmacocinétique des données de l’IRC.
- Ajoutez une colonne intitulée heure.
- Pour Image0001, calculez la durée entre l’application de formulation et la première image.
REMARQUE : Il s’agit du premier point temporel. La durée du cycle est utilisée pour calculer les images restantes (et donc les points temporels) qui augmentent avec le temps. Par exemple, si le temps depuis l’application est de 30 min, Image0001 aura un point de temps de 30 min et avec une durée de cycle de 8 min, Image0002 aura un point de temps de 38 min, Image0003 aura un point de temps de 46 min et ainsi de suite. - Exécutez NCA dans RStudio (à l’aide du package NonCompart)37 sur les données intensité-temps importées à partir de la feuille de calcul, avec l’appel suivant pour une couche/région :
sNCA(x = temps, y = moyenne, dose = 1, tempsUnité = « s », doseUnité ="mg »)
Lorsque x fait référence à des points temporels, y fait référence à l’intensité, et la dose peut être laissée comme un, calculée comme la dose mM du médicament dans la formulation ou la dose du produit.
REMARQUE: La sortie NCA fournira des paramètres tels que Cmax et AUCtous. Cependant, comme il s’agit d’une peau ex vivo , ces paramètres sont, en fait, Jmax et LEFLUX ASC-all. - Comparez visuellement les mesures de flux Jmax et AUC-all en les traçant (Figure 6) en plus des comparaisons statistiques entre les conditions expérimentales. Voir la figure 6 pour un exemple d’analyse d’études ex vivo sur l’IRC.
REMARQUE : Le ou les tests statistiques appropriés dépendent de chaque ensemble de données spécifique. Il est également important de noter que tous les paramètres pharmacocinétiques sont normalement distribués par logarithme et que toute comparaison doit utiliser les données log-transformées (logarithme naturel ou log10).
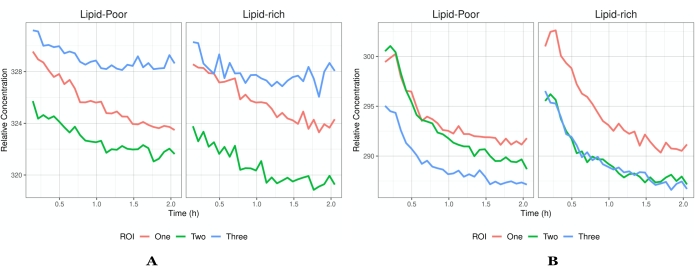
Figure 5 : Intensité vs profils temporels. (A) On voit un exemple de profils de flux qui ont atteint la saturation et donc seulement une diminution de l’intensité. Chaque ROI a un profil de flux différent pour démontrer l’hétérogénéité des données que l’on pourrait acquérir. (B) Un exemple de concentrations qui augmentent après le début de l’imagerie. Chaque ROI est un champ de vision différent (indiqué par les différentes traces de couleur) au sein du même tissu de la même expérience. En plus des concentrations mondiales, il est possible d’élucider l’environnement local qu’une API / formulation préfère, comme indiqué par les régions riches en lipides et pauvres en lipides. Les profils présentés dans A indiquent qu’il n’y a pas d’absorption du médicament dans le tissu car l’IPA a déjà pénétré et a commencé à quitter le tissu une fois que l’imagerie a commencé. Cependant, dans B, le tissu n’a pas atteint la saturation et il y a toujours absorption de l’API suivie d’une élimination. La segmentation des images en lipides riches et lipidiques aidera à élucider la localisation de l’API (ou inactifs) et des voies de perméation dans la peau (c’est-à-dire la couche cornée). Une concentration plus élevée dans les régions riches en lipides indique que l’IPA se localise dans la structure lipidique de la couche étudiée, ce qui facilite l’obtention d’informations ciblées sur l’administration de médicaments. Abréviations : ROI = région d’intérêt ; API = ingrédient pharmaceutique actif. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
L’imagerie est considérée comme réussie si le tissu ne s’est pas déplacé de manière significative dans la direction axiale (<10 μm) ou latérale à la fin de l’expérience (figure 4). Il s’agit d’une indication immédiate si la mesure SRS pour l’API d’intérêt n’est pas représentative de la profondeur initiale, pour laquelle la quantification est spécifique à la couche. Ceci est atténué par l’imagerie des piles z pour chaque position XY d’intérêt, le compro...
Access restricted. Please log in or start a trial to view this content.
Discussion
L’évaluation du BA/BE topique est un domaine de recherche qui nécessite une approche à multiples facettes, car aucune méthode ne peut à elle seule caractériser pleinement la cPK in vivo . Ce protocole présente une méthodologie pour l’évaluation du BA/BE d’un produit pharmaceutique topique basée sur une imagerie Raman cohérente. L’un des premiers points qui pourraient être négligés est la finesse des échantillons de peau, en particulier pour l’imagerie SRS à transmission quantitative. Si...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
CLE est un inventeur de brevets pour la microscopie CARS qui ont été concédés sous licence à plusieurs fabricants de microscopes. Tous les autres auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs tiennent à remercier le Dr Fotis Iliopoulos et Daniel Greenfield du Groupe Evans pour leur discussion et leur relecture de ce manuscrit. En outre, les auteurs tiennent à remercier LEO Pharma pour leur soutien. La figure 2 a été créée avec BioRender.com.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Tissue Preparation | |||
| Autoclavable Biohazard Bags | FisherBrand | 22-044562 | As refered to in text: biohazard bags https://www.fishersci.com/shop/products/fisherbrand-polyethylene-biohazard-autoclave-bags-without-sterilization-indicator-8/22044562?searchHijack=true&searchTerm= 22044562&searchType=RAPID& matchedCatNo=22044562 |
| Cell Culture Buffers: Dulbecco's Phosphate-Buffered Salt Solution 1x | Corning | MT21030CV | As refered to in text: PBS https://www.fishersci.com/shop/products/corning-cellgro-cell-culture-buffers-dulbecco-s-phosphate-buffered-salt-solution-1x-8/MT21030CV?searchHijack=true&searchTerm= 21-030-cv&searchType= RAPID&matchedCatNo=21-030-cv |
| Disposable Scalpels | Exel International | 14-840-00 | As refered to in text: scalpel https://www.fishersci.com/shop/products/exel-international-disposable-scalpels-3/1484000?keyword=true |
| High Precision 45° Angle Broad Point Tweezers/Forceps | Fisherbrand | 12-000-132 | As refered to in text: forceps https://www.fishersci.com/shop/products/high-precision-45-angle-broad-point-tweezers-forceps/12000132#?keyword= |
| Kimwipes Delicate Task Wipers, 1-Ply | Kimberly-Clark Professional Kimtech Science | 06-666 | As refered to in text: task wiper https://www.fishersci.com/shop/products/kimberly-clark-kimtech-science-kimwipes-delicate-task-wipers-7/06666 |
| Parafilm M Laboratory Wrapping Film | Bemis | 13-374-12 | As refered to in text: parafilm https://www.fishersci.com/shop/products/curwood-parafilm-m-laboratory-wrapping-film-4/1337412 |
| Petri Dish (35 mm x 10 mm) | Fisherbrand | FB0875711YZ | As refered to in text: small petri dish https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-specialty-6/FB0875711YZ?keyword=true |
| Petri Dish (60 mm x 15 mm) | Fisherbrand | FB0875713A | As refered to in text: large petri dish https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-12/FB0875713A?keyword=true |
| Surgical Scissors | Roboz | NC9411473 | As refered to in text: scissors https://www.fishersci.com/shop/products/scissors-327/NC9411473?searchHijack=true&searchTerm= RS-5915SC&searchType=RAPID& matchedCatNo=RS-5915SC |
| Laser/microscope | |||
| 650/60 nm BrightLine single-band bandpass filter | Semrock | As refered to in text: CARS filter - CH2 vibrations (645nm/60nm filter) | |
| Control box IX2-UCB | Olympus | As refered to in text: Control Box | |
| D700/30m | Chroma | As refered to in text: CARS filter - deuterated band https://www.chroma.com/products/parts/d700-30m | |
| DeepSee Insight | Spectra-Physics | As refered to in text: Laser https://www.spectra-physics.com/f/insight-x3-tunable-laser | |
| Digital Handheld Optical Power and Energy Meter Console | ThorLabs | PM100D | As refered to in text: power meter https://www.thorlabs.com/newgrouppage9.cfm?objectgroup_id=3341 |
| Fluoview Software | Olympus | As refered to in text: Microscope Control software | |
| Frosted Microscope Slides | FisherBrand | As refered to in text: microscope slides https://www.fishersci.com/shop/products/fisherbrand-frosted-microscope-slides-4/22265446 | |
| FV1000 | Olympus | As refered to in text: Microscope | |
| Incubation Chamber | Tokai Hit | GM-800 | As refered to in text: incubation chamber |
| Integrating Sphere Photodiode Power Sensor | ThorLabs | S142C | As refered to in text: photodiode https://www.thorlabs.com/newgrouppage9.cfm?objectgroup_id=3341 |
| Power supply FV31-PSU | Olympus | As refered to in text: Power Supply | |
| Precision 4063, 80MHz Dual Channel Function Generator | BK Precision | As refered to in text: function generator | |
| ProScan – Precision Microscope Automation | Prior Scientific Instruments | As refered to in text: stage controller https://www.prior.com/microscope-automation/inverted-microscope-systems/proscan-linear-stage-highest-precision-microscope-automation | |
| SecureSeal Imaging Spacers | Grace Biolabs | 654004 | As refered to in text: spacer https://gracebio.com/product/secureseal-imaging-spacers-654004/ |
| SRS Detection Kit | APE | As refered to in text: SRS detector | |
| UPLSAPO 20X NA:0.75 | Olympus | As refered to in text: 20X Objective https://www.olympus-lifescience.com/en/objectives/uplsapo/ | |
| Lipid/Drug Imaging | |||
| 35 mm Dish, No. 0 Uncoated Coverslip, 14 mm Glass Diameter | MatTek Corporation | NC9711297 | As refered to in text: Glass bottom dish https://www.fishersci.com/shop/products/glass-bottom-mircrowell-dish/nc9711297 |
| Cotton-tipped applicators | FisherBrand | As refered to in text: Cotton-tipped applicator | |
| Distriman Postive Displacement Pipette | Gilson | As refered to in text: Postive Displacement Pipette https://www.fishersci.com/shop/products/gilson-distriman-positive-displacement-repetitive-pipette/F164001G#?keyword= | |
| Distriman Postive Displacement Pipette Tips | Gilson | As refered to in text: Tips for pipette https://www.fishersci.com/shop/products/gilson-distritip-syringes-6/f164100g?keyword=true | |
| Data Analysis | |||
| FIJI | Open-source | As refered to in text: FIJI/ImageJ https://imagej.net/software/fiji/ | |
| Jupyter-Lab | open-source | As refered to in text: JupyterLab https://jupyter.org/ | |
| Rstudio | Open-source | As refered to in text: Rstudio https://www.rstudio.com/ |
Références
- Finnin, B., Walters, K. A., Franz, T. J. In vitro skin permeation methodology. In Transdermal and topical drug delivery: principles and methodology. Transdermal and topical drug delivery: principles and practice. Benson, H. E., Watkinson, A. C. , John Wiley & Sons. Hoboken NJ USA. 85-108 (2012).
- Shin, S. H., et al. On the road to development of an in vitro permeation test (IVPT) model to compare heat effects on transdermal delivery systems: exploratory studies with nicotine and fentanyl. Pharmaceutical Research. 34 (9), 1817-1830 (2017).
- Hossain, A., et al. Preparation, characterisation, and topical delivery of terbinafine. Pharmaceutics. 11 (10), 548(2019).
- Santos, L. L., Swofford, N. J., Santiago, B. G. In vitro permeation test (IVPT) for pharmacokinetic assessment of topical dermatological formulations. Current Protocols in Pharmacology. 91 (1), 79(2020).
- Iliopoulos, F., Caspers, P. J., Puppels, G. J., Lane, M. E. Franz cell diffusion testing andquantitative confocal Raman spectroscopy: In vitro-in vivo correlation. Pharmaceutics. 12 (9), 887(2020).
- Cordery, S., et al. Topical bioavailability of diclofenac from locally-acting, dermatological formulations. International Journal of Pharmaceutics. 529 (1-2), 55-64 (2017).
- Pensado, A., et al. Stratum corneum sampling to assess bioequivalence between topicalacyclovir products. Pharmaceutical Research. 36 (12), 1-16 (2019).
- Zhang, Y., et al. Dermal delivery of niacinamide-in vivo studies. Pharmaceutics. 13 (5), 726(2021).
- Bodenlenz, M., et al. Open flow microperfusion as a dermal pharmacokinetic approach to evaluate topical bioequivalence. Clinical Pharmacokinetics. 56 (1), 91-98 (2017).
- Eirefelt, S., et al. Evaluating dermal pharmacokinetics and pharmacodymanic effect of soft topical PDE4 inhibitors:Open flow microperfusion and skin biopsies. Pharmaceutical Research. 37 (12), 1-12 (2020).
- Stagni, G., O'Donnell, D., Liu, Y. J., Kellogg, J. D. L., Shepherd, A. M. Iontophoretic current and intradermal microdialysis recovery in humans. Journal of Pharmacological and Toxicological Methods. 41 (1), 49-54 (1999).
- Garcia Ortiz, P., Hansen, S. H., Shah, V. P., Menne, T., Benfeldt, E. Impact of adultatopic dermatitis on topical drug penetration: assessment by cutaneous microdialysis and tape stripping. Acta Dermato-Venereologica. 89 (1), 33-38 (2009).
- Joshi, A., Patel, H., Joshi, A., Stagni, G. Pharmacokinetic applications of cutaneous microdialysis: Continuous+intermittent vs continuous-only sampling. Journal of Pharmacological and Toxicological Methods. 83, 16-20 (2017).
- Kuzma, B. A., et al. Evaluation of local bioavailability of metronidazole from topical formulations using dermal microdialysis: Preliminary study in a Yucatan mini-pig model. European Journal of Pharmaceutical Sciences. 159, 105741(2021).
- Begley, R., Harvey, A., Byer, R. L.Coherent anti-Stokes Raman spectroscopy. Applied Physics Letters. 25 (7), 387-390 (1974).
- Evans, C. L., et al. Chemical imaging of tissue in vivo with video-rate coherent anti-Stokes Raman scattering microscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (46), 16807-16812 (2005).
- Hill, A. H., Manifold, B., Fu, D. Tissue imaging depth limit of stimulated Raman scattering microscopy. Biomedical Optics Express. 11 (2), 762-774 (2020).
- Feizpour, A., Marstrand, T., Bastholm, L., Eirefelt, S., Evans, C. L. Label-free quantification of pharmacokinetics in skin with stimulated Raman scattering microscopy and deep learning. Journal of Investigative Dermatology. 141 (2), 395-403 (2021).
- Ghosh, B., Reddy, L. H., Kulkarni, R. V., Khanam, J. Comparison of skin permeability of drugs in mice and human cadaver skin. Indian Journal of Experimental Biology. 38 (1), 42-45 (2000).
- Nielsen, J. B., Plasencia, I., Sørensen, J. A., Bagatolli, L. Storage conditions of skin affect tissue structure and subsequent in vitro percutaneous penetration. Skin Pharmacology and Physiology. 24 (2), 93-102 (2011).
- Barbero, A. M., Frasch, H. F. Effect of frozen human epidermis storage duration and cryoprotectant on barrier function using two model compounds. Skin Pharmacology and Physiology. 29 (1), 31-40 (2016).
- Babu, R., et al. The influence of various methods of cold storage of skin on the permeation of melatonin and nimesulide. Journal of Controlled Release. 86 (1), 49-57 (2003).
- Skelly, J. P., et al. FDA and AAPS report of the workshop on principles and practices of in vitro percutaneous penetration studies: relevance to bioavailability and bioequivalence. Pharmaceutical Research. 4 (3), 265-267 (1987).
- OECD. Guidance document for the conduct of skin absorption studies. OECD. , (2004).
- OECD. Test no. 428: Skin absorption: In vitro method. OECD. , (2004).
- Saar, B. G., et al. Video-rate molecular imaging in vivo with stimulated Raman scattering. Science. 330 (6009), 1368-1370 (2010).
- Saar, B. G., Contreras-Rojas, L. R., Xie, X. S., Guy, R. H. Imaging drug delivery to skin with stimulated Raman scattering microscopy. Molecular Pharmaceutics. 8 (3), 969-975 (2011).
- Pence, I. J., Kuzma, B. A., Brinkmann, M., Hellwig, T., Evans, C. L. Multi-windowsparse spectral sampling stimulated Raman scattering microscopy. Biomedical Optics Express. 12 (10), 6095-6114 (2021).
- Herkenne, C., et al. In vivo methods for the assessment of topical drug bioavailability. Pharmaceutical Research. 25 (1), 87-103 (2008).
- Alfonso-Garcıa, A., Mittal, R., Lee, E. S., Potma, E. O. Biological imaging with coherent Raman scattering microscopy: a tutorial. Journal of Biomedical Optics. 19 (7), 071407(2014).
- Osseiran, S., et al. Longitudinal monitoring of cancer cell subpopulations in monolayers, 3D spheroids, and xenografts using the photoconvertible dye DiR. Scientific Reports. 9 (1), 1-10 (2019).
- Evennett, P. Kohler illumination: a simple interpretation. Proceedings of the Royal Microscopical Society. 28 (4), 189-192 (1983).
- Sanderson, J. Fundamentals of microscopy. Current Protocols in Mouse Biology. 10 (2), 76(2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Hunter, J. D. Matplotlib: A 2D graphics environment. Computing in Science & Engineering. 9 (3), 90-95 (2007).
- Wickham, H. ggplot2: Elegant Graphics for Data Analysis. , Springer-Verlag. New York. (2016).
- Kim, H., Han, S., Cho, Y. S., Yoon, S. K., Bae, K. Development of R packages:'Non-Compart' and 'ncar' for noncompartmental analysis (NCA). Translational and Clinical Pharmacology. 26 (1), 10-15 (2018).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon