Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ensayo de la unidad formadora de microcolonias para la evaluación de la eficacia de las vacunas contra la tuberculosis
En este artículo
Resumen
La determinación de las unidades formadoras de colonias (UFC) es la técnica de referencia para cuantificar las bacterias, incluida la Mycobacterium tuberculosis , que puede tardar semanas en formar colonias visibles. Aquí describimos una micro-UFC para la determinación de UFC con mayor eficiencia de tiempo, menor espacio de laboratorio y costo de reactivos, y escalabilidad a experimentos de rendimiento medio y alto.
Resumen
La tuberculosis (TB), la principal causa de muerte en todo el mundo por un agente infeccioso, mató a 1,6 millones de personas en 2022, solo superada por la COVID-19 durante la pandemia de 2019-2021. La enfermedad es causada por la bacteria Mycobacterium tuberculosis (M.tb). La cepa de Mycobacterium bovis Bacillus Calmette-Guérin (BCG), la única vacuna contra la tuberculosis, es la vacuna autorizada más antigua del mundo, todavía en uso. Actualmente, hay 12 vacunas en ensayos clínicos y docenas de vacunas en desarrollo preclínico. El método de elección utilizado para evaluar la eficacia de las vacunas contra la tuberculosis en estudios preclínicos es la enumeración de las colonias bacterianas mediante el ensayo de unidades formadoras de colonias (UFC). Este ensayo, que requiere mucho tiempo, tarda entre 4 y 6 semanas en concluir, requiere un espacio considerable en el laboratorio y la incubadora, tiene altos costes de reactivos y es propenso a la contaminación. Aquí describimos un método optimizado para la enumeración de colonias, la micro-UFC (mCFU), que ofrece una solución simple y rápida para analizar los resultados de eficacia de la vacuna M.tb . El ensayo mCFU requiere diez veces menos reactivos, reduce el período de incubación tres veces, tardando de 1 a 2 semanas en concluir, reduce el espacio de laboratorio y el costo de los reactivos, y minimiza los riesgos para la salud y la seguridad asociados con el trabajo con grandes cantidades de M.tb. Además, para evaluar la eficacia de una vacuna contra la tuberculosis, se pueden obtener muestras de diversas fuentes, incluidos tejidos de animales vacunados infectados con micobacterias. También describimos un método optimizado para producir un cultivo de micobacterias unicelular, uniforme y de alta calidad para estudios de infección. Por último, proponemos que estos métodos se adopten universalmente para los estudios preclínicos de determinación de la eficacia de las vacunas, lo que en última instancia conducirá a una reducción del tiempo en el desarrollo de vacunas contra la tuberculosis.
Introducción
La tuberculosis (TB) es la principal causa de muerte en todo el mundo por un solo agente infeccioso, la bacteria Mycobacterium tuberculosis (M.tb), que mata a más personas que cualquier otro patógeno. En 2021, la tuberculosis fue responsable de 1,6 millones de muertes y fue superada por la COVID-19 durante la pandemia de 2019-20211. Además, según el informe mundial sobre la tuberculosis de 2022 de la Organización Mundial de la Salud, la pandemia de COVID-19 fue responsable de un aumento de los nuevos casos de tuberculosis. La OMS también informa de grandes descensos en el número de personas diagnosticadas con tuberculosis durante este período, lo que podría aumentar aún más el número de casos de tuberculosis1.
El Bacillus Calmette-Guérin (BCG) es una cepa viva atenuada de la patógena Mycobacterium bovis, utilizada por primera vez como vacuna hace más de 100 años. Esta es la única vacuna contra la tuberculosis y es la vacuna autorizada más antigua en el mundo que aún se utiliza 2,3. Actualmente, hay 12 vacunas en diferentes fases de ensayos clínicos4, y decenas de vacunas están en desarrollo preclínico 5,6. La evaluación preclínica de las vacunas contra la TB incluye la evaluación de la seguridad e inmunogenicidad7, que se puede obtener en diversos modelos animales como pez cebra, ratones, cobayas, conejos, bovinos y primates no humanos 8,9,10. Además, la evaluación de la capacidad de una vacuna para inducir protección contra la infección y/o transmisión por M.tb, es decir, la eficacia de la vacuna, requiere un desafío con M.tb in vivo 5,11. Curiosamente, la vacunación con BCG induce efectos inespecíficos que afectan la supervivencia de otros patógenos bacterianos y virales12,13 a través del mecanismo de inmunidad entrenada14. Para cuantificar la carga bacteriana viable en un animal infectado, el método de elección es la enumeración de colonias bacterianas a través del ensayo de unidades formadoras de colonias (UFC) 5,15. La UFC es una unidad que estima el número de microorganismos (bacterias u hongos) que forman colonias en condiciones específicas de crecimiento. Las UFC se originan a partir de microorganismos viables y replicativos, y el número absoluto de microorganismos vivos dentro de cada colonia es difícil de estimar. No se sabe con certeza si una colonia se ha originado a partir de uno o más microorganismos. La unidad UFC refleja esta incertidumbre, por lo que se puede observar una gran variabilidad en las réplicas de una misma muestra. Este ensayo, que requiere mucho tiempo, requiere técnicos especializados capacitados para trabajar en una instalación de nivel de bioseguridad 3 (BSL3), un espacio considerable en el laboratorio y la incubadora, tarda de 4 a 6 semanas en concluir y es propenso a la contaminación.
En este estudio, describimos un método optimizado para la enumeración de colonias, la micro-UFC (mCFU), y ofrecemos una solución simple y rápida para analizar los resultados 15,16,17,18,19,20. El ensayo mCFU requiere diez veces menos reactivos, reduce el período de incubación tres veces, tardando de 1 a 2 semanas en concluir, reduce el espacio de laboratorio y el costo de los reactivos, y minimiza los riesgos para la salud y la seguridad asociados con el trabajo con grandes cantidades de M.tb. Proponemos que este método se adopte universalmente para los estudios preclínicos de determinación de la eficacia de las vacunas, lo que en última instancia conducirá a una reducción del tiempo en el desarrollo de vacunas contra la tuberculosis. Finalmente, este método optimizado de enumeración de UFC se ha utilizado para cuantificar no solo micobacterias sino también otras bacterias, como Escherichia coli y Ralstonia solanacearum21.
Protocolo
NOTA: El protocolo descrito aquí es para BCG, pero se puede aplicar a cualquier micobacteria. La BCG puede utilizarse como bacteria sustituta para experimentos de tuberculosis cuando no se dispone de instalaciones BSL322. Los siguientes procedimientos con BCG deben realizarse en un laboratorio de nivel de bioseguridad 2 (BSL2) y seguir las directrices de bioseguridad y las buenas prácticas de laboratorio adecuadas para la manipulación de microorganismos del grupo de riesgo 2.
1. Preparación de los medios de cultivo
- Prepare el caldo Middlebrook 7H9 suplementado con enriquecimiento de ácido oleico, albúmina, dextrosa y catalasa (OADC) al 10% (v/v), de acuerdo con las instrucciones del proveedor. Complementar el caldo con 0,05% (v/v) de tyloxapol.
NOTA: Tyloxapol es un polímero líquido no iónico que se ha utilizado como tensioactivo para prevenir la formación de grumos bacterianos16. - Prepare el medio sólido Middlebrook 7H10 suplementado con un enriquecimiento de OADC al 10% (v/v) de acuerdo con las instrucciones del proveedor.
- Distribuya 40 ml de medio por placa de Petri cuadrada (120 mm x 120 mm). Deje que las placas se sequen para minimizar la condensación en la superficie del agar.
NOTA: Este tamaño específico de placa de Petri es fundamental para permitir la transposición directa de al menos 96 gotas de una placa de 96 pocillos. El secado efectivo de las placas facilitará posteriormente el recubrimiento de pequeñas gotas de suspensión bacteriana y evitará que las gotas se propaguen. - Prepare el medio del Roswell Park Memorial Institute (RPMI 1640) o el medio Eagle modificado de Dulbecco (DMEM) para producir el medio de infección. En cualquier caso, complemente el medio con un 10% de suero fetal de ternera, un 1% de L-glutamina y 1 mM de piruvato de sodio. No agregue penicilina ni estreptomicina al medio.
2. Preparación de la muestra
- Obtenga muestras de una variedad de fuentes. Por lo general, para cuantificar las UFC y evaluar la eficacia de una vacuna contra la tuberculosis, se adquieren muestras de tejidos animales vacunados y no vacunados. Por ejemplo, pulmón y bazo de ratón11 o pulmón de macaco, ganglios linfáticos torácicos y periféricos, bazo, hígado, piel, sangre, médula ósea y lavado broncoalveolar23. Alternativamente, obtener muestras de cultivos in vitro de macrófagos/células dendríticas/neutrófilos infectados con BCG 18,19,20,24,25,26.
3. Producción de cultivo de BCG
NOTA: En el caso de los estudios in vivo de vacunas contra la tuberculosis, el objetivo es mejorar la eficacia de la BCG. Por lo tanto, los grupos vacunados con BCG suelen utilizarse como control. Las cepas de BCG utilizadas para la vacunación humana son ideales para realizar pruebas en modelos animales. En este caso, se debe reconstituir un cultivo de BCG de acuerdo con las instrucciones del proveedor27. Sin embargo, también se puede producir internamente un cultivo de BCG para estudios in vivo 11. La producción de cultivos unicelulares, uniformes y de alta calidad de BCG para protocolos de infección in vitro se ha producido con mucho éxito en varios estudios 11,16,18,19,20,26,28,29, utilizando el siguiente protocolo, que también puede ser utilizado para estudios de desafío con animales.
- Cultivo 50 mL de BCG en caldo 7H9, a 37 °C, con agitación a 200 rpm. Varía el volumen según las necesidades del experimento.
- Todos los días, durante 8-10 días, recoja 100 μL del cultivo y dilúyalo agregando 900 μL de PBS en una cubeta de 1 mL. A continuación, proceda a medir la densidad óptica de las bacterias (OD a λ=600 nm; OD600) en un espectrofotómetro. Dibuje una curva de crecimiento a partir de esos valores. Identifique la fase logarítmica media del cultivo (cuando el DO se duplica constantemente por unidad de tiempo).
- Preparar un cultivo posterior e incubar hasta llegar a la fase media/tardía de crecimiento de los troncos como en los pasos 3.1 y 3.2. Utilice los valores obtenidos en el paso anterior como guía. Asegúrese de que el cultivo no llegue a la fase de crecimiento estacionario (cuando la OD comienza a estabilizarse) para mantener un cultivo de bacterias viables de buena calidad.
- Recoja el cultivo en la fase de crecimiento del tronco medio/tardío. Centrifugar a 3000 x g durante 10 min. Retire el sobrenadante.
- Agregue 10 ml de PBS para lavar las bacterias. Centrifugar a 3000 x g durante 10 min. Retire el sobrenadante.
- Vuelva a suspender las bacterias con 5 ml de medio infeccioso. Coloque el tubo en un baño de ultrasonido durante 15 minutos, a máxima potencia a 80 Hz.
- Centrifugar a 1000 x g durante 10 min. Recoja el sobrenadante evitando el gránulo ya que es rico en grumos bacterianos que deben evitarse en un cultivo de BCG de alta calidad y deséchelo.
- Mida el diámetro exterior del sobrenadante. Aquí, los cultivos en la fase de crecimiento exponencial, con un DO600 de 0,1, equivalen a 1 x 107 UFC/mL.
NOTA: Cada laboratorio debe producir sus propias curvas de crecimiento de BCG antes de comenzar los experimentos para establecer una regresión lineal entre OD600 y UFC utilizando el espectrofotómetro. Tenga en cuenta que los espectrofotómetros tienen diferentes distancias de trayectoria de luz, lo que puede variar las lecturas obtenidas para la misma muestra. - Realice cálculos sencillos para establecer el número de bacterias que se añadirán a cada cultivo de células huésped. El número de bacterias por célula huésped es la Multiplicidad de Infección (MOI). Utilice un MOI de 10 bacterias por célula huésped, que es el MOI más común utilizado para los experimentos de infección por BCG.
4. Ensayo de la unidad formadora de microcolonias
NOTA: Después de completar un experimento de infección in vivo o in vitro , la enumeración de bacterias se puede realizar mediante mCFU. Para los estudios in vivo , las muestras deben homogeneizarse primero en un batidor de perlas u otro homogeneizador de tejidos. Para cultivos in vitro de macrófagos/células dendríticas/neutrófilos infectados con BCG, las muestras deben lisarse utilizando un detergente no iónico (por ejemplo, una solución al 0,05% de detergente no iónico y no desnaturalizante).
- Diluciones seriadas utilizando una placa de 96 pocillos: realizar diluciones seriadas de 10 veces de los lisados, en una placa estéril de 96 pocillos de acuerdo con el esquema de la Figura 1A. Distribuya los lisados en las filas A y E. Para cada placa, el número máximo de muestras y/o réplicas es de 24.
- Agregue 180 μL de dH2O a los pocillos restantes para realizar la dilución en serie.
- Con una pipeta de 12 canales, vuelva a suspender los lisados en la fila A y transfiera 20 μL a la fila B (20 μL lisado + 180 μL dH2O). Homogeneizar bien. Repita secuencialmente este paso para las filas B y C hasta llegar a la última dilución en la fila D.
NOTA: Solemos realizar tres diluciones (100, 101, 102, 103), utilizando así 4 filas de la placa (A-D o E-H) para cada conjunto de 12 muestras y/o réplicas. - Micro recubrimiento de gotas: utilice una pipeta multicanal de 0,5-10 μL (se prefieren puntas finas) para transferir 5 μL de cada fila de la placa de 96 pocillos a la placa cuadrada mediana sólida, de acuerdo con la Figura 1B.
- Mientras pipetea lentamente las gotas de 5 μL, deje que toquen ligeramente el agar. Esto ayudará a despegar la gota de la punta hacia el agar y reducirá la posibilidad de retención del líquido dentro de la punta.
- Deje que las gotas se sequen, cierre la placa de agar e incube a 37 °C mientras controla el crecimiento bacteriano. Opcionalmente, incube las placas de agar en una bolsa de plástico sellada para evitar que las placas se sequen.
- Recuento de microcolonias: después de aproximadamente 6-10 días de incubación, comprobar si hay colonias individuales, visibles a simple vista (Figura 2).
- Cuente las colonias utilizando el objetivo de aumento más bajo (4x o menos) de un microscopio óptico invertido o una lupa. Los recuentos deben realizarse en las diluciones donde el número de colonias sea inferior a 300 y superior a 30. Alternativamente, use una cámara para tomar una foto de la gota para contar manualmente las colonias en la computadora o use un software como ImageJ para automatizar el conteo de colonias.
- Para expresar el número de celdas en UFC/ml, utilice la siguiente ecuación:
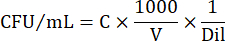
Donde C = número de colonias contadas, V = volumen sembrado en μL y Dil = dilución donde se contaron las colonias (100, 101, 102, 103). Por ejemplo, si se contaron 30 colonias en una gota de 5 μL en la dilución 102, entonces: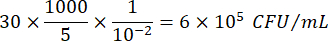
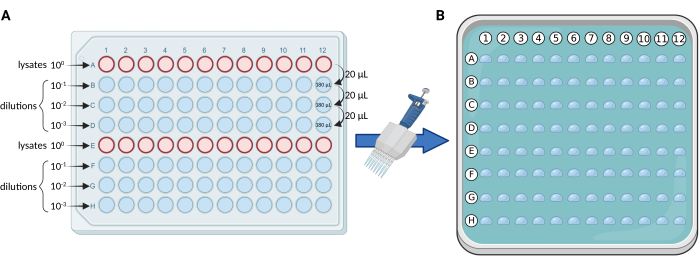
Figura 1. Representación esquemática del protocolo mCFU. (A) Diluciones seriadas de 10 veces de los lisados que contienen BCG en una placa de 96 pocillos. (B) Placa de Petri cuadrada que contiene medio de cultivo sólido y recubierta por 96 gotas de 5 μL cada una. Las gotas se pipetean directamente desde la placa de 96 pocillos utilizando una pipeta multicanal. Creado con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
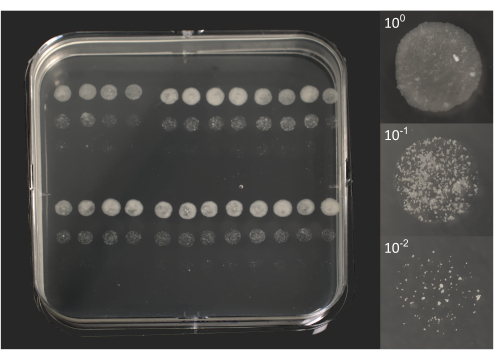
Figura 2. Unidades formadoras de microcolonias de BCG después de 10 días de incubación. A la izquierda, una foto de una placa de Petri cuadrada superpuesta por 96 gotas de 5 μL cada una, como se representó anteriormente en la Figura 1B. A la derecha, fotos individuales de 3 gotas corresponden a un lisado original (100) y dos diluciones (101, 102). Las fotos se tomaron con una cámara DSRL equipada con una lente de zoom de 18-55 mm (placa) o una lente macro de 105 mm (gotas). Haga clic aquí para ver una versión más grande de esta figura.
5. Recuento de unidades formadoras de microcolonias en Fiji (ImagenJ)
NOTA: El método mCFU permite la cuantificación de UFC de grandes conjuntos de muestras. Se pueden registrar imágenes de las gotitas para su posterior análisis y facilitar el recuento de colonias. Varios dispositivos fotográficos pueden producir imágenes con la calidad suficiente para este propósito. Estos incluyen cámaras digitales, cámaras web, microscopios y lupas conectados a la cámara y teléfonos celulares. El software gratuito de análisis de imágenes, como ImageJ, ofrece la posibilidad de realizar un recuento manual o automatizado de colonias en esas imágenes. Para demostrar ambos métodos, se utilizará Fiji, que es una distribución de ImageJ que empaqueta varias herramientas para el análisis científico de imágenes30. Fiji se puede descargar desde https://fiji.sc/.
- Método de conteo manual
- Abra la imagen que contiene la mCFU en Fiji. Seleccione Plugins > analizar > contador de celdas.
- En el menú Contador de celdas, seleccione Inicializar y, a continuación, seleccione un contador (por ejemplo, Tipo 1).
- Proceda haciendo clic en cada colonia. Cada clic se mostrará en la imagen y actualizará el contador (Figura 3). Para deshacer clics accidentales, seleccione Eliminar.
- Registre el valor que se muestra en el contador. Haga clic en el botón Restablecer para restablecer el recuento y abrir una nueva imagen para contar muestras adicionales.
NOTA: Puede encontrar más instrucciones sobre este complemento en https://imagej.net/plugins/cell-counter.
- Método de conteo automatizado
- Abra la imagen que contiene la mCFU en Fiji. Seleccione Imagen > Escriba > 8 bits. Esto convertirá la imagen en una imagen en escala de grises de 8 bits.
- Seleccione la herramienta Óvalo en la barra de herramientas y dibuje un óvalo alrededor del área con las colonias (Figura 4A). El óvalo se puede ajustar después de ser dibujado.
- Seleccione Editar > Borrar exterior para eliminar cualquier interferencia del área exterior (Figura 4B). Seleccione Imagen > ajuste > umbral.
- Mueva los controles deslizantes en el menú de umbral hasta que las colonias aparezcan en rojo y se minimice el ruido de fondo (Figura 4C).
- Seleccione Aplicar y salga de la ventana de umbral. Se genera una imagen en blanco y negro (Figura 4D).
- Seleccione Analizar > Analizar partículas. En la ventana analizar partículas, especifique el rango del área de colonia (entre 1 e infinito, medido en píxeles al cuadrado) y la circularidad (entre 0 y 1, donde 1 es un círculo perfecto; Figura 4E).
- Seleccione Contornos en el menú emergente Mostrar. Marque Mostrar resultados para obtener mediciones detalladas de cada colonia en la ventana de resultados. Marque Borrar resultados para borrar cualquier medición anterior. Marque la casilla Resumir para mostrar los resultados resumidos de las mediciones (Figura 4E).
- Inicie el analizador seleccionando Aceptar. Aparece una nueva ventana que muestra todas las colonias delineadas que se detectaron y contaron. La ventana de resultados muestra los detalles de cada colonia y la ventana de resultados resumidos muestra el total de colonias contadas (Figura 4F).
NOTA: Los ajustes de tamaño y circularidad variarán según la resolución y el aumento de la imagen y el tamaño y la forma de las colonias. Repita el proceso varias veces hasta que se encuentren los mejores ajustes que detecten todas las colonias. Puede encontrar más instrucciones sobre el complemento de análisis de partículas en https://imagej.nih.gov/ij/docs/menus/analyze.html#ap.
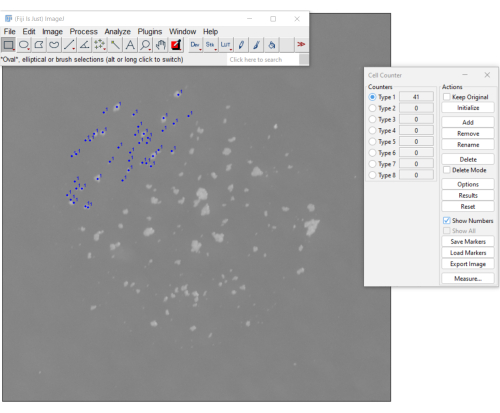
Figura 3. Un método manual para contar mCFU utilizando el complemento de contador de celdas en el software Fiji. Los puntos azules indican colonias en las que el usuario ya ha hecho clic. El menú de la derecha muestra el número de colonias contadas hasta el momento (el recuento es 41). Haga clic aquí para ver una versión más grande de esta figura.

Figura 4. Un método automatizado para contar mCFU utilizando el software Fiji. (A, B) La región de interés con las colonias se selecciona con la herramienta de selección ovalada y el área exterior se elimina con el comando borrar exterior. (C, D) Se genera una imagen en blanco y negro de las colonias utilizando la herramienta de umbral. (E, F) El número de colonias se cuantifica con la herramienta analizar partículas. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
El ensayo mCFU descrito aquí aumenta la cantidad de información que se puede recuperar de una sola placa de Petri a al menos 96 veces. En la figura 5 se muestra una comparación de dos métodos de administración de fármacos para el uso reutilizado de saquinavir (SQV)31,32 como fármaco dirigido al huésped para tratar la tuberculosis. En este ensayo, se utilizaron cuatro cepas diferentes de Mycobacterium tuberculosis par...
Discusión
La tuberculosis es un importante problema de salud pública con una importancia creciente, especialmente en los países de ingresos bajos y medios. La interrupción de los entornos sanitarios para diagnosticar y tratar la tuberculosis durante la pandemia de COVID-19 tuvo un impacto negativo en la incidencia de nuevos casos1. Además, las cepas de M.tb multirresistentes y ampliamente resistentes, así como la coinfección de M.tb y VIH, deben abordarse con urgencia para controlar ...
Divulgaciones
DP y PJGB declaran que el estudio se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Agradecimientos
Este trabajo contó con el apoyo de financiación interna de la Facultad de Medicina de la Universidad Católica Portuguesa y de la Fundación para la Ciencia y Tecnología (FCT), en el marco de las subvenciones UIDP/04279/2020, UIDB/04279/2020 y EXPL/SAU-INF/0742/2021.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well plates | VWR | 734-2781 | |
| DSLR 15-55 mm lens | Nikon | AF-P DX NIKKOR 18-55mm f/3.5-5.6G VR | |
| DSLR camera | Nikon | D3400 | |
| DSLR macro lens | Sigma | MACRO 105mm F2.8 EX DG OS HSM | |
| Fetal calf serum | Gibco | 10270106 | |
| Fiji Software | https://fiji.sc/ | Fiji is an open-source software supported by several laboratories, institutions, and individuals. All the required plugins are included. | |
| Igepal CA-630 | Sigma-Aldrich | 18896 | |
| L-glutamine | Gibco | 25030-081 | |
| Middlebrook 7H10 | BD | 262710 | |
| Middlebrook 7H9 | BD | 271310 | |
| Multichannel pipette (0.5 - 10 µl) | Gilson | FA10013 | |
| Multichannel pipette (20 - 200 µl) | Gilson | FA10011 | |
| Mycobacterium bovis BCG | American Type Culture Collection | ATCC35734 | strain TMC 1011 [BCG Pasteur] |
| OADC enrichment | BD | 211886 | |
| Phosphate buffered saline (PBS) | NZYTech | MB25201 | |
| RPMI 1640 medium | Gibco | 21875091 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Spectrophotometer UV-6300PC | VWR | 634-6041 | |
| Square Petri dish 120 x 120 mm | Corning | BP124-05 | |
| Tyloxapol | Sigma-Aldrich | T8761 | |
| Ultrasound bath Elma P 30 H | VWR | 142-0051 |
Referencias
- World Health Organization. . Global Tuberculosis Report 2022. , (2022).
- Bettencourt, P. J. G., Joosten, S. A., Lindestam Arlehamn, C. S., Behr, M. A., Locht, C., Neyrolles, O. 100 years of the Bacillus Calmette-Guérin vaccine. Vaccine. 39 (50), 7221-7222 (2021).
- Bettencourt, P. J. G. The 100th anniversary of bacille Calmette-Guérin (BCG) and the latest vaccines against COVID-19. The International Journal of Tuberculosis and Lung Disease. 25 (8), 611-613 (2021).
- Scriba, T. J., Netea, M. G., Ginsberg, A. M. Key recent advances in TB vaccine development and understanding of protective immune responses against Mycobacterium tuberculosis. Seminars in Immunology. 50, 101431 (2020).
- McShane, H., Williams, A. A review of preclinical animal models utilised for TB vaccine evaluation in the context of recent human efficacy data. Tuberculosis. 94 (2), 105-110 (2014).
- Voss, G., et al. Progress and challenges in TB vaccine development. F1000Research. 7, 199 (2018).
- Satti, I., McShane, H. Current approaches toward identifying a correlate of immune protection from tuberculosis. Expert Review of Vaccines. 18 (1), 43-59 (2019).
- Young, D. Animal models of tuberculosis. European Journal of Immunology. 39 (8), 2011-2014 (2009).
- Pedroza-Roldán, C., Flores-Valdez, M. A. Recent mouse models and vaccine candidates for preventing chronic/latent tuberculosis infection and its reactivation. Pathogens and disease. 75 (6), (2017).
- Gong, W., Liang, Y., Wu, X. Animal Models of Tuberculosis Vaccine Research: An Important Component in the Fight against Tuberculosis. BioMed Research International. 2020, 1-21 (2020).
- Bettencourt, P., et al. Identification of antigens presented by MHC for vaccines against tuberculosis. NPJ vaccines. 5 (1), 2 (2020).
- Moorlag, S. J. C. F. M., Arts, R. J. W., van Crevel, R., Netea, M. G. Non-specific effects of BCG vaccine on viral infections. Clinical Microbiology and Infection. 25 (12), 1473-1478 (2019).
- Wilkie, M., et al. Functional in-vitro evaluation of the non-specific effects of BCG vaccination in a randomised controlled clinical study. Scientific Reports. 12 (1), 7808 (2022).
- Netea, M. G., et al. Trained immunity: A program of innate immune memory in health and disease. Science. 352 (6284), aaf1098 (2016).
- Bettencourt, P., Pires, D., Carmo, N., Anes, E. Application of Confocal Microscopy for Quantification of Intracellular Mycobacteria in Macrophages. Microscopy: Science, Technology, Applications and Education. 1, 614-621 (2010).
- Bettencourt, P., Carmo, N., Pires, D., Timóteo, P., Anes, E. Mycobacterial infection of macrophages: the effect of the multiplicity of infection. Antimicrobial research: Novel bioknowledge and educational programs. , 651-664 (2017).
- Pires, D., Bettencourt, P., Carmo, N., Niederweis, M., Anes, E. Role of Mycobacterium tuberculosis outer-membrane porins in bacterial survival within macrophages. Drug Discovery Today. 15 (23-24), 1112-1113 (2010).
- Pires, D., et al. Mycobacterium tuberculosis Modulates miR-106b-5p to Control Cathepsin S Expression Resulting in Higher Pathogen Survival and Poor T-Cell Activation. Frontiers in immunology. 8 (DEC), 1819 (2017).
- Pires, D., et al. Role of Cathepsins in Mycobacterium tuberculosis Survival in Human Macrophages. Scientific reports. 6 (August), 32247 (2016).
- Bettencourt, P., et al. Actin-binding protein regulation by microRNAs as a novel microbial strategy to modulate phagocytosis by host cells: the case of N-Wasp and miR-142-3p. Frontiers in cellular and infection microbiology. 3 (June), 19 (2013).
- Bhuyan, S., et al. Microliter spotting and micro-colony observation: A rapid and simple approach for counting bacterial colony forming units. Journal of Microbiological Methods. 207, 106707 (2023).
- Jackson, S., McShane, H. Challenges in Developing a Controlled Human Tuberculosis Challenge Model. Current topics in microbiology and immunology. , 1-27 (2022).
- Darrah, P. A., et al. Prevention of tuberculosis in macaques after intravenous BCG immunization. Nature. 577 (7788), 95-102 (2020).
- Madura Larsen, J., et al. BCG stimulated dendritic cells induce an interleukin-10 producing T-cell population with no T helper 1 or T helper 2 bias in vitro. Immunology. 121 (2), 276-282 (2007).
- Bickett, T. E., et al. Characterizing the BCG-Induced Macrophage and Neutrophil Mechanisms for Defense Against Mycobacterium tuberculosis. Frontiers in immunology. 11, 1202 (2020).
- Pires, D., et al. Interference of Mycobacterium tuberculosis with the endocytic pathways on macrophages and dendritic cells from healthy donors: role of cathepsins. Drug Discovery Today. 15 (23-24), 1112-1112 (2010).
- Betts, G., et al. Optimising Immunogenicity with Viral Vectors: Mixing MVA and HAdV-5 Expressing the Mycobacterial Antigen Ag85A in a Single Injection. PLoS ONE. 7 (12), e50447 (2012).
- Tanner, R., et al. The influence of haemoglobin and iron on in vitro mycobacterial growth inhibition assays. Scientific reports. 7 (1), 43478 (2017).
- McNeill, E., et al. Regulation of mycobacterial infection by macrophage Gch1 and tetrahydrobiopterin. Nature communications. 9 (1), 5409 (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Pereira, M., Vale, N. Saquinavir: From HIV to COVID-19 and Cancer Treatment. Biomolecules. 12 (7), 944 (2022).
- Pires, D., et al. Repurposing Saquinavir for Host-Directed Therapy to Control Mycobacterium Tuberculosis Infection. Frontiers in immunology. 12, 647728 (2021).
- Pires, D., et al. Liposomal Delivery of Saquinavir to Macrophages Overcomes Cathepsin Blockade by Mycobacterium tuberculosis and Helps Control the Phagosomal Replicative Niches. International journal of molecular sciences. 24 (2), (2023).
- Maartens, G., Wilkinson, R. J. Tuberculosis. The Lancet. 370 (9604), 2030-2043 (2007).
- Matarazzo, L., Bettencourt, P. J. G. mRNA vaccines: a new opportunity for malaria, tuberculosis and HIV. Frontiers in Immunology. 14, 1172691 (2023).
- Young, D., Dye, C. The Development and Impact of Tuberculosis Vaccines. Cell. 124 (4), 683-687 (2006).
- Kommareddi, S., Abramowsky, C. R., Swinehart, G. L., Hrabak, L. Nontuberculous mycobacterial infections: Comparison of the fluorescent auramine-o and Ziehl-Neelsen techniques in tissue diagnosis. Human Pathology. 15 (11), 1085-1089 (1984).
- Sabiiti, W., et al. A Tuberculosis Molecular Bacterial Load Assay (TB-MBLA). Journal of visualized experiments: JoVE. (158), e60460 (2020).
- Somoskövi, A., et al. Comparison of Recoveries of Mycobacterium tuberculosis Using the Automated BACTEC MGIT 960 System, the BACTEC 460 TB System, and Löwenstein-Jensen Medium. Journal of Clinical Microbiology. 38 (6), 2395-2397 (2000).
- Tanner, R., et al. The in vitro direct mycobacterial growth inhibition assay (MGIA) for the early evaluation of TB vaccine candidates and assessment of protective immunity: a protocol for non-human primate cells. F1000Research. 10, 257 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados