È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Saggio dell'unità formante microcolonie per la valutazione dell'efficacia dei vaccini contro la tubercolosi
In questo articolo
Riepilogo
La determinazione delle unità formanti colonie (CFU) è la tecnica gold standard per quantificare i batteri, tra cui il Mycobacterium tuberculosis che può richiedere settimane per formare colonie visibili. Qui descriviamo un micro-CFU per la determinazione di CFU con una maggiore efficienza temporale, una riduzione dello spazio di laboratorio e del costo dei reagenti e la scalabilità per esperimenti a media e alta produttività.
Abstract
La tubercolosi (TB), la principale causa di morte in tutto il mondo per un agente infettivo, ha ucciso 1,6 milioni di persone nel 2022, superata solo dal COVID-19 durante la pandemia 2019-2021. La malattia è causata dal batterio Mycobacterium tuberculosis (M.tb). Il ceppo di Mycobacterium bovis Bacillus Calmette-Guérin (BCG), l'unico vaccino contro la tubercolosi, è il più antico vaccino autorizzato al mondo, ancora in uso. Attualmente, ci sono 12 vaccini in fase di sperimentazione clinica e dozzine di vaccini in fase di sviluppo preclinico. Il metodo di scelta utilizzato per valutare l'efficacia dei vaccini contro la tubercolosi negli studi preclinici è l'enumerazione delle colonie batteriche mediante il test delle unità formanti colonie (CFU). Questo test richiede dalle 4 alle 6 settimane per essere concluso, richiede un notevole spazio in laboratorio e nell'incubatore, ha costi elevati per i reagenti ed è soggetto a contaminazione. Qui descriviamo un metodo ottimizzato per l'enumerazione delle colonie, il micro-CFU (mCFU), che offre una soluzione semplice e rapida per analizzare i risultati dell'efficacia del vaccino contro la tubercolosi . Il test mCFU richiede un numero dieci volte inferiore di reagenti, riduce di tre volte il periodo di incubazione, impiegando da 1 a 2 settimane per concludersi, riduce lo spazio di laboratorio e il costo dei reagenti e riduce al minimo i rischi per la salute e la sicurezza associati al lavoro con un gran numero di M.tb. Inoltre, per valutare l'efficacia di un vaccino contro la tubercolosi, è possibile ottenere campioni da una varietà di fonti, compresi tessuti di animali vaccinati infettati da micobatteri. Descriviamo anche un metodo ottimizzato per produrre una coltura micobatterica unicellulare, uniforme e di alta qualità per gli studi sulle infezioni. Infine, proponiamo che questi metodi siano universalmente adottati per gli studi preclinici sulla determinazione dell'efficacia dei vaccini, portando in ultima analisi a una riduzione dei tempi di sviluppo dei vaccini contro la tubercolosi.
Introduzione
La tubercolosi (TBC) è la principale causa di morte in tutto il mondo a causa di un singolo agente infettivo, il batterio Mycobacterium tuberculosis (M.tb), che uccide più persone di qualsiasi altro agente patogeno. Nel 2021, la tubercolosi è stata responsabile di 1,6 milioni di decessi ed è stata superata dalla COVID-19 durante la pandemia 2019-20211. Inoltre, secondo il rapporto globale sulla tubercolosi del 2022 dell'Organizzazione Mondiale della Sanità, la pandemia di COVID-19 è stata responsabile di un aumento dei nuovi casi di tubercolosi. L'OMS segnala anche un forte calo del numero di persone a cui è stata diagnosticata la tubercolosi durante questo periodo, il che potrebbe aumentare ulteriormente il numerodi casi di tubercolosi.
Il Bacillus Calmette-Guérin (BCG) è un ceppo vivo attenuato del patogeno Mycobacterium bovis, utilizzato per la prima volta come vaccino più di 100 anni fa. Questo è l'unico vaccino contro la tubercolosi ed è il più antico vaccino autorizzato al mondo ancora in uso 2,3. Attualmente, ci sono 12 vaccini in diverse fasi di sperimentazione clinica4 e dozzine di vaccini sono in fase di sviluppo preclinico 5,6. La valutazione preclinica dei vaccini contro la tubercolosi include la valutazione della sicurezza e dell'immunogenicità7, che possono essere ottenute in diversi modelli animali come pesce zebra, topi, porcellini d'India, conigli, bovini e primati non umani 8,9,10. Inoltre, la valutazione della capacità di un vaccino di indurre protezione contro l'infezione e/o la trasmissione di M.tb, cioè l'efficacia del vaccino, richiede un challenge di M.tb in vivo 5,11. È interessante notare che la vaccinazione BCG induce effetti non specifici che influenzano la sopravvivenza di altri patogeni batterici e virali12,13 attraverso il meccanismo dell'immunità addestrata14. Per quantificare la carica batterica vitale in un animale infetto, il metodo di scelta è l'enumerazione delle colonie batteriche attraverso il test delle unità formanti colonie (CFU) 5,15. La CFU è un'unità che stima il numero di microrganismi (batteri o funghi) che formano colonie in specifiche condizioni di crescita. I CFU provengono da microrganismi vitali e replicativi e il numero assoluto di microrganismi viventi all'interno di ciascuna colonia è difficile da stimare. Non è chiaro se una colonia abbia avuto origine da uno o più microrganismi. L'unità CFU riflette questa incertezza, quindi si può osservare una grande variabilità nelle repliche dello stesso campione. Questo test richiede molto tempo e tecnici specializzati formati per lavorare in una struttura di livello di biosicurezza 3 (BSL3), un ampio spazio di laboratorio e incubatore, richiede da 4 a 6 settimane per concludersi ed è soggetto a contaminazione.
In questo studio, descriviamo un metodo ottimizzato per l'enumerazione delle colonie, il micro-CFU (mCFU), e offriamo una soluzione semplice e rapida per analizzare i risultati 15,16,17,18,19,20. Il test mCFU richiede un numero dieci volte inferiore di reagenti, riduce di tre volte il periodo di incubazione, impiegando da 1 a 2 settimane per concludersi, riduce lo spazio di laboratorio e il costo dei reagenti e riduce al minimo i rischi per la salute e la sicurezza associati al lavoro con un gran numero di M.tb. Proponiamo che questo metodo sia universalmente adottato per gli studi preclinici sulla determinazione dell'efficacia dei vaccini, portando in ultima analisi a una riduzione dei tempi di sviluppo dei vaccini contro la tubercolosi. Infine, questo metodo ottimizzato di enumerazione CFU è stato utilizzato per quantificare non solo i micobatteri ma anche altri batteri, come Escherichia coli e Ralstonia solanacearum21.
Protocollo
NOTA: Il protocollo qui descritto è per BCG ma può essere applicato a qualsiasi Micobatterio. Il BCG può essere utilizzato come batterio surrogato per gli esperimenti sulla tubercolosi quando le strutture BSL3 non sono disponibili22. Le seguenti procedure che utilizzano BCG devono essere eseguite in un laboratorio di livello di biosicurezza 2 (BSL2) e seguire le linee guida di biosicurezza appropriate e le buone pratiche di laboratorio per la manipolazione di microrganismi del gruppo di pericolo 2.
1. Preparazione dei terreni di coltura
- Preparare il brodo Middlebrook 7H9 integrato con il 10% (v/v) di acido oleico, albumina, destrosio e arricchimento di catalasi (OADC), secondo le istruzioni del fornitore. Integrare il brodo con lo 0,05% (v/v) di tyloxapol.
NOTA: Tyloxapol è un polimero liquido non ionico che è stato utilizzato come tensioattivo per prevenire la formazione di grumi batterici16. - Preparare il terreno solido Middlebrook 7H10 integrato con il 10% (v/v) di arricchimento OADC secondo le istruzioni del fornitore.
- Distribuire 40 mL di terreno per capsula di Petri quadrata (120 mm x 120 mm). Lasciare asciugare le piastre per ridurre al minimo la formazione di condensa sulla superficie dell'agar.
NOTA: Questa dimensione specifica della capsula di Petri è fondamentale per consentire il trasposizione diretto di almeno 96 goccioline da una piastra a 96 pozzetti. Un'efficace asciugatura delle piastre faciliterà in seguito la placcatura di piccole goccioline di sospensione batterica e impedirà la diffusione delle goccioline. - Preparare il terreno di infezione del Roswell Park Memorial Institute (RPMI 1640) o il terreno DMEM (Modified Eagle Medium) di Dulbecco. In entrambi i casi, integrare il terreno con il 10% di siero fetale di vitello, l'1% di L-glutammina e 1 mM di piruvato di sodio. Non aggiungere penicillina e streptomicina al terreno.
2. Preparazione del campione
- Ottenere campioni da una varietà di fonti. Tipicamente, per quantificare la CFU per valutare l'efficacia di un vaccino contro la tubercolosi, è necessario acquisire campioni da tessuti animali vaccinati e non vaccinati. Ad esempio, polmone e milza di topo11 o polmone di macaco, linfonodi toracici e periferici, milza, fegato, pelle, sangue, midollo osseo e lavaggio broncoalveolare23. In alternativa, prelevare campioni da colture in vitro di macrofagi/cellule dendritiche/neutrofili infettati con BCG 18,19,20,24,25,26.
3. Produzione di coltura BCG
NOTA: Per gli studi in vivo sui vaccini contro la tubercolosi, l'obiettivo è quello di migliorare l'efficacia del BCG. Pertanto, i gruppi vaccinati con BCG sono solitamente utilizzati come controllo. I ceppi di BCG utilizzati per la vaccinazione umana sono ideali per i test su modelli animali. In questo caso, una coltura di BCG deve essere ricostituita secondo le istruzioni del fornitore27. Tuttavia, una coltura BCG per studi in vivo può anche essere prodotta internamente11. La produzione di colture BCG unicellulari, uniformi e di alta qualità per protocolli di infezione in vitro è stata prodotta con grande successo in diversi studi 11,16,18,19,20,26,28,29, utilizzando il seguente protocollo, che può essere utilizzato anche per studi di provocazione su animali.
- Coltura 50 mL di BCG in brodo 7H9, a 37 °C, con agitazione a 200 giri/min. Variare il volume in base alle esigenze dell'esperimento.
- Ogni giorno, per 8-10 giorni, raccogliere 100 μL di coltura e diluirli aggiungendo 900 μL di PBS in una cuvetta da 1 mL. Procedere quindi misurando la densità ottica dei batteri (OD a λ=600 nm; OD600) in uno spettrofotometro. Disegna una curva di crescita a partire da questi valori. Identificare la fase intermedia logaritmica della coltura (quando l'OD raddoppia in modo coerente per unità di tempo).
- Preparare una coltura successiva e incubare fino a raggiungere la fase di crescita logaritmica medio/tardiva come nei passaggi 3.1 e 3.2. Utilizzare i valori ottenuti nel passaggio precedente come guida. Assicurarsi che la coltura non raggiunga la fase di crescita stazionaria (quando l'OD inizia a stabilizzarsi) per mantenere una coltura di buona qualità di batteri vitali.
- Raccogliere la coltura nella fase di crescita del log medio/tardivo. Centrifugare a 3000 x g per 10 min. Rimuovere il surnatante.
- Aggiungere 10 ml di PBS per lavare i batteri. Centrifugare a 3000 x g per 10 min. Rimuovere il surnatante.
- Risospendere i batteri con 5 ml di terreno di infezione. Immergere il tubo in un bagno ad ultrasuoni per 15 minuti, a piena potenza a 80 Hz.
- Centrifugare a 1000 x g per 10 min. Raccogliere il surnatante evitando il pellet in quanto ricco di grumi batterici che dovrebbero essere evitati in una coltura BCG di alta qualità e scartarlo.
- Misurare l'OD del surnatante. In questo caso, le colture in fase di crescita esponenziale, con un OD600 di 0,1, equivalgono a 1 x 107 CFU/mL.
NOTA: Ogni laboratorio dovrebbe produrre le proprie curve di crescita BCG prima di iniziare gli esperimenti per stabilire una regressione lineare tra OD600 e CFU utilizzando lo spettrofotometro. Si prega di notare che gli spettrofotometri hanno diverse distanze del percorso della luce, che possono variare le letture ottenute per lo stesso campione. - Esegui semplici calcoli per stabilire il numero di batteri da aggiungere a ciascuna coltura cellulare ospite. Il numero di batteri per cellula ospite è la molteplicità dell'infezione (MOI). Utilizzare un MOI di 10 batteri per cellula ospite, che è il MOI più comune utilizzato per gli esperimenti di infezione da BCG.
4. Saggio dell'unità formante microcolonie
NOTA: Dopo aver completato un esperimento di infezione in vivo o in vitro , l'enumerazione dei batteri può essere eseguita mediante mCFU. Per gli studi in vivo , i campioni devono essere prima omogeneizzati in un battitore di perline o in un altro omogeneizzatore di tessuti. Per le colture in vitro di macrofagi/cellule dendritiche/neutrofili infettati da BCG, i campioni devono essere lisati utilizzando un detergente non ionico (ad esempio, una soluzione allo 0,05% di detergente non ionico e non denaturante).
- Diluizioni seriali utilizzando una piastra a 96 pozzetti: eseguire diluizioni seriali di 10 volte dei lisati, in una piastra sterile a 96 pozzetti secondo lo schema della Figura 1A. Distribuire i lisati sulle righe A ed E. Per ogni piastra, il numero massimo di campioni e/o repliche è 24.
- Aggiungere 180 μL di dH2O ai pozzetti rimanenti per eseguire la diluizione seriale.
- Utilizzando una pipetta a 12 canali, sospendere nuovamente i lisati nella fila A e trasferire 20 μL nella fila B (20 μL lisato + 180 μL dH2O). Omogeneizzare bene. Ripetere in sequenza questo passaggio per le righe B e C fino a raggiungere l'ultima diluizione nella riga D.
NOTA: Di solito eseguiamo tre diluizioni (100, 101, 102, 103), utilizzando quindi 4 file della piastra (A-D o E-H) per ogni set di 12 campioni e/o repliche. - Placcatura a microgoccia: utilizzare una pipetta multicanale da 0,5-10 μL (sono preferibili puntali sottili) per trasferire 5 μL da ciascuna fila della piastra a 96 pozzetti alla piastra quadrata media solida, secondo la Figura 1B.
- Pipettando lentamente le goccioline da 5 μL, lasciare che tocchino leggermente l'agar. Ciò contribuirà a rimuovere la gocciolina dalla punta verso l'agar e a ridurre la possibilità di ritenzione del liquido all'interno della punta.
- Lasciare asciugare le goccioline, chiudere la piastra di agar e incubarla a 37 °C monitorando la crescita batterica. Facoltativamente, incubare le piastre di agar in un sacchetto di plastica sigillato per evitare che le piastre si secchino.
- Conteggio delle microcolonie: dopo circa 6-10 giorni di incubazione, verificare la presenza di singole colonie, visibili ad occhio nudo (Figura 2).
- Contare le colonie utilizzando l'obiettivo con l'ingrandimento più basso (4x o inferiore) di un microscopio ottico invertito o di una lente d'ingrandimento. I conteggi devono essere eseguiti nelle diluizioni in cui il numero di colonie è inferiore a 300 e superiore a 30. In alternativa, utilizzare una fotocamera per scattare una foto della gocciolina per contare manualmente le colonie sul computer o utilizzare un software come ImageJ per automatizzare il conteggio delle colonie.
- Per esprimere il numero di celle in CFU/mL, utilizzare la seguente equazione:
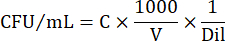
Dove C = numero di colonie contate, V = volume placcato in μL e Dil = diluizione dove sono state contate le colonie (100, 101, 102, 103). Ad esempio, se 30 colonie sono state contate in una gocciolina da 5 μL nella diluizione 102, allora: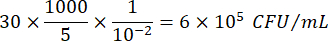
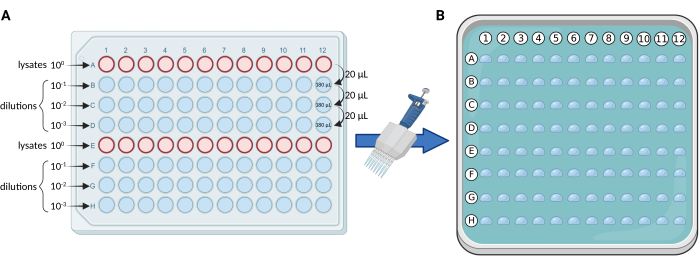
Figura 1. Rappresentazione schematica del protocollo mCFU. (A) Diluizioni seriali di 10 volte dei lisati contenenti BCG in una piastra a 96 pozzetti. (B) Piastra di Petri quadrata contenente terreno di coltura solido e ricoperta da 96 goccioline da 5 μL ciascuna. Le goccioline vengono pipettate direttamente dalla piastra a 96 pozzetti utilizzando una pipetta multicanale. Creato con BioRender.com. Fare clic qui per visualizzare una versione più grande di questa figura.
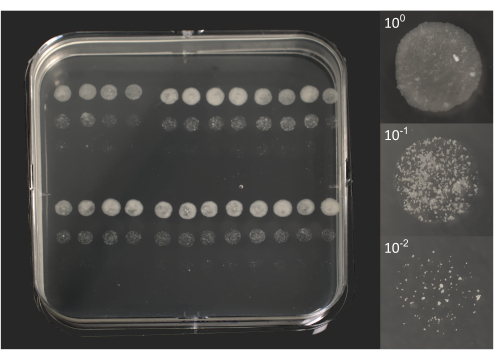
Figura 2. Unità formanti microcolonie di BCG dopo 10 giorni di incubazione. A sinistra, una foto di una capsula di Petri quadrata sovrapposta a 96 goccioline da 5 μL ciascuna, come precedentemente rappresentato nella Figura 1B. A destra, le singole foto di 3 goccioline corrispondono a un lisato originale (100) e due diluizioni (101, 102). Le foto sono state scattate utilizzando una fotocamera DSRL dotata di un obiettivo zoom 18-55 mm (piastra) o di un obiettivo macro da 105 mm (goccioline). Fare clic qui per visualizzare una versione più grande di questa figura.
5. Conteggio delle unità formanti microcolonie nelle Figi (ImageJ)
NOTA: Il metodo mCFU consente la quantificazione CFU di grandi insiemi di campioni. Le immagini delle goccioline possono essere registrate per l'analisi posteriore per facilitare il conteggio delle colonie. Diversi dispositivi fotografici possono produrre immagini con una qualità sufficiente per questo scopo. Questi includono fotocamere digitali, webcam, microscopi e lenti d'ingrandimento collegati alla fotocamera e telefoni cellulari. Un software gratuito di analisi delle immagini come ImageJ offre la possibilità di contare manualmente o automaticamente le colonie in quelle immagini. Per dimostrare entrambi i metodi, verrà utilizzato Fiji, che è una distribuzione di ImageJ che include diversi strumenti per l'analisi scientifica delle immagini30. Le Figi possono essere scaricate da https://fiji.sc/.
- Metodo di conteggio manuale
- Aprire l'immagine contenente l'mCFU in Figi. Seleziona Plugin > Analizza > contatore di celle.
- Nel menu Contatore di celle, selezionare Inizializza , quindi selezionare un contatore (ad esempio, Tipo 1).
- Procedi cliccando su ogni colonia. Ogni clic verrà visualizzato sull'immagine e aggiornerà il contatore (Figura 3). Per annullare i clic accidentali, selezionare Elimina.
- Registrare il valore visualizzato sul contatore. Fare clic sul pulsante Ripristina per reimpostare il conteggio e aprire una nuova immagine per contare altri campioni.
NOTA: Ulteriori istruzioni su questo plugin sono disponibili all'https://imagej.net/plugins/cell-counter.
- Metodo di conteggio automatizzato
- Aprire l'immagine contenente l'mCFU in Figi. Selezionare Immagine > Tipo > 8 bit. Questo convertirà l'immagine in un'immagine in scala di grigi a 8 bit.
- Selezionate lo strumento Ovale nella barra degli strumenti e disegnate un ovale attorno all'area con le colonie (Figura 4A). L'ovale può essere regolato dopo essere stato disegnato.
- Selezionare Modifica > Cancella esterno per rimuovere qualsiasi interferenza dall'area esterna (Figura 4B). Selezionare Immagine > Regola > soglia.
- Spostare i cursori nel menu della soglia fino a quando le colonie non vengono visualizzate in rosso e il rumore di fondo è ridotto al minimo (Figura 4C).
- Selezionare Applica e uscire dalla finestra della soglia. Viene generata un'immagine in bianco e nero (Figura 4D).
- Selezionare Analizza > Analizza particelle. Nella finestra Analizza particelle, specificare l'intervallo per l'area della colonia (compresa tra 1 e infinito, misurata in pixel quadrati) e la circolarità (tra 0 e 1, dove 1 è un cerchio perfetto; Figura 4E).
- Selezionare Contorni nel menu a comparsa Mostra. Controllare Visualizza risultati per misurazioni dettagliate per ogni colonia nella finestra dei risultati. Selezionare Cancella risultati per cancellare eventuali misurazioni precedenti. Selezionare la casella Riepiloga per visualizzare i risultati riepilogativi delle misurazioni (Figura 4E).
- Avviare l'analizzatore selezionando OK. Viene visualizzata una nuova finestra che mostra tutte le colonie delineate che sono state rilevate e conteggiate. Nella finestra dei risultati vengono visualizzati i dettagli di ogni colonia, mentre nella finestra dei risultati riepilogati vengono visualizzati i conteggi totali delle colonie (Figura 4F).
NOTA: Le impostazioni per le dimensioni e la circolarità variano in base alla risoluzione e all'ingrandimento dell'immagine, nonché alle dimensioni e alla forma delle colonie. Ripetere il processo più volte fino a trovare le impostazioni migliori che rilevano tutte le colonie. Ulteriori istruzioni sul plug-in per l'analisi delle particelle sono disponibili all'indirizzo https://imagej.nih.gov/ij/docs/menus/analyze.html#ap.
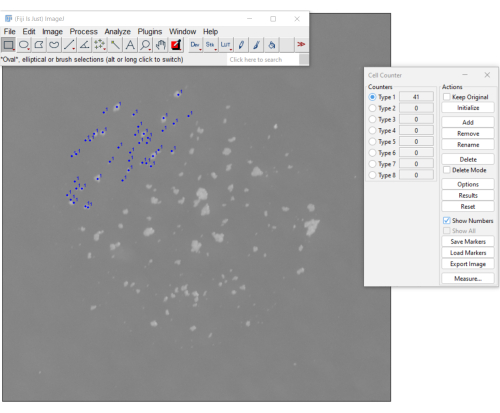
Figura 3. Un metodo manuale per il conteggio di mCFU utilizzando il plug-in del contatore di cellule sul software Fiji. I punti blu indicano le colonie già cliccate dall'utente. Il menu a destra mostra il numero di colonie contate fino a quel momento (il conteggio è 41). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4. Un metodo automatizzato per il conteggio di mCFU utilizzando il software Fiji. (A, B) L'area di interesse con le colonie viene selezionata utilizzando lo strumento di selezione ovale e l'area esterna viene rimossa utilizzando il comando Cancella esterno. (C, D) Un'immagine in bianco e nero delle colonie viene generata utilizzando lo strumento soglia. (E, F) Il numero di colonie viene quantificato utilizzando lo strumento Analizza particelle. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
Il test mCFU qui descritto aumenta la quantità di informazioni che possono essere recuperate da una singola capsula di Petri ad almeno 96 volte. La Figura 5 illustra un confronto tra due metodi di somministrazione del farmaco per l'uso riproposto di saquinavir (SQV)31,32 come farmaco diretto all'ospite per il trattamento della tubercolosi. In questo test, quattro diversi ceppi di Mycobacterium tuberculosis sono stati utiliz...
Discussione
La tubercolosi è un importante problema di salute pubblica con un'importanza crescente, in particolare nei paesi a basso e medio reddito. L'interruzione delle strutture sanitarie per la diagnosi e il trattamento della tubercolosi durante la pandemia di COVID-19 ha causato un impatto negativo sull'incidenza di nuovi casi1. Inoltre, i ceppi di M.tb multi-farmaco ed estensivamente resistenti ai farmaci e la co-infezione di M.tb e HIV devono essere affrontati con urgenza per control...
Divulgazioni
DP e PJGB dichiarano che lo studio è stato condotto in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Riconoscimenti
Questo lavoro è stato sostenuto da finanziamenti interni della Facoltà di Medicina, Universidade Católica Portuguesa, e da finanziamenti esterni della Fundação para a Ciência e a Tecnologia (FCT), nell'ambito delle sovvenzioni UIDP/04279/2020, UIDB/04279/2020 e EXPL/SAU-INF/0742/2021.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well plates | VWR | 734-2781 | |
| DSLR 15-55 mm lens | Nikon | AF-P DX NIKKOR 18-55mm f/3.5-5.6G VR | |
| DSLR camera | Nikon | D3400 | |
| DSLR macro lens | Sigma | MACRO 105mm F2.8 EX DG OS HSM | |
| Fetal calf serum | Gibco | 10270106 | |
| Fiji Software | https://fiji.sc/ | Fiji is an open-source software supported by several laboratories, institutions, and individuals. All the required plugins are included. | |
| Igepal CA-630 | Sigma-Aldrich | 18896 | |
| L-glutamine | Gibco | 25030-081 | |
| Middlebrook 7H10 | BD | 262710 | |
| Middlebrook 7H9 | BD | 271310 | |
| Multichannel pipette (0.5 - 10 µl) | Gilson | FA10013 | |
| Multichannel pipette (20 - 200 µl) | Gilson | FA10011 | |
| Mycobacterium bovis BCG | American Type Culture Collection | ATCC35734 | strain TMC 1011 [BCG Pasteur] |
| OADC enrichment | BD | 211886 | |
| Phosphate buffered saline (PBS) | NZYTech | MB25201 | |
| RPMI 1640 medium | Gibco | 21875091 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Spectrophotometer UV-6300PC | VWR | 634-6041 | |
| Square Petri dish 120 x 120 mm | Corning | BP124-05 | |
| Tyloxapol | Sigma-Aldrich | T8761 | |
| Ultrasound bath Elma P 30 H | VWR | 142-0051 |
Riferimenti
- World Health Organization. . Global Tuberculosis Report 2022. , (2022).
- Bettencourt, P. J. G., Joosten, S. A., Lindestam Arlehamn, C. S., Behr, M. A., Locht, C., Neyrolles, O. 100 years of the Bacillus Calmette-Guérin vaccine. Vaccine. 39 (50), 7221-7222 (2021).
- Bettencourt, P. J. G. The 100th anniversary of bacille Calmette-Guérin (BCG) and the latest vaccines against COVID-19. The International Journal of Tuberculosis and Lung Disease. 25 (8), 611-613 (2021).
- Scriba, T. J., Netea, M. G., Ginsberg, A. M. Key recent advances in TB vaccine development and understanding of protective immune responses against Mycobacterium tuberculosis. Seminars in Immunology. 50, 101431 (2020).
- McShane, H., Williams, A. A review of preclinical animal models utilised for TB vaccine evaluation in the context of recent human efficacy data. Tuberculosis. 94 (2), 105-110 (2014).
- Voss, G., et al. Progress and challenges in TB vaccine development. F1000Research. 7, 199 (2018).
- Satti, I., McShane, H. Current approaches toward identifying a correlate of immune protection from tuberculosis. Expert Review of Vaccines. 18 (1), 43-59 (2019).
- Young, D. Animal models of tuberculosis. European Journal of Immunology. 39 (8), 2011-2014 (2009).
- Pedroza-Roldán, C., Flores-Valdez, M. A. Recent mouse models and vaccine candidates for preventing chronic/latent tuberculosis infection and its reactivation. Pathogens and disease. 75 (6), (2017).
- Gong, W., Liang, Y., Wu, X. Animal Models of Tuberculosis Vaccine Research: An Important Component in the Fight against Tuberculosis. BioMed Research International. 2020, 1-21 (2020).
- Bettencourt, P., et al. Identification of antigens presented by MHC for vaccines against tuberculosis. NPJ vaccines. 5 (1), 2 (2020).
- Moorlag, S. J. C. F. M., Arts, R. J. W., van Crevel, R., Netea, M. G. Non-specific effects of BCG vaccine on viral infections. Clinical Microbiology and Infection. 25 (12), 1473-1478 (2019).
- Wilkie, M., et al. Functional in-vitro evaluation of the non-specific effects of BCG vaccination in a randomised controlled clinical study. Scientific Reports. 12 (1), 7808 (2022).
- Netea, M. G., et al. Trained immunity: A program of innate immune memory in health and disease. Science. 352 (6284), aaf1098 (2016).
- Bettencourt, P., Pires, D., Carmo, N., Anes, E. Application of Confocal Microscopy for Quantification of Intracellular Mycobacteria in Macrophages. Microscopy: Science, Technology, Applications and Education. 1, 614-621 (2010).
- Bettencourt, P., Carmo, N., Pires, D., Timóteo, P., Anes, E. Mycobacterial infection of macrophages: the effect of the multiplicity of infection. Antimicrobial research: Novel bioknowledge and educational programs. , 651-664 (2017).
- Pires, D., Bettencourt, P., Carmo, N., Niederweis, M., Anes, E. Role of Mycobacterium tuberculosis outer-membrane porins in bacterial survival within macrophages. Drug Discovery Today. 15 (23-24), 1112-1113 (2010).
- Pires, D., et al. Mycobacterium tuberculosis Modulates miR-106b-5p to Control Cathepsin S Expression Resulting in Higher Pathogen Survival and Poor T-Cell Activation. Frontiers in immunology. 8 (DEC), 1819 (2017).
- Pires, D., et al. Role of Cathepsins in Mycobacterium tuberculosis Survival in Human Macrophages. Scientific reports. 6 (August), 32247 (2016).
- Bettencourt, P., et al. Actin-binding protein regulation by microRNAs as a novel microbial strategy to modulate phagocytosis by host cells: the case of N-Wasp and miR-142-3p. Frontiers in cellular and infection microbiology. 3 (June), 19 (2013).
- Bhuyan, S., et al. Microliter spotting and micro-colony observation: A rapid and simple approach for counting bacterial colony forming units. Journal of Microbiological Methods. 207, 106707 (2023).
- Jackson, S., McShane, H. Challenges in Developing a Controlled Human Tuberculosis Challenge Model. Current topics in microbiology and immunology. , 1-27 (2022).
- Darrah, P. A., et al. Prevention of tuberculosis in macaques after intravenous BCG immunization. Nature. 577 (7788), 95-102 (2020).
- Madura Larsen, J., et al. BCG stimulated dendritic cells induce an interleukin-10 producing T-cell population with no T helper 1 or T helper 2 bias in vitro. Immunology. 121 (2), 276-282 (2007).
- Bickett, T. E., et al. Characterizing the BCG-Induced Macrophage and Neutrophil Mechanisms for Defense Against Mycobacterium tuberculosis. Frontiers in immunology. 11, 1202 (2020).
- Pires, D., et al. Interference of Mycobacterium tuberculosis with the endocytic pathways on macrophages and dendritic cells from healthy donors: role of cathepsins. Drug Discovery Today. 15 (23-24), 1112-1112 (2010).
- Betts, G., et al. Optimising Immunogenicity with Viral Vectors: Mixing MVA and HAdV-5 Expressing the Mycobacterial Antigen Ag85A in a Single Injection. PLoS ONE. 7 (12), e50447 (2012).
- Tanner, R., et al. The influence of haemoglobin and iron on in vitro mycobacterial growth inhibition assays. Scientific reports. 7 (1), 43478 (2017).
- McNeill, E., et al. Regulation of mycobacterial infection by macrophage Gch1 and tetrahydrobiopterin. Nature communications. 9 (1), 5409 (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Pereira, M., Vale, N. Saquinavir: From HIV to COVID-19 and Cancer Treatment. Biomolecules. 12 (7), 944 (2022).
- Pires, D., et al. Repurposing Saquinavir for Host-Directed Therapy to Control Mycobacterium Tuberculosis Infection. Frontiers in immunology. 12, 647728 (2021).
- Pires, D., et al. Liposomal Delivery of Saquinavir to Macrophages Overcomes Cathepsin Blockade by Mycobacterium tuberculosis and Helps Control the Phagosomal Replicative Niches. International journal of molecular sciences. 24 (2), (2023).
- Maartens, G., Wilkinson, R. J. Tuberculosis. The Lancet. 370 (9604), 2030-2043 (2007).
- Matarazzo, L., Bettencourt, P. J. G. mRNA vaccines: a new opportunity for malaria, tuberculosis and HIV. Frontiers in Immunology. 14, 1172691 (2023).
- Young, D., Dye, C. The Development and Impact of Tuberculosis Vaccines. Cell. 124 (4), 683-687 (2006).
- Kommareddi, S., Abramowsky, C. R., Swinehart, G. L., Hrabak, L. Nontuberculous mycobacterial infections: Comparison of the fluorescent auramine-o and Ziehl-Neelsen techniques in tissue diagnosis. Human Pathology. 15 (11), 1085-1089 (1984).
- Sabiiti, W., et al. A Tuberculosis Molecular Bacterial Load Assay (TB-MBLA). Journal of visualized experiments: JoVE. (158), e60460 (2020).
- Somoskövi, A., et al. Comparison of Recoveries of Mycobacterium tuberculosis Using the Automated BACTEC MGIT 960 System, the BACTEC 460 TB System, and Löwenstein-Jensen Medium. Journal of Clinical Microbiology. 38 (6), 2395-2397 (2000).
- Tanner, R., et al. The in vitro direct mycobacterial growth inhibition assay (MGIA) for the early evaluation of TB vaccine candidates and assessment of protective immunity: a protocol for non-human primate cells. F1000Research. 10, 257 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon