Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aislamiento de núcleos de tejido adiposo intermuscular humano y secuenciación de ARN de núcleos individuales posteriores
En este artículo
Resumen
La biología del tejido adiposo intermuscular (IMAT) está en gran parte inexplorada debido a la limitada accesibilidad del tejido humano. Aquí, presentamos un protocolo detallado para el aislamiento de núcleos y la preparación de bibliotecas de IMAT humano congelado para la secuenciación de ARN de núcleos individuales para identificar la composición celular de este depósito adiposo único.
Resumen
El tejido adiposo intermuscular (IMAT) es un depósito adiposo relativamente poco estudiado situado entre las fibras musculares. El contenido de IMAT aumenta con la edad y el IMC y se asocia con enfermedades metabólicas y degenerativas musculares; sin embargo, la comprensión de las propiedades biológicas de IMAT y su interacción con las fibras musculares circundantes es muy deficiente. En los últimos años, la secuenciación de ARN de una sola célula y de núcleos nos ha proporcionado atlas específicos del tipo de célula de varios tejidos humanos. Sin embargo, la composición celular del IMAT humano sigue siendo en gran medida inexplorada debido a los desafíos inherentes a su accesibilidad a partir de la recolección de biopsias en humanos. Además de la cantidad limitada de tejido recolectado, el procesamiento del IMAT humano es complicado debido a su proximidad al tejido muscular esquelético y la fascia. La naturaleza cargada de lípidos de los adipocitos lo hace incompatible con el aislamiento de una sola célula. Por lo tanto, la secuenciación de ARN de un solo núcleo es óptima para obtener transcriptómica de alta dimensión con resolución de una sola célula y proporciona el potencial de descubrir la biología de este depósito, incluida la composición celular exacta de IMAT. Aquí, presentamos un protocolo detallado para el aislamiento de núcleos y la preparación de bibliotecas de IMAT humano congelado para la secuenciación de ARN de núcleos individuales. Este protocolo permite el perfil de miles de núcleos utilizando un enfoque basado en gotas, lo que proporciona la capacidad de detectar tipos de células raras y poco abundantes.
Introducción
El tejido adiposo intermuscular (IMAT) es un depósito adiposo ectópico que reside entre y alrededor de las fibras musculares1. Como se describe en detalle en una revisión reciente de Goodpaster et al., la IMAT se puede detectar mediante tomografía computarizada (TC) de alta resolución y resonancia magnética (RMN) (Figura 1A, B) y se encuentra alrededor y dentro de las fibras musculares en todo el cuerpo1. La cantidad de IMAT varía mucho entre individuos y está influenciada por el IMC, la edad, el sexo, la raza y el sedentarismo 2,3,4. Además, la deposición de IMAT se observa comúnmente en condiciones patológicas asociadas con la degeneración muscular5, y numerosos estudios han documentado un aumento de la masa de IMAT en individuos con obesidad, diabetes tipo 2, síndrome metabólico y resistencia a la insulina 6,7,8,9. Sin embargo, las propiedades celulares y biológicas de IMAT apenas están comenzando a desentrañarse. La accesibilidad limitada y la variación en las ubicaciones y el contenido de IMAT en todo el cuerpo han dificultado la recolección de muestras de este depósito adiposo único2. Además, las muestras se "contaminan" fácilmente con músculo esquelético (MS) en el momento de la recolección, lo que dificulta el desciframiento de la separación entre la contribución biológica de los diferentes tejidos (Figura 1C). Con este fin, la secuenciación de ARN de un solo núcleo (snRNA-seq), que ha ganado considerable atención durante la última década, sirve como una metodología ideal para permitir la separación de patrones de expresión génica derivados de IMAT y SM con resolución de una sola célula. Además, el aislamiento de núcleos es particularmente útil para el tejido adiposo debido a los grandes adipocitos cargados de lípidos, que son imposibles de disociar en una suspensión unicelular sin comprometer la integridad de las células. Por último, esta tecnología tiene el potencial de descubrir nuevos marcadores de adipocitos específicos de IMAT y descubrir la composición y presencia de diferentes poblaciones de células progenitoras, así como estudiar la variación de la composición celular en condiciones patológicas y normales.
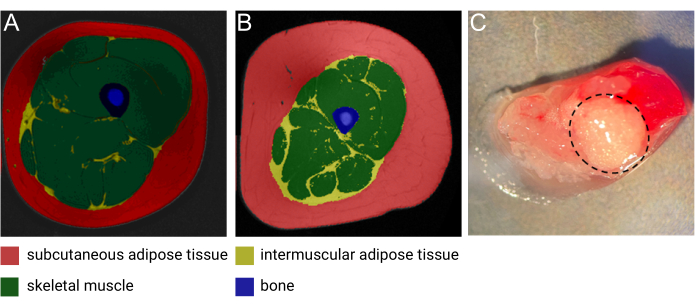
Figura 1: Imágenes de IMAT. Imagen representativa de resonancia magnética (RM) de IMAT de (A) una mujer delgada de mediana edad y (B) un hombre de mediana edad con obesidad. Rojo: tejido adiposo subcutáneo, amarillo: tejido adiposo intermuscular, verde: músculo esquelético, azul: hueso. Imagen cortesía de Heather Cornnell, Instituto de Investigación Traslacional AdventHealth. (C) Muestra de tejido fresco con IMAT (rodeado por una línea negra discontinua). Imagen cortesía de Meghan Hopf, del Instituto de Investigación Traslacional AdventHealth y Bryan Bergman, de la Universidad de Colorado. Esta figura ha sido modificada con permiso de Goodpaster et al.1. Haga clic aquí para ver una versión más grande de esta figura.
Se han publicado varios estudios de la industria ganadera que investigan el marmoleado de la carne (IMAT en particular) en cerdos, pollos y ganado utilizando células individuales (sc) y snRNA-seq10. Estos estudios han identificado varias subpoblaciones de adipocitos y marcadores de potenciales células progenitoras de IMAT 11,12,13; sin embargo, se desconoce si estas composiciones celulares se traducen al IMAT humano. Hasta donde sabemos, solo un estudio ha analizado la heterogeneidad celular del músculo humano con infiltración grasa, obtenido de pacientes masculinos con artrosis de cadera, utilizando snRNA-seq14. Los investigadores reportaron una pequeña población de adipocitos y varias subpoblaciones de progenitores fibroadipogénicos (FAP) dentro de la gran población de mionúcleos14. Nuestro estudio es el primero en desarrollar un método para interrogar directamente IMAT diseccionado manualmente de músculo humano para la composición celular utilizando snRNA-seq.
Es importante destacar que los protocolos para la secuenciación de snRNA deben personalizarse para el tejido específico estudiado, ya que la cantidad de tejido disponible y las propiedades físicas del tejido específico dictarán los pasos de procesamiento óptimos. La producción de tejido para IMAT suele ser pequeña, a menudo no excede los 50 mg, incluso cuando se realizan biopsias guiadas por ecografía. Por lo tanto, el procesamiento adecuado de este tejido escaso es esencial. Creemos que este protocolo servirá como un recurso valioso para los investigadores que estudian el IMAT humano.
Access restricted. Please log in or start a trial to view this content.
Protocolo
La muestra utilizada para este protocolo formó parte del Estudio de Músculo, Movilidad y Envejecimiento (SOMMA)15, que fue aprobado por el Western IRB-Copernicus Group (WCG) Institutional Review Board y se llevó a cabo de acuerdo con la Declaración de Helsinki. Los participantes dieron su consentimiento informado por escrito para su participación en el estudio.
NOTA: Este protocolo es una adaptación de un protocolo anterior utilizando 100 mg de tejido adiposo subcutáneo abdominal humano en una plataforma basada en nanopocillos16. El protocolo actual está optimizado para 50 mg de IMAT humano y la preparación de bibliotecas utilizando una plataforma basada en gotas. Es posible que se requiera una mayor optimización de este protocolo para los aislamientos de núcleos de IMAT no humanos u otros depósitos adiposos.
1. Preparación de tampones y reactivos (Tabla 1 y Tabla 2)
NOTA: Prepare los tampones frescos el día del experimento y no los reutilice.
- Enfriar previamente una centrífuga a 4 °C.
- Preparar el tampón de homogeneización y el medio de aislamiento de núcleos.
- Obtenga dos cubos de hielo y enfríe previamente 2 tubos cónicos de 15 ml.
- Mezcle todos los reactivos para el tampón de homogeneización (HB) en un tubo cónico de 15 mL en el orden indicado en la Tabla 1. Mantener en hielo. Mezclar por vórtice.
- Mezcle todos los reactivos para el medio de aislamiento de núcleos (NIM) en un tubo cónico de 15 mL en la lista ordenada de la Tabla 2. Mantener en hielo. Mezclar por vórtice.
- Prepare Triton-X al 10% añadiendo 100 μL de Triton X-100 a 900 μL de agua libre de nucleasas. Vórtice para asegurar una mezcla adecuada. Mantener a temperatura ambiente (RT).
| Reactivo | Volumen (μL) | Concentración final (mM) | |
| 1 vez | 2 veces | ||
| 1 M MgCl2 | 10 | 20 | 5 |
| 1 m tampón Tris, pH 8.0 | 20 | 40 | 10 |
| 2 M KCl | 25 | 50 | 25 |
| 1,5 m de sacarosa (-4oC) | 334 | 668 | 250 |
| TDT de 1 mM | 2 | 4 | 0,001 (~1 μM) |
| Inhibidor de la proteasa 100x | 20 | 40 | 1 vez |
| Superasin 20 U/μL | 40 | 80 | 0,4 U/μL |
| Agua libre de nucleasas | 1549 | 3098 | - |
| Volumen total | 2000 | 4000 | - |
Tabla 1: Tampón de homogeneización (HB). Mantener en hielo. Mezclar por vórtice.
| Reactivo | Volumen (μL) | Concentración final (mM) | |
| 1 vez | 2 veces | ||
| EDTA | 0.4 | 0.8 | 0.1 |
| Inhibidor de la ARNasa Ribolock (40U/μL) | 40 | 80 | 0,8 U/μL |
| 1% BSA-PBS (-/-) | 1959.6 | 3919.2 | - |
| Volumen total | 2000 | 4000 | - |
Tabla 2: Medio de aislamiento de núcleos (NIM). Mantener en hielo. Mezclar por vórtice.
2. Pulverización de tejido congelado (Figura 2A)
- Configure la estación de trabajo para la homogeneización.
- Llene el bote con nitrógeno líquido (LN2).
PRECAUCIÓN: cuando trabaje con LN2, use siempre gafas y guantes criogénicos. - Obtenga 2 morteros, 1 mortero, 1 espátula de microcuchara, 1 doblón de vidrio y 1 mortero de acero inoxidable para el tapón automático.
- Configura el dosuncer automático.
- Llene un vaso de precipitados con hielo y enfríe previamente la ducha de vidrio.
- Llene el bote con nitrógeno líquido (LN2).
- Llene los 2 morteros (que contienen el mortero y la espátula) con LN2 para enfriar los instrumentos. Deja que el LN2 se evapore y repite.
- Mientras los instrumentos se enfrían, agregue 1 ml de HB a la ducha de vidrio.
- Llene ambos morteros con LN2 por última vez y vierta la muestra de 50 mg de IMAT en uno de los morteros.
- Pulveriza el IMAT con el mortero presionándolo suavemente sobre el trozo de tejido para romperlo en trozos pequeños. Asegúrate de que todas las piezas estén pulverizadas.
- El LN2 se evaporará lentamente mientras pulveriza el tejido. Cuando el tejido esté correctamente pulverizado, y todavía quede 1/4 - 1/2 de un mortero de LN2 , incline el mortero hacia el borde del mortero para recoger el tejido pulverizado por el labio. Deje que el LN2 se evapore por completo.
- Inmediatamente después de que se haya evaporado el último LN2 , coloque el tejido pulverizado en el vaso que contiene 1 mL de HB.
3. Homogeneización del tejido pulverizado
- Homogeneizar el tejido pulverizado con el dosificador automático. Lleve el vidrio hacia arriba y hacia abajo del mortero de acero inoxidable durante 10 golpes en la dirección hacia adelante, seguidos de 10 golpes en la dirección inversa.
- Asegúrese de que la solución esté turbia después de la homogeneización y no contenga trozos visibles de tejido. A menudo se espera un color rosa claro debido a la contaminación con tejido muscular.
- Transfiera el homogeneizado a un tubo de baja unión preenfriado de 1,7 mL en hielo.
- Utilice 400 μL de HB para enjuagar la onza y asegurarse de que todo el material se transfiere y añádalo al tubo.
NOTA: Se pueden procesar dos muestras a la vez. Para hacer esto, duplique la cantidad de HB y NIM. Pulverizar y homogeneizar una muestra de tejido e inmediatamente después pulverizar y homogeneizar la segunda muestra de tejido para poder realizar los pasos de aislamiento y limpieza en paralelo.
4. Aislamiento y limpieza de núcleos (Figura 2B)
- Añadir 14 μL de Triton-X (10%) al homogeneizado para una concentración del 0,1%.
- Mantenga el tubo en hielo y en la oscuridad durante 10-15 minutos mientras realiza un vórtice cada 3 minutos.
- Humedecer previamente un filtro de células de 100 μm y otro de 40 μm (por muestra) con 100 μL de RT DPBS para cada uno en un tubo cónico de 50 mL.
- Filtre el homogeneizado a través del filtro de células de 100 μm.
- Enjuague el tubo de 1,7 ml con 400 μl de HB y filtre a través del filtro de células de 100 μm.
- A continuación, filtre la solución a través del filtro de células de 40 μm.
- Transfiera una cantidad igual de solución a dos tubos de baja unión preenfriados de 1,7 mL, lo que corresponde a ~ 900 μL en cada tubo.
- Centrifugar los tubos durante 10 min a 2700 x g a 4 °C. Debe haber una pequeña bolita visible después de la centrifugación.
- Retire y deseche la capa lipídica superior y el sobrenadante restante, dejando ~50 μL de solución del primer tubo.
- Repita para el segundo tubo.
- Vuelva a suspender completamente el pellet en el primer tubo pipeteando suavemente hacia arriba y hacia abajo 20 veces y transfiéralo a un nuevo tubo de baja fijación de 1,7 ml. Evita crear burbujas.
- Repita este procedimiento para el segundo tubo y transfiera la solución resuspendida al mismo tubo.
- Añadir 500 μL de NIM y mezclar con pipeteo.
- Centrifugar el tubo con una balanza a 1000 x g durante 10 min a 4 °C.
- Retire el sobrenadante, dejando ~50 μL, y pipetee suavemente hacia arriba y hacia abajo hasta que el gránulo se vuelva a suspender. Opcionalmente, transfiera el gránulo resuspendido a un nuevo tubo limpio si queda algún resto de lípidos en el costado del tubo.
- Añadir 200 μL de NIM y mezclar mediante pipeteo.
5. Tinción y recuento de núcleos (Figura 2C y Figura 3)
NOTA : Para facilitar el recuento, establezca un protocolo de "conteo de núcleos" en un contador de células automatizado, ya que el ajuste del campo claro y los canales DAPI puede afectar en gran medida el recuento. Ajuste los canales para que solo se capturen los núcleos y no los residuos. Asegúrese de que el canal de campo claro solo marque los "objetos" que también tengan una mancha DAPI.
- Agregue 1 gota de la solución de tinción de células vivas y déjela en la oscuridad, sobre hielo, durante 15 minutos.
- Filtre la solución a través de un filtro de células de 30 μm.
- Mezcle la solución de núcleos pipeteando y agregue 10 μL de la solución a un portaobjetos de la cámara de recuento de células.
- Cuente los núcleos mediante un contador de células automatizado.
NOTA: La concentración óptima es de 1000 núcleos/μL correspondiente a 1,0 x 106/mL.- Asegúrese de que no haya grumos de núcleos, ya que esto podría obstruir el chip para generar gotas de núcleos individuales (Figura 3).
- Si la concentración de núcleos no es lo suficientemente alta, centrifugar la solución a 1000 x g durante 10 minutos a 4 °C para obtener un pellet, retirar el sobrenadante y volver a suspender en un volumen más pequeño.
- Si el grado de residuos en la solución es alto, vuelva a suspender la solución de los núcleos en un volumen mayor de NIM (es decir, 1 mL) y vuelva a filtrar a través de un filtro de células de 30 μm. A continuación, centrifugar a 1000 x g durante 10 min a 4 °C y volver a suspender en el volumen adecuado en relación con la concentración de núcleos.
- Después de obtener la concentración de núcleos, proceda directamente al primer paso en la preparación de la biblioteca.
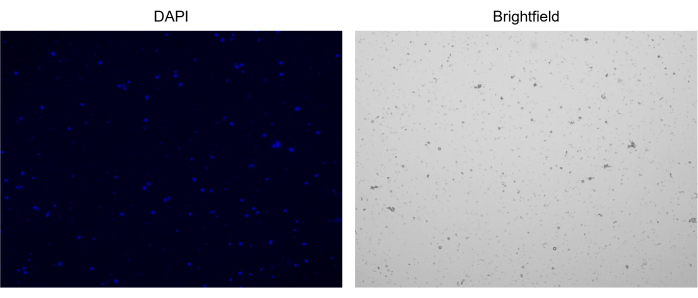
Figura 3: Tinción de núcleos aislados. Imagen del contador celular de núcleos teñidos con NucBlue/DAPI (imagen de la izquierda) y la imagen de campo claro correspondiente (imagen de la derecha). La presencia de pequeñas cantidades de escombros es evidente en la imagen de campo claro. El contador de celdas automatizado utilizado aquí no tiene una opción para incluir barras de escala. Haga clic aquí para ver una versión más grande de esta figura.
6. Parámetros de preparación y secuenciación de la biblioteca
- Consulte un protocolo completo para la preparación de bibliotecas utilizando el enfoque de núcleo único basado en gotas disponible en la página web del proveedor17.
- Apunta a una recuperación de núcleos objetivo de 10.000. Sin embargo, en el caso de muestras con un alto nivel de escombros o núcleos frágiles, se anticiparía un menor número de núcleos recuperados.
- Almacene las muestras a 4 °C durante un máximo de 72 horas después del paso 2.3. en el protocolo de preparación de la biblioteca para combinar el procesamiento de más muestras en paralelo. Para ello, procese dos muestras hasta el paso 2.3 en dos días consecutivos y, al tercer día, procese las 4 muestras juntas desde el paso 3 en adelante en el protocolo de preparación de la biblioteca.
- Parámetros de secuenciación: Secuencia en una plataforma de secuenciación con el objetivo de 50.000 lecturas de extremos emparejados por núcleo.
NOTA: Los datos presentados en este protocolo se secuenciaron en la plataforma NovaSeq 6000, con el objetivo de realizar 50.000 lecturas de extremos emparejados por núcleo.
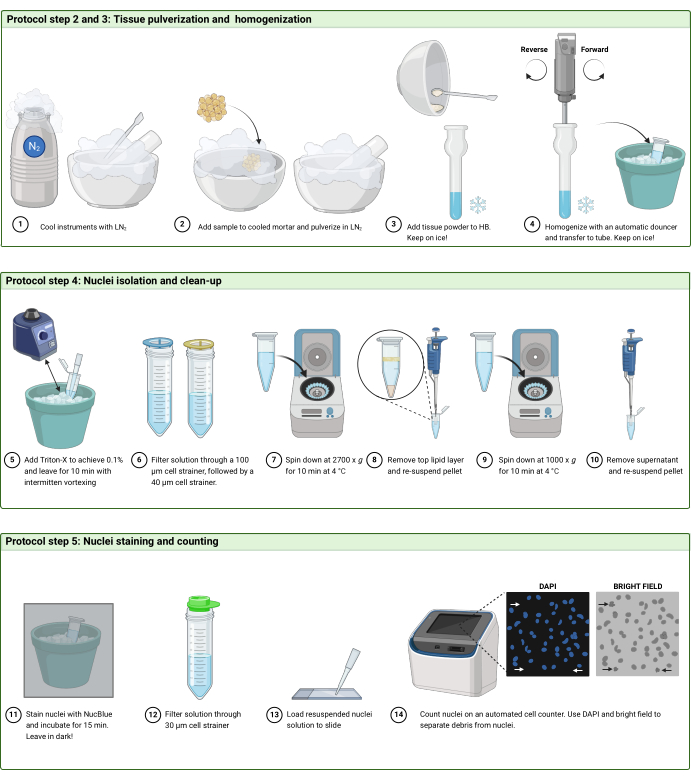
Figura 2: Flujo de trabajo del protocolo. Ilustración esquemática del flujo de trabajo en (A) pasos 2 y 3, (B) paso 4 y (C) paso 5 del protocolo. La figura fue creada con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
7. Procesamiento y análisis de datos
NOTA : En este protocolo, se introducen brevemente algunos de los programas informáticos y paquetes de R recomendados para procesar los datos de secuenciación resultantes, centrándose en los pasos posteriores al preprocesamiento inicial (Tabla 3). Este estudio proporciona métricas generales de control de calidad (QC) y un ejemplo de aproximación y proyección uniforme de variedades (UMAP) en la Figura 4. Sin embargo, una descripción en profundidad del análisis bioinformático está fuera del alcance de este protocolo. Por lo tanto, los lectores pueden consultar la reciente revisión sobre las mejores prácticas para el análisis de células individuales de Heumos et al.18.
- Preprocesamiento de los datos de secuenciación
- Mapea las lecturas de núcleos individuales al genoma humano de referencia GRCh38.
- Incluya las lecturas de intrones en el recuento.
- Realice el control de calidad y el filtrado de los datos utilizando el paquete Seurat R 19.
- Calcule una puntuación de complejidad celular dividiendo el número log(10) de genes detectados por el número log(10) de lecturas detectadas.
- Represente las métricas de control de calidad más importantes mediante un histograma o un gráfico de violín, incluido el número de genes detectados por núcleo, el porcentaje de lectura mitocondrial y la puntuación de complejidad celular.
- Filtre los núcleos con menos de 200 o más de 10.000 genes por núcleo, más del 10% de lecturas mitocondriales y una puntuación de complejidad inferior a 0,8.
- Normalice los datos y realice la reducción de la dimensionalidad.
- Utilice la función SCTransform de Seurat para normalizar los datos utilizando 2000 características variables.
- Agrupe los datos mediante las siguientes funciones del paquete R de Seurat: RunPCA, FindNeighbors, FindClusters y RunUMAP.
- Trace un UMAP para visualizar la agrupación en clústeres de los datos.
- Filtre los dobletes pronosticados con el paquete DoubletFinder R 20 y vuelva a agrupar los datos.
- Anote los grupos utilizando marcadores genéticos conocidos de los tipos de células que se espera que estén presentes en el tejido (enfoque supervisado) o en función de los 5 genes principales expresados diferencialmente entre los grupos (enfoque no supervisado).
- Utilice decontX21para determinar el grado de contaminación del ARN ambiental y para ajustar la matriz de expresión génica para el ARN ambiental.
- Incluya la matriz genética en bruto como antecedente.
- Guarde el objeto Seurat para explorarlos en el futuro.
NOTA: El código para el control de calidad y el análisis de agrupamiento está disponible en el archivo complementario 1.
| Paquetes de software/R utilizados en el flujo de trabajo de datos | Software/paquetes alternativos | Etapa de procesamiento |
| CellRanger (Guardabarros) | STARsolo, kallisto | Recorte, alineación, mapeo |
| Seurat | SingleCellExperiment, Cellranger | Control de calidad, análisis y exploración de datos |
| DoubletFinder | scds, scdblFinder, Scrublet | Detección de dobletes |
| DecontX | SoupX, CellBender | Ajuste del ARN ambiental |
Tabla 3: Software/herramientas para el flujo de trabajo de datos.
Access restricted. Please log in or start a trial to view this content.
Resultados
Este flujo de trabajo fue diseñado para guiar el procesamiento de muestras humanas IMAT congeladas para obtener perfiles de expresión génica con una resolución de un solo núcleo, lo que permite la identificación del tipo de célula. Aquí se presenta una muestra representativa de IMAT de un participante en el estudio SOMMA.
El primer paso de cualquier análisis de los datos de snRNA-seq es evaluar la calidad de los datos para identificar los núcleos de mala calidad, que potencialmente d...
Access restricted. Please log in or start a trial to view this content.
Discusión
Hay varios desafíos inherentes a trabajar con IMAT. Además de su limitada accesibilidad, la producción de material de muestra suele ser muy escasa y la "contaminación" del músculo esquelético es casi imposible de evitar. Para obtener una muestra de la mejor calidad, se debe penetrar en la fascia muscular al insertar la aguja de biopsia (para asegurarse de que no se recolecta tejido adiposo subcutáneo) y eliminar la mayor cantidad posible de tejido muscular mediante la disección de la muestra bajo un microscopio i...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer a Bryan Bergman, PhD de la Universidad de Colorado, por proporcionar la imagen de la biopsia IMAT en la Figura 1C del estudio MoTrIMAT (R01AG077956). Agradecemos que el Estudio de la Musculatría, la Movilidad y el Envejecimiento haya facilitado la muestra IMAT de la que se muestran los datos en el apartado de resultados representativos. El Instituto Nacional sobre el Envejecimiento (NIA, por sus siglas en inglés) financió el Estudio del Músculo, la Movilidad y el Envejecimiento (SOMMA; R01AG059416) y sus estudios auxiliares SOMMA AT (R01AG066474) y SOMMA Knee OA (R01AG070647). El apoyo a la infraestructura del estudio fue financiado en parte por los Centros Claude D. Pepper de la Independencia Americana Antigua del NIA en la Universidad de Pittsburgh (P30AG024827) y la Universidad de Wake Forest (P30AG021332) y los Institutos de Ciencias Clínicas y Traslacionales, financiados por el Centro Nacional para el Avance de la Ciencia Traslacional, en la Universidad de Wake Forest (UL1 0TR001420).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 µm corning syringe filters | Millipore Sigma | CLS431229 | |
| 1.7 mL DNA LoBind tubes | Eppendorf | 22431021 | low-bind tubes |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 100x protease inhibitor | Thermo Fisher Scientific | 78437 | |
| 10X Magnetic Separator | 10X Genomics | 230003 | |
| 10X Vortex Adapter | 10X Genomics | 330002 | |
| 15 mL canonical tubes | Sarstedt | 6,25,54,502 | |
| 2100 Bioanalyzer | Agilent | G2939BA | |
| 50 mL conical tubes | Sarstedt | 6,25,47,254 | |
| CellRanger | Genomics | N/A | |
| Chromium iX accesory kit | 10X Genomics | PN1000323 | |
| Chromium iX Controller | 10X Genomics | PN1000326 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | PN1000127 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1 | 10X Genomics | PN1000129 | |
| Chromium Next GEM Single Cell GEM Kit v3.1 | 10X Genomics | PN1000130 | |
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | AMQAX2000 | Automated cell counter |
| Countess cell counting chamber slides | Thermo Fisher Scientific | C10228 | |
| DoubletFinder | R | N/A | |
| DPBS (no calcium, no magnesium) | Thermo Fisher Scientific | 14190144 | |
| DTT | Thermo Fisher Scientific | R0861 | |
| Dual Index Kit TT Set A, 96 rxns | 10X Genomics | PN1000215 | |
| Dynabeads MyOne SILANE | 10X Genomics | PN2000048 | |
| Falcon 100 µm Cell strainer | Corning Life Science | 352360 | |
| Falcon 40 µm Cell strainer | Corning Life Science | 352340 | |
| Glycerin (glycerol), 50% (v/v) Aqueous Solution | Ricca Chemical Company | 3290-32 | |
| KCL | Thermo Fisher Scientific | AM9640G | |
| Library Construction Kit v3.1 | 10X Genomics | PN1000196 | |
| MACS SmartStrainers (30µm) | Miltenyi Biotec | 130-098-458 | |
| Mastercycler Nexus Gradient Thermal cycler | Eppendorf | 6331000017 | |
| MgCl2 | Ambion | AM9530G | |
| Mortar and pestel | Health care logistics | 14075 | |
| NucBlue Live Ready Probes Reagent | Thermo Fisher Scientific | R37605 | |
| Nuclease Free Water (not DEPC treated) | Thermo Fisher Scientific | AM9930 | |
| Probumin Bovine Serum Albumin Fatty Acid Free, Powder | Sigma-Aldrich | 820024 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Ribolock RNAse inhibitor | Thermo Fisher Scientific | EO0382 | |
| Seurat | R | N/A | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SUPERasin 20 U/µL | Thermo Fisher Scientific | AM2695 | |
| ThermoMixer C | Eppendorf | 5382000015 | |
| Tissue homogenizer | Glass-Col | 099C K54 | |
| Tris buffer pH 8.0 | Thermo Fisher Scientific | AM9855G | |
| Triton X-100 | Thermo Fisher Scientific | AC327372500 | |
| UltraPure 0.5M EDTA pH 8.0 | Gibco | 15575020 |
Referencias
- Goodpaster, B. H., Bergman, B. C., Brennan, A. M., Sparks, L. M. Intermuscular adipose tissue in metabolic disease. Nat Rev Endocrinol. 19 (5), 285-298 (2023).
- Sparks, L. M., Goodpaster, B. H., Bergman, B. C. The metabolic significance of intermuscular adipose tissue: Is IMAT a friend or a foe to metabolic health. Diabetes. 70 (11), 2457-2467 (2021).
- Gallagher, D., et al. Adipose tissue in muscle: A novel depot similar in size to visceral adipose tissue. Am J Clin Nutr. 81 (4), 903-910 (2005).
- Manini, T. M., et al. Reduced physical activity increases intermuscular adipose tissue in healthy young adults. Am J Clin Nutr. 85 (2), 377-384 (2007).
- Addison, O., Marcus, R. L., LaStayo, P. C., Ryan, A. S. Intermuscular fat: A review of the consequences and causes. Int J Endocrinol. 2014, 309570(2014).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Arch Intern Med. 165 (7), 777-783 (2005).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. Am J Clin Nutr. 71 (4), 885-892 (2000).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men. Diabetes Care. 26 (2), 372-379 (2003).
- Sachs, S., et al. Intermuscular adipose tissue directly modulates skeletal muscle insulin sensitivity in humans. Am J Physiol Endocrinol Metab. 316 (5), E866-E879 (2019).
- Ford, H., Liu, Q., Fu, X., Strieder-Barboza, C. White adipose tissue heterogeneity in the single-cell era: From mice and humans to cattle. Biology (Basel). 12 (10), 1289(2023).
- Wang, L., et al. Single-nucleus and bulk RNA sequencing reveal cellular and transcriptional mechanisms underlying lipid dynamics in high marbled pork. NPJ Sci Food. 7 (1), 23(2023).
- Li, J., et al. Identification of diverse cell populations in skeletal muscles and biomarkers for intramuscular fat of chicken by single-cell RNA sequencing. BMC Genomics. 21 (1), 752(2020).
- Lyu, P., Qi, Y., Tu, Z. J., Jiang, H. Single-cell RNA sequencing reveals heterogeneity of cultured bovine satellite cells. Front Genet. 12, 742077(2021).
- Fitzgerald, G., et al. MME+ fibro-adipogenic progenitors are the dominant adipogenic population during fatty infiltration in human skeletal muscle. Commun Biol. 6 (1), 111(2023).
- Cummings, S. R., et al. The study of muscle, mobility and aging (SOMMA): A unique cohort study about the cellular biology of aging and age-related loss of mobility. J Gerontol A Biol Sci Med Sci. 78 (11), 2083-2093 (2023).
- Whytock, K. L., et al. Isolation of nuclei from frozen human subcutaneous adipose tissue for full-length single-nuclei transcriptional profiling. STAR Protoc. 4 (1), 102054(2023).
- 10x Genomics. Chromium Single Cell 3' Reagent Kits User Guide (v3.1 Chemistry Dual Index), Document Number CG000315 RevE. , Available from: https://cdn.10xgenomics.com/image/upload/v1668017706/support-documents/CG000315_ChromiumNextGEMSingleCell3-_GeneExpression_v3.1_DualIndex__RevE.pdf (2022).
- Heumos, L., et al. Best practices for single-cell analysis across modalities. Nat Rev Genet. 24 (1), 550-572 (2023).
- Hao, Y., et al. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 42 (2), 293-304 (2023).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet detection in single-cell RNA sequencing data using artificial nearest neighbors. Cell Syst. 8 (4), 329-337 (2019).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (2), 57(2020).
- Common considerations for quality control filters for single cell RNA-seq data. 10X Genomics. , Available from: https://www.10xgenomics.com/analysis-guides/common-considerations-for-quality-control-filters-for-single-cell-rna-seq-data (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 15 (6), e8746(2019).
- Emont, M. P., et al. A single-cell atlas of human and mouse white adipose tissue. Nature. 603 (7903), 926-933 (2022).
- Hildreth, A. D., et al. Single-cell sequencing of human white adipose tissue identifies new cell states in health and obesity. Nat Immunol. 22 (5), 639-653 (2021).
- Whytock, K. L., et al. Single cell full-length transcriptome of human subcutaneous adipose tissue reveals unique and heterogeneous cell populations. iScience. 25 (8), 104772(2022).
- Probst, V., et al. Benchmarking full-length transcript single cell mRNA sequencing protocols. BMC Genomics. 23 (1), 860(2022).
- CG000148 Rev A Technical Note - Resolving cell types as a function of read depth and cell number. Technical note. 10X Genomics. , Available from: https://assets.ctfassets.net/an68im79xiti/6gDArDPBTOg4IIkYEO2Sis/803be2286bb a5ca67f353e6baf68d276/CG000148_10x_Technical _Note_Resolving_Cell_Types_as_Function_of_ Read_Depth_Cell_Number_RevA.pdf (2018).
- Gupta, A., et al. Characterization of transcript enrichment and detection bias in single-nucleus RNA-seq for mapping of distinct human adipocyte lineages. Genome Res. 32 (2), 242-257 (2022).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648(2018).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. J Am Soc Nephrol. 30 (1), 23-32 (2019).
- Kim, N., Kang, H., Jo, A., Yoo, S. -A., Lee, H. -O. Perspectives on single-nucleus RNA sequencing in different cell types and tissues. J Pathol Transl Med. 57 (1), 52-59 (2023).
- Avila Cobos, F., Alquicira-Hernandez, J., Powell, J. E., Mestdagh, P., De Preter, K. Benchmarking of cell type deconvolution pipelines for transcriptomics data. Nat Commun. 11 (1), 5650(2020).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados