A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בידוד גרעינים מרקמת שומן בין-שרירית אנושית וריצוף RNA חד-גרעיני במורד הזרם
In This Article
Summary
הביולוגיה של רקמת השומן הבין-שרירית (IMAT) לא נחקרה במידה רבה בשל הנגישות המוגבלת של רקמת האדם. כאן, אנו מציגים פרוטוקול מפורט לבידוד גרעינים והכנת ספרייה של IMAT אנושי קפוא לריצוף RNA של גרעינים בודדים כדי לזהות את ההרכב התאי של מחסן שומן ייחודי זה.
Abstract
רקמת שומן בין-שרירית (IMAT) היא מחסן שומן לא נחקר יחסית הממוקם בין סיבי שריר. תוכן IMAT עולה עם הגיל וה- BMI וקשור למחלות מטבוליות וניווניות שרירים; עם זאת, הבנה של התכונות הביולוגיות של IMAT ויחסי הגומלין שלו עם סיבי השריר שמסביב חסרה מאוד. בשנים האחרונות, ריצוף RNA חד-תאי וגרעיני סיפק לנו אטלסים ספציפיים לסוג התא של מספר רקמות אנושיות. עם זאת, ההרכב התאי של IMAT אנושי נותר במידה רבה לא נחקר בשל האתגרים המובנים של נגישותו מאיסוף ביופסיה בבני אדם. בנוסף לכמות המוגבלת של רקמות שנאספות, עיבוד IMAT אנושי מסובך בשל קרבתו לרקמת שריר השלד והפאשיה. האופי עמוס השומנים של האדיפוציטים הופך אותו לבלתי תואם לבידוד של תא יחיד. לפיכך, ריצוף RNA של גרעינים בודדים הוא אופטימלי להשגת שעתוק ממדי גבוה ברזולוציה של תא בודד ומספק את הפוטנציאל לחשוף את הביולוגיה של מחסן זה, כולל ההרכב התאי המדויק של IMAT. כאן, אנו מציגים פרוטוקול מפורט לבידוד גרעינים והכנת ספרייה של IMAT אנושי קפוא לריצוף RNA של גרעינים בודדים. פרוטוקול זה מאפשר יצירת פרופיל של אלפי גרעינים בגישה מבוססת טיפות, ובכך מספק את היכולת לזהות סוגי תאים נדירים ובעלי תפוצה נמוכה.
Introduction
רקמת שומן בין-שרירית (IMAT) היא מחסן שומן חוץ רחמי השוכן בין סיבי שרירוסביבם 1. כפי שתואר בפירוט בסקירה שנערכה לאחרונה על ידי Goodpaster et al., ניתן לזהות IMAT באמצעות טומוגרפיה ממוחשבת ברזולוציה גבוהה (CT) והדמיית תהודה מגנטית (MRI) (איור 1A,B) והוא נמצא סביב ובתוך סיבי שריר בכל הגוף1. כמות ה- IMAT משתנה מאוד בין אנשים ומושפעת מ- BMI, גיל, מין, גזע וישיבה 2,3,4. יתר על כן, תצהיר IMAT נראה בדרך כלל במצבים פתולוגיים הקשורים לניוון שרירים5, ומחקרים רבים תיעדו מסת IMAT מוגברת אצל אנשים עם השמנת יתר, סוכרת מסוג 2, תסמונת מטבולית ותנגודת לאינסולין 6,7,8,9. עם זאת, התכונות הסלולריות והביולוגיות של IMAT רק מתחילות להיחשף. הנגישות המוגבלת והשונות במיקומים ובתוכן של IMAT בכל הגוף אתגרו את איסוף הדגימות ממחסן השומןהייחודי הזה 2. יתר על כן, דגימות "מזוהמות" בקלות בשרירי השלד (SM) בעת האיסוף, מה שהופך את ההפרדה בין התרומה הביולוגית מהרקמות השונות לקשה לפענוח (איור 1C). לשם כך, ריצוף RNA של גרעינים בודדים (snRNA-seq), שזכה לתשומת לב רבה בעשור האחרון, משמש כמתודולוגיה אידיאלית המאפשרת הפרדה של דפוסי ביטוי גנים שמקורם ב-IMAT וב-SM ברזולוציה של תא יחיד. יתר על כן, בידוד גרעינים שימושי במיוחד עבור רקמת שומן בשל אדיפוציטים גדולים עמוסי שומנים, אשר בלתי אפשרי לנתק לתוך תרחיף תא יחיד מבלי לפגוע בשלמות התאים. לבסוף, טכנולוגיה זו טומנת בחובה פוטנציאל לגלות סמנים חדשים של אדיפוציטים ספציפיים ל- IMAT ולחשוף את ההרכב והנוכחות של אוכלוסיות תאי אב שונות, כמו גם לחקור את השונות של הרכב התא בתנאים פתולוגיים ונורמליים.
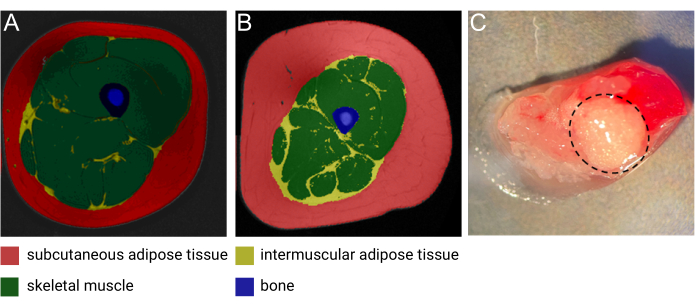
איור 1: תמונות של IMAT. תמונת תהודה מגנטית מייצגת (MRI) של IMAT מ (A) נקבה רזה בגיל העמידה ו (B) זכר בגיל העמידה עם השמנת יתר. אדום: רקמת שומן תת עורית, צהוב: רקמת שומן בין-שרירית, ירוק: שריר השלד, כחול: עצם. התמונה באדיבות הת'ר קורנל, מכון המחקר התרגומי AdventHealth. (C) דגימת רקמה טרייה עם IMAT (מוקפת בקו שחור מקווקו). התמונה באדיבות מייגן הוף, מכון המחקר התרגומי AdventHealth ובריאן ברגמן, אוניברסיטת קולורדו. נתון זה שונה באישור Goodpaster et al.1. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
מספר מחקרים פורסמו מתעשיית בעלי החיים וחקרו את גידול הבשר (IMAT בפרט) בחזירים, תרנגולות ובקר באמצעות תא יחיד (sc) ו-snRNA-seq10. מחקרים אלה זיהו מספר תת-אוכלוסיות של אדיפוציטים וסמנים של תאי אב פוטנציאליים של IMAT 11,12,13; עם זאת, לא ידוע אם הרכבים תאיים אלה מיתרגמים ל- IMAT אנושי. למיטב ידיעתנו, רק מחקר אחד בדק את ההטרוגניות התאית של שריר אנושי עם הסננה שומנית, המתקבלת מחולים גברים עם דלקת מפרקים ניוונית בירך, באמצעות snRNA-seq14. החוקרים דיווחו על אוכלוסיית אדיפוציטים קטנה ועל מספר תת-אוכלוסיות של אבות פיברו-אדיפוגניים (FAP) בתוך האוכלוסייה הגדולה של מיונוקליי14. המחקר שלנו הוא הראשון שפיתח שיטה לחקור ישירות IMAT שנותח ידנית משריר אנושי להרכב תאי באמצעות snRNA-seq.
חשוב לציין, פרוטוקולים עבור snRNA-seq צריכים להיות מותאמים אישית עבור הרקמה הספציפית שנחקרה, שכן כמות הרקמה הזמינה והתכונות הפיזיות של הרקמה הספציפית יכתיבו את שלבי העיבוד האופטימליים. תפוקת הרקמה עבור IMAT היא בדרך כלל קטנה, לעתים קרובות לא עולה על 50 מ"ג, גם בעת ביצוע ביופסיות מונחות אולטרסאונד. לפיכך, עיבוד נכון של רקמה נדירה זו הוא חיוני. אנו מאמינים כי פרוטוקול זה ישמש משאב רב ערך לחוקרים החוקרים IMAT אנושי.
Access restricted. Please log in or start a trial to view this content.
Protocol
הדגימה ששימשה לפרוטוקול זה הייתה חלק ממחקר השרירים, הניידות וההזדקנות (SOMMA)15, שאושר על ידי מועצת הביקורת המוסדית של Western IRB-Copernicus Group (WCG) ובוצע בהתאם להצהרת הלסינקי. המשתתפים סיפקו הסכמה מדעת בכתב להשתתפותם במחקר.
הערה: פרוטוקול זה מותאם מפרוטוקול קודם באמצעות 100 מ"ג של רקמת שומן תת עורית בטנית אנושית על פלטפורמה מבוססת ננו-באר16. הפרוטוקול הנוכחי מותאם ל-50 מ"ג של IMAT אנושי והכנת ספרייה באמצעות פלטפורמה מבוססת טיפות. ייתכן שיהיה צורך באופטימיזציה נוספת של פרוטוקול זה עבור בידוד גרעינים מ- IMAT לא אנושי או מחסני שומן אחרים.
1. הכנת מאגרים וריאגנטים (טבלה 1 וטבלה 2)
הערה: הכינו מאגרים טריים ביום הניסוי ואל תעשו בהם שימוש חוזר.
- מצננים מראש צנטריפוגה ל-4°C.
- הכנת חוצץ הומוגניזציה ומדיום בידוד גרעינים.
- להשיג שני דליים של קרח מראש מגניב 2 x 15 מ"ל צינורות חרוטיים.
- ערבבו את כל הריאגנטים עבור מאגר ההומוגניזציה (HB) בצינור חרוטי של 15 מ"ל בסדר המפורט בטבלה 1. שמור על קרח. מערבבים על ידי מערבולת.
- ערבב את כל הריאגנטים עבור תווך בידוד גרעינים (NIM) בצינור חרוטי של 15 מ"ל ברשימה המסודרת בטבלה 2. שמור על קרח. מערבבים על ידי מערבולת.
- הכינו 10% Triton-X על ידי הוספת 100 μL של Triton X-100 עד 900 μL של מים נטולי Nuclease. מערבולת כדי להבטיח ערבוב נכון. יש לשמור בטמפרטורת החדר (RT).
| מגיב | נפח (μL) | ריכוז סופי (mM) | |
| 1x | פי 2 | ||
| 1 מטר MgCl2 | 10 | 20 | 5 |
| 1 מטר Tris חוצץ, pH 8.0 | 20 | 40 | 10 |
| 2 מטר KCl | 25 | 50 | 25 |
| 1.5 מטר סוכרוז ( -4oC) | 334 | 668 | 250 |
| 1mM DTT | 2 | 4 | 0.001 (~ 1 מיקרומטר) |
| מעכב פרוטאז פי 100 | 20 | 40 | 1x |
| סופראסין 20 U/μL | 40 | 80 | 0.4 U/μL |
| מים ללא Nuclease | 1549 | 3098 | - |
| נפח כולל | 2000 | 4000 | - |
טבלה 1: מאגר הומוגניזציה (HB). שמור על קרח. מערבבים על ידי מערבולת.
| מגיב | נפח (μL) | ריכוז סופי (mM) | |
| 1x | פי 2 | ||
| EDTA | 0.4 | 0.8 | 0.1 |
| מעכב RNAse ריבולוק (40U/μL) | 40 | 80 | 0.8 U/μL |
| 1% BSA-PBS (-/-) | 1959.6 | 3919.2 | - |
| נפח כולל | 2000 | 4000 | - |
טבלה 2: מדיום בידוד גרעינים (NIM). שמור על קרח. מערבבים על ידי מערבולת.
2. כתישה של רקמה קפואה (איור 2A)
- הגדר את תחנת העבודה להומוגניזציה.
- מלאו את המכל בחנקן נוזלי (LN2).
אזהרה: בעת עבודה עם LN2, יש ללבוש תמיד משקפי מגן וכפפות קריו. - השג 2x מרגמות, 1x pestle, 1x micro-scoop spatula, 1x זכוכית לקפוץ, 1x נירוסטה pestle עבור סדרן אוטומטי.
- הגדר את המשדר האוטומטי.
- ממלאים בקרח ומקררים מראש את הזכוכית.
- מלאו את המכל בחנקן נוזלי (LN2).
- ממלאים את 2 המרגמות (המכילות את הפשט והמרית) ב-LN2 כדי לקרר את המכשירים. תנו ל-LN2 להתאדות ולחזור על עצמו.
- בזמן שהמכשירים מתקררים, הוסיפו 1 מ"ל HB לקפיצת הזכוכית.
- מלא את שתי המרגמות ב- LN2 בפעם האחרונה ושפך את דגימת ה- IMAT של 50 מ"ג לאחת המרגמות.
- כתש את ה- IMAT באמצעות המזיק על ידי לחיצה עדינה עליו על פיסת הרקמה כדי לשבור אותו לחתיכות קטנות. ודא שכל החתיכות כתושות.
- LN2 יתאדה לאט תוך כדי כתישת הרקמה. כאשר הרקמה נמעכת כראוי, ועדיין נותר 1/4 - 1/2 של טיט של LN2 , הטה את הטיט לכיוון שפת הטיט כדי לאסוף את הרקמה המרוסקת על ידי השפה. תנו ל-LN2 להתאדות לחלוטין.
- מיד לאחר שה-LN2 האחרון התאדה, גרפו את הרקמה המרוסקת לתוך קופצת הזכוכית המכילה 1 מ"ל HB.
3. הומוגניזציה של רקמה כתושה
- הומוגניזציה של הרקמה המרוסקת באמצעות המשדר האוטומטי. הביאו את הזכוכית למעלה ולמטה את משטח הנירוסטה במשך 10 פעימות בכיוון קדימה, ולאחר מכן 10 פעימות בכיוון ההפוך.
- ודא כי הפתרון הוא עכור לאחר הומוגניזציה ואינו מכיל חתיכות גלויות של רקמה. צבע ורוד בהיר צפוי לעתים קרובות עקב זיהום ברקמת שריר.
- מעבירים את ההומוגנט לצינור מקורר מראש בנפח 1.7 מ"ל על קרח.
- השתמש 400 μL של HB כדי לשטוף את הנקישה כדי לוודא שכל החומר מועבר ולהוסיף אותו לצינור.
הערה: ניתן לעבד שתי דוגמאות בכל פעם. לשם כך, הכפילו את כמות HB ו-NIM. כתשו והומוגניזציה של דגימת רקמה אחת ומיד לאחר מכן כתשו והומוגניזציה של דגימת הרקמה השנייה כדי שתוכלו לבצע את שלבי הבידוד והניקוי במקביל.
4. בידוד וניקוי גרעינים (איור 2B)
- הוסף 14 μL של Triton-X (10%) להומוגנאט לקבלת ריכוז של 0.1%.
- שמור את הצינור על קרח ובחושך במשך 10-15 דקות תוך ערבול כל 3 דקות.
- טרום רטוב אחד 100 מיקרומטר ואחד 40 מיקרומטר מסננת תאים (לכל דגימה) עם 100 μL של RT DPBS עבור כל אחד בצינור חרוטי 50 מ"ל.
- סנן את ההומוגנט דרך מסננת התאים של 100 מיקרומטר.
- שטפו את צינור ה-1.7 מ"ל ב-400 מיקרוליטר HB וסננו דרך מסננת התאים בגודל 100 מיקרומטר.
- לאחר מכן, סנן את התמיסה דרך מסננת התאים 40 מיקרומטר.
- העבר כמות שווה של תמיסה לשני צינורות מקוררים מראש של 1.7 מ"ל בעלי קישור נמוך המתאימים ל~900 μL בכל צינור.
- צנטריפוגה את הצינורות במשך 10 דקות ב 2700 x גרם ב 4 ° C. צריך להיות גלולה קטנה גלויה לאחר צנטריפוגה.
- הסר והשלך את שכבת השומנים העליונה ואת הסופרנטנט שנותר, והשאר ~ 50 μL פתרון מן הצינור הראשון.
- חזור על הפעולה עבור הצינור השני.
- השהה מחדש ביסודיות את הגלולה בצינור הראשון על ידי פיפטציה עדינה מעלה ומטה פי 20 והעבר לצינור חדש בעל קשירה נמוכה של 1.7 מ"ל. הימנעו מיצירת בועות.
- חזור על הליך זה עבור הצינור השני והעבר את התמיסה המושעה לאותו צינור.
- מוסיפים 500 μL של NIM ומערבבים עם pipetting.
- צנטריפוגה את הצינור עם איזון ב 1000 x גרם במשך 10 דקות ב 4 ° C.
- הסר supernatant, משאיר ~ 50 μL, ואת פיפטה בעדינות למעלה ולמטה עד גלולה הוא resuspended. לחלופין, העבירו את הגלולה המרחפת לצינור נקי חדש אם נותרו שאריות שומנים בצד הצינור.
- מוסיפים 200 μL של NIM ומערבבים על ידי pipetting.
5. צביעה וספירה של גרעינים (איור 2C ואיור 3)
הערה: כדי להקל על הספירה, הגדר פרוטוקול 'ספירת גרעינים' על מונה תאים אוטומטי, מכיוון שהתאמת השדה הבהיר וערוצי DAPI יכולה להשפיע מאוד על הספירה. התאימו את התעלות כך שיילכדו רק גרעינים ולא פסולת. ודא שערוץ השדה הבהיר מסמן רק 'אובייקטים' שיש להם גם כתם DAPI.
- הוסיפו טיפה אחת של תמיסת צביעת התאים החיים והשאירו אותה בחושך, על קרח, למשך 15 דקות.
- סנן את התמיסה דרך מסננת תאים של 30 מיקרומטר.
- מערבבים את תמיסת הגרעינים על ידי פיפטינג ומוסיפים 10 μL של התמיסה לשקופית תא ספירת תאים.
- ספירת גרעינים באמצעות מונה תאים אוטומטי.
הערה: הריכוז האופטימלי הוא 1000 גרעינים/μL המתאימים ל-1.0 x 106/mL.- ודאו שאין גושים של גרעינים, מאחר שזה עלול לסתום את השבב ליצירת טיפות גרעינים בודדות (איור 3).
- אם ריכוז הגרעינים אינו גבוה מספיק, סובבו את התמיסה ב 1000 x גרם במשך 10 דקות ב 4 °C כדי לקבל כדור, להסיר את supernatant, ו resuspend בנפח קטן יותר.
- אם דרגת הפסולת בתמיסה גבוהה, השהה מחדש את תמיסת הגרעינים בנפח גדול יותר של NIM (כלומר, 1 מ"ל) וסנן שוב דרך מסננת תאים של 30 מיקרומטר. לאחר מכן סובב למטה ב 1000 x גרם במשך 10 דקות ב 4 ° C והשהה מחדש בנפח המתאים ביחס לריכוז הגרעינים.
- לאחר קבלת ריכוז הגרעינים, המשיכו ישירות לשלב הראשון בהכנת הספרייה.
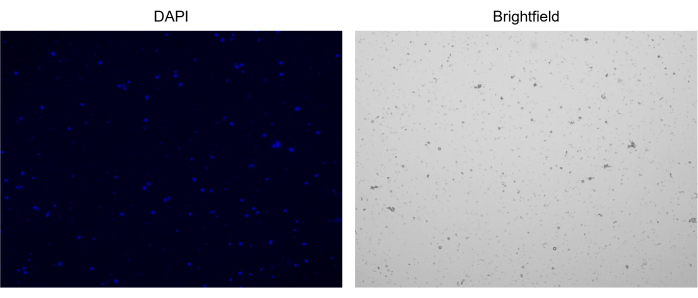
איור 3: צביעה של גרעינים מבודדים. תמונה ממונה תאים של גרעינים המוכתמים ב- NucBlue/DAPI (תמונה שמאלית) ובתמונת השדה הבהיר המתאימה (תמונה מימין). נוכחותם של כמויות קטנות של פסולת ניכרת בתמונת השדה הבהיר. למונה התאים האוטומטי המשמש כאן אין אפשרות לכלול פסי קנה מידה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
6. פרמטרים של הכנת הספרייה וריצוף
- עיין בפרוטוקול יסודי להכנת הספרייה באמצעות גישת גרעינים בודדים מבוססי טיפות הזמין בדף האינטרנט של הספק17.
- יש לשאוף להתאוששות גרעינים ממוקדת של 10,000. עם זאת, עבור דגימות עם רמה גבוהה של פסולת או גרעינים שבירים, צפוי מספר נמוך יותר של גרעינים שנמצאו.
- אחסנו את הדגימות בטמפרטורה של 4°C למשך עד 72 שעות לאחר שלב 2.3. בפרוטוקול הכנת הספרייה לשלב עיבוד של דגימות נוספות במקביל. בצע זאת על ידי עיבוד שתי דגימות עד שלב 2.3 בשני ימים רצופים, וביום השלישי, עבד את 4 הדגימות יחד משלב 3 ואילך בפרוטוקול הכנת הספרייה.
- פרמטרי ריצוף: רצף על פלטפורמת רצף המכוון ל-50,000 קריאות זוגיות לכל גרעינים.
הערה: הנתונים המוצגים בפרוטוקול זה רוצפו על פלטפורמת NovaSeq 6000, במטרה להגיע ל-50,000 קריאות זוגיות לכל גרעינים.
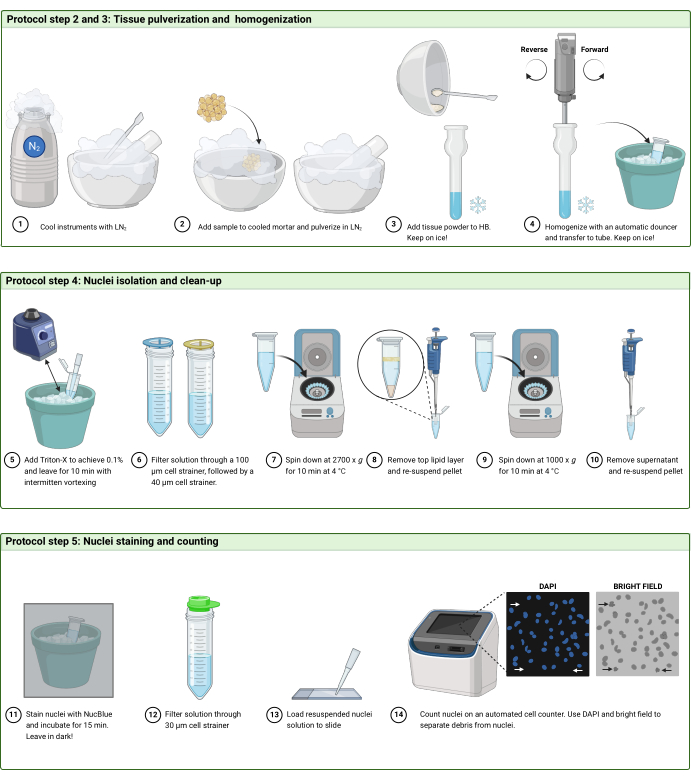
איור 2: זרימת עבודה של פרוטוקול. המחשה סכמטית של זרימת העבודה בשלבים (A) 2 ו- 3, (B) שלב 4 ו- (C) שלב 5 של הפרוטוקול. הדמות נוצרה עם BioRender.com. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
7. עיבוד וניתוח נתונים
הערה: בפרוטוקול זה, חלק מחבילות התוכנה וה-R המומלצות המשמשות לעיבוד נתוני הרצף המתקבלים מוצגות בקצרה, תוך התמקדות בשלבים שלאחר העיבוד המקדים הראשוני (טבלה 3). מחקר זה מספק מדדי בקרת איכות כלליים (QC) ודוגמה לקירוב והיטל סעפת אחידים (UMAP) באיור 4. עם זאת, תיאור מעמיק של הניתוח הביואינפורמטי אינו במסגרת פרוטוקול זה. לכן, הקוראים יכולים לעיין בסקירה האחרונה על שיטות עבודה מומלצות לניתוח תא יחיד על ידי Heumos et al.18.
- עיבוד מראש של נתוני רצף
- מפה את הגרעינים הבודדים לגנום הייחוס האנושי GRCh38.
- כלול קריאות אינטרון בספירה.
- בצע QC וסינון הנתונים באמצעות חבילת Seurat R 19.
- חשב ציון סיבוכיות תא על-ידי חלוקת log(10) מספר הגנים שזוהו על ידי log(10) מספר הקריאות שזוהו.
- שרטט את מדדי QC החשובים ביותר באמצעות היסטוגרמה או תרשים כינור, כולל מספר הגנים שזוהו לכל גרעינים, אחוז קריאה מיטוכונדריאלי וציון מורכבות התא.
- סנן גרעינים עם פחות מ-200 או יותר מ-10,000 גנים לכל גרעינים, יותר מ-10% קריאות מיטוכונדריאליות וציון מורכבות נמוך מ-0.8.
- נרמל נתונים ובצע הפחתת ממדיות.
- השתמש בפונקציה SCTransform מ - Seurat כדי לנרמל את הנתונים באמצעות תכונות משתנות 2000.
- אשכול נתונים באמצעות הפונקציות הבאות מחבילת Seurat R : RunPCA, FindNeighbors, FindClusters ו - RunUMAP.
- התווה UMAP כדי להציג באופן חזותי את קיבוץ הנתונים באשכולות.
- סנן כפילויות חזויות באמצעות חבילת DoubletFinder R 20 וקבץ מחדש נתונים.
- ביאור אשכולות באמצעות סמנים גנטיים ידועים של סוגי התאים הצפויים להימצא ברקמה (גישה מפוקחת) או בהתבסס על 5 הגנים המובילים המבוטאים באופן דיפרנציאלי בין הצבירים (גישה ללא פיקוח).
- השתמש decontX21כדי לקבוע את מידת זיהום RNA הסביבה ולהתאים את מטריצת ביטוי הגנים עבור RNA הסביבה.
- כלול את מטריצת הגן הגולמי כרקע.
- שמור את אובייקט Seurat לחקירה עתידית של הנתונים.
הערה: הקוד עבור ניתוח QC וקיבוץ באשכולות זמין בקובץ משלים 1.
| חבילות תוכנה/R המשמשות בזרימת עבודה של נתונים | תוכנה/חבילות חלופיות | שלב העיבוד |
| סל-ריינג'ר | STARsolo , קליסטו | חיתוך, יישור, מיפוי |
| סרה | SingleCellExperiment, Cellranger | QC, ניתוח וחקר נתונים |
| DoubletFinder | scds, scdblFinder, Scrublet | זיהוי כפול |
| DecontX | SoupX, CellBender | כוונון RNA סביבתי |
טבלה 3: תוכנות/כלים לזרימת עבודה של נתונים.
Access restricted. Please log in or start a trial to view this content.
תוצאות
זרימת עבודה זו תוכננה להנחות את העיבוד של דגימות IMAT אנושיות קפואות כדי להשיג פרופילי ביטוי גנים ברזולוציית גרעינים יחידים, המאפשרים זיהוי סוג תא. כאן מוצג מדגם IMAT מייצג אחד ממשתתף במחקר SOMMA.
הצעד הראשון של כל ניתוח של נתוני snRNA-seq הוא להעריך את איכות הנתונים כדי לזהות גרעינים ...
Access restricted. Please log in or start a trial to view this content.
Discussion
ישנם מספר אתגרים מובנים בעבודה עם IMAT. בנוסף לנגישותו המוגבלת, התפוקה של חומר הדגימה היא לעתים קרובות נדירה מאוד, וכמעט בלתי אפשרי להימנע מ"זיהום" של שרירי השלד. כדי לקבל את הדגימה האיכותית ביותר, יש לחדור לשריר הפאשיה בעת החדרת מחט הביופסיה (כדי להקפיד שלא לאסוף רקמת שומן תת עורית) ולהסיר כמ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
המחברים רוצים להודות לבריאן ברגמן, PhD מאוניברסיטת קולורדו על שסיפק את התמונה של ביופסיית IMAT באיור 1C ממחקר MoTrIMAT (R01AG077956). אנו אסירי תודה על מחקר השרירים, הניידות וההזדקנות המספק את מדגם IMAT שממנו מוצגים הנתונים בסעיף התוצאות המייצגות. המכון הלאומי להזדקנות (NIA) מימן את המחקר של שרירים, ניידות והזדקנות (SOMMA; R01AG059416) והמחקרים הנלווים שלה SOMMA AT (R01AG066474) ו- SOMMA Knee OA (R01AG070647). התמיכה בתשתית המחקר מומנה בחלקה על ידי NIA Claude D. Pepper Older American Independence Centers באוניברסיטת פיטסבורג (P30AG024827) ואוניברסיטת ווייק פורסט (P30AG021332) והמכונים למדע קליני ותרגומי, שמומנו על ידי המרכז הלאומי לקידום מדע תרגומי, באוניברסיטת ווייק פורסט (UL1 0TR001420).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2 µm corning syringe filters | Millipore Sigma | CLS431229 | |
| 1.7 mL DNA LoBind tubes | Eppendorf | 22431021 | low-bind tubes |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 100x protease inhibitor | Thermo Fisher Scientific | 78437 | |
| 10X Magnetic Separator | 10X Genomics | 230003 | |
| 10X Vortex Adapter | 10X Genomics | 330002 | |
| 15 mL canonical tubes | Sarstedt | 6,25,54,502 | |
| 2100 Bioanalyzer | Agilent | G2939BA | |
| 50 mL conical tubes | Sarstedt | 6,25,47,254 | |
| CellRanger | Genomics | N/A | |
| Chromium iX accesory kit | 10X Genomics | PN1000323 | |
| Chromium iX Controller | 10X Genomics | PN1000326 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | PN1000127 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1 | 10X Genomics | PN1000129 | |
| Chromium Next GEM Single Cell GEM Kit v3.1 | 10X Genomics | PN1000130 | |
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | AMQAX2000 | Automated cell counter |
| Countess cell counting chamber slides | Thermo Fisher Scientific | C10228 | |
| DoubletFinder | R | N/A | |
| DPBS (no calcium, no magnesium) | Thermo Fisher Scientific | 14190144 | |
| DTT | Thermo Fisher Scientific | R0861 | |
| Dual Index Kit TT Set A, 96 rxns | 10X Genomics | PN1000215 | |
| Dynabeads MyOne SILANE | 10X Genomics | PN2000048 | |
| Falcon 100 µm Cell strainer | Corning Life Science | 352360 | |
| Falcon 40 µm Cell strainer | Corning Life Science | 352340 | |
| Glycerin (glycerol), 50% (v/v) Aqueous Solution | Ricca Chemical Company | 3290-32 | |
| KCL | Thermo Fisher Scientific | AM9640G | |
| Library Construction Kit v3.1 | 10X Genomics | PN1000196 | |
| MACS SmartStrainers (30µm) | Miltenyi Biotec | 130-098-458 | |
| Mastercycler Nexus Gradient Thermal cycler | Eppendorf | 6331000017 | |
| MgCl2 | Ambion | AM9530G | |
| Mortar and pestel | Health care logistics | 14075 | |
| NucBlue Live Ready Probes Reagent | Thermo Fisher Scientific | R37605 | |
| Nuclease Free Water (not DEPC treated) | Thermo Fisher Scientific | AM9930 | |
| Probumin Bovine Serum Albumin Fatty Acid Free, Powder | Sigma-Aldrich | 820024 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Ribolock RNAse inhibitor | Thermo Fisher Scientific | EO0382 | |
| Seurat | R | N/A | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SUPERasin 20 U/µL | Thermo Fisher Scientific | AM2695 | |
| ThermoMixer C | Eppendorf | 5382000015 | |
| Tissue homogenizer | Glass-Col | 099C K54 | |
| Tris buffer pH 8.0 | Thermo Fisher Scientific | AM9855G | |
| Triton X-100 | Thermo Fisher Scientific | AC327372500 | |
| UltraPure 0.5M EDTA pH 8.0 | Gibco | 15575020 |
References
- Goodpaster, B. H., Bergman, B. C., Brennan, A. M., Sparks, L. M. Intermuscular adipose tissue in metabolic disease. Nat Rev Endocrinol. 19 (5), 285-298 (2023).
- Sparks, L. M., Goodpaster, B. H., Bergman, B. C. The metabolic significance of intermuscular adipose tissue: Is IMAT a friend or a foe to metabolic health. Diabetes. 70 (11), 2457-2467 (2021).
- Gallagher, D., et al. Adipose tissue in muscle: A novel depot similar in size to visceral adipose tissue. Am J Clin Nutr. 81 (4), 903-910 (2005).
- Manini, T. M., et al. Reduced physical activity increases intermuscular adipose tissue in healthy young adults. Am J Clin Nutr. 85 (2), 377-384 (2007).
- Addison, O., Marcus, R. L., LaStayo, P. C., Ryan, A. S. Intermuscular fat: A review of the consequences and causes. Int J Endocrinol. 2014, 309570(2014).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Arch Intern Med. 165 (7), 777-783 (2005).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. Am J Clin Nutr. 71 (4), 885-892 (2000).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men. Diabetes Care. 26 (2), 372-379 (2003).
- Sachs, S., et al. Intermuscular adipose tissue directly modulates skeletal muscle insulin sensitivity in humans. Am J Physiol Endocrinol Metab. 316 (5), E866-E879 (2019).
- Ford, H., Liu, Q., Fu, X., Strieder-Barboza, C. White adipose tissue heterogeneity in the single-cell era: From mice and humans to cattle. Biology (Basel). 12 (10), 1289(2023).
- Wang, L., et al. Single-nucleus and bulk RNA sequencing reveal cellular and transcriptional mechanisms underlying lipid dynamics in high marbled pork. NPJ Sci Food. 7 (1), 23(2023).
- Li, J., et al. Identification of diverse cell populations in skeletal muscles and biomarkers for intramuscular fat of chicken by single-cell RNA sequencing. BMC Genomics. 21 (1), 752(2020).
- Lyu, P., Qi, Y., Tu, Z. J., Jiang, H. Single-cell RNA sequencing reveals heterogeneity of cultured bovine satellite cells. Front Genet. 12, 742077(2021).
- Fitzgerald, G., et al. MME+ fibro-adipogenic progenitors are the dominant adipogenic population during fatty infiltration in human skeletal muscle. Commun Biol. 6 (1), 111(2023).
- Cummings, S. R., et al. The study of muscle, mobility and aging (SOMMA): A unique cohort study about the cellular biology of aging and age-related loss of mobility. J Gerontol A Biol Sci Med Sci. 78 (11), 2083-2093 (2023).
- Whytock, K. L., et al. Isolation of nuclei from frozen human subcutaneous adipose tissue for full-length single-nuclei transcriptional profiling. STAR Protoc. 4 (1), 102054(2023).
- 10x Genomics. Chromium Single Cell 3' Reagent Kits User Guide (v3.1 Chemistry Dual Index), Document Number CG000315 RevE. , Available from: https://cdn.10xgenomics.com/image/upload/v1668017706/support-documents/CG000315_ChromiumNextGEMSingleCell3-_GeneExpression_v3.1_DualIndex__RevE.pdf (2022).
- Heumos, L., et al. Best practices for single-cell analysis across modalities. Nat Rev Genet. 24 (1), 550-572 (2023).
- Hao, Y., et al. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 42 (2), 293-304 (2023).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet detection in single-cell RNA sequencing data using artificial nearest neighbors. Cell Syst. 8 (4), 329-337 (2019).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (2), 57(2020).
- Common considerations for quality control filters for single cell RNA-seq data. 10X Genomics. , Available from: https://www.10xgenomics.com/analysis-guides/common-considerations-for-quality-control-filters-for-single-cell-rna-seq-data (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 15 (6), e8746(2019).
- Emont, M. P., et al. A single-cell atlas of human and mouse white adipose tissue. Nature. 603 (7903), 926-933 (2022).
- Hildreth, A. D., et al. Single-cell sequencing of human white adipose tissue identifies new cell states in health and obesity. Nat Immunol. 22 (5), 639-653 (2021).
- Whytock, K. L., et al. Single cell full-length transcriptome of human subcutaneous adipose tissue reveals unique and heterogeneous cell populations. iScience. 25 (8), 104772(2022).
- Probst, V., et al. Benchmarking full-length transcript single cell mRNA sequencing protocols. BMC Genomics. 23 (1), 860(2022).
- CG000148 Rev A Technical Note - Resolving cell types as a function of read depth and cell number. Technical note. 10X Genomics. , Available from: https://assets.ctfassets.net/an68im79xiti/6gDArDPBTOg4IIkYEO2Sis/803be2286bb a5ca67f353e6baf68d276/CG000148_10x_Technical _Note_Resolving_Cell_Types_as_Function_of_ Read_Depth_Cell_Number_RevA.pdf (2018).
- Gupta, A., et al. Characterization of transcript enrichment and detection bias in single-nucleus RNA-seq for mapping of distinct human adipocyte lineages. Genome Res. 32 (2), 242-257 (2022).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648(2018).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. J Am Soc Nephrol. 30 (1), 23-32 (2019).
- Kim, N., Kang, H., Jo, A., Yoo, S. -A., Lee, H. -O. Perspectives on single-nucleus RNA sequencing in different cell types and tissues. J Pathol Transl Med. 57 (1), 52-59 (2023).
- Avila Cobos, F., Alquicira-Hernandez, J., Powell, J. E., Mestdagh, P., De Preter, K. Benchmarking of cell type deconvolution pipelines for transcriptomics data. Nat Commun. 11 (1), 5650(2020).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved