Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mondial Analyse expression génique utilisant une plateforme biopuces oligonucléotidiques Zebrafish
Résumé
Microarrays Gene sont des outils puissants en matière de profilage d'expression génique au niveau du génome entier. Cette technologie a des applications dans une variété de disciplines biologiques, y compris la biologie du développement et de la toxicologie. Dans cette vidéo, nous détaillons un protocole d'analyse d'expression génique globale à l'aide d'une plateforme complète d'oligonucléotides biopuces pour le poisson zèbre.
Résumé
Gene la technologie des microréseaux permet la mesure quantitative et le profilage d'expression génique des niveaux de transcription sur une base du génome entier. Gene la technologie des puces est utilisée dans de nombreuses disciplines biologiques dans une variété d'applications incluant l'analyse d'expression génique globale par rapport au stade de développement, à un état de maladie, et dans les réponses toxiques. Ici, nous incluons une démonstration de l'analyse de l'expression des gènes en utilisant une échelle mondiale complète zebrafish spécifiques plateforme biopuces oligonucléotides. La plate-forme de poisson zèbre microréseaux d'expression contient 385 000 sondes, 60 paires de base de longueur, interrogeant 37 157 cibles avec jusqu'à 12 sondes par cible. Pour cette plateforme, toutes les informations d'ADNc et génomiques disponibles pour le poisson zèbre ont été recueillies auprès de divers y compris les bases de données génomiques Ensembl (http://www.ensembl.org), VEGA (http://vega.sanger.ac.uk), UCSC ( http://genome.ucsc.edu), et ZFIN (http://www.zfin.org). En conséquence, cette gamme offre une couverture complète d'expression du transcriptome zèbre actuel. Les microréseaux d'expression poisson zèbre a été imprimé par Roche NimbleGen (Madison, WI). Cette démonstration technique comprend le marquage fluorescent d'un produit d'ADNc, l'hybridation de l'ADNc produit étiqueté à la plateforme biopuces, et la numérisation de tableau pour l'acquisition du signal en utilisant la stratégie d'une couleur d'analyse.
Protocole
Partie 1: marquage fluorescent de l'ADNc
Le colorant Cy3 utilisés dans l'expérience microarray est sensible à la photo-dégradation. La procédure devrait avoir lieu en un minimum de lumière. Le protocole de marquage a été adapté à partir du système d'étiquetage CGH array BioPrime génomique manuelle 1.
- Les réactifs pour la réaction de marquage sont stockés à -20 ° C. De la CGH-array génomique BioPrime système d'étiquetage de dégel 2.5X Tampon amorce aléatoire, mélanger dCTP 10X, et une eau sans nucléase. Aussi le dégel le colorant Cy3-dCTP. Après décongélation, centrifuger brièvement tous les réactifs avant utilisation.
- Une réaction de marquage séparé devra être rempli pour chaque échantillon d'ADNc pour être analysés. Dans un 0,6 ml à paroi épaisse du tube ajouter 500 ng d'ADNc, 40 pl de tampon 2.5X amorce aléatoire et sans nucléase l'eau pour atteindre un volume total de 86 ul.
- Mélangez bien et centrifuger brièvement. Incuber de réaction pendant 5 minutes à 100 ° C sur un thermocycleur.
- Immédiatement réaction de transfert dans une boue bain de glace pendant 10 minutes.
- Pour chaque tube, puis ajouter 10 ul mélange de dCTP 10X, 2 pl 1 mM Cy3-dCTP et 2 polymérase ADN ul Exo Klenow. La polymérase Klenow Exo l'ADN doit être laissé dans le congélateur jusqu'à ce que nécessaire et doit toujours être gardé sur la glace.
- Mélangez bien et la réaction centrifuger brièvement.
- Incuber réaction à 37 ° C pendant 16 heures dans un thermocycleur.
Partie 2: Précipitation des ADNc marqué
- Avant de continuer dans le tour du protocole sur le système d'hybridation NimbleGen et permettent au système de s'équilibrer à 42 ° C.
- Pour chaque réaction, placer une colonne Microcon dans un tube de 1,5 ml. Ajouter 300 ul 0,1 X SSC à chaque colonne suivies par le contenu de la réaction de marquage (100 pi).
- Centrifuger le tube contenant la colonne à 16 100 xg pendant 10 minutes à 4 ° C.
- Jeter le débit à travers, ce qui peut être légèrement de couleur rose, et ajouter un autre 300 pi 0,1 X SSC à la colonne.
- Répétez centrifugation à 16100 xg pendant 10 minutes à 4 ° C.
- Jeter le débit à travers et d'ajouter un supplément de 300 pl 0,1 X SSC à la colonne.
- Centrifuger à 16100 xg pendant 12 minutes à 4 ° C.
- Jeter les traverser. Le débit à travers devrait être clair après ce dernier lavage et vous devriez être en mesure de voir l'ADNc roses marqués sur la colonne.
- Appliquer 100 ul eau sans nucléase à la colonne. Retirer la colonne du tube de 1,5 ml et inverser la colonne dans un nouveau tube de 1,5 ml.
- Centrifuger à 2300 xg pendant 2 minutes à 4 ° C. L'ADNc marqué doit être réhydraté dans l'eau qui s'accumule au fond du tube.
- Utilisez un NanoDrop ND-1000 spectrophotomètre conformément aux instructions du fabricant pour calculer la concentration d'ADN et d'évaluer l'incorporation de colorants en calculant le ratio de la base pour la teinture, où le nucléotide à la Cy3 ratio est égal à l'A 260 / A 550 multiplié par 23,15. La base de ratio de teinture doit être entre 50 et 80.
- Si ADNc s'ils sont étiquetés suffisamment, aliquot 6 ug d'ADNc marqué dans un nouveau tube de 1,5 ml.
- Mélanger un volume de 3 M 0,1 X NaOAc, pH 5,2 et 1x volume d'isopropanol. Laisser le mélange à incuber dans l'obscurité pendant 30 minutes à température ambiante.
- Suivant centrifuger les échantillons à 16100 xg pendant 20 minutes à température ambiante.
- Soigneusement jeter le surnageant sans perturber le culot d'ADN, qui devraient être de couleur rose.
- Effectuer un lavage d'éthanol par addition de 500 ul EtOH 70% et mélanger par inversion douce.
- Centrifuger l'échantillon à 16 100 xg pendant 5 minutes à température ambiante.
- Retirer le surnageant, inverser le tube, et de permettre le culot sécher pendant 5 minutes. À cette étape, le culot peut être stocké à -20 ° C si désiré ou le protocole peut être poursuivi à l'étape d'hybridation.
Partie 3: Hybridation
La procédure de l'hybridation est adapté du Guide de l'utilisateur Roche NimbleGen Arrays pour 2 Analyse expression génique.
- Dissoudre le culot dans 5 pi d'eau sans nucléase.
- Une fois la pastille est dissoute ajouter des composants croisements énumérés au Tableau 1. Composants d'hybridation sont dans le Kit de Roche NimbleGen hybridation. Après tous les composants sont ajoutés au volume total devrait maintenant total de 18 ul. Mélangez bien et faites tourner la brève tube pour recueillir le contenu vers le bas.
- L'étape suivante consiste à dénaturer l'ADNc marqué en le plaçant sur un bloc chauffant à 95 ° C pendant 5 minutes.
- Transférer immédiatement le tube à la système d'hybridation NimbleGen tandis que les tableaux sont préparés dans les étapes suivantes.
- Microarrays devraient toujours être manipulés avec soin et stockés conformément aux instructions du fabricant. Roche NimbleGen recommande de stocker les tableaux d'expression dans le noir avec déshydratant à température ambiante.
- Chargez le tableau dans l'outil d'alignement de précision Mixer (PMAT) fourni dans les accessoires de NimbleGen le traitement de tableau.
- Charger une table de mixage sur le X1 PMAT et retirer la bande adhésive avec une pince. Fermez la PMAT sécurisée. Appliquer une pression vers le mélangeur à travers le trou dans la PMAT et ouvrir lentement pour que le tableau et mélangeur se désengager de la PMAT.
- Fermement appliquer une pression avec le brayer autour de l'extérieur de la console afin d'assurer l'étanchéité au tableau est complet et d'enlever toutes les bulles. Placez le complexe matrice-mélangeur sur le système d'hybridation à se réchauffer.
- Avec une pipette de déplacement, ajouter lentement 15 pi de l'échantillon marqué dans l'orifice de remplissage. C'est le trou situé dans le centre de la table de mixage. Appliquer une pression lente et régulière à la pipette afin que l'échantillon se répand uniformément à travers le mélangeur sans bulles d'air. Essuyer tout excès d'échantillon par les trous de port et d'étanchéité des hublots avec des bandes adhésives.
- Fermez le loquet et commencer à mélanger de la solution d'hybridation en utilisant le programme B sur le système d'hybridation NimbleGen. Autoriser puces d'hybridation pour les 16-20 heures.
Partie 4: Post-hybridation lave et de balayage des biopuces
La procédure de lavage et de balayage est adapté du Guide de l'Roche NimbleGen tableaux de l'utilisateur pour l'analyse de l'expression des gènes 2.
- Une fois que l'hybridation est presque terminé de préparer les tampons de lavage comme indiqué au tableau 2. Préchauffer IA tampon de lavage à 42 ° C dans un bain d'eau.
- Allumez le scanner GenePix array 4000B et le scanner permettent de se réchauffer pendant environ 15 minutes avant utilisation.
- Verser l'IA préchauffé tampon de lavage dans un plat.
- Retirez le complexe array-mélangeur à partir du système d'hybridation et de NimbleGen glisser dans le gabarit (fourni dans les accessoires de NimbleGen le traitement de tableau). Assemblée submerger dans préchauffé IA tampon de lavage et retirez soigneusement mélangeur en retirant de la diapositive.
- Retirer tableau à partir de gabarit et agiter pendant 15 secondes dans le tampon de lavage IA.
- Transférez rapidement tableau dans un rack glisser dans la température du tampon IB salle de lavage. Agiter le rack glisser vigoureusement pendant 2 minutes.
- Transfert du rack glisser dans du Wash Buffer II et agiter pendant 1 minute.
- Transfert du rack glisser dans le tampon de lavage III et agiter pendant 15 secondes.
- Transférer le tableau dans la diapositive de séchage par centrifugation et de spin 2 minutes pour enlever toute humidité. Procéder à la numérisation.
- Il est recommandé de scanner le réseau immédiatement, car le colorant est sensible à la fois la lumière et l'ozone.
- Chargez le tableau, code à barres vers le bas, dans le scanner GenePix 4000B array.
- Ouvrez le logiciel GenePix Pro et de procéder à la numérisation de la matrice. Les tableaux Roche NimbleGen l'expression des gènes d'utiliser une stratégie d'une couleur d'hybridation. En conséquence une seule longueur d'onde est nécessaire pour la numérisation. Numérisation du signal de fluorescence Cy3 utilisant le laser à 532 nm. Ouvrez la boîte de dialogue de matériel et de mise en valeur de consigne PMT du laser à 532 nm pour une valeur de 500.
- Dans la boîte de dialogue du matériel réglage, régler la puissance à 100%, la taille des pixels à 5 microns, les lignes à moyenne à 1, la position et de se concentrer à 0 microns.
- Effectuez une numérisation de prévisualisation et de mettre la zone de numérisation.
- Effectue un balayage.
- Une fois la numérisation est terminée vérifiez l'histogramme de l'image. Idéalement 1e-5 compte normalisée devrait être à 65000 signifie que la limite de la saturation ~ 1-2% des fonctionnalités sont saturés. Si les comptages normalisés sont moins 1e-5, augmenter le gain du PMT et de réanalyser la diapositive. Si les comptages normalisés sont supérieurs à 1e-5, diminuer le gain du PMT et de réanalyser la diapositive.
- Une fois la numérisation est terminée enregistrer l'image. Il est recommandé de choisir une convention d'appellation standard pour les images pour en faciliter le renvoi. L'image peut désormais être chargé dans le programme d'analyse des données de préférence pour la normalisation des données et le calcul des valeurs d'intensité.
Les résultats représentatifs
Vous pouvez tout d'abord commencer à évaluer l'efficacité de la réaction de fluorescence Cy3 étiquetage de l'ADNc lorsque l'on commence l'lavages avec 0,1 X SSC. Lors de chaque lavage subséquent du flux grâce devrait devenir moins roses de couleur avec le dernier flux grâce clair. En outre, le Cy3 étiqueté produit d'ADNc doit être visible sur la colonne au cours de ces étapes de lavage. Si le débit à travers est rose vif dans la couleur ou il n'ya pas de produit de rose sur la colonne, c'est une bonne indication que la réaction de marquage n'a pas procédé correctement. La réaction de marquage peuvent être évalués quantitativement en utilisant le ND-1000 NanoDrop spectrophotomètre pour déterminer la concentration de l'ADN marqué et de la base au rapport de colorant (figure 1). La réaction de marquage des rendements régulièrement au moins 10 ug d'ADN marqué dans notre laboratoire. L'incorporation de colorants peuvent être calculées en utilisant une simple base / colorant où le ratio de nucléotide Cy3 ratio est égal à l'A 260 / A 550 multiplié par 23,15. Des valeurs allant de 50 à 80 indiquent suffisamment l'incorporation du colorant dans la réaction de marquage. Si la réaction de marquage donne une faible concentration de l'ADN ou l'incorporation de colorants pauvres, l'hybridation de l'échantillon sur le microroarray n'est pas recommandée et réaction de marquage doit être répété.
Évaluation de la qualité de l'hybridation peut être réalisée en évaluant l'histogramme d'image et l'image numérisée du microréseau (figure 2). Hybridations acceptable aura 1-2% des fonctionnalités saturés (ie, ce qui donne 1e-5 compte normalisé à la limite de saturation 65000) avec la valeur du PMT mis près de 500. Si la valeur du PMT doit être ajusté de façon drastique à partir de 500 pour atteindre 1e-5 compte normalisé à la limite de saturation 65000, cela indique généralement une hybridation pauvres de l'ADNc marqué au microarray. La qualité des données sera constituée si ces données se poursuit par une analyse ultérieure. La qualité de l'hybridation peut également être évaluée qualitativement en regardant les images du tableau numérisée (figure 3). Le kit de Roche NimbleGen hybridation contient un mélange d'oligo alignement de Cy3 et Cy5-oligonucléotides marqués 48mer qui s'hybrident à des fonctions d'alignement sur les tableaux. Évaluation qualitative de l'intensité de l'alignement des oligos d'autres caractéristiques sur le tableau permet un contrôle de qualité sur l'efficacité de l'hybridation de l'ADNc marqué par fluorescence sur le tableau. Suite à une analyse plus poussée, un nuage de points de vos données analysées peuvent être utilisées pour déterminer les niveaux relatifs d'expression des gènes et d'identifier les gènes qui sont différentiellement exprimés entre les échantillons (figure 4).
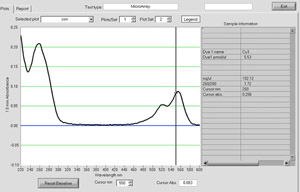
Figure 1. Un spectre NanoDrop à partir d'ADNc de rendement étiquetés avec Cy3. ADNc marqué et l'incorporation de colorants peuvent être évaluées en utilisant un ND-1000 NanoDrop spectrophotomètre.
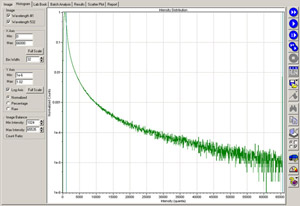
Figure 2. Un histogramme des puces GenePix numérisation. Qualité de l'hybridation peut être évaluée en évaluant l'histogramme de l'image. Hybridations acceptable aura 1-2% des fonctionnalités saturé de 1e-5 compte normalisé à la limite de saturation 65000 avec la valeur du PMT mis près de 500.

Figure 3. Une image scannée biopuces. Hybridation de qualité peut être évaluée par visualisation de l'image numérisée du microréseau. L'image doit être examiné pour la présence de poussière ou d'autres facteurs interférant interdisant la visualisation de chaque fonctionnalité et ainsi, empêcher le calcul précis de l'intensité de fluorescence de la fonction. L'intensité de la oligos alignement peut être comparé aux autres caractéristiques de la matrice pour évaluer l'efficacité d'hybridation.

Figure 4. Un diagramme de dispersion d'une expérience microarray analysés. Le but de cette procédure est d'identifier les gènes qui sont exprimés différentiellement dans vos échantillons. Données de biopuces peuvent encore être analysées et visualisées dans un nuage de points de visualiser les gènes qui sont différentiellement exprimés.
Tableau 1. Composants du kit Roche NimbleGen hybridation à ajouter pour 2 étape d'hybridation.
| Composante | Volume |
| Exemples (dissous dans l'eau) | 5 pl |
| Tampon d'hybridation 2X | 9 ul |
| Un Hyb | 3,6 ul |
| Oligo Alignement | 0,4 pi |
| Le volume total | 18 ul |
Tableau 2. Laver tampons pour être prêt pour le lavage des tableaux suivants hybridation.
| Composante | Tampon de lavage IA * | Tampon de lavage IB | Wash Buffer II | Tampon de lavage III |
| Eau | 225 ml | 225 ml | 225 ml | 225 ml |
| Tampon de lavage 10X I, II ou III | 25 ml | 25 ml | 25 ml | 25 ml |
| 1 M de DTT | 25 ul | 25 ul | 25 ul | 25 ul |
| Le volume total | 250 ml | 250 ml | 250 ml | 250 ml |
* Pré-chaude à 42 ° C
Discussion
Protocoles microarray Beaucoup, y compris celui détaillé ici, l'utilisation de colorants fluorescents comme un moyen de mesurer et de quantifier l'hybridation. Le colorant fluorescent Cy3 est sensible à la dégradation de la photo et l'ozone. Il est recommandé que la procédure soit menée dans un minimum d'éclairage. En outre, les petites particules de poussière peuvent interférer avec l'hybridation et la numérisation. Une attention particulière devrait être accordée à s'assurer que ...
matériels
| Name | Company | Catalog Number | Comments |
| Array Microcentrifuge Dryer | ISC Bioexpress | C-1303-T | |
| BioPrime Array CGH Genomic Labeling System | Invitrogen | 18095-011 | |
| Cyanine 3-dCTP | PerkinElmer, Inc. | NEL576 | |
| GenePix 4000B | Molecular Devices | GENEPIX 4000B | |
| Microcon YM-30 | EMD Millipore | 42411 | |
| NimbleGen Array Processing Accessories | Roche Group | 5223539001 | |
| NimbleGen Hybridization Kit | Roche Group | 5223474001 | |
| NimbleGen Hybridization System 4 | Roche Group | 5223652001 | |
| NimbleGen Wash Buffer Kit | Roche Group | 5223504001 | |
| X1 Mixers | Roche Group | 5223725001 |
Références
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon