Method Article
Aide d'un compteur de cellules automatisé pour simplifier les études d'expression génique: Knockdown siRNA de l'IL-4 dans l'expression des gènes dépendant cellules Namalwa
Dans cet article
Résumé
Cette procédure décrit un workflow rapide et facile à introduire dans les siARN difficiles à transfecter des lignées cellulaires et de suivre l'expression génique par PCR en temps réel. L'utilisation d'un compteur de cellules automatisé, plaque électroporation multi-puits et la station d'électrophorèse automatisé de fournir des résultats rapides et fiables, sans la nécessité pour la manipulation robotisée cher.
Résumé
L'utilisation d'inactivation génique siARN médiation continue à être un outil important dans les études d'expression génique. Des études sont menées siARN non seulement d'étudier les effets de la régulation négative des gènes uniques, mais aussi d'interroger les voies de signalisation et d'autres réseaux d'interactions complexes. Ces analyses voie exigent à la fois l'utilisation de modèles cellulaires pertinents et des méthodes qui causent moins de perturbations de la physiologie cellulaire. L'électroporation est de plus en plus utilisé comme un moyen efficace d'introduire siRNA et d'autres acides nucléiques dans les difficiles à transfecter des lignées cellulaires et cellules primaires, sans modifier la voie de signalisation de l'enquête. Il ya plusieurs étapes essentielles à une expérience réussie siARN, et il ya des façons de simplifier le travail tout en améliorant la qualité des données à plusieurs étapes expérimentales. Pour vous aider à démarrer votre projet de siRNA médiation inactivation génique, nous allons démontrer comment effectuer une étude complète de la voie collecte et le comptage des cellules avant l'électroporation par transfection après l'analyse PCR en temps réel l'expression des gènes. L'étude qui suit examine le rôle de l'activateur transcriptionnel STAT6 de l'IL-4 expression des gènes dépendant de CCL17 dans une ligne de lymphome de Burkitt (Namalwa). Les techniques démontrées sont utiles pour un large éventail de siARN basé sur des expériences sur les deux adhérentes et les cellules en suspension. Nous montrerons également comment rationaliser le comptage de cellules avec le compteur de cellules automatisé TC10, comment électroporation plusieurs échantillons simultanément en utilisant le système d'électroporation MXcell, et comment évaluer simultanément la qualité et la quantité d'ARN avec le système Experion électrophorèse automatisé.
Protocole
1. Préparation et comptage des cellules Namalwa
- Éliminer les cellules du flacon de culture et centrifuger les cellules à granules. Retirer les cellules surnageant et remettre dans un volume connu de PBS. Les cellules utilisées dans ce protocole sont les cellules en suspension. Si vous utilisez des cellules adhérentes, vous devrez trypsiniser avant de recueillir.
- Pour déterminer le nombre de cellules vivantes, aux taches une aliquote de la suspension cellulaire en mélangeant l'1:1 avec une solution de 0,4% de bleu trypan et pipeter 10 ul du mélange sur une lame de comptage, puis insérer la lame dans la cellule TC10 automatisé compteur (Bio-Rad Laboratories, Inc.)
- Le compteur de cellules TC10 sera autofocus pour assurer le résultat le plus précis, et puis procéder au comptage. Il détermine automatiquement la densité des cellules vivantes et cellules totales dans l'échantillon.
- Sélectionnez Afficher l'image pour afficher les cellules sur la viewscreen. Ensuite, sélectionnez le calculateur de dilution pour le compteur de cellules TC10 calculer automatiquement la quantité de mélange de cellules qui seront nécessaires.
- Cette expérience est d'utiliser un total de trois siRNA: un siRNA contrôle et deux siRNA différents ciblant le gène STAT6, plus les contrôles non transfectées. Cela nécessite 3,6 ml à une concentration finale de 5 x 10 6 cellules par ml.
- Entrez la concentration désirée et des valeurs volume final de dilution dans le calculateur, et il calcule le volume de suspension cellulaire nécessaire basée sur le comptage de cellules vivantes pour cet échantillon. Sinon, effectuer le calcul manuellement. Puis transférer le volume requis de mélange de cellules pour ces expériences dans un nouveau tube.
- Faites tourner les mélanges de cellules d'un culot cellulaire et de supprimer tous les CPE. Remettre en suspension dans 3,6 ml de tampon de Gene Pulser électroporation (Bio-Rad Laboratories). Transfert 800 aliquotes dans des tubes contenant les siRNA approprié et mélanger délicatement. Les cellules sont maintenant prêts pour l'électroporation.
2. Électroporation dans les cellules des plaques 96 puits
- La plaque de 96 puits électroporation permet les répétitions pour les quatre traitements à électroporées ensemble.
- Transfert 150 pi de la suspension de cellules dans les puits appropriés d'une plaque de 96 puits électroporation.
- Insérer la plaque dans la chambre de la plaque MXcell électroporation et fermer le couvercle.
- Électroporation en utilisant des paramètres optimisés pour la lignée cellulaire et le tampon utilisé. Ces cellules sont électroporées utilisant une vague de décroissance exponentielle avec réglages de l'instrument de 250 V, 350 uF, et la résistance 1000 Ω.
- Après électroporation est terminée, une pipette de haut en bas doucement dans chaque puits pour mélanger et ensuite transférer les cellules à une plaque de culture de tissus contenant préchauffé médias. Cellules Namalwa sont cultivées dans du RPMI (Life Technologies) supplémenté avec 7,5% de SVF et 1% de pyruvate.
- Les échantillons de contrôle qui n'ont pas été électroporées sont tirées de la suspension de cellules restantes et ajouté à des boîtes de culture de milieu de culture pré-chauffé à l'identique pour les cellules par électroporation.
- Incuber les cellules pendant la nuit à 37 ° C avec 5% de CO 2.
- Après 24 h, un ensemble de cellules a été induite avec 10 concentration finale ng / ml d'IL-4 recombinante (R & D Systems). La deuxième série de cellules de contrôle ne reçoivent que des supports supplémentaires. Toutes les cellules sont incubées une supplémentaire de 24 heures à 37 ° C et 5% de CO 2.
3. Extrait et vérifier l'ARN
- Après un total de 48 heures de croissance, de compter les cellules dans chaque puits, et le transfert une aliquote de chaque puits d'un tube de micro pour l'extraction de l'ARN, et de geler une seconde aliquote d'analyse western blot des protéines ou autres.
- Centrifuger les échantillons pour culotter les cellules, puis retirez et jetez le surnageant de chaque tube.
- Procéder à l'extraction d'ARN. Nous utilisons généralement l'Aurum ARN total kit (Bio-Rad) selon le protocole inclus.
- Lors de la surveillance inactivation génique médiée siARN il est particulièrement important de vérifier la qualité de l'ARN, car l'ARN dégradé va donner des résultats trompeurs. Le rapport n'indique pas 260/280 intégrité de l'ARN par conséquent, il est important d'exécuter les échantillons sur un gel ou d'un dispositif microfluidique pour confirmer l'ARN ne sont pas dégradés.
- Pour évaluer la qualité et la quantité en une seule étape, ces échantillons seront analysés en utilisant le système Experion électrophorèse automatisé (Bio-Rad).
- Tout d'abord, des échantillons de la chaleur. Puis, le premier, la charge, et le vortex de la puce Experion ARN. Insérer la puce dans la station d'électrophorèse Experion et commencer courir.
- Après la course est terminée, le logiciel affichera Experion entraîne automatiquement sous la forme d'un électrophérogramme, tableau des résultats (qui affiche la concentration d'ARN ribosomal rapport, et le nombre d'ARN indicateur de qualité), et un gel virtuel (qui ressemble à un traditionnel ARN gel). Haute qualité totale échantillons d'ARN ont en général une RQI de 8-10.
4. PCR en temps réel
- Après vérification de la qualité de l'ARN, procéder à des réactions d'ADNc. Cette expérience a utilisé le iScript une étape de RT-PCR mélange (Bio-Rad) pour combiner la synthèse d'ADNc et PCR en temps réel dans une réaction.
- Faites un mélange maître contenant tous les composants de la réaction autres que les matrices d'ARN pour chaque ensemble d'amorces, et distribuer ces dans la plaque PCR.
- Après dilution de l'ARN à une concentration uniforme, ajouter les échantillons d'ARN dans les puits appropriés. Chaque réaction est mis en place en trois exemplaires.
- Sceller la plaque, et placez-le dans la CFX384 système en temps réel de PCR (Bio-Rad). Sélectionnez le protocole approprié et commencer la course.
- Une fois la course terminée, l'expression des gènes est déterminé en comparant l'expression génique du gène d'intérêt normalisé à un gène de référence dans les cellules par électroporation avec le siRNA expérimentales pour la même normalisation des cellules électroporation avec un contrôle siARN.
5. Analyse des knockdown siARN
- L'expression de CCL17 a été contrôlée à l'aide quantitative PCR en temps réel pour enquêter sur le rôle de l'activateur transcriptionnel STAT6 de l'IL-4 par induction dépendante de CCL17. Pour cette expérience, GAPDH a été utilisé comme un gène de référence. Gènes de référence suppléant ou plusieurs gènes de référence peut être utilisé de la même manière.
- Le logiciel Gestionnaire CFX automatiquement normalisées CCL17 expression à l'expression du gène de référence à partir du même échantillon et automatiquement comparés aux valeurs normalisées résultant de contrôler les traitements en sélectionnant simplement la GAPDH comme «référence» et le traitement de contrôle (siRNA contrôle, sans induction de l'IL-4) comme «contrôle» dans l'expérience la fenêtre des paramètres du logiciel CFX gestionnaire.
- Sans l'IL-4 par induction, la transcription de CCL17 est resté à peu, les niveaux constitutifs comparable au traitement de contrôle.
- Cellules induites par l'IL-4 et en conservant une normale STAT6 voie (ie, le siRNA contrôle ou le contrôle des traitements non transfectées) a montré l'induction 80-10 pli de CCL17 expression.
- Les cellules induite par l'IL-4, mais avec STAT6 expression inhibée par l'introduction de siARN soit STAT6 montré une expression CCL17 seulement environ 2 fois plus élevé que les cellules non induits confirmant le rôle de STAT6 dans l'IL-4 expression induite de CCL17.
6. Les résultats représentatifs
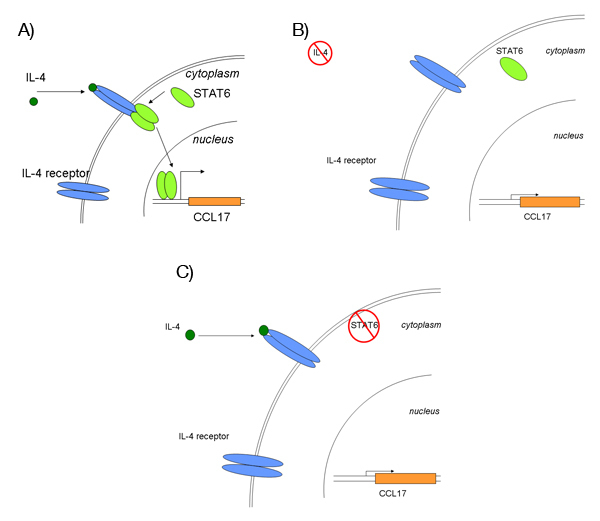
Figure 1. Aperçu de la voie de signalisation de l'IL-4.
Une reliure) de l'IL-4 à son récepteur entraîne la dimérisation et l'activation de STAT6. Activé STAT6 translocation vers le noyau et active la transcription de ses gènes cibles, tels que CC17.
B) Sans l'IL-4 par induction, la transcription de CCL17 restera à peu, les niveaux constitutifs.
C) Après siARN médiée taire du gène STAT6, le traitement par l'IL4 devrait conduire à augmenter peu ou pas de CCL17 expression.
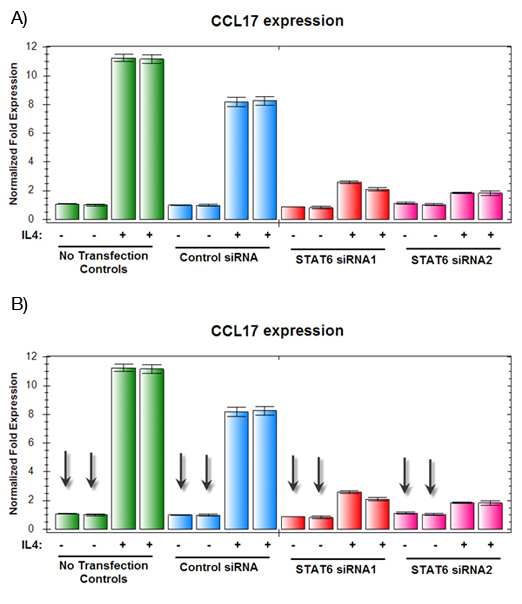
Figure 2. L'induction de CCL17 par IL4 mesurée par des conditions différentes par quantitative PCR en temps réel.
A) Les échantillons cultivés sans IL-4 avait un faible niveau d'expression constitutive indépendamment de l'expression génique.
B) Comme indiqué en vert et bleu, des échantillons de contrôle réagi normalement à un traitement par l'IL-4, avec une induction de 80 à 10 fois de CCL17. Cependant, les cellules transfectées avec des siRNA ciblant STAT6, en rouge et rose, avaient diminué CCL17 expression en réponse à l'IL-4, soutenant l'idée que STAT6 joue un rôle dans l'expression de l'IL-4 induit de CCL17.
Discussion
Cet article a montré comment électroporation dans un siRNA difficiles à transfecter une lignée cellulaire de suspension et la façon de suivre l'expression des gènes dans ces cellules. Lorsque vous effectuez cette procédure, il est important de se rappeler de garder les choses aussi cohérentes que possible dans tous les échantillons.
Déclarations de divulgation
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon