Method Article
Pinces magnétiques pour la mesure de la torsion et de couple
Dans cet article
Résumé
Pinces magnétiques, une technique de manipulation de molécule unique puissante, peuvent être adaptés pour les mesures directes de la torsion (en utilisant une configuration appelée librement orbite pinces magnétiques) et le couple (en utilisant une configuration appelée pinces de couple magnétique) dans les macromolécules biologiques. Lignes directrices pour l'exécution de ces mesures sont donnés, y compris les applications à l'étude de l'ADN et des filaments nucléo-protéines associées.
Résumé
Techniques molécule unique permettent d'enquêter sur le comportement de molécules biologiques individuelles en solution en temps réel. Ces techniques comprennent des approches dites de spectroscopie de force tels que microscopie à force atomique, pinces optiques, l'écoulement d'étirement et pinces magnétiques. Parmi ces approches, pinces magnétiques se sont distingués par leur capacité à appliquer un couple tout en maintenant une force d'étirement constant. Ici, il est illustré comment une telle configuration expérimentale des pinces magnétiques "classique" peut, par une modification simple de la configuration de son champ de réduire au minimum l'amplitude du champ transversal, être adapté à mesurer le degré de torsion dans une molécule biologique. La configuration qui en résulte est appelé les pinces magnétiques librement en orbite. En outre, il est démontré comment une autre modification de la configuration du champ peut produire un champ transverse avec une magnitude intermédiaire entre celle de l'& #8220; «pinces magnétiques classiques et les pinces magnétiques librement en orbite, ce qui permet de mesurer directement le couple stocké dans une molécule biologique. Cette configuration est appelée la pince à épiler de couple magnétiques. La vidéo d'accompagnement explique en détail comment la conversion de pinces magnétiques classiques en libre-orbite pinces magnétiques et des pinces de couple magnétique peut être accompli, et illustre l'utilisation de ces techniques. Ces adaptations maintiennent toutes les forces de pinces magnétiques classiques tout en élargissant considérablement la polyvalence de cet instrument puissant.
Introduction
Au cours des dernières années, les techniques de molécule unique ont prouvé leur large application dans l'étude des protéines motrices processive et d'autres enzymes, ce qui donne un aperçu de leur cinétique et la mécanochimie sous-jacent. Dans le contexte de la spectroscopie de force, d'importantes contributions ont été apportées par les flux de microscopie à force atomique étirements, et des pinces optiques et magnétiques. Pinces optiques et magnétiques (MT) ont notamment réussi à combiner une grande flexibilité en termes de manipulation moléculaire à haute résolution spatiale et temporelle. Ici, nous nous concentrons sur MT, qui peut appliquer les deux forces d'étirement et des couples de molécules biologiques attachés entre une surface et perles superparamagnétiques 1-3.
Pinces magnétiques (MT, Figure 1a) sont une technique unique de molécule très polyvalent qui a été utilisé pour surveiller à la fois les propriétés mécaniques des acides nucléiques, ainsi que leurs interactions avec les protéines. MT ont beaucoup de forces, notamment la simplicité et la robustesse globale de la mise en œuvre expérimentale, une application facile de couple, le fonctionnement normal et en mode calibration simple à force constante 4, parallèlement à l'extension des mesures 5, 6, et en l'absence de chauffage de l'échantillon et le photovieillissement. Comparé à d'autres approches de molécule unique, MT fournir un moyen d'effectuer des mesures de force à la dépendance des forces aussi faibles que ≈ 10 fN et avoir la capacité de contrôler carrément le degré de surenroulement. Alors que MT ont principalement été utilisé comme un outil expérimental pour étudier les processus biologiques impliquant des acides nucléiques 7, 8, ils ont également trouvé une application dans l'étude des propriétés mécaniques des protéines ou des cellules 9-13 10, 14-17. De nombreuses références utiles sont disponibles qui décrivent comment construire et gérer une MT 4, 18-20.
Cepener, MT conventionnelle ne pas suivre le mouvement de rotation directement, et, alors qu'ils s'appliquent couple, ils ne mesurent pas directement le couple. De plus, elles limitent la rotation libre de l'attache d'acide nucléique. Ici, nous présentons deux extensions de pinces magnétiques. Les premiers, librement orbite pinces magnétiques appelés (FOMT, figure 1b) 21, permet des mesures de fluctuations et les changements dans la torsion de molécules d'acide nucléique captifs angle équilibre, sans contraindre le mouvement de rotation autour de l'axe d'attache. Le second, appelé magnétiques pinces de couple (MTT, figure 1c), qui a la capacité d'appliquer et de mesurer directement les forces et les couples de biomolécules uniques 22-27.
Dans le protocole suivant, nous supposons que le lecteur a à sa / son caractère un instrument «classique» de MT. Nous renvoyons le lecteur à la discussion des références sur la façon de construire et gérer un MT mis en place, ainsi que considérations qui doivent être prises en compte dans la sélection des billes magnétiques, les aimants, et les routines de suivi. En outre, les articles 1 et 2 du Protocole texte décrivent comment nous préparons généralement et incuber un échantillon d'ADN pour utilisation dans la MT ainsi que les mesures préliminaires qui peuvent être exécutées sur un seul ADN dans le MT classique. Les articles 3 et 4 du Protocole texte illustrent comment un instrument MT peut être facilement adapté et utilisé pour les mesures FOMT et MTT.
Protocole
1. Préparation et incubation d'un échantillon d'ADN
- Préparer des constructions d'ADN qui sont ligaturés au duplex extrémités (≈ utiliser habituellement des fragments de 600 pb de l'ADN de PCR) qui sont fonctionnalisés avec de la biotine et digoxigénine groupes, respectivement 18 multiples. Pour commencer, une longueur d'ADN d'attache> 1 pm, par exemple, un kbp 7.9 correspondant à une longueur étirée de ~ 2,7 um comme utilisée ici, est recommandé pour la facilité d'utilisation; en particulier, en utilisant une longueur d'ADN qui est similaire à ou plus courte que le rayon de la bille est problématique en raison de la géométrie de la pièce jointe dans le MTT et FOMT. Voir la discussion pour une description de la façon dont la longueur d'ADN influe sur la réponse temporelle dans le domaine angulaire.
- Assembler les cellules d'écoulement pour les expériences d'une seule molécule. Pour les cellules d'écoulement, utilisez deux microscope en verre lamelles couvre séparés par un double-couche Parafilm entretoise. La lamelle supérieure microscope doit avoir deux trous pour le fluide en entrée et sortie de la cellule. Il est commode d'utiliser une sableuse pour percer les trous. La lamelle inférieure est revêtue avec de la nitrocellulose (0,1% poids / volume dans de l'acétate d'amyle). Placer les entretoises Parafilm sur le côté de nitrocellulose revêtu des lames de fond et fermer le haut avec les meilleurs lames propres.
- Sceller les cellules d'écoulement. En utilisant des pinces physiques, placer la cellule d'écoulement monté sur une plaque chauffante mis à 80-100 ° C pendant environ 1 min. Veillez à ce que la cellule d'écoulement est bien scellé, que le Parafilm ne ferme pas les trous qui se connectent à l'entrée et la sortie, et que les lames de verre sont bien alignés.
Remarque: Pour assurer une bonne étanchéité, il est recommandé de course sur bulles dans le Parafilm en utilisant un grand coton-tige. La cellule d'écoulement peut alors être montée sur l'instrument de pinces magnétiques. - Préparation des tampons. Préparer le tampon TE attache (10 mM Tris-HCl, pH 8,0, 1 mM d'acide éthylènediaminetétraacétique (EDTA) et 200 mM de NaCl). En variante, on peut utiliser un tampon PBS (137 mM NaCl, 2,7 mM KCl, 10 mM de tampon phosphate, pH 7,4) supplémenté avecith 100 pg / ml de BSA, 0,1% de Tween et 5 mM d'azoture de sodium (PBS +) en tant que tampon d'attache. Volumes 2-3 cellulaires Rincer TE tampon d'attache dans la cellule d'écoulement.
- Incuber 0,5 ou 1,5 des billes de latex non-magnétiques um rayon dans la cellule d'écoulement pendant ~ 30 min. Ces billes vont agir comme des perles de référence lors de pinces magnétiques mesures qui permettent de minimiser l'effet de la dérive entre l'objectif et le porte-échantillon (la cellule d'écoulement). Rincez billes non magnétiques seules en rinçant avec 2-3 volumes de cellules de tampon d'attache TE.
- Fonctionnaliser la surface du fond de la cuve à circulation par incubation avec 100 ug / ml d'anti-digoxigénine dans du PBS pendant au moins 1 heure (de préférence plus longue; incubation peut être réalisée pendant la nuit), pour assurer la fixation de l'ADN. Rincer avec 2-3 volumes de cellules de tampon d'attache TE. Enfin incuber la cellule d'écoulement avec 2 mg / ml d'albumine de sérum bovin (BSA) dans un tampon TE d'attache pendant 30 min pour la passivation de surface.
- Prélever2 ml de perles de MyOne superparamagnétiques revêtues de streptavidine (voir Discussion et table des matières) et diluer avec 10 ml de tampon d'attache TE. Laver deux fois avec 10 ml de tampon TE d'attache en utilisant un concentrateur de particules magnétiques, et remettre en suspension dans 10 ml de tampon TE d'attache. Attacher ~ 1 ml des molécules d'ADN (environ 1 ng) à ces billes par incubation dans du tampon TE d'attache pendant 30 min.
- Diluer la solution de l'ADN des billes superparamagnétiques-captif de dix fois en ajoutant 90 ml de tampon TE d'attache. Enfin, l'injection de la solution dans la cellule d'écoulement et incuber pendant ~ 1 heure pour permettre la fixation de l'ADN à la surface anti-digoxigénine-enduit. Laver la cellule d'écoulement à fond avec du tampon TE de l'attache. Après incubation des constructions d'ADN-longe, rincer abondamment avec le tampon expérimental (cela peut être un tampon d'attache TE) pour supprimer toutes les billes non-inscrits.
- Pour des mesures qui utilisent un protocole de repérage angulaire qui nécessite des billes de repère de calage fixés à des billes magnétiques 23 (voir Discussion), incuber la cellule d'écoulement avec 1000 fois dilué magasin de perles de marqueurs dans le tampon d'attache TE pendant au moins 30 minutes et rincer à fond avec le tampon.
2. Mesures sur une simple molécule d'ADN dans les pinces magnétiques classiques
- L'utilisation d'un MT classique (voir discussion) avec une configuration de champ approprié (figure 1a) et deux de translation et de contrôle de la position de l'aimant de rotation, la recherche de molécules d'ADN en rotation limitée dans la cellule d'écoulement. À des forces de traction de ≥ 1 pN (consulter les références 4, 19, 20, 28, 29 en ce qui concerne la force étalonnage pinces magnétiques), des perles captifs peuvent être facilement distingués des perles collées à la surface de la lame de fond par leurs différentes hauteurs dans la mise au point . Si une molécule d'ADN est contraint en rotation peut être évaluée par l'introduction de 20 à 30 son tour,s des aimants à une force de ≈ 0,25 PN: ici, la longueur de la sangle devrait diminuer de 0,4-0,5 um.
Remarque: Pour exécuter pinces magnétiques expériences, traitement de l'image est utilisé pour déterminer le x, y, et z la position de perles d'ADN-captif. logiciel Labview personnalisé à cet effet est disponible auprès des auteurs sur demande.- Vérifiez que le bourrelet est fixé par une attache unique d'ADN. Cela peut être fait en comparant le comportement sous tours positifs et négatifs de forces de> 1 pN (Figure 2a). Dans ce régime de force, la présence de multiples attaches d'ADN donne lieu à une diminution approximativement symétrique dans le prolongement à l'introduction de tours positifs et négatifs, tandis que les attaches d'ADN simple vont donner lieu à une réaction asymétrique.
- Quoi billes fixes appropriées collé à la surface inférieure au voisinage de l'élément d'attache d'intérêt qui peut servir de billes de référence.
- Calibrer la longueur de til ADN, l. La position de la surface de la cuve à circulation peut être déterminée en mettant le talon attaché en contact avec la surface (par exemple, en faisant tourner l'aimant de ~ 60 spires avec une force inférieure à 0,2 pN). Les mesures de la position verticale du cordon attaché à l'égard de cette surface ensuite un rapport sur la valeur absolue de l.
Remarque: Afin de minimiser les effets ultérieurs de dérive, il est conseillé d'effectuer des mesures de l par rapport à la position d'un bourrelet de référence fixé à la surface. - Enregistrer une courbe de rotation (par exemple une mesure de l'extension de l'ADN en tant que fonction du nombre de tours) à une force d'étirage de 0,25 ≈ pN (Figure 2a).
- Déterminer le nombre de tours au cours de laquelle l'extension est maximale, car cela correspond à l'état dans lequel la molécule d'ADN est en torsion détendue. Pour ce faire, il est utile pour s'adapter à la courbe de rotation localement avec un parabolique ou une fonction de Gauss pour déterminer le centre positisur. Définir ce point «zéro» à tour de rôle.
Remarque: Une routine écrite sur mesure à cet effet est disponible auprès des auteurs sur demande.
- Déterminer le nombre de tours au cours de laquelle l'extension est maximale, car cela correspond à l'état dans lequel la molécule d'ADN est en torsion détendue. Pour ce faire, il est utile pour s'adapter à la courbe de rotation localement avec un parabolique ou une fonction de Gauss pour déterminer le centre positisur. Définir ce point «zéro» à tour de rôle.
- Pour une série de ~ 20 positions d'aimant, déterminer l'extension moyenne de la molécule en torsion détendue (c'est à dire à «zéro» à tour de rôle, voir l'étape 2.4.1) de la z-trace.
- Pour chaque point dans l'étape 2.5 de mesure, de déterminer avec précision la force d'étirage à partir des fluctuations de l'x ou y la position 20, 28, 29, ou, à condition que l'aimantation de la bille est bien connue, en utilisant la connaissance du gradient local de champ 4. Tracer la force d'étirement par rapport aux résultats de vulgarisation moyenne dans une courbe force-extension (Figure 2b).
- Monter les données force-extension résultant de l'équation de la chaîne comme un ver utilisant l'approximation polynomiale par Bouchiat et al 30.
- Si la préparation de mesures FOMT suivantes, tourner lentement les aimants lors de l'enregistrement des excursions de la bille magnétique dans (x, y).
Remarque: Le plus petit rayon de l'anneau résultant de la configuration des MT classique, le plus près de la molécule d'ADN est attaché plus près du "pôle sud" de la bille magnétique. Quand on bascule sur la configuration FOMT, une telle molécule d'ADN sera attaché étroitement à la "équateur" de la bille magnétique, qui permet un suivi fiable de l'angle de rotation de la (x, y) position (voir Discussion).
3. Mesures d'ADN Twist aide des pinces magnétiques librement en orbite
- Remplacer manuellement les aimants carrés des pinces magnétiques classiques par un aimant cylindrique qui est utilisé pour FOMT (figure 1b). Cette opération doit être réalisée de telle manière que l'attache d'ADN choisie reste dans le champ de vision.
- Ceci peut être accompli en moins de 1 min, en dévissant simplement la tête magnétique complète qui contient les aimants de la configuration d'une pince à épiler classiques et en le remplaçant par une tête magnétique qui contient un aimant cylindrique pour FOMT.
- Les excursions en (x, y) d'une bille magnétique attaché par une seule attache ADNdb dépendent fortement de la position de l'attache par rapport à l'axe de l'aimant cylindrique (figure 1b, figure 3a). Enregistrer les excursions (x, y) afin de déterminer l'emplacement correspondant dans le motif caractéristique de fluctuation (Figure 3a, Discussion).
- Effectuez un alignement grossier de l'aimant dans le FOMT. Ceci peut être réalisé en déplaçant l'aimant cylindrique au-dessus de la cuve à circulation en utilisant (x, y) des platines de translation. Si les excursions (x, y) suivent un arc de cercle, l'aimant cylindrique n'est pas correctement alignée et doit être déplacéedans le sens approprié (figure 3b).
- Alignement grossier peut être effectué dans les 15 min pour le cas des billes MyOne avec 7,9 attaches kbp, et est complète lorsque la mesure des (x, y) excursions résultats dans l'observation du mouvement circulaire (Figure 3b, centre).
Remarque: alignement grossier est généralement suffisante pour observer les changements occasionnés par torsion dans la liaison aux protéines à ADN simple attachés dans la configuration FOMT 21, 31 (résultats représentatifs, figure 5), malgré le fait que l'histogramme bidimensionnel d'accompagnement peuvent ne pas avoir ses chiffres absolument uniformément répartis le long de l'anneau circulaire (figure 3c).
- Alignement grossier peut être effectué dans les 15 min pour le cas des billes MyOne avec 7,9 attaches kbp, et est complète lorsque la mesure des (x, y) excursions résultats dans l'observation du mouvement circulaire (Figure 3b, centre).
- Si nécessaire pour d'autres expériences, procéder à l'alignement amende FOMT. Ceci peut être réalisé à l'aide des vis micrométriques haute résolution ou une étape automatisée à haute résolution soit de déplacer l'aimant ou la cellule d'écoulement pour center l'aimant cylindrique sur le cordon à l'intérieur de ~ 10 um. Dans l'étape d'alignement fin, l'aimant est soigneusement positionné de telle sorte que les fluctuations sur l'anneau de cercle sont presque uniforme, ce qui correspond à une situation dans laquelle la barrière d'énergie à la pleine rotation du fait de l'aimant est k B T (Figure 4).
Remarque: Un script MATLAB pour tracer les fluctuations dans un histogramme ou thermogramme comme dans la figure 4 est disponible auprès des auteurs sur demande.
Remarque: alignement fine peut être réalisée dans 45 min pour le cas des billes MyOne avec 7,9 attaches kbp, et une réduction des délais pour les petites perles et les attaches courtes sont employées (voir Discussion).
Remarque: alignement fin est généralement nécessaire d'effectuer des mesures de la rigidité en torsion de l'ADN nu ou revêtu de protéine (résultats représentatifs, figure 4). - Si nécessaire pour l'analyse, de calibrer la force dans le FOMT. Ceci peut être réalisé ina manière analogue à la TA, en utilisant soit des fluctuations radiales de la perle 2> (où les supports angulaires indiquent la moyenne dans le temps), comme illustré dans la vidéo d'accompagnement et détaillée dans Lipfert et al 21, ou à condition que la magnétisation de la perle est bien connu, en utilisant les connaissances de la pente locale de la zone 21.
4. Mesures d'ADN Couple aide de la pince de couple magnétique
- Remplacer manuellement l'aimant cylindrique qui est utilisé pour FOMT par un aimant cylindrique, plus un côté aimant (permanent) pour le MTT (figure 1c). Cette opération doit être réalisée de telle manière que l'attache d'ADN choisie reste dans le champ de vision.
- La façon la plus simple d'y parvenir est d'ajouter manuellement l'aimant de côté à son emplacement approprié, qui peut être accompli en 1 min. Aucune autre réalignement est nécessaire.
Remarque: Une alternative à un aimant de côté est l'utilisationd'électro-aimants 32.
- La façon la plus simple d'y parvenir est d'ajouter manuellement l'aimant de côté à son emplacement approprié, qui peut être accompli en 1 min. Aucune autre réalignement est nécessaire.
- En cas de besoin aux fins d'analyse, de calibrer la force d'une manière analogue à la TA, en utilisant soit des x de la chenille ou les fluctuations y ou, à condition que l'aimantation de la bille est bien connue, en utilisant la connaissance de la pente locale du terrain 21.
- Suivre les fluctuations angulaires en fonction de θ de temps (t) en utilisant soit le repère basé sur le protocole de suivi 23 ou, comme le montre la vidéo qui l'accompagne, le protocole de suivi angulaire basée sur le contrôle de l'(x, y) position (voir Discussion). Dans le premier cas, l'enregistrement des images complètes de la bille en fonction du temps pour un traitement d'image ultérieur. Dans ce dernier cas, il suffit d'enregistrer (x, y) les fluctuations de la perle à cette étape.
Remarque: Un script MATLAB pour déterminer θ (t) à partir des images pleines de la perle en fonction du temps dans le protocole de suivi de repère sur la base est unvailable des auteurs sur demande.- Comme décrit dans la discussion, pour le protocole de suivi angulaire sur la base de la surveillance de l'(x, y) en position, il est également souhaitable d'enregistrer une trace de temps où les aimants sont lentement (typiquement à 0,1 Hz) mis en rotation de plusieurs tours. Cela permettra de convertir votre précision des coordonnées cartésiennes (x, y) en coordonnées polaires (r, θ) en utilisant les équations 3-5 de la discussion.
Remarque: Un script MATLAB pour le script de suivi angulaire basé sur le suivi des (x, y) position est disponible auprès des auteurs sur demande.
Remarque: Le temps de mesure dépend surtout de la résolution de couple désiré. Une argumentation détaillée est donnée dans Lipfert et al 24. Pour perles MyOne et 8 kbp attaches d'ADN, de mesure de 30-100 secondes devraient suffire à donner une résolution de couple dans la plage de 1 ~ pN · nm.
- Comme décrit dans la discussion, pour le protocole de suivi angulaire sur la base de la surveillance de l'(x, y) en position, il est également souhaitable d'enregistrer une trace de temps où les aimants sont lentement (typiquement à 0,1 Hz) mis en rotation de plusieurs tours. Cela permettra de convertir votre précision des coordonnées cartésiennes (x, y) en coordonnées polaires (r, θ) en utilisant les équations 3-5 de la discussion.
- Déterminer la rigidité en torsion de la trappe de the variance des fluctuations angulaires (σ θ 2, en radians) à l'aide:
k θ = k B T / σ θ 2 (1)
Remarque: Les raideurs des pièges typiques de rotation obtenus dans le MTT sont dans la gamme de 10-1000 pN · nm / rad, inférieur à celui des pinces magnétiques classiques. - En outre, enregistrer la position z de la bille et l'utiliser pour déterminer la longueur d'attache l (voir les étapes 2.4 à 2.7 aussi).
- Rotation N tours et enregistrer θ (t) et l (t) à nouveau.
Remarque: La rigidité de piège de rotation réduite du MTT par rapport à MT rend approprié pour les mesures de moment de rotation de la molécule unique, mais implique que le couple maximum qui peut être exercée soit réduite. Cela implique que le MTT peut ne pas être en mesure de contrebalancer les couples de glisser élevés causés par la rotation rapide. Il faut donc veiller à ne pas dépasser la vitesse maximale; ttourner abituellement à des taux proches de 0,1 Hz. - Déterminer le couple accumulé dans l'attache de l'acide nucléique après N tours à l'aide:
Γ = - k θ <θ N - θ 0> (2)
Où <...> désigne la moyenne et θ 0 et θ N sont l'angle à zéro tours (correspondant à une attache en torsion détendue, cf. L'étape 2.3 et N tours, respectivement. - Répétez les étapes 4.5 et 4.6 comme nécessaire afin de déterminer pleinement la réponse du couple d'une molécule en une seule opération de mesure (résultats représentatifs, figure 6).
Résultats
Les résultats représentatifs de la MT (Figure 1a) sont présentés dans la figure 2. Figure 2a montre les courbes de rotation extension pour un ADN de 7,9 kb prise à F = 0,25, 0,5, et 2,0 pN. La réponse d'un seul ADN de rotation doit être symétrique par rapport à des forces plus faibles (0,25 Pn), avec l'extension de l'ADN diminuant par suite de la formation de supercoils plectonemic positifs ou négatifs. Connaissance qualitative de cette réponse est utile lors de la recherche d'abord pour une attache de l'ADN en rotation limitée (étape 2.1). A noter que l'inspection de l'attache supplémentaire est nécessaire pour vérifier qu'il est constitué d'une molécule d'ADN simple: ici, la réaction asymétrique d'un seul ADN à une rotation à des forces supérieures à 0,5 pN permet de le distinguer de multiples ADN (étape 2.1.1). Une fois que cela a été vérifiée, on retourne à la réponse de rotation à 0,25 pN afin de déterminer le nombre exact d'aimant qui tourne à l'ADN simple is torsion détendue, où l'on prend une courbe force-extension, qui devrait ressembler à la Figure 2 b. Pour cette mesure particulière, un ajustement des données au modèle de chaîne comme un ver (trait plein) a donné un contour équipée longueur L C = 2,71 um et pliage longueur de persistance L P = 45 nm. Pour les ADN double brin, les valeurs ajustées de la longueur de persistance doivent se situent dans la plage de 40 à 55 nm, en fonction des conditions de tampon 33, et la longueur de contour équipée devraient être proches (typiquement moins de 10%) à la valeur attendue pour le produit d'assemblage d'ADN qui est utilisé dans les mesures, en utilisant la relation L = 0,34 nm ADN / bp · nombre de paires de bases.
La figure 3 montre les procédures et les résultats de l'alignement dans le FOMT (Figure 1b). Les excursions initiales (x, y) ont été enregistrés à l'étape 3.2 peuvent être comparées à la vue d'ensemble de la variation en fonction of la position de l'aimant transversal représenté sur la figure 3a, qui présente un motif «vortex» qui peut être utilisé pour guider un déplacement relatif ultérieur entre l'aimant et la bille d'ADN-captif tenue à la FOMT. Lorsque l'alignement grossier subséquente est terminée, (x, y) du bourrelet fluctuations-tracent une trajectoire circulaire, comme cela est également illustré par le tracé en noir sur la figure 3b. A ce point, le couple à partir des aimants autour de l'axe-z est réduite au point que les fluctuations thermiques suffisent pour faire tourner la bille autour de son point d'attache. Le rayon R cercle de l'anneau circulaire résultant (cercle équipée est affichée en rouge) représente la distance radiale entre le point de fixation de l'ADN et du centre de la bille (figure 1b). Comme le montre la figure 3c, cependant, un histogramme des données de la figure 3b montre que l'alignement grossier ne garantit pas une couverture uniformede toutes les positions possibles le long de l'anneau circulaire. Même si les fluctuations thermiques sont suffisantes pour explorer tout l'angle de rotation sur le cercle, il reste une barrière d'énergie faible (de l'ordre de l'énergie thermique k B T) à rotation libre.
Quand l'alignement de la plus fine est réalisée dans le FOMT (étape 3.4), l'instrument peut être utilisé pour déterminer le module de torsion de l'ADN (figure 4). Tout d'abord, bien alignement de l'échantillon est utilisé pour obtenir un mouvement circulaire (figure 4a) dont les deux dimensions histogramme devrait maintenant montrer une couverture uniforme (figure 4b). La trace q correspondant de temps (t) des fluctuations angulaires (obtenus à partir de la conversion de l'(x, y) positions, voir ci-dessous) ne montre aucune périodicité correspondant à 360 ˚ (Figure 4c) et révèle de grandes excursions correspondant à plusieurs tours complets (Figure 4d). Le paysage énergétique impliciteest harmonique sur une plage de> 1000 ˚ (figure 4e). L'écart-type des fluctuations est σ θ = 223 °, ce qui correspond à une rigidité de piège angulaire de k θ = k B T / σ θ 2 = 0,27 pN · nm / rad, qui à son tour donne une estimation de la longueur de torsion efficace de persistance de l'ADN égal à C = C L / σ θ 2 ~ 76 nm (L C = 1,150 nm pour l'ADN de 3,4 kb utilisé dans cette mesure) à la force mesurée.
Un exemple de la façon dont FOMT peut être utilisé pour mesurer la variation de la torsion induite dans la molécule d'ADN attaché à travers la liaison de protéines 31, 34 est représenté sur la figure 5. Ici, nous avons surveillé la liaison de la protéine RAD51 doublerBrin d'ADN; RAD51 est connu à la fois allonger et détendre l'ADN, en forme d'un filament de nucléoprotéine 31. Après rinçage RAD51 dans le flux de cellules, nous observons que le bourrelet subit une trajectoire en spirale dans le FOMT (figure 5a). En convertissant trace de (x, y) le mouvement en fonction du temps de q (t) tel que décrit ci-dessus, nous pouvons co-tracé de l'effet que RAD51 a sur la longueur de l'ADN d'attache et son degré de déroulage (figure 5b, c) .
Une autre approche pour mesurer les propriétés de torsion de l'ADN sont le MTT (figure 1c, figure 6). Le schéma de la figure 6a illustre le principe de la mesure: à partir de la surtension (ou sous-renvidage) l'ancrage de l'ADN par N spires, l'ADN exerce un couple de rappel sur le bourrelet qui conduit à un changement dans la position angulaire d'équilibre à partir de θ 0 et θ N. Dans le MTT la composante transversale du champ magnétique est réduite par rapport à la TA, ce qui facilite la mesure de ces déplacements angulaires bourrelet tout en permettant la rotation (figure 1). L'amplitude du décalage angulaire mesurée après l'application de N = 45 tourne à un ADN de 7,9 kpb est représenté sur la figure 6b. La séquence complète du protocole de mesure MTT et le résultat obtenu d'un couple de rotation par rapport à la courbe de l'ADN sont présentés à la Figure 6c-f. Ici, les mesures de l'écart type (figure 6c) et la moyenne (figure 6d) de la coordonnée angulaire sont indiqués en fonction de sur-et sous-renvidage, avec l'écart type est inversement proportionnel à la rigidité de piège angulaire (équation 1). Pris ensemble, ces quantités permettent de construire un couple contre la courbe de rotation de l'ADN (figure 6f), qui devrait montrer une région de la réponse linéaire centrée sur 0 désactive unend deux plateaux au cours de laquelle les acides gras saturés, de couple à la rotation positive et négative, respectivement. Un tel couple de rotation par rapport à la courbe complète les informations en une extension par rapport à la courbe de rotation (figure 6e), la quantification de ce fait les transitions qui accompagnent le flambage et la dénaturation de l'ADN.
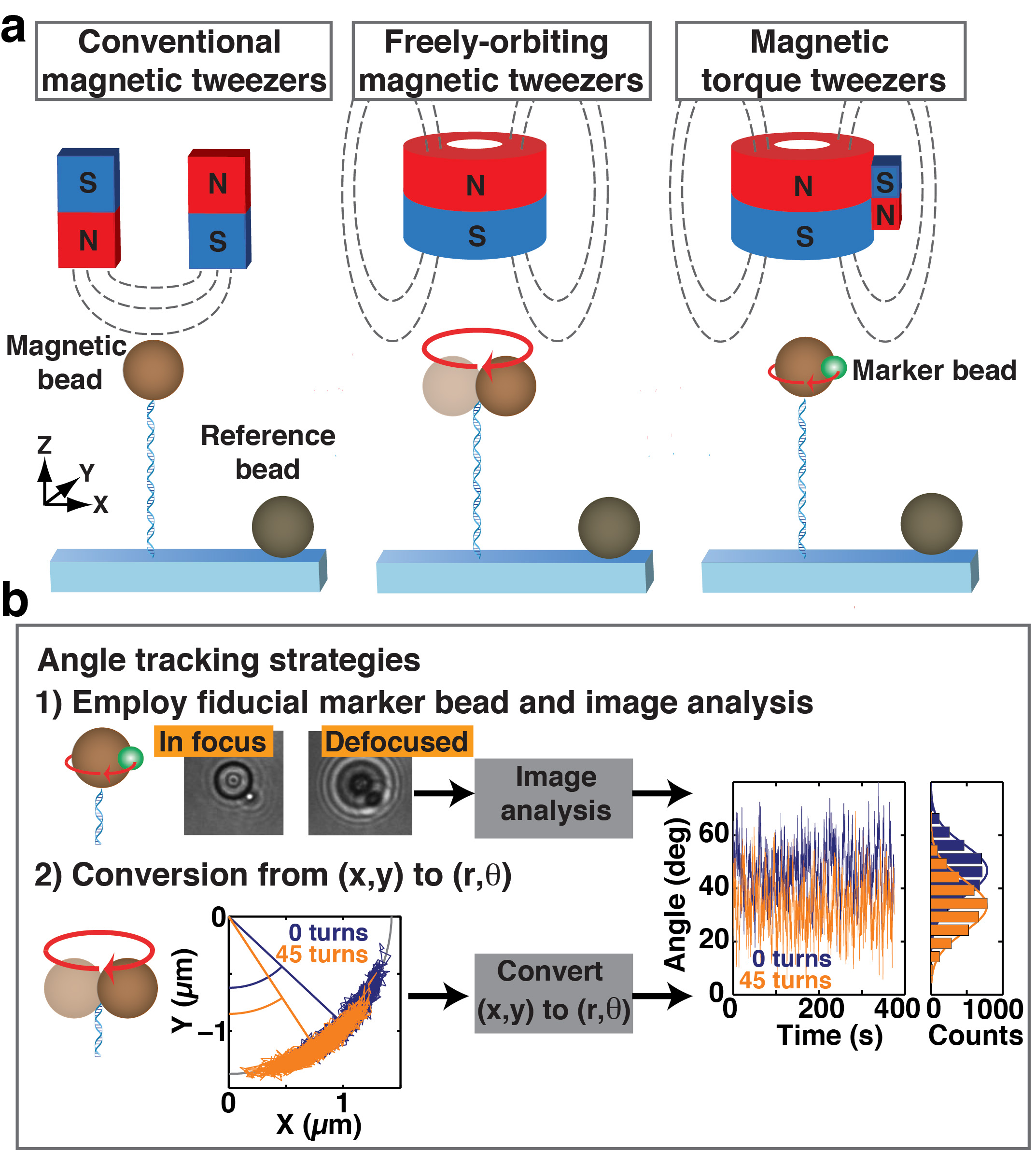
Figure 1. Schéma de pinces magnétiques classiques (MT), librement orbite pinces magnétiques (FOMT), une pince à épiler de couple magnétique (MTT), et deux stratégies pour le suivi de l'angle de rotation. (A) Dans les trois réalisations de pinces magnétiques, les billes magnétiques sont attachés à une surface de la cellule d'écoulement par des macromolécules fonctionnalisés, par exemple, les molécules d'ADN double brin représentés schématiquement. des billes de référence sont attachés à la surface de la cellule d'écoulement et suivis pour drift correction. Tous les trois MT set-ups utilisent des aimants à appliquer une force d'étirement vers le haut sur la bille magnétique et, par conséquent, l'ADN attache. Dans MT classique, une paire d'aimants exerce un champ magnétique qui est orienté transversalement par rapport à l'axe d'attache, ce qui limite fortement la rotation de la bille autour de l'axe de l'ADN-attache. Dans FOMT, un aimant de forme cylindrique fournit un champ magnétique orienté selon que la direction de la sangle. Lorsque l'attache est aligné sur le centre de l'aimant de forme cylindrique, les champs transverses restantes sont réduites au minimum, permettant la libre rotation autour de l'axe d'attache En MTT, un aimant latéral est ajouté à l'aimant de forme cylindrique utilisé dans FOMT afin de fournir un petit champ transversal (réduite en grandeur par rapport à la MT). Ce petit domaine transversal permet l'application d'un couple, ainsi que de sa mesure. (B) Deux stratégies pour mesurer l'angle d'une bille magnétique autour de l'axe de rotation de l'ADN-attache sont représentés. 1): un bourrelet de repérage (degrén) attaché à la bille magnétique (brun) donne une image asymétrique qui permet la poursuite en angle par analyse imaginer. Deux images CCD d'une bille magnétique de 1,4 um-rayon avec un marqueur de repère de 0,5 um-rayon sont affichés, la mise au point et out-of-focus. 2): lorsque l'ADN est attaché à la bille magnétique à une position éloignée du pôle sud de la perle, le centre de la bille oscille le long d'un arc dont le centre définit une position angulaire. Soit stratégie peut être utilisée pour suivre l'angle de rotation et de surveiller les changements dans la position de l'angle que l'attache est en torsion tendu (traces sur la droite), permettant ainsi des mesures de couple de la molécule unique.

Mesures d'étalonnage de l'ADN Figure 2. Dans la MT classique. (A) courbes de rotation-extension pour un ADN de 7,9 kb prise à F = 00,25, 0,5, et 2,0 pN. La réaction asymétrique en rotation à tour de rôle positifs et négatifs de simples attaches d'ADN double brin peut être utilisé comme un test pratique de l'attache de retenue. (B) la courbe Force-extension pour un ADN kb 7,9, avec un ajustement de la vis sans fin comme modèle de chaîne (ligne solide), ce qui donne un contour équipée longueur L C = 2,71 um et pliage longueur persistance L P = 45 nm. Toutes les mesures ont été effectuées dans du tampon PBS.
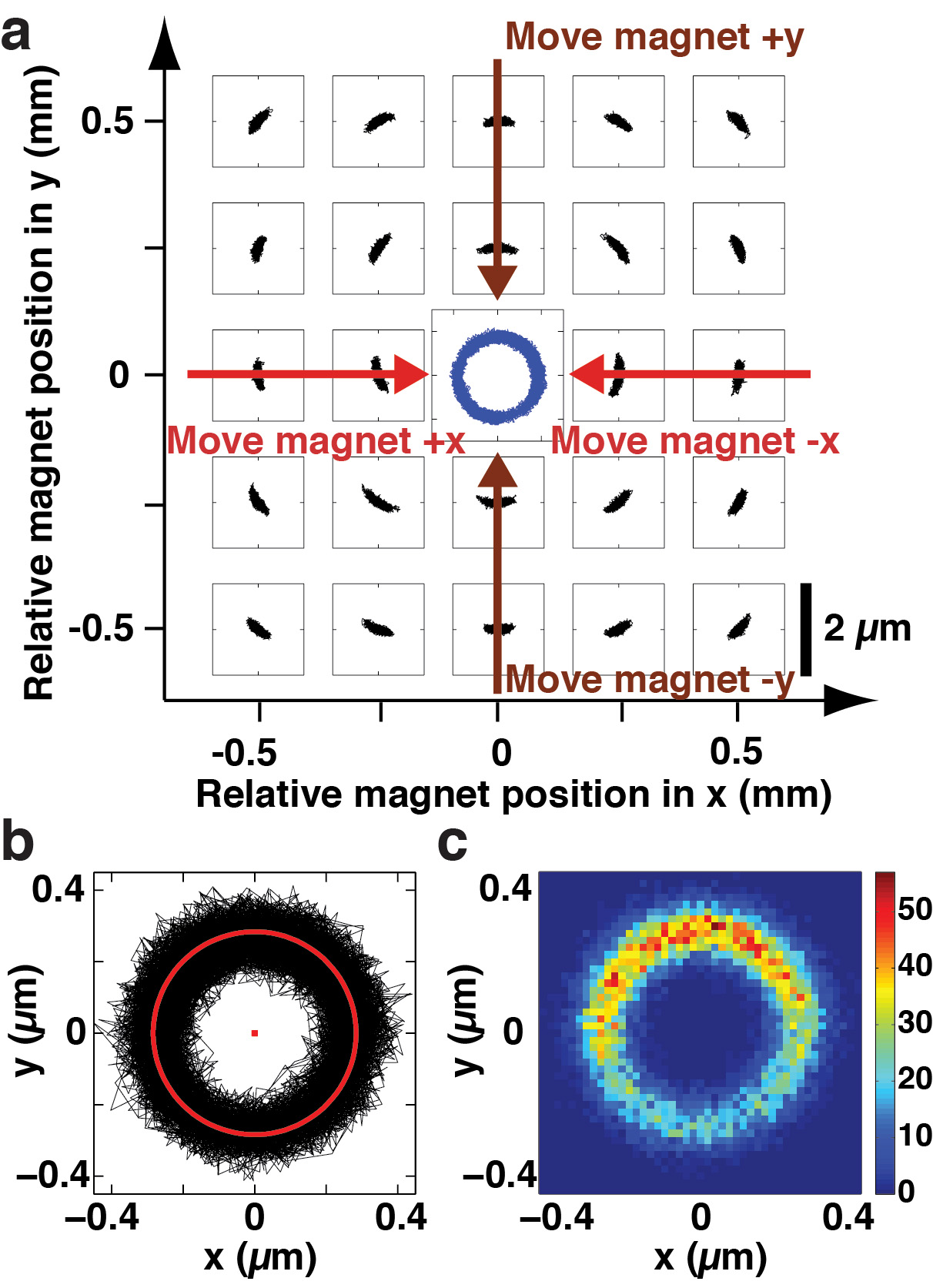
Figure 3. L'alignement dans le FOMT. (A) (x, y) des fluctuations de bourrelet d'ADN-captif détenues dans le FOMT en fonction de la position de l'aimant. La position de l'aimant cylindrique a été scannée à une hauteur constante de 3 mm à travers la surface de la cellule d'écoulement par pas de 250 um de x et Y et est indiqué sur les axes de l'intrigue extérieures. A chaque (x, y) position de l'aimant, les fluctuations de la même bille d'ADN-captif ont été enregistrés et sont tracées dans les petits systèmes de coordonnées (la barre d'échelle en bas à droite s'applique à tous les sous-systèmes de coordonnées). Variations systématiques de (x, y) de la moulure motif de fluctuation de la position de l'aimant qui ressemble à un cyclone ou vortex sont apparents. Ce motif de "vortex" peut être utilisé pour guider le déplacement de l'aimant (ou en variante l'élément d'attache tout en maintenant l'aimant fixe) en x et y (indiqué par les grosses flèches) pour réaliser l'alignement. Lorsque l'alignement grossier est terminée, (x, y) de la perle-fluctuations tracent une trajectoire circulaire (trace bleue dans le centre de la parcelle). Cette trace a été enregistré dans une expérience séparée après alignement des aimants dans les petites étapes sur le centre et est affiché à titre d'illustration dans ce complot. (B) (x, y) des fluctuations d'un cordon d'ADN-captif qui s'est tenue à til FOMT après le succès grossière alignement de l'aimant (trace noire). Les fluctuations sont situés sur un anneau circulaire et les fluctuations thermiques sont suffisantes pour explorer tous les angles des rotations sur le cercle. Un cercle en place est représenté en rouge. (C) Un histogramme correspondant aux données (b), montrant que l'alignement grossier ne garantit pas une couverture uniforme de toutes les positions possibles le long de l'anneau circulaire. Même si les fluctuations thermiques sont suffisantes pour explorer tout l'angle de rotation sur le cercle, il reste une barrière d'énergie (sur de l'ordre de l'énergie thermique k B T) à rotation libre.
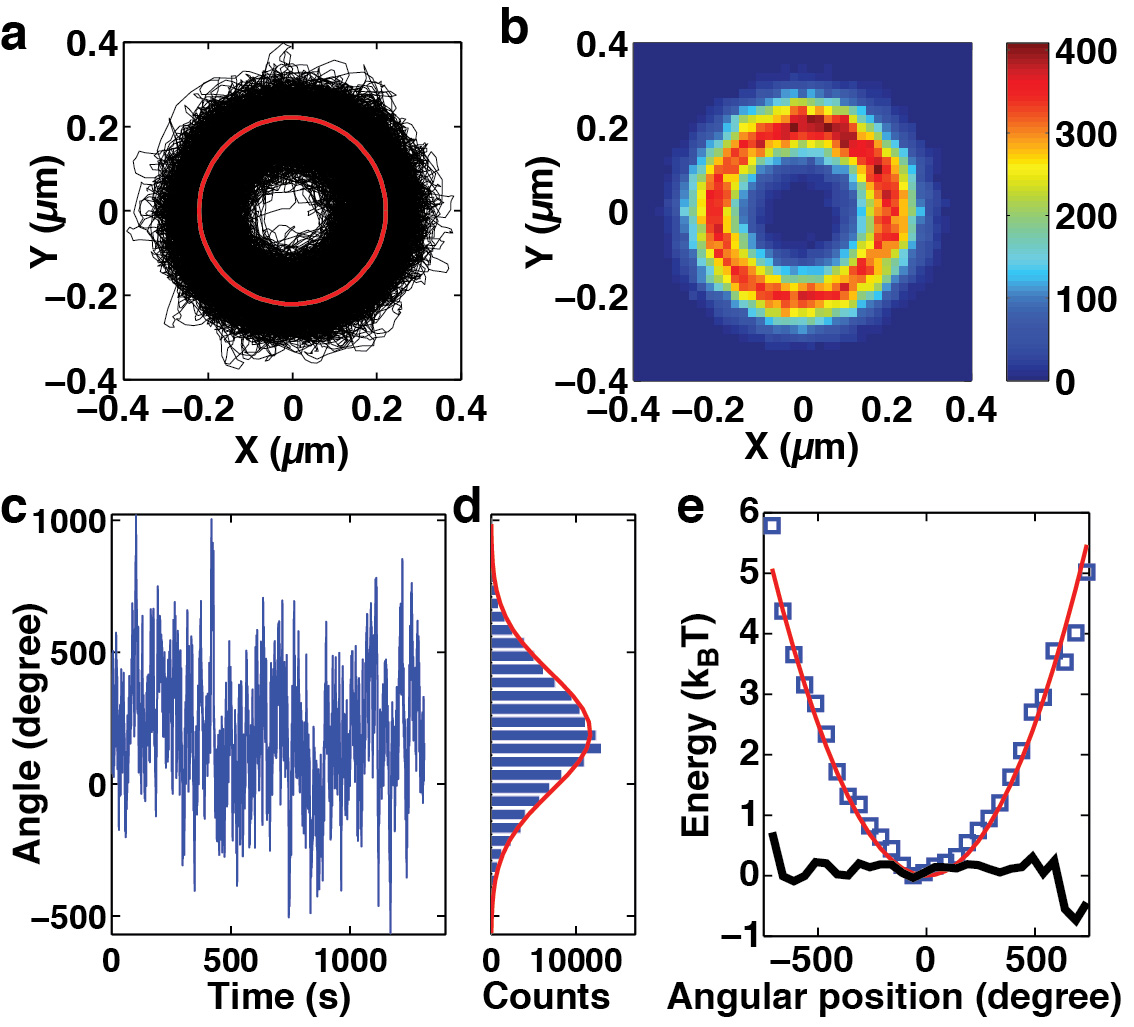
Figure 4. Mesure de la rigidité en torsion de l'ADN en utilisant FOMT. (X, y)-trajectoire (a) et l'histogramme (b) d'un ADN-tethEred les fluctuations de perles après alignement fin de la position de l'aimant-attache par rapport à la FOMT. Dans ces circonstances, l'histogramme révèle couverture sensiblement uniforme de la position sur le cercle (C). Fluctuations de rotation de la bille déterminée à partir de la (x, y)-positions. (D) de l'histogramme des fluctuations rotationnelles. La ligne rouge est un ajustement gaussien avec σ θ = 223 °. (E) Le paysage de l'énergie impliquée par la densité de fluctuation de rotation de (c) et (d). La différence entre le paysage énergétique implicite par les fluctuations de rotation et une approximation harmonique (k θ = k B T / σ θ 2 = 0,27 pN-nm/rad) est beaucoup plus petit que l'énergie thermique k B T sur plusieurs tours. Les données sont décalées pour plus de clarté telle que θ 0 = 0. La largeur deles variations peuvent être utilisées pour déterminer la rigidité en torsion de l'ADN, voir le texte principal. La mesure a été effectuée dans du tampon PBS à une force d'étirage de 1 ~ pN. Les données sont adaptés de Lipfert et al 21.

Figure 5. L'liaison de la protéine RAD51 à l'ADN mesurée à l'aide FOMT. (A) de la protéine RAD51 Assemblée sur un captif 7,9 kbp ADNds contrôlée à 3,5 pN. (X, y, z)-trajectoire exécuté par la bille magnétique (1,0 mm de diamètre) au cours de la 200 premières secondes de l'ensemble est représenté, avec le temps un code de couleur du bleu au rouge. (B) L'extension de la ADNds déduit à partir de la composante z de la trajectoire de la bille dans (a) comme une fonction du temps. (c) L'angle de rotation autour de l'axe d'attache ADNdb déduitesà partir de x, y d'éléments de la trajectoire de talon en (a) en fonction du temps.
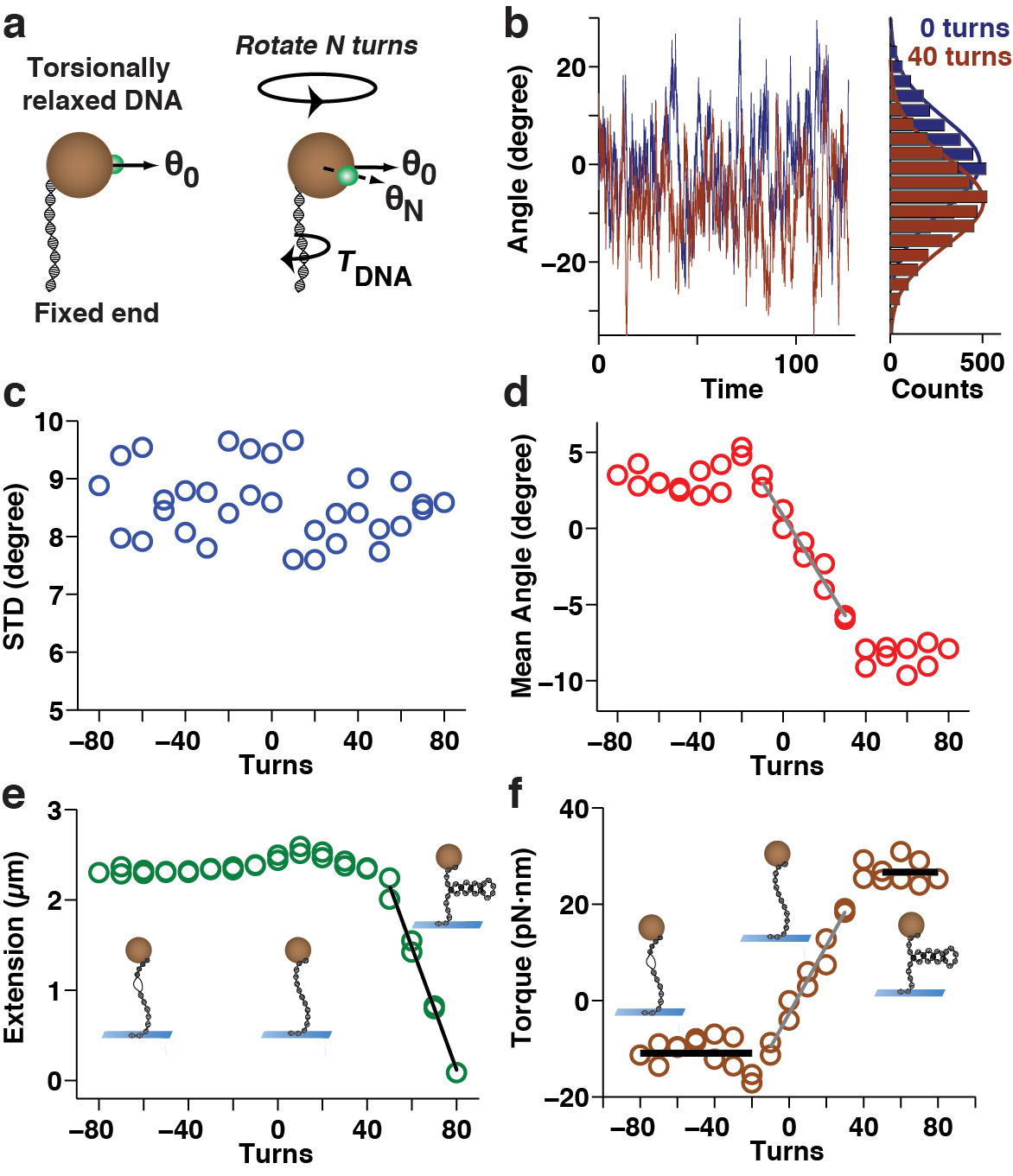
Figure 6. Mesures de couple sur une seule attache de l'ADN dans le MTT. (A) Représentation schématique montrant le principe de la mesure du couple. Après over-(ou sous-) enrouler la longe de l'ADN par N spires, l'ADN exerce un couple de rappel sur le bourrelet qui conduit à un changement dans la position angulaire d'équilibre à partir de θ 0 pour θ N. (B) Exemple de traces d'angles utilisés pour mesurer le couple:. fluctuations angulaires d'un cordon attaché à une molécule de 7,9 ADN kbp torsion détendu avant (bleu) et après l'introduction de 40 tours (rouge foncé) (cf) La mesure du couple sur une molécule d'ADN de 7,9 kbp en tampon PBS tenue à une stvigueur de ~ 3 PN vomir en utilisant le cordon de repère de calage à base de protocole de suivi angulaire. Fluctuations angulaires comme indiqué en (b) ont été enregistrées en fonction du nombre de tours appliqués. (C) L'écart type des variations angulaires en fonction de spires appliquées. La largeur des fluctuations est approximativement constante, ce qui indique la raideur du piège angulaire constante. (D) Le décalage de l'angle de rotation moyen en fonction de spires appliquées. Décalages systématiques de l'angle moyen à sur-et sous-renvidage sont apparents. (E) Le prolongement d'ancrage de l'ADN surveillés simultanément en fonction de spires appliquées. (F) Le couple exercé par l'élément d'attache d'ADN déterminée à partir de l'angle moyen représenté en (d) , voir le texte principal. Sur-et sous-renvidage tourne autour de zéro donne naissance à un couple en fonction de la réponse linéaire tours de l'ADN-attache (pentes gris équipés ionique (d) et (f)) qui peut être utilisé pour déterminer la longueur effective de torsion de persistance (~ 77 nm pour cet ensemble de données). Surtension outre conduit au flambage et la formation de supercoils plectonemic (représentés schématiquement dans les encarts), correspondant à un plateau de couple (ligne noire à tour de rôle positifs dans (f) à ~ 26 pN · nm) et une diminution linéaire de l'extension d'attache avec le nombre de tours (piste noire en (e)). Dénouement au-delà du régime linéaire provoque l'ADN pour faire fondre localement (montré dans les encarts sur la gauche), marquée par un plateau de couple égal au couple de fusion (ligne noire au tournant négatif dans (f) à ~ -11 pN · nm).
Discussion
Lors de l'exécution des expériences utilisant le MTT ou la FOMT, un certain nombre de choix doivent être faits en ce qui concerne des perles, des aimants, des protocoles de suivi, etc Les meilleurs choix à faire dépendront de l'expérience d'intérêt. Ci-dessous, nous décrivons les compromis qui accompagnent des choix différents, ce qui devrait faciliter la sélection d'une expérience particulière. Ensuite, nous décrivons plusieurs étapes critiques qui accompagnent l'alignement et le fonctionnement des expériences MTT et FOMT. Enfin, nous discutons de l'importance de la MTT et FOMT par rapport aux méthodes existantes ainsi que les applications futures.
Considérations avant le début de MTT et FOMT expériences
Toute expérience oblige à sélectionner un type de bille magnétique pour utilisation. On peut choisir entre plusieurs disponibles dans le commerce des perles recouvertes de streptavidine superparamagnétiques, par exemple, de 0,25 um billes de rayon, 0,5 um billes de rayon, soit 1,4 um billes de rayon (de see la table des matières). Grandes perles auront un moment magnétique accrue par rapport aux petites perles (environ échelle que le volume) et donc leur utilisation facilitera l'application de forces supérieures (pour les forces typiques obtenus dans nos instruments, voir le tableau 1). Lorsque le suivi angulaire en utilisant des billes de marqueur est souhaitée, nous travaillons généralement avec 1,4 um rayon et utilisons 0,5 um rayon perles biotinylés non-magnétiques comme des perles de marqueurs (voir le paragraphe 1.9 du protocole de fixation correspondante). L'utilisation de petites perles est particulièrement recommandé pour la FOMT, comme le temps caractéristique de rotation de perle τ C est égal au rapport de la traînée du système sur son ressort constante γ / k θ; Surtout, le coefficient de traînée de rotation pertinentes pour les échelles de l'échelle de temps de mesure angulaire que R ~ bourrelet 3, c'est à dire avec la troisième puissance du rayon (voir le tableau 2 pourles échelles de temps caractéristique pour plusieurs combinaisons de perles ADN dans FOMT et des mesures de MTT). Réductions d'accompagnement à la force maximale pouvant être appliquées peuvent être traitées en utilisant une pile renversé des aimants cylindriques 27. Néanmoins, dans les mesures FOMT il peut parfois être nécessaire de faire des compromis entre la meilleure résolution temporelle réalisable et la force maximale appliquée.
En outre, une expérience requiert la sélection d'une configuration d'aimant. Dans la configuration classique des pinces magnétiques (figure 1a), on utilise typiquement une paire de 5x5x5 mm aimants cubes en orientation verticale avec un écart de 0,5 ou 1 mm entre les aimants 4. Lorsque les aimants sont espacés le long de l'axe x (y), on obtient un champ magnétique qui est essentiellement dirigé le long de l'axe x (y). Pour les expériences FOMT, un aimant de forme cylindrique est sélectionné au centre de laquelle le champ magnétique est essentiellement dirigéle long de l'axe z (figure 1b). Dans la pratique, on utilise un empilement de trois de ces aimants de forme cylindrique, chacune ayant un diamètre de 6 mm et un trou central d'un diamètre de 2 mm, pour une épaisseur totale de 6 mm. Lorsque des forces de traction plus élevées sont souhaitées, une configuration magnétique "de la pile renversé", dans lequel l'aimant inférieur est empilée avec une aimantation opposée est préféré. Pour atteindre la configuration de MTT (Figure 1c), on ajoute un petit aimant supplémentaire sur le côté de la pile principale de l'aimant de la configuration FOMT, typiquement un cylindre solide ayant un diamètre de 4 mm et une hauteur de 7 mm. Pour voir comment les forces maximales obtenus dans nos instruments dépendent de la configuration de l'aimant, voir le tableau 1.
L'alignement de MTT et FOMT expériences
Etant donné que les billes magnétiques ont une surface (approximativement) uniformément fonctionnalisé (typiquement la streptavidine) et depuis la fixation à la fois du n fonctionnaliséattaches d'acide ucleic et perles de marqueurs (au cas où la poursuite angulaire à base de billes marqueur est utilisé) se produit via un simple incubation dans une solution, on ne contrôle pas où l'attache et / ou marqueur perle attachent à la bille magnétique. Les billes magnétiques ont un axe de magnétisation préféré qui tend à aligner le long de la direction du champ externe. Si l'on note les points où l'axe de magnétisation préférée croise la surface de la perle que les pôles nord et sud, puis perles où l'ADN-attache est fixé près de l'équateur seront tracer un anneau circulaire avec un rayon proche ou légèrement supérieure à la rayon de la bille dans le FOMT; en revanche, les perles qui sont attachés près du pôle sud fluctuent sur un anneau circulaire très petit rayon dans la FOMT, ce qui peut empêcher le montage du cercle en utilisant les équations 3-5. Nous notons que par la géométrie sphérique simple, la probabilité de fixation à proximité de l'équateur est beaucoup plus grande que un attachement exactement aux pôles; par conséquent, les plus bEADS être attaché de telle sorte que le suivi angulaire basée sur le plan (x, y) peut être effectuée avec succès.
Un argument similaire vaut pour la fixation des perles de marqueurs pour le marqueur à base de poursuite angulaire de repère. Le cordon de marqueur est utilisé pour créer une asymétrie dans l'image de la bille magnétique qui permet le suivi de l'angle. Si le cordon de marqueur est fixé exactement au niveau du pôle nord ou sud de la bille (à savoir directement au-dessus ou sur le dessous), l'image obtenue est toujours symétrique en rotation et le protocole de repérage angulaire échoue. Cependant, par le même argument de géométrie sphérique, la possibilité pour un cordon de marqueur pour attacher directement à l'un des pôles est relativement faible; nous constatons que dans la pratique la plupart des perles de marqueurs donnent une asymétrie suffisante pour permettre le suivi angulaire. Enfin, nous notons que dans les pinces magnétiques classiques, la direction du champ est en (x, y)-plan; par conséquent, l'axe du cordon d'aimantation préférés s'aligneront dans ee (x, y)-plan et les pôles nord et sud, tel que défini ci-dessus, vont être aux côtés de la perle, peu probable que la situation dans le FOMT ou MTT, où les pôles sont en haut et en bas.
Dans des expériences FOMT, une étape essentielle est l'alignement de l'aimant cylindrique de telle sorte que le champ magnétique radial est négligeable au voisinage de la perle. Cet alignement est effectuée pour une seule bille à la fois. Pour juger si un mouvement de talon dans le FOMT est répartie uniformément sur un anneau circulaire, le temps de mesure doit être supérieure à 20 · C τ. Comme τ est égal à C ~ 45 sec à 8 kpb de l'ADN et une bille de 0,5 mm de rayon, le temps de mesure est d'environ 900 secondes dans la phase finale de l'alignement. A titre de comparaison, l'utilisation de l'ADN 1,9 kb et 0,25 mm perles de rayon réduit τ C vingt fois à ~ 2 sec (voir également le tableau 2).
Étapes critiques et des considérations pour le suivi des cours de FOMT et MTT expériences
Pour suivre les variations du talon dans le plan, c'est à dire sa position (x, y) en position, on emploie une analyse de corrélation croisée des profils d'intensité affichée par un bourrelet à des intervalles de temps suivants 35, 36. Ceci peut être effectué à une résolution inférieure au pixel avec une précision de quelques nanomètres à 20. Pour suivre le mouvement de la bille en z, nous utilisons généralement une méthode d'abord conçue par Gosse et Croquette, dans lequel le plan focal de l'objectif (OFP) est précisément déplacé dans la direction verticale tandis que l'imagerie des anneaux de diffraction du cordon attaché à l'acide nucléique 20 . De cette manière, un profil d'étalonnage est générée à corréler le spectre de diffraction de la nervure à la distance entre le bourrelet et le PFO 19. Lorsque ce profil de calibrage est interpolée, les déplacements verticaux du bourrelet peuvent être également mesurées avec une précision allant jusqu'à quelques nm 20.Nous renvoyons le lecteur aux références supplémentaires qui décrivent des algorithmes plus raffinés de suivi 37, 38 ainsi que leur application au suivi de plusieurs perles 5, 6, 37 parallèles.
Lorsque vous utilisez le suivi angulaire qui repose sur la conversion de (x, y) positions en coordonnées angulaires, nous vous conseillons de procéder comme suit. À partir d'une trace de temps dans laquelle le bourrelet trace une couronne circulaire, utilise le (x i, y i) positions (où l'indice i désigne les points de mesure ultérieurs) en fonction du centre du cercle (x 0, y 0) et cercle de rayon R (figure 2a) en réduisant au minimum:
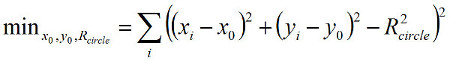 (3)
(3)
où la somme s'étend sur tous les points de données. Après Fitting x 0, y 0, et R cercle, déterminer les coordonnées polaires (r i, θ i) de chaque point de données dans la trace de temps à l'aide:
 (4)
(4)
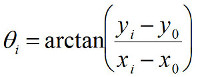 (5)
(5)
Notez que l'on doit prendre soin de "déballer" l'angle θ, c'est à dire d'ajouter des sauts de phase de ± π le cas échéant. Code personnalisé écrit pour le montage et la conversion de (x, y) à (r, θ) coordonne est disponible auprès des auteurs sur demande. Dans le FOMT, une trace temporelle dans laquelle le bourrelet trace une couronne circulaire peut être obtenue par la réalisation de l'alignement grossier (cf. étape 3.3) et en enregistrant les variations thermiques de la perle. Dans le MTT, fluctuation thermiquetions sont insuffisantes pour tracer l'anneau circulaire; à la place, utiliser une trace du temps où les aimants sont lentement (généralement à 0,1 Hz) tourné par plusieurs tours pour s'adapter le cercle en utilisant les équations 3-5.
Nous notons que pour le MTT, il est important de choisir l'approche de suivi angulaire appropriée, soit par l'intermédiaire d'un marqueur de suivi angulaire (figure 1c, figure 1d, figure 3a) ou via la conversion de (x, y) positions en coordonnées angulaires ( Figure 1d, figure 2b). Bien que généralement la précision de la poursuite angulaire à partir de (x, y)-positions et l'utilisation de billes de marquage sont comparables, il est important de réaliser que la diaphonie se produit entre les fluctuations d'une perle en (x, y) et dans l'angle, comme décrit dans l' Janssen et al 32: ainsi, le suivi angulaire de (x, y) positions n'est valable que pour autant que les fluctuations browniennes en (x, Y) ne contribuent que de façon négligeable à l'incertitude de la coordonnée angulaire, et son utilisation appropriée de (x, y) de suivi peut nécessiter de réglage de la raideur de la trappe de rotation par l'intermédiaire de l'ajustement de la position de l'aimant latéral. En règle générale, l'utilisation de la raideur plus élevée piège requiert l'utilisation de poursuite angulaire en utilisant des billes de marquage. L'utilisation de billes de marquage nécessite une étape supplémentaire de fixation, ce qui peut réduire le nombre d'attaches utilisables (voir le protocole d'attachement à l'étape 1.9). Lors de l'utilisation du suivi en fonction de talon-terrain, il est important de sélectionner des billes magnétiques qui ont un bourrelet de marqueur est fixé près de l'équateur pour obtenir les meilleurs résultats.
Importance de la FOMT et MTT Approches par rapport aux méthodes et applications existantes
Dans ce qui précède, nous avons montré comment on peut, à partir de MT classique, facile à modifier les configurations magnétiques pour convertir l'instrument en MTT ou FOMT. Cette m simpleodification, qui peut être accompagné par la mise en place du suivi angulaire lorsque l'utilisation d'un marqueur de suivi angulaire est souhaitée, est un point fort immédiate des deux configurations, car il permet à l'utilisateur d'appliquer un couple, mesurer le couple, ou de mesurer la torsion en fonction de la expérimenter à portée de main. Comme mentionné dans l'introduction, à la fois FOMT et MTT profit de la plupart des forces existantes de MT, notamment leur simplicité, avec le MTT en particulier en bénéficiant de la capacité de mesures parallèles 5, 6 (ce ne sont pas aussi facilement atteints en FOMT donné l'exigence d'alignement de l'élément d'attache par rapport au centre de l'aimant cylindrique). Notamment, MTT et FOMT ne nécessitent pas, contrairement à d'autres techniques, spécialement des particules de nano-fabriqué 22, 39, 40, conception optique complexe 41, ou l'introduction de perles supplémentaires dans le captif (ADN) 42. Cette outres techniques peuvent néanmoins fournir d'autres avantages comme la haute résolution temporelle 27, 43, 44. Les deux FOMT et MTT devraient trouver des applications futures dans l'étude de la transformation du génome, comme le comportement des moteurs moléculaires sur l'ADN est à la fois influencé par et a des conséquences sur la touche locale et de couple. Des applications supplémentaires peuvent être trouvés dans le domaine émergent de la nanotechnologie de l'ADN 27 ou dans le domaine plus large de moteurs rotatifs actifs dans le traitement biologique 7, 45.
| M270 (R bourrelet = 1,4 um) | MyOne (R bourrelet = 0,5 um) | Ademtech (R bourrelet = 0,25 pm) | |
| MT classique (paire de cubes de 5 x 5 x 5 mm 3 aimants, 1 écart mm, alignement vertical) | 70 pN | 8 pN | 1,6 pN |
| FOMT ou MTT * (empilement de trois aimants cylindriques, de diamètre 6 mm, écart 2 mm de diamètre) | 9 pN | 1 pN | 0,2 pN |
| FOMT ou MTT * (empilement de trois aimants cylindriques, un diamètre de 6 mm, une fente de mm de diamètre) | 18 pN | 2 pN | 0,4 pN |
| FOMT ou MTT * (pile de trois aimants cylindriques dernier retournées, espace de 1 mm de diamètre) | ~ 50 pN | 9 pN | 1,8 pN |
* La présence du petit aimant latéral dans le MTT a un effet négligeable sur la force d'étirement
Tableau 1. Forces maximales généralement obtenus pour les configurations magnétiques différentes et de types de perles.
| R bourrelet = 1,4 um | R bourrelet = 0,5 pm | R perle =0,25 um | |
| Coefficient de frottement * | 120 pN · nm · s | 5,5 pN · nm · s | 0,7 pN · nm · s |
| Échelle de temps caractéristique: FOMT, 10 kbp ADN ** | 1200 sec | 55 sec | 7 s |
| Échelle de temps caractéristique: FOMT, ADN de 1 kbp | 120 sec | 5.5 sec | 0,7 sec |
| Échelle de temps caractéristique: MTT, k q = 100 pN · nm / rad | 1,2 s | 0,06 sec | 0,007 sec |
| Échelle de temps caractéristique: MTT, k q = 1000 pN · nm / rad | 0,12 sec | 0,006 sec = 6 ms | 0.0007 s = 0,7 ms |
* Coefficient de friction pour une rotation autour d'un axe passant par le "équateur" (c'est à dire la situation représentée sur la figure 1b), Donnée par 14 · p · h · R bourrelet 3, où h est la viscosité du tampon.
** Dans le FOMT, la raideur de la trappe de rotation est donnée par la rigidité en torsion de l'ADN, k q, ADN = C · k B T / L C, où C est la longueur de torsion efficace de persistance, qui est supposé être de 80 nm ici ( qui est caractéristique d'un régime de force intermédiaire, F ~ 1 pN) et C L est la longueur du contour de l'ADN, de 0,34 nm par paire de bases.
Tableau 2. Coefficients de frottement et temps caractéristique des échelles pour FOMT et MTT.
Déclarations de divulgation
Un brevet relatives à ces travaux a été déposée en vertu PCT/NL2011/050446 de référence.
Remerciements
Ce travail a été soutenu par la TU Delft, l'Organisation néerlandaise pour la recherche scientifique (NWO), la Fondation pour la recherche fondamentale sur la matière, et par la Fondation européenne de la science.
matériels
| Name | Company | Catalog Number | Comments |
| Sandblaster | Great Lake Orthodontics | 190-070 Microetcher II | |
| Nitrocellulose | Life Technologies | LC2001 | |
| Magnetic particle concentrator | Life Technologies | 12002D | |
| Non-magnetic latex beads (0.5 μm radius) | Polysciences | 17010 | |
| Non-magnetic latex beads (1.5 μm radius) | Sanbio | PV05N/2179 | |
| Antidigoxigenin | Roche | 11 214 667 001 | |
| Streptavidin-coated superparamagnetic beads (0.25 μm radius) | Ademtech | 3150 | |
| Streptavidin-coated superparamagnetic beads (0.5 μm radius, “MyOne”) | Life Technologies | 650.01 | |
| Streptavidin-coated superparamagnetic beads (1.4 μm radius, “M270”) | Life Technologies | 653.05 | |
| Biotin-coated latex beads (0.5 μm radius) | Life Technologies | F-8768 | |
| Cubic magnets for conventional tweezers | Supermagnete | W-05-N50-G | |
| Cylindrical magnet for MTT and FOMT | Supermagnete | R-06-02-02G | |
| Side magnet for MTT | Supermagnete | S-04-07-N | |
| Linear stage | Physik Instrumente | M-126.PD | |
| Rotary stage | Physik Instrumente | C-150 | |
| High-resolution automated sample stage | Physik Instrumente | P-733.2D | |
| Software for coding analysis routines | The Mathworks | MATLAB | custom-written routines are available from the authors |
Références
- Strick, T. R., Allemand, J. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271, 1835-1837 (1996).
- Bustamante, C., Bryant, Z., Smith, S. B. Ten years of tension: single-molecule DNA mechanics. Nature. 421, 423-427 (2003).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature methods. 5, 491-505 (2008).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical journal. 96, 5040-5049 (2009).
- Ribeck, N., Saleh, O. A. Multiplexed single-molecule measurements with magnetic tweezers. The Review of scientific instruments. 79, (2008).
- De Vlaminck, I., et al. Highly parallel magnetic tweezers by targeted DNA tethering. Nano letters. 11, 5489-5493 (2011).
- Koster, D. A., Crut, A., Shuman, S., Bjornsti, M. A., Dekker, N. H. Cellular strategies for regulating DNA supercoiling: a single-molecule perspective. Cell. 142, 519-530 (2010).
- Dulin, D., Lipfert, J., Moolman, M. C., Dekker, N. H. Studying genomic processes at the single-molecule level: introducing the tools and applications. Nature reviews. Genetics. 14, 9-22 (2013).
- Ajjan, R., et al. Common variation in the C-terminal region of the fibrinogen beta-chain: effects on fibrin structure, fibrinolysis and clot rigidity. Blood. 111, 643-650 (2008).
- Mierke, C. T., et al. Mechano-coupling and regulation of contractility by the vinculin tail domain. Biophysical journal. 94, 661-670 (2008).
- Shang, H., Lee, G. U. Magnetic tweezers measurement of the bond lifetime-force behavior of the IgG-protein A specific molecular interaction. Journal of the American Chemical Society. 129, 6640-6646 (2007).
- Shang, H. K. P., et al. The application of magnetic force differentiation for the measurement of the affinity of peptide libraries. J Magn Magn Mater. 293, 382-388 (2005).
- Lee, G. U., Metzger, S., Natesan, M., Yanavich, C., Dufrene, Y. F. Implementation of force differentiation in the immunoassay. Analytical biochemistry. 287, 261-271 (2000).
- Smith, A. S., Sengupta, K., Goennenwein, S., Seifert, U., Sackmann, E. Force-induced growth of adhesion domains is controlled by receptor mobility. Proceedings of the National Academy of Sciences of the United States of America. 105, 6906-6911 (2008).
- Kanger, J. S., Subramaniam, V., van Driel, R. Intracellular manipulation of chromatin using magnetic nanoparticles. Chromosome research : an international journal on the molecular, supramolecular and evolutionary aspects of chromosome biology. 16, 511-522 (2008).
- Tanase, M., Biais, N., Sheetz, M. Magnetic tweezers in cell biology. Methods in cell biology. 83, 473-493 (2007).
- Bausch, A. R., Moller, W., Sackmann, E. Measurement of local viscoelasticity and forces in living cells by magnetic tweezers. Biophysical journal. 76, 573-579 (1999).
- Lipfert, J., Koster, D. A., Vilfan, I. D., Hage, S., Dekker, N. H. Single-molecule magnetic tweezers studies of type IB topoisomerases. Methods Mol Biol. 582, 71-89 (2009).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H., Hinterdorder, P., van Oijen, A. . Handbook of Single-Molecule Biophysics. , (2009).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical journal. 82, 3314-3329 (2002).
- Lipfert, J., Wiggin, M., Kerssemakers, J. W., Pedaci, F., Dekker, N. H. Freely orbiting magnetic tweezers to directly monitor changes in the twist of nucleic acids. Nature communications. 2, 439 (2011).
- Celedon, A., et al. Magnetic tweezers measurement of single molecule torque. Nano letters. 9, 1720-1725 (2009).
- Lipfert, J., Kerssemakers, J. J., Rojer, M., Dekker, N. H. A method to track rotational motion for use in single-molecule biophysics. The Review of scientific instruments. 82, (2011).
- Lipfert, J., Kerssemakers, J. W., Jager, T., Dekker, N. H. Magnetic torque tweezers: measuring torsional stiffness in DNA and RecA-DNA filaments. Nature. 7, 977-980 (2010).
- Mosconi, F., Allemand, J. F., Bensimon, D., Croquette, V. Measurement of the torque on a single stretched and twisted DNA using magnetic tweezers. Physical review letters. , 102 (2009).
- Mosconi, F., Allemand, J. F., Croquette, V. Soft magnetic tweezers: A proof of principle. Review of Scientific Instruments. 82 (12), (2011).
- Kauert, D. J., Kurth, T., Liedl, T., Seidel, R. Direct mechanical measurements reveal the material properties of three-dimensional DNA origami. Nano letters. 11, 5558-5563 (2011).
- Velthuis, A., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative Guidelines for Force Calibration through Spectral Analysis of Magnetic Tweezers Data. Biophysical journal. 99, 1292-1302 (2010).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. The Review of scientific instruments. 83, (2012).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76, 409-413 (1999).
- Lee, M., Lipfert, J., Sanchez, H., Wyman, C., Dekker, N. H. Structural and torsional properties of the RAD51-dsDNA nucleoprotein filament. Nucleic acids research. 41, (2013).
- Janssen, X. J., et al. Electromagnetic torque tweezers: a versatile approach for measurement of single-molecule twist and torque. Nano letters. 12, 3634-3639 (2012).
- Baumann, C. G., Smith, S. B., Bloomfield, V. A., Bustamante, C. Ionic effects on the elasticity of single DNA molecules. Proc Natl Acad Sci U S A. 94, 6185-6190 (1997).
- Lipfert, J., Wiggin, M., Kerssemakers, J. W., Pedaci, F., Dekker, N. H. Freely orbiting magnetic tweezers to directly monitor changes in the twist of nucleic acids. Nat Commun. 2, 439 (2011).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophys. J. 81, 2378-2388 (2001).
- Gelles, J., Schnapp, B. J., Sheetz, M. P. Tracking kinesin-driven movements with nanometre-scale precision. Nature. 331, 450-453 (1988).
- Loenhout, M. T., Kerssemakers, J. W., De Vlaminck, I., Dekker, C. Non-bias-limited tracking of spherical particles, enabling nanometer resolution at low magnification. Biophysical journal. 102, 2362-2371 (2012).
- Kim, K., Saleh, O. A. A high-resolution magnetic tweezer for single-molecule measurements. Nucleic acids research. 37, 136 (2009).
- Deufel, C., Forth, S., Simmons, C. R., Dejgosha, S., Wang, M. D. Nanofabricated quartz cylinders for angular trapping: DNA supercoiling torque detection. Nature methods. 4, 223-225 (2007).
- Huang, Z., Pedaci, F., van Oene, M., Wiggin, M. J., Dekker, N. H. Electron beam fabrication of birefringent microcylinders. ACS nano. 5, 1418-1427 (2011).
- La Porta, A., Wang, M. D. Optical torque wrench: angular trapping, rotation, and torque detection of quartz microparticles. Physical review letters. 92, (2004).
- Gore, J., et al. DNA overwinds when stretched. Nature. 442, 836-839 (2006).
- Bryant, Z., Oberstrass, F. C., Basu, A. Recent developments in single-molecule DNA mechanics. Curr Opin Struct Biol. 22, 304-312 (2012).
- Oberstrass, F. C., Fernandes, L. E., Bryant, Z. Torque measurements reveal sequence-specific cooperative transitions in supercoiled DNA. Proceedings of the National Academy of Sciences of the United States of America. 109, 6106-6111 (2012).
- Forth, S., Sheinin, M. Y., Inman, J., Wang, M. D. Torque measurement at the single-molecule level. Annu Rev Biophys. 42, 583-604 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon