Method Article
Pinzetta magnetici per la misurazione di Twist and Torque
In questo articolo
Riepilogo
Pinzette magnetiche, una potente tecnica di manipolazione singola molecola, possono essere adattati per le misure dirette della torsione (utilizzando una configurazione denominata liberamente orbitante pinzette magnetiche) e coppia (utilizzando una configurazione definita pinzette coppia magnetica) in macromolecole biologiche. Linee guida per l'esecuzione di tali misure sono espresse, comprese le applicazioni allo studio del DNA e associati filamenti nucleo-proteine.
Abstract
Tecniche di singola molecola permettono di studiare il comportamento delle singole molecole biologiche in soluzione in tempo reale. Queste tecniche comprendono cosiddetti approcci spettroscopia di forza come microscopia a forza atomica, pinzette ottiche, flusso di stretching, e pinzette magnetiche. Tra questi approcci, pinzette magnetiche si sono distinti per la loro capacità di applicare una coppia mantenendo una forza di allungamento costante. Qui, è illustrato come un tale "convenzionale" configurazione sperimentale pinzette magnetiche può, attraverso una semplice modifica della sua configurazione di campo per minimizzare la grandezza del campo trasversale, essere atto a misurare il grado di torsione in una molecola biologica. La configurazione risultante è definito pinzette magnetiche liberamente orbitanti. Inoltre, è mostrato come ulteriore modifica della configurazione del campo può produrre un campo trasversale di magnitudo intermedia fra quella del & #8220; convenzionali "pinzette magnetiche e le pinzette magnetiche liberamente orbitanti, che rende possibile di misurare direttamente la coppia memorizzato in una molecola biologica. Questa configurazione viene denominata la pinzetta coppia magnetici. Il video che accompagna spiega in dettaglio come la conversione dei tradizionali pinzette magnetiche in banche-orbitanti pinzette magnetiche e pinzette coppia magnetica può essere realizzato, e dimostra l'uso di queste tecniche. Questi adattamenti mantengono tutti i punti di forza di pinzette magnetici convenzionali, mentre ampliando notevolmente la versatilità di questo potente strumento.
Introduzione
Negli ultimi anni, le tecniche di singola molecola hanno dimostrato la loro ampia applicabilità nello studio delle proteine motrici processive ed altri enzimi, cedendo spaccato la cinetica e la meccanochimica sottostante. Nel contesto di spettroscopia di forza, contributi importanti sono state fatte da microscopia a forza atomica flusso stiramento, e pinzette ottiche e magnetiche. Pinzette ottiche e magnetiche (MT) sono in particolare riusciti a coniugare una grande flessibilità in termini di manipolazione molecolare ad alta risoluzione spaziale e temporale. Qui, ci concentriamo su MT, che può applicare entrambe le forze di stretching e coppie di molecole biologiche impastoiati tra una superficie e perline superparamagnetici 1-3.
Pinzette magnetiche (MT, Figura 1a) sono una tecnica singola molecola molto versatile che è stato usato per monitorare sia le proprietà meccaniche degli acidi nucleici e le loro interazioni con proteine. MT hanno molti forzas, compresi semplicità e robustezza complessiva dell'attuazione sperimentale, l'applicazione facile di coppia, naturale funzionamento e taratura semplice in modalità forza costante 4, ampliamento parallele misure 5, 6, e l'assenza di riscaldamento del campione e fotodanneggiamento. Rispetto ad altri approcci molecola singola, MT fornisce un modo per effettuare misure di forza-dipendenza in forze a partire da ≈ 10 fN e hanno la capacità di controllare semplicemente il grado di superavvolgimento. Mentre MT sono prevalentemente state utilizzate come strumento sperimentale per studiare processi biologici che coinvolgono acidi nucleici 7, 8, hanno anche trovato applicazione in studi sulle proprietà meccaniche di proteine 9-13 o celle 10, 14-17. Numerosi riferimenti utili sono disponibili che descrivono come costruire e gestire un MT 4, 18-20.
Tuter, MT convenzionale non tiene moto rotatorio direttamente, e, mentre si applicano coppia, non misurano direttamente la coppia. Inoltre, essi limitano la libera rotazione del tether acido nucleico. Qui vi presentiamo due proroghe di pinzette magnete. I primi, chiamati liberamente orbitanti pinzette magnetiche (FOMT, figura 1b) 21, consente le misure delle fluttuazioni angolo di equilibrio e cambiamenti nel giro di molecole di acido nucleico frenati, senza vincolare il moto di rotazione attorno all'asse cavezza. Il secondo, definito pinzette coppia magnetici (MTT, Figura 1c), che ha la capacità di applicare e direttamente misurare sia forze e coppie di singole biomolecole 22-27.
Nel seguente protocollo, si presume che il lettore abbia al suo / sua disposizione uno strumento MT 'convenzionale'. Rimandiamo il lettore alla discussione per i riferimenti su come costruire e gestire un MT istituito, così come considerazioni che devono essere presi in considerazione nella selezione dei grani magnetici, magneti, e le routine di rilevamento. Inoltre, le sezioni 1 e 2 del protocollo Testo descrivono come tipicamente preparando e incubare un campione di DNA per l'uso nel MT nonché le misure preliminari che possono essere eseguite su un singolo DNA nel MT convenzionale. Le sezioni 3 e 4 del protocollo Testo illustrano come uno strumento MT può essere facilmente adattato e utilizzato per FOMT e MTT misure.
Protocollo
1. Preparazione e incubazione di un campione di DNA
- Preparare costrutti di DNA che sono legatura duplex estremità (generalmente impiegano ≈ 600 frammenti di PCR DNA bp) che vengono funzionalizzati con più gruppi, rispettivamente 18 biotina e digoxigenin. Per iniziare, una lunghezza tether DNA> 1 micron, per esempio, un 7,9 kbp corrispondente ad una lunghezza compresa di ~ 2,7 micron come impiegato qui, è raccomandato per la facilità d'uso; in particolare, la lunghezza con un DNA che è simile o inferiore rispetto al raggio perla è problematico a causa della geometria allegato nel MTT e FOMT. Vedere la discussione per una descrizione di come lunghezza del DNA influenzi la risposta temporale nel dominio angolare.
- Assemblare le celle di flusso per esperimenti singola molecola. Per le celle di flusso, usare due microscopio vetro coprioggetti separati da una Parafilm distanziale a doppio strato. Il microscopio coprioggetto superiore dovrebbe avere due fori per il fluido in-e sbocchi alla cella. È convenienteutilizzare una sabbiatrice per praticare i fori. Il coprioggetto inferiore è rivestito con nitrocellulosa (0,1% peso / volume in acetato di amile). Posizionare i distanziali parafilm sul lato nitrocellulosa rivestite delle diapositive di fondo e chiudere la parte superiore con il massimo dei vetrini puliti.
- Sigillare le celle di flusso. Usando le pinzette fisici, posizionare la cella di flusso montato su una piastra di riscaldamento impostato a 80-100 ° C per circa 1 min. Prestare attenzione che la cella di flusso sia ben sigillato, che il parafilm non chiude i fori che collegano alla a-e presa, e che i vetrini sono ben allineati.
Nota: Per assicurare una buona tenuta, si consiglia di corsa fuori bolle nel Parafilm utilizzando un grande batuffolo di cotone. La cella di flusso può essere montato sullo strumento pinzette magnetiche. - Preparare buffer. Preparare tampone TE tethering (10 mM Tris-HCl, pH 8,0, 1 mM acido etilendiamminotetraacetico (EDTA), e NaCl 200 mM). In alternativa, si può usare tampone PBS (137 mM NaCl, 2,7 mM KCl, 10 mM tampone fosfato, pH 7.4) addizionato wesima 100 pg / ml BSA, 0,1% Tween e 5 mM sodio azide (PBS +) come tampone di tethering. Lavare i volumi 2-3 cellule TE buffer di tethering nella cella di flusso.
- Incubare 0,5 o 1,5 micron Area di perle di lattice non magnetici nella cella di flusso per ~ 30 min. Queste perle agiranno come perline di riferimento durante le pinzette magnetiche misure che permettono di ridurre al minimo l'effetto di deriva tra l'obiettivo e il supporto del campione (ovvero la cella di flusso). Lavare perle non magnetici senza legami con il risciacquo con 2-3 volumi cellulari di tampone tethering TE.
- Funzionalizzare la superficie inferiore della cella di flusso mediante incubazione con 100 pg / ml di anti-digossigenina in PBS per almeno 1 ora (preferibilmente maggiore; incubazione può essere effettuata durante la notte), prevedere attaccamento DNA. Risciacquare con 2-3 volumi cellulari di tampone tethering TE. Infine incubare la cella di flusso con 2 mg / ml di sieroalbumina bovina (BSA) in tampone TE tethering per 30 min per passivazione della superficie.
- Prelevare un 'aliquota di2 ml perline MYONE superparamagnetiche rivestite di streptavidina (vedi la discussione e la tabella dei materiali) e diluire con 10 ml di TE buffer di tethering. Lavare due volte con 10 ml di TE buffer di tethering utilizzando un concentratore di particelle magnetiche, e risospendere in 10 ml TE buffer di tethering. Attaccare ~ 1 ml delle molecole di DNA (circa 1 ng) a queste perle mediante incubazione in tampone TE tethering per 30 min.
- Diluire la soluzione del DNA-tethered perle superparamagnetiche dieci volte con l'aggiunta di 90 ml di tampone tethering TE. Infine, iniettare la soluzione nella cella di flusso e incubare per ~ 1 ora per consentire fissaggio DNA alla superficie anti-digossigenina-rivestita. Lavare accuratamente la cella di flusso con tampone tethering TE. Dopo l'incubazione dei costrutti di DNA-tether, risciacquare a lungo con tampone sperimentale (questo può essere tampone tethering TE) per rimuovere tutte le perline non iscritti.
- Per le misure che utilizzano un protocollo di monitoraggio angolare che richiede fiduciali perline marcatori attaccati alle sfere magnetiche 23 (vedi la discussione), incubare la cella di flusso con 1.000 volte magazzino diluita di perline marcatori nel buffer tethering TE per almeno 30 minuti e risciacquare con il tampone di fondo.
2. Misure su una singola molecola di DNA nelle pinzette magnetici convenzionali
- Utilizzando una MT convenzionale (vedi Discussione) con configurazione di campo appropriato (Figura 1a) e sia traslazionale e rotazionale controllo della posizione del magnete, verificare la molecole di DNA rotazione vincolati nella cella di flusso. Alle forze di trazione ≥ 1 pN (consultare i riferimenti 4, 19, 20, 28, 29 per la taratura in forza pinzette magnetiche), perline frenati possono essere facilmente distinguibili da perline attaccate alla superficie del vetrino fondo dalle loro diverse altezze nel fuoco . Se una molecola di DNA è rotazionalmente vincolato può essere valutata introducendo 20-30 turnos dei magneti a una forza di ≈ 0,25 pN: qui, la lunghezza cavezza dovrebbe ridursi di 0,4-0,5 micron.
Nota: Per eseguire pinzette magnetiche esperimenti, elaborazione delle immagini viene utilizzato per determinare la x, y, z e la posizione di DNA-tethered perline. LabVIEW personalizzato per questo scopo è disponibile su richiesta degli autori.- Verificare che il tallone è attaccato da un singolo cavezza DNA. Questo può essere fatto confrontando il comportamento sotto giri positivo e negativo forze> 1 pN (Figura 2a). In questo regime forza, la presenza di più attacchi DNA darà luogo ad una diminuzione di circa simmetrica nell'estensione upon introducendo turni positivi e negativi, mentre singoli attacchi DNA daranno luogo ad una risposta asimmetrica.
- Ricerca di appropriate perline fissi attaccato alla superficie inferiore in prossimità del tether di interesse che può servire come perline di riferimento.
- Calibrare la lunghezza di tegli DNA, l. La posizione della superficie cella a flusso può essere determinata portando il tallone legato in contatto con la superficie (ad esempio, ruotando il magnete ~ 60 giri con una forza inferiore allo 0,2 pN). Misure di posizione verticale del branello legato rispetto a questa superficie poi riferire in valore assoluto di l.
Nota: Per ridurre al minimo gli effetti successivi di deriva, si consiglia di effettuare misurazioni di L rispetto alla posizione di un cordone di riferimento apposte sulla superficie. - Registrare una curva di rotazione (cioè una misura dell'estensione DNA in funzione del numero di giri) in una forza di stiramento di ≈ 0,25 pN (Figura 2a).
- Determinare il numero di giri a cui l'estensione è massima, in quanto questo corrisponde allo stato in cui la molecola di DNA è torsionalmente rilassato. Per fare ciò, è utile per adattare la curva di rotazione localmente con un parabolico o una funzione Gaussiana per determinare il centro position. Definire questo punto come "zero giri".
Nota: Una routine-custom scritto per questo scopo è disponibile su richiesta degli autori.
- Determinare il numero di giri a cui l'estensione è massima, in quanto questo corrisponde allo stato in cui la molecola di DNA è torsionalmente rilassato. Per fare ciò, è utile per adattare la curva di rotazione localmente con un parabolico o una funzione Gaussiana per determinare il centro position. Definire questo punto come "zero giri".
- Per una serie di ~ 20 posizioni del magnete, determinare l'estensione media della molecola torsionalmente-rilassato (cioè a "zero giri", vedere il punto 2.4.1) dalla z-traccia.
- Per ciascun punto di misurazione nel passaggio 2.5, determinare precisamente la forza che si estende dalle fluttuazioni del xoy posizione 20, 28, 29, o, purché la magnetizzazione del tallone è ben noto, utilizzando la conoscenza del gradiente di campo locale 4. Tracciare la forza di allungamento rispetto ai risultati medi di estensione in una curva forza-estensione (Figura 2b).
- Montare i dati risultanti forza di estensione per l'equazione catena vermiforme usando l'approssimazione polinomiale da Bouchiat et al 30.
- Se la preparazione per le successive misurazioni FOMT, ruotare lentamente i magneti durante la registrazione escursioni del tallone magnetico in (x, y).
Nota: minore è il raggio dell'anello conseguente configurazione MT convenzionale, il più strettamente la molecola di DNA è legato vicino al "polo sud" della perlina magnetica. Quando si passa alla configurazione FOMT, tale molecola di DNA verrà legato strettamente alla "equatore" del branello magnetico, che permette la tracciabilità efficace dell'angolo di rotazione dalla (x, y)-posizione (vedi Discussione).
3. Misure di Twist DNA utilizzando le Liberamente-orbitanti pinzette magnetiche
- Sostituire manualmente i magneti quadrati delle pinzette magnetici convenzionali da un magnete cilindrico utilizzato per FOMT (Figura 1b). Questa operazione deve essere eseguita in modo tale che il tether DNA selezionato rimane all'interno del campo di vista.
- Ciò può essere eseguito in meno di 1 min semplicemente svitando la testa del magnete completo che contiene i magneti per la configurazione pinzette convenzionali e sua sostituzione con un magnete testa che contiene un magnete cilindrico per FOMT.
- Le escursioni a (x, y) di un cordone magnetico legato da un unico tether dsDNA dipendono fortemente dalla posizione del tether rispetto all'asse del magnete cilindrico (Figura 1b, Figura 3a). Registrare le escursioni (x, y) per determinare la posizione corrispondente nel modello caratteristico di fluttuazione (figura 3a, Discussione).
- Eseguire l'allineamento grossolano del magnete nel FOMT. Ciò può essere ottenuto spostando il magnete cilindrico sopra la cella di flusso utilizzando (x, y) Ciclo di traduzione. Se le escursioni (x, y) seguono un arco, il magnete cilindrico non è correttamente allineato e deve essere spostatonella direzione appropriata (Figura 3b).
- Allineamento Coarse può essere realizzato entro 15 minuti per il caso di perline MYONE con 7.9 pastoie KBP, ed è completa quando la misurazione delle (x, y) escursioni comporta l'osservazione del moto circolare (figura 3b, al centro).
Nota: l'allineamento di massima è in genere sufficiente per osservare i cambiamenti di torsione causate da proteine di legame alle singole DNA impastoiati nella configurazione FOMT 21, 31 (Rappresentante dei risultati, Figura 5), nonostante il fatto che la accompagna istogramma bidimensionale non può avere i suoi conti assolutamente distribuiti uniformemente lungo l'anello circolare (figura 3c).
- Allineamento Coarse può essere realizzato entro 15 minuti per il caso di perline MYONE con 7.9 pastoie KBP, ed è completa quando la misurazione delle (x, y) escursioni comporta l'osservazione del moto circolare (figura 3b, al centro).
- Se necessario, per ulteriori esperimenti, eseguire l'allineamento bene in FOMT. Ciò può essere ottenuto utilizzando viti micrometriche ad alta risoluzione oppure una fase automatizzata ad alta risoluzione o spostare il magnete o la cella di flusso per centoer il magnete cilindrico sul tallone entro ~ 10 micron. Nella fase di allineamento fine, il magnete è posizionato con attenzione in modo tale che le fluttuazioni del annulus cerchio sono quasi uniforme, corrispondente ad una situazione in cui la barriera energetica alla rotazione integralmente a causa del magnete è k B T (Figura 4).
Nota: Uno script MATLAB per tracciare le fluttuazioni in un istogramma o termogramma come in figura 4 è disponibile su richiesta degli autori.
Nota: Fine di allineamento può essere eseguita entro 45 min per il caso di perline MYONE con 7.9 pastoie KBP, e in tempi ridotti per perline più piccole e attacchi più brevi sono impiegati (vedi discussione).
Nota: Fine di allineamento è in genere necessario per eseguire misure della rigidità torsionale del DNA nudo o di proteine rivestite (Rappresentante Risultati, Figura 4). - Se necessario per l'analisi, calibrare la forza della FOMT. Questo può essere effettuato ina maniera analoga a MT, utilizzando sia oscillazioni radiali del tallone 2> (dove le parentesi angolari indicano la media temporale) come mostrato nel video che accompagna e dettagliata in Lipfert et al 21, o, purché la magnetizzazione del tallone è ben noto, utilizzando la conoscenza del gradiente di campo locale 21.
4. Misure di coppia DNA con le pinze Torque magnetici
- Sostituire manualmente il magnete cilindrico utilizzato per FOMT da un magnete cilindrico e un lato (permanente) magnete per la MTT (figura 1c). Questa operazione deve essere eseguita in modo tale che il tether DNA selezionato rimane all'interno del campo di vista.
- Il modo più semplice per ottenere questo risultato è necessario aggiungere manualmente il magnete lato alla sua posizione corretta, che può essere effettuata entro 1 min. Nessun ulteriore riallineamento è necessario.
Nota: Un'alternativa a un magnete laterale è l'usodi elettromagneti 32.
- Il modo più semplice per ottenere questo risultato è necessario aggiungere manualmente il magnete lato alla sua posizione corretta, che può essere effettuata entro 1 min. Nessun ulteriore riallineamento è necessario.
- Se necessario per l'analisi, calibrare la forza in un modo analogo a MT, utilizzando sia x del tallone o fluttuazioni y o, a condizione che la magnetizzazione del tallone è ben noto, utilizzando la conoscenza del gradiente di campo locale 21.
- Monitorare le oscillazioni angolari in funzione di θ tempo (t) utilizzando il protocollo fiduciali basato inseguimento 23 o, come mostrato nel video che accompagna, il protocollo di inseguimento angolare basato sul controllo (x, y)-posizione (vedi Discussione). Nel primo caso, registrare immagini complete del tallone in funzione del tempo per l'elaborazione delle immagini successive. In quest'ultimo caso, è sufficiente registrare (x, y) fluttuazioni del tallone in questa fase.
Nota: Uno script MATLAB per determinare θ (t) a partire da immagini complete del tallone in funzione del tempo nel protocollo inseguimento fiduciali basato è unvailable dagli autori su richiesta.- Come descritto nella discussione, per il protocollo inseguimento angolare basato sul controllo (x, y)-posizione è anche consigliabile registrare una traccia di tempo in cui i magneti sono lentamente (tipicamente a 0.1 Hz) ruotato di alcuni giri. Questo permetterà uno a convertire con precisione le coordinate cartesiane (x, y) in coordinate polari (r, θ) utilizzando le equazioni 3-5 della discussione.
Nota: Uno script MATLAB per script di monitoraggio angolare basato sul controllo (x, y)-position è disponibile su richiesta degli autori.
Nota: Il tempo di misura dipende in gran parte dalla risoluzione coppia desiderata. Un argomento dettagliata è riportata nella Lipfert et al 24. Per perline MYONE e 8 KBP attacchi del DNA, di misura per 30-100 secondi dovrebbero essere sufficienti per fornire una risoluzione di coppia nella gamma di ~ 1 pN · nm.
- Come descritto nella discussione, per il protocollo inseguimento angolare basato sul controllo (x, y)-posizione è anche consigliabile registrare una traccia di tempo in cui i magneti sono lentamente (tipicamente a 0.1 Hz) ruotato di alcuni giri. Questo permetterà uno a convertire con precisione le coordinate cartesiane (x, y) in coordinate polari (r, θ) utilizzando le equazioni 3-5 della discussione.
- Determinare la rigidezza torsionale della trappola da the varianza delle fluttuazioni angolari (σ θ 2, in radianti) utilizzando:
k θ = k B T / σ θ 2 (1)
Nota: Tipici rigidezze trappola rotazionale conseguiti nella MTT sono nell'intervallo 10-1,000 pN · nm / rad, inferiore per pinzette magnetici convenzionali. - Inoltre, registrare la z-posizione del cordone e utilizzare questo per determinare la lunghezza cavezza l (vedi anche passi 2,4-2,7).
- Ruota N gira e di nuovo registrare θ (t) e l (t).
Nota: Il ridotta rotazione trappola rigidità del MTT rispetto a MT rende idoneo per misure di coppia singola molecola, ma implica che la coppia massima che può essere esercitata è diminuito. Ciò implica che il MTT non può essere in grado di controbilanciare coppie elevate trascinamento causati dalla rotazione rapida. Si deve perciò essere presa per non superare la velocità massima; typically girare a tassi vicini a 0,1 Hz. - Determinare la coppia accumulato nel tether acido nucleico dopo N spire utilizzando:
Γ = - k θ <θ N - θ 0> (2)
Dove <...> indica la media e θ 0 e θ N sono l'angolo a zero giri (corrispondente a una pastoia torsione rilassato, cfr. Passo 2.3 e N si trasforma, rispettivamente. - Ripetere i punti 4.5 e 4.6, se necessario per determinare pienamente risposta della coppia di una molecola in un singolo ciclo di misura (Risultati rappresentativi, Figura 6).
Risultati
Risultati rappresentativi della MT (Figura 1a) sono mostrati nella Figura 2. Figura 2a mostra curve di rotazione-estensione di 7,9 kb di DNA scattata a F = 0,25, 0,5, e 2,0 pN. La risposta di un singolo DNA di rotazione deve essere simmetrica a forze più basse (0,25 Pn), con l'estensione del DNA diminuire a seguito della formazione di supercoils plectonemic positivi o negativi. Conoscenza qualitativa di questa risposta è utile quando si cerca inizialmente per una pastoia DNA rotazione vincolata (punto 2.1). Si noti che un controllo supplementare della cavezza è tenuto a verificare che consiste di una singola molecola di DNA: qui, la risposta asimmetrica di un singolo DNA a rotazione a forze superiori a 0,5 pN aiuta a distinguerlo da più DNA (fase 2.1.1). Una volta che questo è stato verificato, si ritorna alla risposta rotazionale a 0.25 pN per determinare il numero esatto di magnete gira in cui il DNA a singolo is torsionally rilassato, dove si prende una curva forza-estensione, che dovrebbe assomigliare alla Figura 2 b. Per questa particolare misura, un adattamento dei dati al modello della catena vermiforme (linea continua), ha prodotto un profilo aderente lunghezza L C = 2.71 micron e piegatura lunghezza di persistenza L P = 45 nm. Per dsDNA, i valori stimati della lunghezza di persistenza dovrebbe essere compresa nell'intervallo 40-55 nm, a seconda delle condizioni del tampone 33, e la lunghezza del profilo adattata devono essere vicini (tipicamente entro 10%) al valore atteso per il costrutto di DNA che viene utilizzato nelle misurazioni, utilizzando la relazione L DNA = 0,34 nm / bp · numero di coppie di basi.
La figura 3 mostra le procedure e risultati di allineamento nel FOMT (Figura 1b). I (x, y) escursioni iniziali registrate nella fase 3.2 può essere paragonato alla visione d'insieme delle fluttuazioni in funzione of la posizione del magnete trasversale mostrata in figura 3a, che mostra un modello "vortice" che può essere usato per guidare conseguente spostamento relativo tra il magnete e il tallone DNA-legato tenuto in FOMT. Quando successivo posizionamento grossolano è completa, del tallone (x, y)-fluttuazioni tracciano una traiettoria circolare, come è dimostrato anche dalla traccia nera nella figura 3b. A questo punto, la coppia di magneti circa l'asse z è ridotto al punto che le fluttuazioni termiche sufficienti per ruotare il cordone attorno al suo punto di attacco. Il cerchio di raggio R dell'anello circolare risultante (cerchio montato è mostrato in rosso) rappresenta la distanza radiale tra il punto di attacco DNA e il centro del tallone (Figura 1b). Come mostrato in figura 3c, tuttavia, un istogramma dei dati della Figura 3b mostra che il posizionamento grossolano non garantisce una copertura uniformedi tutte le possibili posizioni lungo l'anello circolare. Anche se le fluttuazioni termiche sono sufficienti per esplorare tutti angolo rotazioni sul cerchio, rimane una piccola barriera di energia (dell'ordine di energia termica k B T) a rotazione libera.
Quando allineamento più fine avviene nel FOMT (punto 3.4), lo strumento può essere utilizzato per determinare il modulo torsionale del DNA (Figura 4). In primo luogo, allineamento fine del campione viene utilizzato per ottenere movimento circolare (figura 4a), il cui istogramma bidimensionale dovrebbe ora mostrare una copertura uniforme (Fig. 4b). La corrispondente q traccia temporale (t) delle oscillazioni angolari (ottenuti dalla conversione del (x, y)-posizioni, vedi sotto) non mostra alcuna periodicità corrispondente a 360 ˚ (Fig. 4c) e rivela le grandi escursioni corrispondenti a diversi giri completi (Figura 4d). Il panorama energetico implicitaè armonico in un intervallo di> 1.000 ˚ (Figura 4e). La deviazione standard delle fluttuazioni è σ θ = 223 °, corrispondente ad una trappola rigidezza angolare θ k = k B T / σ θ 2 = 0,27 · pN nm / rad, che a sua volta fornisce una stima della lunghezza effettiva persistenza torsionale di DNA pari a C = L C / σ θ 2 ~ 76 nm (L C = 1.150 nm per il DNA 3.4 kbp utilizzato in questa misura) alla forza misurata.
Un esempio di come FOMT può essere utilizzato per misurare la variazione di torsione indotta nella molecola di DNA legato attraverso il legame delle proteine 31, 34 è illustrato nella Figura 5. Qui, abbiamo monitorato il legame della proteina RAD51 raddoppiareFilamento di DNA; RAD51 è noto sia per allungare e distendersi DNA come si forma un filamento nucleoprotein 31. Al lavaggio RAD51 nella cella di flusso, si osserva che il tallone subisce una traiettoria a spirale nella FOMT (Figura 5a). Convertendo traccia di (x, y) di movimento in funzione del tempo per q (t) come descritto sopra, possiamo co-plot l'effetto che RAD51 ha sulla lunghezza tether DNA e del grado di svolgimento (Figura 5b, c) .
Un approccio alternativo per misurare le proprietà torsionali del DNA sono la MTT (Figura 1c, Figura 6). Lo schema in Figura 6a illustra il principio della misurazione: dopo overwinding (o underwinding) il tether DNA da N spire, il DNA esercita una coppia ripristino sul tallone che porta ad uno spostamento nella posizione angolare di equilibrio θ da 0 a θ N. Nella MTT componente trasversale di campo magnetico è ridotto rispetto al MT, che facilita la valutazione di tali spostamenti angolari pur consentendo la rotazione del branello (Figura 1). L'ampiezza dello spostamento angolare misurato dopo l'applicazione N = 45 si rivolge a un DNA 7,9 kbp è illustrato nella Figura 6b. La sequenza completa del protocollo misurazione MTT e l'esito risultante di una coppia contro curva di rotazione per DNA sono mostrati in Figura 6c-f. Qui, misure della deviazione standard (figura 6c) e la (Figura 6d) medio della coordinata angolare sono visualizzate in funzione di sovra e underwinding, con la deviazione standard essendo inversamente proporzionale alla rigidezza trappola angolare (Equazione 1). Presi insieme, questi quantitativi permettono di costruire una coppia contro la curva di rotazione per il DNA (Figura 6f), che dovrebbe mostrare una regione risposta lineare centrata su 0 giri unnd due plateau in cui satura di coppia, a rotazioni positivi e negativi rispettivamente. Tale coppia contro curva rotazione integra le informazioni in un interno contro curva di rotazione (figura 6e), quantificando così le transizioni che accompagnano l'instabilità e la denaturazione del DNA.
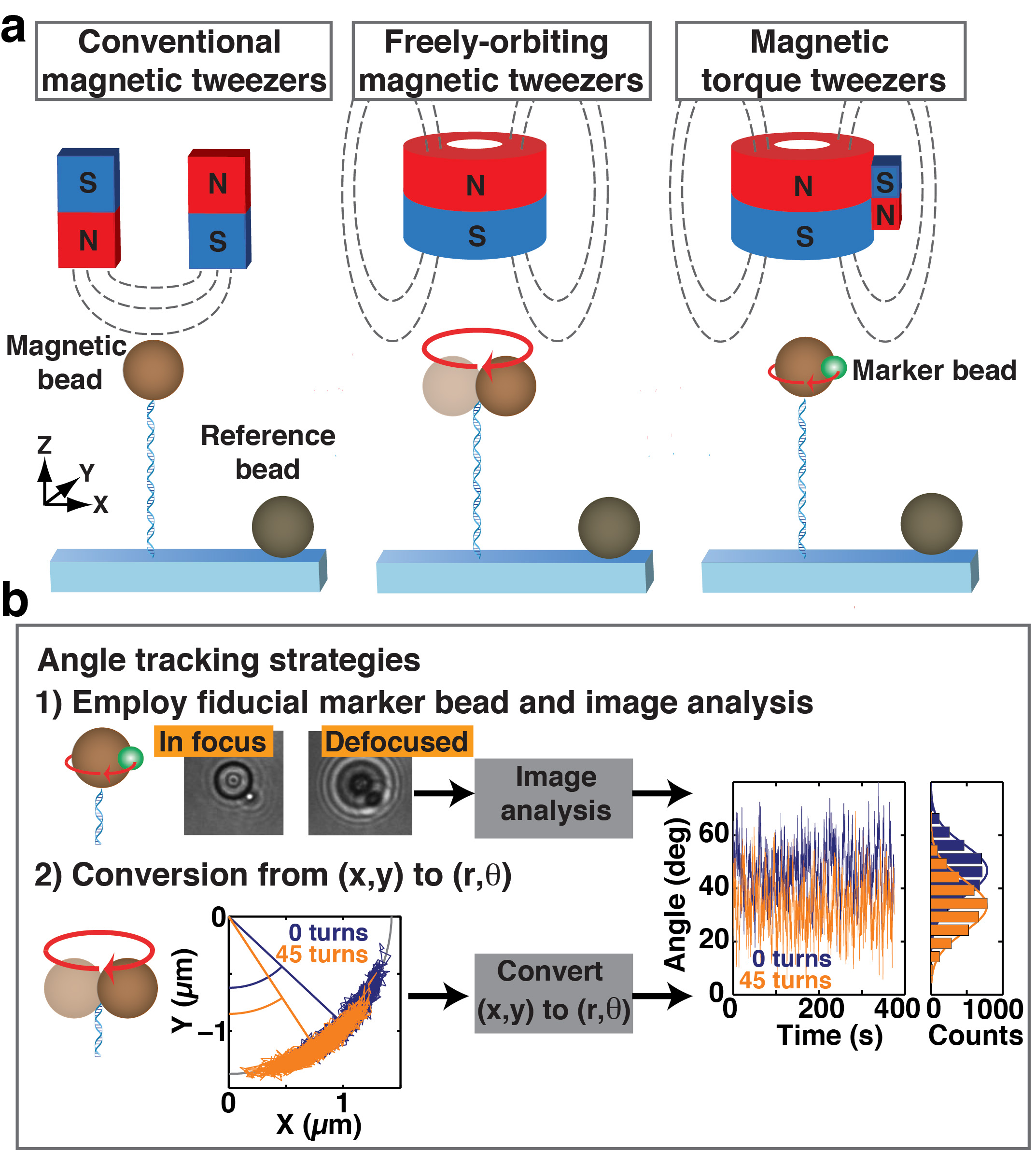
Figura 1. Schema di pinzette magnetici convenzionali (MT), liberamente orbitanti pinzette magnetiche (FOMT), pinzette coppia magnetica (MTT), e le due strategie per il monitoraggio angolo di rotazione. (A) In tutte e tre le implementazioni di pinzette magnetiche, biglie magnetiche sono tenuti legati ad una superficie della cella di flusso da macromolecole funzionalizzati, per esempio, le molecole di DNA a doppia elica mostrate schematicamente. Perline di riferimento sono attaccati alla superficie delle cellule di flusso e monitorati per DRIFt correzione. Tutti e tre MT allestimenti impiegano magneti per applicare una forza che si estende verso l'alto sul tallone magnetico e, quindi, tether DNA. In MT convenzionale, una coppia di magneti esercita un campo magnetico che è orientato trasversalmente rispetto all'asse tether, strettamente vincolare la rotazione del tallone attorno all'asse DNA-tether. In FOMT, un magnete di forma cilindrica fornisce un campo magnetico che orientata lungo la direzione tether. Quando il tether è allineato al centro del magnete di forma cilindrica, eventuali campi trasversali rimanenti sono minimizzati, consentendo la libera rotazione attorno all'asse tether In MTT, un magnete laterale viene aggiunto al magnete a forma cilindrica utilizzato in FOMT per fornire un piccolo campo trasversale (ridotto in grandezza rispetto a MT). Questo piccolo campo trasversale consente l'applicazione di coppia e la sua misurazione. (B) due strategie per misurare l'angolo di rotazione di una perlina magnetica intorno all'asse DNA-tether sono mostrate. 1): un cordone marcatore (green) attaccata al cordone magnetico (marrone) dà un'immagine asimmetrica che consente angolo inseguimento da immaginare analisi. Due immagini CCD di una perla magnetica 1.4-micron-radius con un 0,5 micron raggio di servirlo fiduciale sono mostrati, a fuoco e out-of-focus. 2): quando il DNA è legato alla perlina magnetica in una posizione lontana dal polo sud del tallone, il centro del tallone oscilla lungo un arco il cui centro definisce una posizione angolare. Ciascuna strategia può essere utilizzato per monitorare l'angolo di rotazione e per monitorare spostamenti nella posizione angolare come la cordicella è torsionalmente tesa (tracce a destra), permettendo così misurazioni di coppia singola molecola.

Misurazioni di calibrazione DNA Figura 2. Nel MT tradizionale. (A) Rotazione-estensione curve per un 7,9 kb di DNA scattata a F = 0.25, 0.5, e 2.0 pN. La risposta asimmetrica in rotazione a turni positivi e negativi delle singole attacchi DNA a doppia elica può essere utilizzato come una comoda prova dell'attacco tether. (B) Curva forza-estensione di 7,9 kb di DNA, insieme ad una forma di verme- come il modello di catena (linea continua), ottenendo un profilo aderente lunghezza L C = 2.71 micron e piegando la persistenza lunghezza L P = 45 nm. Tutte le misurazioni sono state effettuate in tampone PBS.
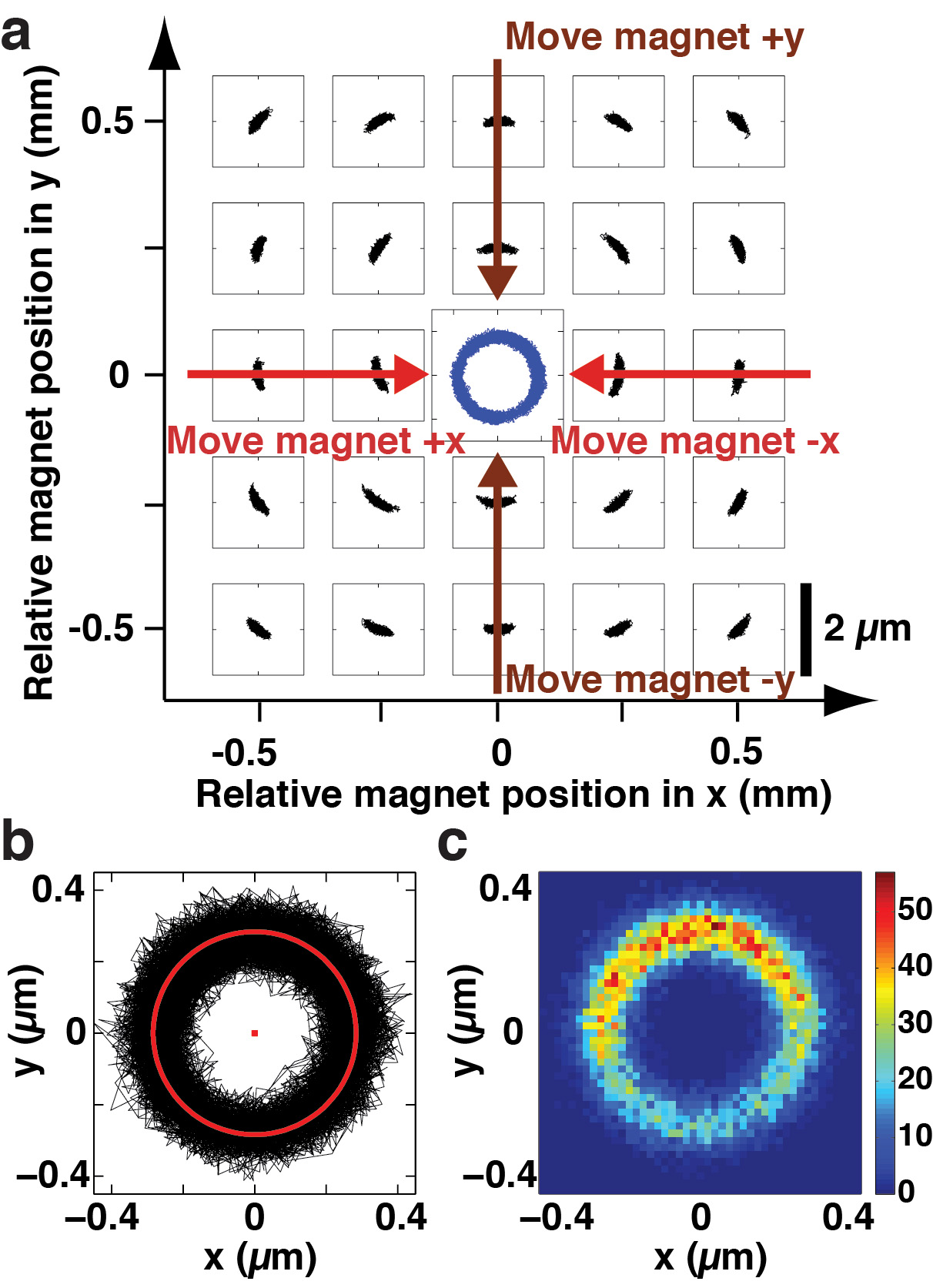
Figura 3. Allineamento in FOMT. (A) (x, y) fluttuazioni del branello DNA-legato organizzati nel FOMT in funzione della posizione del magnete. La posizione del magnete cilindrico stato scansionato ad un'altezza costante di 3 mm, la superficie della cella di flusso a passi di 250 micron di x e Y ed è indicato sugli assi trama esterni. Ad ogni (x, y)-posizione del magnete, fluttuazioni dello stesso tallone DNA-legati sono stati registrati e sono riportati nei piccoli sistemi di coordinate (barra della scala in basso a destra si applica a tutti i sistemi sub-coordinate). Variazioni sistematiche del tallone (x, y) modello-oscillazione con la posizione del magnete simile a un ciclone o vortice sono evidenti. Questo pattern "vortice" può essere usato per guidare lo spostamento del magnete (o in alternativa la cordicella mantenendo il magnete fisso) in x e y (indicato dalle frecce grandi) per ottenere l'allineamento. Quando il posizionamento grossolano è completa, del tallone (x, y)-fluttuazioni tracciano una traiettoria circolare (traccia blu al centro della trama). Questa traccia è stata registrata in un esperimento separato dopo aver allineato i magneti piccoli passi per il centro ed è evidenziato per l'illustrazione in questa trama. (B) (x, y)-fluttuazioni di un cordone di DNA-tethered tenuto in tegli FOMT dopo successo grossolana-allineamento del magnete (traccia nera). Le fluttuazioni giacciono su un anello circolare e fluttuazioni termiche sono sufficienti per esplorare tutte le rotazioni gli angoli sul cerchio. Un cerchio montato è mostrato in rosso. (C) Un istogramma corrispondente ai dati in (b), mostra che il posizionamento grossolano non garantisce una copertura uniforme di tutte le possibili posizioni lungo l'anello circolare. Anche se le fluttuazioni termiche sono sufficienti per esplorare tutti angolo rotazioni sul cerchio, rimane una barriera energetica (sul dell'ordine della energia termica k B T) a rotazione libera.
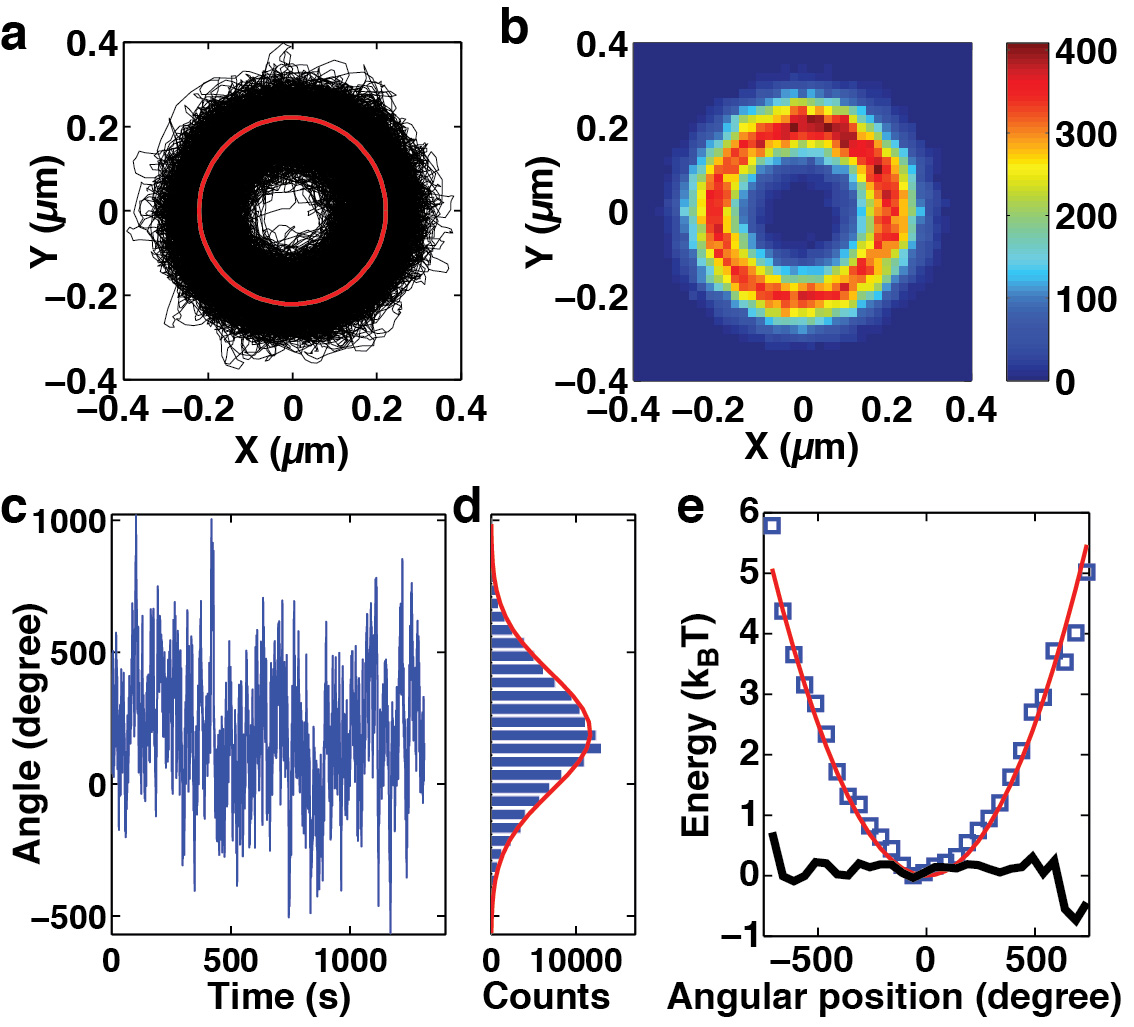
Figura 4. Misurazione di DNA rigidezza torsionale utilizzando FOMT. (X, y)-traiettoria (a) e istogramma (b) di una DNA-TethEred fluttuazioni del tallone dopo l'allineamento di precisione della posizione del magnete-tether relativa nel FOMT. In queste circostanze, l'istogramma rivela una copertura sostanzialmente uniforme delle posizioni sul cerchio. (C) fluttuazioni di rotazione del tallone determinata dalla (x, y)-posizioni. (D) Istogramma delle fluttuazioni rotazionali. La linea rossa è una misura gaussiana con σ ° θ = 223. (E) Il contesto energetico implicita la densità fluttuazione di rotazione da (c) e (d). La differenza tra il panorama energetico implicita fluttuazioni rotazionali e una approssimazione armonica (con k θ = k B T / σ θ 2 = 0.27 pN-nm/rad) è molto più piccola della energia termica k B T su più turni. I dati sono compensate per chiarezza tale che θ 0 = 0. La larghezza dile fluttuazioni possono essere utilizzati per determinare la rigidità torsionale del DNA, cfr. testo. La misurazione è stata effettuata in tampone PBS a una forza di stiramento di ~ 1 pN. I dati sono adattati da Lipfert et al 21.

Figura 5. L'associazione della proteina RAD51 di DNA misurata utilizzando FOMT. (A) Assemblea dei proteina RAD51 su un tethered 7,9 kbp dsDNA monitorato a 3,5 pN. La (x, y, z)-traiettoria eseguita dal tallone magnetico (diametro 1,0 millimetri) durante il primo 200 sec del gruppo si onda con codice colore dal blu al rosso. (B) L'estensione del dsDNA dedotta dalla componente z della traiettoria tallone in (a) in funzione del tempo. (c) L'angolo di rotazione attorno all'asse tether dsDNA dedottedalla x, y componenti della traiettoria tallone in (a) in funzione del tempo.
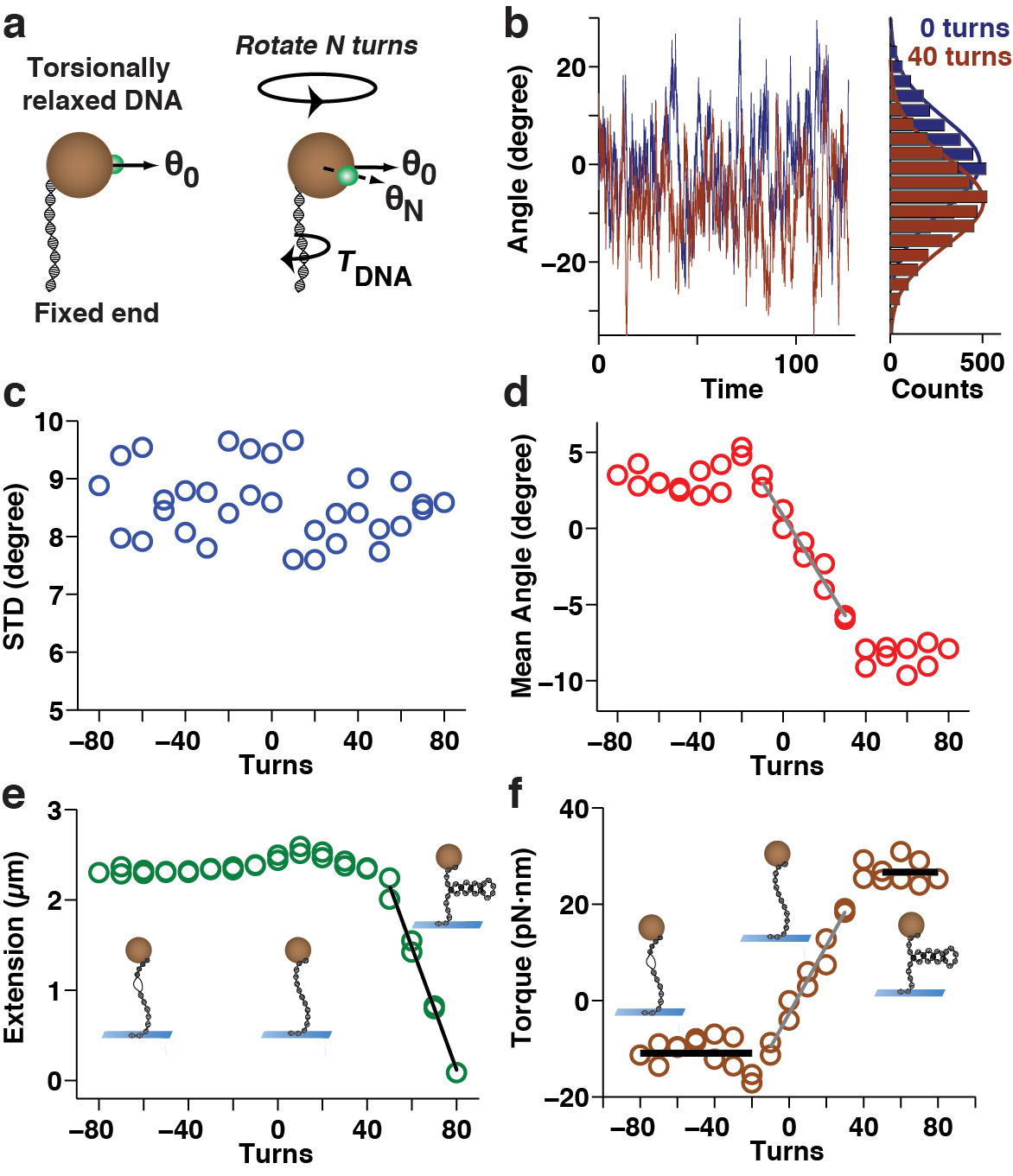
Misurazioni della coppia Figura 6. Su un singolo tether DNA nella MTT. (A) schematica che mostra il principio della misurazione della coppia. Dopo oltre-(o sotto-) avvolgimento del tether DNA da N spire, il DNA esercita una coppia ripristino sul tallone che porta ad uno spostamento nella posizione angolare di equilibrio θ da 0 a N θ. (B) Esempio di tracce angolari utilizzati per misurare la coppia:. oscillazioni angolari di un cordone legato ad una torsione rilassato molecola di 7.9 kbp DNA prima (blu) e dopo l'introduzione di 40 giri (rosso scuro) (cf) misurazione della coppia su una molecola di DNA 7,9 kbp in tampone PBS presso una stvigore del ~ 3 PN conati di vomito con il tallone marcatore fiduciali basato su protocollo di monitoraggio angolare. Oscillazioni angolari come mostrato in (b) sono stati registrati in funzione del numero di giri applicati. (C) La deviazione standard delle oscillazioni angolari in funzione di giri applicati. La larghezza delle fluttuazioni è approssimativamente costante, indicando costante trappola angolare rigidità. (D) Lo spostamento dell'angolo di rotazione medio in funzione di giri applicati. Errori sistematici dell'angolo medio su over-e underwinding sono evidenti. (E) Il DNA estensione tether monitorati contemporaneamente in funzione di giri applicati. (F) La coppia esercitata dal tether DNA determinata dall'angolo medio mostrato in (d) , vedi testo principale. Over-e underwinding circa zero attiverà dà luogo ad una coppia lineare vs giri risposta del DNA-tether (piste grigi montati ion (d) e (f)) che può essere utilizzata per determinare la lunghezza effettiva persistenza torsionale (~ 77 nm per questo insieme di dati). Ulteriori overwinding porta a instabilità e formazione di supercoils plectonemic (schematicamente illustrati nella inserti), corrispondente ad un plateau di coppia (linea nera a svolte positive in (f) a ~ 26 pN · nm) e una diminuzione lineare dell'estensione tether con numero di giri (pista nera in (e)). Svolgitore là regime lineare provoca il DNA a fondere localmente (mostrato nella inserti a sinistra), caratterizzato da un plateau coppia uguale alla coppia di fusione (linea nera diventa negativo in (f) a -11 ~ pN · nm).
Discussione
Quando si esegue esperimenti utilizzando il MTT o FOMT, una serie di scelte devono essere fatte per quanto riguarda perline, magneti, protocolli di monitoraggio, ecc Le scelte migliori da compiere dipenderà l'esperimento di interesse. Qui di seguito descriviamo i compromessi che accompagnano scelte diverse, che dovrebbe agevolare la selezione di un particolare esperimento. Avanti, descriviamo diversi passaggi critici che accompagnano l'allineamento e la gestione di MTT e FOMT esperimenti. Infine, discuteremo il significato della MTT e FOMT rispetto ai metodi esistenti così come le applicazioni future.
Considerazioni Prima dell'inizio di MTT e FOMT esperimenti
Ogni esperimento richiede di selezionare un tipo di perlina magnetica per l'utilizzo. Si può scegliere tra diversi perline disponibili in commercio sono rivestite di streptavidina superparamagnetiche, ad esempio, 0,25 micron perle raggio, 0,5 micron perline raggio, o 1,4 micron perline raggio (eee la tabella dei materiali). Perle più grandi avranno un maggiore momento magnetico rispetto alle perle più piccole (all'incirca scala come volume) e quindi il loro utilizzo faciliteranno l'applicazione di forze maggiori (per le forze tipici ottenuti nei nostri strumenti, vedi Tabella 1). Quando si desidera inseguimento angolare con perline marcatori, noi di solito lavoriamo con 1,4 micron di raggio e l'uso di 0,5 micron raggio perline biotinilati non magnetici come perle marcatori (cfr. paragrafo 1.9 per il protocollo allegato corrispondente). L'uso di perline più piccole è particolarmente indicato per la FOMT, come i tempi caratteristici per la rotazione tallone τ C è uguale al rapporto di resistenza del sistema rispetto al suo costante elastica γ / k θ; soprattutto, il coefficiente di resistenza rotazionale rilevante ai angolari scala scale di tempo di misura da ~ R tallone 3, cioè con la terza potenza del raggio (vedi Tabella 2 perscale il tempo caratteristico per diverse combinazioni bead-DNA in FOMT e misure MTT). Riduzioni accompagnamento della forza massima che può essere applicata possono essere affrontate mediante una pila capovolto di magneti cilindrici 27. Tuttavia, nelle misurazioni FOMT volte può essere necessario un compromesso tra la migliore risoluzione temporale realizzabile e la forza massima esercitata.
Inoltre, un esperimento richiede la selezione di una configurazione di magnete. Nel tradizionale configurazione pinzette magnetiche (Figura 1a), si utilizzano in genere un paio di 5x5x5 mm magneti cubi in orientamento verticale con un gap di 0,5 o 1 mm tra i magneti 4. Quando i magneti sono distanziati lungo l'asse x (y), questo produce un campo magnetico che è diretto principalmente lungo l'asse x (y). Per gli esperimenti FOMT, un magnete di forma cilindrica è selezionato al centro della quale il campo magnetico è principalmente direttolungo l'asse z (Figura 1b). In pratica, si usa una pila di tre di tali magneti a forma cilindrica, ciascuna con un diametro di 6 mm e un foro centrale 2 mm di diametro, per uno spessore complessivo di 6 mm. Quando le forze di trazione più elevati sono desiderati, una configurazione magnete "pila capovolto" nel quale il magnete inferiore è raggruppato con magnetizzazione opposta è preferito. Per realizzare la configurazione MTT (Figura 1c), si aggiunge un magnete addizionale al lato della pila magnete principale della configurazione FOMT, tipicamente un cilindro con diametro di 4 mm e un'altezza di 7 mm. Per vedere come le forze massimi raggiunti nei nostri strumenti dipendono dalla configurazione magnete, vedere Tabella 1.
L'allineamento di MTT e FOMT esperimenti
Poiché biglie magnetiche hanno una superficie (circa) uniformemente funzionalizzato (tipicamente streptavidina) e poiché l'attaccamento sia del n funzionalizzatoattacchi acidi ucleic e perline marcatori (nel caso in cui è impiegato il marcatore a base tallone-tracking angolare) avviene tramite semplice incubazione in soluzione, uno non controlla dove la cavezza e / o marcatore tallone attribuiscono al tallone magnetico. Le biglie magnetiche hanno un asse di magnetizzazione preferito che tende ad allinearsi lungo la direzione del campo esterno. Se indichiamo i punti in cui l'asse di magnetizzazione preferito interseca la superficie del branello come i poli nord e sud, poi perline dove il DNA-tether è attaccato vicino all'equatore traccerà un anello circolare con un raggio vicino o leggermente più grande raggio tallone in FOMT; al contrario, perle che sono attaccati vicino al polo sud oscilleranno su un anello circolare con raggio molto piccolo in FOMT, che può precludere il montaggio del cerchio utilizzando Equazioni 3-5. Notiamo che per semplice geometria sferica, la probabilità di attaccare vicino all'equatore è molto più grande di un allegato esattamente ai poli; Pertanto, la maggior parte bpotranno essere legati eads tale che l'inseguimento angolare (x, y)-based può essere effettuata con successo.
Un discorso simile vale per il fissaggio delle perline marcatore per il marcatore sulla base inseguimento angolare fiduciale. Il tallone marcatore viene utilizzato per creare una asimmetria nell'immagine del branello magnetico che consente di inseguimento angolo. Se il tallone marcatore è fissato esattamente al nord o sud polo del tallone (vale a dire direttamente in alto o in basso), l'immagine risultante è ancora in rotazione simmetrica e il protocollo di monitoraggio angolare fallisce. Tuttavia, lo stesso argomento geometria sferica, la possibilità di un cordone marcatore per collegare direttamente ad uno dei poli è relativamente piccolo; troviamo che in pratica la maggior parte perline marcatori danno una asimmetria sufficiente a consentire il monitoraggio angolare. Infine, si nota che nel pinzette magnetici convenzionali la direzione del campo è in (x, y)-piano; Pertanto, l'asse di magnetizzazione preferite della perlina si allineeranno in the (x, y)-piano e le poli nord e sud, come sopra definito, stanno per essere ai lati del tallone, improbabile la situazione nella FOMT o MTT, in cui i poli sono in alto e in basso.
In esperimenti FOMT, un passaggio critico è l'allineamento del magnete cilindrico tali che il campo magnetico radiale è trascurabile in prossimità del tallone. Questo allineamento è eseguita per una singola goccia alla volta. Per giudicare se il movimento tallone in FOMT è uniformemente distribuito su un anello circolare, il tempo di misurazione deve essere superiore a 20 · τ C. Come τ C equivale ~ 45 sec per 8 kbp DNA e una perlina 0,5 millimetri raggio, il tempo di misurazione è ~ 900 sec nelle fasi finali di allineamento. Per raffronto, l'utilizzo di 1,9 kbp DNA e 0,25 millimetri perline raggio riduce τ C venti volte a ~ 2 sec (vedi anche tabella 2).
Fasi critiche e considerazioni per monitoraggio Durante FOMT e MTT esperimenti
Per monitorare il tallone di oscillazioni nel piano, cioè la sua (x, y)-posizione, impieghiamo una analisi di correlazione incrociata dei profili di intensità visualizzati da un cordone a successivi intervalli di tempo 35, 36. Questo può essere effettuato a risoluzione sub-pixel con una precisione di pochi nanometri 20. Per monitorare il movimento del tallone in z, si utilizzano in genere un primo metodo creazione di Gosse e Arancino, in cui il piano focale della oggettiva (OFP) è spostato esattamente nella direzione verticale, mentre l'imaging gli anelli di diffrazione del tallone attaccato all'acido nucleico 20 . In questo modo, un profilo di calibrazione viene generato correlando la figura di diffrazione del tallone alla distanza tra il tallone ed il OFP 19. Quando questo profilo calibrazione viene interpolato, gli spostamenti verticali del cordone può essere misurata anche con una precisione fino a pochi nm 20.Ci riferiamo al lettore di riferimenti aggiuntivi che descrivono più raffinati algoritmi di tracking 37, 38, nonché la loro applicazione in parallelo monitoraggio di molteplici perline 5, 6, 37.
Quando utilizzi il monitoraggio angolare che si basa sulla conversione di (x, y) posizioni in coordinate angolari, si consiglia di procedere come segue. Da una traccia temporale in cui il tallone traccia un anello circolare, utilizzare la (x i, y i) posizione (dove l'indice i indica successivi punti di misura) per adattarle al centro del cerchio (x 0, y 0) e raggio R del cerchio (Figura 2a) minimizzando:
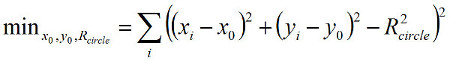 (3)
(3)
dove la somma corre su tutti i punti di dati. Dopo racng x 0, y 0, e R cerchio, determinare le coordinate polari (r i, θ i) di ciascun punto di dati nella traccia tempo utilizzando:
 (4)
(4)
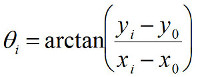 (5)
(5)
Si noti che si dovrebbe prendere cura di "scartare" il θ l'angolo, cioè aggiungere salti di fase ± π, se del caso. Codice personalizzato scritto per il montaggio e la conversione da (x, y) a (r, θ) coordina è disponibile su richiesta degli autori. Nel FOMT, un tempo traccia in cui il tallone traccia un anello circolare può essere ottenuta realizzando il posizionamento grossolano (cfr. punto 3.3) e registrare le fluttuazioni termiche del tallone. Nella MTT, fluctu termicozioni sono insufficienti per tracciare l'anello circolare; invece, utilizzare una traccia temporale in cui i magneti sono lentamente (in genere a 0.1 Hz) ruotato di alcuni giri per adattarsi al cerchio utilizzando Equazioni 3-5.
Notiamo che per la MTT, è importante scegliere il giusto approccio inseguimento angolare, cioè tramite un indicatore di inseguimento angolare (Figura 1c, 1d Figura, Figura 3a) o tramite la conversione di (x, y) posizioni in coordinate angolari ( Figura 1d, Figura 2b). Mentre tipicamente della precisione degli inseguimento angolare da (x, y)-posizioni e l'uso di perline marcatori sono paragonabili, è importante rendersi conto che diafonia si verifica tra le fluttuazioni di un tallone in (x, y) e dell'angolo, come descritto nella Janssen et al 32: in tal modo, inseguimento angolare da (x, y)-posizioni è valida solo a condizione che le fluttuazioni browniano in (x, Y) contribuiscono solo minimamente l'incertezza nella coordinata angolare, e il suo uso corretto di (x, y)-tracking può richiedere una regolazione della trappola rigidezza rotazionale tramite regolazione della posizione del magnete lato. In genere, l'utilizzo di una maggiore rigidità trappola necessario l'utilizzo di inseguimento angolare con perline marcatori. L'uso di perline marcatori richiede un passo ulteriore allegato, che può ridurre il numero di attacchi utilizzabili (vedi protocollo allegato al punto 1.9). Quando si utilizza il monitoraggio basato tallone-marcatore, è importante scegliere biglie magnetiche che hanno una perlina marcatore è attaccato vicino all'equatore per i migliori risultati.
Significato della FOMT e MTT Approcci rispetto ai metodi esistenti e Applicazioni
In precedenza, abbiamo dimostrato come si possa, partendo da MT convenzionale, facilmente modificare le configurazioni magnete per convertire lo strumento in MTT o FOMT. Questo semplice mODIFICA, che può essere accompagnata dall'introduzione di inseguimento angolare quando si desidera l'uso di un marcatore di inseguimento angolare, è un punto di forza immediata di entrambe le configurazioni, in quanto consente all'utente di applicare una coppia, misurare la coppia, o misurare torsione seconda delle sperimentare a portata di mano. Come accennato nell'introduzione, sia FOMT e MTT beneficio da molti dei punti di forza esistenti di MT, in particolare per la loro semplicità, con l'MTT, in particolare, beneficiando anche la capacità di misurazioni parallele 5, 6 (questi non sono così facilmente raggiunti in FOMT data il requisito di allineamento del tether rispetto al centro del magnete cilindrico). In particolare, MTT e FOMT non richiedono, in contrasto con altre tecniche, specialmente nano-fabbricazione di particelle 22, 39, 40, complessa progettazione ottica 41, o l'introduzione di ulteriori perle ai tethered (DNA) molecola 42. Tale otecniche ther possono comunque fornire altri vantaggi quali maggiore risoluzione temporale 27, 43, 44. Sia FOMT e MTT dovrebbero trovare future applicazioni nello studio della trasformazione del genoma, come il comportamento dei motori molecolari sul DNA è sia influenzata da e ha conseguenze per la torsione locale e coppia. Ulteriori applicazioni possono essere trovati nel campo emergente delle nanotecnologie DNA 27 o nel campo più ampio di motori rotativi attivi nella trasformazione biologica 7, 45.
| M270 (R perlina = 1.4 micron) | MyOne (R perlina = 0,5 micron) | Ademtech (R perlina = 0.25 micron) | |
| Convenzionale MT (coppia di cubi 5 x 5 x 5 mm 3 magneti, 1 mm di aria, allineamento verticale) | 70 pN | 8 pN | 1.6 pN |
| FOMT o MTT * (pila di tre magneti cilindrici, diametro di 6 mm, 2 mm di diametro gap) | 9 pN | 1 pN | 0,2 pN |
| FOMT o MTT * (pila di tre magneti cilindrici, diametro di 6 mm, 1 mm di diametro gap) | 18 pN | 2 PN | 0.4 pN |
| FOMT o MTT * (pila di tre magneti cilindrici con ultimo capovolte, 1 mm di aria diametro) | ~ 50 pN | 9 pN | 1.8 pN |
* La presenza del piccolo magnete laterale nel MTT ha un effetto trascurabile sulla forza di allungamento
Tabella 1. Forze massime in genere realizzati per le diverse configurazioni del magnete e tipi di perline.
| R tallone = 1.4 micron | R tallone = 0,5 micron | R tallone =0.25 micron | |
| Coefficiente di attrito * | 120 pN · nm · sec | 5.5 pN · nm · sec | 0.7 pN · nm · sec |
| Scala di tempo caratteristico: FOMT, 10 kbp DNA ** | 1.200 sec | 55 sec | 7 sec |
| Scala di tempo caratteristico: FOMT, 1 kbp DNA | 120 sec | 5.5 sec | 0,7 sec |
| Scala di tempo caratteristico: MTT, k q = 100 pN · nm / rad | 1.2 sec | 0.06 sec | 0.007 sec |
| Scala di tempo caratteristico: MTT, k q = 1000 pN · nm / rad | 0.12 sec | 0.006 sec = 6 msec | 0.0007 s = 0.7 msec |
* Coefficiente di attrito per la rotazione attorno ad un asse attraverso il "equatore" (ossia la situazione mostrata in Figura 1b), In 14 · p · h · R tallone 3, dove h è la viscosità del buffer.
** Nella FOMT, la trappola rigidezza rotazionale è data dalla rigidezza torsionale del DNA, k q, DNA = C · k B T / L C, dove C è la lunghezza efficace persistenza torsionale, considerato uguale a 80 nm here ( che è caratteristica di un regime forza intermedia, F ~ 1 pN) e C L è la lunghezza del profilo del DNA, 0,34 nm per coppia di basi.
Coefficienti di attrito Tabella 2. Ei tempi caratteristici scale per FOMT e MTT.
Divulgazioni
Un brevetto relativo a questo lavoro è stato classificato in riferimento PCT/NL2011/050446.
Riconoscimenti
Questo lavoro è stato supportato dal TU Delft, l'Organizzazione olandese per la ricerca scientifica (NWO), la Fondazione per la Ricerca Fondamentale sulla Materia, e dalla Fondazione europea della scienza.
Materiali
| Name | Company | Catalog Number | Comments |
| Sandblaster | Great Lake Orthodontics | 190-070 Microetcher II | |
| Nitrocellulose | Life Technologies | LC2001 | |
| Magnetic particle concentrator | Life Technologies | 12002D | |
| Non-magnetic latex beads (0.5 μm radius) | Polysciences | 17010 | |
| Non-magnetic latex beads (1.5 μm radius) | Sanbio | PV05N/2179 | |
| Antidigoxigenin | Roche | 11 214 667 001 | |
| Streptavidin-coated superparamagnetic beads (0.25 μm radius) | Ademtech | 3150 | |
| Streptavidin-coated superparamagnetic beads (0.5 μm radius, “MyOne”) | Life Technologies | 650.01 | |
| Streptavidin-coated superparamagnetic beads (1.4 μm radius, “M270”) | Life Technologies | 653.05 | |
| Biotin-coated latex beads (0.5 μm radius) | Life Technologies | F-8768 | |
| Cubic magnets for conventional tweezers | Supermagnete | W-05-N50-G | |
| Cylindrical magnet for MTT and FOMT | Supermagnete | R-06-02-02G | |
| Side magnet for MTT | Supermagnete | S-04-07-N | |
| Linear stage | Physik Instrumente | M-126.PD | |
| Rotary stage | Physik Instrumente | C-150 | |
| High-resolution automated sample stage | Physik Instrumente | P-733.2D | |
| Software for coding analysis routines | The Mathworks | MATLAB | custom-written routines are available from the authors |
Riferimenti
- Strick, T. R., Allemand, J. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271, 1835-1837 (1996).
- Bustamante, C., Bryant, Z., Smith, S. B. Ten years of tension: single-molecule DNA mechanics. Nature. 421, 423-427 (2003).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature methods. 5, 491-505 (2008).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical journal. 96, 5040-5049 (2009).
- Ribeck, N., Saleh, O. A. Multiplexed single-molecule measurements with magnetic tweezers. The Review of scientific instruments. 79, (2008).
- De Vlaminck, I., et al. Highly parallel magnetic tweezers by targeted DNA tethering. Nano letters. 11, 5489-5493 (2011).
- Koster, D. A., Crut, A., Shuman, S., Bjornsti, M. A., Dekker, N. H. Cellular strategies for regulating DNA supercoiling: a single-molecule perspective. Cell. 142, 519-530 (2010).
- Dulin, D., Lipfert, J., Moolman, M. C., Dekker, N. H. Studying genomic processes at the single-molecule level: introducing the tools and applications. Nature reviews. Genetics. 14, 9-22 (2013).
- Ajjan, R., et al. Common variation in the C-terminal region of the fibrinogen beta-chain: effects on fibrin structure, fibrinolysis and clot rigidity. Blood. 111, 643-650 (2008).
- Mierke, C. T., et al. Mechano-coupling and regulation of contractility by the vinculin tail domain. Biophysical journal. 94, 661-670 (2008).
- Shang, H., Lee, G. U. Magnetic tweezers measurement of the bond lifetime-force behavior of the IgG-protein A specific molecular interaction. Journal of the American Chemical Society. 129, 6640-6646 (2007).
- Shang, H. K. P., et al. The application of magnetic force differentiation for the measurement of the affinity of peptide libraries. J Magn Magn Mater. 293, 382-388 (2005).
- Lee, G. U., Metzger, S., Natesan, M., Yanavich, C., Dufrene, Y. F. Implementation of force differentiation in the immunoassay. Analytical biochemistry. 287, 261-271 (2000).
- Smith, A. S., Sengupta, K., Goennenwein, S., Seifert, U., Sackmann, E. Force-induced growth of adhesion domains is controlled by receptor mobility. Proceedings of the National Academy of Sciences of the United States of America. 105, 6906-6911 (2008).
- Kanger, J. S., Subramaniam, V., van Driel, R. Intracellular manipulation of chromatin using magnetic nanoparticles. Chromosome research : an international journal on the molecular, supramolecular and evolutionary aspects of chromosome biology. 16, 511-522 (2008).
- Tanase, M., Biais, N., Sheetz, M. Magnetic tweezers in cell biology. Methods in cell biology. 83, 473-493 (2007).
- Bausch, A. R., Moller, W., Sackmann, E. Measurement of local viscoelasticity and forces in living cells by magnetic tweezers. Biophysical journal. 76, 573-579 (1999).
- Lipfert, J., Koster, D. A., Vilfan, I. D., Hage, S., Dekker, N. H. Single-molecule magnetic tweezers studies of type IB topoisomerases. Methods Mol Biol. 582, 71-89 (2009).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H., Hinterdorder, P., van Oijen, A. . Handbook of Single-Molecule Biophysics. , (2009).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical journal. 82, 3314-3329 (2002).
- Lipfert, J., Wiggin, M., Kerssemakers, J. W., Pedaci, F., Dekker, N. H. Freely orbiting magnetic tweezers to directly monitor changes in the twist of nucleic acids. Nature communications. 2, 439 (2011).
- Celedon, A., et al. Magnetic tweezers measurement of single molecule torque. Nano letters. 9, 1720-1725 (2009).
- Lipfert, J., Kerssemakers, J. J., Rojer, M., Dekker, N. H. A method to track rotational motion for use in single-molecule biophysics. The Review of scientific instruments. 82, (2011).
- Lipfert, J., Kerssemakers, J. W., Jager, T., Dekker, N. H. Magnetic torque tweezers: measuring torsional stiffness in DNA and RecA-DNA filaments. Nature. 7, 977-980 (2010).
- Mosconi, F., Allemand, J. F., Bensimon, D., Croquette, V. Measurement of the torque on a single stretched and twisted DNA using magnetic tweezers. Physical review letters. , 102 (2009).
- Mosconi, F., Allemand, J. F., Croquette, V. Soft magnetic tweezers: A proof of principle. Review of Scientific Instruments. 82 (12), (2011).
- Kauert, D. J., Kurth, T., Liedl, T., Seidel, R. Direct mechanical measurements reveal the material properties of three-dimensional DNA origami. Nano letters. 11, 5558-5563 (2011).
- Velthuis, A., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative Guidelines for Force Calibration through Spectral Analysis of Magnetic Tweezers Data. Biophysical journal. 99, 1292-1302 (2010).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. The Review of scientific instruments. 83, (2012).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76, 409-413 (1999).
- Lee, M., Lipfert, J., Sanchez, H., Wyman, C., Dekker, N. H. Structural and torsional properties of the RAD51-dsDNA nucleoprotein filament. Nucleic acids research. 41, (2013).
- Janssen, X. J., et al. Electromagnetic torque tweezers: a versatile approach for measurement of single-molecule twist and torque. Nano letters. 12, 3634-3639 (2012).
- Baumann, C. G., Smith, S. B., Bloomfield, V. A., Bustamante, C. Ionic effects on the elasticity of single DNA molecules. Proc Natl Acad Sci U S A. 94, 6185-6190 (1997).
- Lipfert, J., Wiggin, M., Kerssemakers, J. W., Pedaci, F., Dekker, N. H. Freely orbiting magnetic tweezers to directly monitor changes in the twist of nucleic acids. Nat Commun. 2, 439 (2011).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophys. J. 81, 2378-2388 (2001).
- Gelles, J., Schnapp, B. J., Sheetz, M. P. Tracking kinesin-driven movements with nanometre-scale precision. Nature. 331, 450-453 (1988).
- Loenhout, M. T., Kerssemakers, J. W., De Vlaminck, I., Dekker, C. Non-bias-limited tracking of spherical particles, enabling nanometer resolution at low magnification. Biophysical journal. 102, 2362-2371 (2012).
- Kim, K., Saleh, O. A. A high-resolution magnetic tweezer for single-molecule measurements. Nucleic acids research. 37, 136 (2009).
- Deufel, C., Forth, S., Simmons, C. R., Dejgosha, S., Wang, M. D. Nanofabricated quartz cylinders for angular trapping: DNA supercoiling torque detection. Nature methods. 4, 223-225 (2007).
- Huang, Z., Pedaci, F., van Oene, M., Wiggin, M. J., Dekker, N. H. Electron beam fabrication of birefringent microcylinders. ACS nano. 5, 1418-1427 (2011).
- La Porta, A., Wang, M. D. Optical torque wrench: angular trapping, rotation, and torque detection of quartz microparticles. Physical review letters. 92, (2004).
- Gore, J., et al. DNA overwinds when stretched. Nature. 442, 836-839 (2006).
- Bryant, Z., Oberstrass, F. C., Basu, A. Recent developments in single-molecule DNA mechanics. Curr Opin Struct Biol. 22, 304-312 (2012).
- Oberstrass, F. C., Fernandes, L. E., Bryant, Z. Torque measurements reveal sequence-specific cooperative transitions in supercoiled DNA. Proceedings of the National Academy of Sciences of the United States of America. 109, 6106-6111 (2012).
- Forth, S., Sheinin, M. Y., Inman, J., Wang, M. D. Torque measurement at the single-molecule level. Annu Rev Biophys. 42, 583-604 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon