Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse d’Interaction psychophysiologique généralisée (PPI) de mémoire qui connectivité chez les personnes à risque génétique pour la maladie d’Alzheimer
Dans cet article
Résumé
Cet article décrit comment implémenter une analyse de l’interaction psychophysiologique pour révéler des changements de tâche dépendante de la connectivité fonctionnelle entre une région de semences sélectionnées et les voxels dans d’autres régions du cerveau. Analyse de l’interaction psychophysiologique est une méthode populaire pour étudier les effets de tâche sur la connectivité de cerveau, distincte des effets d’activation univariée traditionnel.
Résumé
Neuro-imagerie, imagerie de résonance magnétique fonctionnelle (IRMf) mesure le signal (BOLD) dépendant de sang-oxygénation-niveau dans le cerveau. Le degré de corrélation entre le "BOLD" signal spatialement en régions indépendantes du cerveau définit la connectivité fonctionnelle de ces régions. Au cours d’une tâche cognitive IRMf, une analyse de l’interaction psychophysiologique (PPI) permet d’analyser les changements dans la connectivité fonctionnelle au cours de contextes spécifiques définis par la tâche cognitive. Un exemple d’une telle mission est celui qui engage le système de mémoire, demandant aux participants d’apprendre des paires de mots indépendants (codage) et rappeler le deuxième mot dans une paire quand présenté avec le premier mot (récupération). Dans la présente étude, nous avons utilisé ce type de tâche de mémoire associative et une analyse généralisée de PPI (gPPI) pour comparer les changements en matière de connectivité hippocampe chez les adultes âgés qui sont porteurs de la maladie d’Alzheimer (ma) facteur de risque génétique apolipoprotéine-E epsilon-4 ( APOEΕ4). Plus précisément, nous montrons que la connectivité fonctionnelle des sous-régions des changements hippocampe pendant l’encodage et de récupération, les deux phases actives de la tâche de mémoire associative. Dépendant du contexte variations connectivité fonctionnelle de l’hippocampe ont été significativement différentes chez les porteurs de APOEε4 par rapport aux non-porteurs. Analyses PPI permettent d’analyser les changements dans la connectivité fonctionnelle, distincte d’univariée effets principaux et de comparer ces changements entre groupes. Ainsi, une analyse PPI peut révéler des effets de tâche complexe en cohortes spécifiques que les méthodes traditionnelles univariée ne tiennent pas comprennent. Analyses PPI ne peut pas, cependant, déterminer directionnalité ou causalité entre régions fonctionnellement reliées. Néanmoins, analyses PPI fournissent des moyens puissants pour générer des hypothèses spécifiques au sujet des relations fonctionnelles, qui peuvent être testées à l’aide de modèles de causalité. Comme de plus en plus, le cerveau est décrite en termes de connectivité et réseaux, PPI est une importante méthode d’analyse des données de tâche IRMf qui s’inscrit dans la conception actuelle du cerveau humain.
Introduction
Le terme « connectome » a été inventé en 2005, marquant un changement de paradigme en neurosciences qui continue à ce jour1. Le cerveau est de plus en plus décrit en termes de réseaux fonctionnels, la connectivité et les interactions entre régions et entre elles sur une grande échelle. Néanmoins, la délimitation de la spécialisation fonctionnelle régionale et les associations entre l’activité mesurée de l’IRMf et les demandes de tâche sont des approches restent valables et utiles. Compte tenu de l’intérêt croissant pour les connectomics, connectivité fonctionnelle des approches d’analyse des tâches IRMf gagnent en popularité. Une approche pour mesurer les variations de la connectivité fonctionnelle dépendantes de la tâche exige fait usage du concept de PPI. Un IPP est l’interaction entre une tâche active phase ou de la demande de tâche particulière (« psycho ») et la connectivité fonctionnelle (« physio ») d’une région d’intérêt ou de la « graine » dans le cerveau. PPI diffère de l’analyse bivariée, axée sur la corrélation de la connectivité fonctionnelle, qui mesure généralement le degré de corrélation entre l’activité dans deux régions sans toutes les contraintes liées aux exigences de la tâche.
Le concept et le cadre d’une analyse PPI a été initialement décrite par Friston et ses collègues en 19972. Les auteurs ont affirmé que leur approche était importante, car elle permettrait à l’enquête de la connectivité d’être plus fonctionnelle spécifique et permettre des inférences qu’activité dans une graine distale pourrait être modulant l’activité résultant d’une demande de tâche. En 2012, McLaren et ses collaborateurs a ajouté à ce cadre original et décrit une approche gPPI dans lequel toutes les phases de travail et leurs interactions sont incluses dans un seul modèle3. Cette approche conduit à des résultats qui sont plus sensibles et plus spécifiques à la phase opérationnelle et l’interaction étudiée. C’est cette approche gPPI actualisé que nous employons dans la présente étude (Voir l’étape 6.2.2 de protocole). L’approche gPPI a maintenant été citée dans plus de 200 études. Pour plus de clarté, ci-après, nous utilisons « PPI » pour décrire les caractéristiques communes de la version standard et généralisée. « gPPI » serviront pour discuter des progrès spécifiques associés au cadre plus récent.
L’objectif global d’une analyse PPI est de comprendre comment les exigences d’une tâche cognitive influencent ou modulent la connectivité fonctionnelle d’une région de semences. Une analyse PPI nécessite une hypothèse fort a priori . Activité dans la région de semences doit être modulée par la tâche afin que l’approche PPI à travailler efficacement4. Par exemple, dans la présente étude, nous avons fondé notre sélection de semences sur les éléments de preuve solide que hippocampe activité est modulée par les exigences cognitives d’une tâche de mémoire. À l’aide de PPI, régions qui sont significativement plus ou moins fonctionnellement reliées à l’hippocampe au cours des phases de tâche spécifique peuvent être identifiées. En bref, nous posons la question, « dans quelles régions est l’activité plus corrélé avec la semence pendant contexte un par rapport à la base ? » Nous pouvons également demander le contraire logique (tel qu’il est important de comprendre la différence) : « dans quelles régions est activité moins corrélées avec la semence pendant contexte un par rapport à la base ? » Lors de l’interprétation des différences de groupe des effets de PPI, il est important d’examiner les données et savoir si un changement positif ou négatif dans la connectivité fonctionnelle, ou les deux, est le moteur de différences entre les groupes.
L’approche PPI a été utilisée pour étudier les centres de contrôle de tâche dynamique à témoins sains, comment la modulation de la connectivité fonctionnelle est liée à des performances cognitives dans la maladie d’Alzheimer (ma), intelligence chez les individus atteints d’autisme, connectivité réseau moteur chez les personnes atteintes de la maladie de Parkinson, visage de traitement chez les personnes souffrant de dysmorphophobie et anorexie, règlement de l’émotion, la mémoire et bien d’autres questions spécifiques liées à la connectivité5,6,7 ,8,9,10,11. Dans la présente étude, nous comparer les changements dans la connectivité fonctionnelle des sous-régions de l’hippocampe au cours de la mémoire d’encodage et de récupération entre un groupe de personnes à risque génétique pour annonce un groupe sans le facteur de risque12. Ce qui suit décrit le protocole que nous avons utilisé, conformément à la méthode gPPI, pour nous permettre de vérifier si les variations induites à la tâche connectivité fonctionnelle diffèrent en liaison avec la présence de APOEε4, un facteur de risque génétique pour AD.
Access restricted. Please log in or start a trial to view this content.
Protocole
la présente étude a été réalisée en conformité avec les protocoles de UCLA Institutional Review Board (IRB) et approuvée par le Comité de Protection des sujets humains UCLA. Tous les participants ont donné le consentement éclairé afin de s’inscrire dans cette étude.
1. participant sélection
- approbation de la CISR à obtenir pour effectuer l’étude de. Individus d’écran
- âgés de 55 ans et plus pour le déclin cognitif à l’aide d’une batterie neuropsychologique normalisée. Comprendre les tests d’Intelligence générale (sous-tests du WAIS-III) 13, aisance (Fruits et légumes) 14, Attention (chiffres Forward et Backward) 13, langue (Test Naming de Boston ) 15, 16 la mémoire verbale (Buschke-Fuld sélectifs rappelant tâche), apprentissage WMS-III mémoire logique et verbale jumelé associe 13 et mémoire visuelle (test de Rey-Osterrieth Figure) 17.
- ont les participants de remplir les questionnaires d’humeur comme les inventaires d’anxiété et de dépression de Hamilton 18 , 19 ainsi que le (Mini Mental State Exam MMSE) 20.
- Comprennent les participants qui obtiennent un score 26 ou supérieur sur l’ESGI et obtiennent de meilleurs résultats que deux écarts-types au-dessous de la normale pour leur âge dans les tests cognitifs. Exclure les participants avec la clinique anxiété, de dépression ou de toute autre maladie neuropsychiatrique ou neurologique. Exclure les participants qui ne satisfont pas aux critères de sécurité de MRI ou qui ne consentez pas à un sang dessiner.
Remarque : Dans la présente étude, 93 participants se sont réunis ces critères (âge moyen = 67,4 ans, 31M/49F).
2. Génotypage
- avez un prélèvement formé ou autre professionnel de la santé prélever du sang de chaque participant.
- Isoler 200 µg d’ADN génomique de 10 mL de l’échantillon comme décrit 21.
- Réaliser de polymorphisme nucléotidique simple génotypage (SNP) à l’aide de PCR en temps réel à deux loci, rs429358 et rs7412 de discriminer APOE allèles 22.
- Incorporate journaliste colorants pour rs429358 et rs7412 dans un test de génotypage SNP. Après chaque cycle d’amplification PCR, tracer des signaux fluorescents sur un graphique montrant la répartition des colorants reporter et extincteur. Réaliser l’expérience en double exemplaire pour confirmer les résultats.
- Analyser les données de génotypage SNP à l’aide d’un logiciel mis au point pour la procédure PCR en temps réel de sortie 23.
Remarque : Le programme utilisé dans la présente étude calcule l’affinité de l’échantillon à l’un des colorants deux journaliste qui, à son tour, représente un SNP APOE sur l’autre. Dans la présente étude, 34 transporteurs de l’annonce des risques allèle ε4 APOE (hétérozygotes ε3/ε4) et 46 non-porteurs (homozygote ε3/ε3) ont été inscrits pour un total de 80 participants à l’étude. Exclure les porteurs de l’allèle APOEε2 parce qu’il y a preuve que cet allèle peut avoir un effet protecteur lié à AJ.
3. Fonctionnelles et collecte de données d’imagerie structurale
- utilisation d’une IRM 3 Tesla (3 t) système d’acquérir des données d’imagerie du cerveau entier.
- Pour l’imagerie fonctionnelle, recueillir des tranches axiales utilisant un écho planar d’imagerie (EPI) séquence. Pour faciliter l’enregistrement des images fonctionnelles, acquérir des tranches axiales d’images structurelles pondérées en T2, coplanaires. Pour une imagerie structurale, recueillir des tranches axiales utilisant un 3D séquences pondérées en T1.
Remarque : Dans la présente étude, un aimant de 3 t a été utilisé avec une bobine de tête 12 canaux. Les paramètres ci-dessous ont été conçus pour un scanner spécifique et la bobine. Pour plus d’informations, consultez Table des matières.- Acquérir les données en utilisant les paramètres suivants de la séquence d’imagerie fonctionnelle : temps de répétition (TR) = 2 500 ms, écho de temps (TE) = 21 ms, champ de vision (FOV) = 200 mm x 200 mm, angle flip = 75°, matrice = 64 x 64, 33 tranches, tranches épaisseur = 3 mm, espace entre les tranches = 0 .75 mm, taille du voxel = 3,125 x 3.125 x 3,75 mm.
- Déclencher la tâche de mémoire associative de mots indépendants pour commencer avec le troisième volume de la séquence d’imagerie fonctionnelle. Pour tenir compte de l’équilibre stationnaire, exclure les deux premiers volumes de chaque analyse fonctionnelle analyses.
Remarque : Les mots sans rapport tâche de mémoire associative a été décrit ailleurs 12 , 24. Brièvement, c’est une tâche de bloc-conception fonctionnelle avec l’encodage et l’extraction de blocs. Les participants sont chargés d’apprendre des paires de mots indépendants. - Acquire pondérées en T2, coplanaires structurels d’imagerie des données en utilisant les paramètres suivants de la séquence : TR = 5 000 ms, TE = 34 ms, FOV = 200 mm x 200 mm, angle flip = 90°, matrice = 128 x 128, 28 tranches, épaisseur de tranche = 3 mm, espace entre les tranches = taille de 1 mm et voxel = 1,56 x 1 .56 x 4 mm.
- Acquérir haute résolution structurale (anatomique) d’imagerie utilisant les paramètres de séquence de magnétisation préparé rapide Gradient Echo (MPRAGE) suivants : TR = 1 900 ms, TE = 2,26 ms, TI = 900 ms, FOV = 250 mm x 218 mm, angle flip = 9°, matrice = 256 x 215, 176 tranches , épaisseur de tranche = 1 mm, zéros à une matrice de 256 x 224 aboutissant à une taille de voxel = 1 x 0,976 x 0,976 mm.
- Pour l’imagerie fonctionnelle, recueillir des tranches axiales utilisant un écho planar d’imagerie (EPI) séquence. Pour faciliter l’enregistrement des images fonctionnelles, acquérir des tranches axiales d’images structurelles pondérées en T2, coplanaires. Pour une imagerie structurale, recueillir des tranches axiales utilisant un 3D séquences pondérées en T1.
4. IRMf BOLD prétraitement de données
- Prétraiter les données fonctionnelles à l’aide de l’IRM fonctionnelle de la cerveau (FMRIB) Software Library (FLS) version 6.0 (http://fsl.fmrib.ox.ac.ul) comme suit :
- Pour chaque participant ' s dataset, artefact de mouvements de la tête supprimer des données à l’aide de la Motion Correction FMRIB ' s outil de d’enregistrement des images linéaires (MCFLIRT) 25.
- Enlever les tissus non brain à partir des images à l’aide de l’outil d’extraction cerveau (BET) avec l’option -F drapeau 26.
- Utiliser l’outil de FLS Motion valeurs aberrantes pour identifier tous les volumes de données fonctionnelles dans lesquelles sévit un mouvement excessif, basée sur la cylindrée de trame entre les volumes. Drapeau des volumes où la motion est mesurée comme une valeur aberrante (au-dessus de la 75 ème percentile + 1,5 fois l’intervalle interquartile) par rapport au reste de l’analyse et utilisez le résultat de ce programme pour downweight ces volumes dans les analyses.
Remarque : Avant d’exécuter des comparaisons de groupe, vérifiez que mouvement moyen, tel que mesuré par le FSL Motion aberrantes, ne diffère pas entre les deux groupes. Cela aidera à faire en sorte que les résultats ne sont pas poussés par différence liée au groupe dans la motion.
- Mis en place le prétraitement et premier niveau modèle linéaire général (GLM) à l’aide de l’interface utilisateur graphique (GUI) pour FLS IRMf Expert Analysis Tool (FEAT) pour le premier participant.
Remarque : Répétez cette étape pour chaque participant à l’étude. Pour gagner du temps, après la mise en place d’un seul passage pour un participant, écrire un script pour exécuter le prétraitement pour les autres participants de l’étude ' données en modifiant la " design.fsf " fichier (FSL FEAT sortie) pour chaque participant à faire référence à ce participant, ' s données spécifiques.- Dans l’onglet données, cliquez sur " ajouter des données de 4D " et naviguez jusqu’au fichier motion-corrigés et extraits au cerveau. La valeur du TR 2,5 s (correspondant à la TR de la séquence fonctionnelle acquise). Utilisez le filtre par défaut (la valeur 100 s).
NOTE : Filtrage passe-haut supprimera les signaux basse fréquence sans intérêt. - Dans l’onglet stats avant, cliquez sur " aucun " sous " correction de mouvement " (comme il a été déjà effectuée à l’étape 4.1). Décochez la case " BET cerveau extraction " (comme il était déjà achevé à l’étape 4.1). Type " 5 " dans la boîte de réglage 5 mm pleine largeur moitié-maximum (FWHM) gaussienne noyau pour le lissage spatial.
NOTE : La FWHM pour le noyau lissage doit généralement être réglé à environ deux fois la taille de la taille du voxel analyse fonctionnelle. - Utiliser la sortie (6 colonnes, lignes = Nbre de TRs dans l’analyse) de MCFLIRT pour créer des fichiers de texte colonne 6 qui décrivent la correction de mouvement réalisées dans chaque volume dans le dataset. Celles-ci s’ajouteront au modèle comme régresseurs dans l’étape suivante.
- Dans l’onglet stats sous " complet d’installation avec le modèle ", ajouter les paramètres de 6 requête et leurs dérivés temporelles comme régresseurs ou variables explicatives (EVs) dans le MLG. Pour chaque mouvement EV choisir " personnalisée " (1 entrée par volume) pour la forme de base, " aucun " convolution et cocher " appliquer le filtrage temporel. "
Remarque : paramètres de mouvement ne doivent pas être convolé par n’importe quelle fonction car elles référencent le réalignement obtenu à chaque volume fonctionnel pendant la correction de mouvement et donc n’ont pas besoin d’être ajusté.
- Dans l’onglet stats sous " complet d’installation avec le modèle ", ajouter les paramètres de 6 requête et leurs dérivés temporelles comme régresseurs ou variables explicatives (EVs) dans le MLG. Pour chaque mouvement EV choisir " personnalisée " (1 entrée par volume) pour la forme de base, " aucun " convolution et cocher " appliquer le filtrage temporel. "
- Dans l’onglet statistiques, sélectionnez la sortie des valeurs aberrantes de la FSL Motion d’étape 4.1 sous la " ajouter supplémentaires confondre EVs ".
Remarque : Cette sortie est une matrice indiquant chaque volume qui a été marqué pour mouvement excessif et, en ajoutant le fichier confondez, va être deweighted dans le MLG. - Dans l’onglet statistiques, cliquez sur " Set-up modèle complet ". Créez la tâche des fichiers de texte calendrier indiquant l’apparition et le décalage des phases différentes tâches et ajoutez-les comme EVs dans le MLG en choisissant format de colonne 1 et naviguer vers le fichier texte correspondant (inclure un pour la phase de codage de la tâche et l’autre pour la phase de récupération). Pour " convolution " choisir le " double-gamma FRH " option dans la liste déroulante pour chacun d’eux. Ne pas poser la base ou des parties non actifs de la tâche dans la MLG.
NOTE : HRF est fonction de la réponse hémodynamique. Convolution la tâche EV par le FRH décale le calendrier de la tâche EV à être plus cohérent avec les changements attendus de signal "BOLD" induite par la tâche dans le cerveau. - Dans l’onglet Enregistrement, cochez " élargi image fonctionnelle " et " image structurelle principale " pour une inscription en deux étapes.
- Sélectionnez le participant ' s coplanaires pondérées en T2 structurels scan pour la première étape, dans lequel les données fonctionnelles sont enregistrées dans les données structurales coplanaires. Choisir 6 degrés de liberté (DDL) pour cette étape en cliquant sur la deuxième liste déroulante case sous cette étape et en choisissant " 6 DOF ".
- Pour l’étape suivante, dans laquelle les images pondérées en T2 est enregistré à la haute résolution pondérées en T1 MPRAGE, sélectionne l’enregistrement de limite basée (BBR) dans le menu déroulant zone 27.
NOTE : BBR utilise les différences d’intensité entre substance blanche et substance grise pour inscrire des analyses structurales et fonctionnelles et s’est avéré fonctionner mieux que FLIRT et autres méthodes alternatives. - Pour l’étape finale, dans laquelle les données structurales à haute résolution sont enregistrées pour le modèle MNI152 standard, choisissez 12 degrés de liberté et d’une transformation linéaire en choisissant " 12 DOF ".
Remarque : Lorsque toutes les étapes dans la section 4 sont complètes données fonctionnelles sont prétraités et prêt pour une analyse ultérieure.
- Dans l’onglet données, cliquez sur " ajouter des données de 4D " et naviguez jusqu’au fichier motion-corrigés et extraits au cerveau. La valeur du TR 2,5 s (correspondant à la TR de la séquence fonctionnelle acquise). Utilisez le filtre par défaut (la valeur 100 s).
5. Hippocampe graines
- générer un masque de l’hippocampe gauche dans chaque participant ' s haute résolution structurale espace aide FSL ' algorithme de segmentation FMRIB inscription intégrée et outil de Segmentation (premières) s 28 .
Remarque : Les autres régions, dont l’hippocampe droit, serait intéressantes et graines valables pour les autres analyses. - à l’aide d’une plate-forme de logiciels statistiques, écrire du code dans pour calculer la longueur des tiers antérieurs et postérieurs de la structure 29. En particulier, utiliser la longueur du masque hippocampe volumétrique dans le plan antéro-postérieur pour trouver les coordonnées aborner les tiers antérieurs et postérieurs de ce plan.
Remarque : Une méthode récemment publiée de segmentation de l’hippocampe le long de l’axe longitudinal est peut-être une autre graine création approche 30. - Basé sur ces coordonnées, créer des images de masque hippocampe antérieures et postérieures. Enregistrer les masques hippocampe antérieurs et postérieurs en espace fonctionnel natif en utilisant la " example_func2highres " matrice dans le répertoire d’enregistrement de la sortie FEAT.
Remarque : En utilisant les tiers antérieurs et postérieurs empêché signal flou à travers les deux graines hippocampe après son inscription à l’espace fonctionnel. Il y a preuve de spécialisation fonctionnelle selon l’axe longitudinal de l’hippocampe 31 , 32 , 33 , 34. Régions antérieures sont des régions d’entrée et associé de l’encodage, alors que l’hippocampe postérieure est une région de production associée à mémoire récupération et consolidation 35 , 36 , 37. ainsi, l’utilisation des ces régions permet d’évaluer de participation fonctionnelle des antérieure versus postérieur hippocampe en encodage par rapport aux phases de recherche de la tâche de mémoire. - Utilisation FLS signifie timeseries (fslmeants) pour extraire le bruit timeseries moyenne des graines hippocampe antérieur et postérieur ( Figure 1). Suivez les instructions de programme et utilisez la graine hippocampe d’antérieure ou postérieure comme le masque et les bruit, prétraitées données fonctionnelles l’image principale.
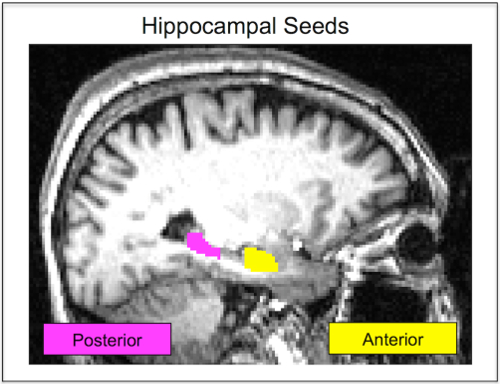
figure 1 : graines hippocampiques. Dans l’espace natif, un seul participant ' semences hippocampe antérieur s s’affiche en jaune. La graine de l’hippocampe postérieur pour le même participant est indiquée en rose. Les graines sont définis dans chaque participant ' image structurelle unique s et ensuite enregistrée à leur analyse fonctionnelle. Les graines sont jamais dans un espace normalisé, ce qui améliore la précision de la segmentation hippocampe. Ce chiffre a été réimprimé avec la permission de 12. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
6. PPI modèle
- utiliser le GUI pour FLS FEAT charger les données fonctionnelles prétraitées.
- Dans l’onglet données, sélectionnez le " filtered_func_data " DEBRUITE image (sortie d’étapes franchies dans la section 4) que le fichier d’entrée. Dans l’onglet stats avant, affectez à extraction de correction et de cerveau de motion " none. " décochez les cases pour effectuer un filtrage temporel et le lissage spatial.
- PPI ModEl Set-up (tableau 1).
- Dans l’onglet statistiques, sélectionnez " Set-up modèle complet ". Dans l’onglet EVs, ajoutez tous les EVs depuis le premier modèle de niveau : 6 motion correction EVs, confondre EV matrice de valeurs aberrantes de la FSL Motion et la tâche de synchronisation EVs. Cliquez sur la flèche haut pour ajouter le serveur virtuel Exchange. Inclure dans ce modèle une EV pour le timecourse physiologique de la graine (la sortie du fichier texte de fslmeants à l’étape 5.4) comme covariable sans intérêt en cliquant sur la flèche vers le haut.
- Créer les conditions PPI.
- Choisir " interaction " dans le menu base forme et choisissez la semence timecourse EV et une tâche EV. Pour le " faire le zéro " option, choisissez " veut dire " pour le semences timecourse EV et " centre " pour la tâche EV. Répétez cette procédure pour l’autre tâche ou phases. Exécuter un modèle distinct pour chaque région de semence.
Remarque : Ces nouvelles EVs sont les termes PPI pour la phase de la tâche sélectionnée (psycho) et la graine (physio). Dans la présente étude, un terme PPI pour la phase de codage et un second mandat PPI pour la phase de récupération ont été inclus dans chaque modèle PPI. Le " centre " option veille à ce que le " sur " et " hors " phases de la tâche de conception de bloc sont traités de manière égale. Le " signifie " option est toujours appliquée à la timecourse semences et se traduit par la moyenne étant soustraite ce régresseur.
- Choisir " interaction " dans le menu base forme et choisissez la semence timecourse EV et une tâche EV. Pour le " faire le zéro " option, choisissez " veut dire " pour le semences timecourse EV et " centre " pour la tâche EV. Répétez cette procédure pour l’autre tâche ou phases. Exécuter un modèle distinct pour chaque région de semence.
- Dans les contrastes et l’onglet tests F, modéliser les effets spécifiques suivants en saisissant " 1 " dans l’EV correspondant cell : psych_enc (phase de tâche codage), psych_ret (phase de tâche de récupération), phys (semences timecourse), PPI_enc (PPI des semences et encodage), PPI_ret (PPI de semences et de la récupération). Enfin, entrer dans un " -1 " pour modéliser des IPP négatif pour chaque phase de travail.

tableau 1 : installation du modèle gPPI.
7. comparaisons de groupe
- Select " niveau supérieur analyse " en FLS FEAT pour exécuter un modèle simple groupe comparant APOEε4 transporteurs aux non-porteurs pour chaque combinaison de tâche-semence.
Remarque : Ces comparaisons sont exécutés pour générer les images de résidus de 4D groupe concerné (" res4d ") nécessaires pour estimer la finesse de l’objet dataset. Des résultats statistiquement significatifs de cette comparaison de groupe sont valides, mais utilisant AFNI et SPM8 pour définir un cluster significatif minimal basé sur des simulations de Monte-Carlo est décrite dans les étapes ci-dessous une autre méthode de seuillage. - Utilisation analyse de Functional Neuroimaging (AFNI)
- usage AFNI ' s 3dFWHMx (n’importe quelle version après décembre 2015) sur la ligne de commande pour estimer la douceur des résidus 4D groupe images générées à l’aide de FLS.
Remarque : Un bug a été découvert en AFNI ' s 3dClustSim et corrigée en mai 2015. En décembre 2015, AFNI ' s 3dFWHMx a été mis à jour avec plus de précision modèle auto-corrélations. Ainsi, les versions de ces outils sorti en décembre 2015 ou plus tard doit être utilisées. - Usage AFNI ' s 3dClustSim (n’importe quelle version après décembre 2015) afin de déterminer le minimum de mesure de cluster atteignant importance à différents seuils de voxel-niveau. Inclure les estimations de la douceur de l’étape précédente dans l’invocation de la ligne de commande de 3dClustSim. À partir de la table générée par 3dClustSim, basé sur les hypothèses de l’étude concernant les effets attendus ' hauteur et l’étendue, choisissez un voxel-niveau seuil et la taille minimale correspondante cluster.
Remarque : en général, plus grandes grappes minimiser les faux positifs.
- usage AFNI ' s 3dFWHMx (n’importe quelle version après décembre 2015) sur la ligne de commande pour estimer la douceur des résidus 4D groupe images générées à l’aide de FLS.
- Utilisation Statistical Parametric Mapping (SPM8)
- à l’aide de l’interface graphique SPM8, sélectionnez " spécifier 2 nd-niveau ". L’éditeur de commandes s’ouvre. Sélectionnez " deux échantillon test t " en cours de conception. Naviguez jusqu’au répertoire avec les images d’estimation de paramètre pour le groupe 1 (APOEε4, transporteurs) et sélectionnez en cliquant dessus. Ensuite, ajoutez des images (APOEε4 non-porteurs) groupe 2. Exécutez cette comparaison en cliquant sur le bouton vert play.
- Revenir à l’interface graphique de SPM, sélectionnez " estimer ", puis accédez au fichier SPM.mat créé à l’étape précédente pour exécuter le processus d’estimation de modèle.
- Select " résultats " et exécutez groupe comparaison contrastes : transporteurs APOEε4 > APOEε4 non-porteurs, non-porteurs APOEε4 > APOEε4 transporteurs.
- Cliquez sur " définir un nouveau contraste ", choisissez " T-contraste " sous " type " et entrez " 1 -1 " dans la " contraste " boîte pour les transporteurs de APOEε4 > APOEε4 non-porteurs. Cliquez sur " fait ". Choisissez " aucun " pour appliquer le masque et manuellement ensemble le voxel-niveau seuil et le minimum de taille de cluster selon la décision rendue dans l’étape 7.2.2.Enter " 1-1 " pour APOEε4 non-porteurs > transporteurs APOEε4.
Remarque : dans la présente étude, un seuil de voxelwise de p < 0,005 a été utilisé et grappes seuillée à alpha < 0,05.
- Cliquez sur " définir un nouveau contraste ", choisissez " T-contraste " sous " type " et entrez " 1 -1 " dans la " contraste " boîte pour les transporteurs de APOEε4 > APOEε4 non-porteurs. Cliquez sur " fait ". Choisissez " aucun " pour appliquer le masque et manuellement ensemble le voxel-niveau seuil et le minimum de taille de cluster selon la décision rendue dans l’étape 7.2.2.Enter " 1-1 " pour APOEε4 non-porteurs > transporteurs APOEε4.
Access restricted. Please log in or start a trial to view this content.
Résultats
Avec deux phases différentes tâche active (encodage et récupération) et deux régions (hippocampe antérieure et postérieure), il y a quatre conditions à soumettre des rapports pour chaque groupe. Les cartes d’activation intra-groupe de travail (non illustrée ici, voir Harrison et al., 201612) montrent que le lobe occipital, cortex auditif, grandes régions du lobe pariétal, les zones de langue frontale, gyrus temporal supérieur et le noyau caud...
Access restricted. Please log in or start a trial to view this content.
Discussion
Les premières études d’IRMf basée sur les tâches ont été conçus afin de découvrir les relations statistiques entre les processus cognitifs particulières ou exigences et les changements dans le "BOLD" du signal par rapport à une mesure de base. Cette approche traditionnelle est utile pour identifier des régions spécifiques du cerveau dont l’activité est modulée par une tâche expérimentale. En revanche, une analyse PPI est principalement préoccupée avec la modulation de la connectivité fonctionnelle,...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
DGM est qu'un employé de Biospective, Biospective, Inc. a fait pas traiter des données présentées.
Remerciements
Ce travail a été soutenu par le National Institute of Aging (numéro de licence R01AG013308 à SYB, F31AG047041 à TMH). Les auteurs ont utilisé computationnelle et services de stockage associé de Cluster partagé Hoffman2 prévues par UCLA Institute Research Technology Group Digital Research et de l’éducation.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 3T manetic resonance imaging scanner | Siemens Medical Solutions | MAGNETOM Trio, A Tim System | 3T MRI Scanner |
| FSL (FMRIB Software Library) | Oxford University | Version 6.0 | Functional Imaging Processing Software |
| AFNI (Analysis of Functional Neuroimaging) | National Institute of Mental Health, National Institutes of Health | Any version after May 2015 | Functional Imaging Processing Software |
| SPM8 (Statistical Parametric Mapping) | University College of London | SPM8 | Functional Imaging Processing Software |
| Matlab Software | The Mathworks, Inc | Version R2012a | Computing Software |
| SDS Software | Applied Biosystems, Inc | 7900HT Fast Real-Time PCR System | Real Time PCR |
| Taqman Assays | ThermoFisher Scientific | Specific to SNP | SNP Genotyping |
Références
- Sporns, O., Tononi, G., Kötter, R. The Human Connectome: A Structural Description of the Human Brain. PLoS Comput Biol. 1 (4), 42(2005).
- Friston, K. J., Buechel, C., Fink, G. R., Morris, J., Rolls, E., Dolan, R. J. Psychophysiological and modulatory interactions in neuroimaging. NeuroImage. 6 (3), 218-229 (1997).
- McLaren, D. G., Ries, M. L., Xu, G., Johnson, S. C. A generalized form of context-dependent psychophysiological interactions (gPPI): a comparison to standard approaches. NeuroImage. 61 (4), 1277-1286 (2012).
- O'Reilly, J. X., Woolrich, M. W., Behrens, T. E. J., Smith, S. M., Johansen-Berg, H. Tools of the trade: psychophysiological interactions and functional connectivity. Soc Cogn Affect Neurosci. 7 (5), 604-609 (2012).
- Moody, T. D., Sasaki, M. A., et al. Functional connectivity for face processing in individuals with body dysmorphic disorder and anorexia nervosa. Psychol Med. 45 (16), 3491-3503 (2015).
- Simard, I., Luck, D., Mottron, L., Zeffiro, T. A., Soulières, I. Autistic fluid intelligence: Increased reliance on visual functional connectivity with diminished modulation of coupling by task difficulty. NeuroImage Clin. 9, 467-478 (2015).
- Yan, L. -R., Wu, Y. -B., Zeng, X. -H., Gao, L. -C. Dysfunctional putamen modulation during bimanual finger-to-thumb movement in patients with Parkinson's disease. Front Hum Neurosci. 9, 516(2015).
- Cole, M. W., Reynolds, J. R., Power, J. D., Repovs, G., Anticevic, A., Braver, T. S. Multi-task connectivity reveals flexible hubs for adaptive task control. Nat Neurosci. 16 (9), 1348-1355 (2013).
- McLaren, D. G., Sperling, R. A., Atri, A. Flexible modulation of network connectivity related to cognition in Alzheimer's disease. NeuroImage. 100, 544-557 (2014).
- Morawetz, C., Bode, S., Baudewig, J., Heekeren, H. R. Effective amygdala-prefrontal connectivity predicts individual differences in successful emotion regulation. Soc Cogn Affect Neurosci. , 169(2016).
- Takashima, A., Bakker, I., van Hell, J. G., Janzen, G., McQueen, J. M. Richness of information about novel words influences how episodic and semantic memory networks interact during lexicalization. NeuroImage. 84, 265-278 (2014).
- Harrison, T. M., Burggren, A. C., Small, G. W., Bookheimer, S. Y. Altered memory-related functional connectivity of the anterior and posterior hippocampus in older adults at increased genetic risk for Alzheimer's disease. Hum Brain Mapp. 37 (1), 366-380 (2016).
- Wechsler, D. Wecshler Adult Intelligence Scale, 3rd Edition. , Harcourt Assessement. San Antonio, TX. (1997).
- Cauthen, N. R. Verbal fluency: normative data. J Clin Psychol. 34 (1), 126-129 (1978).
- Goodglass, H. P., Kaplan, E. P. Boston Naming Test, 3rd Edition. , (2001).
- Buschke, H., Fuld, P. A. Evaluating storage, retention, and retrieval in disordered memory and learning. Neurol. 24 (11), 1019-1025 (1974).
- Osterrieth, P. A. Le test de copie d'une figure complex: Contribution à l'étude de la perception et de la memoir. Archives de Psychologie. 30, 286-356 (1944).
- Hamilton, M. The assessment of anxiey states by rating. Br J Med Psychol. 32, (August) 50-55 (1959).
- Hamilton, M. A rating scale for depression. J Neurol Neurosurg Psychiatry. 23, 56-62 (1960).
- Folstein, M. F., Robins, L. N., Helzer, J. E. The Mini-Mental State Examination. Arch Gen Psychiatry. 40 (7), 812(1983).
- O'Brien, D., Campbell, K. A., Morken, N. W., Bair, R. J., Heath, E. M. Automated Nucleic Acid Purification for Large Samples. J Lab Autom. 6 (2), 67-70 (2001).
- Lehmann, M., Ghosh, P. M., et al. Greater medial temporal hypometabolism and lower cortical amyloid burden in ApoE4-positive AD patients. J Neurol Neurosurg Psychiatry. 85 (3), 266-273 (2014).
- TaqMan® SNP Genotyping Assays User Guide. , Thermo Fisher Scientific. Available from: https://tools.thermofisher.com/content/sfs/manuals/TaqMan_SNP_Genotyping_Assays_man.pdf (2014).
- Bookheimer, S. Y., Strojwas, M. H., et al. Patterns of brain activation in people at risk for Alzheimer's disease. N Engl J Med. 343 (7), 450-456 (2000).
- Jenkinson, M., Bannister, P., Brady, M., Smith, S. Improved optimization for the robust and accurate linear registration and motion correction of brain images. NeuroImage. 17 (2), 825-841 (2002).
- Smith, S. M. Fast robust automated brain extraction. Hum Brain Mapp. 17 (3), 143-155 (2002).
- Greve, D. N., Fischl, B. Accurate and robust brain image alignment using boundary-based registration. NeuroImage. 48 (1), 63-72 (2009).
- Patenaude, B., Smith, S. M., Kennedy, D. N., Jenkinson, M. A Bayesian model of shape and appearance for subcortical brain segmentation. NeuroImage. 56 (3), 907-922 (2011).
- Learn MATLAB Basics. , Available from: https://www.mathworks.com/support/learn-with-matlab-tutorials.html?s_tid=hp_ff_I_tutorials (2017).
- Lerma-Usabiaga, G., Iglesias, J. E., Insausti, R., Greve, D. N., Paz-Alonso, P. M. Automated segmentation of the human hippocampus along its longitudinal axis. Hum Brain Mapp. 37 (9), 3353-3367 (2016).
- Salami, A., Eriksson, J., Nyberg, L. Opposing effects of aging on large-scale brain systems for memory encoding and cognitive control. J Neurosci. 32 (31), 10749-10757 (2012).
- Schacter, D. L., Wagner, A. D. Medial temporal lobe activations in fMRI and PET studies of episodic encoding and retrieval. Hippocampus. 9 (1), 7-24 (1999).
- Strange, B., Dolan, R. Functional segregation within the human hippocampus. Mol Psychiatry. 4 (6), 508-511 (1999).
- Strange, B. A., Fletcher, P. C., Henson, R. N., Friston, K. J., Dolan, R. J. Segregating the functions of human hippocampus. Proc Natl Acad Sci U S A. 96 (7), 4034-4039 (1999).
- Eldridge, L. L., Engel, S. A., Zeineh, M. M., Bookheimer, S. Y., Knowlton, B. J. A dissociation of encoding and retrieval processes in the human hippocampus. J Neurosci. 25 (13), 3280-3286 (2005).
- Strange, B. A., Witter, M. P., Lein, E. S., Moser, E. I. Functional organization of the hippocampal longitudinal axis. Nat Rev Neurosci. 15 (10), 655-669 (2014).
- Zeineh, M. M., Engel, S. A., Thompson, P. M., Bookheimer, S. Y. Dynamics of the hippocampus during encoding and retrieval of face-name pairs. Science. 299 (5606), 577-580 (2003).
- Nieuwenhuis, S., Forstmann, B. U., Wagenmakers, E. -J. Erroneous analyses of interactions in neuroscience: a problem of significance. Nat Neurosci. 14 (9), 1105-1107 (2011).
- Cisler, J. M., Bush, K., Steele, J. S. A comparison of statistical methods for detecting context-modulated functional connectivity in fMRI. NeuroImage. 84, 1042-1052 (2014).
- Friston, K. J., Harrison, L., Penny, W. Dynamic causal modelling. NeuroImage. 19 (4), 1273-1302 (2003).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon