Method Article
Enrichissement phosphopeptide couplée à la spectrométrie masse Quantitative exempte d’étiquette d’enquêter sur le Phosphoproteome dans le Cancer de la Prostate
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit un procédé pour extraire et enrichir des phosphopeptides de lignées cellulaires de cancer de la prostate ou de tissus pour une analyse de la phosphoproteome via basé sur la spectrométrie de masse protéomique.
Résumé
Phosphoprotéomique implique l’étude à grande échelle des protéines phosphorylées. La phosphorylation des protéines est une étape critique dans nombreuses voies de transduction de signal et est étroitement contrôlée par les kinases et phosphatases. Par conséquent, caractérisant le phosphoproteome peut donner un aperçu identifier de nouvelles cibles et biomarqueurs pour la thérapie oncologique. Spectrométrie de masse permet globalement détecter et quantifier des milliers d’événements uniques de phosphorylation. Cependant, phosphopeptides sont beaucoup moins abondants que les non-phosphopeptides, faire une analyse biochimique plus difficile. Pour contourner cette limitation, les méthodes d’enrichissement des phosphopeptides avant l’analyse de spectrométrie de masse sont nécessaires. Nous décrivons une procédure pour extraire et de digérer les protéines du tissu pour produire des peptides, suivies d’un enrichissement de la phosphotyrosine (pY) et de peptides phosphosérine/thréonine (pST) en utilisant une base d’anticorps et/ou de dioxyde de titane (TiO2)-base méthode d’enrichissement. Après la préparation de l’échantillon et la spectrométrie de masse, nous a par la suite identifier et quantifier des phosphopeptides utilisant la spectrométrie de masse-chromatographie en phase liquide et logiciel d’analyse.
Introduction
Environ 165 000 nouveaux cas et environ 29 000 décès aura lieu en 2018 en raison du cancer de la prostate, ce qui représente le cancer le plus fréquent et la deuxième cause de décès liés au cancer chez les hommes dans les États-Unis1. Premiers stades du cancer de la prostate sont traitables avec résection ou rayonnement thérapie de maladie orgue confiné, où le taux de récurrence de dix ans est entre 20 % et 40 % pour les patients qui subissent une prostatectomie et entre 30 % et 50 % pour les patients qui reçoivent le rayonnement 2de thérapie. Parce que le cancer de la prostate repose sur des androgènes de signalisation pour la croissance, des thérapies de castration chirurgicale et chimiques sont également employées pour les patients à haut risque. Cependant, rechute se produit lorsque le cancer ne répond plus au traitement de privation androgénique comme en témoigne la récidive biochimique, où renaît l’antigène prostatique spécifique dans le sérum. À ce stade dans la progression, les métastases sont souvent détectés ainsi. Ce stade avancé, appelé cancer de la prostate métastatique résistant à la castration, représente la forme mortelle de la maladie où le pronostic est une durée médiane de survie inférieure à deux ans3. Il existe peu d’options thérapeutiques dans la maladie de stades, y compris la seconde génération anti-androgènes tels qu’enzalutamide et abiratérone, ainsi que de chimiothérapie à base de taxane comme docetaxel. Malgré les traitements disponibles, la maladie évolue souvent. Par conséquent, la découverte et le développement de nouveaux traitements sont nécessaires pour améliorer les soins des patients de cancer de la prostate avec une maladie avancée.
Spectrométrie de masse (MS)-selon les approches offrent une analyse globale du protéome grâce à la détection des centaines de milliers de peptide analytes4. En particulier, protéomique de découverte, une acquisition de données dépendantes (DDA), également connu sous le nom peut permettre l’identification et la quantification des milliers de peptides4,5. Protéomique axée sur le MS découverte peut être délimité davantage en protéomique haut vers le bas, où les protéines intactes sont caractérisés, et protéomique (également connu sous le nom de fusil) de bas en haut, où les peptides sont analysées afin de caractériser les protéines5. Ainsi, en protéomique de fusil de chasse, une étape de protéolyse se déroule dans la préparation de l’échantillon qui précède l’analyse MS en conjonction avec les protéines en peptides. À la fin, une recherche de base de données est exécutée pour remapper les peptides à des protéines pour l’identification. Label-free, ainsi que plusieurs isotopes-étiquetage [p. ex., isotope stable étiquetant par des acides aminés dans la culture de cellules (SILAC)] méthodes peuvent être utilisées pour comparer quantitativement les peptides entre échantillons6,7. Alors que les techniques de marquage isotopique sont l’étalon-or, exempte d’étiquette méthodes ont démontré similaires quantification exactitudes8,9 et ont compromis comparables entre la sensibilité et la spécificité de10. Quantification exempte d’étiquette fournit une plus grande couverture et permet des comparaisons entre les échantillons plus nombreux, alors que les méthodes basées sur les étiquettes sont limités par les coûts et multiplexage capacités6,7,8.
En outre, fusil MS peut être aussi utilisé pour interroger les modifications post-traductionnelles (PTMs) tel que la phosphorylation11. En raison de la nature inférieure stoechiométrique des phosphopeptides par rapport aux peptides totales, plusieurs méthodes sont utilisées pour enrichir des phosphopeptides, y compris à base d’anticorps immunoprécipitation des peptides de la phosphotyrosine (pY), dioxyde de titane (TiO2 ) et immobilisée en métal affinité chromatographie (IMAC)5,12. Parce que la phosphorylation des protéines est une étape clé dans plusieurs cell signaling pathways, fusil de chasse phosphoprotéomique permet aux chercheurs d’étudier les changements dans les différents cancers, notamment du sein13, la prostate14, rénale15, de la signalisation cellulaire et ovarien,16,17 afin de mieux comprendre la biologie du cancer et d’identifier de nouvelles cibles potentielles pour la thérapie.
Cette méthode de phosphoproteomic exempte d’étiquette de fusil de chasse a été construit et raffinée basée sur les travaux antérieurs de le Graeber groupe18,19,20. Ce protocole commence par décrire l’extraction et la digestion des protéines et des phosphoprotéines du tissu en peptides. Ensuite, nous détaillons l’enrichissement de pY peptides utilisant des anticorps spécifiques phosphotyrosine et TiO2. Nous décrivons également l’enrichissement des peptides phosphosérine/thréonine (pST) à l’aide de forte échangeuse de cations (SCX) suivie de TiO2. Ce protocole se termine par la présentation des échantillons à un laboratoire de MS et l’utilisation du logiciel d’analyse de MS pour identifier et quantifier des phosphopeptides et leurs phosphoprotéines correspondantes. L’application du présent protocole peut s’étendre au-delà de la prostate dans d’autres cancers et les champs en dehors de l’oncologie.
Protocole
Expériences utilisant des tumeurs de la xénogreffe ont été approuvées par le Comité de l’emploi et de Rutgers University Institutional Animal Care énoncés suite conformément aux lignes directrices de la National Institutes of Health.
1. Extraction des protéines
- Préparer le tampon de lyse (tableau 1). (Le volume dépend du nombre d’échantillons à récolter.) Pour in vitro des échantillons cellulaires, passez à l’étape 1.2. Pour le tissu tumoral, passez à l’étape 1.3.
-
Récolte des cellules
- Recueillir les cellules dans un tube conique de 50 mL et les essorer à 700 x g pendant 5 min à 4 ° C. Jeter le surnageant et garder le diabolo sur la glace. Répétez cette étape pour tous les plats recueillir les cellules par un plomb. (En général, environ 5 plats de 15 cm presque confluentes de cellules sont nécessaires pour 5 mg de protéine, mais cela peut dépendre de la lignée cellulaire et doit être déterminé empiriquement par chaque chercheur.)
- Laver le culot avec 30 mL de réfrigérés solution saline tamponnée au phosphate (PBS) et un essorage à 700 x g pendant 5 min à 4 ° C avant d’aspirer le PBS. Ajouter 1,5 mL de tampon de lyse par 5 mg de protéine utilisée pour le culot cellulaire. Pipette et descendre une couple de fois. Passez à l’étape 1.4.
-
Récolte de tissus
- Peser la tumeur et ajouter 2 mL de tampon de lyse glacée pour chaque 100 mg de tissu dans un tube de culture. (En règle générale, 50 à 150 mg de poids humide tissulaire est nécessaire).
- Homogénéiser le lysat utilisant un homogénéisateur à main ou sur table (impulsion 2 x pour 15 s.) Nettoyer l’homogénéisateur avant le premier échantillon et entre les échantillons à l’aide de 10 % eau de Javel, 70 % d’éthanol et d’eau déionisée dans la succession.
- Pour réduire et alkylat, chauffer les échantillons homogénéisés à 95 ° C pendant 5 min. Puis refroidir sur glace pendant 15 minutes. Sur la glace, laisser agir le lysat x 3 (p. ex., impulsions pendant 30 s à 60 s pauses entre les impulsions). L’échantillon ne devrait pas être à ce point visqueux ou grumeleux. Faire chauffer le lysat à 95 ° C pendant 5 min21.
- Centrifuger le lysat dans la sonication même tube à l’aide d’un rotor de seau swing à 3 500 g à 15 ° C pendant 15 min. récupérer le surnageant et jeter le culot.
- Déterminer la concentration de protéine en effectuant un essai de Bradford22. Si nécessaire, diluer le lysat à 5 mg/mL avec un tampon de lyse. Conserver à-20 ° C.
Remarque : L’expérience peut être suspendue ici. Congeler les échantillons à-80 ° C et poursuivre à une date ultérieure.
2. lysate Digestion
- Diluer l’échantillon 12 fois en utilisant 100 mM Tris (pH = 8,5) pour réduire la quantité de guanidinium. Diluer les échantillons tous sur le même volume pour minimiser les effets de la digestion inégale. Enregistrez 12,5 µg de la non digérés lysat pour le confirmer sur un gel teinté bleu de Coomassie23.
- Pour 5 mg de protéine, ajouter 10 µg de Lysyl Endopeptidase (Lys-C) et il incuber à température ambiante pour ajuster le pH 5-6 h. à 8.0 en ajoutant 1 M untitrated Tris (pH ~ 11).
- Préparer 1 mg/mL de L-1-tosylamino-2-phényléthyle chlorométhyl cétone (TPCK)-traité de la trypsine dans HCl à 1 mM (avec 20 mM CaCl2). Ajouter la trypsine dans une proportion de trypsine : protéine au 1/100 et il incuber à 37 ° C pendant 3 h.
- Ajouter la même quantité de trypsine fraîche comme au point 2.3. Incuber à 37 ° C pendant la nuit.
- Enregistrez 12,5 µg de la digéré lysat pour confirmer la digestion complète sur un gel teinté bleu de Coomassie23.
3. inverser l’Extraction en Phase
- Noter le volume de lysat. Filtrer l’échantillon en utilisant un filtre de coupure 15 mL 10 kDa. Centrifuger l’échantillon à 3500 x g par le rotor de seau swing (ou 3 500 g x dans un rotor à angle fixe) à 15 ° C jusqu'à ce que le volume de rétentat est inférieure à 250 µL (cela prend environ 45-60 min). Recueillir le cheminement et jeter le rétentat.
Remarque : L’expérience peut être suspendue ici. Congeler les échantillons à-80 ° C et poursuivre à une date ultérieure. - Pour acidifier l’échantillon, ajouter environ 20 µL de 5 % l’acide trifluoroacétique (TFA) par mL de lysate. Mélangez-les bien et mesurer le pH de l’échantillon à l’aide de bandelettes de pH. Ajuster le pH à 2,5 à l’aide de 5 % TFA.
- Branchez l’extrémité plus courte d’une colonne de C-18 à une tubulure de vide. Définir le vide entre 17 et 34 kPa (ou selon les instructions du fabricant). À l’aide de pipettes en verre , mouillez la colonne avec 3 mL de 100 % d’acétonitrile (ACN). Ne laissez pas la colonne sèche.
- À l’aide de pipettes en verre , équilibrer la colonne avec 6 mL de 0,1 % TFA appliqué que 2 x 3 mL. Charger l’échantillon acidifié dans la colonne. N’ajoutez pas plus de 3 mL à la fois. Ajuster le vide afin de cibler environ 1 à 2 gouttes par seconde.
- À l’aide de pipettes en verre , laver la colonne avec 9 mL de 0,1 % TFA a postulé 3 x 3 mL. Éluer la colonne avec 2 mL de 40 % ACN, 0,1 % TFA. Recueillir deux fractions de 2 mL dans des tubes de culture de verre . Jeter la colonne.
- Couvrir les tubes de l’éluat avec parafilm et percer des trous de 3-5 sur le couvercle à l’aide d’une aiguille de 20G. Geler l’éluat sur la glace sèche pendant au moins 30 min jusqu'à ce qu’il est complètement plein.
- Lyophiliser les fractions du jour au lendemain. Le lendemain, s’assurer que les échantillons soient complètement secs avant d’arrêter le lyophilisateur. Stocker les tubes dans un tube conique de 50 mL avec des lingettes délicats à-80 ° C.
Remarque : L’expérience peut être suspendue ici.
4. immunoprécipitation et enrichissement du pY Peptides24
- Resuspendre le poudre lyophilisée avec 0,5 mL de tampon de liaison glacee immunoprécipitation (IP) dans chacune des fractions. Mettre en commun les fractions en transférant le volume de remise en suspension de 0,5 mL de la fraction de seconde à la première fraction et sauver l’embout de la pipette. Vigoureusement les vortex (au lieu de pipetage de haut en bas) pour s’assurer que l’échantillon est complètement dissous avant de transférer à un cryotube de bouchon à vis 3,6 mL.
- Comme au point 4.1., rincer les tubes de lyophilisation avec un autre 0,5 mL de tampon de liaison IP (tableau 1) dans chaque tube. Transvaser la solution dans le tube avec bouchon à vis 3,6 mL en utilisant la même pipette pour minimiser toute perte d’échantillon. Répétez le rinçage 1 x plus, rendant la resuspension finale volume 2 mL (pour 5 mg de protéine). Mesurer le pH de l’échantillon pour s’assurer que c’est environ 7,4. Si c’est trop acide, itérativement ajouter 10µl de 1 M Tris (untitrated, pH ~ 11). Si c’est trop basique, itérativement ajouter 10µl de HCl dilué (01:25 ou au 1/100).
-
Prélaver les perles de pY (pour 5 mg de démarrage lysat)
- Il faut 25 µg d’anticorps 4 10 et 12,5 µg d’anticorps 27B10.4 par exemple. Après avoir utilisé un p200 pipette avec une pointe coupée à transférer les anticorps dans des tubes de microcentrifuge distinct, laver les anticorps avec 450 µL de tampon de liaison IP glacee 2 x. Les centrifuger à 100 g pendant 1 min à 4 ° C et aspirer par le surnageant.
- Remettre en suspension les perles à une concentration de stock de 0,5 mg/mL à l’aide du tampon de liaison IP. (Ne pas vortexer talons.) Après aliquotage la boue nécessaire (50 µL de lisier 10 4 anticorps et la boue de l’anticorps 27B10.4 de 25 µL par échantillon) dans un tube unique, tournez en bas des tubes à centrifuger stock à 200 x g pendant 1 min à 4 ° C. Laver les parois latérales avec surnageant avant de regagner les perles de stockage dans le réfrigérateur.
- Ajouter pY prélavée perles à la solution de l’échantillon remis en suspension dans les cryotubes bouchon à vis. Les incuber à 4 ° C sur une coiffe sur la fin du jour au lendemain.
- Placez les bouchon à vis cryotubes dans un tube à centrifuger 50 mL doublé avec une lingette délicate. Tournez en bas les perles à 100 g pendant 1 min. enregistrer le surnageant, qui servira à enrichir des peptides de pST. (L’enrichissement pour pST commence à l’étape 7 et peut être effectuée en parallèle au traitement peptide pY).
- Remettre en suspension les perles avec 300 µL de tampon de liaison IP. Transférez-les sur un tube de microcentrifuge de 2 mL et de les tourner vers le bas à 100 x g pendant 1 min à 4 ° C.
- Rincer l’incubation tube 3 x avec 200 µL de tampon de liaison IP. Transférer le contenu dans le tube de Microcentrifuge même chaque fois. Puis les tourner vers le bas.
- Laver les perles dans la microcentrifugeuse tube 3 x avec 500 µL de tampon de liaison IP et leur filer vers le bas à 100 x g pendant 1 min. Laver ensuite les perles 4 x 450 µl de 25 mM NH4HCO3pH 7,5 et essorage eux vers le bas à 100 g pendant 1 min. Utilisez un frais de 25 mM NH4HCO3 solution de poudre à chaque fois.
- Centrifuger les perles à 1 500 g pendant 1 min. utiliser une pointe de gel-chargement pour éliminer le liquide surnageant complètement en trempant l’extrémité de la pointe de gel-chargement légèrement sous la surface des perles.
- Ajouter 4 x le volume de perle de 0,1 % TFA aux talons (c'est-à-direajouter 300 µL de 0,1 % TFA pour 75 µg de lisier perle pY). Bien mélanger et laisser incuber le mélange dans un thermomixer à 1 000 tr/min pendant 15 min à 37 ° C.
- Transférer la remise en suspension dans un filtre de spin de 0,2 µm. Tournez en bas du tube d’élution et transférer le volume résiduel sur le même filtre de spin à l’aide d’une pipette de P10 rapidement. Centrifuger le filtre essorage à 850 x g pendant 1 min. transférer l’élution dans un tube de microcentrifuge de faible - la liaison aux protéines . Vide concentrer l’éluat jusqu'à dessiccation complète du jour au lendemain à 40 ° C et avec un temps de chaleur de 300 min.
Remarque : L’expérience peut être suspendue ici. Congeler les échantillons à-80 ° C et poursuivre à une date ultérieure.
5. dioxyde de titane enrichissement25 de pY Peptides
- Resuspendre le séchées bas phosphopeptides dans 200 µL de 50 % ACN, 0,1 % TFA. Vortex et centrifuger à 10 000 g pour 30 s. répéter ce 1 x pour remettre en suspension les bien.
- Préparer les perles de2 TiO contenues dans les conseils qui ont une capacité de 200 échantillons µL.
- Tapotez doucement sur le petit bout-côté de la pointe de déplacer le matériel à cette fin. Rincez l’embout en ajoutant 200 µL de 100 % ACN, suivi en inversant la pointe et en effleurant le petit bout pour déplacer le liquide vers le cap.
- À l’aide d’une lame de rasoir, couper le petit bout de la pointe et placez-le sur une faible liaison aux protéines tube. (Évitez d’utiliser des tubes de polystyrène comme le TiO2 s’en tiendra aux côtés du tube). Enlever le bouchon et insérer une micropipette de plonger à l’ACN restant. Répéter le lavage avec 200 µL de 100 % ACN. Les perles de TiO2 sont maintenant situés dans le tube de liaison aux protéines faible pour les étapes suivantes.
- Préconditionner TiO2 avec 500 µL d’ACN 100 % 2 x. Pipette à mélanger les billes avec le solvant. Les centrifuger à 100 g pendant 1 min.
- La condition de TiO2 500 µl de tampon de phosphate de sodium 0,2 M (pH ~ 7) x 2. Laver les billes avec 300 µL de l’équilibration du tampon x 3. TiO2 étant très dense, les perles seront déposent rapidement.
- Ajouter 400 µL de 50 % ACN, 0,1 % TFA dans le tube de liaison aux protéines faible, suivi par ajout 84 µL d’acide lactique. Transférer phosphopeptides resuspendues dans le tube de liaison aux protéines faible et les incuber pendant 1 heure à température ambiante à l’aide d’une coiffe sur la fin.
- Centrifuger les perles à 100 x g pendant 1 min pour les granuler. Lavez-les avec 300 µL de tampon de l’équilibration (tableau 1), 2 x et les tourner vers le bas à 100 x g pendant 1 min.
- Rincer les perles avec 300 µL de rinçage tampon 2 x. Transférez-les sur un filtre de spin de 0,2 µm. Les tourner à 1 500 g pendant 1 min.
- Transfert du filtre dans un tube de liaison aux protéines faible propre 1,5 mL. Éluer le contenu 2 x avec 200 µL de 0,9 % NH3 H2O. mesure du pH avec des bandelettes de pH, qui devrait se situer entre 10 et 11. Vide me concentrer l’éluat jusqu'à siccité du jour au lendemain pour évaporer l’ammoniac.
6. dessalement pY Peptides pour Analyses MS
- Reconstituer les phosphopeptides 15 µl de 0,1 % TFA par Vortex et eux centrifugation à 10 000 g pendant 30 s pour remettre en suspension les. Répétez cette 1 x pour remettre en suspension les bien. Ne pas pipeter en haut et en bas.
- Nettoyer à l’aide d’une pointe de C-18 avec une capacité de liaison de 5 µg et suivre le protocole du fabricant.
- Sécher complètement le volume d’élution par concentration sous vide. Cela prend 1-2 h. Resuspendre phosphopeptides séchées dans 12,5 µL de solution de spectrométrie de masse (voir tableau 1) (ou tel que recommandé par MS du chercheur protéomique core facility). Vortex et essorer brièvement la solution vers le bas à 10 000 g pendant 30 s. répéter ce x 2 remettre en suspension les bien. Les échantillons sont prêts à être soumis à une installation de spectrométrie de masse (étape 11).
Remarque : Les étapes suivantes ci-dessous sont liées à l’enrichissement de peptide pST seulement.
7. inverser la Phase d’Extraction des Peptides pST
- Mesurer la concentration de peptide de surnageant a acquis de l’étape 4,6 en effectuant un essai de peptide. Une quantité suffisante pour la spectrométrie de masse de pST est de 2,5 mg.
- Ajuster le pH de 3,5 à 5 % TFA.
- Branchez l’extrémité plus courte d’une colonne de C-18 à une tubulure de vide. Définir le vide entre 17 et 34 kPa (ou selon les instructions du fabricant). Mouiller la colonne avec 3 mL de 100 % ACN. Ne laissez pas la colonne sèche.
- Equilibrer la colonne avec 6 mL de 0,1 % TFA appliqué que 2 x 3 mL. Charger l’échantillon acidifié dans la colonne. N’ajoutez pas plus de 3 mL à la fois. Ajuster le vide afin de cibler environ 1 à 2 gouttes par seconde.
- Laver la colonne avec 9 mL de 0,1 % TFA a postulé 3 x 3 mL. Éluer la colonne avec 2 mL de 40 % ACN, 0,1 % TFA. Recueillir deux fractions de 2 mL dans des tubes de culture de verre. Jeter la colonne.
- Couvrir les tubes de l’éluat avec parafilm et percer des trous de 3-5 sur le couvercle à l’aide d’une aiguille de 20G. Geler l’éluat sur la glace sèche pendant au moins 30 min jusqu'à ce qu’il est complètement plein.
- Lyophiliser les fractions sélectionnées du jour au lendemain. Le lendemain, s’assurer que les échantillons soient complètement secs avant d’arrêter le lyophilisateur. Stocker les tubes dans un tube conique de 50 mL avec des lingettes délicats à-80 ° C.
Remarque : L’expérience peut être suspendue ici.
8. fort échangeuse de cations (SCX) des Peptides pST
- Remettre en suspension les peptides lyophilisées dans 2 mL de tampons A (tableau 1). Mettre en commun les fractions pour chaque échantillon. (La solution sera nuageuse).
- Préparer la tubulure de vide. Connecter une colonne SCX à une seringue de 3 mL avec le plongeur supprimé. Définir le vide entre 17 et 34 kPa (ou selon les instructions du fabricant).
- Conditionner la colonne SCX avec 4 mL d’ACN, suivie de 4 mL de tampon A.
- Charger les 2 mL de l’échantillon de l’étape 8.1 et recueillir l’éluat immédiatement. Charge de 3 mL de tampon d’a : b (80.9:19.1) et recueillir l’éluat. Centraliser les éluats de chaque échantillon et aliquote eux dans tubes de 2 mL basse-la liaison aux protéines.
- Vide se concentrent tous les échantillons jusqu'à environ 30 % du volume. (Cette étape prend environ 2-4 h.) Centraliser les aliquotes dans 1 tube de liaison aux protéines faible pour chaque échantillon.
- Branchez l’extrémité plus courte d’une colonne de C-18 à une tubulure de vide. Définir le vide entre 17 et 34 kPa (ou selon les instructions du fabricant). Mouiller la colonne avec 3 mL de 100 % ACN 2 x. Faire pas laissez la colonne sèche.
- Équilibrer la colonne avec 3 mL de 0,1 % TFA 2 x. Charger l’échantillon dans la colonne. N’ajoutez pas plus de 3 mL à la fois. Ajuster le vide afin de cibler environ 1 à 2 gouttes par seconde.
- Laver la colonne avec 3 mL de 0,1 % TFA 2 x. Éluer la colonne avec 4 mL de 50 % ACN, 0,1 % TFA.
9. dioxyde de titane enrichissement des Peptides pST
-
Préparer les perles de2 TiO contenues dans les conseils qui ont une capacité de 200 échantillons µL
- Tapoter légèrement sur le côté de la petite pointe de la pointe pour déplacer les billes à cette fin. Enlever le bouchon et verser les billes dans un tube conique polypropylène 15 mL.
- Rincez l’embout en ajoutant 200 µL de 100 % ACN, inversant la pointe une couple de fois et appuyer sur le petit bout pour déplacer le liquide vers le cap. À l’aide d’une lame de rasoir, couper le petit bout de la pointe et placez-le sur le tube à fond conique polypropylène 15 mL. Enlever le bouchon et insérer une micropipette de plonger à l’ACN restant. Répéter le lavage avec 200 µL de 100 % ACN. Les perles de2 TiO sont maintenant situés dans le tube à fond conique 15 mL pour les étapes suivantes.
- Préconditionner TiO2 avec 500 µL d’ACN 100 % 2 x. Pipette à mélanger les billes avec le solvant. Les centrifuger à 100 g pendant 1 min.
- La condition de TiO2 500 µl de tampon de phosphate de sodium 0,2 M (pH ~ 7) deux fois. Laver les billes avec 300 µL de l’équilibration du tampon x 3.
- Transférer les phosphopeptides éluées dans le tube conique polypropylène 15 mL. Ajouter 560 µL d’acide lactique et il incuber pendant 1 heure à température ambiante à l’aide d’une coiffe sur la fin.
- Centrifuger le mélange à 100 x g pendant 1 min granuler les perles. Lavez-les avec 300 µL de tampon de l’équilibration (tableau 1), 3 x. Elles tournent vers le bas à 100 x g pendant 1 min.
- Rincer les perles avec 300 µL de rinçage tampon 2 x. Transférez-les sur un filtre de spin de 0,2 µm. Tourner vers le bas à 1 500 g pendant 1 min.
- Transfert du filtre dans un tube de liaison aux protéines faible propre 1,5 mL. Éluer le contenu 2 x avec 200 µL de 0,9 % NH3 H2O. Laisser la solution sur phosphopeptides pendant 2 min avant leur élution. Mesurer le pH, ce qui devrait se situer entre 10 et 11.
- Vide me concentrer l’éluat jusqu'à siccité du jour au lendemain pour évaporer l’ammoniac.
10. dessalage des Peptides de pST pour les Analyses de MS
- Tapotez doucement sur le petit bout-côté de la pointe de déplacer le matériel à cette fin. Rincez l’embout en ajoutant 200 µL de 100 % ACN, suivi en inversant la pointe et en effleurant le petit bout pour déplacer le liquide vers le cap.
- À l’aide d’une lame de rasoir, couper le petit bout de la pointe et placez-le sur un tube conique polypropylène 15 mL. Enlever le bouchon et insérer une micropipette de plonger à l’ACN restant. Répéter le lavage avec 200 µL de 100 % ACN. Les perles de TiO2 sont maintenant situés dans le tube à fond conique polypropylène 15 mL pour les étapes suivantes.
- Nettoyer à l’aide d’une pointe de C-18 avec une capacité de fixation de 100 µg. (suivre les instructions du fabricant.)
- Sécher complètement le volume d’élution par concentration sous vide. Cela prend 1-2 h.
- Resuspendre les phosphopeptides séchées dans 12,5 µL de solution de spectrométrie de masse (ou tel que recommandé par l’installation de base du chercheur MS protéomique). Vortex et les centrifuger à 10 000 x g pendant 30 s. répéter 2 x pour remettre en suspension les bien. (Ne pas pipeter haut et en bas.)
11. mass Spectrometry analyse
- Soumettre les échantillons à l’installation de base MS protéomique pour effectuer liquide MS chromatographie en tandem (LC-MS/MS) à l’aide de leurs paramètres recommandés. Paramètres de l’exemple sont les suivantes (voir tableau 2 pour le résumé) :
- Charger 5 µL des échantillons sur une colonne de piège (2 cm long x 75 µm de diamètre) et lavez-les avec 0,1 % TFA pendant 5 min avec un débit de 5 µL/min.
- Apporter le piège en ligne avec une colonne analytique nano (20 cm x 75 µm) avec un débit de 300 nL/min.
- Les dégradés linéaires segmentés (pourcentage d’acide formique de 0,16 %, 80 % ACN à 0,2 % d’acide formique) diffèrent entre les échantillons pY et la TVP :
- Pour les échantillons de pY, éluer à l’aide d’un dégradé de 4 à 15 % en 5 min, 15-50 % en 40 min et 50-90 % en 5 min.
- Pour les échantillons de pST, éluer à l’aide d’un dégradé de 4 à 15 % en 30 min, 15-25 % en 40 min, 25 à 50 % en 44 min et 50-90 % en 11 min.
- Acquisition de données de MS en mode d’acquisition de données-dépendante avec une série cyclique d’un scan complet avec une résolution de 120 000 suivi par SM/SM (HCD, énergie de collision relative de 27 %) des 20 ions plus intenses et une durée de dynamique d’exclusion de 20 s.
- Après la MS exécuter achèvement, importer les fichiers raw de MS dans un logiciel d’analyse MS pour identifier et quantifier des phosphopeptides. (MaxQuant logiciel8,26,27 a été utilisé dans cette expérience. Sauf si spécifié dans le tableau 3, les paramètres par défaut ont été utilisés.)
Résultats
Ce protocole décrit en détail une méthode d’extraction de protéine et digestion suivie d’enrichissement de le phosphopeptide et l’analyse ultérieure de MS (Figure 1). Les compositions de tous les tampons et les solutions qui sont utilisés dans le présent protocole sont énumérées au tableau 1. L’utilisation séquentielle de Lys-C et la trypsine fournit une digestion efficace. Un coloration bleu de Coomassie gel de prédigéré lysat confirme la présence de protéines, tandis que la coloration des études digérées lysat confirme la digestion complète (Figure 2 a). Pour une digestion complète, aucune bande ne doit apparaître au-dessus de 15 kDa, sauf les 30 kDa et 23,3 kDa bandes pour Lys-C et la trypsine, respectivement. L’ajout de Lys-C réduit également le nombre des clivages manquées (Figure 2 b). Parce que les peptides pY ne représentent que 2 % du phosphoproteome28, immunoprécipitation des peptides pY à l’aide d’un anticorps spécifique pY est la première étape de l’enrichissement de peptide de pY. Le surnageant qui en résulte devient l’entrée pour l’enrichissement de peptide pST. L’immunoprécipitation pY sépare efficacement pY peptides de peptides pST où en moyenne 85 % des phosphopeptides identifiées dans la préparation de pY sont pY (Figure 3 a) et plus de 99 % des phosphopeptides identifiées dans la préparation de pST sont pST (Figure 3 b). Dioxyde de titane est utilisé pour enrichir des phosphopeptides dans les deux préparations. Le pourcentage prévu de peptides dans la préparation prête à MS qui sont phosphorylées est entre 30 et 50 % (Figure 4 a). La variabilité du pourcentage d’enrichissement phosphopeptide peut être supérieure à la préparation de pY à la suite qu’il y ait beaucoup moins pY peptides que les peptides de pST. En ce qui concerne les espèces de phosphopeptide, la plupart des phosphopeptides détectés ont un groupe phosphoryle simple ou double (Figure 4 b).
Après avoir effectué la spectrométrie de masse, les fichiers raw de MS sont chargés dans un logiciel d’analyse de MS. Le réglage des paramètres utilisé dans l’expérience est répertorié dans le tableau 3 mais variera de logiciel à logiciel et peut-être varier d’une version à l’autre. Les paramètres qui ne sont pas répertoriés ont été laissés comme par défaut, y compris un FDR coupure de 1 % pour le peptide-spectre correspondant (PSM) avec une note minimale de Andromeda de 40 pour l’identification des peptides modifiés27. Définissant un seuil de probabilité de localisation de plus de 0,75 filtre dehors environ 5 % des peptides pY et 15 % et 34 % de la pS et la pT peptides, respectivement (Figure 5 a). Après l’application de ces filtres, le nombre d’identifications phosphopeptide à la fin de l’analyse de MS est environ 300 pY peptides (pour 5 mg de la protéine de départ) et environ 7 500 peptides pS et peptides pT 640 (pour 2,5 mg du peptide départ montant) de la préparation d’enrichissement respectifs (Figure 5 b). Le nombre de répétitions et de la variabilité de l’intensité du signal phosphopeptide déterminent l’alimentation adéquate pour les comparaisons statistiques. Dans quatre expériences distinctes avec les groupes contenant des doublons biologiques ou réanalysés, les coefficients de pourcentage de variation (% CV) pour phosphopeptides détectés ont été calculées. Les distributions de variabilité moindre (p. ex., groupes de pST 1-5 dans la Figure 5) indiquent que le prélèvement, préparation et spectrométrie de masse séries était conforme. En revanche, les distributions de variabilité plus élevée (p. ex., groupe pST 6 dans la Figure 5) indique des données plus bruyantes qui exigeraient plus grands pli-modifications pour détecter des différences significatives dans les analyses différentielles en aval.
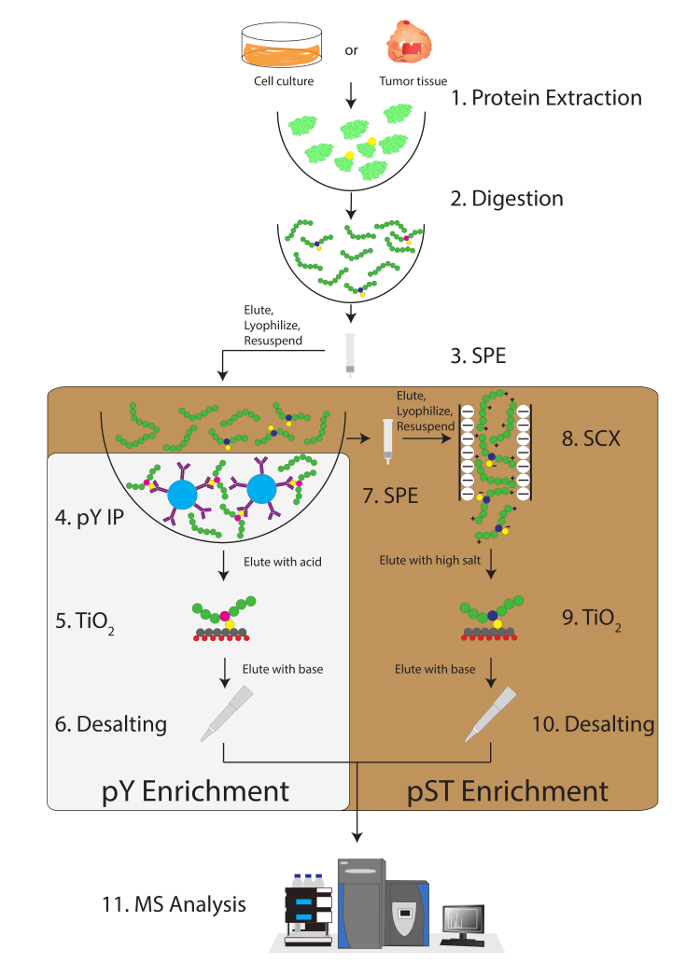
Figure 1 : diagramme de flux de travail. Les protéines provenant d’échantillons sont extraites et digérés. Peptides sont obtenus par extraction en phase solide (SPE), et les peptides phosphotyrosine (pY) sont immunoprécipitée. En parallèle, les peptides phosphosérine/thréonine (pST) sont enrichis du surnageant à l’étape de l’immunoprécipitation de pY. Échange de cations fort (SCX) est effectuée sur le surnageant pour enlever les peptides survoltées pour réduire la suppression d’ion12. Les deux préparations subissent phosphopeptide enrichissement via dioxyde de titane (TiO2). Après nettoyage de l’échantillon, liquide chromatography-spectrométrie de masse (LC-MS/MS) est effectuée afin de mesurer l’abondance phosphopeptide. Les données brutes sont ensuite chargées dans un logiciel d’analyse de MS pour identifier des phosphopeptides. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
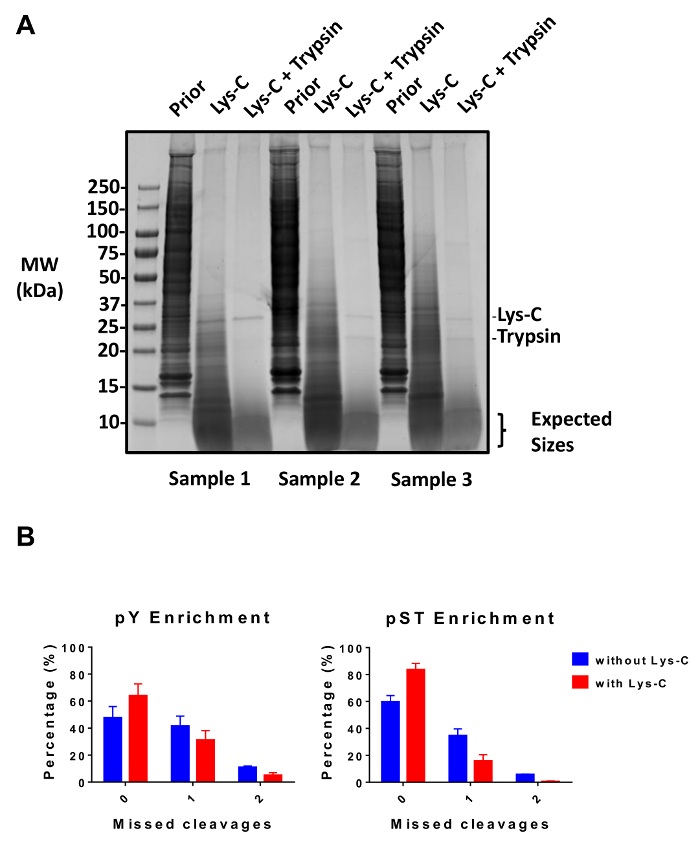
Figure 2 : évaluation de la digestion. (A) les trois échantillons au moyen de 12,5 µg de lysate digestion prédigestion, post-Lys-C, et la digestion trypsique après apparaissent. Un test de gel-tache bleu de Coomassie révèle une digestion propre après utilisation séquentielle de Lys-C et la trypsine. Les marqueurs de taille masse moléculaire (mm) sont en kilodaltons (kDa). (B) A réduction de clivages manquées est observée après Lys-C a été ajoutée au protocole. Le pourcentage des phosphopeptides sans clivages manqués ont augmenté de 48 % à 64 % et de 60 % à 84 % en moyenne pour les préparations d’enrichissement pY et la TVP, respectivement. Les graphiques résument les données obtenues des deux expériences réalisées sans Lys-C et cinq expériences réalisées avec Lys-C. Les barres d’erreur sont les écarts-types représentant pY 38 et 38 échantillons de pST de 2 expériences distinctes (sans Lys-C) et pY 62 60 échantillons de pST de 5 expériences distinctes (avec Lys-C). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
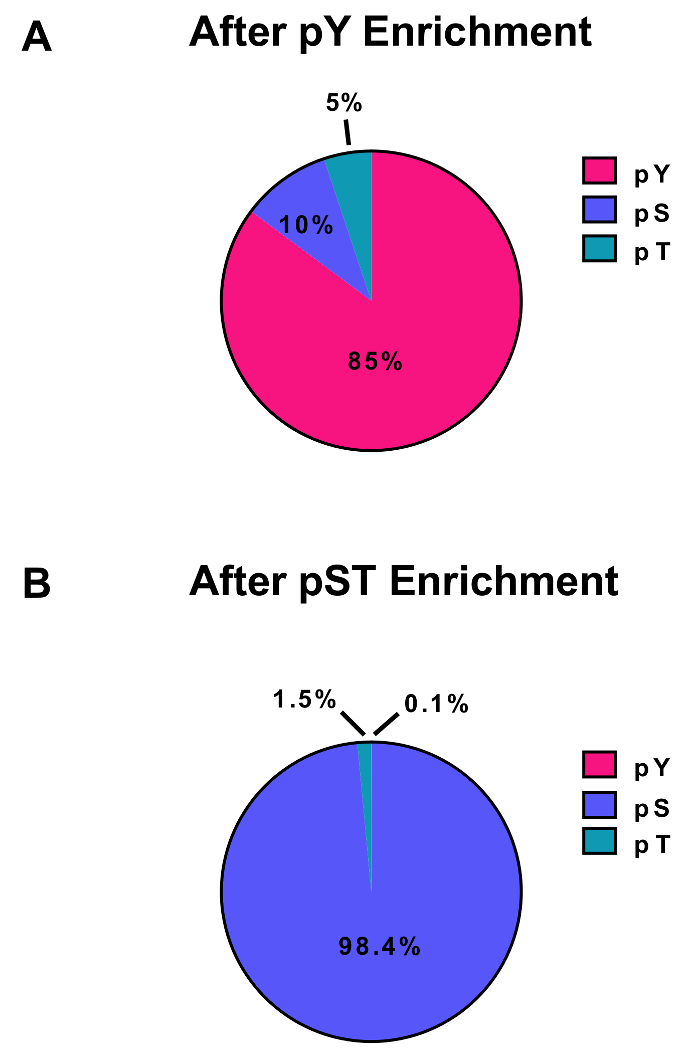
Figure 3 : enrichissement des phosphopeptides pY et pST. Ces panneaux indiquent les pourcentages des phosphopeptides pSTY de soit (A) le pY ou (B) les préparatifs de l’enrichissement de pST. L’enrichissement de pY par immunoprécipitation de la pY et dioxyde de titane résultait phosphopeptides 85 % étant des peptides de pY, tandis que seulement 0,1 % des phosphopeptides dans l’enrichissement de pST sont pY. Ces valeurs ont été tirées examinant la Phospho (fichier de STY)Sites.txt d’une expérience représentative après filtrage des contaminants et des séquences inversées phosphopeptides les probabilités de localisation inférieure à 0,75. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
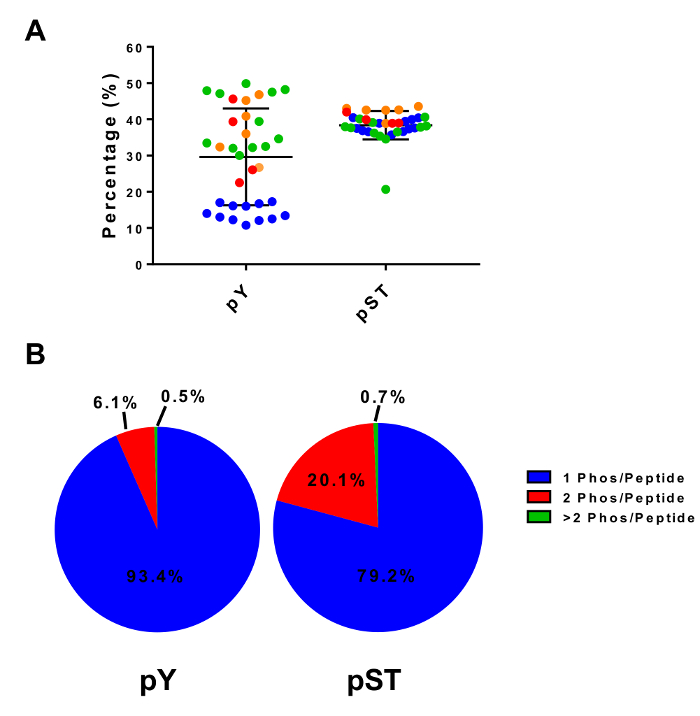
Figure 4 : enrichissement de Phosphopeptide avec le dioxyde de titane. (A) montre le pourcentage des phosphopeptides détecté (par rapport aux peptides totales) des échantillons de quatre expériences distinctes. (B), ce panneau affiche la composition moyenne de mono, double et les peptides phosphorylés multi dans quatre expériences distinctes. Les barres d’erreur dans le groupe A sont les écarts-types. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
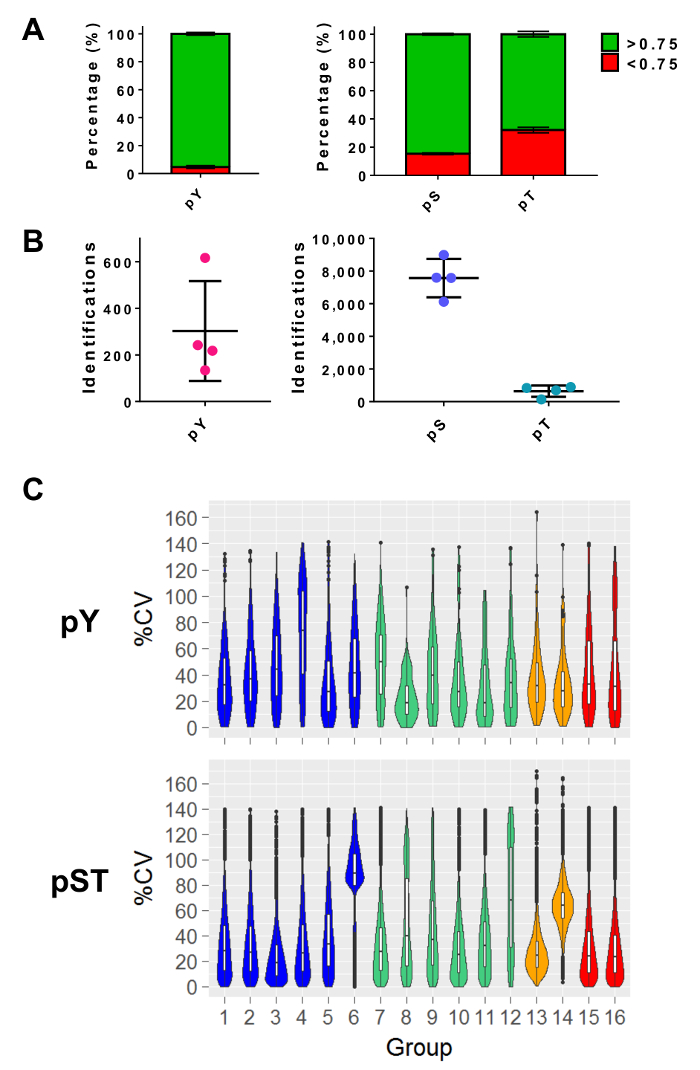
Figure 5 : prévu identifications phosphoresidue. (A), ce panneau indique les probabilités de localisation de phosphorylation des ID de pY enrichissement (à gauche) et l’enrichissement de pST (à droite). Le pourcentage moyen des identifiants qui respectent le seuil de probabilité > 0,75 est de 93 %, 75 % et 52 % pour pY, pS et pT, respectivement. (B) la moyenne nombre d’ID avec un > 0,75 localisation probabilité est 300 pour pY, 7 500 pour le pS et 640 pour pT. (C), ce panneau montre les parcelles de violon du coefficient de variation (% CV) des phosphopeptides pourcentage. Une évaluation du % de CV a été effectuée seulement si une valeur d’intensité de signal a été détectée dans chaque réplicat biologique ou d’un groupe en triple. Données sont issues de quatre expériences distinctes. Les barres d’erreur dans les groupes A et B sont les écarts-types de pY 34 et 34 échantillons de pST de 4 expériences distinctes. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Mémoire tampon | Volume | Composition | |
| Mémoire tampon lyse de chlorure guanidinium 6 M | 50 mL | Chlorure de guanidinium 6 M, 100 mM tris pH 8,5, phosphine de tris (2-carboxyethyl) de 10 mM, 40 mM chloroacétamide, orthovanadate de sodium 2 mM, pyrophosphate de sodium 2,5 mM, 1 mM β-glycérophosphate, 500 mg n-octyl-glycoside ultra pure eau au volume | |
| pyrophosphate de sodium 100 mM | 50 mL | pyrophosphate de sodium décahydraté de 2,23 g, eau ultra pure volume | |
| Β-glycérophosphate de 1M | 50 mL | 15,31 g β-glycérophosphate, eau ultra pure volume | |
| 5 % d’acide trifluoroacétique | 20 mL | Ajouter 1 mL d’acide trifluoroacétique 100 % dans 19 mL d’eau ultra pure | |
| 0,1 % d’acide trifluoroacétique | 250 mL | Ajouter 5 mL d’acide trifluoroacétique 5 % à 245 mL d’eau ultra pure | |
| tampon d’élution pY | 250 mL | 0,1 % trifluoroacétique acide, 40 % acétonitrile, eau ultra pure volume | |
| tampon d’élution pST | 250 mL | 0,1 % trifluoroacétique acide, 50 % acétonitrile, eau ultra pure volume | |
| Tampon de liaison IP | 200 mL | 50 mM tris de pH 7,4, 50 mM de chlorure de sodium, eau ultra pure à volume | |
| 25 mM, bicarbonate d’ammonium, pH 7.5 | 10 mL | Dissoudre 19,7 mg dans 10 mL d’eau ultra pure stérile, pH de 7,5 à l’acide chlorhydrique 1 N (~ 10-15 µL/10 ml de solution), faire des frais | |
| 1M tampon phosphate à pH 7 | 1 000 mL | dihydrogénophosphate de sodium des 1 M 423 mL, mL 577 1 M sodium hydrogénophosphate | |
| Tampon d’équilibration | 14 mL | 6,3 mL d’acétonitrile, 280 acide trifluoroacétique 5 % µL, 1740 µL d’acide lactique, 5,68 mL d’eau ultra pure | |
| Tampon de lavage | 20 mL | 9 mL d’acétonitrile, 400 acide trifluoroacétique 5 % µL, 10,6 mL d’eau ultra pure | |
| Solution de spectrométrie de masse | 10 mL | 500 µL acétonitrile, 200 µL 5 % d’acide trifluoroacétique, 9,3 mL d’eau ultra pure | |
| Tampon A | 250 mL | phosphate monopotassique de 5 mM (pH 2,65), 30 % d’acétonitrile, chlorure de potassium de 5 mM, eau ultra pure volume | |
| Mémoire tampon B | 250 mL | phosphate monopotassique de 5 mM (pH 2,65), 30 % d’acétonitrile, 350 mM chlorure de potassium, eau ultra pure volume | |
| 0,9 % d’hydroxyde d’ammonium | 10 mL | 300 μL 29.42 % hydroxyde d’ammonium, 9,7 mL d’eau ultra pure | |
Tableau 1 : tampons et solutions de. Ce tableau montre les compositions des tampons et solutions utilisées dans le présent protocole.
| Paramètres de LC-MS/MS | ||
| Paramètre | pY réglage | pST réglage |
| Exemple de chargement (µL) | 5 | |
| Débit de chargement (µL/min) | 5 | |
| Débit dégradé (nL/min) | 300 | |
| Dégradé linéaire (pourcentage 0,16 % d’acide formique, 80 % ACN à 0,2 % d’acide formique) | 4 - 15 % pendant 5 min | 4 - 15 % pendant 30 min |
| 15 - 50 % pendant 40 min | 15 - 25 % pendant 40 min | |
| 50 - 90 % pendant 5 min | 25 - 50 % pour les 44 min | |
| 50 - 90 % pendant 11 min | ||
| Résolution de numérisation complète | 120 000 | |
| Nombre d’ions plus intenses sélectionnés | 20 | |
| Énergie de collision relative (%) (HCD) | 27 | |
| Exclusion de dynamique (s) | 20 | |
Tableau 2 : paramètres de LC-MS. Il s’agit d’un exemple de paramètres de LC-MS lors d’une expérience de phosphoproteomic typique de fusil de chasse. Les échantillons ont été chargées sur une colonne de piège. Le piège a été mis en ligne avec une colonne analytique. Ces paramètres ont été optimisés pour utiliser le système de SM-répertorié dans Table des matières et réactifs. Ces paramètres devront être ajustés pour d’autres systèmes de LC-MS.
| MaxQuant des paramètres | ||
| Réglage | Action | |
| Paramètres spécifiques à un groupe | ||
| Type de | Type de | Sélectionnez Standard |
| Multiplicité | La valeur 1 | |
| Mode de digestion | Enzyme | Sélectionnez la trypsine/P |
| Max. clivages manqués | La valeur 2 | |
| Modifications | Modification de variable | Ajouter Phospho (STY) |
| Quantification d’étiquette-gratuit | Quantification d’étiquette-gratuit | Sélectionnez LFQ |
| Comte de ratio LFQ min. | La valeur 1 | |
| LFQ rapide | Cochez les | |
| Divers | Re-quantifier les | Cochez les |
| Paramètres globaux | ||
| Séquences | Fichiers FASTA | Fasta sélectionnez fichier téléchargé depuis UniProt |
| Modifications fixes | Ajouter Carbamidomethyl (C) | |
| Identification de l’ADV. | Match de football entre les deux s’exécute | Cochez les |
| Fenêtre de temps de correspondance | A 5 min | |
| Fenêtre de temps de l’alignement | La valeur 20 min | |
| Correspondent à des caractéristiques non identifiés | Cochez les | |
| Quantification des protéines | Comte de rapport / min. | La valeur 1 |
| Emplacements des dossiers | Modifier en conséquence | |
Tableau 3 : paramètres d’analyse du logiciel MS. Dans MaxQuant, les paramètres spécifiques à un groupe et globales dans ce tableau ont été sélectionnés ou ajustés. Tous les autres paramètres sont restés à défaut. Ces expériences ont été réalisées à l’aide de la version 1.5.3.30. Les paramètres peuvent varier d’une version à l’autre et d’un logiciel à un logiciel.
Discussion
Avant d’utiliser ce protocole pour enrichir des phosphopeptides, un examen attentif du plan expérimental est critique. À l’aide de réplicats biologiques est une utilisation plus rentable des ressources de la spectrométrie de masse que les répétitions techniques. Le nombre de répétitions nécessaires dépendra en partie de la variabilité des données. Une étude récente a démontré que, tout en augmentant le nombre de répétitions en sus de la troisième que marginalement augmente le nombre d’identifications, le nombre d’identification significative entre groupes augmente plus réplique10.
En raison de la plus faible abondance des phosphoprotéines dans la cellule, une quantité suffisante de protéine départ est nécessaire pour obtenir un phosphoproteome global des échantillons de cancer de la prostate en mode découverte. Dans ces expériences, 5 mg de protéine a été utilisé. Environ cinq plats de 15 cm presque confluentes de cellules fournissent suffisamment de protéines comme entrée dans ce protocole, bien qu’il s’agit de cellule dépendante de ligne. En ce qui concerne le tissu tumoral, le rendement de protéine est environ 6 à 8 % du poids du tissu. Dans le milieu in vitro , un échantillon de contrôle positif à considérer est l’ajout de vanadate de 1 mM pour 30 min avant la récolte des cellules. Vanadate, un inhibiteur de la phosphatase concurrentiel protéine phosphotyrosyl, préservera la phosphorylation de la tyrosine, augmentant ainsi le nombre de pY peptide identifications29.
La digestion propre est une étape clé pour optimiser l’identification phosphopeptide. En plus de l’essai de tache de bleu de Coomassie, le pourcentage des clivages manquées dans les données peut servir à évaluer l’efficacité de la digestion (Figure 2). Logiciel de contrôle de qualité est disponible qui analyse les clivages manquées et autres mesures pour évaluer les données de MS qualité30. Alors que la trypsine est que les plus courantes, d’autres protéases sont disponibles5 pour combler les lacunes couverture dans le protéome où des peptides tryptiques optimales ne peut pas être généré31. Les paramètres du logiciel d’analyse MS devra alors être modifiée en conséquence pour s’ajuster à des changements dans les protéases.
Le protocole utilise immunoprécipitation (pour l’enrichissement de pY) ainsi que le dioxyde de titane (TiO2) afin d’enrichir des phosphopeptides. Les approches alternatives pour enrichir des peptides incluent chromatographie d’affinité métallique immobilisée (IMAC), autres oxydes métalliques pour la chromatographie d’affinité métallique (MOAC) tels que l’hydroxyde d’aluminium et de capture d’affinité d’ion métallique à base de polymères (PolyMAC) 5,,12. Des études antérieures ont montré que les méthodes d’enrichissement différents enrichissent pour différentes populations des phosphopeptides32. Par exemple, IMAC enrichit plusieurs peptides phosphorylés multi tandis que MOAC préférence enrichit de peptides phosphorylés mono33. Les Résultats de représentant du présent protocole reflètent cette observation (Figure 4 b). Une publication récente a démontré que combinant IMAC et Mac en utilisant un matériau hybride pourrait fournir éventuellement une plus grande couverture de phosphopeptide espèces34. Ainsi, le présent protocole peut être modifié pour utiliser d’autres méthodes d’enrichissement en parallèle pour permettre des analyses phosphoproteomic encore plus complètes.
La suite logicielle de MaxQuant26 est utilisée pour analyser les données de MS dans le présent protocole, mais les applications commerciales35 sont également disponibles pour la quantification et identification de phosphopeptide. Pour l’identification de phosphopeptide, une probabilité de localisation coupure est appliquée. Ce filtre est effectué pour sélectionner des phosphopeptides avec un degré de confiance élevé (c'est-à-diresupérieur à 0.75) l’identification de phosphoresidue10,28. En d’autres termes, la probabilité cumulée de tous les autres résidus qui pourraient contenir le phospho-groupe est inférieure à 0,25. Ce seuil de décision pourrait être augmenté afin d’accroître la rigueur de la sélection de phosphopeptide. En ce qui concerne le nombre d’identifications, le nombre de peptides de pY est dans les centaines, tandis que le nombre de peptides pST est dans les milliers de hauts. Ces valeurs reflètent déjà observée phosphoproteome distribution où environ 2 %, 12 % et 86 % de la phosphosites sont pY, pT et pS, respectivement28.
Si les étapes d’enrichissement pY et la TVP sont exécutés en parallèle, les étapes de préparation d’échantillon dans le protocole peuvent être complétées en six jours. En s’associant avec le puissant outil de MS, protocoles d’enrichissement phosphopeptide comme celui-ci offrent une approche globale pour les scientifiques recueillir des données pour analyser le phosphoproteome dans leurs domaines respectifs.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions les membres du laboratoire de Drake pour fournir des conseils et des commentaires sur le manuscrit. Nous remercions également les membres du biologique masse spectrométrie installation de Robert Wood Johnson Medical School et Rutgers, The State University of New Jersey, pour donner des conseils et exécution de spectrométrie de masse sur nos échantillons. Larry C. Cheng est pris en charge par le National Institute of General Medical Sciences, de la National Institutes of Health, sous le numéro prix T32 GM008339. Thomas G. Graeber est pris en charge par le NCI/NIH (des spores dans le Cancer de la Prostate P50 CA092131 ; P01 CA168585) et l’American Cancer Society Research Scholar Award (RSG-12-257-01-TBE). Justin M. Drake est pris en charge par le ministère de la défense Prostate Cancer recherche programme W81XWH-15-1-0236, Prostate Cancer Foundation Young Investigator Award, la Fondation de santé du New Jersey et une précision médecine Initiative pilote Award de la Rutgers Cancer Institute du New Jersey.
matériels
| Name | Company | Catalog Number | Comments |
| Ultra-Low Temperature Freezer | Panasonic | MDF-U76V | |
| Freezer -20 °C | VWR | scpmf-2020 | |
| Swing rotor bucket | ThermoFisher Scientific | 75004377 | |
| Vacuum manifold | Restek | 26080 | |
| Lyophilizer | Labconco | 7420020 | |
| CentriVap Benchtop Vacuum Concentrator | Labconco | 7810010 | |
| End-over-end rotator | ThermoFisher Scientific | 415110Q | |
| Razor blade | Fisher Scientific | 620177 | |
| Amicon Ultra-15 Centrifugal Filter Units | Millipore Sigma | UFC901024 | |
| Glass culture tubes | Fisher Scientific | 14-961-26 | |
| Parafilm | Fisher Scientific | 13-374-12 | |
| 20G needle | BD | B305175 | |
| Kimwipes | Fisher Scientific | 06-666A | |
| Screw cap cryotube | ThermoFisher Scientific | 379189 | |
| Nunc 15 mL conical tubes | ThermoFisher Scientific | 12-565-268 | |
| Gel loading tips | Fisher Scientific | 02-707-181 | |
| Millipore 0.2 µm spin filter | Millipore Sigma | UFC30GVNB | |
| Low protein-binding Eppendorf tubes | Eppendorf | 22431081 | |
| anti-Phosphotyrosine, Agarose, Clone: 4G10 | Millipore Sigma | 16101 | |
| 27B10.4 antibody | Cytoskeleton | APY03-beads | |
| Peptide assay kit | Thermo Scientific | 23275 | Step 7 |
| TopTip | PolyLC Inc | TT200TIO.96 | Steps 5 and 9 |
| SCX columns (PolySULFOETHYL A) | PolyLC Inc | SPESE1203 | |
| 3 mL syringe | BD | 309657 | |
| Trifluoracetic Acid (TFA) | Fisher Scientific | PI-28904 | |
| Acetonitrile (ACN) | Fisher Scientific | A21-1 | |
| Lactic acid | Sigma-Aldrich | 69785-250ML | |
| Ammonium Hydroxide | Fisher Scientific | A669S-500 | |
| Potassium Phosphate Monobasic | Fisher Scientific | BP362-500 | |
| Potassium Chloride | Fisher Scientific | BP366-500 | |
| Calcium Chloride Dihydrate | Fisher Scientific | BP510-500 | |
| Tris Base | Fisher Scientific | BP152-5 | |
| Trypsin, TPCK Treated | Worthington Biochemicals | LS003740 | |
| Lysyl Endopeptidase | Wako Pure Chemical Industries, Ltd. | 125-05061 | |
| MonoTip | PolyLC Inc | TT200TIO.96 | Step 10 |
| ZipTip | MilliporeSigma | ZTC18S096 | Step 6 |
| nanoEase, MZ peptide BEH C18, 130A, 1.7 μm, 75 μm x 20 cm | Waters | 186008794 | Step 11: analytical column |
| Acclaim PepMap 100 C18 LC Columns | ThermoFisher Scientific | 164535 | Step 11: trap column |
| Ultimate 3000 RLSCnano System | Dionex | ULTIM3000RSLCNANO | Step 11 |
| Q Exactive HF | ThermoFisher Scientific | IQLAAEGAAPFALGMBFZ | Step 11 |
| MilliQ water | deionized water used to prepare all solutions and bufferes | ||
| Sonic Dismembrator | Fisher Scientific | FB-120 | sonicator |
| Polytron System PT | Kinematica AG | PT 10-35 GT | homogenizer |
Références
- Siegel, R. L., Miller, K. D., Jemal, A. Cancer statistics, 2018. CA: A Cancer Journal for Clinicians. 68 (1), 7-30 (2018).
- Paller, C. J., Antonarakis, E. S. Management of biochemically recurrent prostate cancer after local therapy: evolving standards of care and new directions. Clinical Advances in Hematology & Oncology. 11 (1), 14-23 (2013).
- Lowrance, W. T., Roth, B. J., Kirkby, E., Murad, M. H., Cookson, M. S. Castration-resistant prostate cancer: AUA guideline amendment 2015. The Journal of Urology. 195 (5), 1444-1452 (2016).
- Domon, B., Aebersold, R. Options and considerations when selecting a quantitative proteomics strategy. Nature Biotechnology. 28 (7), 710-721 (2010).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Bantscheff, M., Schirle, M., Sweetman, G., Rick, J., Kuster, B. Quantitative mass spectrometry in proteomics: a critical review. Analytical and Bioanalytical Chemistry. 389 (4), 1017-1031 (2007).
- Bantscheff, M., Lemeer, S., Savitski, M. M., Kuster, B. Quantitative mass spectrometry in proteomics: critical review update from 2007 to the present. Analytical and Bioanalytical Chemistry. 404 (4), 939-965 (2012).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Rubbi, L., et al. Global phosphoproteomics reveals crosstalk between Bcr-Abl and negative feedback mechanisms controlling Src signaling. Science Signaling. 4 (166), ra18(2011).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045(2018).
- Rush, J., et al. Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nature Biotechnology. 23 (1), 94-101 (2005).
- Fila, J., Honys, D. Enrichment techniques employed in phosphoproteomics. Amino Acids. 43 (3), 1025-1047 (2012).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Drake, J. M., et al. Phosphoproteome integration reveals patient-specific networks in prostate cancer. Cell. 166 (4), 1041-1054 (2016).
- Lue, H. W., et al. Metabolic reprogramming ensures cancer cell survival despite oncogenic signaling blockade. Genes & Development. 31 (20), 2067-2084 (2017).
- Francavilla, C., et al. Phosphoproteomics of primary cells reveals druggable kinase signatures in ovarian cancer. Cell Reports. 18 (13), 3242-3256 (2017).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Skaggs, B. J., et al. Phosphorylation of the ATP-binding loop directs oncogenicity of drug-resistant BCR-ABL mutants. Proceedings of the National Academy of Sciences of the United States of America. 103 (51), 19466-19471 (2006).
- Zimman, A., et al. Activation of aortic endothelial cells by oxidized phospholipids: a phosphoproteomic analysis. Journal of Proteome Research. 9 (6), 2812-2824 (2010).
- Zimman, A., Berliner, J. A., Graeber, T. G. Phosphoproteomic analysis of aortic endothelial cells activated by oxidized phospholipids. Methods in Molecular Biology. , 53-69 (2013).
- Humphrey, S. J., Azimifar, S. B., Mann, M. High-throughput phosphoproteomics reveals in vivo insulin signaling dynamics. Nature Biotechnology. 33 (9), 990-995 (2015).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Meyer, T. S., Lamberts, B. L. Use of coomassie brilliant blue R250 for the electrophoresis of microgram quantities of parotid saliva proteins on acrylamide-gel strips. Biochimica el Biophysica Acta. 107 (1), 144-145 (1965).
- Bergstrom Lind, S., et al. Immunoaffinity enrichments followed by mass spectrometric detection for studying global protein tyrosine phosphorylation. Journal of Proteome Research. 7 (7), 2897-2910 (2008).
- Pinkse, M. W., Uitto, P. M., Hilhorst, M. J., Ooms, B., Heck, A. J. Selective isolation at the femtomole level of phosphopeptides from proteolytic digests using 2D-NanoLC-ESI-MS/MS and titanium oxide precolumns. Analytical Chemistry. 76 (14), 3935-3943 (2004).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Cox, J., et al. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Olsen, J. V., et al. Global, in vivo, and site-specific phosphorylation dynamics in signaling networks. Cell. 127 (3), 635-648 (2006).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. The Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Bielow, C., Mastrobuoni, G., Kempa, S. Proteomics quality control: quality control software for MaxQuant results. Journal of Proteome Research. 15 (3), 777-787 (2016).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Bodenmiller, B., Mueller, L. N., Mueller, M., Domon, B., Aebersold, R. Reproducible isolation of distinct, overlapping segments of the phosphoproteome. Nature Methods. 4 (3), 231-237 (2007).
- Leitner, A., Sturm, M., Lindner, W. Tools for analyzing the phosphoproteome and other phosphorylated biomolecules: a review. Analytica Chimica Acta. 703 (1), 19-30 (2011).
- Yang, D. S., et al. Design and synthesis of an immobilized metal affinity chromatography and metal oxide affinity chromatography hybrid material for improved phosphopeptide enrichment. Journal of Chromatography A. 1505, 56-62 (2017).
- Al Shweiki, M. R., et al. Assessment of label-free quantification in discovery proteomics and impact of technological factors and natural variability of protein abundance. Journal of Proteome Research. 16 (4), 1410-1424 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon