Method Article
Enriquecimento de Fosfopeptida acoplado com espectrometria de massa livre de rótulo quantitativa para investigar o Phosphoproteome no câncer de próstata
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo descreve um procedimento para extrair e enriquecer o phosphopeptides de linhas de células de câncer de próstata ou tecidos para uma análise da phosphoproteome através de massa baseado em espectrometria proteômica.
Resumo
Phosphoproteomics envolve o estudo em larga escala de proteínas fosforiladas. Fosforilação de proteínas é uma etapa crítica em muitas vias de transdução de sinal e é fortemente regulamentada por quinases e fosfatases. Portanto, caracterizar o phosphoproteome pode fornecer insights sobre identificação de novos alvos e biomarcadores para terapia oncológica. Espectrometria de massa fornece uma maneira para globalmente detectar e quantificar milhares de eventos de fosforilação exclusivo. No entanto, phosphopeptides são muito menos abundantes do que não-phosphopeptides, fazendo a análise bioquímica mais desafiador. Para contornar essa limitação, métodos para enriquecer phosphopeptides antes da análise de espectrometria de massa são necessários. Descrevemos um procedimento para extrair e digerir as proteínas do tecido para produzir peptídeos, seguidos por um enriquecimento para phosphotyrosine (pY) e peptídeos de phosphoserine/treonina (pST) usando um anticorpo-baseado e/ou dióxido de titânio (TiO2)-com base método de enriquecimento. Após a preparação da amostra e espectrometria de massa, posteriormente identificar e quantificar phosphopeptides usando líquida cromatografia / espectrometria de massa e software de análise.
Introdução
Um estimado de 165.000 novos casos e cerca de 29.000 mortes ocorrerão em 2018 devido a câncer de próstata, que representa o câncer mais comum e a segunda causa principal de morte por câncer em homens no Estados Unidos1. Estágios iniciais de câncer de próstata são tratáveis com terapia de ressecção ou radiação de doença órgão-confinado, onde a taxa de recorrência de 10 anos está entre 20% e 40% para pacientes submetidos a prostatectomia e entre 30% e 50% para os pacientes que recebem a radiação terapia2. Porque o câncer de próstata depende de andrógeno sinalização para crescimento, terapias de castração cirúrgica e químicos também são empregadas para pacientes de alto risco. No entanto, recidiva ocorre quando o câncer já não responde à terapia de privação do andrógeno, como evidenciado pela recorrência bioquímica, onde o antígeno específico da próstata em soro sobe novamente. Neste momento na progressão, metástases são detectados frequentemente também. Este estágio avançado, chamado de câncer de próstata metastático resistente à castração, representa a forma letal da doença onde o prognóstico é um tempo de sobrevida média de menos de dois anos3. Poucas opções de tratamento estão disponíveis na doença do tarde-estágio, incluindo Antiandrogénios segunda geração como enzalutamide e abiraterone, bem como a quimioterapia baseada em taxano como docetaxel. Apesar de tratamentos disponíveis, a doença progride, muitas vezes. Portanto, a descoberta e o desenvolvimento de modalidades de tratamento romance são necessários para melhorar o cuidado de pacientes com câncer de próstata com doença avançada.
Espectrometria de massa (MS)-baseado em abordagens fornecem uma análise global do proteome através da detecção de centenas a milhares de peptídeo analitos4. Em particular, descoberta proteomics, aquisição de dados dependentes também conhecido como (DDA), pode render a identificação e quantificação de milhares de peptídeos4,5. Proteomics descoberta baseada em MS pode ser ainda mais delineado em proteômica de cima para baixo, onde as proteínas intactas são caracterizadas, e proteomics ascendente (também conhecido como espingarda), onde os peptídeos são analisados para caracterizar proteínas5. Assim, na espingarda proteomics, um passo de proteólise tem lugar na preparação amostra anterior a análise de MS para clivar as proteínas em peptídeos. No final, é feita uma pesquisa de banco de dados para mapear os peptídeos volta para as proteínas para identificação. Etiqueta-livre, bem como vários isótopos-rotulagem [por exemplo, isótopo estável rotulagem por aminoácidos na cultura de pilha (SILAC)] métodos podem ser usados para comparar quantitativamente peptídeos entre amostras6,7. Enquanto técnicas de rotulagem de isótopo são o padrão-ouro, livre de rótulo métodos demonstraram semelhante quantificação exatidões8,9 e tem compensações comparáveis entre sensibilidade e especificidade10. Quantificação da etiqueta livre proporciona maior cobertura e permite comparações entre muitos mais amostras, Considerando que os métodos baseados no rótulo são limitados pelo custo e multiplexação capacidades6,7,8.
Além disso, espingarda MS pode também ser usada para interrogar modificações borne-translational (PTMs) como fosforilação11. Devido à natureza estequiométrica inferior dos phosphopeptides em relação ao totais peptídeos, vários métodos são empregados para enriquecer para phosphopeptides, incluindo os baseados em anticorpo da imunoprecipitação de peptídeos phosphotyrosine (pY), dióxido de titânio (TiO2 ) e imobilizada afinidade metal cromatografia (IMAC)5,12. Porque a fosforilação de proteínas é uma etapa chave em muitos celulares, sinalização de caminhos, espingarda phosphoproteomics permite que os pesquisadores investigar mudanças em diferentes tipos de câncer, incluindo mama13,14de próstata, renal,15, de sinalização celular e ovário,16,17 para entender melhor a biologia do câncer e identificar potenciais novos alvos para a terapia.
Este método de phosphoproteomic livre de rótulo espingarda foi construído e refinado com base em trabalho anterior por Graeber grupo18,19,20. Este protocolo começa por descrever a extração e a digestão de proteínas e as fosfoproteínas do tecido em peptídeos. Em seguida, detalhamos o enriquecimento dos peptides pY usando anticorpos específicos phosphotyrosine e TiO2. Descrevemos também o enriquecimento de peptídeos de phosphoserine/treonina (pST) usando catiónica forte (SCX) seguida pelo TiO2. Este protocolo conclui com a apresentação das amostras a uma instalação de MS e o uso de software de análise de MS para identificar e quantificar phosphopeptides e seus correspondentes fosfoproteínas. A aplicação do presente protocolo pode estender além do câncer de próstata em outros campos fora da Oncologia e cânceres.
Protocolo
Experimentos utilizando tumores do enxerto foram aprovados pelo Comitê de uso e Rutgers University institucional Cuidado Animal como conjunto adiante sob as diretrizes do National Institutes of Health.
1. extração de proteínas
- Prepare o tampão de lise (tabela 1). (O volume depende do número de amostras a serem colhidas). Para amostras de células em vitro , prossiga para a etapa 1.2. Para o tecido do tumor, prossiga para a etapa 1.3.
-
Colheita de células
- Coletar as células em um tubo cónico de 50 mL e girá-los a 700 x g por 5 min a 4 ° C. Desprezar o sobrenadante e manter a pelota no gelo. Repita este passo para todos os pratos coletar as células em uma pelota. (Normalmente, cerca de 5 pratos quase confluentes de 15 cm de células são necessários para 5 mg de proteína, mas isto pode ser dependente da linhagem celular e precisa ser determinado empiricamente por cada investigador.)
- Lave a pelota com 30 mL de refrigerados fosfato salino (PBS) e girar a 700 x g por 5 min a 4 ° C antes de aspirar a PBS. Adicione 1,5 mL de tampão de Lise por 5 mg de proteína usada para o centrifugado. Pipeta e descer algumas vezes. Pule para a etapa 1.4.
-
Colheita de tecidos
- Pesar o tumor e adicionar 2 mL de tampão de Lise gelada para cada 100 mg de tecido em um tubo de ensaio de cultura. (Normalmente, 50 a 150 mg de peso molhado tecido é necessário.)
- Homogeneizar o lisado usando um homogeneizador portátil ou de bancada (pulso 2 x 15 s.) Limpe o homogenizador antes da primeira amostra e entre as amostras, usando água sanitária 10%, 70% de etanol e água desionizada na sucessão.
- Para reduzir e alkylate, aquecer as amostras homogeneizadas a 95 ° C por 5 min. Depois resfriá-los no gelo por 15 min. No gelo, proceda à sonicação o lisado x 3 (ou seja, pulso por 30 s 60 s pausas entre pulsos). A amostra não deve ser viscoso ou agrupadas neste ponto. Aqueça o lisado a 95 ° C por 5 min21.
- Centrifugar o lisado na mesma sonication tubo usando um balde de balanço do rotor a 3.500 x g a 15 ° C durante 15 min. recolher o sobrenadante e descartar a pelota.
- Determine a concentração de proteína através da realização de um ensaio de Bradford22. Se necessário, dilua o lisado a 5 mg/mL com um tampão de Lise. Armazená-lo a-20 ° C.
Nota: O experimento pode ser pausado aqui. Congelar as amostras a-80 ° C e continuar em uma data posterior.
2. lisado digestão
- Diluir a amostra 12-fold usando 100 mM Tris (pH = 8,5) para reduzir a quantidade de guanidínio. Dilua todas as amostras para o mesmo volume para minimizar os efeitos da digestão desigual. Salve a 12,5 µ g do não digerida lisado para confirmá-la em um gel de Coomassie manchada23.
- 5 mg de proteína, adicionar 10 µ g de lisil Endopeptidase (Lys-C) e incube-lo à temperatura de pH de 5-6 h. ajustar a 8.0 por adição de 1m untitrated Tris (pH ~ 11).
- Prepare-se 1 mg/mL de L-1-tosylamido-2-feniletílico clorometil cetona (TPCK)-Tratado de tripsina em 1mm HCl (com 20 mM CaCl2). Adicionar a tripsina na proporção de 1: 100 de tripsina: proteína e incubar a 37 ° C por 3 h.
- Adicione a mesma quantidade de tripsina fresca como na etapa 2.3. Incube-a 37 ° C durante a noite.
- Salve a 12,5 µ g do digerido lisado para confirmar a digestão completa em um gel de Coomassie manchada23.
3. inverter a fase de extração
- O volume de lisado de registro. Filtre a amostra usando um filtro de corte 15 mL 10 kDa. Centrifugar a amostra em 3.500 x g usando o balde de balanço do rotor (ou 3.500 x g em um rotor de ângulo fixo) a 15 ° C até que o volume de retentate é inferior a 250 µ l (isto leva aproximadamente 45-60 min). Coletar o escoamento e descartar o retentate.
Nota: O experimento pode ser pausado aqui. Congelar as amostras a-80 ° C e continuar em uma data posterior. - Para acidificar a amostra, adicione cerca de 20 µ l de ácido trifluoroacético (TFA) de 5% de lisado por mL. Misture-os bem e medir o pH da amostra usando tiras de pH. Ajustar o pH a 2.5 usando 5% TFA.
- Ligue a extremidade mais curta de uma coluna de C-18 para um distribuidor de vácuo. Defina o vácuo entre 17 e 34 kPa (ou de acordo com as instruções do fabricante). Usar pipetas de vidro , molhe a coluna com 3 mL de 100% acetonitrila (ACN). Não deixe a coluna seca.
- Usar pipetas de vidro , equilibrar a coluna com 6 mL de 0.1% TFA aplicado como 2 x 3 mL. Carrega a amostra acidificada na coluna. Não adicione mais de 3 mL de cada vez. Ajuste o vácuo para atingir cerca de 1 a 2 gotas por segundo.
- Usar pipetas de vidro , lave a coluna com 9 mL de 0.1% TFA aplicado como 3 x 3 mL. Eluir a coluna com 2 mL de 40% ACN, 0,1% TFA. Colete duas frações 2 mL em tubos de cultura do vidro . Descarte a coluna.
- Cobrir os tubos eluído com parafilm e 3-5 buracos na capa usando uma agulha 20g. Congele o eluído em gelo seco durante pelo menos 30 minutos até que esteja completamente sólida.
- As frações lyophilize durante a noite. No dia seguinte, certifique-se que as amostras estão completamente secas antes de interromper o liofilizador. Armazenar os tubos em um tubo cônico de 50 mL com toalhitas delicadas a-80 ° C.
Nota: O experimento pode ser pausado aqui.
4. imunoprecipitação e enriquecimento de pY peptídeos24
- Resuspenda o pó liofilizado com 0,5 mL de tampão de ligação de imunoprecipitação gelada (IP) em cada fração. As frações do pool transferindo o volume de ressuspensão de 0,5 mL de fração de segundo para a primeira fração e salvar a ponta da pipeta. Vórtice vigorosamente (em vez de pipetagem para cima e para baixo) para certificar-se que a amostra é completamente dissolvida antes de transferi-lo para um 3,6 mL tampa de rosca criotubo.
- Como na etapa 4.1., lave os tubos de liofilização com outro 0,5 mL de tampão de ligação de IP (tabela 1) em cada tubo. Transferi a solução para o tubo de tampa de rosca de 3,6 mL usando a mesma ponta da pipeta para minimizar qualquer perda de amostra. Repita o enxágue 1 x mais, fazendo o volume final ressuspensão 2 mL (para 5 mg de proteína). Medir o pH da amostra para certificar-se de que é aproximadamente 7,4. Se for muito ácida, iterativamente, adicione 10 µ l de 1 M Tris (untitrated, pH ~ 11). Se é muito básico, iterativamente, adicione 10 µ l de HCl diluído (01:25 ou 1: 100).
-
Pre-lave os grânulos de pY (para 5 mg de lisado a começar)
- são necessários 25 µ g de anticorpo 4 10 e 12,5 µ g de anticorpos 27B10.4 por amostra. Depois de usar um p200 pipete com uma dica de corte para transferir os anticorpos para tubos de microcentrifuga separado, lavar os anticorpos com 450 µ l de tampão de ligação gelada IP 2x. Centrifugá-los a 100 x g por 1 min a 4 ° C e aspirado para fora o sobrenadante.
- Resuspenda as contas de estoque concentração de 0,5 mg/mL com tampão de ligação IP. (Do vórtice não os grânulos). Depois de alíquotas o chorume necessária (50 µ l de chorume de anticorpo 4 10 e 25 µ l 27B10.4 anticorpo da pasta por exemplo) em um único tubo, spin para baixo os tubos de centrífuga de estoque a 200 x g por 1 min a 4 ° C. Lave as paredes laterais com sobrenadante antes de retornar os grânulos de armazenamento na geladeira.
- Adicione pre-lavado pY grânulos para a solução de amostra ressuspensão em cryotubes a tampa de rosca. Incube-os a 4 ° C em uma extremidade-sobre-extremidade rotator durante a noite.
- Coloque a tampa de rosca cryotubes em um tubo de centrífuga de 50 mL forrado com uma limpeza delicada. Spin para baixo os grânulos a 100 x g por 1 min. salvar o sobrenadante, que será usado para enriquecer por peptídeos de pST. (O enriquecimento para pST começa no passo 7 e pode ser executado em paralelo ao processamento de peptídeo de pY).
- Resuspenda os grânulos com 300 µ l de tampão de ligação IP. Transfira para um tubo de microcentrifugadora de 2 mL e girá-los para baixo a 100 x g por 1 min a 4 ° C.
- Enxágue a incubação do tubo 3 x com 200 µ l de tampão de ligação IP. Transferi o conteúdo para o mesmo tubo microcentrifuga cada vez. Em seguida, girá-los para baixo.
- Lave os grânulos no microcentrifuga tubo 3 x com 500 µ l de tampão de ligação IP e girá-los para baixo a 100 x g por 1 min. Em seguida, lave os grânulos 4 x com 450 µ l de 25mm NH4HCO3, pH 7,5 e spin-los a 100 x g por 1 min. Use um fresco 25mm NH4HCO3 solução de pó de cada vez.
- Centrifugue os grânulos a 1.500 x g por 1 min. Use uma ponta de gel de carregamento para remover o sobrenadante completamente mergulhando a ponta da ponta de gel de carregamento ligeiramente abaixo de superfície dos grânulos.
- Adicionar 4 vezes o volume do grânulo de 0,1% TFA aos talões (i.e., adicione 300 µ l de 0.1% TFA para 75 µ g de chorume de grânulo de pY). Misture-os bem e incubar a mistura em uma thermomixer a 1.000 rpm por 15 min a 37 ° C.
- Transferi a ressuspensão de um filtro de rotação de 0,2 µm. Rapidamente girar para baixo o tubo de eluição e transferir o volume residual para o mesmo filtro de rotação usando uma pipeta de P10. Spin-down o filtro volta a 850 x g por 1 min. Transfira a eluição para um tubo de microcentrifugadora de baixa proteína ligadora . Vácuo concentrar-se o eluato de secura durante a noite a 40 ° C e com um tempo de calor de 300 min.
Nota: O experimento pode ser pausado aqui. Congelar as amostras a-80 ° C e continuar em uma data posterior.
5. o dióxido de titânio enriquecimento25 de pY peptídeos
- Resuspenda as secas em baixo phosphopeptides em 200 µ l de 50% ACN, 0,1% TFA. Vórtice e centrifugar--los a 10.000 x g durante 30 s. repetir este 1x para ressuspendê-los bem.
- Preparando os TiO2 grânulos contidos em dicas que têm uma capacidade de 200 amostras µ l.
- Bata levemente no lado pequeno ponta da ponta para mover o material para esse fim. Enxágue a ponta adicionando-se 200 µ l de 100% ACN, seguido por invertendo a ponta e passando rapidamente a extremidade pequena para mover o líquido para o cap.
- Usando uma lâmina de barbear, cortar a ponta pequena da ponta e coloque sobre um tubo de proteína ligadora de baixo. (Evitar o uso de tubos de poliestireno como o TiO2 furará para os lados do tubo). Retire a tampa e insira uma micropipeta para mergulhar para fora o restante do Grupo ACN. Repita a lavagem com 200 µ l de 100% ACN. Agora, os grânulos de TiO2 localizam-se no tubo de baixa proteína ligadora para as etapas seguintes.
- Condição de TiO2 com 500 µ l de 100% do Grupo ACN 2 x. Pipetar a mistura de grânulos com o solvente. Centrifugá-los a 100 x g por 1 min.
- Condição de TiO2 com 500 µ l de tampão de fosfato de sódio 0,2 M (pH ~ 7) x 2. Lave os grânulos com 300 µ l de equilíbrio do buffer 3x. Porque TiO2 é muito densa, os grânulos vão resolver rapidamente.
- Adicione 400 µ l de 50% ACN, 0,1% TFA no tubo de baixa proteína ligadora, seguido por adição µ l 84 de ácido láctico. Transferir a ressuspensão phosphopeptides no tubo de baixa proteína ligadora e incube-os por 1h à temperatura ambiente usando um rotador extremidade-sobre-extremidade.
- Centrifugue os grânulos a 100 x g por 1 min a pelota-los. Lave-os com 300 µ l de tampão de equilíbrio (tabela 1), 2 x e girá-los para baixo a 100 x g por 1 min.
- Lave os grânulos com 300 µ l de lavagem buffer 2x. Transferi-los para um filtro de rotação de 0,2 µm. Girá-los a 1.500 x g por 1 min.
- Transferi a unidade de filtro para um tubo de proteína ligadora de baixo limpa 1,5 mL. Eluir o conteúdo 2 x com 200 µ l de 0,9% de NH3 em H2O. medida do pH com tiras de pH, que deve ser entre 10 e 11. Vácuo concentrar o eluato de secura durante a noite para evaporar o amoníaco.
6. dessalinização pY peptídeos para MS Analyses
- Reconstituir o phosphopeptides com 15 µ l de 0,1% TFA pela utilização do Vortex e eles centrifugação a 10.000 x g, durante 30 s para ressuspendê-los. Repita este 1x para ressuspendê-los bem. Não pipete acima e para baixo.
- Limpar a amostra usando uma ponta de C-18 com uma capacidade de ligação de 5 µ g e seguir o protocolo do fabricante.
- Seque completamente o volume de eluição por concentração de vácuo. Isto leva 1-2 h. Ressuspender phosphopeptides secas em 12,5 µ l de solução de espectrometria de massa (ver tabela 1) (ou como recomendado pelo MS do pesquisador proteomics core facilidade). Vórtice e spin brevemente a solução para baixo a 10.000 x g, durante 30 s. repetir este 2x ressuspendê-los bem. As amostras estão prontas para a apresentação de uma instalação de espectrometria de massa (passo 11).
Nota: Os seguintes passos abaixo estão relacionados ao enriquecimento de peptídeo pST somente.
7. inverter a fase de extração de peptídeos de pST
- Medir a concentração de peptídeo de sobrenadante adquirida da etapa 4.6 através da realização de um ensaio de peptídeo. Uma quantidade suficiente para espectrometria de massa de pST é de 2,5 mg.
- Ajustar o pH de 3,5 com 5% TFA.
- Ligue a extremidade mais curta de uma coluna de C-18 para um distribuidor de vácuo. Defina o vácuo entre 17 e 34 kPa (ou de acordo com as instruções do fabricante). Molhar a coluna com 3 mL de 100% ACN. Não deixe a coluna seca.
- Equilibrar a coluna com 6 mL de 0.1% TFA aplicado como 2 x 3 mL. Carrega a amostra acidificada na coluna. Não adicione mais de 3 mL de cada vez. Ajuste o vácuo para atingir cerca de 1 a 2 gotas por segundo.
- Lave a coluna com 9 mL de 0.1% TFA aplicado como 3 x 3 mL. Eluir a coluna com 2 mL de 40% ACN, 0,1% TFA. Colete duas frações 2 mL em tubos de cultura do vidro. Descarte a coluna.
- Cobrir os tubos eluído com parafilm e 3-5 buracos na capa usando uma agulha 20g. Congele o eluído em gelo seco durante pelo menos 30 minutos até que esteja completamente sólida.
- Lyophilize as frações selecionadas durante a noite. No dia seguinte, certifique-se que as amostras estão completamente secas antes de interromper o liofilizador. Armazenar os tubos em um tubo cônico de 50 mL com toalhitas delicadas a-80 ° C.
Nota: O experimento pode ser pausado aqui.
8. forte catiónica (SCX) de peptídeos de pST
- Resuspenda os peptídeos liofilizados em 2 mL de tampão A (tabela 1). Piscina as frações para cada amostra. (A solução será nublada).
- Prepare o distribuidor de vácuo. Conecte uma coluna SCX uma seringa de 3 mL com o êmbolo removido. Defina o vácuo entre 17 e 34 kPa (ou de acordo com as instruções do fabricante).
- Condição a coluna SCX com 4 mL de ACN, seguido de 4 mL de tampão A.
- Carregar a 2 mL da amostra de passo 8.1 e recolher o eluato imediatamente. Carregar 3 mL de tampão de A:B (80.9:19.1) e recolher o eluato. Piscina os eluídos de cada amostra e alíquota-los em tubos de ligação às proteínas baixa 2 mL.
- Vácuo concentrar todas as amostras até cerca de 30% do volume permanece. (Esta etapa leva cerca de 2-4 h.) As alíquotas do pool 1 baixa proteína ligadora de tubo para cada amostra.
- Ligue a extremidade mais curta de uma coluna de C-18 para um distribuidor de vácuo. Defina o vácuo entre 17 e 34 kPa (ou de acordo com as instruções do fabricante). Molhar a coluna com 3 mL de 100% ACN 2 x. Fazer não deixe a coluna seca.
- Equilibrar a coluna com 3 mL de 0.1% TFA 2 x. Carrega a amostra para a coluna. Não adicione mais de 3 mL de cada vez. Ajuste o vácuo para atingir cerca de 1 a 2 gotas por segundo.
- Lavar a coluna com 3 mL de 0.1% TFA 2 x. Eluir a coluna com 4 mL de 50% ACN, 0,1% TFA.
9. dióxido de titânio enriquecimento de peptídeos de pST
-
Preparando os TiO2 grânulos contidos em dicas que têm uma capacidade de 200 amostras µ l
- Bata levemente no lado pequeno ponta da ponta para mover as contas para esse fim. Retire a tampa e despeje os grânulos em um tubo cônico de polipropileno de 15 mL.
- Enxágue a ponta adicionando-se 200 µ l de 100% ACN, invertendo a ponta de um par de vezes e passando rapidamente a extremidade pequena para mover o líquido para o cap. Usando uma lâmina de barbear, cortar a ponta pequena da ponta e coloque-o sobre o tubo cónico de polipropileno de 15 mL. Retire a tampa e insira uma micropipeta para mergulhar para fora o restante do Grupo ACN. Repita a lavagem com 200 µ l de 100% ACN. Os grânulos de2 TiO agora estão localizados no tubo cônico de 15 mL para as etapas seguintes.
- Condição de TiO2 com 500 µ l de 100% do Grupo ACN 2 x. Pipetar a mistura de grânulos com o solvente. Centrifugá-los a 100 x g por 1 min.
- Condição de TiO2 com 500 µ l de tampão de fosfato de sódio 0,2 M (pH ~ 7) duas vezes. Lave os grânulos com 300 µ l de equilíbrio do buffer 3x.
- Transferi o phosphopeptides eluted no tubo cónico de polipropileno de 15 mL. Adicionar 560 µ l de ácido láctico e incube-lo por 1h à temperatura ambiente usando um rotador extremidade-sobre-extremidade.
- Centrifugue a mistura a 100 x g por 1 min granular os grânulos. Lave-os com 300 µ l de tampão de equilíbrio (tabela 1), 3 x. Girá-los para baixo a 100 x g por 1 min.
- Lave os grânulos com 300 µ l de lavagem buffer 2x. Transferi-los para um filtro de rotação de 0,2 µm. Girá-los para baixo a 1.500 x g por 1 min.
- Transferi a unidade de filtro para um tubo de proteína ligadora de baixo limpa 1,5 mL. Eluir o conteúdo 2 x com 200 µ l de 0,9% de NH3 em H2O. Deixe a solução em phosphopeptides por 2 min antes de eluição-los. Medir o pH, que deve ser entre 10 e 11.
- Vácuo concentrar o eluato de secura durante a noite para evaporar o amoníaco.
10. dessalinização pST peptídeos para análises de MS
- Bata levemente no lado pequeno ponta da ponta para mover o material para esse fim. Enxágue a ponta adicionando-se 200 µ l de 100% ACN, seguido por invertendo a ponta e passando rapidamente a extremidade pequena para mover o líquido para o cap.
- Usando uma lâmina de barbear, cortar a ponta pequena da ponta e coloque sobre um tubo cónico de polipropileno 15 mL. Retire a tampa e insira uma micropipeta para mergulhar para fora o restante do Grupo ACN. Repita a lavagem com 200 µ l de 100% ACN. Agora, os grânulos de TiO2 localizam-se no tubo cónico de polipropileno 15 mL para as etapas seguintes.
- Limpar a amostra usando uma ponta de C-18 com uma capacidade de ligação de 100 µ g. (siga as instruções do fabricante.)
- Seque completamente o volume de eluição por concentração de vácuo. Isto leva 1-2 h.
- Resuspenda phosphopeptides secas em 12,5 µ l da solução de espectrometria de massa (ou como recomendado por instalação de núcleo do pesquisador MS proteômica). Vórtice e centrifugá-los a 10.000 x g, durante 30 s. repetir 2x para ressuspendê-los bem. (Não pipete acima e para baixo.)
11. espectrometria análise
- Submeta as amostras para a instalação de núcleo de proteomics MS para executar MS de tandem-cromatografia líquida (LC-MS/MS) usando as configurações recomendadas. Exemplo de configurações é as seguintes (ver tabela 2 para obter o resumo):
- Carregar 5 µ l das amostras em uma coluna de armadilha (2 cm de comprimento x 75 µm de diâmetro) e lave-os com 0,1% TFA por 5 min com uma taxa de fluxo de 5 µ l/min.
- Trazer a armadilha em linha com uma coluna analítica nano (20 cm x 75 µm) com uma vazão de 300 nL/min.
- Os gradientes lineares segmentados (percentagem de ácido fórmico de 0,16%, 80% ACN em 0,2% de ácido fórmico) são diferentes entre as amostras de pY e pST:
- Para as amostras de pY, elui-los usando um gradiente de 4-15% em 5 min, 15-50% em 40 min e 50-90% em 5 min.
- Para amostras de pST, elui-los usando um gradiente de 4-15% em 30 min, 15-25% em 40 min, 25-50% em 44 min e 50-90% em 11 min.
- Aquisição de dados de MS no modo de aquisição de dados dependentes uma série cíclica de uma varredura completa com uma resolução de 120.000 seguido por MS/MS (HCD, energia de colisão relativo de 27%) dos 20 íons mais intensos e uma duração de exclusão dinâmica de 20 s.
- Após a MS execute conclusão, importe os arquivos raw de MS para um programa de software de análise de MS para identificar e quantificar phosphopeptides. (Software de MaxQuant8,26,27 foi utilizado neste experimento. A menos que especificado na tabela 3, as configurações padrão foram usadas.)
Resultados
Este protocolo descreve detalhadamente um método para a extração de proteínas e digestão seguido de enriquecimento Fosfopeptida e posterior análise de MS (Figura 1). As composições de todos os buffers e soluções que são utilizados neste protocolo são listadas na tabela 1. O uso sequencial de Lys-C e tripsina fornece uma digestão eficiente. Um manchado de Coomassie gel de pré-digerida lisado confirma a presença de proteínas, enquanto a coloração de pós digerido lisado confirma a digestão completa (Figura 2A). Para uma digestão completa, sem bandas devem aparecer acima 15 kDa, exceto a 30 kDa e 23,3 kDa bandas por Lys-C e tripsina, respectivamente. A adição de Lys-C também reduz o número de clivagens perdidas (Figura 2B). Porque pY peptídeos representam apenas 2% do phosphoproteome28, imunoprecipitação dos peptides usando um anticorpo específico de pY pY é o primeiro passo de enriquecimento de peptídeo de pY. O sobrenadante resultante torna-se a entrada para o enriquecimento de peptídeo de pST. A imunoprecipitação pY efetivamente separa pY peptídeos peptídeos pST onde em média 85% das phosphopeptides identificadas a partir da preparação de pY pY (Figura 3A) e mais de 99% dos phosphopeptides identificados a partir da preparação de pST são pST (Figura 3B). Dióxido de titânio é usado para enriquecer para phosphopeptides em ambas as preparações. O percentual esperado de peptídeos na preparação MS-pronto que são fosforiladas é entre 30 a 50% (Figura 4A). A variabilidade da percentagem de enriquecimento Fosfopeptida pode ser maior na preparação pY como resultado de haver muitos menos pY peptídeos que peptídeos de pST. Em termos de espécies de Fosfopeptida, a maioria dos phosphopeptides detectado tem um grupo de fosforilo simples ou dupla (Figura 4B).
Depois de realizar a espectrometria de massa, os arquivos raw de MS são carregados em um software de análise de MS. As configurações de parâmetro usadas no experimento estão listadas na tabela 3 , mas irão variar de um software para o software e podem variar de versão para versão. Os parâmetros que não estão listados foram deixados como padrão, incluindo um FDR corte de 1% para peptídeo-espectro correspondente (PSM) com uma pontuação mínima de Andrômeda de 40 para a identificação de peptídeos modificado27. Definindo um limite de probabilidade de localização de superior a 0,75 filtra aproximadamente 5% dos peptides pY e 15% e 34% do pS e pT peptídeos, respectivamente (Figura 5A). Depois de aplicar estes filtros, o número esperado de identificações de Fosfopeptida no final da análise de MS é aproximadamente 300 pY peptídeos (para 5 mg de proteína a partida) e cerca de 7.500 peptídeos de pS e pT 640 peptídeos (para 2,5 mg do peptide partido o montante) de preparações o respectivo enriquecimento (Figura 5B). O número de repetições e a variabilidade da intensidade de sinal Fosfopeptida determinam a alimentação adequada para comparações estatísticas. Em quatro experimentos separados com grupos contendo duplicatas biológicas ou triplica, foram calculados os porcentagem coeficientes de variação (% CV) para phosphopeptides detectado. Distribuições de menor variabilidade (por exemplo, grupos de pST 1-5 na Figura 5) indicam que a coleta de amostra, preparação e execuções de espectrometria de massa foram consistentes. Por outro lado, as distribuições de maior variabilidade (por exemplo, pST grupo 6 na Figura 5) indica dados ruidosos que exigiriam maiores alterações-dobra para detectar diferenças significativas nas análises diferenciais a jusante.
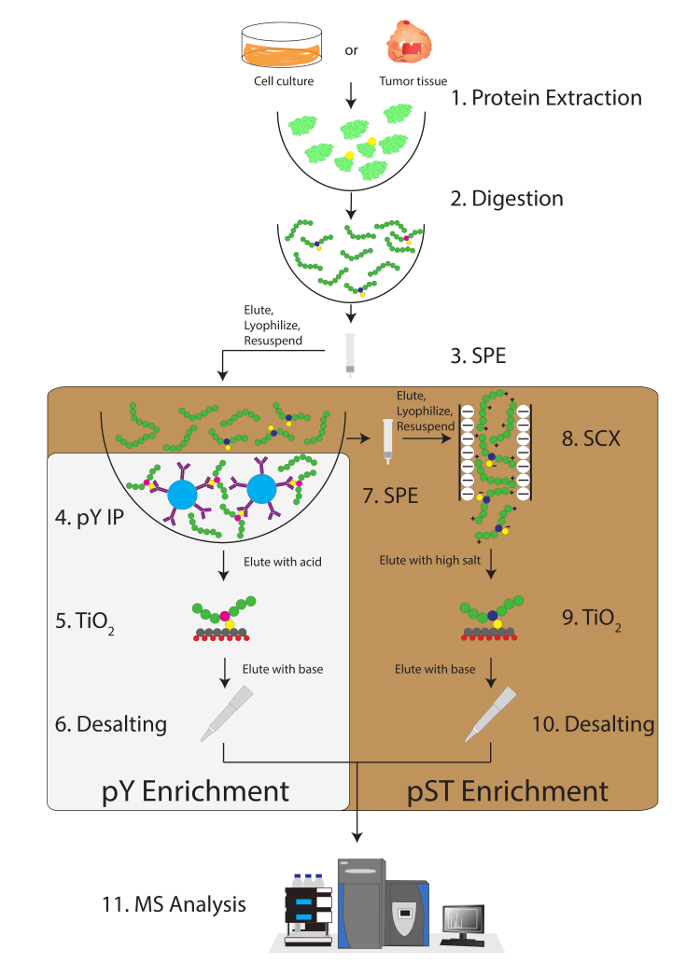
Figura 1: diagrama de fluxo de trabalho. Proteínas de amostras são extraídas e digeridas. Peptídeos são extraídos por extração de fase sólida (SPE) e peptídeos phosphotyrosine (pY) são immunoprecipitated. Em paralelo, os peptídeos de phosphoserine/treonina (pST) são enriquecidos de sobrenadante na etapa de imunoprecipitação pY. Troca de cátion forte (SCX) é executada no sobrenadante para remover peptídeos altamente carregados para reduzir o íon supressão12. Ambas as preparações passam por Fosfopeptida enriquecimento através de dióxido de titânio (TiO2). Após a limpeza da amostra, líquida espectrometria de massa em tandem-cromatografia (LC-MS/MS) é realizada para medir a abundância de Fosfopeptida. Os dados brutos é então carregados em um software de análise de MS para identificar phosphopeptides. Clique aqui para ver uma versão maior desta figura.
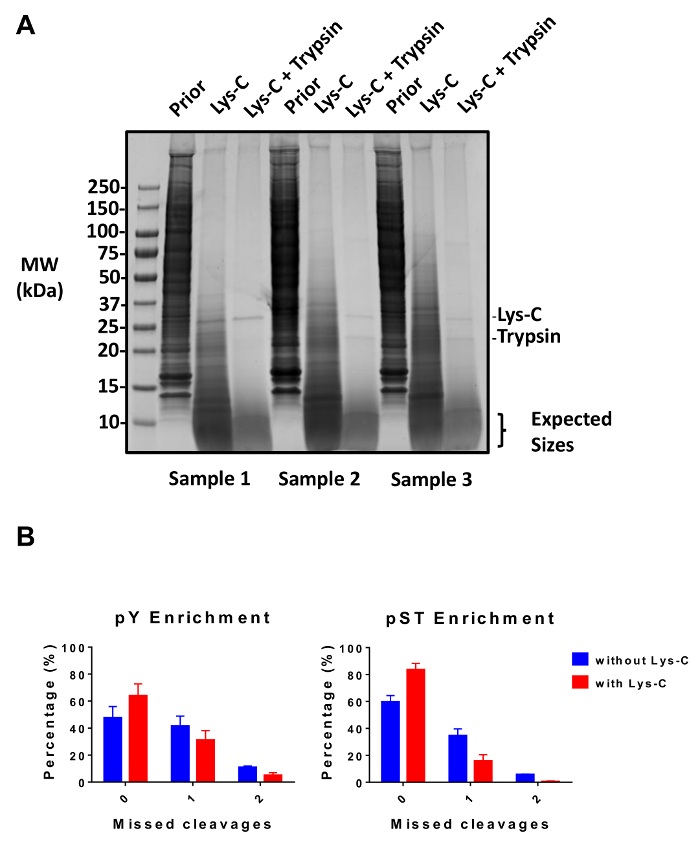
Figura 2: avaliação da digestão. (A) três amostras com 12,5 µ g de lisado digestão pre- digestão, post-Lys-C, e pós-tripsina digestão são mostrados. Um teste de gel-mancha de Coomassie mostra uma digestão limpa após o uso sequencial de Lys-C e tripsina. Os marcadores de tamanho molecular peso (MW) estão em kilodaltons (kDa). (B) A redução em decotes perdidas é observada após Lys-C foi adicionada ao protocolo. A porcentagem dos phosphopeptides sem decotes não atendidas aumentou de 48% a 64% e de 60% para 84% em média para preparações de enriquecimento pY e pST, respectivamente. Os gráficos resumem os dados obtidos de dois experimentos realizados sem Lys-C e cinco experimentos realizados com Lys-C. As barras de erro são desvios-padrão que representa pY 38 38 amostras de pST de 2 experimentos separados (sem Lys-C) e 62 pY e 60 amostras de pST de 5 experimentos separados (com Lys-C). Clique aqui para ver uma versão maior desta figura.
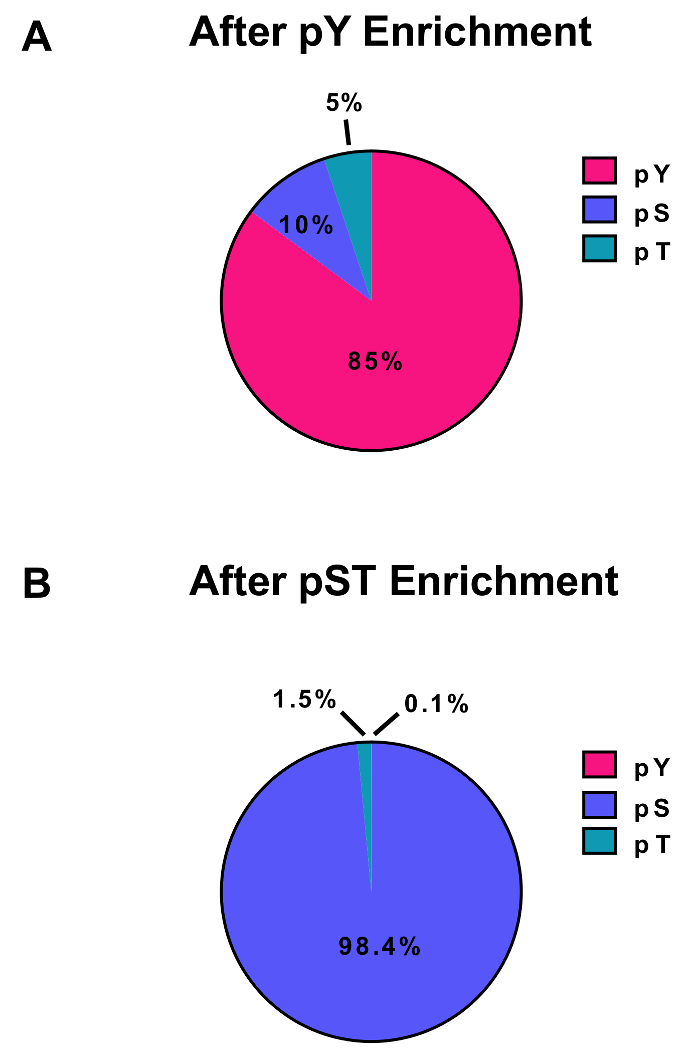
Figura 3: enriquecimento dos phosphopeptides pY e pST. Estes painéis mostram as porcentagens de pSTY phosphopeptides de (A) o pY ou (B) os preparativos de enriquecimento do pST. O enriquecimento de pY pY imunoprecipitação e dióxido de titânio resultou em 85% phosphopeptides sendo para pY peptídeos, enquanto apenas 0,1% dos phosphopeptides no enriquecimento de pST são pY. Esses valores foram retirados de examinar o Phospho (arquivo STY)Sites.txt experiência representativa após filtragem de contaminantes, sequências reversos e phosphopeptides com probabilidades de localização inferior a 0,75. Clique aqui para ver uma versão maior desta figura.
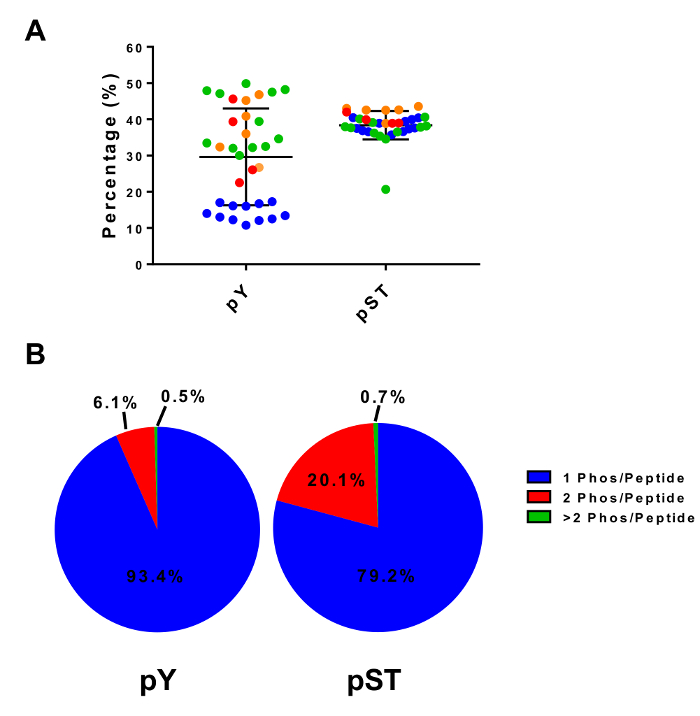
Figura 4: Fosfopeptida enriquecimento com dióxido de titânio. (A) a porcentagem de phosphopeptides detectados (em relação ao totais peptídeos) de amostras em quatro experimentos separados é mostrada. (B), este painel mostra a composição média de mono - double- e peptídeos multi-fosforilada em quatro experimentos separados. As barras de erro no painel A são desvios-padrão. Clique aqui para ver uma versão maior desta figura.
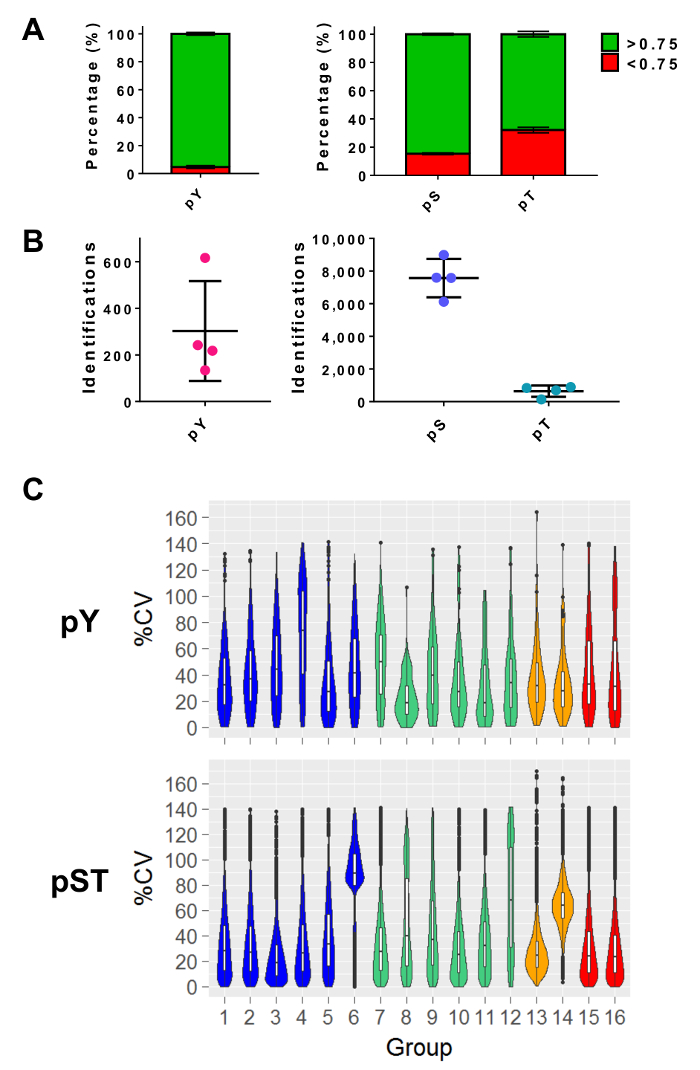
Figura 5: esperado identificações phosphoresidue. (A), este painel mostra as probabilidades de localização de fosforilação de IDs de enriquecimento pY (à esquerda) e enriquecimento de pST (à direita). A percentagem média de IDs que atendem o limite de probabilidade > 0,75 é 93%, 75% e 52% para pY, pS e pT, respectivamente. (B) a média número de identificações com um > 0,75 probabilidade de localização é 300 para pY, 7.500 para pS e 640 em pT. (C), este painel mostra parcelas do violino do percentual do coeficiente de variação (% CV) dos phosphopeptides. Só foi realizada uma avaliação de % CV se um valor de intensidade de sinal foi detectado em cada replicar biológica ou grupo de três vias. Dados foram retirados de quatro experimentos separados. As barras de erro em painéis A e B são desvios-padrão de 34 pY e 34 amostras de pST de 4 experimentos separados. Clique aqui para ver uma versão maior desta figura.
| Buffer de | Volume de | Composição | |
| Tampão de lise de cloreto de guanidínio 6m | 50 mL | Cloreto de guanidínio 6m, 100 mM tris pH 8,5, fosfina tris (2-sesquióxido) de 10 mM cloroacetamida 40mm, 2mm ortovanadato de sódio, pirofosfato de sódio 2,5 mM, β-glicerofosfato de 1 mM, 500 mg n-octil-glicosídeo, ultra pura água ao volume | |
| Pirofosfato de sódio de 100 mM | 50 mL | 2,23 g sódio pirofosfato decahidrato, água ultra-pura para volume | |
| Β-glicerofosfato de 1m | 50 mL | g 15,31 β-glicerofosfato, água ultra-pura para volume | |
| 5% de ácido trifluoroacético | 20 mL | Adicionar 1 mL de ácido trifluoroacético de 100% em 19 mL água ultra pura | |
| 0,1 o ácido trifluoroacético % | 250 mL | Adicionar 5 mL 5% de ácido trifluoroacético para 245 mL de água ultra pura | |
| tampão de eluição pY | 250 mL | 0,1% ácido trifluoroacético 40% acetonitrilo, água ultra-pura para volume | |
| tampão de eluição de pST | 250 mL | 0,1% ácido trifluoroacético 50% acetonitrilo, água ultra-pura para volume | |
| Tampão de ligação IP | 200 mL | pH de tris 50mm 7.4, 50 mM de cloreto de sódio, água ultra-pura para volume | |
| bicarbonato de amónio de 25 mM, pH 7,5 | 10 mL | Dissolver 19,7 mg em 10 mL de água ultra-pura estéril, pH de 7.5 com 1 N de ácido clorídrico (~ 10-15 µ l/10 ml de solução), fazer fresco | |
| 1M tampão fosfato, pH 7 | 1.000 mL | hidrogenofosfato de sódio de 1 M 423 mL, hidrogenofosfato de sódio de 1 M mL 577 | |
| Buffer de equilibração | 14 mL | 6,3 mL acetonitrila, 280 µ l trifluoroacético 5% ácido lácticas µ l de 1740, 5,68 mL de água ultra pura | |
| Tampão de lavagem | 20 mL | 9 mL acetonitrila, o ácido trifluoroacético 5% µ l 400, 10,6 mL de água ultra pura | |
| Solução de espectrometria de massa | 10 mL | 500 µ l acetonitrilo, 200 µ l 5% trifluoroacético ácido, 9,3 mL de água ultra pura | |
| Tampão A | 250 mL | Fosfato monopotássico de 5 mM (pH 2,65), 30% acetonitrila, 5 mM de cloreto de potássio, água ultrapura para volume | |
| Tampão B | 250 mL | Fosfato monopotássico de 5 mM (pH 2,65), 30% acetonitrila, 350 mM de cloreto de potássio, água ultra-pura para volume | |
| 0,9% de hidróxido de amônio | 10 mL | 300 μL 29.42% de hidróxido de amónio, 9,7 mL de água ultra pura | |
Tabela 1: Buffers e soluções. Esta tabela mostra as composições dos amortecedores e soluções utilizados neste protocolo.
| Configurações de LC-MS/MS | ||
| Parâmetro | pY configuração | pST configuração |
| Amostra (µ l) de carregamento | 5 | |
| Taxa de fluxo de carga (µ l/min) | 5 | |
| Taxa de fluxo de gradiente (nL/min) | 300 | |
| Gradiente linear (percentagem de ácido fórmico 0,16%, 80% ACN em 0,2% de ácido fórmico) | 4 - 15% por 5 min | 4 - 15% por 30 min |
| 15 - 50% para 40 min | 15 - 25% para 40 min | |
| 50 - 90% por 5 min | 25 - 50% para 44 min | |
| 50 - 90% para 11 min | ||
| Resolução de digitalização completa | 120.000 | |
| Número de íons mais intensas selecionado | 20 | |
| Energia de colisão relativa (%) (HCD) | 27 | |
| Exclusão dinâmica (s) | 20 | |
Tabela 2: configurações de LC-MS. Este é um exemplo de configurações de LC-MS em um experimento de phosphoproteomic típica de espingarda. As amostras foram carregadas em uma coluna de armadilha. A armadilha foi trazida em linha com uma coluna analítica. Essas configurações foram otimizadas para usar o sistema de LC-MS, listado na tabela de materiais e reagentes. Essas configurações precisa ser ajustada para outros sistemas de LC-MS.
| Configurações de parâmetro de MaxQuant | ||
| Configuração | Ação | |
| Parâmetros específicos do grupo | ||
| Tipo | Tipo | Selecione padrão |
| Multiplicidade | Definido como 1 | |
| Modo de digestão | Enzima | Selecione a tripsina/P |
| Max. clivagens perdidas | Conjunto de 2 | |
| Modificações | Modificações de variáveis | Adicionar Phospho (STY) |
| Quantificação de etiqueta-livre | Quantificação de etiqueta-livre | Selecione LFQ |
| Contagem de rácio de min. LFQ | Definido como 1 | |
| LFQ rápido | Verifique | |
| Diversos | Re-quantificar | Verifique |
| Parâmetros globais | ||
| Sequências de | Arquivos FASTA | Fasta Selecione arquivo baixado UniProt |
| Modificações fixas | Adicionar Carbamidomethyl (C) | |
| Identificação de adv. | Transferências entre as execuções | Verifique |
| Janela de tempo de jogo | Situado a 5 min | |
| Janela de tempo de alinhamento | Definido como 20 min | |
| Combinar recursos não identificados | Verifique | |
| Quantificação da proteína | Contagem de rácio de min. | Definido como 1 |
| Locais de pasta | Modificar em conformidade | |
Quadro 3: definições de software de análise de MS. Em MaxQuant, os parâmetros específicos do grupo e globais nesta tabela foram selecionados ou ajustados. Todos os outros parâmetros permaneceram no padrão. Estes experimentos foram realizados utilizando a versão 1.5.3.30. Os parâmetros podem variar de uma versão para e de um software para o software.
Discussão
Antes de utilizar este protocolo para enriquecer para phosphopeptides, uma consideração cuidadosa do projeto experimental é fundamental. Usar repetições biológicas é um uso mais rentável dos recursos de espectrometria de massa do que repetições de técnicas. O número de repetições necessárias dependerá em parte a variabilidade dos dados. Um estudo recente demonstrou que, enquanto o aumento do número de repetições, além de três apenas marginalmente aumenta o número de identificações, o número de identificações significativas entre grupos aumenta com mais repetições10.
Devido à baixa abundância de fosfoproteínas na célula, quantidades suficientes de proteína inicial são necessárias para obter um phosphoproteome global das amostras de câncer de próstata no modo de descoberta. Nesses experimentos, utilizou-se 5 mg de proteína. Aproximadamente cinco pratos de 15 cm quase confluentes de células fornecem proteína suficiente como entrada no presente protocolo, embora este será célula dependente da linha. Quanto ao tecido do tumor, o rendimento previsto de proteína é cerca de 6-8% do peso do tecido. No cenário em vitro , uma amostra de controlo positivo a considerar é a adição de Vanadato de 1 mM para 30 min antes da colheita das células. Vanadato, um inibidor da fosfatase do competidor proteínas phosphotyrosyl, preservará a fosforilação da tirosina, aumentando assim o número de identificações de peptídeo pY29.
Digestão limpo é um passo chave para maximizar a identificação de Fosfopeptida. Além do teste de mancha de Coomassie, a porcentagem de segmentações perdidas nos dados pode ser usada para avaliar a eficiência da digestão (Figura 2). Software de controle de qualidade está disponível que analisa decotes perdidas e outras métricas para avaliar MS dados qualidade30. Enquanto a tripsina é que as mais comuns, alternativas proteases estão disponíveis5 para endereço cobertura lacunas do proteome onde peptídeos tryptic ideais não podem ser gerado31. As configurações de software de análise do MS seria então necessário ser modificado de acordo para ajustar para alterações em proteases.
O protocolo emprega imunoprecipitação (para enriquecimento do pY) bem como de dióxido de titânio (TiO2) enriquecer para phosphopeptides. Abordagens alternativas para enriquecer para peptídeos incluem cromatografia de afinidade de metal imobilizado (IMAC), outros óxidos de metal para cromatografia de afinidade de óxido de metal (MOAC) tais como hidróxido de alumínio e captura de afinidade do polímero do íon do metal (PolyMAC) 5,12. Estudos anteriores mostraram que os métodos diferentes de enriquecimento enriquecem para diferentes populações de phosphopeptides32. Por exemplo, IMAC enriquece mais multi-fosforilada peptídeos enquanto MOAC enriquece de preferência para mono-fosforilada peptídeos33. Os Resultados do representante do presente protocolo refletem esta observação (Figura 4B). Uma publicação recente demonstrou que combinar IMAC e MOAC usando um material híbrido potencialmente poderia proporcionar maior cobertura de espécies de Fosfopeptida34. Assim, este protocolo pode ser modificado para utilizar outros métodos de enriquecimento em paralelo para permitir análises de phosphoproteomic ainda mais abrangentes.
A suíte de softwares de26 MaxQuant é usada para analisar os dados do MS no presente protocolo, mas aplicações comerciais35 também estão disponíveis para quantificação e identificação de Fosfopeptida. Para a identificação de Fosfopeptida, uma probabilidade de localização corte é aplicada. Este filtro é realizado para selecionar para phosphopeptides com uma alta confiança (ou seja, superior a 0,75) na identificação de phosphoresidue10,28. Em outras palavras, a probabilidade somada de todos os outros resíduos que potencialmente pode conter o phospho-grupo é inferior a 0,25. Este corte pode ser aumentado para aumentar o rigor da seleção Fosfopeptida. No que se refere o número de identificações, o número esperado de peptídeos de pY é na ordem das centenas, enquanto o número esperado de peptídeos de pST é alta milhares. Esses valores refletem a distribuição phosphoproteome observadas anteriormente, onde cerca de 2%, 12% e 86% da phosphosites são pY, pT e pS, respectivamente,28.
Se as etapas de enriquecimento pY e pST são executadas em paralelo, as etapas de preparação de amostra no protocolo podem ser concluídas em seis dias. Emparelhando com a poderosa ferramenta do MS, protocolos de enriquecimento Fosfopeptida como este fornecem uma abordagem global para os cientistas a recolher dados para analisar o phosphoproteome em seus campos respectivos de pesquisa.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos a membros do laboratório de Drake para fornecer conselhos e entrada no manuscrito. Agradecemos também os membros do biológico massa espectrometria de instalações de Robert Wood Johnson medicina e Rutgers, The estado University de New Jersey, para aconselhamento e executando a espectrometria de massa em nossas amostras. Larry C. Cheng é suportado pelo Instituto Nacional de General Medical Ciências, dos institutos nacionais de saúde, sob número prêmio T32 GM008339. Thomas G. Graeber é suportado pelo NCI/NIH (SPORE no câncer de próstata P50 CA092131; P01 CA168585) e um prêmio de estudioso de pesquisa de sociedade americana do câncer (RSG-12-257-01-TBE). Justin M. Drake é suportado pelo departamento de defesa próstata câncer pesquisa programa W81XWH-15-1-0236, câncer de próstata Foundation Young Investigator Award, a Fundação de saúde de Nova Jersey e uma precisão medicina iniciativa piloto prêmio da Rutgers Instituto do câncer de Nova Jersey.
Materiais
| Name | Company | Catalog Number | Comments |
| Ultra-Low Temperature Freezer | Panasonic | MDF-U76V | |
| Freezer -20 °C | VWR | scpmf-2020 | |
| Swing rotor bucket | ThermoFisher Scientific | 75004377 | |
| Vacuum manifold | Restek | 26080 | |
| Lyophilizer | Labconco | 7420020 | |
| CentriVap Benchtop Vacuum Concentrator | Labconco | 7810010 | |
| End-over-end rotator | ThermoFisher Scientific | 415110Q | |
| Razor blade | Fisher Scientific | 620177 | |
| Amicon Ultra-15 Centrifugal Filter Units | Millipore Sigma | UFC901024 | |
| Glass culture tubes | Fisher Scientific | 14-961-26 | |
| Parafilm | Fisher Scientific | 13-374-12 | |
| 20G needle | BD | B305175 | |
| Kimwipes | Fisher Scientific | 06-666A | |
| Screw cap cryotube | ThermoFisher Scientific | 379189 | |
| Nunc 15 mL conical tubes | ThermoFisher Scientific | 12-565-268 | |
| Gel loading tips | Fisher Scientific | 02-707-181 | |
| Millipore 0.2 µm spin filter | Millipore Sigma | UFC30GVNB | |
| Low protein-binding Eppendorf tubes | Eppendorf | 22431081 | |
| anti-Phosphotyrosine, Agarose, Clone: 4G10 | Millipore Sigma | 16101 | |
| 27B10.4 antibody | Cytoskeleton | APY03-beads | |
| Peptide assay kit | Thermo Scientific | 23275 | Step 7 |
| TopTip | PolyLC Inc | TT200TIO.96 | Steps 5 and 9 |
| SCX columns (PolySULFOETHYL A) | PolyLC Inc | SPESE1203 | |
| 3 mL syringe | BD | 309657 | |
| Trifluoracetic Acid (TFA) | Fisher Scientific | PI-28904 | |
| Acetonitrile (ACN) | Fisher Scientific | A21-1 | |
| Lactic acid | Sigma-Aldrich | 69785-250ML | |
| Ammonium Hydroxide | Fisher Scientific | A669S-500 | |
| Potassium Phosphate Monobasic | Fisher Scientific | BP362-500 | |
| Potassium Chloride | Fisher Scientific | BP366-500 | |
| Calcium Chloride Dihydrate | Fisher Scientific | BP510-500 | |
| Tris Base | Fisher Scientific | BP152-5 | |
| Trypsin, TPCK Treated | Worthington Biochemicals | LS003740 | |
| Lysyl Endopeptidase | Wako Pure Chemical Industries, Ltd. | 125-05061 | |
| MonoTip | PolyLC Inc | TT200TIO.96 | Step 10 |
| ZipTip | MilliporeSigma | ZTC18S096 | Step 6 |
| nanoEase, MZ peptide BEH C18, 130A, 1.7 μm, 75 μm x 20 cm | Waters | 186008794 | Step 11: analytical column |
| Acclaim PepMap 100 C18 LC Columns | ThermoFisher Scientific | 164535 | Step 11: trap column |
| Ultimate 3000 RLSCnano System | Dionex | ULTIM3000RSLCNANO | Step 11 |
| Q Exactive HF | ThermoFisher Scientific | IQLAAEGAAPFALGMBFZ | Step 11 |
| MilliQ water | deionized water used to prepare all solutions and bufferes | ||
| Sonic Dismembrator | Fisher Scientific | FB-120 | sonicator |
| Polytron System PT | Kinematica AG | PT 10-35 GT | homogenizer |
Referências
- Siegel, R. L., Miller, K. D., Jemal, A. Cancer statistics, 2018. CA: A Cancer Journal for Clinicians. 68 (1), 7-30 (2018).
- Paller, C. J., Antonarakis, E. S. Management of biochemically recurrent prostate cancer after local therapy: evolving standards of care and new directions. Clinical Advances in Hematology & Oncology. 11 (1), 14-23 (2013).
- Lowrance, W. T., Roth, B. J., Kirkby, E., Murad, M. H., Cookson, M. S. Castration-resistant prostate cancer: AUA guideline amendment 2015. The Journal of Urology. 195 (5), 1444-1452 (2016).
- Domon, B., Aebersold, R. Options and considerations when selecting a quantitative proteomics strategy. Nature Biotechnology. 28 (7), 710-721 (2010).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Bantscheff, M., Schirle, M., Sweetman, G., Rick, J., Kuster, B. Quantitative mass spectrometry in proteomics: a critical review. Analytical and Bioanalytical Chemistry. 389 (4), 1017-1031 (2007).
- Bantscheff, M., Lemeer, S., Savitski, M. M., Kuster, B. Quantitative mass spectrometry in proteomics: critical review update from 2007 to the present. Analytical and Bioanalytical Chemistry. 404 (4), 939-965 (2012).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Rubbi, L., et al. Global phosphoproteomics reveals crosstalk between Bcr-Abl and negative feedback mechanisms controlling Src signaling. Science Signaling. 4 (166), ra18(2011).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045(2018).
- Rush, J., et al. Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nature Biotechnology. 23 (1), 94-101 (2005).
- Fila, J., Honys, D. Enrichment techniques employed in phosphoproteomics. Amino Acids. 43 (3), 1025-1047 (2012).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Drake, J. M., et al. Phosphoproteome integration reveals patient-specific networks in prostate cancer. Cell. 166 (4), 1041-1054 (2016).
- Lue, H. W., et al. Metabolic reprogramming ensures cancer cell survival despite oncogenic signaling blockade. Genes & Development. 31 (20), 2067-2084 (2017).
- Francavilla, C., et al. Phosphoproteomics of primary cells reveals druggable kinase signatures in ovarian cancer. Cell Reports. 18 (13), 3242-3256 (2017).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Skaggs, B. J., et al. Phosphorylation of the ATP-binding loop directs oncogenicity of drug-resistant BCR-ABL mutants. Proceedings of the National Academy of Sciences of the United States of America. 103 (51), 19466-19471 (2006).
- Zimman, A., et al. Activation of aortic endothelial cells by oxidized phospholipids: a phosphoproteomic analysis. Journal of Proteome Research. 9 (6), 2812-2824 (2010).
- Zimman, A., Berliner, J. A., Graeber, T. G. Phosphoproteomic analysis of aortic endothelial cells activated by oxidized phospholipids. Methods in Molecular Biology. , 53-69 (2013).
- Humphrey, S. J., Azimifar, S. B., Mann, M. High-throughput phosphoproteomics reveals in vivo insulin signaling dynamics. Nature Biotechnology. 33 (9), 990-995 (2015).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Meyer, T. S., Lamberts, B. L. Use of coomassie brilliant blue R250 for the electrophoresis of microgram quantities of parotid saliva proteins on acrylamide-gel strips. Biochimica el Biophysica Acta. 107 (1), 144-145 (1965).
- Bergstrom Lind, S., et al. Immunoaffinity enrichments followed by mass spectrometric detection for studying global protein tyrosine phosphorylation. Journal of Proteome Research. 7 (7), 2897-2910 (2008).
- Pinkse, M. W., Uitto, P. M., Hilhorst, M. J., Ooms, B., Heck, A. J. Selective isolation at the femtomole level of phosphopeptides from proteolytic digests using 2D-NanoLC-ESI-MS/MS and titanium oxide precolumns. Analytical Chemistry. 76 (14), 3935-3943 (2004).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 26 (12), 1367-1372 (2008).
- Cox, J., et al. Andromeda: a peptide search engine integrated into the MaxQuant environment. Journal of Proteome Research. 10 (4), 1794-1805 (2011).
- Olsen, J. V., et al. Global, in vivo, and site-specific phosphorylation dynamics in signaling networks. Cell. 127 (3), 635-648 (2006).
- Huyer, G., et al. Mechanism of inhibition of protein-tyrosine phosphatases by vanadate and pervanadate. The Journal of Biological Chemistry. 272 (2), 843-851 (1997).
- Bielow, C., Mastrobuoni, G., Kempa, S. Proteomics quality control: quality control software for MaxQuant results. Journal of Proteome Research. 15 (3), 777-787 (2016).
- Swaney, D. L., Wenger, C. D., Coon, J. J. Value of using multiple proteases for large-scale mass spectrometry-based proteomics. Journal of Proteome Research. 9 (3), 1323-1329 (2010).
- Bodenmiller, B., Mueller, L. N., Mueller, M., Domon, B., Aebersold, R. Reproducible isolation of distinct, overlapping segments of the phosphoproteome. Nature Methods. 4 (3), 231-237 (2007).
- Leitner, A., Sturm, M., Lindner, W. Tools for analyzing the phosphoproteome and other phosphorylated biomolecules: a review. Analytica Chimica Acta. 703 (1), 19-30 (2011).
- Yang, D. S., et al. Design and synthesis of an immobilized metal affinity chromatography and metal oxide affinity chromatography hybrid material for improved phosphopeptide enrichment. Journal of Chromatography A. 1505, 56-62 (2017).
- Al Shweiki, M. R., et al. Assessment of label-free quantification in discovery proteomics and impact of technological factors and natural variability of protein abundance. Journal of Proteome Research. 16 (4), 1410-1424 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados