Method Article
Un test haut débit pour évaluer et quantifier les neutrophiles piège extracellulaire Formation
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit un dosage très sensible et très haut débit de neutrophiles piège extracellulaire (NET) pour la quantification semi-automatisé des ex vivo NET formation par microscopie confocale tridimensionnelle par immunofluorescence. Ce protocole peut être utilisé pour évaluer la formation nette et dégradation après différents stimuli et peut être utilisé pour l’étude des thérapies potentielles axés sur le NET.
Résumé
Les pièges extracellulaires neutrophiles (filets) sont immunogènes structures extracellulaires d’ADN qui peuvent être libérées par les neutrophiles sur une grande variété de déclencheurs. Des filets ont été démontrés pour servir comme un mécanisme de défense du hôte important qui emprisonne et tue les micro-organismes. En revanche, ils ont été impliqués dans diverses maladies auto-immunes systémiques. Les filets sont immunogènes et toxiques des structures contenant un pool d’auto-antigènes pertinentes, y compris les anticorps anti-neutrophile cytoplasmiques (ANCA)-vascularite (AAV) et lupus érythémateux systémique (les) associés. Différentes formes de filets peuvent être induites selon le stimulus. La quantité de filets peut être quantifiée à l’aide de différentes techniques y compris mesure de libération d’ADN dans les surnageants, mesure ADN complexée avec NET-molécules comme la myéloperoxydase (MPO) ou de l’élastase neutrophile (NE), mesurer la présence des citrullinés histones par microscopie de fluorescence, ou détection de cytométrie en flux NET-composants, qui tous ont des caractéristiques différentes au sujet de leur spécificité, la sensibilité, l’objectivité et la quantité. Voici un protocole pour quantifier ex vivo NET formation de manière très sensible et à haut débit à l’aide de la microscopie confocale immunofluorescence en trois dimensions. Ce protocole peut être appliqué pour répondre à diverses questions de recherche sur la formation nette et de la dégradation de la santé et la maladie.
Introduction
La formation de pièges extracellulaires neutrophiles (filets) est le processus dans lequel neutrophiles libèrent leur ADN dans un réseau tridimensionnel extracellulaire de (3D) comme structure, complexé avec un large éventail de molécules antimicrobiennes et dangereux, granulaires et enzymes cytoplasmiques, des peptides et des protéines. Ces structures immunogènes et toxiques ont un rôle physiologique important dans la défense immunitaire innée des individus sains par piégeage et tuer les pathogènes infectieux1. Cependant, ils ont aussi prouvés à être impliqué dans diverses maladies auto-immunes systémiques, y compris les anticorps anti-neutrophile cytoplasmiques (ANCA) et de thrombose2 -associés de vascularite (AAV)3, le lupus érythémateux disséminé (SLE )4,5, syndrome des antiphospholipides (APS)2,6, la polyarthrite rhumatoïde (pr), psoriasis et la goutte7,8,9.
Formation nette in vitro a été largement étudiée avec le phorbol composé chimique 12-myristate 13-acétate (PMA), qui induit la formation nette massive. Toutefois plus de stimuli physiologiques induisent des niveaux beaucoup plus bas de formation nette10. D’étude NET-déclencheurs dans, par exemple, un cadre d’une maladie auto-immune, un test quantitatif normalisé, sensible et à haut débit est nécessaire pour détecter et quantifier la formation nette. Quantification des filets s’est avéré pour être un défi et est actuellement réalisée par des méthodes différentes, chacune avec leurs propres avantages et limitations11. Une méthode couramment utilisée est la détection de l’ADN dans les surnageants12, qui est l’objectif, mais ne fait aucune distinction entre l’origine de l’ADN (apoptose, nécrotique, filets) et n’est donc pas très spécifiques pour les filets. Deuxièmement, les dosages d’immuno-enzymatique (Elisa) d’ADN-complexé avec des protéines spécifiques NET, par exemple, contre la myéloperoxydase (MPO) ou l’élastase neutrophile (NE), sont une approche plus spécifique pour détecter les filets et ont été démontrées à la pour corrélation entre les citrullinés histone-3 (CitH3) positif filets13. Cependant, on ne sait pas si cette méthode est assez sensible pour ramasser tous les filets (p. ex., MPO, NE et CitH3 NETs négatifs). Une troisième approche est la microscopie en immunofluorescence qui sert à détecter la co-localisation des molécules associées aux NET (NE, MPO, CitH3) avec l’ADN extracellulaire pour quantifier les filets. Cette méthode est généralement spécifique pour les filets, mais il ne peut être appliquée comme une méthode de haut-débit et n’est pas objectif en raison de la partialité de l’observateur. En outre, cette méthode ne tient pas compte FTU-, NE-, filets CitH3-négatifs qui sont fréquemment présentes selon les NET-déclencheur utilisé14,15. Flow cytometry approches détectent les filets grâce à une diffusion avant/côté changée (FSC/SSC) indiquant un gonflement du noyau dans NET-ting neutrophiles16. Cette méthode ne tient pas compte des différentes formes de formation nette qui ont été identifiées, qui pourraient impliquer pas de gonflement du noyau, telles que formation nette vital17. Enfin, la microscopie confocale immunofluorescence a été appliquée afin de visualiser et de quantifier la formation nette par coloration directement l’ADN extracellulaire avec un colorant cellulaire-imperméable qui taches extracellulaire ADN12,18. Généralement, 5 à 10 champs haute puissance sont manuellement cueillis et appréciées, qui couvre 1 à 5 % de chaque puits d’une plaque à 96 puits11,17. Sélection manuelle des images n’est pas toujours objective, sujettes à un biais et pas intéressant pour l’analyse de haut-débit. Un essai de quantification NET automatisé et à haut débit a été récemment développé, qui imagés 11 % du puits de façon 3D couvrant 13 µm par microscopie confocale immunofluorescence Z-stack, entraînant ainsi une technique très sensible pour évaluer les filets par rapport aux méthodes conventionnelles10. Le présent rapport décrit le protocole plus récent afin de quantifier la formation nette grâce à un dosage automatisé et très sensible à l’aide de la microscopie confocale 3D, qui atteint une superficie totale de représentation de 45 % de chaque puits et couvre 27 µm par Z-cheminées. Ce protocole est conçu pour quantifier, avec une sensibilité élevée, faibles niveaux de formation nette de façon objective et impartiale.
Protocole
Tous les patients et les témoins sains ont consenti à participer à la biobanque LUMC. Les deux études de biobanques ont été approuvées par le Comité d’éthique LUMC.
1. isolement des neutrophiles sains
- Provenir d’un donneur sain dans deux tubes EDTA-revêtus de 10 mL 20 mL de sang périphérique.
- Mettre 10 mL de sang dans un tube stérile de 50 mL et ajouter en solution saline tamponnée au phosphate (PBS) à 32,5 mL.
-
Ajouter le gradient de densité (par exemple,., gradient de Ficoll-amidotrizoaat) dans les cellules.
- Relever 14 mL de densité de gradient avec une pipette de 10 mL et le régulateur de la pipette.
- Placez la pipette sur la partie inférieure du tube de 50 mL.
- Tenir la pipette contrôleur au large de la pipette, autorisant le gradient de densité s’écouler de la pipette par gravité jusqu'à ce que le maximum est atteint par effet de capillarité (1 à 2 mL restera-t-il), sans utiliser le moteur.
- Enlever pipette en plaçant un pouce sur le dessus de la pipette, empêchant ainsi le gradient de densité de fuir au moment du retrait de la pipette.
- Faites tourner les tubes pendant 20 min à 912 x g et à température ambiante (RT) sans accélération ou de freinage.
Remarque : Globules rouges (RBC) et les neutrophiles ont une densité élevée et sont au fond du tube de 50 mL. Les cellules mononucléaires de sang périphérique (PBMC) sont séparés et sur le dessus du gradient de densité comme un anneau de couleur blanche. PBS-dilué plasma sera au-dessus les PBMC. Si nécessaire, les PBMC peut être isolés en transférant l’anneau blanc dans un nouveau tube de 50 mL avec étapes de lavage supplémentaire avec du PBS. - Retirer délicatement l’anneau blanc contenant des PBMC tout d’abord, suivie de l’élimination du plasma PBS dilué et enfin la couche de gradient de densité autant que possible.
-
Pour isoler les neutrophiles du mélange neutrophile/érythrocytes, lyse érythrocytes par froid eau distillée stérile.
- Prendre la bouteille d’eau distillée stérile froide et un 10 x concentré flacon de PBS dans le réfrigérateur.
- Travailler rapidement pour cette étape. Ajouter 36 mL d’eau distillée stérile froide directement sur le dessus de la pastille et mélanger une fois soigneusement. Ajouter 4 mL de 10 x PBS après 20 s pour faire une solution isotonique. Mélanger une fois soigneusement.
- Tourner le tube pendant 5 min à 739 x g et 4 ° C (pour l’élimination des globules rouges). Neutrophiles sera dans la pastille blanche.
- Soigneusement éliminer le surnageant. Effectuez de nouveau étape 1.6.2 et veillez à ce que la pastille est suspendue correctement.
- Tourner le tube pendant 5 min à 328 x g et 4 ° C.
- Avec précaution, retirez le surnageant et resuspendre le culot dans 5 mL de PBS. Compter les neutrophiles et les garder sur la glace.
Remarque : Rendement prévu de 1 tube de sang (10 mL) est à environ 15 millions de neutrophiles.
2. rouge Fluorescent cellule étiquetage des neutrophiles
- Préparer une suspension neutrophile des neutrophiles 10 millions dans 2 mL de PBS dans un tube de 15 mL.
- Préparer une solution de 2 mL de PBS avec 4 µL de linker de cellule fluorescente rouge 2 µM (voir Table des matières) dans un tube différentes 15 mL. Ajouter délicatement à la suspension de neutrophile et mélanger soigneusement.
- Incuber dans l’obscurité pendant exactement 25 minutes à 37 ° C, d’étiqueter les neutrophiles avec l’éditeur de liens cellule fluorescente rouge.
- Inactiver l’étiquetage en ajoutant un milieu RPMI 1640 contenant 10 % de chaleur inactivé (FCS) de sérum de veau fœtal au RT jusqu'à 15 mL et mélanger une fois soigneusement. Veillez à ce que la pastille est resuspendue soigneusement si une pastille a formé.
- Tourner le tube pendant 5 min à 328 x g et RT.
- Retirez le surnageant et resuspendre le culot dans 5 mL de phénol rouge-libre RPMI 1640 moyen contenant FCS 2 % et 10 % P/S à RT. compter les neutrophiles.
Remarque : Une perte cellulaire de 50 % peut se produire après marquage de cellules fluorescentes rouges.
3. induction de la Formation de neutrophiles Trap extracellulaire
- Faire une suspension de cellules de 0,42 x 106 cellules/mL dans le rouge de phénol gratuit RPMI 1640 moyen contenant 2 % FCS et 10 % P/S.
- Ajouter 37 500 neutrophiles dans 90 µL / puits dans une plaque 96 puits, plat noir.
- Ajouter 10 µL du stimulus choisi (par exemple, le sérum du patient) en trois exemplaires pour atteindre une concentration de 10 % dans le puits. Toujours inclure un contrôle négatif (moyen) en trois exemplaires.
- Incuber à 37 ° C dans l’obscurité pour la durée désirée, allant de 30 min à 2, 4 ou 6 h. incubation pour 4 h est suggéré.
- Calculer le volume nécessaire d’ajouter 25 µL de 5 µM imperméable ADN teindre (voir Table des matières), pour atteindre une concentration finale de 1 µM dans le puits. Faire une dilution si nécessaire en milieu RPMI 1640 moyen contenant 2 % FCS et 10 % P/S pour obtenir une concentration de 5 µM.
- Ajouter 25 µL de 5 colorant µM à ADN imperméable 15 min avant la fin de la période d’incubation. Poursuivre l’incubation pendant 15 min à 37 ° C dans l’obscurité.
- Retirer le surnageant (~ 125 µL) très soigneusement et entreposer si nécessaire. Ajouter 100 µL de paraformaldéhyde à 4 % (PFA). Maintenir dans l’ignorance et passez immédiatement à l’étape 4.
4. NET visualisation avec la microscopie confocale Immunofluorescence 3D de haute résolution, haute teneur
-
Configurer les paramètres sur la microscopie confocale immunofluorescence en cliquant sur le paramétrage de l’Acquisition.
- Cliquez sur l’onglet configurer .
- Sélectionner l’objectif et la caméra. Le choix de l’objectif de 10 X Apo Lambda avec un mode d’acquisition d’un trou d’épingle confocal 60 µm.
- Cliquez sur la plaque et choisissez la plaque 96 puits en plastique. Sélectionner les sites à visiter et de choisir un nombre fixe de sites. Remplir 3 colonnes et 3 lignes sans chevauchement (0 µm), qui couvrent un total de 45 % du puits.
- Cliquez sur l’Acquisition. Sélectionnez activer par Laser mise au point. Le cas échéant, sélectionnez Acquérir série Z/chronologiques .
- Cliquez sur le Site Autofocus.
- Cliquez sur Focus sur fond de plaque | Déport de fond épaisseur. Pour le puits initial trouver de l’échantillon, choisissez le premier puits d’acquis. Pour la mise au point automatique de site, choisissez tous les sites.
- Cliquez sur les longueurs d’onde.
- Pour le nombre de longueurs d’onde, choisissez 2. Pour le niveau de raffinement TL ombrage correction, choisissez 2.
- Pour la longueur d’onde 1, sélectionnez Texas Red.
- Pour les options de mise au point automatique, sélectionnez laser avec décalage Z, poste laser offset 1,1 µm. utilisation Z-pile avec une gamme personnalisée de 200-10.
- Pour la longueur d’onde 2, sélectionnez FITC.
- Pour les options de mise au point automatique, sélectionnez imprimante laser Z offset de w1 0 µm. utilisation Z-pile avec une gamme personnalisée de 200-10.
- Pour les options d’acquisition, sélectionnez série Z et projection 2D image maximale. Pour les options d’acquisition, sélectionnez Shading Correction | Hors.
- Sélectionnez la série Z. Sélectionnez le nombre d’étapes : 10. Sélectionnez la taille d’étape : 3 mm (gamme totale sera 27 µm).
-
Mettre la plaque dans la microscopie confocale immunofluorescence.
- Cliquez sur l’onglet exécuter .
- Remplir de la description et le nom de plaque et choisissez l’emplacement de stockage.
- Sélectionnez les puits qui doivent être acquises.
- Cliquez sur l’onglet exécuter .
- Choisir les temps d’exposition pour Texas Red et FITC.
- Cliquez sur la Plaque d’acquérir pour démarrer l’acquisition, qui prendra environ 1 h par plaque.
5. l’analyse de formation nette
- Utilisez une programme conçu pour l’analyse des images multidimensionnelles scientifiques (voir Table des matières) pour analyser la formation nette de traitement d’image.
- Transférer les données de l’image acquise sur un disque dur distinct.
-
Sélectionnez la couleur, l’ajout d’outil.
- Sélectionnez w1 et choisissez le dossier dans le disque dur où les données sont stockées.
- Sélectionnez w2 et choisissez le dossier dans le disque dur où les données sont stockées.
Remarque : Utiliser une macro standard qui utilise w1 dans le nom du fichier à ajouter de la couleur rouge aux images Texas Red et w2 pour ajouter de la couleur verte aux images FITC.
-
Sélectionnez analyse Macro.
- Sélectionnez w1, choisissez la valeur de seuil (seuil d’intensité), qui est habituellement de 10. Sélectionnez la valeur désirée pixel (seuil de la taille, par exemple, 100).
- Sélectionnez w2, choisissez la valeur de seuil (seuil d’intensité), qui est habituellement de 10. Sélectionnez la valeur désirée pixel (seuil de la taille, par exemple, 500).
- Choisissez la destination pour le fichier de feuille de calcul, exécuter une analyse et enregistrer des fichiers journaux par la suite.
- Analyser les données dans une feuille de calcul.
Résultats
Formation de neutrophiles trap extracellulaire (NET) est quantifiée de façon 3D en quantifiant l’ADN extracellulaire tachée sur 10 Z-piles avec 3 µm distance départ dans le plan focal dans chaque puits. En mesurant la superficie cumulée, la sensibilité de l’augmentation de dosage (Figure 1 a). Les neutrophiles isolés ont une pureté moyenne 98,7 % avec la déviation standard (SD) de 1,10 %, mesurée en 14 différents échantillons d’isolations différentes. Le pourcentage moyen des globules rouges est de 1,04 % ± 1,1 % SD et le pourcentage moyen des monocytes est 0,085 % ± 0,17 % SD (données non présentées). La superficie totale des neutrophiles tachées imagés sont quantifiées que dans le plan focal dans chaque puits, qui sont corrélés significativement avec le nombre de neutrophiles total dans chaque puits avec un coefficient de corrélation de Pearson de 0,99 (95 % intervalle de confiance [IC] 0,985-0,997, p < 0,0001) (Figure 1 b). Les résultats représentatifs de quantification formation nette dans les neutrophiles stimulent avec 10 % des sérums de patients de l’AAV ou moyen (MED) comme témoin négatif, exprimé en cumulé zone tachée d’ADN extracellulaire sur 10 Z-piles par imagés neutrophile (Figure 1 C). instantanés d’images représentatives des neutrophiles non stimulées (Figure 2 a) et des filets dans les neutrophiles stimulée par l’AAV (Figure 2 b) sont indiquées.
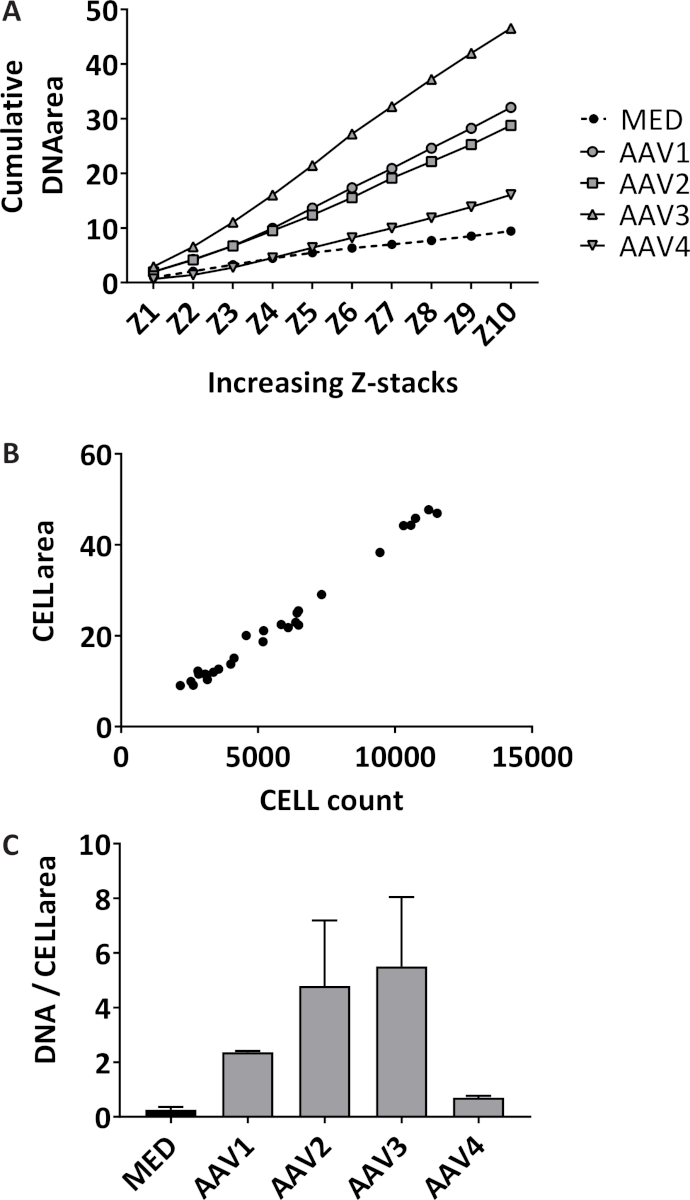
Figure 1 : Quantification de formation nette en mesurant l’ADN extracellulaire et neutrophiles. (A) zone est quantifiée cumulativement sur les 10 Z-piles pour chaque bien pour neutrophiles non stimulées (MED) et pour les neutrophiles stimulée avec 10 % de sérum de vascularites associées aux ANCA patients (AAV) (n = 4) quantifié avec un programme de traitement d’image. Chaque stimulus a été testé en trois exemplaires, chaque point représente la valeur médiane. (B) rouge fluorescent étiqueté zone et cellule globules ont été quantifiés dans le plan focal de chaque puits par le programme de traitement d’image (R2 = 0,99, p < 0,0001). (C) formation nette est exprimée en superficie cumulée par imagés neutrophile (région de la cellule). La moyenne ± erreur-type de la moyenne (SEM) de chaque trois exemplaires sont tracée par stimulation. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
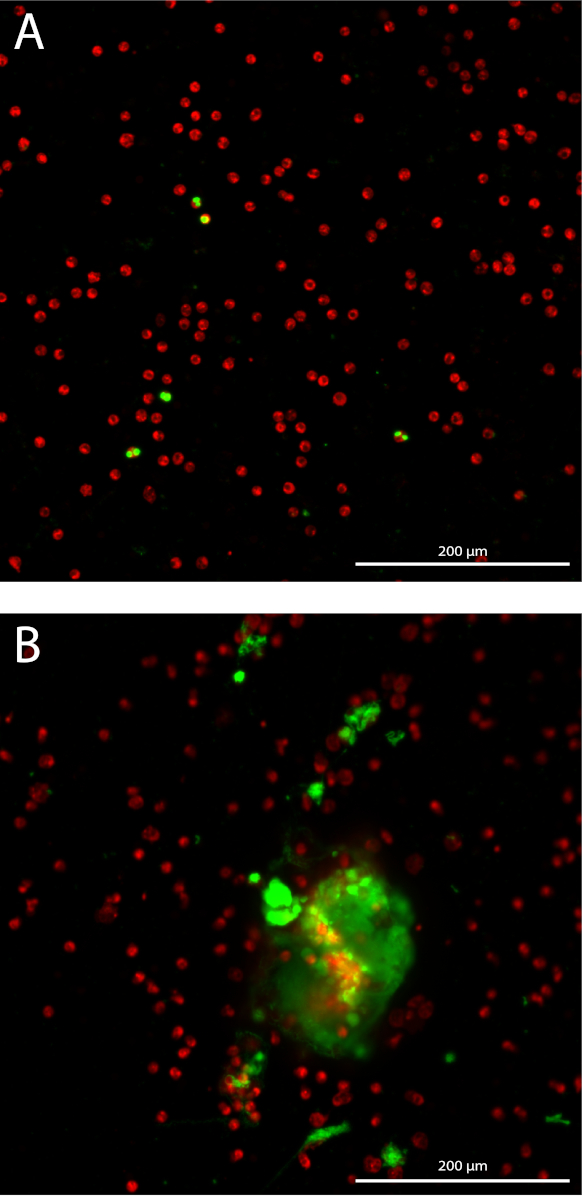
Figure 2 : instantanés d’essai de quantification NET. Fluorescents étiquetés neutrophiles sont indiquées en rouge et tachée ADN extracellulaire s’affiche en vert. 10 x objectif Plan Apo Lambda. Neutrophiles non stimulées (A). (B) neutrophiles stimulés avec sérum AAV. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Le volet essentiel de ce test est la nécessité d’utiliser des neutrophiles fraîchement isolées pour chaque expérience, parce que les neutrophiles sont de courte durées et meurent lorsque congelés. Cela nécessite un donneur sain chaque fois, ce qui pourrait impliquer quelques variations en raison des caractéristiques du donneur. Une de ces variantes est l’état d’activation des neutrophiles. Neutrophiles pourraient être activées déjà in vivo avant l’isolation. En outre, les neutrophiles peuvent être activées dans les étapes d’isolement, notamment lors de la lyse des érythrocytes, un gestionnaire expérimenté des neutrophiles est donc requise pour minimiser l’activation des neutrophiles. En général, isolement des neutrophiles doit être effectuée dès que possible après le dessin de sang et l’expérience ne devrait pas être suspendue afin d’éviter une activation spontanée excessive. Deuxièmement, une manipulation brutale des neutrophiles doit être évitée. Par conséquent, l’avantage notable du protocole décrit est les interventions minimales de pipetage une fois que les neutrophiles sont ensemencées dans une plaque à 96 puits. Ce qui est important, l’état d’activation des neutrophiles est mieux appréciée dans la condition non stimulée, dans lequel ce test peut détecter avec sensibilité de faibles niveaux de formation nette. Un autre facteur qui pourrait influencer le dosage est l’utilisation des centres d’amitié dans le milieu. Le pourcentage de FCS a été réduit de 10 % à 2 % pour éviter la suppression éventuelle formation nette d’antioxydant activité19,20 ou l’activation possible des neutrophiles malgré l’inactivation par la chaleur. Culture sans FCS ou avec l’utilisation de différents types de médias n’a pas été jugée. Un contrôle non stimulée ou moyen est toujours emmené lors de l’exécution de l’essai d’avoir une idée du signal de fond (par exemple, état d’activation des neutrophiles). L’augmentation de pli pour chaque stimulus par rapport à l’échantillon non stimulée s’affiche pour obtenir des résultats cohérents sur différentes expériences à l’aide d’un même stimulus.
Un facteur important pour un fond important possible est la coloration des ADN extracellulaire qui n’est pas lié au processus de formation nette. Le test actuel essaye de réduire cela en supprimant la coloration de l’ADN extracellulaire immédiatement après la courte période d’incubation de 15 min et en analysant la plaque directement après fixation. Par conséquent, il est indispensable d’utiliser un microscope confocal avancé qui a suffisamment de vitesse et puissance d’analyse pour capturer la plaque 96 puits dans 1 à 2 h. automatique réglage de la durée d’exposition et mise au point est recommandé. Ainsi, le paramètre microscope peut varier entre chaque échantillon et expérimenter en ce qui concerne le seuil d’intensité de couleur qui est nécessaire pour une qualité d’image optimale dans l’ensemble. Les influences de ce dernier devrait donc confirmer la capacité éventuelle de quantifier correctement les neutrophiles et les filets et le réglage optimal en utilisant plusieurs échantillons de contrôle (par exemple, le sérum de sujets sains). Lors de l’analyse des images capturées, l’utilisation d’un seuil de pixel et le seuil de la taille dans le programme d’analyse permet une meilleure sélection formation nette.
Extracellulaire ADN dérivé de formation nette peut être le résultat des voies distinctes de la mort, y compris NETosis, necroptosis, pyroptosis, ferroptosis ou même non-lytique processus vital formation nette1. À ce titre, une limitation de l’analyse actuelle est que par coloration seulement pour ADN extracellulaire il n’y aucune distinction possible entre formation nette et autres voies cellulaires pertinents de la mort. Il est possible d’y parvenir en utilisant deux inhibiteurs sélectifs des voies distinctes mort pour distinguer les différentes formes qui sous-tendent la formation nette ou immunostainings séparé pour confirmer la présence de marqueurs NET spécifiques, tels que citH3 et NE, a localisé avec l’ADN. La co-localisation d’ADN extracellulaire avec citH3 et NE a récemment été confirmée pour ce dosage10. L’avantage d’éviter des marqueurs spécifiques NET dans ce test, permet d’évaluer toutes les formes de NET formation menant à l’extrusion de l’ADN par les neutrophiles aussi complets et aussi objective que possible avec le potentiel de criblage à haut débit. L’application du présent protocole a été démontrée dans l’étude de faible induction NET niveau médiée par les complexes immuns dans la maladie auto-immune dans laquelle la capacité à détecter des différences qualitatives et quantitatives peut être plus importante que le type de processus participant10,21,22. Illustrant que cet essai de roman quantification NET peut être de valeur ajoutée pour les différents chercheurs étudier divers aspects de la formation nette. De petits ajustements à l’essai sont facilement mises en œuvre : ajustement de la période de stimulation, l’utilisation d’un marqueur NET favori pour se concentrer sur la voie d’un décès spécifique conduisant à la formation nette, l’utilisation d’un grossissement différent ou l’utilisation des différents critères nets dans la quantification et l’analyse.
En conclusion, le protocole fourni est très sensibles d’un test largement applicable pour la quantification semi-automatisé formation nette pour l’évaluation de l’induction ex vivo des filets à différents stimuli.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le travail de Eline J. Arends et Y.K. Onno Teng est soutenu par la Fondation néerlandaise du rein (17OKG04), bourse de recherche clinique de l’organisation néerlandaise pour la recherche scientifique (90713460). Travail de Laura S. van Dam est soutenu par la Fondation pour la recherche en rhumatologie (FOREUM).
matériels
| Name | Company | Catalog Number | Comments |
| Aqua Sterile H2O | B. Braun, Melsungen, Germany | 12604052 | |
| Fetal bovine serum (FCS) | Bodinco, Alkmaar, The Netherlands | Used in high concentrations it could influcence NET formation | |
| Ficoll 5.7% - amidotrizoaat 9% density 1.077 g/mL | LUMC, Leiden, The Netherlands | 97902861 | |
| Immunofluorescence confocal microscope | Image Xpress Micro Confocal (Molecular Devices, Sunnyvale, CA, USA) | ||
| Neutralization PBS (10x) | Gibco, Paisley, UK | 70011-036 | |
| Penicillin/streptomycin (p/s) | Gibco, Paisley, UK | 15070063 | |
| Phenol red free RPMI 1640 (1x) | Gibco, Paisley, UK | 11835-063 | Phenol red can interfere with the immunofluorescence signal |
| Phosphate-buffered saline (PBS) | B. Braun, Melsungen, Germany | 174628062 | |
| PKH26 2 μM Red fluorescent cell linker | Sigma Aldrich Saint-Louis, MO, USA | PKH26GL-1KT | PKH are patented fluorescent dyes and a cell labeling technology named after their discoverer Paul Karl Horan |
| Program for scientific multidimensional images analysis | ImageJ, Research Services Branch, National Institute of Mental Health, Bethesda, Maryland, USA | ||
| RPMI medium 1640 (1x) | Gibco, Paisley, UK | 52400-025 | |
| Sytox green 5 mM Impermeable DNA dye | Gibco, Paisley, UK | 57020 | |
| Trypan blue stain 0.4% | Sigma Aldrich, Germany | 17942E | |
| 96 well, black, flat bottom, tissue culture treated plate | Falcon, NY, USA | 353219 |
Références
- Jorgensen, I., Rayamajhi, M., Miao, E. A. Programmed cell death as a defence against infection. Nature Reviews Immunology. 17 (3), 151-164 (2017).
- Rao, A. N., Kazzaz, N. M., Knight, J. S. Do neutrophil extracellular traps contribute to the heightened risk of thrombosis in inflammatory diseases?. World Journal of Cardiology. 7 (12), 829-842 (2015).
- Kessenbrock, K., et al. Netting neutrophils in autoimmune small-vessel vasculitis. Nature Medicine. 15 (6), 623-625 (2009).
- Garcia-Romo, G. S., et al. Netting neutrophils are major inducers of type I IFN production in pediatric systemic lupus erythematosus. Science Translational Medicine. 3 (73), 73ra20 (2011).
- Lande, R., et al. Neutrophils activate plasmacytoid dendritic cells by releasing self-DNA-peptide complexes in systemic lupus erythematosus. Science Translational Medicine. 3 (73), 73ra19 (2011).
- Meng, H., Yalavarthi, S., et al. In Vivo Role of Neutrophil Extracellular Traps in Antiphospholipid Antibody-Mediated Venous Thrombosis. Arthritis & Rheumatology. 69 (3), 655-667 (2017).
- Desai, J., et al. Particles of different sizes and shapes induce neutrophil necroptosis followed by the release of neutrophil extracellular trap-like chromatin. Scientific Reports - Nature. 7 (1), 15003 (2017).
- Desai, J., et al. PMA and crystal-induced neutrophil extracellular trap formation involves RIPK1-RIPK3-MLKL signaling. European Journal of Immunology. 46 (1), 223-229 (2016).
- Apel, F., Zychlinsky, A., Kenny, E. F. The role of neutrophil extracellular traps in rheumatic diseases. Nature Reviews Rheumatology. , (2018).
- Kraaij, T., et al. A novel method for high-throughput detection and quantification of neutrophil extracellular traps reveals ROS-independent NET release with immune complexes. Autoimmunity Reviews. 15 (6), 577-584 (2016).
- Masuda, S., et al. NETosis markers: Quest for specific, objective, and quantitative markers. Clinica Chimica Acta. 459, 89-93 (2016).
- Brinkmann, V., Goosmann, C., Kuhn, L. I., Zychlinsky, A. Automatic quantification of in vitro NET formation. Frontiers in Immunology. 3, 413 (2012).
- Nakazawa, D., et al. Enhanced formation and disordered regulation of NETs in myeloperoxidase-ANCA-associated microscopic polyangiitis. Journals of the American Society of Nephrology. 25 (5), 990-997 (2014).
- Konig, M. F., Andrade, F. A Critical Reappraisal of Neutrophil Extracellular Traps and NETosis Mimics Based on Differential Requirements for Protein Citrullination. Frontiers in Immunology. 7, 461 (2016).
- Kenny, E. F., et al. Diverse stimuli engage different neutrophil extracellular trap pathways. Elife. 6, (2017).
- Zhao, W., Fogg, D. K., Kaplan, M. J. A novel image-based quantitative method for the characterization of NETosis. Journal of Immunological Methods. 423, 104-110 (2015).
- Pilsczek, F. H., et al. A novel mechanism of rapid nuclear neutrophil extracellular trap formation in response to Staphylococcus aureus. The Journal of Immunology. 185 (12), 7413-7425 (2010).
- Tanaka, K., et al. In vivo characterization of neutrophil extracellular traps in various organs of a murine sepsis model. PLoS One. 9 (11), e111888 (2014).
- Fuchs, T. A., et al. Novel cell death program leads to neutrophil extracellular traps. Journal of Cell Biology. 176 (2), 231-241 (2007).
- von Köckritz-Blickwede, M., Chow, O., Nizet, V. Fetal calf serum contains heat-stable nucleases that degrade neutrophil extracellular traps. Blood. 114 (25), 5245-5246 (2009).
- Kraaij, T., et al. Excessive neutrophil extracellular trap formation in ANCA-associated vasculitis is independent of ANCA. Kidney International. , (2018).
- Kraaij, T., et al. The NET-effect of combining rituximab with belimumab in severe systemic lupus erythematosus. Journal of Autoimmunity. , (2018).
- Hahn, J., Schauer, C., et al. Aggregated neutrophil extracellular traps resolve inflammation by proteolysis of cytokines and chemokines and protection from antiproteases. The FASEB Journal. (Aug 21), (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon