Method Article
Application de l’empreinte génétique à l’aide du locus D1S80 dans les classes de laboratoire
Dans cet article
Résumé
Nous décrivons ici un protocole simple pour générer des profils d’empreintes génétiques en amplifiant le locus VNTR D1S80 à partir de l’ADN des cellules épithéliales.
Résumé
En sciences biologiques, l’empreinte génétique a été largement utilisée pour les tests de paternité, les applications médico-légales et les études phylogénétiques. Ici, nous décrivons une méthode fiable et robuste pour génotypage des individus par l’analyse du nombre variable de répétition en tandem (VNTR) dans le contexte des classes de laboratoire de premier cycle. Le locus humain D1S80 VNTR est utilisé dans ce protocole comme marqueur hautement polymorphe basé sur la variation du nombre de séquences répétitives.
Ce protocole simple transmet des informations utiles pour les enseignants et la mise en œuvre de l’empreinte génétique dans les cours pratiques de laboratoire. Dans l’exercice de laboratoire présenté, l’extraction de l’ADN suivie d’une amplification par PCR est utilisée pour déterminer la variation génétique au niveau du locus D1S80 VNTR. Les différences dans la taille des fragments des produits de PCR sont visualisées par électrophorèse sur gel d’agarose. La taille des fragments et le nombre de répétitions sont calculés sur la base d’une régression linéaire de la taille et de la distance de migration d’une norme de taille de l’ADN.
En suivant ce guide, les élèves devraient être en mesure de :
• Récolter et extraire l’ADN des cellules épithéliales de la muqueuse buccale
• Effectuer une expérience PCR et comprendre la fonction de divers composants de réaction
• Analyser les amplicons par électrophorèse sur gel d’agarose et interpréter les résultats
• Comprendre l’utilisation des VNTR dans la prise d’empreintes génétiques et son application en sciences biologiques
Introduction
L’empreinte moléculaire, également appelée empreinte génétique, a été introduite par Sir Alec Jeffreys alors qu’il travaillait au département de génétique de l’Université de Leicester en 19841. Il est basé sur le 0,1% du génome humain qui diffère entre les individus et est composé d’environ trois millions de variantes. Ces différences uniques dans le génotype permettent la différenciation entre les individus et peuvent donc fonctionner comme une empreinte génétique à l’exception des jumeaux monozygotiques. Par conséquent, l’empreinte génétique est utilisée pour l’estimation de la relation de différents individus qui est appliquée par exemple dans les tests de paternité ou dans les études sur la diversité des populations. Dans nos cours de laboratoire, nous avons cherché à transmettre le concept d’empreintes génétiques et de fréquences d’allèles. La méthode décrite ici démontre une méthode fiable et robuste pour l’empreinte génétique en analysant le nombre variable de répétitions en tandem (VNTR) au locus D1S80. La méthode comprend l’extraction de l’ADN des cellules épithéliales de la muqueuse buccale et la réaction en chaîne de la polymérase (PCR) subséquente pour amplifier le locus D1S80, suivie de la visualisation des différences de longueur des fragments sur un gel d’agarose.
Le lieu de minisatellite de VNTR D1S80 est fortement variable et situé à la région telomeric du chromosome 1 (1p36-p35). Il a été identifié et décrit par Karsai et ses collègues en 1990 comme un lieu avec une unité de répétition centrale de 16 pb, permettant la séparation des allèles différant d’une seule unité2. En outre, D1S80 montre un degré élevé de variation avec un taux de mutation d’environ 7,77 x 10-53. D1S80 a un degré élevé d’hétérozygosité4,5 (par exemple, 80,8% d’hétérozygosité pour les Caucasiens6 et jusqu’à 87% d’hétérozygosité pour les Afro-Américains7). De plus, le locus D1S80 est polymorphe dans la plupart des populations, avec généralement plus de 15 allèles différents portant 14 à 41 répétitions chacun. La fréquence des allèles D1S80 varie selon les populations. L’allèle à 24 unités répétées (allèle 24) est le plus fréquent dans les populations européennes et asiatiques, tandis que l’allèle 21 est le plus fréquent dans les populations africaines4,7,8,9,10. Par conséquent, les distributions de fréquence des allèles sont diagnostiques pour différentes populations humaines et doivent être prises en compte pour l’estimation de la célérité (p. ex. dans les tests de paternité).
L’amplification par PCR du locus D1S80 VNTR a été une méthode très utile en science médico-légale, tests de paternité, analyse de maladies et études de diversité de population11,12,13,14. Alors qu’en médecine légale aujourd’hui l’utilisation des VNTRs a été remplacée par de courtes répétitions en tandem, le locus VNTR D1S80 est largement utilisé dans la détermination des origines et des relations génétiques entre et entre les populations4,8,9,11. En outre, il est souvent utilisé pour enseigner l’empreinte génétique dans les classes de laboratoire pratique15,16. La méthode décrite ici représente une méthode robuste, rentable et facile à utiliser avec un taux de réussite très élevé dans les classes de laboratoire de premier cycle. Le but de cet article est de fournir une vue d’ensemble du flux de travail pour l’analyse moléculaire du lieu humain de minisatellite D1S80 des cellules épithéliales buccales de mucosa. Il comprend la démonstration de techniques, de protocoles simplifiés et de suggestions pratiques décrites dans des travaux précédemment publiés2,17.
Protocole
REMARQUE: Ce protocole ne doit être utilisé que si les étudiants ou les tuteurs légaux respectifs ont accepté la mise en œuvre de ce protocole, car les profils génotypiques donnent un aperçu des relations génétiques. L’empreinte génétique est une méthode courante de biologie moléculaire principalement appliquée aux études de population et aux questions médico-légales. Par conséquent, il faut s’attacher à maintenir les risques de contamination aussi bas que possible. Pour éviter la contamination de l’échantillon par de l’ADN provenant d’une source extérieure ou de DNases, des gants doivent être portés, les instruments doivent être soigneusement nettoyés ou stérilisés et les solutions doivent être stérilisées par filtre ou autoclavées avant utilisation.
1. Récolte des cellules épithéliales de la muqueuse buccale
ATTENTION : Le travail avec la salive et les cellules épithéliales peut entraîner la transmission de maladies infectieuses. Par conséquent, des précautions normalisées et fondées sur la transmission devraient être appliquées (p. ex. l’utilisation d’un équipement de protection individuelle approprié).
NOTA : Inclure un témoin positif, qui est fourni par l’instructeur (p. ex., l’ADN extrait par l’instructeur) et inclus dans les étapes de traitement en aval.
- Attendez au moins 1 h après avoir mangé ou brossé les dents avant le prélèvement de l’échantillon.
- Étiqueter un tube de microcentrifugation vide et stérile de 2,0 mL.
- Retirez un écouvillon buccal stérile de l’emballage et frottez vigoureusement à l’intérieur de la joue pendant 30 à 40 fois ou 30 à 40 secondes pour récolter les cellules épithéliales de la muqueuse buccale.
- Placez la pointe de l’écouvillon de collecte dans le tube de microcentrifugation stérile précédemment étiqueté et cassez la longueur du plastique qui s’étend au-delà du bord, soit à la main, soit à l’aide de ciseaux stériles.
- Placez le capuchon solidement sur le tube, en scellant l’écouvillon de collecte à l’intérieur.
2. Extraction de l’ADN génomique des cellules humaines
- Avant l’extraction, régler un mélangeur thermique ou un bloc chauffant à 65 °C pour la lyse de l’échantillon.
ATTENTION : Certains produits chimiques utilisés sont classés comme dangereux. Lisez attentivement la fiche de données de sécurité et prenez les mesures de sécurité appropriées avant de la manipuler.- Préparer et stériliser en filtrant ou en autoclavant tous les tampons et solutions nécessaires (solution de lyse, acétate de potassium 8 M, 2-propanol, éthanol à 70% et tampon d’élution).
NOTA: Toutes les étapes de centrifugation doivent être effectuées à température ambiante (20-30 °C), sauf indication contraire.
- Préparer et stériliser en filtrant ou en autoclavant tous les tampons et solutions nécessaires (solution de lyse, acétate de potassium 8 M, 2-propanol, éthanol à 70% et tampon d’élution).
- Ajouter 500 μL de solution de lyse (50 mM Tris/HCl, pH 8,0; acide tétraacétique (EDTA) d’éthylènediamine 10 mM; 2 % de dodécyl sulfate de sodium (FDS)) à l’écouvillon buccal, en s’assurant que l’échantillon est complètement immergé dans la solution de lyse.
- Vortex vigoureusement pendant au moins 5 s.
- Incuber des échantillons à 65 °C dans un mélangeur thermique pendant 10 min.
- Mélanger l’échantillon 3-4 fois par impulsion-vortexing pendant 5 s pendant l’incubation.
- Retirez l’écouvillon du tampon de lyse, appuyez sur l’écouvillon contre l’intérieur du tube pour obtenir le volume d’échantillon maximal.
- Ajouter 100 μL d’acétate de potassium 8 M aux cellules lysées.
- Bien mélanger en inversant le tube jusqu’à ce qu’il y ait un précipité blanc.
- Incuber pendant 5 min à température ambiante.
- Centrifuger l’échantillon pendant 5 min à 18 000 x g.
- Transférer 450 μL du surnageant dans un tube microcentrifuge propre et stérile de 1,5 mL.
- Ajouter 450 μL de 2-propanol et bien mélanger en inversant le tube (précipitation de l’ADN).
- Incuber pendant 5 min à température ambiante.
- Centrifuger pendant 5 min à 18 000 x g.
- Jetez le surnageant et inversez le tube sur une serviette en papier propre pour sécher le granulé et éviter la contamination croisée.
- Incuber l’ADN pendant 5 min à 65 °C dans un bloc chauffant pour sécher complètement la pastille.
- Pour laver l’ADN, ajoutez 500 μL d’éthanol à 70 %.
- Centrifuger pendant 1 min à 18 000 x g.
- Jetez le surnageant et inversez le tube sur une serviette en papier propre pour sécher le granulé.
- Ajouter 30 μL de tampon de remise en suspension (10 mM Tris/HCl pH 8,0, 1 mM d’EDTA) à la pastille.
- Incuber l’ADN pendant 10 min à 65 °C dans un bloc chauffant pour inactiver les adn.
3. Amplifier le locus D1S80 VNTR à l’aide de la PCR
NOTA : Consigner le nombre d’échantillons qui seront utilisés et préparer une feuille de travail avec les réactifs requis et leurs volumes avant de recueillir les plastiques et autres matières nécessaires. Étiquetez les tubes/bandes/plaques stériles à utiliser pour la PCR avec les numéros d’échantillon. N’oubliez pas d’inclure un témoin négatif utilisant H2O au lieu de l’ADN et un témoin positif (p. ex., en utilisant l’ADN fourni par l’instructeur) pour valider la PCR.
- Préparer le mélange maître 1x PCR contenant 10 μL de 5x tampon de réaction PCR (contenant 15 mMMgCl2),1 μL de triphosphate de désoxyribonucléotide (dNTPs) (10 mM), 5 μL (10 pmol) de chaque amorce pMCT118-f et pMCT118-r (avant - 5'-GAAACTGGCCTCCAAACACTGCCCGCCG-3', inverse - 5'-GTCTTGTTGGAGATGCACGTGCCCCTTGC-3') selon Kasaï et coll.2,23,8 μL d’ultrapureH2O,et 0,2 μL d’ADN polymérase Taq (5 U/μL).
REMARQUE: Les amorces pMCT118-f/pMCT118-r ont été conçues sur les régions flanquantes de la région D1S80 VNTR amplifiant l’ensemble du locus. - Étiquetez les tubes/bandes/plaques de PCR avec les numéros d’échantillon à utiliser.
- Aliquote 45 μL du mélange maître à chaque tube ou puits d’échantillon PCR marqué.
- Ajouter 5 μL du gabarit d’ADN au mélange maître dans chaque tube/plaque PCR pour acquérir un volume total de 50 μL. Changer la pointe de la pipette pour chaque échantillon d’ADN afin d’éviter la contamination croisée.
- Inclure un contrôle sans gabarit (NTC) en utilisantH2O ultrapure au lieu de l’ADN.
- Fermez les tubes/bandes de PCR ou scellez la plaque et mélangez.
- Centrifuger les tubes/bandes/plaques de PCR pendant 20 s à l’aide d’une centrifugeuse de table.
REMARQUE: Les conditions de PCR données dans ce protocole ont été optimisées à l’aide de l’ADN polymérase et du cycleur thermique PCR utilisés. En général, les conditions de PCR doivent être adaptées à l’ADN polymérase. Le temps de prolongation standard pour une ADN polymérase Taq est de 1 min/kb. - Placer les tubes/bandes/plaques de l’échantillon dans le thermocycleur et incuber les réactions en utilisant les conditions suivantes (temps d’extension de l’ADN polymérase): 1 cycle de 95 °C pendant 2 min; 25 cycles de 94 °C pendant 15 s, 60 °C pour 15 s, 72 °C pour 30 s; 1 cycle de 72 °C pendant 10 min.
- Lorsque le programme est terminé, retirez les produits du thermocycleur et conservez-les à 4 °C pendant la nuit ou à -20 °C jusqu’à l’électrophorèse.
4. Électrophorèse sur gel d’agarose des produits pcr
- Préparez un gel d’agarose à 1,5%.
- Utiliser 1,5 g de poudre d’agarose et la mélanger avec 100 mL de 1 solution tampon tris-acétate-EDTA (TAE) dans une fiole.
- Chauffer le mélange pendant environ 1,5-2 min dans un four à micro-ondes (600 W). Faites tourbillonner le contenu et chauffer à nouveau, si nécessaire, pour dissoudre complètement l’agarose. Refroidir légèrement et ajouter 2 μL de PeqGreen à l’agarose.
- Versez le gel sous une forme à l’aide d’un peigne avec suffisamment de puits pour tous les échantillons et ajoutez au moins un marqueur de poids moléculaire.
ATTENTION: Retard d’ébullition peut se produire. Agiter soigneusement le ballon lors de la remise en suspension de l’agarose qui n’a pas encore été dissous.
REMARQUE: PeqGreen est un colorant non toxique pour la détection des acides nucléiques. Il est applicable pour la coloration de l’ADN double brin (ADNdb) et de l’ADN simple brin (ADNss) ainsi que de l’ARN. La sensibilité est comparable à celle du bromure d’éthidium.
- Retirer les produits PCR de 4 °C et centrifuger pendant environ 10 s.
- Chargez les puits du gel avec un échantillon de 10 μL. Ne surchargez pas le gel.
REMARQUE: Le tampon d’ADN polymérase comprend également des composés qui augmentent la densité de l’échantillon afin que les échantillons puissent être chargés directement sur des gels sans avoir besoin d’un colorant de chargement. Cela permet à l’échantillon de s’enfoncer dans le puits et aux colorants d’aider à suivre la distance dans la migration de l’échantillon d’ADN.- Ajouter un étalon de poids moléculaire aux puits flanquants (de préférence un étalon de poids moléculaire de 50 pb).
REMARQUE: Conserver les échantillons résiduels de PCR à -20 °C au cas où l’électrophorèse sur gel d’agarose devait être répétée.
- Ajouter un étalon de poids moléculaire aux puits flanquants (de préférence un étalon de poids moléculaire de 50 pb).
- Faire fonctionner le gel dans un tampon d’exécution TAE 1x à 150 V (constante) pendant environ 40 min ou jusqu’à ce que le front de colorant jaune inférieur qui se déplace à environ 50 pb atteigne l’extrémité inférieure du gel.
- Imagez le gel pendant que la lumière bleue ou la lumière ultraviolette (UV) est appliquée et enregistrez une image pour l’analyse de la longueur des fragments.
ATTENTION: La lumière UV peut endommager vos yeux et votre peau. Portez toujours des vêtements de protection et des lunettes de sécurité UV lorsque vous utilisez une boîte à lumière UV. - Éliminer le gel conformément à la politique de l’établissement sur les matières dangereuses.
5. Analyse des résultats de longueur de fragment
Remarque : utilisez l’analyse de régression linéaire pour estimer les longueurs des fragments.
- Pour le dimensionnement des fragments de PCR D1S80, placez une règle sur la photographie de gel sur la voie standard de poids moléculaire de 50 pb de sorte que le haut de la règle s’aligne avec le bas du puits dans lequel l’échantillon a été chargé(Figure 1).
Remarque : Pour calculer l’équation de régression linéaire pour la norme de poids moléculaire (norme de poids moléculaire de 50 pb), un tableur a été utilisé. - Consigner la distance par rapport au puits (p. ex., en cm) pour chaque bande de l’étalon de poids moléculaire de 50 pb dans un tableau à l’aide d’un tableur(figure 2).
- Déterminez le log (p. ex., en base 10) de chaque taille de fragment de la norme de poids moléculaire de 50 pb et entrez les valeurs logarithmiques dans le tableau(figure 2).
- Tracez chaque point de données sur un graphique avec le log des tailles de bande sur l’axe vertical (axe des y) et la distance de course mesurée entre le haut du gel et chaque bande de l’étalon de poids moléculaire sur l’axe horizontal (axe des x) à l’aide d’un nuage de points(Figure 3).
- Ajuster une courbe de tendance (droite de régression linéaire) et afficher l’équation de régression (y = ax + b) et la valeur R2 sur le graphique(Figure 4).
- Mesurer la distance migrée (p. ex., en cm) pour chaque amplicon D1S80 (figure 5).
- Estimer la taille de chaque amplicon D1S80 à l’aide de l’équation de régression : y = ax + b
où y = le journal de la taille du fragment
a = la pente de la ligne (calculée au point 5)
x = la distance du puits (en cm)
b = le point où la droite de régression intercepte l’axe des y (calculé au point 5)
Remarque : étant donné que la valeur y représente le log de la taille de fragment, l’antilog (10y)doit être calculé pour obtenir la taille de fragment en bp des amplicons D1S80.
6. Estimation du nombre d’unités répétées dans les allèles des individus testés
REMARQUE: L’unité de répétition D1S80 est de 16 bp de longueur. Le plus petit allèle connu pour D1S80 a 14 répétitions. Le schéma d’amplicons décrit ici produit des amplicons avec des régions flanquantes additionnant 145 pb supplémentaires à la taille finale.
- Utilisez le tableau 1 pour estimer le nombre d’unités de répétition contenues dans chaque fragment de PCR. La taille extrapolée utilisant la régression linéaire devrait être dans les 8 points d’ébullition de n’importe quel allèle particulier.
- Enregistrer le génotype de chaque individu testé sous la forme d’une combinaison de numéros de taille de répétition d’allèles.
Résultats
En utilisant le protocole décrit, l’analyse du marqueur D1S80 VNTR a été réalisée sur de l’ADN génomique humain extrait de cellules épithéliales de la muqueuse buccale récoltées par prélèvement par écouvillonnage(figure 6). Suite à l’amplification par PCR, une représentation typique d’un gel d’agarose contenant les amplicons D1S80 est représentée sur la figure 1. La voie 1 montre la norme de poids moléculaire de 50 pb. À côté de la norme de poids moléculaire, les produits PCR de huit échantillons d’étudiants sont visualisés. La voie 10 montre le CNT dans lequel l’eau a été utilisée à la place de l’ADN modèle. La plupart des échantillons analysés présentent clairement deux bandes, représentant des individus hétérozygotes pour le locus D1S80. La voie 2 et la voie 9 montrent une seule bande représentant des individus homozygotes pour le locus D1S80. La voie 5 montre une bande unique ambiguë qui est beaucoup plus large par rapport aux autres bandes. Cela pourrait être le résultat de deux allèles D1S80 différant dans une seule unité de répétition. Pour une analyse plus approfondie, l’échantillon dans la voie 5 sera considéré comme une seule bande, représentant un individu homozygote.
L’analyse de fragments d’électrophorèse sur gel en aval effectuée sur des produits de PCR vérifiés a été utilisée pour l’appel de taille du lieu D1S80 VNTR de différents échantillons d’étudiants. L’interprétation des tailles d’amplicons obtenues par analyse PCR repose sur la norme de poids moléculaire utilisée. La distance du puits pour chaque bande de l’étalon de poids moléculaire a été mesurée(figure 1)et enregistrée(figure 2). En fonction de la taille et de la distance de migration, la taille des canaux individuels peut être calculée à l’aide d’une analyse de régression linéaire. Le log (base 10) a été déterminé pour chaque bande dans les échantillons d’étudiants(figure 5)et l’antilogue respectif représente le nombre de pb pour chaque amplicon. Le nombre d’unités de répétition peut être calculé en fonction de la taille de l’amplicon obtenu(tableau 2).
Pour chaque échantillon d’élèves, les tailles des amplicons ont été calculées par régression linéaire et chaque bande pouvait être facilement attribuée à un allèle particulier, comme le montre le tableau 2. Les informations sur la taille des allèles D1S80 peuvent ensuite être utilisées pour déterminer les fréquences des allèles parmi le petit sous-ensemble de population des étudiants de premier cycle. L’allèle à 28 répétitions est le plus fréquent chez les étudiants en raison de deux individus homozygotes pour cet allèle répétitif (individus 1 et 4). Le deuxième allèle le plus fréquent est l’allèle à 18 répétitions, qui est porté par l’individu homozygote 8 et par l’individu hétérozygote 6. Les allèles de basse fréquence sont les allèles 21, 26 et 30 répétitions trouvés qu’une seule fois chez les individus 6, 5 et 3, respectivement. Les modèles de fréquence des allèles du locus D1S80 VNTR de la petite population étudiante peuvent en outre être comparés à la fréquence des allèles des populations humaines plus importantes, par exemple des populations provenant de différents continents4,7,8,9,10.
Parmi les élèves, les individus 2 et 3 partagent l’allèle à 24 répétitions, les individus 2 et 7 partagent l’allèle à 20 répétitions et les individus 5 et 7 partagent l’allèle à 23 répétitions. Fait intéressant, les deux individus homozygotes D1S80 (individus 1 et 4) partagent l’allèle 28-repeat. L’appariement des allèles entre deux individus peut indiquer une relation potentielle. Cependant, les allèles partagés peuvent également se produire par hasard. Ainsi, il est crucial de déterminer la probabilité d’une correspondance d’allèle entre deux individus. La probabilité dépend de la fréquence des allèles individuels dans la population générale et des approches statistiques devraient être utilisées pour attacher un poids statistique à l’analyse des marqueurs VNTR, comme l’approche bayésienne. En résumé, la méthode d’empreintes digitales présentée peut être utilisée dans les pratiques pratiques de cours pratiques pour enseigner l’utilisation des VNTR dans l’empreinte génétique et son application en sciences biologiques.
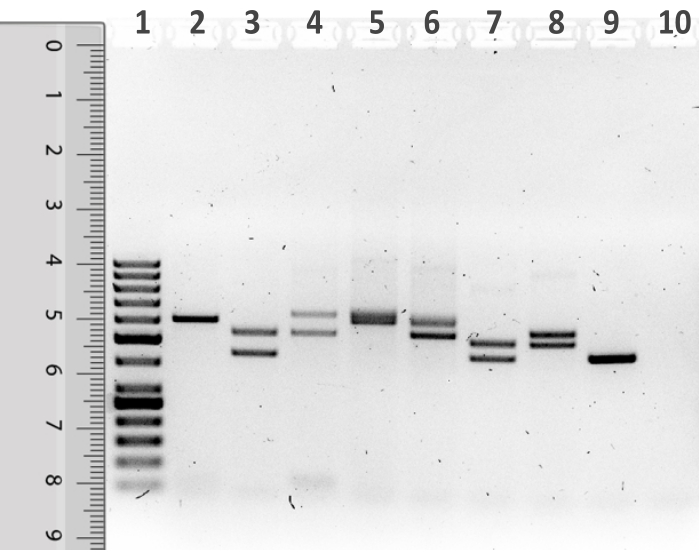
Figure 1: Représentation d’un gel d’agarose après électrophorèse de produits d’amplification D1S80 avec une règle placée à côté de la voie standard de poids moléculaire de 50 pb. La voie 1 contient la norme de poids moléculaire de 50 pb, tandis que les voies 2 à 9 contiennent des produits de réaction PCR. La voie 10 contient le contrôle sans modèle (NTC). Veuillez cliquer ici pour voir une version plus grande de cette figure.
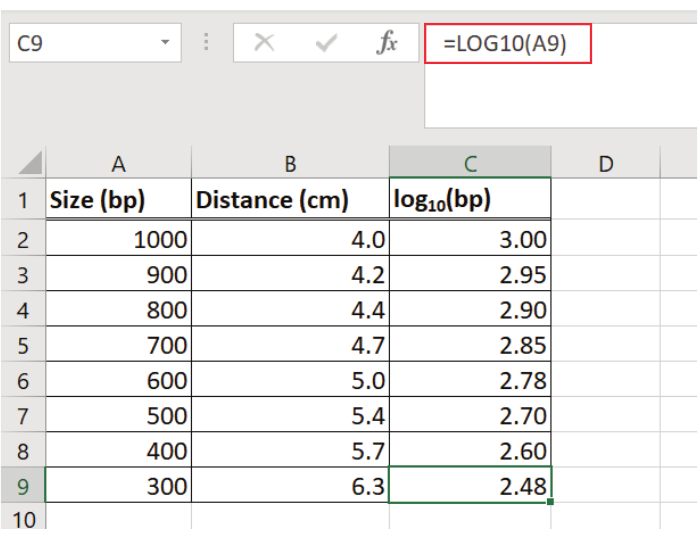
Figure 2: Détermination de la distance de migration des fragments d’ADN et du log de chaque taille de fragment de la norme de poids moléculaire de 50 pb. La distance du puits (en cm) pour chaque bande de l’étalon de poids moléculaire de 50 pb est enregistrée et le log (base 10) pour chaque fragment est déterminé. Les valeurs sont entrées dans un tableau à l’aide d’un tableur. Veuillez cliquer ici pour voir une version plus grande de cette figure.
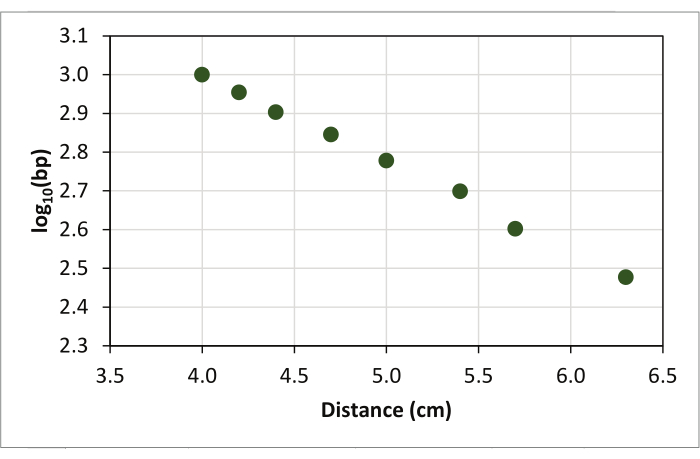
Figure 3: Nuage de points généré en traçant la distance de migration des fragments d’ADN de l’étalon de poids moléculaire sur l’axe des x par rapport au logarithme (base 10) de la taille de chaque fragment sur l’axe des y. Veuillez cliquer ici pour voir une version plus grande de cette figure.
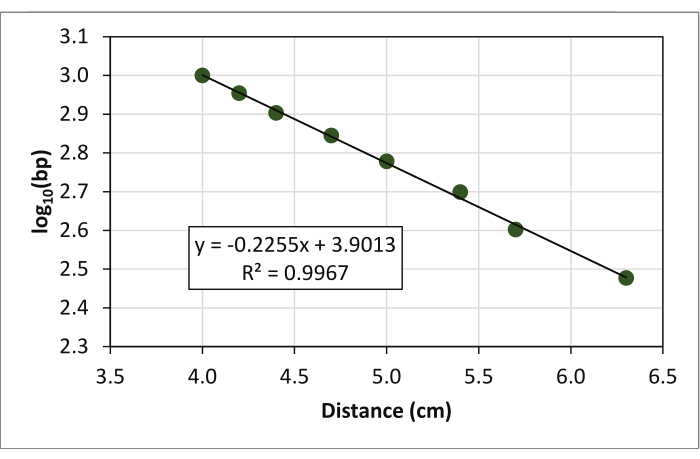
Figure 4: Exploitation de la droite de régression linéaire et de l’équation de régression ainsi que de la valeur R2 pour les données. Veuillez cliquer ici pour voir une version plus grande de cette figure.
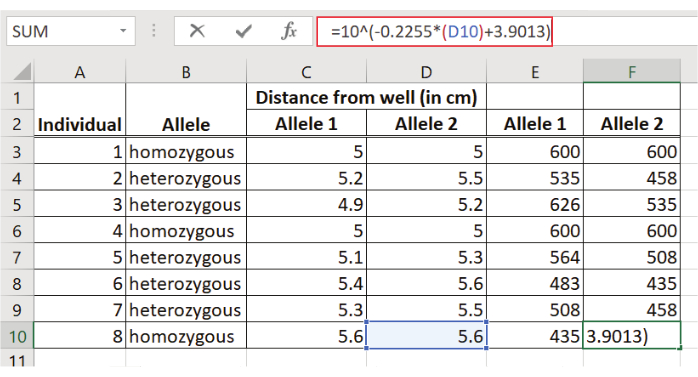
Figure 5: Détermination de la taille des amplicons D1S80. La distance du puits pour chaque fragment est entrée dans l’équation de régression. L’antilog de la valeur y représente la taille du fragment en bp. Veuillez cliquer ici pour afficher une version plus grande de cette figure.
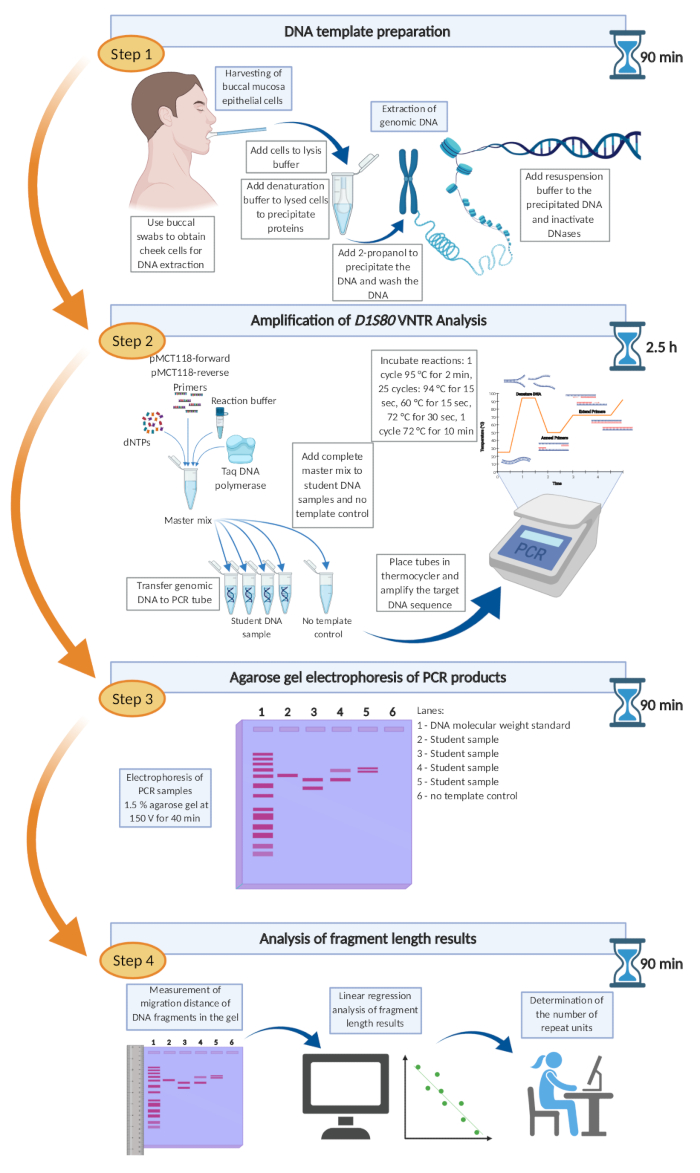
Figure 6: Chronologie et flux de travail suggérés pour l’analyse VNTR D1S80. La procédure entière d’analyse D1S80 VNTR présentée ici, de la récolte buccale de cellules épithéliales à l’évaluation de longueur de fragment, peut être accomplie dans un jour ouvrable simple. Veuillez cliquer ici pour voir une version plus grande de cette figure.
| Taille des allèles (en pb) | Nombre de répétitions |
| 369 | 14 |
| 385 | 15 |
| 401 | 16 |
| 417 | 17 |
| 433 | 18 |
| 449 | 19 |
| 465 | 20 |
| 481 | 21 |
| 497 | 22 |
| 513 | 23 |
| 529 | 24 |
| 545 | 25 |
| 561 | 26 |
| 577 | 27 |
| 593 | 28 |
| 609 | 29 |
| 625 | 30 |
| 641 | 31 |
| 657 | 32 |
| 673 | 33 |
| 689 | 34 |
| 705 | 35 |
| 721 | 36 |
| 737 | 37 |
| 753 | 38 |
| 769 | 39 |
| 785 | 40 |
| 801 | 41 |
Tableau 1: Taille de l’amplicon pour chaque locus VNTR D1S80.
| individu | allèle | Distance (en cm) | Taille (en pb) | Nombre d’unités de répétition |
| 1 | homozygote | 5 | 600 | 28 |
| 2 | hétérozygote | 5.2 ; 5.5 | 535, 458 | 24, 20 |
| 3 | hétérozygote | 4.9 ; 5.2 | 626, 535 | 30, 24 |
| 4 | homozygote | 5 | 600 | 28 |
| 5 | hétérozygote | 5.1 ; 5.3 | 564, 508 | 26, 23 |
| 6 | hétérozygote | 5.4 ; 5.6 | 483, 435 | 21, 18 |
| 7 | hétérozygote | 5.3 ; 5.5 | 508, 458 | 23, 20 |
| 8 | homozygote | 5.6 | 435 | 18 |
Tableau 2 : Composition des allèles des différents individus testés. Le nombre d’unités répétées peut être approché en fonction de la taille des amplicons obtenue par analyse de régression linéaire.
Discussion
Ici, nous avons décrit une méthode simple et rentable pour mettre en œuvre l’empreinte moléculaire dans les cours pratiques de premier cycle.
Pour dépister la variation génétique au locus D1S80, la collecte d’échantillons biologiques humains ainsi que l’extraction et l’analyse de l’ADN sont nécessaires. Il est essentiel que l’utilisation éthique des échantillons biologiques humains soit assurée tout au long du processus. La gestion des échantillons est contrôlée dans un cadre réglementaire complet qui garantit l’utilisation correcte des échantillons et des données connexes18 (p. ex. consentement à l’utilisation de matériel biologique humain). Les participants doivent être bien informés de l’utilisation de leurs échantillons, du risque de découverte d’anomalies dans les relations génétiques (p. ex. pour les personnes apparentées), de la protection de la vie privée et des intentions de stockage futur des spécimens et des données biologiques. Tous les donateurs (étudiants ou collègues) doivent donner leur consentement librement et comprendre le droit de se retirer sans donner de raison. En général, il est indispensable de se familiariser avec les directives et réglementations respectives pour la gestion des échantillons humains avant de mener ce cours de laboratoire.
Pour la collecte de cellules épithéliales de la muqueuse buccale dans les cours de laboratoire de premier cycle, il faut prendre soin d’éviter la contamination des échantillons d’ADN par l’ADN de l’opérateur ou par des DNases. Il est recommandé d’utiliser des sarraus de laboratoire, des gants et des lunettes de protection ainsi que des écouvillons stériles et des tubes à microcentrifugation. La récolte de cellules à base d’écouvillons buccaux est considérée comme une méthode pratique et rentable pour la collecte de matériel génétique adapté à l’analyse VNTR basée sur pcr, car elle est relativement peu coûteuse et non invasive. Outre les écouvillons buccaux, la salive est la méthode d’échantillonnage oral la plus courante pour la recherche médicale. Il a été démontré que les écouvillons buccaux contiennent une proportion plus élevée de cellules épithéliales que la salive, ce qui les rend plus fiables pour fournir une quantité et une qualité suffisantes d’ADN pour l’analyse D1S80 VNTR basée sur pcr19,20. De plus, les écouvillons buccaux peuvent être envoyés par la poste après l’auto-collecte en surmontant les obstacles géographiques lorsqu’ils sont utilisés, par exemple, dans les études sur la diversité despopulations 21.
L’extraction de l’ADN est devenue relativement facile grâce au développement de kits d’extraction de différentes entreprises. Néanmoins, l’utilisation de ces trousses peut ne pas convenir dans les classes de laboratoire en raison de ressources financières limitées. Ici, nous avons présenté une méthode simple, rapide et rentable pour l’extraction de l’ADN à partir d’échantillons humains sans avoir besoin d’un kit d’extraction d’ADN disponible dans le commerce. La méthode d’extraction de l’ADN décrite n’utilise pas de solvants organiques, mais les protéines cellulaires sont éliminées en augmentant la concentration en sel à l’aide d’une solution d’acétate de potassium 8 M. Cette méthode permet également de manipuler plusieurs échantillons à la fois et donne suffisamment d’ADN pour plusieurs analyses de génotype pour chaque échantillon.
La PCR est une technique courante dans de nombreux laboratoires moléculaires. Bien qu’ils soient généralement exempts de problèmes, il existe des pièges qui peuvent compliquer la réaction produisant des résultats fallacieux, qui ont été discutés ailleurs22. Les conditions de PCR présentées ici ont semblé très robustes face à la mauvaise qualité d’ADN de modèle, mais le manque d’amplification de PCR a néanmoins été occasionnellement observé en utilisant des modèles avec des concentrations extrêmement basses d’ADN dues à la mauvaise récolte buccale de cellules épithéliales. Les amorces d’ADN utilisées dans cette étude peuvent être commandées auprès de différentes entreprises, telles que celles citées dans le tableau des matériaux à la fin de cet article. Des précautions particulières doivent être prises lorsque vous travaillez avec des amorces d’ADN afin d’éviter la contamination par des DNases ou l’ADN de l’opérateur. Les produits PCR doivent également être conservés au froid lorsqu’ils ne sont pas utilisés.
Une autre étape critique est l’électrophorèse sur gel d’agarose. Les fragments différant d’une seule unité de répétition de 16 pb peuvent ne pas être séparés dans l’électrophorèse sur gel et apparaissent ainsi comme une seule bande. Dans ce cas, une détermination correcte du nombre d’unités répétées des allèles D1S80 testés ne peut être assurée. Par conséquent, le temps d’exécution du gel doit être augmenté tandis que la tension doit être réduite et la concentration du gel peut être augmentée pour fournir une meilleure résolution et séparation des bandes simples.
Il convient de mentionner que tous les allèles d’un locus VNTR ne contiennent pas d’unités de répétition complètes. Les allèles non consensuels (microvariants) qui contiennent des unités de répétition incomplètes sont communs à la plupart des loci VNTR et leurs tailles tombent entre les tailles des allèles avec des unités de répétition complète. Ces microvariants sont à peine détectables par électrophorèse sur gel d’agarose. En revanche, des techniques comme les gels de polyacrylamide ou l’électrophorèse capillaire peuvent résoudre des allèles qui diffèrent d’une à quelques unités de répétition ou microvariants12,23,24,25,26. Cependant, ces dernières techniques conviennent moins aux classes de laboratoire de premier cycle car elles présentent de nombreux inconvénients, notamment l’utilisation de composés dangereux, la préparation complexe et le manque d’équipement. Pour la détermination de la taille des fragments, une attention particulière doit être prise lors de la mesure de la distance des fragments d’ADN migrés. Si les bandes sont trop diffuses pour être mesurées avec précision, le calcul de la taille du fragment d’allèle D1S80 par régression linéaire peut être incorrect, ce qui entraîne une mauvaise estimation des nombres unitaires répétés. Dans ce cas, une réexécuter l’électrophorèse sur gel d’agarose après optimisation des conditions de gel est conseillée comme décrit précédemment par Lorenz et ses collègues22.
Le procédé entier d’analyse de D1S80 VNTR présenté ici, de la récolte buccale de cellules épithéliales à l’évaluation de longueur de fragment, peut être accompli en une seule journée de travail. Ce protocole est une méthode robuste, rentable et facile à utiliser adaptée aux cours de laboratoire pratique de premier cycle.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs tiennent à remercier Kevin Graham pour sa voix off et tous les participants qui ont fait don d’échantillons pour ce travail.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Sarstedt | 72,706 | autoclaved |
| 2 mL microcentrifuge tube | Sarstedt | 7,26,95,500 | autoclaved |
| 2-propanol | Fisher chemical | PI7508/17 | |
| 5x PCR buffer | Promega | M7845 | |
| Acetic acid | Sigma/Merck | A6283-500ML | |
| Agarose | BioBudget | 10-35-1020 | |
| Buccal swabs | BioBudget | 57-BS-05-600 | |
| Centrifuge | Eppendorf | 5430 | |
| Combs | BioRad | ||
| dNTPs (10 mM) | Invitrogen | R0192 | |
| EDTA | Carl Roth | 8043.1 | |
| Ethanol | Honeywell | 32221-2.5L | |
| Gel caster | BioRad | ||
| Gel chamber | BioRad | SubCell GT | |
| Gel Doc XR+ | BioRad | ||
| Geltray | BioRad | SubCell GT | |
| GeneRuler 50 bp DNA Ladder | Thermo Fisher | SM0371 | |
| Go-Taq Polymerase | Promega | M7845 | |
| Heating block | Eppendorf | Thermomixer | 1.5 mL and 2 mL |
| Microwave | Sharp | R-941STW | |
| peqGreen | VWR | 732-3196 | |
| Pipettes | Gilson | 1000 µL, 200 µL, 20 µL, 2 µL | |
| Potassium acetate | Arcos Organics | 217100010 | |
| PowerPac power supply | BioRad | 100-120/220-240V | |
| Primers | Sigma/Merck | ||
| Scale | Kern | PCB 2.5 kg | |
| SDS | Arcos Organics | 218591000 | |
| Shaker | IKA labortechnik | IKA RH basic | |
| Tabletop centrifuge | myFuge | ||
| Thermal Cycler | BioRad | C100 Touch | |
| Tris | VWR | 103156x | |
| Vortex mixer | LMS | VTX-3000L |
Références
- Jeffreys, A. J., Wilson, V., Thein, S. L. Hypervariable 'minisatellite' regions in human DNA. Nature. 314 (6006), 67-73 (1985).
- Kasai, K., Nakamura, Y., White, R. Amplification of a variable number of tandem repeats (VNTR) locus (pMCT118) by the polymerase chain reaction (PCR) and its application to forensic science. Journal of Forensic Science. 35 (5), 1196-1200 (1990).
- Balamurugan, K., Tracey, M. L., Heine, U., Maha, G. C., Duncan, G. T. Mutation at the human D1S80 minisatellite locus. The Scientific World Journal. 2012 (917235), (2012).
- Herrera, R. J., Adrien, L. R., Ruiz, L. M., Sanabria, N. Y., Duncan, G. D1S80 single-locus discrimination among African populations. Human Biology. 76 (1), 87-108 (2004).
- Lauritzen, S. L., Mazumder, A. Informativeness of genetic markers for forensic inference - An information theoretic approach. Forensic Science International: Genetics Supplement Series. 1 (1), 652-653 (2008).
- Budowle, B., Chakraborty, R., Giusti, A. M., Eisenberg, A. J., Allen, R. C. Analysis of the VNTR locus D1S80 by the PCR followed by high-resolution PAGE. American Journal of Human Genetics. 48 (1), 137 (1991).
- Budowle, B., et al. D1S80 population data in African Americans, Caucasians, southeastern Hispanics, southwestern Hispanics, and Orientals. Journal of Forensic Science. 40 (1), 38-44 (1995).
- Verbenko, D. A., et al. Polymorphisms at locus D1S80 and other hypervariable regions in the analysis of Eastern European ethnic group relationships. Annals of Human Biology. 33 (5-6), 570-584 (2006).
- Walsh, S. J., Eckhoff, C. Australian Aboriginal population genetics at the D1S80 VNTR locus. Annals of Human Biology. 34 (5), 557-565 (2007).
- Limborska, S. A., Khrunin, A. V., Flegontova, O. V., Tasitz, V. A., Verbenko, D. A. Specificity of genetic diversity in D1S80 revealed by SNP-VNTR haplotyping. Annals of Human Biology. 38 (5), 564-569 (2011).
- Sajib, A. A., Yeasmin, S., Akter, M., Uddin, M. A., Akhteruzzaman, S. Phylogenetic analysis of Bangladeshi population with reference to D1S80 VNTR locus. Bioresearch Communications. 2 (1), 146-151 (2016).
- Köseler, A., Atalay, A., Atalay, E. &. #. 2. 1. 4. ;. Allele frequency of VNTR locus D1S80 observed in Denizli province of Turkey. Biochemical genetics. 47 (7-8), 540-546 (2009).
- Mastana, S. S., Papiha, S. S. D1S80 distribution in world populations with new data from the UK and the Indian sub-continent. Annals of Human Biology. 28 (3), 308-318 (2001).
- Okuda, H., et al. A Japanese propositus with D-- phenotype characterized by the deletion of both the RHCE gene and D1S80 locus situated in chromosome 1p and the existence of a new CE-D-CE hybrid gene. Journal of Human Genetics. 45 (3), 142-153 (2000).
- Campbell, A. M., Williamson, J. H., Padula, D., Sundby, S. Use PCR & a Single Hair to Produce a "DNA Fingerprint.". The American Biology Teacher. 59 (3), 172-178 (1997).
- Jackson, D. D., Abbey, C. S., Nugent, D. DNA profiling of the D1S80 locus: A forensic analysis for the undergraduate biochemistry laboratory. Journal of Chemical Education. 83 (5), 774 (2006).
- Mansoor, S. K., Hussein, E. F., Ibraheem, A. K. Use the Variable Number of Tandem Repeats (VNTR) in DNA fingerprinting and its application biological sciences. EurAsian Journal of BioSciences. 14 (2), 2835-2839 (2020).
- Beier, K., Schnorrer, S., Hoppe, N., Lenk, C. The ethical and legal regulation of human tissue and biobank research in Europe. Proceedings of the Tiss. EU Project. , (2011).
- Aida, J., et al. Telomere length variations in 6 mucosal cell types of gastric tissue observed using a novel quantitative fluorescence in situ hybridization method. Human Pathology. 38 (8), 1192-1200 (2007).
- Theda, C., et al. Quantitation of the cellular content of saliva and buccal swab samples. Scientific Reports. 8 (1), 1-8 (2018).
- McMichael, G. L., et al. DNA from buccal swabs suitable for high-throughput SNP multiplex analysis. Journal of biomolecular techniques: JBT. 20 (5), 232 (2009).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Destro-Bisol, G., Capelli, C., Belledi, M. Inferring microevolutionary patterns from allele-size frequency distributions of minisatellite loci: a worldwide study of the APOB 3'hypervariable region polymorphism. Human Biology. 72 (5), 733-751 (2000).
- Chen, B., et al. Structure and function of alleles in the 3'end region of human apoB gene. Chinese Medical Journal. 112 (3), 221-223 (1999).
- Renges, H. -. H., Peacock, K., Dunning, A. M., Talmud, P., Humphries, S. E. Genetic relationship between the 3 '-VNTR and diallelic apolipoprotein B gene polymorphisms: Haplotype analysis in individuals of European and South Asian origin. Annals of Human Genetics. 56 (1), 11-33 (1992).
- Kravchenko, S. A., Maliarchuk, O. S., Livshits, L. A. A population genetics study of the allelic polymorphism in the hypervariable region of the apolipoprotein B gene in the population of different regions of Ukraine. Tsitologiia i Genetika. 30 (5), 35-41 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon