Method Article
Surveillance des interactions protéine-ligand dans les cellules humaines par RMN quantitative intracellulaire en temps réel à l’aide d’un bioréacteur à haute densité cellulaire
Dans cet article
Résumé
Ce protocole décrit la configuration d’un bioréacteur RMN pour maintenir les cellules humaines encapsulées viables jusqu’à 72 h, suivie de l’acquisition et de l’analyse de données RMN dans les cellules résolues dans le temps. La méthodologie est appliquée pour surveiller les interactions protéine-ligand intracellulaire en temps réel.
Résumé
La RMN cellulaire est une approche unique pour observer les propriétés structurelles et dynamiques des macromolécules biologiques à résolution atomique directement dans les cellules vivantes. Le repliement des protéines, les modifications chimiques et les changements conformationnels induits par la liaison au ligand peuvent être observés. Par conséquent, cette méthode a un grand potentiel dans le contexte du développement de médicaments. Cependant, la courte durée de vie des cellules humaines confinées dans le spectromètre RMN limite la plage d’application de la RMN dans les cellules. Pour surmonter ce problème, des bioréacteurs RMN sont utilisés qui peuvent grandement améliorer la stabilité de l’échantillon cellulaire au fil du temps et, surtout, permettre l’enregistrement en temps réel des spectres RMN dans les cellules. De cette façon, l’évolution des processus tels que la pénétration du ligand et la liaison à la cible protéique intracellulaire peut être surveillée en temps réel. Les bioréacteurs sont souvent limités par une faible viabilité cellulaire à un nombre élevé de cellules, ce qui entraîne un compromis entre la sensibilité globale de l’expérience et la viabilité cellulaire. Nous avons récemment signalé un bioréacteur RMN qui maintient un grand nombre de cellules humaines métaboliquement actives pendant de longues périodes, jusqu’à 72 h. Cette configuration a été appliquée pour surveiller les interactions protéine-ligand et la modification chimique des protéines. Nous avons également introduit un flux de travail pour l’analyse quantitative des données RMN en temps réel, basé sur la résolution de courbe multivariée. La méthode fournit des profils de concentration des espèces chimiques présentes dans les cellules en fonction du temps, qui peuvent être analysés plus en détail pour obtenir des paramètres cinétiques pertinents. Nous fournissons ici une description détaillée de la configuration du bioréacteur RMN et de son application à la surveillance des interactions protéine-ligand dans les cellules humaines.
Introduction
La spectroscopie par résonance magnétique nucléaire (RMN) dans les cellules est récemment apparue comme une approche puissante pour étudier les propriétés structurelles et dynamiques des macromolécules dans l’environnement cellulaire1,2,3,4,5,6. La RMN cellulaire a réussi à étudier des processus fonctionnellement pertinents tels que le repliement/mauvais repliement des protéines7,8,9, la liaison aux métaux7,10, la formation de liaisons disulfures11,12 et l’interaction protéine-protéine13, l’interaction protéine-ligand14,15,16 et l’interaction acide nucléique-ligand17 ,18 dans des cellules humaines vivantes. L’un des facteurs limitants des applications de RMN dans les cellules est la courte durée de vie des cellules pendant l’expérience. La solution à ce problème implique l’utilisation de bioréacteurs RMN. Dans ces dispositifs, un flux constant de milieu de croissance est appliqué aux cellules, qui sont maintenues confinées dans le spectromètre RMN, afin de fournir de l’oxygène et des nutriments et d’éliminer les sous-produits toxiques. Suite à l’avènement de la RMN cellulaire, plusieurs conceptions de bioréacteurs RMN ont été développées pour améliorer la viabilité cellulaire pendant de plus longues périodes, dans lesquelles des bactéries ou des cellules de mammifères sont encapsulées dans un hydrogel19,20,21,22 ou maintenues en suspension et perfusées à l’aide d’une membrane de microdialyse23 . De tels bioréacteurs ont permis l’acquisition d’expériences RMN plus longues avec un rapport signal/bruit (S/N)5 accru et, plus important encore, pourraient être utilisés pour étudier les processus cellulaires en temps réel22,23,24. Grâce à la haute sensibilité chimique et conformationnelle de la RMN, cette dernière application peut fournir des informations précieuses sur la cinétique des processus fonctionnels dans les cellules vivantes à résolution atomique.
Dans ce protocole, nous montrons comment mettre en place et exploiter un bioréacteur amélioré récemment rapporté25, qui a été obtenu en combinant une conception de bioréacteur modulaire existante23 avec l’approche reposant sur l’encapsulation cellulaire dans l’hydrogel qui ont été lancées par d’autres groupes19,20,21,22,26,27 . Nous décrivons l’application du bioréacteur à des études RMN intracellulaires en temps réel de la liaison intracellulaire des ligands observés par les protéines dans les cellules HEK293T. Dans le bioréacteur, les cellules sont encapsulées à haute densité dans des fils de gel d’agarose et sont maintenues très viables et métaboliquement actives jusqu’à 72 h, au cours desquelles des expériences de RMN cellulaire en temps réel sont enregistrées. Le bioréacteur est composé d’un tube en verre qui s’adapte aux sondes RMN standard de 5 mm, étanche à l’eau et relié à un porte-tube, de sorte que la chambre d’échantillonnage interne ait un diamètre intérieur de 4,2 mm, une hauteur de 38 mm et un volume de 526 μL. L’entrée est un capillaire en PEEK de 7 mètres de long (o.d. = 1/32 », i.d. = 0,5 mm) inséré dans la chambre d’échantillonnage jusqu’à ~6 mm à partir du bas, tandis que la sortie est un capillaire PTFE de 7 mètres de long (o.d. = 1/32 », i.d. = 0,5 mm) fixé en haut du porte-tube (Figure 1). Le tube est inséré coaxialement dans une conduite à température contrôlée reliée à un bain-marie. L’entrée et la sortie sont reliées par un tube en PEEK à une vanne à 4 voies à 2 positions fixée à une pompe FPLC pour contrôler le débit moyen et un conteneur à déchets.
Le bioréacteur est appliqué pour étudier la cinétique de l’interaction, précédemment rapportée14,25, entre deux médicaments, l’acétazolamide (AAZ) et le méthazolamide (MZA), dans des cellules humaines avec la deuxième isoforme de l’anhydrase carbonique humaine (CA II), une cible pharmacologiquement pertinente28,29,30, et la cinétique de la formation de la liaison disulfure intramoléculaire, favorisée par la petite molécule ebselen25, 31, de la forme sans cuivre et liée au zinc du cuivre humain, la superoxyde de zinc dismutase (SOD1), une enzyme antioxydante impliquée dans l’apparition de la sclérose latérale amyotrophique7,8,32. Enfin, l’analyse quantitative des données RMN en temps réel est effectuée dans MATLAB à l’aide de l’algorithme MCR-ALS (Multivariate Curve Resolution-Alternating Least Squares)33, grâce auquel les composants spectraux purs et les profils de concentration en fonction du temps sont obtenus pour les espèces observées, qui peuvent être analysés plus avant pour obtenir des paramètres cinétiques pertinents.
Le protocole commence à partir d’une fiole T75 de cellules HEK293T (~3 x 107 cellules par flacon) surexprimant transitoirement soit l’AC II humaine (non marquée), soit la SOD1 humaine (marquée 15N). Les cellules ont été cultivées et maintenues dans des flacons de T75 avec un taux de glucose élevé en DMEM par 1:10 passages tous les 3-4 jours et transfectées avec l’ADNc codant pour la protéine d’intérêt 48 h avant l’expérience. Les étapes de cette phase sont décrites en détail ailleurs34.
Protocole
1. Configuration du réactif et de la solution
- Pour préparer le DMEM complet, ajouter 5 mL de L-glutamine 200 mM, 5 mL de pénicilline-streptomycine 100x et 50 mL de sérum fœtal bovin (FBS, concentration finale de 10 % vol/vol) à 440 mL de DMEM.
REMARQUE: Cette solution peut être conservée à 4 °C pendant 1 mois. - Préparer une solution d’agarose en dissolvant 150 mg d’agarose à faible teneur en gélification dans 10 mL de solution saline tamponnée au phosphate (PBS) à 85 °C pour obtenir une solution à 1,5 % (p/v). Stériliser par filtration avec un filtre de 0,22 μm. Préparer 1 mL d’aliquotes de solution d’agarose dans des tubes coiffés de 1,5 mL et conserver à 4 °C.
- Préparer le milieu du bioréacteur.
- Dissoudre 13,4 g de poudre de DMEM dans 1 L de H2O ultrapur.
REMARQUE: Selon l’application, le volume final requis peut différer (par exemple, pour un milieu de 500 mL, dissoudre 6,7 g de poudre dans 500 mL de H2O). - Ajouter 2 % de FBS, 10 mM de NaHCO3, 1x pénicilline-streptomycine (100x) et 2 % de D2O (p. ex., pour un milieu de 500 mL, ajouter 10 mL de FBS, 0,4 g de NaHCO3, 5 mL de pénicilline-streptomycine 100x et 10 mL de D2O).
- Mesurez le pH à l’aide d’un pH-mètre et, si nécessaire, ajustez-le à 7,4 en ajoutant du HCl.
REMARQUE: En règle générale, le pH initial est très proche de 7,4. - Filtrer le milieu du bioréacteur à l’aide d’un filtre stérile sous vide dans une bouteille en verre stérile de 250 mL ou 500 mL.
- Dans la hotte à écoulement laminaire, scellez la bouteille avec un casque en acier stérile avec deux buses de tuyau et connectez-les à un tube FEP (o.d. = 1/8 », i.d. = 1,6 mm) qui sera connecté à la pompe et à un filtre à seringue PTFE de 0,22 μm pour l’admission d’air.
- Dissoudre 13,4 g de poudre de DMEM dans 1 L de H2O ultrapur.
2. Configuration du bioréacteur
- Assemblez l’unité d’écoulement à l’aide d’un deuxième tube RMN d’unité d’écoulement, qui sera remplacé plus tard par celui contenant les cellules. Reportez-vous aux instructions d’utilisation de l’unité de débit pour connaître l’assemblage correct.
REMARQUE: À ce stade, l’unité de débit doit déjà être nettoyée (sinon, effectuez l’étape 4.2). - Réglez le bain-marie connecté au contrôle de la température de l’unité de débit à 37 °C. Placez la bouteille du réservoir dans le bain-marie.
- Connectez le tube FEP de la bouteille du réservoir à la pompe.
- Tournez la vanne du bioréacteur pour « contourner » et préremplissez la pompe avec du milieu.
- Tournez la vanne du bioréacteur en « débit » et préremplissez le bioréacteur avec un milieu à 0,1 mL/min.
3. Préparation de l’échantillon de cellule
- Prélever les cellules de l’incubateur à CO2 .
- Prenez une fiole T75 de cellules HEK293T transfectées de l’incubateur à CO2 et retirez le milieu usé.
- Lavez les cellules deux fois avec 7 mL (chacun) de PBS à température ambiante (~20 °C).
- Utilisez 2 mL de trypsine/EDTA pour détacher les cellules. Après avoir ajouté la solution, incuber pendant 5 min à température ambiante pour détacher les cellules.
REMARQUE: Les cellules transfectées peuvent prendre un peu plus de temps à se détacher. Si nécessaire, incuber les cellules à 37 °C. - Inactiver la trypsine avec 20 mL de DMEM complet; remettre soigneusement en suspension les cellules en les pipetisant de haut en bas et les transférer dans un tube de centrifugeuse de 50 mL.
- Centrifugez les cellules à 800 x g pendant 5 min à température ambiante et jetez le surnageant.
- Lavez les cellules avec 10 mL de PBS à température ambiante pour éliminer le milieu résiduel.
- Centrifugez les cellules à 800 x g pendant 5 min à température ambiante et jetez le surnageant.
- Transférer la pastille de cellule dans un tube de microcentrifugation bouché de 1,5 mL.
- Incorporer des cellules dans des fils d’agarose.
- Faire fondre une aliquote d’agarose solidifiée à 85 °C au bain-marie et ensuite la conserver en solution à 37 °C dans un chauffe-blocs.
- Avec une pipette Pasteur, remplissez le fond du tube RMN de l’unité d’écoulement avec 60-70 μL de gel d’agarose à 1,5% et placez-le dans de la glace. Cela créera un bouchon inférieur d’environ 5 mm de haut qui permettra de placer l’échantillon de cellule dans le volume actif de la bobine RMN 1H.
- Chauffer la pastille de cellules obtenue à l’étape 3.1.8 à 37 °C pendant 15−20 s dans le thermobloc.
- Remettre en suspension des cellules dans 450 μL de solution d’agarose. Attention à éviter la formation de bulles.
- Aspirer la suspension cellule-agarose dans un tube peek chromatographique d’environ 30 cm de long (i.d. = 0,75 mm) relié à une seringue de 1 mL.
REMARQUE: Avant l’aspiration, le tube et le volume mort de la seringue doivent être préremplis avec du PBS à température ambiante pour éviter la formation de bulles. La longueur du tube n’est pas critique. - Laissez le tube refroidir à température ambiante pendant 2 min.
- Préremplissez le tube RMN de l’unité de débit avec 100 μL de PBS à température ambiante.
- Faites couler des fils de cellules incorporés dans l’agarose dans le tube RMN de l’unité d’écoulement en poussant doucement la seringue.
REMARQUE: Pour remplir le tube RMN de manière homogène, commencez par placer l’extrémité du tube PEEK au bas du tube RMN et continuez vers le haut tout en se balançant lentement de gauche à droite. - Répétez les étapes 3.2.5, 3.2.6 et 3.2.8 jusqu’à ce que toute la suspension d’agarose cellulaire ait été coulée.
- Insérez des cellules dans le bioréacteur.
- Retirez le tube RMN vide de l’unité de débit et augmentez le débit à 2 mL/min pendant quelques minutes pour éliminer les bulles de gaz résiduelles dans le tuyau d’entrée.
- Réglez le débit à 0,2 mL/min et insérez le tube RMN contenant les cellules en le poussant vers le haut lentement mais régulièrement.
REMARQUE: L’écoulement actif du fluide évite le refoulement du contenu du tube à travers l’entrée, qui se produirait autrement lors de l’insertion.
4. Fonctionnement et nettoyage du bioréacteur
- Fonctionnement du bioréacteur pendant l’expérience RMN.
- Réglez la température dans le spectromètre RMN à 310 K.
- Insérez l’unité de débit dans le spectromètre.
- Fournir le milieu du bioréacteur à un débit de 0,1 mL/min pendant toute la durée des expériences de RMN dans les cellules.
- Au moment souhaité pendant l’expérience, injecter une solution concentrée de molécule externe dans le flacon à réservoir moyen en perçant le tube de silicone avec une seringue stérile à aiguille longue.
REMARQUE: La concentration finale de la molécule dans le milieu doit être choisie en fonction des connaissances antérieures sur la toxicité cellulaire et, si disponible, sur le taux de diffusion prévu / estimé à travers la membrane cellulaire. - À la fin de l’expérience rmnographique, remplacez le tube contenant les cellules par un tube vide et rincez l’unité d’écoulement avec de l’eau.
- Bioréacteur propre en place.
- Nettoyer l’unité d’écoulement en faisant couler les solutions suivantes à 1 mL/min : 0,2 M d’hydroxyde de sodium (NaOH) ; 3 M d’acide citrique; 0,2 M NaOH, pendant au moins 30 min chacun, suivi d’eau ultrapure filtrée stérile pendant >2 h.
- Nettoyez et autoclavez la bouteille du réservoir et l’ensemble des tubes après chaque passage.
5. Expériences de RMN
- Mise en place des expériences RMN.
REMARQUE: Effectuez ces étapes au préalable, avant la préparation de l’échantillon de RMN dans la cellule, afin d’éviter tout retard entre la collecte des cellules et l’acquisition des données.- Créez un nouvel ensemble de données au spectromètre RMN et définissez les paramètres des expériences RMN souhaitées.
- Définissez les paramètres des expériences de RMN 1D 1H.
- Centrez la fréquence porteuse 1H à 4,7 ppm sur le signal d’eau.
- Sélectionnez le programme d’impulsions zgesgp, réglez la largeur spectrale sur 20 ppm et une impulsion carrée de 1 000 μs à 180 ° pour la suppression de l’eau. Définissez un délai d’analyse intermédiaire de 1 s. Acquérez le spectre avec 32 scans.
- Pour les cellules exprimant de l’AC II non marquée : sélectionnez le programme d’impulsions p3919gp, réglez la largeur spectrale à 30 ppm pour couvrir la région imino du spectre et ajustez le délai de suppression de l’eau binomiale de sorte que l’excitation maximale soit centrée sur les décalages chimiques des signaux d’intérêt (d7 = 20 μs à 950 MHz). Définissez un délai d’analyse intermédiaire de ≥1 s. Acquérir avec 512 scans.
- Pour les cellules exprimant la SOD1 marquée 15N : sélectionnez le programme d’impulsions sfhmqf3gpph, réglez les largeurs spectrales 1H et 15N sur 16 et 50 ppm, respectivement, le décalage d’impulsion en forme et la bande passante d’excitation à 8,5 et 6 ppm, respectivement, et une impulsion de 350 μs pour le schéma de découplage (garp4 ou autre selon l’instrument). Définissez un délai d’analyse intermédiaire de 0,3 s. Acquérir avec 16 scans et 128 incréments dans la dimension 15N.
- Acquisition de spectres RMN en temps réel.
- Une fois le bioréacteur inséré dans le spectromètre RMN, attendez quelques minutes pour permettre l’échange du milieu.
REMARQUE: Ce processus est facilement surveillé à partir de l’apparition du signal de verrouillage car le PBS est remplacé par un support contenant 2% de D2O. - Ajustez la correspondance et le réglage du canal 1H, calez l’aimant et calculez la longueur d’impulsion dure 1H 90 °.
- Ajustez les niveaux de puissance 1H dans chaque séquence d’impulsions en fonction de l’impulsion dure 1H.
- Enregistrez un premier spectre zgesgp 1H pour vérifier le contenu de l’échantillon et l’homogénéité du champ.
- Copiez les expériences zgesgp et p3919gp/sfhmqcf3gpph au nombre souhaité et mettez-les en file d’attente dans le spouleur d’acquisition.
REMARQUE: Les spectres zgesgp ne sont utilisés que pour contrôler l’état de l’échantillon et l’homogénéité du champ; par conséquent, ils peuvent être ignorés ou enregistrés moins fréquemment. - Pour les cellules exprimant du CA II non marqué : traiter les spectres p3919gp en appliquant la fonction de remplissage zéro et la fonction de fenêtre d’élargissement de ligne exponentielle (LB = 20 Hz).
- Pour les cellules exprimant la SOD1 marquée 15N : traiter les spectres sfhmqcf3gpph en appliquant le remplissage zéro et la fonction de fenêtre en cloche sinusoïdale carrée (SSB = 2) dans les deux dimensions.
REMARQUE: La taille des spectres traités peut être encore réduite en supprimant les régions exemptes de signaux (dans Topspin, cela se fait en définissant les valeurs STSR et STSI souhaitées).
- Une fois le bioréacteur inséré dans le spectromètre RMN, attendez quelques minutes pour permettre l’échange du milieu.
6. Analyse MCR-ALS
- Pour l’analyse des spectres CA II, importez les régions spectrales 1D dans MATLAB R2019b.
- Dans le logiciel, créez une liste d’expériences à exporter dans le menu Liste des jeux de données de processus .
- À l’aide d’une version modifiée du programme au convbin2asc, exportez la région spectrale d’intérêt au format ASCII pour chaque spectre.
Remarque : Cela crée un fichier texte nommé ascii-spec.txt dans chaque sous-répertoire de spectre. - Dans MATLAB, importez les régions spectrales à l’aide du script personnalisé Load_ascii_spectra.
REMARQUE: Ce script nécessite le répertoire du jeu de données comme entrée et produit un spectre de réseau 2D contenant les spectres 1D empilés et un tableau 1D cs contenant les décalages chimiques. - Exécutez le script Load_acqus pour extraire les horodatages des spectres 1D.
REMARQUE: Ce script produit un tableau 1D times_hours contenant l’incrément de temps pour chaque spectre exprimé en heures, avec le spectre initial à l’heure = 0.
- Pour l’analyse des spectres SOD1, importez des spectres 2D dans MATLAB R2020b.
- Dans Topspin, créez une liste d’expériences à exporter dans le menu Liste des jeux de données de processus .
- Dans MATLAB, importez les spectres 2D à l’aide du script personnalisé Load_2D_spectra.
REMARQUE: Ce script nécessite le répertoire du jeu de données comme entrée et produit un tableau 3D Spectra contenant les spectres 2D empilés et un tableau 1D cs contenant les décalages chimiques. - Exécutez le script Load_acqus pour extraire les horodatages des spectres 2D.
- Spécifiez les régions spectrales d’intérêt dans le script personnalisé Cut_2D_spectra et exécutez le script pour couper des sous-tableaux 3D [(1H x 15N) intensités spectrales) x temps]; les remodeler en tableaux 2D (points temporels x intensités spectrales) et les assembler.
REMARQUE: Cela produit un réseau 2D JoinSpec_flat contenant les régions spectrales remodelées et jointes.
- Exécutez MCR-ALS 2.0 en mode GUI.
- Ouvrez l’interface graphique MCR-ALS 2.0 en exécutant le script mcr_main.
- Dans l’onglet Sélection des données , chargez les spectres ou la matrice JoinSpec_flat. Les données peuvent être tracées pour vérification.
- Évaluez le nombre de composants par décomposition de valeur singulière (SVD) ou manuellement.
NOTE: Le nombre de composants doit correspondre au nombre d’espèces distinctes présentes dans l’expérience. Dans ce cas n = 2, correspondant à la protéine libre et liée. - Sélectionnez une méthode pour l’estimation initiale des spectres purs. La détection de variables la plus pure ou l’analyse factorielle évolutive (EFA) peuvent être utilisées.
- Dans la fenêtre Sélection de l’ensemble de données , sélectionnez Continuer.
- Définissez les contraintes pour les concentrations dans la fenêtre Contraintes : mode ligne . Appliquez une contrainte de non-négativité, sélectionnez fnnls (Fast nonnegativity-constrained least-squares) comme « implémentation et 2 espèces ». Appliquez 1 contrainte de fermeture, définissez la contrainte sur 1, la condition de fermeture comme « égale à » et appliquez-la à toutes les espèces.
NOTE: Cela force la somme des concentrations de chaque espèce à être égale à 1, de sorte que les profils obtenus de chaque espèce sont normalisés par rapport à la concentration totale en protéines. - Définissez les contraintes pour les spectres dans la fenêtre Contraintes : mode colonne . Appliquez une contrainte de non-négativité, sélectionnez fnnls comme « implémentation et 2 espèces ».
REMARQUE: Cette contrainte ne doit pas être appliquée si des signaux négatifs sont présents dans les spectres RMN. - Dans la dernière fenêtre, définissez 50 itérations et 0,01 critère de convergence. Spécifiez les noms de sortie pour Concentrations, Spectres et Écart Std. Cliquez sur Continuer pour exécuter le raccord MCR-ALS.
REMARQUE: Une fenêtre graphique affichera le résultat du raccord avec des graphiques des profils de concentration et des spectres des composants purs. - Pour le jeu de données SOD1 : utilisez le script personnalisé Rebuild_2D_spectra pour reconstruire les régions spectrales 2D à partir de la sortie 1D de MCR-ALS et les tracer.
7. Test du bleu trypan
- Récupérez le contenu du tube RMN à l’aide d’un pipet Pasteur et transférez les fils d’agarose dans un tube bouché de 1,5 mL.
- Retirer le milieu résiduel en rinçant les fils d’agarose avec 600 μL de PBS et les centrifuger à 4 000 x g pendant 1 min à température ambiante. Jetez le surnageant.
- Ajouter 250 μL de PBS et 50 μL de solution de bleu de trypan à 0,4 %.
- Incuber pendant 2 min avec pipetage continu.
- Laver deux fois avec 600 μL de PBS en jetant le surnageant.
- Placez quelques fils d’agarose sur une lame de microscope et hachez-les avec des lames de rasoir pour créer de petites tranches de gel. Sélectionnez les tranches les plus fines (épaisseur < 0,4 mm, idéalement ~0,2 mm) pour l’analyse.
- Transférer les tranches de gel dans une chambre de comptage de cellules auto-fabriquée composée de deux lames de verre espacées de trois couches de film de paraffine (~ 0,4 mm d’épaisseur totale) de chaque côté.
REMARQUE: Une glissière de chambre pourrait également être utilisée; cependant, les tranches de gel plus épaisses que la hauteur de la chambre (0,1 mm) seraient pressées, rompant les cellules incorporées. - Acquérir des images de cellules à l’intérieur de l’agarose et compter les globules blancs et bleus.
- Calculer la viabilité cellulaire comme (cellules totales - cellules bleues) / cellules totales.
Résultats
Le protocole ci-dessus permet l’encapsulation des cellules dans des fils d’hydrogel pour maximiser la viabilité cellulaire pendant de longues périodes de temps, nécessaires pour étudier en temps réel les processus intracellulaires. Dans le bioréacteur, les cellules sont maintenues vivantes et métaboliquement actives jusqu’à 72 h, comme le confirme le test du bleu de Trypan (Figure 2a-c). En principe, ce protocole peut être appliqué pour observer une protéine intracellulaire d’intérêt subissant des changements conformationnels ou chimiques. Dans la première application décrite ci-dessus, le bioréacteur est appliqué pour surveiller en temps réel la liaison de deux inhibiteurs, AAZ et MZA, à CA II surexprimé dans le cytosol des cellules HEK293T. Le premier spectre de sculpture d’excitation 1H (zgesgp) enregistré est utilisé pour évaluer l’intensité globale du signal (qui est proportionnelle au nombre de cellules), la présence de signaux provenant de la protéine surexprimée et l’homogénéité du champ (Figure 2d). Dans le cas de l’AC II, la liaison intracellulaire des deux inhibiteurs peut être surveillée par les spectres RMN WATERGATE 3-9-1935 1D 1H (p3919gp), en observant des signaux 1H dans la région comprise entre 11 et 16 ppm. Ces signaux proviennent des histidines coordonnant le zinc et d’autres résidus aromatiques de CA II36 et sont perturbés lors de la liaison au ligand14,15. Les concentrations de ligands peuvent être choisies en fonction de la vitesse de diffusion ou, si disponible, à partir de valeurs de perméabilité préalablement déterminées14. La liaison réussie est confirmée visuellement par l’apparition d’un ensemble supplémentaire de signaux dans la région spectrale d’intérêt, qui remplace progressivement les signaux d’origine (Figure 3). Les courbes de liaison dépendantes du temps sont obtenues par analyse MCR-ALS, qui sépare les deux ensembles de signaux RMN provenant de l’AC II libre et lié (figure 4a) et fournit simultanément les profils de concentration relatifs des deux espèces (figure 4b). Dans la deuxième application, le bioréacteur est appliqué pour surveiller la formation de liaison disulfure intramoléculaire SOD1 liée au zinc favorisée par ebselen, un mimétique de glutathion peroxydase, dans les cellules humaines. Ce processus est surveillé en observant les changements dans les spectres 1H-15N 2D SOFAST-HMQC37 (qui fournit une empreinte digitale de la conformation de l’épine dorsale protéique) causés par des perturbations de la structure protéique induites par la formation de liaisons disulfures. Des signaux supplémentaires provenant de la SOD1 oxydée par le disulfure apparaissent dans le spectre 1H-15N et remplacent progressivement ceux de la SOD1 réduite en disulfure. L’analyse MCR-ALS sur des régions sélectionnées du spectre 2D sépare les signaux provenant des deux espèces (figure 5a) et fournit leurs profils de concentration relatifs (figure 5b). Les courbes de concentration obtenues peuvent être analysées plus avant par ajustement non linéaire afin de fournir des informations sur la cinétique des processus étudiés25.
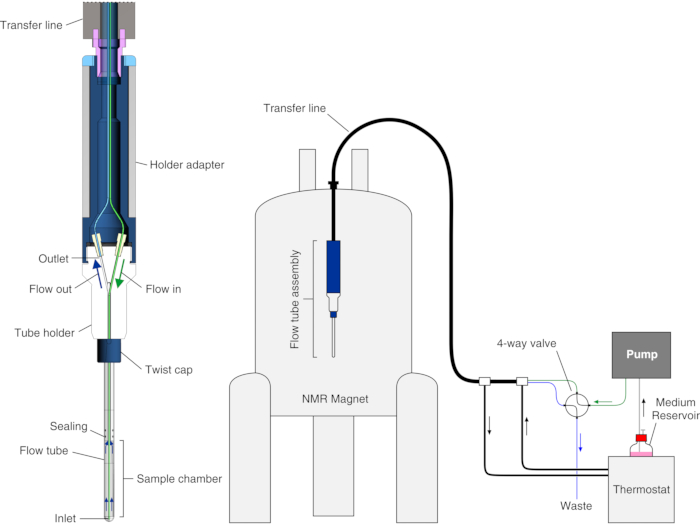
Figure 1 : Schéma du bioréacteur. Gauche : vue en coupe de l’unité d’écoulement vide. Droite : schéma de la configuration du bioréacteur. Le tube d’entrée en PEEK est représenté en vert; le tube de sortie en PTFE est représenté en bleu. Le panneau de gauche est reproduit avec la permission de Luchinat et al.25. Veuillez cliquer ici pour voir une version agrandie de cette figure.
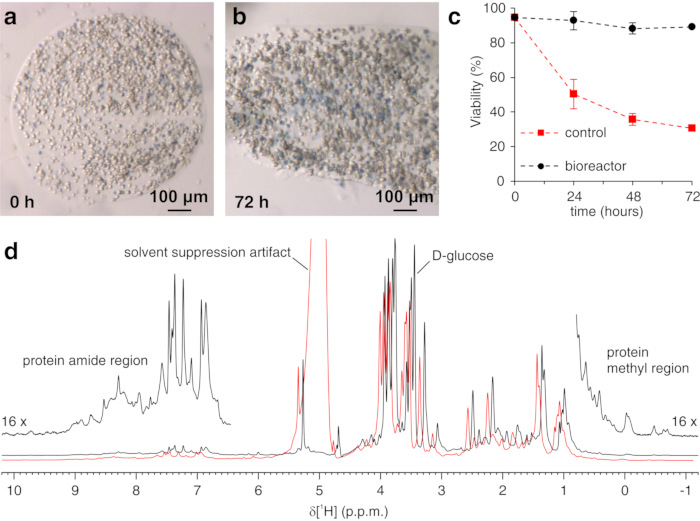
Figure 2 : Test trypan blue sur cellules encapsulées et contrôle de l’échantillon par RMN 1H. Tranches représentatives d’agarose contenant des cellules incorporées et colorées avec du bleu de trypan (a) immédiatement après la coulée et (b) après 72 h dans le bioréacteur; c) viabilité cellulaire en fonction du temps dans le bioréacteur RMN en flux actif (noir) et en conditions statiques (rouge), mesurée par trypan blue test. d) spectres RMN zgesgp 1H enregistrés sur des cellules incorporées dans l’agarose surexprimant CA II en l’absence (noir) et en présence (rouge) de bulles de gaz dans le bioréacteur. Dans ce dernier cas, la diminution de l’homogénéité du champ entraîne l’élargissement de la ligne et l’apparition d’artefacts de suppression des solvants. Des caractéristiques spectrales intéressantes sont étiquetées. Les panneaux (a-c) sont reproduits avec la permission de Luchinat et al.25. Veuillez cliquer ici pour voir une version agrandie de cette figure.
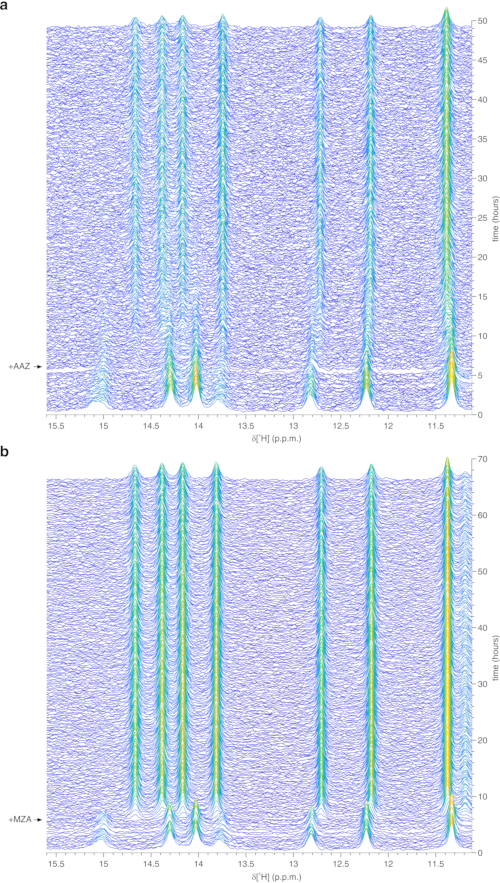
Figure 3 : Données RMN 1H en temps réel dans les cellules obtenues sur des cellules encapsulées dans l’agarose dans le bioréacteur. Diagrammes en cascade de spectres RMN 1D 1H (région comprise entre 15,6 et 11,1 ppm) de cellules HEK293T surexprimant CA II et ensuite traités avec (a) 25 μM AAZ et (b) 10 μM MZA, enregistrés en fonction du temps dans le bioréacteur RMN. L’heure du traitement par ligand est indiquée par une flèche. L’intensité spectrale (u.a.) est codée par couleur du bleu (le plus bas) au jaune (le plus élevé). Cette figure est reproduite avec la permission de Luchinat et al.25. Veuillez cliquer ici pour voir une version agrandie de cette figure.
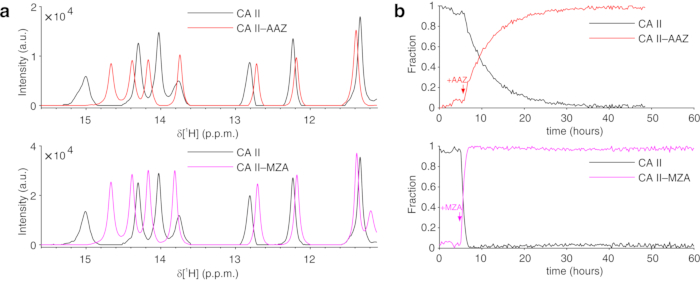
Figure 4 : Sortie MCR-ALS représentative des spectres RMN 1D. (a) Spectres RMN 1H des composants purs reconstruits par MCR-ALS : CA II en l’absence de ligands (noir) et dans le complexe avec AAZ (rouge) ou MZA (magenta) ; b) les profils de concentration relatifs de CA II libre (noir) et lié en fonction du temps lors de l’addition d’AAZ (rouge) ou de MZA (magenta) obtenus par MCR-ALS. Les heures de traitement par ligand sont marquées par des flèches. Cette figure est reproduite avec la permission de Luchinat et al.25. Veuillez cliquer ici pour voir une version agrandie de cette figure.
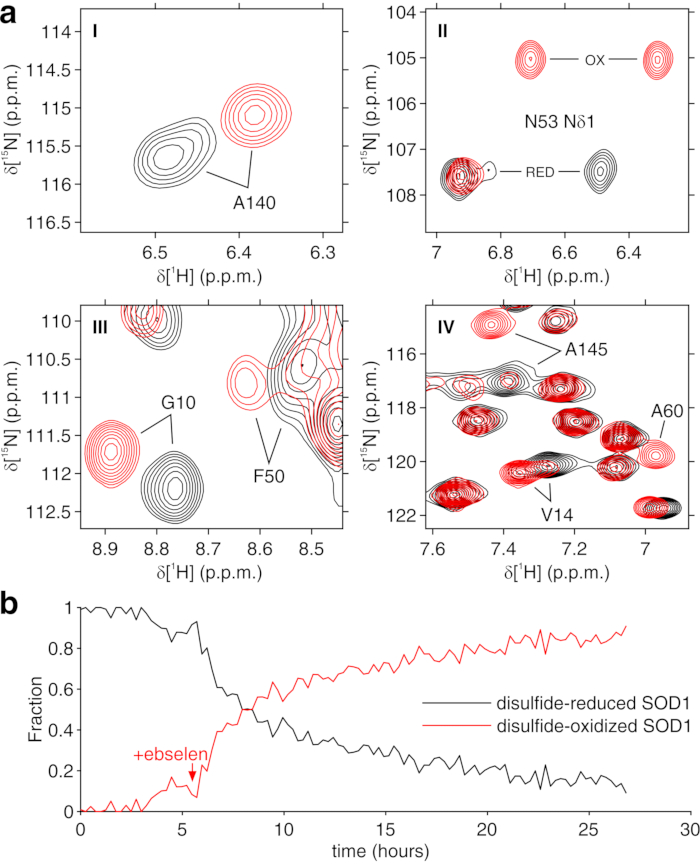
Figure 5 : Sortie MCR-ALS représentative des spectres RMN 2D. (a) Régions spectrales 1H-15N (marquées I-IV) des composants purs reconstruits par MCR-ALS : SOD1 réduite en disulfure (noir) et SOD1 oxydée au disulfure (rouge); b) profil de concentration relatif des composants purs en fonction du temps lors de l’addition d’ebselen (marqué d’une flèche) obtenu par MCR-ALS. Cette figure est reproduite avec la permission de Luchinat et al.25. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
L’objectif de l’utilisation d’un bioréacteur pour l’expérience de RMN dans les cellules est de maintenir les cellules en vie et métaboliquement actives pendant une période prolongée. Un certain nombre d’aspects critiques doivent être pris en considération pour atteindre cet objectif. Tout d’abord, il est primordial d’éviter la contamination bactérienne lors de la préparation de l’échantillon cellulaire et lors de l’acquisition des données RMN. Si des souches d’E. coli ou d’autres bactéries couramment utilisées pour le clonage de gènes et l’expression de protéines recombinantes sont utilisées en laboratoire, elles peuvent contaminer les cellules pendant la préparation de l’échantillon. Une fois dans le bioréacteur, les bactéries se développeront rapidement en exploitant le milieu de croissance frais et provoqueront la mort cellulaire due à la production d’endotoxines. La contamination bactérienne n’est repérée qu’à un stade avancé, lorsqu’elle rend le milieu de croissance jaune et trouble. De plus, un nettoyage incomplet du bioréacteur pourrait entraîner une contamination de la pompe ou du tube par des bactéries, des levures ou des moisissures courantes.
Une condition préalable au succès de l’expérience est d’éviter la formation de bulles de gaz. Les bulles de gaz piégées entre les fils d’agarose dans le volume actif de la bobine RMN introduiraient de grandes inhomogénéités de champ magnétique, provoquant une suppression incomplète du signal H2O et une perte sévère de la qualité spectrale (Figure 2d). Les bulles peuvent être causées par l’air piégé dans le système ou par la formation de CO2 gazeux. Le premier peut être facilement évité en rinçant le système avec un milieu avant d’insérer l’échantillon de cellule, tandis que pour éviter le second, il est recommandé de diminuer la concentration de NaHCO3 dans le milieu de croissance et de maintenir toutes les parties du système à une température constante pour minimiser les différences de solubilité du CO2. Le métabolisme aérobie cellulaire peut également provoquer la formation de CO2 gazeux, qui peut être évité en augmentant le débit.
La viabilité des cellules doit être vérifiée après chaque exécution par Trypan Blue Test. Cependant, cela ne fournit pas d’informations sur l’activité métabolique. Pour obtenir une image plus complète de l’état métabolique des cellules pendant le fonctionnement du bioréacteur, des spectres RMN 31P peuvent être effectués pour évaluer la production d’ATP en fonction du temps23,25. Cependant, une sonde dédiée est souvent nécessaire pour cette mesure, ce qui peut permettre un enregistrement simultané avec RMN 1H.
Dans le cas de l’AC II, la présence de signaux rapporteurs bien résolus dans une région inhabituelle du spectre 1H facilite l’analyse à partir de spectres RMN 1D simples et ne nécessite pas d’enrichissement isotopique lors de l’expression des protéines. En général, d’autres protéines pourraient donner lieu à des signaux 1H utiles pour surveiller les changements spectraux dans d’autres régions, comme celle typique du noyau hydrophobe protéique entre 0 et -1 ppm11; cependant, ces régions ont tendance à être encombrées pour les protéines repliées de plus de ~ 10 kDa. Dans ce cas, comme indiqué pour la SOD1, il est préférable d’enrichir la protéine avec du 15N, en fournissant uniformément un milieu de croissance enrichi en 15N pendant l’expression des protéines, et de surveiller les changements en temps réel dans les spectres RMN 2D 1H-15N. Les spectres 2D sont importés sous forme de tableaux 2D dans MATLAB, réarrangés en tableaux 1D et empilés avant l’analyse MCR-ALS. Cette dernière approche est généralement applicable à toute protéine intracellulaire qui donne lieu à des signaux détectables et fournit des informations sur les changements conformationnels des protéines au niveau de résidu unique. En principe, cette dernière approche peut être généralisée aux spectres nD et à d’autres schémas de marquage isotopique.
En ce qui concerne l’application à différents types de cellules, le protocole doit être facilement adapté à différentes lignées cellulaires et n’exige pas que la protéine d’intérêt soit directement exprimée dans les cellules. Par conséquent, d’autres approches de la RMN cellulaire peuvent être combinées avec ce protocole, dans lequel la macromolécule d’intérêt est produite de manière recombinante, ou synthétisée, puis insérée dans les cellules par électroporation ou par d’autres méthodes d’administration1,9,38. Lorsque vous travaillez avec différentes lignées cellulaires ou différents protocoles de préparation d’échantillons, des paramètres tels que la concentration d’agarose, l’épaisseur du fil et la densité cellulaire finale dans les fils d’agarose peuvent devoir être optimisés empiriquement. De plus, l’applicabilité du protocole décrit ici est limitée aux cellules qui tolèrent l’encapsulation de l’agarose. D’autres types de cellules peuvent nécessiter différentes formulations d’hydrogel, tandis qu’une configuration différente est recommandée lors de l’analyse de cellules qui se développent nativement en suspension, par exemple, en utilisant une membrane de microdialyse coaxiale pour assurer la diffusion des nutriments tout en gardant les cellules en suspension confinées dans le tube RMN23.
Par rapport à d’autres conceptions de bioréacteurs RMN19,20,21,22, le dispositif décrit ici repose sur une unité de débit disponible dans le commerce, adaptée avec des modifications mineures. Par conséquent, l’appareil peut être facilement reproduit dans différents laboratoires avec une reproductibilité élevée. En outre, il permet un fonctionnement standardisé et une conformité totale aux réglementations strictes en matière de sécurité des laboratoires, si nécessaire. Dans l’ensemble, la flexibilité et la facilité d’utilisation du bioréacteur devraient permettre de nombreuses autres applications de la RMN en solution, tant dans les cellules qu’in vitro, en plus de celles déjà signalées23,25. Finalement, la même conception de bioréacteur pourrait être appliquée à des échantillons qui ressemblent davantage à l’environnement physiologique d’un tissu, tels que des sphéroïdes ou des organoïdes, à condition que des échafaudages appropriés soient trouvés pour maintenir ces échantillons en vie - ou même maintenir leur croissance - dans le spectromètre RMN.
Déclarations de divulgation
Les auteurs déclarent qu’il n’y a pas de conflit d’intérêts.
Remerciements
Ce travail a été soutenu par iNEXT-Discovery, subvention numéro 871037, financé par le programme Horizon 2020 de la Commission européenne, par Instruct-ULTRA, numéro de subvention 731005, un projet européen H2020 visant à développer davantage les services d’Instruct-ERIC, et par Ministero dell’Istruzione, dell’Università e della Ricerca PRIN grant 20177XJCHX. Les auteurs reconnaissent le soutien d’Instruct-ERIC, un projet historique de l’ESFRI, à travers le prix JRA numéro 815 et l’utilisation des ressources du Centre CERM/CIRMMP Italie. Nous remercions Matteo Pennestri (Bruker, Royaume-Uni) d’avoir apporté son soutien au fonctionnement de l’unité de débit InsightMR.
matériels
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Citric acid | Sigma-Aldrich | 251275 | |
| D2O | Sigma-Aldrich | 453366 | |
| DMEM, high glucose | Life Technologies | 10313-021 | |
| DMEM, high glucose, powder | Sigma-Aldrich | D5648 | |
| FBS | Life Technologies | 10270 | |
| HCl | Sigma-Aldrich | 30721 | |
| L-glutamine (200 mM) | Life Technologies | 25030 | |
| Low-gelling agarose, powder | Sigma-Aldrich | A4018 | |
| NaHCO3, powder | Carlo Erba | 478537 | |
| PBS | Life Technologies | 10010 | |
| Penicillin–streptomycin (10,000 U/ml) | Life Technologies | 15140-122 | |
| NaOH, pellets | Sigma-Aldrich | 30620 | |
| Trypan Blue solution (0.4% (wt/vol) | Sigma-Aldrich | T8154 | Hazard statement(s): H350 may cause cancer. |
| Trypsin–EDTA (0.05% (wt/vol)) | Life Technologies | 25300-054 | |
| Equipment | |||
| Avance III Spectrometer equipped with a 5 mm CryoProbe | Bruker | n/a | All modern spectrometers and narrow-bore magnets equipped with 5 mm probes are compatible. |
| InsightMR flow unit | Bruker | n/a | |
| P-920 pump module from ÄKTA FPLC | GE Healthcare | n/a | Any FPLC, HPLC peristaltic or syringe pump should be compatible with the flow unit. |
Références
- Inomata, K., et al. High-resolution multi-dimensional NMR spectroscopy of proteins in human cells. Nature. 458 (7234), 106-109 (2009).
- Luchinat, E., Banci, L. In-cell NMR: a topical review. IUCrJ. 4, 108-118 (2017).
- Dzatko, S., et al. Evaluation of the stability of DNA i-motifs in the nuclei of living mammalian cells. Angewandte Chemie (International Ed. in English). 57 (8), 2165-2169 (2018).
- Luchinat, E., Banci, L. In-cell NMR in human cells: direct protein expression allows structural studies of protein folding and maturation. Accounts of Chemical Research. 51 (6), 1550-1557 (2018).
- Tanaka, T., et al. High-resolution protein 3D structure determination in living eukaryotic cells. Angewandte Chemie (International Ed. in English). 58 (22), 7284-7288 (2019).
- Siegal, G., Selenko, P. Cells, drugs and NMR. Journal of Magnetic Resonance. 306, 202-212 (1997).
- Banci, L., et al. Atomic-resolution monitoring of protein maturation in live human cells by NMR. Nature Chemical Biology. 9 (5), 297-299 (2013).
- Luchinat, E., et al. In-cell NMR reveals potential precursor of toxic species from SOD1 fALS mutants. Nature Communications. 5, 5502 (2014).
- Theillet, F. -. X., et al. Structural disorder of monomeric α-synuclein persists in mammalian cells. Nature. 530 (7588), 45-50 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Intracellular metal binding and redox behavior of human DJ-1. Journal of biological inorganic chemistry: JBIC: A Publication of the Society of Biological Inorganic Chemistry. 23 (1), 61-69 (2018).
- Banci, L., Barbieri, L., Luchinat, E., Secci, E. Visualization of redox-controlled protein fold in living cells. Chemistry & Biology. 20 (6), 747-752 (2013).
- Mercatelli, E., Barbieri, L., Luchinat, E., Banci, L. Direct structural evidence of protein redox regulation obtained by in-cell NMR. Biochimica Et Biophysica Acta. 1863 (2), 198-204 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Protein interaction patterns in different cellular environments are revealed by in-cell NMR. Scientific Reports. 5, 14456 (2015).
- Luchinat, E., et al. Drug screening in human cells by NMR spectroscopy allows the early assessment of drug potency. Angewandte Chemie (International Ed. in English). 59 (16), 6535-6539 (2020).
- Luchinat, E., et al. Intracellular binding/unbinding kinetics of approved drugs to carbonic anhydrase II observed by in-cell NMR. ACS Chemical Biology. 15 (10), 2792-2800 (2020).
- DeMott, C. M., et al. Potent inhibitors of mycobacterium tuberculosis growth identified by using in-cell NMR-based screening. ACS Chemical Biology. 13 (3), 733-741 (2018).
- Krafcikova, M., et al. Monitoring DNA-ligand interactions in living human cells using NMR spectroscopy. Journal of the American Chemical Society. 141 (34), 13281-13285 (2019).
- Broft, P., et al. In-cell NMR of functional riboswitch aptamers in eukaryotic cells. Angewandte Chemie (International Ed. in English). , (2020).
- Sharaf, N. G., Barnes, C. O., Charlton, L. M., Young, G. B., Pielak, G. J. A bioreactor for in-cell protein NMR. Journal of magnetic resonance. 202 (2), 140-146 (2010).
- Kubo, S., et al. A gel-encapsulated bioreactor system for NMR studies of protein-protein interactions in living mammalian cells. Angewandte Chemie (International Ed. in English). 52 (4), 1208-1211 (2013).
- Inomata, K., Kamoshida, H., Ikari, M., Ito, Y., Kigawa, T. Impact of cellular health conditions on the protein folding state in mammalian cells. Chemical Communications. 53 (81), 11245-11248 (2017).
- Breindel, L., DeMott, C., Burz, D. S., Shekhtman, A. Real-time in-cell nuclear magnetic resonance: ribosome-targeted antibiotics modulate quinary protein interactions. Biochemistry. 57 (5), 540-546 (2018).
- Cerofolini, L., et al. Real-time insights into biological events: in-cell processes and protein-ligand interactions. Biophysical Journal. 116 (2), 239-247 (2019).
- Breindel, L., Burz, D. S., Shekhtman, A. Active metabolism unmasks functional protein-protein interactions in real time in-cell NMR. Communications Biology. 3, (2020).
- Luchinat, E., Barbieri, L., Campbell, T. F., Banci, L. Real-time quantitative in-cell NMR: ligand binding and protein oxidation monitored in human cells using multivariate curve resolution. Analytical Chemistry. 92 (14), 9997-10006 (2020).
- Koczula, K. M., et al. Metabolic plasticity in CLL: adaptation to the hypoxic niche. Leukemia. 30 (1), 65-73 (2016).
- Alshamleh, I., et al. Real-time NMR spectroscopy for studying metabolism. Angewandte Chemie (International Ed. in English). 59 (6), 2304-2308 (2020).
- Supuran, C. T. Carbonic anhydrases: novel therapeutic applications for inhibitors and activators. Nature Reviews. Drug Discovery. 7 (2), 168-181 (2008).
- Neri, D., Supuran, C. T. Interfering with pH regulation in tumours as a therapeutic strategy. Nature Reviews. Drug Discovery. 10 (10), 767-777 (2011).
- Alterio, V., Di Fiore, A., D'Ambrosio, K., Supuran, C. T., De Simone, G. Multiple binding modes of inhibitors to carbonic anhydrases: how to design specific drugs targeting 15 different isoforms. Chemical Reviews. 112 (8), 4421-4468 (2012).
- Capper, M. J., et al. The cysteine-reactive small molecule ebselen facilitates effective SOD1 maturation. Nature Communications. 9 (1), 1693 (2018).
- Trist, B., Hilton, J. B., Crouch, P. J., Hare, D. J., Double, K. L. Superoxide dismutase 1 in health and disease: How a front-line antioxidant becomes neurotoxic. Angewandte Chemie (International Ed. in English). , (2020).
- Tauler, R. Multivariate curve resolution applied to second order data. Chemometrics and Intelligent Laboratory Systems. 30 (1), 133-146 (1995).
- Barbieri, L., Luchinat, E., Banci, L. Characterization of proteins by in-cell NMR spectroscopy in cultured mammalian cells. Nature Protocols. 11 (6), 1101-1111 (2016).
- Piotto, M., Saudek, V., Sklenár, V. Gradient-tailored excitation for single-quantum NMR spectroscopy of aqueous solutions. Journal of biomolecular NMR. 2 (6), 661-665 (1992).
- Vasa, S. K., Singh, H., Grohe, K., Linser, R. Assessment of a large enzyme-drug complex by proton-detected solid-state NMR spectroscopy without deuteration. Angewandte Chemie (International Ed. in English). 58 (17), 5758-5762 (2019).
- Schanda, P., Brutscher, B. Very fast two-dimensional NMR spectroscopy for real-time investigation of dynamic events in proteins on the time scale of seconds. Journal of the American Chemical Society. 127 (22), 8014-8015 (2005).
- Ogino, S., et al. Observation of NMR signals from proteins introduced into living mammalian cells by reversible membrane permeabilization using a pore-forming toxin, streptolysin O. Journal of the American Chemical Society. 131 (31), 10834-10835 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon