Extraction d'ADN communautaire à partir de colonies bactériennes
Vue d'ensemble
Source : Laboratoires du Dr Ian poivre et Dr Charles Gerba - Université de l’Arizona
Démonstration d’auteur : Luisa Ikner
Les méthodes traditionnelles d’analyse des communautés microbiennes dans les sols comportent habituellement soit culturels dosages utilisant la dilution et méthodologie sur médias sélectifs et différenciés ou dénombrement direct des essais de placage. Les comptes directs offrent des informations sur le nombre total de bactéries présentes, mais ne donnent aucune information sur le nombre ou la diversité des populations présentes au sein de la communauté. Dénombrements permettent d’énumération du total culturel ou certaines populations culturelles et donc fournissent des informations sur les différentes populations présentes. Toutefois, étant donné que moins de 1 % des bactéries du sol sont facilement cultivables, information culturelle offre seulement un morceau de l’image. La fraction réelle de la communauté qui peut être cultivée dépend le moyen choisi pour chefs culturels. N’importe quel support seul choisira pour les populations qui conviennent le mieux à ce support particulier.
Ces dernières années, les avantages d’étudier la communauté ADN extrait d’échantillons de sol sont apparues. Cette approche axée sur la non-culture est considérée comme plus représentatif de la communauté réelle présente qu’approches axées sur la culture. En plus de fournir des informations sur les types de populations présentes, cette approche peut également fournir des informations sur leur potentiel génétique. Comme avec n’importe quelle technique, il y a limites aux données qui peuvent être obtenues par extraction d’ADN. Donc, beaucoup de chercheurs maintenant utilise l’extraction d’ADN en conjonction avec des dénombrements directs et culturels afin d’optimiser les données obtenues auprès d’un échantillon environnemental.
Principles
Extraction de l’ADN du sol peut être effectuée de deux façons (tableau 1). Dans la méthode in situ , une combinaison de techniques de base chimique et mécaniques est utilisée. Pour cette extraction, une masse de sol est combinée avec un volume équivalent d’un tampon d’extraction. Perles de verre sont ensuite ajoutés à la suspension avec un volume de détergent (sodium dodécyl sulfate ou SDS, est généralement utilisé), et l’échantillon est mélangé pour faciliter la séparation des particules de sol suivie d’une incubation à une température élevée pour promouvoir la lyse cellulaire. Après centrifugation, le surnageant est soumis à des mesures supplémentaires d’extraction et d’incubation afin de purifier le produit de l’ADN.
Alternativement, les cellules peuvent d’abord être fractionnés (ou séparés) de la matrice du sol avant l’extraction du matériel génétique. Une masse de l’échantillon de sol subit des cycles successifs de l’assemblage et lent centrifugation. L’étape de perle-battant disparaît ici, cependant, afin de maintenir les cellules intactes, qui sont centrifugés pour obtenir une boulette. Un lysozyme d’extraction est ensuite effectuée en conjonction avec l’incubation de perturber les parois des cellules et de libérer l’ADN pour la purification.
Ce manuscrit et vidéo démontrera la méthode in situ d’extraction de l’ADN du sol, comme l’a démontré cette procédure pour obtenir des concentrations plus élevées de l’ADN d’échantillons de sol par rapport à la méthode de fractionnement cellulaire.
| Question | Fractionnement bactérien | In Situ Lyse |
| Rendement de l’ADN | 1 à 5 μg/g | 1-20 μg/g |
| Représentant de la communauté | Moins représentatifs en raison de la sorption de la cellule | Sorption de cellule plus représentative, pas affectée |
| Source d’ADN récupéré | Que des bactéries | Pour la plupart des bactéries mais aussi les champignons et les protozoaires |
| Degré d’ADN de cisaillement | Moins de cisaillement | Plus de cisaillement |
| Taille moyenne des fragments d’ADN | 50 Ko | 25 Ko |
| Degré de contamination humique | Moins contaminés | Plus contaminés |
| Facilité de méthodologie | Low, laborieux | Plus rapide et moins fastidieux |
Table 1. Comparaison des méthodologies lyse bactériennes à fractionnement et in situ pour la récupération de l’ADN du sol.
Procédure
1. Extraction d’ADN bacterial Community
- Pour commencer la procédure, peser 100 g de sol tamisé. Ajouter ceci à un navire en polypropylène et ajouter 100 mL de tampon d’extraction, composée de tampon Tris modifié avec EDTA pour promouvoir la sortie de bactéries provenant de la matrice du sol, puis serrer à la main.
- Ensuite, peser 100 g de perles de verre et les ajouter à la cuve de mélange. Agiter l’échantillon pendant 5 min avec un filet battant périphérique ou agitateur oscillant mécanique pour 15 min. Ajouter 10 mL 20 % sodium dodécyl sulfate ou SDS, pour le mélange, puis agiter pendant une minute supplémentaire. Incuber à une température élevée de 60-65 ° C pendant 60 min.
- Tout aussi distribuer l’échantillon entre les tubes de 50 mL distincts et centrifugation pendant 10 min à 6 000 x g. transférer le surnageant des tubes dans un seul récipient stérile. Ensuite, répéter l’extraction sur le culot de sol comme décrit précédemment, en utilisant un volume frais de tampon d’extraction.
- Ensuite, ajoutez le volume total de liquide surnageant transformé, environ 200 mL, un tube propre 50 mL rempli à la moitié du volume d’une solution de chlorure de sodium de polyéthylène glycol et 1,6 M de 30 %. Inverser les bouteilles plusieurs fois à la main pour bien mélanger et laisser incuber à température ambiante pendant 2 h. échantillons de centrifugation à 10 000 x g pendant 20 min à l’ADN de granule.
- Retirez soigneusement le surnageant du tube à centrifuger, laissant derrière lui le culot d’acide nucléique partiellement purifiée. Ajouter 20 mL de tampon TE et 1,5 mL d’une solution d’acétate de potassium 7,5 M à Resuspendre le culot, puis vortexer. Placez la suspension sur la glace pendant 5 min. Centrifuger à 16 000 x g pendant 30 min à 4 ° C, précipitation des protéines et des polysaccharides.
- Ensuite, ajouter une RNAse et la protéinase K dans l’échantillon, mélanger doucement à la main et laisser reposer pour le moment. Ajouter un volume équivalent de phénol : chloroforme : isoamylique alcool (mélange de ratio de 25:24:1) à la suspension à extraire et mélanger délicatement à la main. Centrifuger la préparation pendant 10 min à 13 000 x g. Retirer soigneusement le récipient de la centrifugeuse et note les deux couches.
- Au fond, une couche plus lourd est composé de l’alcool de phénol : chloroforme : isoamylique et extraites des débris et la couche supérieure est l’humeur aqueuse et contient de l’ADN. Placez la phase aqueuse dans un récipient stérile, ajouter un volume équivalent d’isopropanol et inverser doucement pour ouvrir la précipitation de l’ADN. Incuber la suspension à température ambiante pendant 2 h. Pellet l’ADN purifié par centrifugation à 16 000 x g pendant 30 min. Retirer délicatement le surnageant l’ADN granulé peut ou pas être visible au fond du bateau, et puis remettre en suspension dans 1 mL de tampon TE.
- À l’aide d’un spectrophotomètre ou un fluorimètre de quantification de l’ADN/ARN, mesurer le niveau de l’ADN extrait de l’échantillon. La quantité d’ADN est estimée à partir de la lecture nm 260. Une lecture d’absorbance de 1.0 équivaut à 50 µg d’ADN par mL de solution. Si la concentration est trop élevée pour une lecture précise, diluer la suspension de 1 à 10, ou 1 à 100 à l’aide de l’eau de qualité moléculaire.
- La pureté de l’ADN est estimée par le rapport entre la lecture à 260 nm à celle à 280 nm. Une valeur > 1.7 indique ADN relativement pur. La valeur maximale théorique est 2.0.
Applications et Résumé
ADN de la communauté de colonies de culture ou extraites de sol peut être soumis à la bio-informatique et « omic » des approches qui permettent pour la caractérisation des bactéries au sein de l’échantillon originales. Les approches omic incluent métagénomique – détermination des « qui » est au sein de la Communauté par l’intermédiaire de séquençage des ARNr 16 s. Cela donne une estimation de la diversité au sein de la communauté.
Le nombre de cellules bactériennes dans l’échantillon de sol original peut également être calculé. Communauté ADN est extraite d’un sol et quantifiée par des analyses spectroscopiques. La quantité estimée d’ADN mesurée en µg ADN par mL de solution est liée au volume total de l’ADN extrait en solution pour donner un montant total de l’ADN par g de sol. En connaissant la valeur théorique de l’ADN par cellule, le nombre total de cellules / g de sol peut être calculé.
Exemple de
Un sol a 0,12 µg ADN par g de sol
Si chaque cellule possède 4 fg de l’ADN
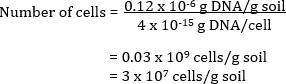
L’ADN extrait de communauté peut être soumis à l’analyse PCR utilisant des amorces spécifiques pour déterminer si une espèce est présente au sein de la communauté. Les exemples incluent spécifiques bactéries pathogènes comme le Clostridium perfringens ou Bacillus anthracis.
Passer à...
Vidéos de cette collection:

Now Playing
Extraction d'ADN communautaire à partir de colonies bactériennes
Environmental Microbiology
28.8K Vues

Détermination de la teneur en humidité du sol
Environmental Microbiology
359.5K Vues

Technique aseptique en sciences de l'environnement
Environmental Microbiology
126.5K Vues

Coloration de Gram des bactéries provenant de sources environnementales
Environmental Microbiology
100.3K Vues

Visualisation des micro-organismes du sol grâce aux lames de contact et à la microscopie
Environmental Microbiology
42.3K Vues

Champignons filamenteux
Environmental Microbiology
57.3K Vues

Détection des micro-organismes environnementaux avec la réaction en chaîne par polymérase et l'électrophorèse sur gel
Environmental Microbiology
44.6K Vues

Analyse d'ARN d'échantillons environnementaux à l'aide de la RT-PCR
Environmental Microbiology
40.4K Vues

Quantification des micro-organismes et des virus environnementaux à l'aide de la qPCR
Environmental Microbiology
47.8K Vues

Analyse de la qualité de l'eau via des organismes indicateurs
Environmental Microbiology
29.5K Vues

Isolement des bactéries fécales à partir des échantillons d'eau par filtration
Environmental Microbiology
39.3K Vues

Détection de bactériophages dans des échantillons environnementaux
Environmental Microbiology
40.7K Vues

Culture et dénombrement des bactéries dans un échantillon de sol
Environmental Microbiology
184.5K Vues

Analyse de la courbe de croissance bactérienne et ses applications environnementales
Environmental Microbiology
296.0K Vues

Dénombrement des algues via une méthodologie de culture
Environmental Microbiology
13.8K Vues