Extração de DNA de comunidades de colônias bacterianas
Visão Geral
Fonte: Laboratórios do Dr. Ian Pepper e Dr. Charles Gerba - Universidade do Arizona
Autora de Demonstração: Luisa Ikner
Os métodos tradicionais de análise para comunidades microbianas dentro dos solos geralmente envolveram ensaios culturais utilizando a metodologia de diluição e plating em mídias seletivas e diferenciais ou ensaios de contagem direta. As contagens diretas oferecem informações sobre o número total de bactérias presentes, mas não dão informações sobre o número ou diversidade de populações presentes na comunidade. As contagens de placas permitem a enumeração de populações culturais ou selecionadas totais e, portanto, fornecem informações sobre as diferentes populações presentes. No entanto, como menos de 1% das bactérias do solo são prontamente culturais, a informação cultural oferece apenas um pedaço da imagem. A fração real da comunidade que pode ser cultivada depende do meio escolhido para contagem cultural. Qualquer meio único selecionará para as populações mais adequadas a esse meio em particular.
Nos últimos anos, as vantagens de estudar DNA comunitário extraído de amostras de solo tornaram-se aparentes. Acredita-se que essa abordagem não-cultural seja mais representativa da comunidade atual presente do que as abordagens baseadas na cultura. Além de fornecer informações sobre os tipos de populações presentes, essa abordagem também pode fornecer informações sobre seu potencial genético. Como em qualquer técnica, há limitações aos dados que podem ser obtidos com extração de DNA. Portanto, muitos pesquisadores agora usam a extração de DNA em conjunto com contagens diretas e culturais para maximizar os dados obtidos a partir de uma amostra ambiental.
Princípios
A extração de DNA do solo pode ser conduzida de duas formas (Tabela 1). No método in situ, é utilizada uma combinação de técnicas químicas e mecânicas. Para esta extração, uma massa de solo é combinada com um volume equivalente de um tampão de extração. As contas de vidro são então adicionadas à suspensão, juntamente com um volume de detergente (sulfato de dodecilo de sódio, ou SDS, é tipicamente usado), e a amostra é misturada para facilitar a separação das partículas do solo seguidas de incubação a uma temperatura elevada para promover a lise celular. Após a centrifugação, o supernascido é submetido a novas etapas de extração e incubação, a fim de purificar o produto de DNA.
Alternativamente, as células podem primeiro ser fracionadas (ou separadas) da matriz do solo antes da extração do material genético. Uma massa de amostras de solo sofre sucessivos ciclos de mistura e centrifugação lenta. O passo de bater contas é eliminado aqui, no entanto, a fim de manter células intactas, que são centrifadas para obter uma pelota. Uma extração à base de lysozyme é então realizada em conjunto com a incubação para interromper as paredes celulares e liberar o DNA para purificação.
Este manuscrito e vídeo demonstrarão o método in situ de extração de DNA do solo, pois este procedimento tem sido demonstrado para produzir maiores concentrações de DNA a partir de amostras de solo em relação ao método de fracionamento celular.
| Questão | Fracionamento bacteriano | Em Situ Lise |
| Rendimento do DNA | 1-5 μg/g | 1-20 μg/g |
| Representante da comunidade | Menos representativo por causa da sorção celular | Mais representativa, sorção celular não afetada |
| Fonte de DNA recuperado | Apenas bactérias | Principalmente bactérias, mas também fungos e protozoários |
| Grau de tesoura de DNA | Menos tesoura | Mais tesouras |
| Tamanho médio de fragmentos de DNA | 50 kb | 25 kb |
| Grau de contaminação húnica | Menos contaminado | Mais contaminado |
| Facilidade de metodologia | Baixo, trabalhoso | Mais rápido, menos mão-de-obra |
Mesa 1. Comparação de fracionamento bacteriano e metodologias in situ lysis para a recuperação do DNA do solo.
Procedimento
1. Extração de DNA da comunidade bacteriana
- Para iniciar o procedimento, pese 100 g de solo peneirado. Adicione isso a um vaso de polipropileno, e adicione 100 mL de tampão de extração composto de tampão Tris alterado com EDTA para promover a liberação de bactérias da matriz do solo, em seguida, aperte à mão.
- Em seguida, pese 100 g de contas de vidro, e adicione-as ao recipiente de mistura. Agitar a amostra por 5 min usando um dispositivo de batida de contas ou um agitador mecânico de ação de pulso por 15 minutos. Adicione 10 mL 20% de sulfato de dodecil de sódio, ou SDS, à mistura e, em seguida, agitar por mais um minuto. Incubar a uma alta temperatura de 60 - 65 °C por 60 min.
- Distribua igualmente a amostra entre tubos separados de 50 mL e centrífuga por 10 minutos a 6.000 x g. Transfira o supernatante dos tubos para um único recipiente estéril. Em seguida, repita a extração na pelota do solo como descrito anteriormente, utilizando um novo volume de tampão de extração.
- Em seguida, adicione o volume total de supernanato processado, aproximadamente 200 mL, a um tubo limpo de 50 mL preenchido a meio volume com uma solução de 30% de polietileno glicol e cloreto de sódio de 1,6 M. Inverta as garrafas várias vezes à mão para misturar e incubar à temperatura ambiente por 2h. Centrífuga amostras a 10.000 x g por 20 minutos para pelotar o DNA.
- Remova o supernatante cuidadosamente do tubo centrífuga, deixando para trás a pelota de ácido nucleico parcialmente purificada. Adicione 20 mL de TE Buffer e 1,5 mL de uma solução de acetato de potássio de 7,5 M para resuspensar a pelota, depois vórtice. Coloque a suspensão no gelo por 5 minutos. Centrifugar a 16.000 x g por 30 min a 4 °C para precipitar proteínas e polissacarídeos.
- Em seguida, adicione um RNAse e proteinase K à amostra, misture suavemente à mão e deixe descansar por momento. Adicione um volume equivalente de fenol:clorofórmio:álcool isoamyl (mistura de razão de 25:24:1) à suspensão a ser extraída, e misture suavemente à mão. Centrifugar a preparação para 10 min a 13.000 x g. Remova cuidadosamente o vaso da centrífuga e observe as duas camadas.
- A camada inferior e mais pesada é composta do fenol:clorofórmio: álcool isoamyl e detritos extraídos, e a camada superior é a aquosa e contém o DNA. Coloque a fase aquosa em um vaso estéril, adicione um volume equivalente de isopropanol e inverta suavemente para iniciar a precipitação do DNA. Incubar a suspensão em temperatura ambiente por 2h. Pellet o DNA purificado por centrifugação a 16.000 x g por 30 min. Remova cuidadosamente o supernasciente, pois o DNA pelleted pode ou não ser visível na parte inferior do vaso e, em seguida, resuspend em 1 mL de TE Buffer.
- Usando um fluorímetro de quantificação de DNA/RNA, meça o nível de DNA extraído da amostra. A quantidade de DNA é estimada a partir da leitura de 260 nm. Uma leitura de absorvância de 1,0 equivale a 50 μg de DNA por mL de solução. Se a concentração for muito alta para leituras precisas, dilui a suspensão de 1 a 10, ou 1 a 100 usando água de grau molecular.
- A pureza do DNA é estimada da razão da leitura em 260 nm para a de 280 nm. Um valor > 1,7 indica DNA relativamente puro. O valor teórico máximo é 2.0.
Aplicação e Resumo
O DNA comunitário de colônias cultivadas ou extraídos do solo pode ser submetido a abordagens bioinformáticas e "omic" que permitem a caracterização das bactérias originais dentro da amostra. As abordagens omicais incluem metagenômica – determinação de "quem" está dentro da comunidade através de sequenciamento de rRNA 16S. Isso dá uma estimativa da diversidade dentro da comunidade.
O número de células bacterianas na amostra original do solo também pode ser calculado. O DNA comunitário é extraído de um solo e quantificado por análises espectroscópicas. A quantidade estimada de DNA medido como DNA μg por mL de solução está relacionada ao volume total de DNA extraído em solução para dar uma quantidade total de DNA por g de solo. Sabendo o valor teórico do DNA por célula, o número total de células por g de solo pode ser calculado.
Exemplo
Um solo tem 0,12 μg de DNA por g de solo
Se cada célula tem 4 fg de DNA
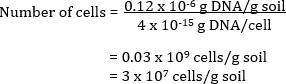
O DNA da comunidade extraído pode ser submetido à análise pcr usando primers específicos para determinar se uma determinada espécie está presente dentro da comunidade. Exemplos incluem patógenos bacterianos específicos, como clostridium perfringens ou Bacillus anthracis.
Pular para...
Vídeos desta coleção:

Now Playing
Extração de DNA de comunidades de colônias bacterianas
Environmental Microbiology
28.8K Visualizações

Determinação do teor de umidade no solo
Environmental Microbiology
359.5K Visualizações

Técnica Asséptica em Ciências Ambientais
Environmental Microbiology
126.4K Visualizações

Coloração de Gram de bactérias de fontes ambientais
Environmental Microbiology
100.3K Visualizações

Visualização de Microrganismos do Solo por Meio do Ensaio de Lâmina de Contato e Microscopia
Environmental Microbiology
42.3K Visualizações

Fungos Filamentosos
Environmental Microbiology
57.3K Visualizações

Detecção de Microrganismos Ambientais com Reação em Cadeia da Polimerase e Eletroforese em Gel
Environmental Microbiology
44.6K Visualizações

Análise de RNA de amostras ambientais usando RT-PCR
Environmental Microbiology
40.4K Visualizações

Quantificando Microorganismos e Vírus Ambientais Usando qPCR
Environmental Microbiology
47.8K Visualizações

Análise da Qualidade da Água por Organismos Indicadores
Environmental Microbiology
29.5K Visualizações

Isolamento de bactérias fecais de amostras de água por filtração
Environmental Microbiology
39.3K Visualizações

Detecção de Bacteriófagos em Amostras Ambientais
Environmental Microbiology
40.7K Visualizações

Cultivo e Enumeração de Bactérias de Amostras de Solo
Environmental Microbiology
184.5K Visualizações

Análise da Curva de Crescimento Bacteriano e suas Aplicações Ambientais
Environmental Microbiology
296.0K Visualizações

Contagem de Algas em Métodos de Cultura
Environmental Microbiology
13.8K Visualizações
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados