Method Article
זיהוי של שותפי אינטראקציה חלבון בתאי יונקים באמצעות SILAC-immunoprecipitation פרוטאומיקה כמותי
In This Article
Summary
ניסויי immunoprecipitation SILAC מייצגים אמצעי רב עוצמה לגילוי חלבון חדש: אינטראקציות חלבון. על ידי המאפשר כימות היחסית מדויקת של שפע חלבון בשליטה והן דגימות בדיקה, אינטראקציות אמיתי ניתן להבחין בקלות ממזהמים ניסיוניים, ואינטראקציות זיקה נמוכות נשמרו באמצעות שימוש בתנאי חיץ פחות מחמירים.
Abstract
פרוטאומיקה כמותית בשילוב עם טיהור חיסונית זיקה, immunoprecipitation SILAC, מייצגת אמצעי רב עוצמה לגילוי חלבון חדש: אינטראקציות חלבון. על ידי המאפשר כימות היחסית מדויקת של שפע חלבון בשליטה והן דגימות בדיקה, אינטראקציות אמיתי ניתן להבחין בקלות ממזהמים ניסיוניים. ניתן לשמר אינטראקציות זיקה נמוכה באמצעות השימוש בתנאי חיץ פחות מחמירים ויישארו לזיהוי בקלות. פרוטוקול זה דן בתיוג של תאים בתרבית רקמה עם חומצות אמינו שכותרתו איזוטופ היציב, transfection וimmunoprecipitation של זיקה מתויג חלבון של עניין, ואחריה ההכנה להגשה למתקן ספקטרומטריית מסה. פרוטוקול זה אז מסביר כיצד לנתח ולפרש את הנתונים שהוחזרו מספקטרומטר המסה על מנת לזהות שותפים סלולריים אינטראקציה עם חלבון של עניין. כדוגמא טכניקה זו תחול על iDENתצדיק את החלבונים מחייבים את גורמי חניכה תרגום אוקריוטים: eIF4AI וeIF4AII.
Introduction
צעד חיוני בהבנת תפקוד חלבון הוא זיהוי של חלבוני אינטראקציה רלוונטיים. איפה חלבונים כאלה אינם ידועים יש מספר טכניקות זמינות, כל אחד עם יתרונות משלהם וחסרונות. אלה כוללים את מערכת השמרים שני ההיברידית, מבחני הנפתח באמצעות חלבון רקומביננטי, כמו גם טיהור זיקת טנדם או TAP תיוג 1, 2.
בנוסף אחרונים יותר לטכניקות אלה הוא שילוב של טיהור זיקה של חלבון של עניין משורת תאים של יונקים רלוונטיים, ואחריו על ידי ספקטרומטריית מסה כמותית באמצעות תיוג איזוטופ יציב של חומצות אמינו בתרבית תאים 3 (SILAC). זו יש יתרונות על פני הגישה של שני ההיברידית שמרים שבלוקליזציה התא ולאחר translational שינויים אינם מוטרדים, כמו גם יתרונות על פני מסורתי בכך שהיא היא גישת כמותית ולא איכותית המאפשרת למשתמש ריאה תיוג TAP dily להבחין שאינו במיוחד באינטראקציה חלבונים וחומרים מזהמים, מגורמי מארח שלהיקשר באופן ספציפי. יתר על כן, כמדגם מנותח בדרך כלל כולו, ולא להקות חלבון בודדות, כמו, חלבונים של עניין אינם רעול פנים על ידי דומה נודדים חלבונים על ג'ל, ואין הם בדרך כלל צריכים להיות נוכחים ברמות מספיקות כדי להיות גלוי לאחר צביעה, מה שמוביל ל מספרים מוגברים של חלבונים שזוהו בביטחון 4.
כדי להדגים את הטכניקה הזו, התכה GFP של גורם החניכה תרגום אוקריוטים הקשור קשר הדוק eIF4AI וeIF4AII מניה שעל זהות חומצת אמין 90% נחקרו על ידי פרוטאומיקה כמותיים SILAC-immunoprecipitation. eIF4AI האדם והשני היו משובטים לתוך pEGFP-C1 ליצירת חלבון היתוך שבו GFP הוא קבע את N-הסופית של eIF4A. כדי למנוע את הצורך ביצירת שורות תאי יציבות transfection חולף שימש כדי לספק המבנים הללו לתאי איזוטופ יציבים שכותרת 293T.
"Jove_content"> תאים תויגו ראשון במשך שבועיים בתקשורת תרבות תא SILAC, ואחרי transfection של DNA פלסמיד קידוד חלבון של עניין. תאים אז היו lysed, ריכוז החלבון מנורמל, וכמויות שווים של זיקת lysate מטוהרים על agarose אנטי-GFP. כמויות שווה של eluate אוחדו לאחר מכן והגישו לניתוח LC-MS/MS. התוצאות של ניתוח זה מעובד מכן לזהות חלבון אמון גבוה: אינטראקציות חלבון (איור 1).
immunoprecipitation SILAC מאפשרת זיהוי לא רק אינטראקציות ישירות אלא גם זיקה נמוכה או אינטראקציות עקיפות עם קומפלקסי חלבונים 4. באמצעות מערכת זו, immunoprecipitations eIF4AI והשני אפשר זיהוי לשעתק ובטוח בעצמו של השותף העיקרי מחייב eIF4G (isoforms I / II ו-III) 5, כמו גם יחסי גומלין עקיפים עם eIF4E, ומספר רבים של רכיבים המורכבים eIF3.
Protocol
1. דור וPassaging של שורות תאים שכותרתו SILAC
שים לב: השימוש בטריפסין-EDTA יש להימנע בכל שלבי passaging והכנת דגימות ניסיוניות לניתוח כטריפסין עשוי להכיל חומצות אמינו ללא תווית, מה שיובילו לתיוג שלם של דוגמאות.
- להכין בקבוק של תקשורת SILAC להוסיף aliquot 0.5 מיליליטר של ארגינין כראוי שכותרתו SILAC (84 מ"ג / מיליליטר PBS) וליזין (146 מ"ג / מיליליטר PBS) לבקבוק 500 מיליליטר של DMEM בחינם Arg / יס (המכיל L-גלוטמין).
- לאחר מכן, להוסיף 50 מיליליטר של FBS dialyzed ו5 מיליליטר של פניצילין / סטרפטומיצין לבקבוק 500 מיליליטר.
הערה: תאי 293T HEK (ATCC) יש לשמור בתקשורת DMEM חסר ארגינין וליזין והשלים עם אור (R0K0), בינוני (R6K4) או כבד (R10K8) חומצות אמינו, סרום dialyzed שור העובר (10 הפסקת KDA), ופניצילין / סטרפטומיצין. תאים חייבים להישמר בתקשורת למינימום של 5 חלוקות תא כדי להבטיח מלאתיוג. ברוב המקרים תאים מסומנים בקלות ב≥ 2 שבועות. - כדי לפצל תאים לpassaging, צריכים להיעקר מmonolayer תאים על ידי להכות את הבקבוק. חלופות כוללות את השימוש של מגרדי תא או על ידי שימוש ב, חיץ תא דיסוציאציה מבוסס PBS ללא אנזים.
שימו לב: כדי לחסוך בתקשורת, יש passaged תאים בצלוחיות T25 ומספרי תא גדולים יותר שנוצרו רק מייד לפני ניסוי. - 24 שעות לפני תאי transfecting, זרע 3.5 x 10 6 תאים שכותרתו SILAC ל10 סנטימטר 2 מנה אחת לכל תנאי ניסוי תחת חקירה.
2. Transfection של תאים שכותרתו עם מבני pEGFP-Fusion
- הסר את המדיה מהתאים ולהחליף אותו עם 9 מיליליטר של DMEM SILAC אנטיביוטיקה ללא מדיה (בהיר, בינוני או כבד).
שים לב: לא צריכה להוסיף אנטיביוטיקה לתקשורת מכיוון שהם עלולים להפריע ליעילות של חומרים כימיים transfection מבוסס ליפוזום. - תכשיריםמחדש שילוב של 10 מיקרוגרם של פלסמיד המתאים (epEGFP-C1, pEGFP-eIF4A-I, pEGFP-eIF4A-II) ב500 μl של SILAC DMEM ללא אנטיביוטיקה ולערבב אותו עם 500 μl של DMEM SILAC ללא אנטיביוטיקה המכיל 10 μl של מגיב transfection (למשל, lipofectamine 2000). מערבבים את התגובה ביסודיות על ידי pipetting אותו מעלה ומטה מספר פעמים.
- דגירה את תערובת התגובה בטמפרטורת חדר למשך 20 דקות ולאחר מכן להוסיף טיפה חכמה לmonolayer התא. רוק הצלחת בעדינות מצד לצד.
- דגירה תאי transfected על 37 מעלות צלזיוס ו10% CO 2 ל24 שעות.
הערה: אם הביטוי של חלבון של תוצאות עניין בכל רעילות נראית לעין, אז זה עשוי להיות נחוץ כדי להפחית את כמות הפלסמיד transfected ו / או משך התקופה שתבוא מאוחר.
3. Lysates תא הקציר
- קציר תאים מהצלחת לקרח הקר PBS באמצעות מגרד תא. איסוף תאים על ידי צנטריפוגה ב220 XG, 476; צלזיוס למשך 5 דקות. לשטוף את התאים 3x נוסף ב10 מיליליטר של PBS קר כקרח.
- Resuspend התא גלולה ב μl 200 של חיץ תמוגה תא (10 מ"מ TrisCl / pH 7.5, 150 mM NaCl, 0.5 mM EDTA, NP40 0.5%) המכיל מעכבי פרוטאז קוקטייל הוסיף טרי III בריכוז 1x וקוקטייל RNase (אופציונלי) בשעה 5 μl לכל מיליליטר.
הערה: לקבלת חלבונים עם חומצות גרעין ידועות מחייבות פעילות, ייתכן שיהיה צורך להוסיף nucleases לlysate לפני משקעים. במקרים בהם nuclease הוא הוסיף, צריכה להיות מודגרות דגימות על קרח ל30 דקות על קרח, עם pipetting כל 10 דקות. חילוץ חומצות גרעין הכוללת מחלק קטן של המדגם והניתוח על ידי ג'ל אלקטרופורזה agarose יכול לבדוק את האפקטיביות של nuclease. - צנטריפוגה דגימות 13,000 XG, 4 ° C עבור 10 דקות לשמר את supernatant כlysate התא מסיס.
- הריכוז של lysate התא צריך להיות מוערך על ידי assay BCA על פי הוראות היצרן.
- השתמש במאגר תמוגה המכיל מעכבי פרוטאז קוקטייל שלישי לנרמל את ריכוז חלבון בנפח סופי של μl 500.
- כוון את עוצמת הקול למ"ל 1 עם התוספת של 500 μl של חיץ דילול (10 מ"מ TrisCl / pH 7.5, 150 mM NaCl, 0.5 mM EDTA) המכיל מעכבי פרוטאז קוקטייל שלישי בריכוז 1x סופי. Aliquot 50 μl של המדגם צריך להישמר כקלט לדוגמא וlysate כל הזמן על קרח תוך הכנת חרוזים אנטי-GFP (למשל, ה-GFP-מלכודת).
הערה: בדרך כלל תשואות משתנות בין 1-3.5 מ"ג של חלבון במדגם 1 מיליליטר הסופי. בעוד המאגרים הנ"ל מתאימים לחלבונים רבים של עניין, לאחרים זה עשוי להיות נחוץ כדי לשנות את רכיבי חיץ כדי להבטיח את חלבון הפיתיון solubilized ולשמור על אינטראקציות בין חלבונים. שינויים אפשריים כוללים סוכן החציצה (פוספט, HEPES), ריכוז מלח (150-500 מ"מ), בחירה של חומרי ניקוי, או תוספים אחרים.
4. כבילה למעסיק Anti-Gחרוזים FP
- בקצרה מערבולת slurry חרוז לresuspend את החרוזים. באמצעות פיפטה קצה 200 μl עם הסוף מנותק, להעביר 25 μl של חרוזים לדגימה לצינור טרי.
שים לב: המשתמש צריך להכין את חרוזים לניסוי SILAC בודד כmastermix כדי למזער וריאציה מדגם למדגם. - עבור כל 25 μl של slurry חרוז, להוסיף 20 כרכים (1,500 μl ל75 slurry μl) של חיץ דילול, ו צנטריפוגות חרוזים ב2,700 XG במשך 5 דקות. בשלב הבא, לשטוף את החרוזים 2x נוסף ב20 כרכים של חיץ דילול.
- הוספת 100 חיץ דילול μl ל25 slurry חרוז μl. באמצעות קצה 200 μl עם הסוף מנותק, להעביר 85 μl של slurry resuspended זה לכל אחת מהדגימות שכותרת SILAC מהצעד 3.6.
- דגירה הדגימות עם חרוזים על הכתף על 4 מעלות צלזיוס במשך שעה 2.
5. כביסה, Elution, והכנת דגימות לטרשת נפוצה ניתוח
- צנטריפוגה דגימות 2,700 XG, 46; צלזיוס למשך 5 דקות. 50 μl של supernatant צריך להישמר כמדגם לא מאוגד, עם השארית של supernatant המושלך.
- חיץ דילול 1ml יש להוסיף לכל מבחנה כדי resuspend את החרוזים ואת המדגם centrifuged ב2,700 XG, 4 מעלות צלזיוס למשך 5 דקות. Supernatant צריך להיות מושלך. זה צריך להתבצע פעמיים.
- Elute חלבון מן החרוזים על ידי התוספת של 50 μl של חיץ טעינת 2x SDS, וחימום ב95 מעלות צלזיוס למשך 10 דקות. גלולה חרוזים על ידי צנטריפוגה ב2,700 XG במשך 2 דקות על 4 מעלות צלזיוס
- שמור את supernatant בצינורות prelubricated, שבו ניתן לאחר מכן מאוחסנים ב-80 ° C עד צורך. להגשה למתקן ספקטרומטריית מסה, לערבב דוגמאות שכותרת 1:01:01 (למשל, 10 μl של כל אחד) ולהגיש את המדגם מעורב.
הערה: בשלב זה דגימות עשויות להיבדק על ידי כתם מערבי, כסף מכתים או באמצעים אחרים לבדיקת אינטראקציה עם שותפים ידועים / לא ידועים המחייבים. דוגמה לכך ניתנת באיוריור 2 מראה מחייב ספציפי של שותף ידוע אינטראקציה - eIF4G על ידי כתם מערבי, ואת המראה של להקות צביעת כסף כיום בדגימות הנפתח מחלבון של עניין, אבל לא מדובר במדגם בקרה. - שלח דגימה לניתוח LC-MS/MS.
הערה: פעם אחת מרוצה כי חלבון מתויג של העניין מחייב בהצלחה שותפי אינטראקציה, כמויות שווה של כל דגימה שכותרתו (קל, בינוני וכבד) משולבים והוגש לניתוח LC-MS/MS. מקובל להגיש 30 כולל μl של מדגם ה-IP לניתוח. זה יהיה כרוך ערבוב 10 μl של מדגם שכותרתו אור עם 10 μl של דגימות שכותרתו כבדות שכותרתו בינונית ו10 μl לתת בסך הכל 30 μl.
6 ניתוח נתוניי:. הבנת התוצאות, והסרה של הזדהויות נמוך ביטחון
הערה: רשימה של כותרות העמודות חזרו על ידי תוכנת Proteome Discoverer היא נתון בטבלה 1 Dif.תוכנת ferent (למשל, MSQuant, MaxQuant) יחזיר כותרות שונות, לעומת זאת, רק קבוצת משנה של אלה הנדרשים לניתוח ואלה משותפים לחבילות תוכנה השונות. נתונים תמיד צריכים לכלול מספר הצטרפות לכל חלבון שזוהה, היחס של השוואה כל יחס מדגם (אור לעומת בינוני, אור לעומת כבד, בינוני לעומת וכו 'כבדים), מספרם של פפטידים ייחודיים שזוהו, וצורה כלשהי של שיעור חיובי כוזב או אינדיקציה ביטחון.
- לפני עובר את הנתונים, להעתיק ראשון את הנתונים הגולמיים לגיליון אלקטרוני חדש. מגיליון אלקטרוני זה להסיר את כל עמודות פרט לאלה הנותנים את מספר הרישום, מספר פפטידים ייחודיים, יחסי השוואת דגימות, השתנות יחס, ותיאור החלבון. אם ניסויי העתק בוצעו, אלה צריכים להיות משולבים לתוך קובץ Excel אחד, עם כל ניסוי מופיע בכרטיסייה נפרדת.
הערה: נתוני אמון נמוך כוללים חלבונים שזוהו על ידי רק פפטיד ייחודי אחת, ואלה wכאן הכימות לא היה אפשרי. - השתמש בפונקציה 'המיון' מצטיין להורות על נתונים במספר פפטידים ולהסיר את הערכים של חלבונים חסרי פפטיד יותר מאחד. לאחר מכן לפי יחס ולהסיר חלבונים שחסרי יחסי SILAC (חלבוני unquantified).
- המרת יחסי SILAC ליומן ערכים 2 תוך שימוש בנוסחה: "= יומן (יחס SILAC, 2) ', שבו" יחס SILAC' מוחלף למזהה תא.
הערה: כתוצאות יחס SILAC בנתונים בשום צורה לחלבונים המראים ירידה בשפע להיות מוגבלת לערכים בין 0 ל -1, הוא כרגיל להמיר יחס SILAC ליחס SILAC יומן 2, כפי שזה אומר שני חלבונים מוגברים או ירידה ב מדגם מיוצגים בקנה מידת יומן שבו 2 או -2 מייצג עלייה של פי 4 או ירידה בשפע, ו3 או -3 עלייה של פי 9 או ירידה בשפע בהתאמה. בעקבות שינוי זה, הנתונים צריכים להתאים Gaהפצת ussian התרכזה סביב יחס יומן 2 SILAC של 0. דוגמא לכך היא נתון באיור 3. - יצירת עמודים חדשים בקובץ Excel ולחשב להיכנס 2 יחסי SILAC עבור כל המדגם / העמודות מדומה. עבור המרה של יחס מדומה / מדגם למדגם / יחס מדומה, השתמש בנוסחא: "= 1/ratio"
7 ניתוח נתונים השני:. בחירת אינטראקציות אמון גבוהה למחקר נוסף
- הפתוחה GraphPad פריזמה, בחר חדש> טבלת נתונים וגרפים ... 'טור' בחר מהרשימה בצד שמאל של החלון, ובחר את הזן / יבוא נתונים> הזן ערכי העתק, שנערמו לאפשרויות עמודות. לחץ על "צור".
- לבחור ולהעתיק לדוגמא / טור יחס מוק SILAC ניתנו יומן 2 מהגיליון האלקטרוני של Excel לקובץ פריזמה החדש.
- לחץ על התפריט הנפתח 'הוספת' ובחר 'ניתוח חדש'. על פי ניתוח עמודה בחר 'חלוקת תדר '. שמירה על אפשרויות ברירת המחדל לחץ על 'אישור'.
הערה: שלב זה יוצר היסטוגרמה הממחיש את מספר החלבונים שזוהו ביחס נתון. זה אמור ליצור הפצת גאוס. - בתיקייה 'התוצאות' הסעיף 'היסטוגרמה' חדש יהיה כבר נוצר. בחר את מקטע חלוקת תדר.
- לחץ על התפריט הנפתח 'הוספת' ובחר ניתוח חדש> XY> רגרסיה ליניארית (בכושר עקום). לחץ על "הפצת גאוס 'ולחץ על אישור.
הערה: צעד זה משתלב עקומה לנתונים חלוקת תדר שנוצרו בשלב 7.3, מניב ערכים שלאחר מכן ניתן להשתמש כדי לחשב את הספים למשמעות. - בחלון התוצאות שמופיע, מקבלים את הממוצע וסטיית תקן. צור סף על ידי הוספת 1.96 סטיות תקן לממוצע.
הערה: ערכים אלה מייצגים את הממוצע וסטיות תקן של dist גאוסribution, לא במערך הכולל. 1.96 סטיות תקן היו מקום סף ב95% גבול הביטחון (p ≤ 0.05). 2.58 SD ייתן 99%, ו3.3 99.9% SD. הסף צריך להיקבע בנפרד עבור כל ניסוי העתק כפי שהוא עשוי להשתנות.
8 ניתוח נתונים III:. מיזוג מערכי נתוני Replica
הערה: כדי להיות בטוח בזיהוי שותפי אינטראקציה ניסוי הנפתח SILAC טיפוסי אידיאלי צריכה להתבצע שלוש פעמים, עם המדיום ושכותרתו דגימות כבדות עבר לאחת מהחזרות כדי לשלוט על השפעה של התקשורת על התוצאות. חלבון שמופיע כאינטראקציה בלפחות שתיים משלושת הניסויים, עם 'מדגם switched' מדיה אידיאלי המייצג את אחד מאלה, הוא אינטראקציה ביטחון גבוהה.
- לחזור לקובץ Excel, צור כרטיסייה חדשה בשם 'בשילוב'. "הצטרפות עמודת תווית ולהעתיק את כל המספרים להצטרפות מכל oו הניסויים הבודדים לתוך עמודה אחת זה.
- בחר בכרטיסייה 'נתונים', ולאחר מכן את האפשרות "להסיר הכפילויות".
- יצירת עמודות עבור יחסי SILAC, והשתנות יחס מכל ניסוי, כמו גם עבור העמודה שם / תיאור החלבון.
- בכרטיסיית התיאור, השתמש בנוסחת VLOOKUP כדי לאסוף את התיאור של כל מספר הצטרפות. גרור את הנוסחה למטה את העמודה כדי למלא את תיאור החלבון. נוסחת VLOOKUP היא: "= VLOOKUP (AccessionNo, WheretoLook, ColumnsAccross, שקר)", שבו: AccessionNo הוא aX $ עם X להיות מספר שורת WheretoLook הוא 'שם כרטיסיית ניסוי' $ 2 $:.! Y $ Z $ שבו Y ו-Z הוא החתיכה הימנית התחתונה של הנתונים שבהפנית הגיליון האלקטרוני. ColumnsAccross הוא מספר העמודות מול num ההצטרפותבער בעמודת ערך רצוי שקרים.
הערה: לדוגמא, אם מספר רישום הוא בעמודת A, ואת הנתונים של עניין טמון בטור א הערך כאן יהיה 5 (עמודה נחשב 1). כמספרי הצטרפות היו נקווים בשלב ב ', סביר להניח שVLOOKUP לא יקבל את כל שמות הנדרשים מניסוי אחד. איפה היא מחזירה N / A, לשנות את הנוסחה כדי להשיג את התיאור מכל ניסוי בתורו עד שכל התיאורים שנרכשו. - כדי להדגיש את האינטראקציה חלבונים במערך הנתונים המשולבים, בחר כל עמודת יחס אישית ולחץ על כרטיסיית הבית, ואחריו כללי תאים> הדגש על העיצוב המותנה> עוד כללים.
- סגנון בחירה 'קלאסית'. עיצוב תאים המכילים רק "ערך תא ',' גדולים או שווה ל". בתיבה, הקלד את ערך סטיית תקן 1.96, ולחץ על 'אישור'.
- להעריך את השתנות יחס לכל אינטראקציה חיובית. אם הפחתת var%iability היה טיפה 'מכה' מתחת לערך הסף, זה צריך להיות מטופל בזהירות.
שים לב: זה מייצג כמה עקבי היחסים האישיים לכל פפטיד שיחד מרכיבים את יחס SILAC 'חלבונים, וניתן כאחוז. כאשר השתנות היחס נלקחת בחשבון וחלבון נותן יחס SILAC מעל הסף, חלבון זה מייצג להיט. אם השתנות היחס נותנת מגוון שנופל מתחת לסף, 'מכה' עשוי לייצג מזהם. - חזור על פעולה זו עבור כל אחת מעמודות התוצאות ולהשוות על פני ניסויים. חלבונים מסומנים שזוהו בשניים או יותר ניסויים מייצגים אינטראקציה ביטחון גבוה.
הערה: בהתאם למאגר המידע המשמש להקצאה מספרי הצטרפות חלבון, בניסויים שונים של חלבון יכול להיות מזוהה על פי מספרי הצטרפות שונים, או אפילו מרובים. חשוב לבדוק זאת על מנת להבטיח כי חלבונים לא הושמטו בטעות.
תוצאות
בניסוי הנפתח SILAC טיפוסי, הרוב המכריע של חלבונים שזוהו (> 90%) מייצג מזהמים כמו גם חלבונים מחייבים שאינם ספציפי למטריצת זיקה וזה מודגם באיור 2 ב ', גם כאשר פרוטוקולי כביסה להסיר רוב המזהמים cytoplasmic כגון (איור 2 א) GAPDH. עם זאת, באשכולות של הלא ספציפי מחייבים חלבונים בהתפלגות נורמלית מאפשר לי חלבונים באופן ספציפי להיקשר לחלבון של עניין יש להבחין כמו אלה מדגם / יחסים מדומים גבוהים יותר מהרקע. בעוד מזהמים אמורים באופן תיאורטי אשכול סביב יחס יומן 2 SILAC של 0, זה לא בהכרח המקרה, דוגמא לכך היא נתון באיור 3. סיבות אפשריות לכך כולל תיוג SILAC מושלם של תאים, טעינת כרכים או ריכוזים של lysate שוויוניים על חרוזים אנטי-GFP, אובדן מקרי של חרוזים במהלך טיהור או שילוב שוויוני ing של דגימות בסוף הליך הטיהור 3. עם זאת, הנתונים בהנחה שהם נותחו על בסיס סטיית תקן סף מהממוצע של המזהמים מתפלגים נורמלי, משמרות קטין במרכוז של נתונים לא אמורה להשפיע על איכות התוצאות.
כאשר משווים את ההבדלים באינטראקציות חלבון בין שני חלבונים קשורים, מצב דומה עלול להתרחש בו חלבון אחד של העניין מופק לרמות גבוהות יותר בתוך תאים משנייה (או בשל הבדלים ביעילות transfection, או נכס מהותי של החלבון או mRNA) . וריאציה מסוימת בביטוי (לדוגמא, איור 2 א) ניתן לתיקון על ידי ניתוח ליחס SILAC לשתי דגימות אלה. בדוגמא זו אלה יהיו דגימות ה-GFP-eIF4AI וה-GFP-eIF4AII. על ידי ניתוח יחס 4AI/4AII SILAC כאמור בסעיף 7, ניתן לזהות חלבונים שמחייבים משתנה באופן משמעותי בין isoforms.
class = "jove_content"> באיור 4, ייצוג מחייב חלבוני eIF4A זיהו באחד הניסויים שנערכו ההעתק מוצג הממחיש את הכיסוי של מתחם גורם ייזום פרוטוקול זה הושג. היחס הגבוה ביותר נצפה בדרך כלל עם eIF4G, אשר נקשר ישירות לeIF4A, עם יחס נמוך יותר עבור eIF4E, אשר נקשר לeIF4G באתר מהאתר מחייב eIF4A. יחס נמוך יותר נצפה עבור חברים של מתחם eIF3. עם זאת, זה ממחיש באופן ברור כי הניסוי באופן משביע רצון זיהה שותפים המחייבים הן ישירים ועקיפים של eIF4AI ושני. כפי שניתן לצפות מהזהות גבוהה רצף 6, חלבון: אינטראקציות חלבון הופיעו נשמרים במידה רבה בין שני isoforms במערכת זו ניסיונית 7, 8. בחירה של חלק מחלבוני האינטראקציה מוצגת בטבלה 2, הממחישה את פורמט הנתונים.
| כותרת עמודה | תיאור |
| הצטרפות | מציג את מספר ההצטרפות לרצף |
| כיסוי | חלקם של רצף החלבון מכוסה על ידי פפטידים זוהו |
| ♯ PSM | התאמה רפאים פפטיד |
| ♯ פפטידים | מספר כולל של פפטידים ייחודיים שזוהו לחלבון |
| ♯ AAS | אורכו של חלבון בחומצות אמינו |
| MW (דה) | המשקל המולקולרי של חלבון בDaltons. לא כולל שינויים |
| calc. PI | הנקודה איזואלקטרית התיאורטית של חלבון |
| ציון | הציון הכולל של חלבון (המייצג את הסכום של indivעשרות פפטיד idual). הציון המדויק הנדרש למשמעות ינוע בין ניסויים. מתקן MS בדרך כלל יחול 5% הפסקת שיעור גילוי שווא. |
| רצף | רצף חומצות האמינו המרכיבות את החלבון |
| יחס | העצמה היחסית של פפטידים בשם מדגם שכותרתו, בהשוואה למדגם שכותרת שנייה |
| רוזן יחס | מספר יחסי פפטיד ששמשו לחישוב יחס חלבון נתון |
| השתנות יחס (%) | ההשתנות של יחסי פפטיד הבודדים המשמשים לחישוב יחס חלבון נתון |
| תיאור | שמו של החלבון |
טבלת 1. כותרות עמודות רגילה מדו"ח Proteome Discoverer.בעוד מידע שימושי ניתן להשיג מכל אחת מהעמודות אלה, אלה קריטיים לניתוח זה מוצגים באותיות מודגשות.
| הצטרפות | פפטידים | 4AI/Mock | 4AII/Mock | שם | ניתוח SILAC |
| A8K7F6 | 21 | 100 | 1 | eIF4AI | חלבון 'פיתיון' |
| Q14240 | 22 | 0.01 | 90.855 | eIF4AII | |
| G5E9S1 | 25 | 47.575 | 30.53 | eIF4GI | אינטראקציה חלבונים |
| Q59GJ0 | 5 | 11.778 | 10.619 | eIF4GII60; | |
| P06730 | 3 | 7.22 | 7.57 | eIF4E | |
| Q5T6W5 | 4 | 0.685 | 0.646 | hnRNPK | מזהמים-במיוחד שאינו מחייבים |
| P62805 | 1 | 0.531 | 0.498 | H4 היסטון | |
| H6VRG2 | 18 | - | - | קראטין-1 | מזהמים סביבתיים |
| P35527 | 11 | 0.01 | 0.01 | cytoskeletal קרטין-1 9 |
טבלה 2. נתונים אופייניים מניסוי immunoprecipitation SILAC. מתן נתונים דוגמא לחלבון של עניין / פיתיון (פפטידים גבוהים, יחס גבוה), חלבוני אינטראקציה עם חלבון של עניין (פפטידים גבוהים / נמוכים, יחס גבוה), שאינו מחייבים באופן ספציפי חלבונים (pept גבוה / נמוךאידו, יחס יורד מתחת לסף - בניסוי זה 0.96), ומזהמים סביבתיים (לעתים קרובות פפטידים גבוהים, יחס שלילי / מתחת לסף).

איור 1. ניסויי תכנית. ראשית תאים גדלים בתקשורת חסרת ארגינין וליזין ולהחליף עם ארגינין איזוטופ היציב שכותרתו ויזין 2 שבועות (1). (2) תאים הם זורעים לתוך 10 סנטימטר 2 מנות וtransiently transfected עם פלסמידים קידוד ה-GFP (מוק) או חלבוני GFP היתוך (דוגמאות). (3) תאים lysed והחלבונים GFP או GFP היתוך הם immunoprecipitated מlysates התא. (4) דוגמאות משולבות בביחס של 1:1 והגישו לניתוח LC-MS/MS. הנתונים נותחו לאחר מכן להסיר את זיהוי חלבון אמון נמוךכישורים, וכדי לבחור ברמה של העשרת החלבון המתאימה לחלבוני אינטראקציה אמיתיים.
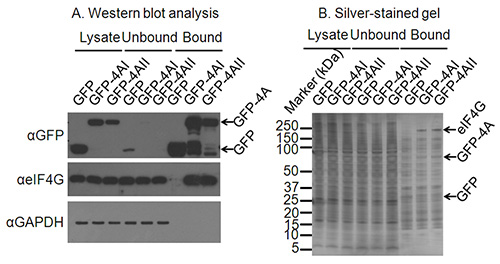
איור 2. אישור תנאי immunoprecipitation מתאימים. א) ניתוח מערבי כתם של lysates תא, כמו גם שברים מאוגדים וכרוך מimmunoprecipitation מאשר ביטוי וimmunoprecipitation של החלבון של עניין. כתם מערבי נגד GAPDH מאשר דלדול של חלבונים שאינן אינטראקציה וכתם מערבי עוד שותפת אינטראקציה ידועה של eIF4A מאשרת immunoprecipitation המוצלחת של חלבוני קשירה לחלבון של עניין. ב) שותפים באינטראקציה של חלבון של עניין אינם ידועים, ג'ל מוכתם כסף רשאי לאשר immunoprecipitation של חלבוני אינטראקציה. על כסף הכתם הזה להקות ג'ל ed עבור ה-GFP וGFP-eIF4AI/II ברורות, ולהקה הנודדת בגודל הנכון עבור eIF4G קיים רק בנתיבי GFP-4AI/II-bound ולא בנתיב שליטת ה-GFP.

איור 3. נציג תוצאות. היסטוגרמה המציגה את ההתפלגות של יחסי חלבון מן חוזר אחד () הנפתח ה-GFP-eIF4AII ה-GFP-eIF4AI או (ב). הפסקת סטיית תקן 1.96 מסומנת בקו מקווקו. חלבוני אינטראקציה נופלים מחוץ למזהמים בדרך כלל המופצים ניכרים מ~ 0.25 ו1 ב (א), ו ~ .3-1.5 ב( ב ').
les/ftp_upload/51656/51656fig4.jpg "/>
איור 4. זיהוי של חברים מורכבים eIF מהעתק אחד של ניסוי SILAC IP. חלבונים בירוק היו החלבון של עניין משמש לנפתח אינטראקציה חלבונים מוצלים מאדום לדברים לבנים להיכנס 2 יחס SILAC בIP SILAC עם אדום להיות החלבון הנפוץ ביותר בניתוח, ולבן להיות הפסקת 1.96 SD. חלבונים המוצלים באפור לא זוהו בניתוח זה.
Discussion
אסטרטגית הנפתח SILAC המתוארת כאן מהווה אמצעי רגיש מאוד ורב עוצמה של גילוי חלבון חדש: אינטראקציות חלבון, ויתר על כן מאפשרת האפליה המהירה ופשוטה של דפוסים המחייבים שינו בין דגימות קשורות קשר הדוק של עניין. בדוגמא זו טכניקה זו הייתה בשימוש כדי לחקור את החלבון: אינטראקציות חלבון של חלבוני eIF4AI וeIF4AII 6. לידיעתו של המחבר, זהו המחקר הראשון בספרות מנצלת את השירות של פרוטאומיקה SILAC לחקור interactome הסלולרי של שני isoforms של eIF4A אלה.
הגישה כפי שתואר לעיל משתמשת GFP-תג ואנטי-GFP חרוזים 9, 10 ולכן שינויים עשויים להידרש כדי לאפשר גישה זו כדי לשמש לחלבון ספציפי של עניין, לדוגמא אם התג ממוקם בN-או C-סופית של חלבון. איפה, כתמים מערביים אפשריים או מבחני פונקציונליים צריכיםיש לבצע כדי לזהות מחייב של שותף אינטראקציה חלבון ידוע. צריך חלבון לא יסבול היתוך עם תג ה-GFP, תיוג אחר או אסטרטגיות הנפתח הוחלו pulldowns SILAC באמצעות שני תגים אחרים (FLAG 11, 12 ביוטין, סטרפטוקוקוס (הנתונים שלו,) לא פורסם), או על ידי שימוש בנוגדנים ראשוניים נגד חלבון עניין שבו מציאה siRNA של חלבון המטרה מספקת מדגם שליטה 13. ניסויים כאלה כבר מתוארים במקומות אחרים בספרות, אבל בקצרה, שלב 1 ושלבים 5.4-8 היינו להיות מיושמים כאמור לעיל, בצעדים 2-5.3 שונה בהתאם למערכת ביטוי / הנפתח של בחירה באמצעות תשומות חלבון שווים כפי שמתואר ב צעדים 2.4-2.5. כשלבי הכימות לאפשר חלבונים המחייבים שאינם ספציפיים שיש להסיר ברמת הניתוח, מומלץ להשמיט מראש דגירה עם חרוזים שליטה, או שטיפות מלח גבוהות על מנת לשמר חלבון דל זיקה: אינטראקציות חלבון עם חלבון של עניין . Ma nucleasey להיכלל או הושמט מפרוטוקול זה על פי הפרטים של ניסוי מסוים. לדוגמא: כמו החלבונים המשמשים בשיטה זו הם helicases RNA, קוקטייל RNase נכלל בפרוטוקול כדי להסיר אינטראקציות עקיפות בתיווך באמצעות RNA (שלב 3.2). במקרים מסוימים עם זאת, יכול להיות שיש להפיק תועלת מביצוע ניסויים במקביל עם ובלי nuclease לזהות אינטראקציות תלויות חומצות גרעין.
בתוך פרוטוקול זה, המיתוג של "בינוני" ודגימות 'כבדות' בניסויים לשכפל מומלץ לשלוט וריאציה הוצגה על ידי הבדלים בתקשורת SILAC או צמיחת תאים. שליטה חלופית כרוכה במעבר הרציף של כל שלושה (, 'הבינוני', 'אור' ו 'הכבדות') כלי התקשורת בניסויי העתק. בעוד גישה זו היא שעלולה להיות מחמירה יותר, זה להגדיל את המורכבות של הניתוח, כמו בלשכפל אחד לפחות, חלבון של עניין wחולה להיות מיוצר בתאים 'אור' שכותרתו ולכן יש צורך להבחין בין החלבונים שזוהו באופן עקבי בדגימות 'אור' (מזהמים סביבתיים כגון keratins), ואלה שמועשרים רק בדגימות 'אור' כאשר חייב חלבון של ריבית.
תוך השימוש בנתוני כימות מאפשר אפליה של ספציפיות מאינטראקציות לא ספציפיות באמצעות שימוש בסף, באופן בלתי נמנע כמה אינטראקציות אמיתיות עשויות להיות מושלכת. הגישה הנ"ל היא גישה פשוטה ומהירה לזיהוי חלבון: אינטראקציות חלבון זה יכול להיות ניסיון בקלות על ידי חוקרים ללא ניסיון קודם עם ספקטרומטריית מסה, או ניתוח של חלבון גדול: מערכי נתונים אינטראקציה חלבון. עבור רוב השימושים זה יותר ממספיק לזיהוי חלבונים חדשים של עניין. שינויים בהמשך לגישה זו כדי לעזור להפחית אובדן נתונים זה מתוארים במקומות אחרים בספרות, וכוללים שימוש ביחסי ציבורספרייה בתדירות otein בי ידועות חלבונים מזהמים לקבוצה מסוימת של פרמטרים ניסיוניים (קו תא, מטריצת חרוז, תנאי חיץ) עשויה להיות שלילית 10, 14. עם זאת, בהתאם לפרמטרים של ניסוי מסוימים זה עשוי להיות נחוץ כדי להפעיל מספר ניסויי שליטה לייצר proteome חרוז וזה ולכן יכול להגביר הן את חשבון והמורכבות של הניסוי. מידע נוסף על טכניקה זו הוא זמין מאתר האינטרנט www.peptracker.co.uk 14.
צריך גם לציין, כי הפרוטוקול שתואר לעיל כרוך בערבוב דגימות שכותרתו שונה בסופו של תהליך immunoprecipitation (שמכונה ערבוב לאחר טיהור - ניסוי SILAC MAP). הדבר נעשה כחלבון: אינטראקציות חלבון להתרחש בשיווי משקל נתון 15. יש לציין שקבוצות אחרות יש בשילוב גישת SILAC מפה זו מתוארת בפרוטוקול זה עם דגירה של הדגימותלפני הנפתח (טיהור לאחר ערבוב - PAM SILAC) עבור אורכים שונים של זמן (20 דקות לשעה 2 היו בשימוש בספרות) 15, 16. בהתבסס על כמה מהר יחס חלבון יורד לכיוון 1:1, ניתן לחקור באופן איכותי זיקות מחייבות ולהגדיר חלבונים כמו חלבוני אינטראקציה יציבים או דינמיים 15.
לסיכום, pulldowns SILAC מייצג אמצעי רב עוצמה לזיהוי חלבונים באינטראקציה עם חלבון נתונה של ריבית, בסביבה רלוונטית מבחינה פיזיולוגית. הטכניקה יכולה להיות מותאמת בקלות למספר אסטרטגיות טיהור שונות, המאפשרת יישום שלה לכל חלבון נתונה של ריבית. כימות של תוצאות בהרבה מפשטת זיהוי של אינטראקציות אמיתיות, ומאפשרת הרפיה של תנאי חיץ מחמירים המשמשים להסרת קלסרים שאינם ספציפיים, ובכך שומרת על אינטראקציות זיקה נמוכות. כפי שניתן לעומת עד שלוש דגימות באמור לעילאסטרטגיה, יש לו את הטכניקה עוצמות ברורות בהשוואת הבדלים בחלבון קושר בין isoforms שונה חלבון, חלבוני מוטציה, או את ההשפעה של מעכבים תרופתיים. כפרוסות ג'ל כל מנותחות ולא להקות בודדות שכתם על ידי Coomassie, המספרים של חלבונים שזוהו בוודאות גבוהה הם בדרך כלל גבוהים יותר מאלה שזוהו בGST / TAP-הנפתח סטנדרטיים, והנסיין הטיה בבחירת חלבונים של עניין מוסר. הטכניקה ולכן משווה מאוד לטובה עם טכניקות נפוצות אחרות המשמשות בזיהוי של חלבונים אינטראקציות רומן (שמרים 2 היברידי, GST / או pulldowns TAP).
Disclosures
החוקרים מצהירים כי אין להם אינטרסים כלכליים מתחרים.
Acknowledgements
עבודה זו נתמכה על ידי מענקים מקרן Wellcome וBBSRC לIG. IG הוא Wellcome עמית בכיר.
Materials
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
References
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved