Method Article
SILAC-immunoprecipitation Kantitatif PROTEOMİK kullanma Memeli Hücrelerde Protein Etkileşim Ortakların Kimlik
Bu Makalede
Özet
Protein etkileşimleri: SILAC immunopresipitasyon deneyler yeni proteini keşfetmek için güçlü bir araç temsil eder. Sağlayarak kontrol ve test örnekleri, gerçek etkileşim hem de protein bolluğu doğru nispi miktar kolaylıkla deneysel kirletici ayırt edilebilir ve düşük afiniteli etkileşimler daha az sıkı tampon koşullarında kullanımı yoluyla korunur.
Özet
Protein etkileşimleri: İmmün afinite temizliğinin, SILAC immüno ile birleştirildi, kantitatif proteomik yeni bir proteinin keşfi için güçlü bir aracı temsil etmektedir. Kontrol ve test örnekleri hem de protein bolluk doğru nispi miktarını vererek, gerçek etkileşimleri kolayca deneysel kirletici ayırt edilebilir. Düşük afiniteli etkileşimler daha az sıkı tampon koşulların kullanılmasıyla boyunca korunmuş ve kolaylıkla tanımlanabilir devam edilebilir. Bu protokol bir kütle spektrometresi tesisine sunulması için hazırlanmasını takiben ilgi proteini, etiketlenmiş bir yakınlık kararlı izotop etiketli amino asitler, nakil ve immunoprecipitation doku kültürü hücreleri etiketleme tartışır. Bu protokol daha sonra ilgi bir protein ile etkileşim hücresel ortakları belirlemek için kütle spektrometresi döndürülen verileri analiz etme ve yorumlama anlatılır. Bir örnek olarak, bu teknik, kimliklerini uygulanıreIF4AI ve eIF4AII: ökaryotik translasyon başlatma faktörleri için bağlama proteinleri haklı göstermeye çalışıyorlar.
Giriş
Protein işlevlerini anlamada önemli bir adım, ilgili etkileşen proteinlerin belirlenmesidir. Bu tür proteinler bilinmeyen Nereye mevcut tekniklerin bir dizi, kendi yararları ve sakıncaları ile her vardır. Bu maya iki-hibrid sistemi, yeniden birleştirici protein kullanılarak açılan deneyleri, hem de tandem afinite arıtma ya da kademe etiketleme 1, 2 içerir.
Bu teknikler için daha yeni bir ek (SILAC) 3 hücre kültürü içinde amino asitlerin izotop etiketleme kullanılarak niceliksel ardından kütle spektrometrisi ile ilgili bir memeli hücre hattından ilgi konusu bir proteinin afinite saflaştırma birleşimidir. Bu, hücre lokalizasyonu ve post-translasyonel modifikasyonlar maya iki-hibridli bir yaklaşım göre avantajları fazla avantaj, hem de tedirgin değildir sahip geleneksel TAP etiketlemeyi rea olanak sağlayan bir nicel değil, nitel yaklaşım olmasıylaDily özellikle bağlanan konak faktörler, proteinler ve kirletici etkileşim spesifik olmayan ayırt. Bir örnek olarak, tipik olarak daha çok tek tek protein bantları dışında, bütün incelendiğinde daha ayrıntılı olarak, ilgi konusu proteinlerin benzer bir protein jeli üzerinde göç eden, ne de, tipik olarak boyanarak görünür olması için yeterli seviyelerde mevcut olması gerekir, yol maskelenir değildir güvenle belirlenen protein 4 artan sayıları.
Bu tekniği göstermek için, yakından ilgili ökaryotik çeviri başlatma faktörü GFP füzyonlarıdır eIF4AI ve eIF4AII% 90 amino asit kimliği üzerindeki payının SILAC-immünopresipitasyon nicel Proteomikte tarafından incelenmiştir. İnsan eIF4AI ve II GFP eIF4A of N-terminaline bağlı bir füzyon proteini oluşturmak için pEGFP-C1 klonlanmıştır. Geçici transfeksiyon izotop işaretli 293T hücreleri, bu yapıları teslim etmek için kullanılmıştır kararlı hücre kuşaklarının üretilmesi için kaçınmak gerekir.
Hücreler ilk olarak ilgi konusu bir proteini kodlayan plasmid DNA transfeksiyonu ardından hücre kültürü ortamı SILAC iki hafta boyunca işaretlendi. Hücreler daha sonra, lize normalleştirilmiş protein konsantrasyonu ve lizat afinite eşit miktarlarda anti-GFP agaroz üzerinde saflaştırılmıştır. Eluatın eşit miktarları daha sonra, bir araya getirilmiş ve LC-MS/MS analizi için gönderilmiştir. Bu analizin sonuçları daha sonra yüksek bir güven proteini tespit etmek için işlenir: protein etkileşimleri için (Şekil 1).
SILAC immüno doğrudan etkileşimleri hem de düşük afinite ya da protein kompleksleri 4 ile doğrudan etkileşimleri sadece tanımlanmasını sağlar. Bu sistemi kullanarak, eIF4AI ve II imüno birincil bağlanma ortağı eIF4G (izoformlar I / II ve III) 5 tekrarlanabilir ve güvenli kimlik, hem de eIF4E ile dolaylı etkileşimleri ve eIF3 kompleksinin çeşitli bileşenler izin verdi.
Protokol
1.. Üretimi ve SILAC etiketli Hücre Hatlarının Pasajlanması
Not: tripsin örneklerin eksik etiketleme yol açacak etiketsiz amino asitleri içerebilir olarak tripsin-EDTA kullanımının analizi için deneysel örneklerin pasaj ve hazırlama tüm aşamalarında kaçınılmalıdır.
- SILAC ortamın hazırlanması için bir şişe içeren (Arg / Lys DMEM bir 500 ml şişeye, bir uygun SILAC etiketli argininin (84 mg / ml PBS içinde) ve lizin (PBS içerisinde 146 mg / ml) içinde bir 0.5 ml bir kısım eklemek L-glutamin).
- Daha sonra, 500 ml şişeye diyaliz edilmiş FBS ve 50 ml penisilin / streptomisin, 5 ml ekleyin.
Not: HEK 293T hücreleri (ATCC) yoksun DMEM ortamı içinde muhafaza edilmelidir arginin ve lisin ve ışık (R0K0) ile desteklenmiş, orta (R6K4) ya da ağır (R10K8) amino asitler, diyaliz edilmiş cenin sığır serumu (10 kDa kesim) ve penisilin / streptomisin. Hücreler, tam sağlamak için 5 hücre bölünmesi en az ortam içinde muhafaza edilmelidiretiketleme. Çoğu durumda, hücreler kolaylıkla ≥ 2 hafta içinde etiketlenir. - Geçiş için hücreleri bölmek için, hücreler şişeyi vurarak tek tabaka yerinden alınmalıdır. Alternatifler hücre kazıyıcı ya da enzim içermez, PBS-tabanlı bir hücre ayırma tamponu ile kullanımını içerir.
Not: ortamını korumak için, hücreler T25 kaplarına aktarılmıştır edilmeli ve büyük hücre sayıları yalnızca hemen önce bir deneme oluşturulur. - Önce transfekte hücrelere 24 saat, inceleme altındaki her deneysel koşul için tek bir 10 cm 2 çanak içine 3.5 x 10 6 SILAC etiketli hücreler tohum.
PEGFP-füzyon Yapıların ile Etiketli Hücreler 2. Transfeksiyonu
- Hücrelerden ortamı çıkarın ve antibiyotik içermeyen SILAC DMEM'den (Hafif, Orta veya Ağır) medya 9 ml ile değiştirin.
Not: lipozom-esaslı transfeksiyon reaktifleri verimliliğine müdahale olabilir çünkü Antibiyotikler ortama ilave edilmemelidir. - PrepaUygun plazmidi 10 ug bir karışımı yeniden (epEGFP-C1, pEGFP-eIF4A-I, pEGFP-eIF4A-II) antibiyotik içermeyen SILAC DMEM 500 ul ve antibiyotik içermeyen DMEM SILAC 10 ihtiva eden 500 ul ile karıştırarak transfeksiyon reaktifi ul (örneğin, lipofektamin 2000). Yukarı ve aşağı pipetleme birkaç kez iyice reaksiyonu karıştırın.
- 20 dakika boyunca, oda sıcaklığında, reaksiyon karışımı inkübe edin ve daha sonra damla damla, hücre mono tabakasına ilave edin. Yanlara doğru hafifçe plaka sallayın.
- 37 ° C'de transfekte hücreler inkübe edin ve% 10 CO2, 24 saat boyunca.
Not: belirgin bir toksisite ilgi sonuç, bir proteinin ifadesi, o zaman transfekte edilen plazmid miktarı ve / veya daha sonraki sentezleme dönemin süresini azaltmak için gerekli olabilir.
3.. Hasat hücre lizatları
- Bir hücre kazıyıcı kullanarak buz gibi soğuk PBS içine çanak Hasat hücreleri. 4, 220 x g'de santrifüj ile hücreler toplayın76, 5 dakika boyunca C. Buz soğukluğunda 10 ml PBS içinde hücreleri bir başka 3 kez yıkayın.
- Hücre parçalama tamponu 200 ul 5 ul 1x konsantrasyon ve RNaz kokteyl (isteğe bağlı) de taze ilave proteaz önleyici kokteyl III ihtiva eden (10 mM TrisCI / pH 7.5, 150 mM NaCl, 0.5 mM EDTA,% 0.5 NP-40) 'de, hücre pelletini ml başına.
Not: bağlanma aktivitesi, bilinen nükleik asit ile proteinler için, önce çökelmesine lizat nükleazlar eklemek için gerekli olabilir. Nükleaz eklenmektedir durumlarda, numuneler her 10 dakikada bir pipetleme ile, buz üzerinde 30 dakika boyunca buz üzerinde inkübe edilmelidir. Agaroz jel elektroforezi ile analiz numunesi ve küçük bir kısmı toplam nükleik asit ayıklama nükleaz etkinliğini test edebilir. - 13,000 x g'de santrifüjleyin örnekleri, 10 dakika boyunca 4 ° C, çözünür hücre lizatı gibi süpernatant saklayın.
- Hücre lizatının konsantrasyonu, üretici firmanın talimatlarına uygun olarak BCA deneyi ile değerlendirilmelidir.
- 500 ul'lik bir nihai hacimde protein konsantrasyonu normalleştirmek için proteaz önleyici kokteyl III ihtiva eden liziz tamponu kullanın.
- Seyreltme tamponu içinde 500 ul eklenmesiyle 1x nihai konsantrasyonda proteaz önleyici kokteyl III ihtiva eden (10 mM TrisCI / pH 7.5, 150 mM NaCl, 0.5 mM EDTA) ile, 1 ml için ses ayarlayın. Numunenin 50 ul alikosu, numune girişi olarak muhafaza edilmeli ve lizat (örneğin, GFP-trap), anti-GFP boncuk hazırlama esnasında buz üzerinde tutuldu.
Not: Tipik verimler, nihai 1 ml numune içinde protein 1-3,5 mg arasında değişir. Yukarıdaki tampon ilgi çekici pek çok protein için uygun iken, diğerleri için yem protein çözünebilir sağlamak için ve protein-protein etkileşimleri korumak için tampon bileşenleri değiştirmek için gerekli olabilir. Olası değişiklikler tamponlama maddesi (fosfat, HEPES), tuz konsantrasyonu (150-500 mM), deterjan seçimi veya diğer katkı maddelerini içerir.
4.. Anti-G BağlamaFP Boncuk
- Kısaca boncuklar tekrar süspansiyon boncuk bulamaç vorteks. Ucu olan bir 200 ul bir pipet ucu kullanılarak kesilmiş, yeni bir tüpe örnek başına 25 ul boncuk aktarın.
Not: kullanıcı örnek-örnek varyasyonu en aza indirmek için bir master olarak tek SILAC deney için boncuk hazırlamalıdır. - Boncuk bulamacın her 25 ul için, 5 dakika boyunca 2,700 x g de boncuk seyreltme tamponu 20 hacim (75 ul bulamaç başına 1500 ul) ekleyin ve santrifüj. Daha sonra, seyreltme tamponunun 20 hacminde boncuklar bir başka 2x yıkayın.
- 25 | il boncuk çamuru başına 100 ul seyreltme tampon çözeltisi ekleyin. Ucu olan bir 200 ul ucunu kullanarak, kesme aşamasında 3,6 SILAC etiketli numunelerinin her biri için bu yeniden süspansiyon haline getirilmiş bulamaç 85 ul transfer.
- 2 saat boyunca 4 ° C'de bir karıştırıcı üzerine boncuklar ile inkübe edin.
MS Analizi için Numune 5. Yıkama, sıyrılması, ve Hazırlama
- 2700 xg santrifüj örnekleri, 46, 5 dakika boyunca C. Süpernatan 50 ul üstte yüzen madde atıldı ve geri kalan, bağlanmamış numune olarak muhafaza edilmelidir.
- 1 ml seyreltme tamponu boncuklar tekrar süspansiyon her bir tüpe ilave edilmelidir ve numune 5 dakika boyunca 4 ° C, 2700 x g'de santrifüj edilir. Süpernatan atılmalıdır. Bu, iki kez uygulanmalıdır.
- 2x SDS yükleme tamponu içinde 50 ul eklenmesi ile boncuklardan proteininin elüt edilmesi, ve 10 dakika boyunca 95 ° C'de ısıtma. 4 ° C'de 2 dakika boyunca 2700 x g'de santrifüjleme ile boncuk Pelet
- Kullanılana kadar daha sonra -80 ° C'de saklanabilir yağlanan tüpler içinde süpernatant saklayın. Bir kütle spektrometresi tesisine sunulması için, etiketli örnekleri 01:01:01 (örneğin, her biri 10 ul) karıştırın ve karışık örneğini göndermek.
Not: Bu noktada numune western blot, gümüş lekeleme ya da bilinen / bilinmeyen bağlanma partnerleri ile etkileşim için test etmek için başka yollarla test edilebilir. Bir örnek şekil l'de verilmiştirwestern blot ile eIF4G ve ilgi konusu bir protein açılan numuneleri içinde bulunan gümüş boyama bantlarının görünüşü, fakat, bir kontrol numunesi, - bilinen bir etkileşim ortağının spesifik bağlanma gösteren ure 2. - LC-MS/MS analizi için örnek gönder.
Not: bir ilgi etiketli protein başarıyla etkileşim ortaklarını, her etiketli örnek eşit hacimlerde (hafif, orta ve ağır) birleştirildi ve LC-MS/MS analiz için gönderilen bağlayıcı olduğunu bir kez memnun. Bu analiz için, bir IP örnek 30 ul toplam gönderme olağandır. Bu, 30 ul toplam vermek üzere orta-etiketlenmiş ve 10 ul ağır etiketli örnekler 10 ul ile hafif etiketli örnek 10 ul karıştırma açmak olur.
6 Veri Analizi I:. Sonuçlar anlama ve Düşük güven Tanınmaları çıkarılması
Not: Proteom Discoverer yazılım tarafından döndürülen sütun başlıklarının bir listesi Tablo 1'de verilmiştir dif.ferent yazılımı (örneğin, MSQuant, MaxQuant), ancak bu yalnızca bir alt kümesini farklı başlıkları döndürecektir analiz için gerekli ve bu çeşitli yazılım paketleri için ortak vardır. Veriler her zaman oranı en her örnek oranını karşılaştırarak tanımlanan her protein için bir erişim numarası, (orta vs hafif, ağır vb vs ağır, orta vs ışık) içermelidir, tanımlanan benzersiz peptidler sayısı ve yanlış pozitif oranı ya da güven göstergesi çeşit.
- Veri üzerinden gitmeden önce, ilk olarak yeni bir elektronik tabloya ham verileri kopyalayın. Bu tablonuzda üyelik numarasını vererek dışındaki tüm sütunları kaldırmak, eşsiz peptidler sayısı, örnekleri, oran değişkenlik ve protein açıklamasını karşılaştırarak oranları. Çoğaltma deneyler olsaydı, bu her deney ayrı bir sekmede görünen ile, tek bir Excel dosyası içine kombine edilmelidir.
Not: Hafif güven veriler sadece tek tek peptid tarafından tanımlanan proteinler içerir ve bu wburada miktar mümkün değildi. - Peptidler sayısına göre verileri sipariş ve birden fazla peptid eksik proteinlerin girdileri kaldırmak için üstünlük 'çeşit' işlevini kullanın. Sonra oranına göre sıralamak ve SILAC oranları (belirlenmemiş proteinler) eksikliği proteinleri uzaklaştırmak.
- 'SILAC Oranı' hücre tanımlayıcı için ikame edilir '= log (SILAC Oran, 2)': formülü kullanarak günlük 2 değerlerine SILAC oranları dönüştürün.
Not: 0 ve 1 arasında bir değere sınırlı olduğunu bolca bir azalma gösteren proteinler için herhangi bir veri oranının bir SILAC Sonuç olarak, bu artmış ya da azalmış proteinleri de anlamına gelir, bir günlük 2 SILAC oranına bir oranını SILAC dönüştürmek olağandır örnek 2 veya -2, 4-kat artış veya azalma bolca ve 3, ya da -3 9-kat bir artış ya da azalma, sırasıyla bolca temsil eden bir log ölçeğinde temsil edilmektedir. Bu dönüşümden sonra, veriler bir Ga oturmalıdır0 ile 2 arasında bir günlük SILAC oranı etrafında ussian dağılımı. Bunun bir örneği, Şekil 3'te verilmiştir. - Excel dosyasında yeni sütunlar oluşturun ve tüm numune / sahte sütunları 2 SILAC oranları log hesaplamak. "= 1/ratio": Bir örnek / sahte oranı bir sahte / numune oranı dönüşüm için, formülü kullanın
7 Veri Analizi II:. Ek Çalışması için Yüksek Güven etkileşimler seçilmesi
- Açık Graphpad Prism, Yeni> Veri Tablo ve Grafik penceresinin sol taraftaki listeden ... Seç 'sütun' seçeneğini seçin ve Enter / İthalat verileri seçin> sütunlar seçenekleri içine yığılmış çoğaltma değerleri girin. Basın 'Oluştur'.
- Seçmek ve yeni Prism dosyasına Excel elektronik verilen bir günlük 2 örnek / Mock SILAC oranı sütun kopyalamak.
- Açılan 'Ekle' menüsünü tıklayın ve 'Yeni analizini' seçeneğini seçin. Sütun analiz altında 'seçeneğiniFrekans dağılımı '. Varsayılan seçenekleri tutulması 'Tamam' ı tıklatın.
Not: Bu adım, belirli bir oranda belirlenen protein sayısını gösteren bir çubuk grafik oluşturur. Bu, Gauss dağılımı oluşturmalıdır. - 'Sonuçlar' klasöründe yeni bir 'histogram' bölümünde oluşturulan edilmiş olacaktır. Frekans dağılımı bölümünü seçin.
- Açılan 'Ekle' menüsünü tıklayın ve Yeni analizi seçin> XY> Doğrusal olmayan regresyon (eğri). 'Gauss dağılımı' üzerine tıklayın ve Tamam'a tıklayın.
Not: Bu adım, daha sonra anlamlılık için eşik hesaplamak için kullanılabilir değerleri elde edildi, aşama 7.3 'de elde edilen frekans dağılımı verilere bir eğri uyar. - Görünen sonuç penceresinde, ortalama ve standart sapma verilmiştir. Ortalamaya 1.96 standart sapma ekleyerek bir eşik oluşturmak.
Not: Bu değerler ortalama ve Gauss dist standart sapmaları temsilmalara değil, toplam veri kümesi. 1.96 standart sapma% 95 güven sınırı (p ≤ 0.05) bir eşik yer olacaktır. 2.58 SD% 99 ve 3.3 SD% 99.9 verecek. Bu değişiklik gibi eşik her kopya deney için ayrı ayrı belirlenmelidir.
8 Veri Analizi III:. Kopya Kümeleri birleştirme
Not: Tipik bir SILAC açılan deney ideal sonuçlar üzerinde herhangi bir medya etkisini kontrol etmek tekrarlar biri için açık orta ve ağır etiketli örnekleri ile, üç kez yapılmalıdır etkileşim ortakları belirlemek emin olmak için. Ideal olarak, bu birini temsil 'switched' ortam numunesi ile üç deneyler en az iki, etkileşim olarak gösteren bir proteini, yüksek bir güven etkileşimdir.
- , Excel dosyası geri dön 'Kombine' adında yeni bir sekme oluşturmak. Etiket sütunu A 'Katılım' ve o her gelen tüm üyelik numaraları kopyalamakBu tek bir sütuna f bireysel deneyler.
- 'Veri' sekmesini ve ardından 'Kaldır Çiftleri' seçeneğini seçiniz.
- SILAC oranları ve her deney değişkenlik oranı hem de protein adı / açıklama sütun için sütunlar oluşturur.
- Açıklama sekmesinde, her bir erişim numarası açıklamasını toplamak için vlookup formülü kullanabilirsiniz. Protein açıklamasında doldurmak için sütunun aşağı formülü sürükleyin. Vlookup formülü: "= DÜŞEYARA (erişim no, WheretoLook, ColumnsAccross, Yanlış)", burada:.! Erişim no X WheretoLook 'Deney sekmesi Adı' $ 2 $ olduğu satır numarası olmak $ aX olan: $ Y $ Z nerede Y ve Z elektronik tablo başvurulan verilerin sağ alt parça vardır. ColumnsAccross katılım num karşısında sütun sayısıber A sütununda istenen değer yatıyor.
Not: Katılım sayısı A sütununda olduğunu ve ilgi veri sütun E. yatıyor Örneğin, burada değeri (sütun A 1 olarak sayılır) 5 olurdu. Erişim numaraları aşama b'de havuzlandı gibi, vlookup bir deneyde tüm gerekli isimlerini elde olmayacağı muhtemeldir. Bu N / A döner nerede, tüm açıklamaları edinilen kadar sırayla her deneyde açıklamasını elde etmek için formülü değiştirin. - Kombine kümesi içinde etkileşen proteinleri vurgulamak için, tek tek her bir oran sütunu seçin ve> Koşullu Biçimlendirme> hücreleri vurgulayın kurallar, ardından Giriş sekmesinde Daha kurallarını tıklayın.
- Seç style 'Klasik'. 'Hücre değeri' içeren Format sadece hücreler, 'Büyüktür veya Eşit'. Kutu, 1.96 standart sapma değeri tip ve 'Tamam' ı tıklatın.
- Her olumlu etkileşim oranı değişkenliği değerlendirin. % Değişkeni çıkarılarak varsaiability bir eşik değerin altında 'hit' düşeceği, bu dikkatle tedavi edilmelidir.
Not: Bu arada, bir proteinlerin SILAC oranını oluşturan her bir peptit için tek tek oranları ne kadar tutarlı temsil eder ve bir yüzde olarak verilir. Bu oran değişkenlik dikkate alınır ve bir protein, eşiğin üstünde bir SILAC oranı verir, bu protein, bir isabet eder. Oran değişkenlik eşiğinin altında kalan bir aralık verirse, 'hit' bir kirletici temsil edebilir. - Sonuçlar sütunların her biri için bu işlemi tekrarlayın ve deneyler arasında karşılaştırma. Iki veya daha fazla deneylerde tespit vurgulanan proteinleri yüksek bir güven etkileşimi temsil eder.
Not: Farklı deneylerde bir protein farklı bir, hatta birden fazla erişim numaraları ile tespit edilmiştir olabilir, protein üyelik numaralarını atamak için kullanılan veritabanına bağlı olarak. Bu proteinler yanlışlıkla ihmal değil emin olmak için bu kontrol etmek önemlidir.
Sonuçlar
Tipik SILAC açılan bir deneyde tespit proteinleri (>% 90) büyük bir çoğunluğu kirleticiler gibi afinite matrisine spesifik olmayan bağlanma proteinleri temsil eder ve yıkama protokolleri sitoplazmik kirletici maddelerin büyük bir kısmını kaldırmak bile bu, Şekil 2B'de gösterilmiştir Bu tür GAPDH (Şekil 2A) elde edildi. Bununla birlikte, spesifik olmayan normal bir dağılım içinde bağlayıcı proteinlerin kümelenme özel olarak bu olarak ayırt edilmesi için ilgi konusu bir proteinine bağlanan proteinler, daha yüksek bir arka plan örnek / mock oranına sahip sağlar. Kirleticiler teorik 0 günlük 2 SILAC oranı kumelendi gerekir iken, bu mutlaka böyle değildir, bu bir örnek Şekil 3'te verilmiştir. Bunun olası nedenler üzerine eşitsiz hacimleri veya lizatının konsantrasyonlarını yükleme, hücrelerin kusurlu SILAC Etiketleme anti-GFP boncuklar, saflaştırılması ya da eşit olmayan boncuk karışımı sırasında kazara zararSaflaştırma prosedürü 3 sonunda numunelerin ing. Bununla birlikte, normal olarak varsayılarak verilerin dağıtılmış kirleticilerin ortalamasından bir standart sapma eşik göre incelendiğinde, verilerin merkezlenmesi ufak kaymalar sonuçların kalitesini etkilemez.
İki ilişkili protein arasındaki protein etkileşimi farklarını karşılaştırırken ilgi duyulan bir protein (ya da bağlı Transfeksiyon etkinliğindeki değişim, ya da protein veya mRNA içsel bir özelliği) bir saniyeden daha hücreler içinde daha yüksek düzeylerde üretilir burada, benzer bir durum ortaya çıkabilir . Bazı ifade değişimi (örneğin, Şekil 2A) Bu iki numune için SILAC oranını analiz tarafından düzeltilebilir. Bu örnekte, bu GFP-eIF4AI ve GFP-eIF4AII örnekleri olacaktır. Bölüm 7'de açıklandığı gibi 4AI/4AII SILAC oranını analiz ederek, bu kimin bağlayıcı izoformlarına arasında anlamlı farklılık proteinleri tespit etmek mümkündür.
Şekil 4'te class = "jove_content">, gerçekleştirilen çoğaltma deneyleri birinde tanımlanan eIF4A bağlayıcı protein bir temsilidir, bu protokol elde başlatma faktörü kompleksinin kapsama gösteren gösterilmiştir. En yüksek oranlar genellikle uzağa eIF4A-bağlanma sitesinden bir bölgede eIF4G bağlanan eIF4E, daha düşük oranlarda, eIF4A doğrudan bağlanan eIF4G ile gözlendi. Alt oranları eIF3 kompleks üyeleri için gözlendi. Ancak, bu açıkça deney tatmin edici eIF4AI ve II doğrudan ve dolaylı ortakları bağlayıcı tespit olduğunu göstermektedir. Yüksek dizi özdeşliği 6, ikinci Beklenebileceği gibi protein: protein etkileşimleri için büyük ölçüde bu deney sistemi 7, 8, iki izoform arasında muhafaza ortaya çıktı. Etkileşen proteinlerin bazı bir seçim veri formatını gösteren, Tablo 2'de verilmiştir.| Sütun Başlığı | Tanım |
| Katılım | Sırası için erişim numarası gösterir |
| Kapsama | Belirlenen peptidler kapsadığı protein dizisinin Oranı |
| ♯ PSM | Peptit spektral maç |
| Peptidler ♯ | Bir protein için tanımlanan benzersiz peptitlerin sayısı |
| ♯ AAs | Amino asitleri, bir proteinin uzunluğu |
| MW (Da) | Dalton bir proteinin molekül ağırlığı. Değişiklikleri içermez |
| hesaplanan. PI | Bir proteinin teorik izoelektrik noktası |
| Skor | Indiv toplamını temsil eden bir proteinin (toplam puanidual peptid puanları). Anlamlılık için gerekli tam puan deneyler arasında değişir. Bir MS tesis genellikle% 5 yanlış keşif oranı cutoff geçerli olacaktır. |
| Sekans | Proteini oluşturan amino asitlerin sekansı, |
| Oran | Ikinci bir işaretli örnek göre adlandırılmış işaretlenmiş numunede peptidlerin nispi yoğunluk, |
| Oranı Sayısı | Belirli bir protein oranını hesaplamak için kullanılan peptid oranları sayısı |
| Oranı değişkenlik (%) | Belirli bir protein oranını hesaplamak için kullanılan tek tek peptid oranlarının değişkenliğinin |
| Tanım | Proteinin adı |
Tablo 1. Bir Proteom Discoverer rapordan Standart sütun başlıkları.Yararlı bilgiler bu sütunların her elde edilebilir iken, bu analiz için kritik olanlar kalın olarak gösterilmiştir.
| Katılım | Peptidler | 4AI/Mock | 4AII/Mock | Isim | SILAC analizi |
| A8K7F6 | 21 | 100 | 1 | eIF4AI | 'Bait' proteini |
| Q14240 | 22 | 0.01 | 90,855 | eIF4AII | |
| G5E9S1 | 25 | 47.575 | 30.53 | eIF4GI | Etkileşen proteinleri |
| Q59GJ0 | 5 | 11.778 | 10.619 | eIF4GII60; | |
| P06730 | 3 | 7.22 | 7.57 | eIF4E | |
| Q5T6W5 | 4 | 0.685 | 0.646 | hnRNPK | Non-spesifik bağlanma kirletici |
| P62805 | 1 | 0.531 | 0,498 | Histon H4 | |
| H6VRG2 | 18 | - | - | Keratin-1 | Çevresel kirleticiler |
| P35527 | 11 | 0.01 | 0.01 | Keratin-1 sitoskeletal 9 |
Tablo 2. Bir SILAC immunopresipitasyon deney Tipik Veri. Faiz / yem bir protein için örnek verileri verilmesi (yüksek peptitler, yüksek oran), faiz (yüksek / düşük peptidler, yüksek oranlı) bir protein ile etkileşen proteinler, non-spesifik bağlayıcı proteinler (yüksek / düşük Pept) Bu deneyde 0.96 olarak ve çevresel kirleticiler (genellikle yüksek peptitler, eşiğin altında negatif oranı /) - ides, oran cutoff'a altına düşer.

Şekil 1. Deneysel Plan. İlk olarak hücreler, 2 hafta içinde arginin ve lisin yoksun ortam içinde büyütüldü ve izotop işaretli arginin ve lisin ile ikame edilir (1). (2) Hücreler 10 cm2 tabaklar içine ekildi ve geçici olarak (Örnekler) (Mock) veya GFP füzyon proteinlerinin GFP'yi kodlayan plazmid ile transfekte edilir. (3) Hücreler lizlenir ve GFP ya da GFP füzyon proteinleri, hücre lisatlanndan imüno-çökeltilir. (4) Numuneler 1:1 oranında bir araya getirilmiş ve LC-MS/MS analizine tabi tutuldu. Veriler daha sonra düşük güven protein idendi kaldırmak için analiz edilirifications ve hakiki etkileşen proteinlere karşılık gelen protein zenginleştirme düzeyini seçin.
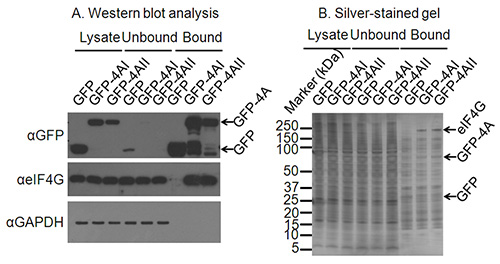
Şekil 2.. Uygun immunopresipitasyon koşulları Onaylanması. A) hücre lizatlarının Western blot analizi, hem de immünopresipitasyon gelen bağlanmamış ve bağlanmış parçacıklar ifade ve ilgilenilen protein imüno doğrulamaktadır. GAPDH karşı bir western blot, eIF4A bir bilinen etkileşim ortak ilgi konusu bir proteinin etkileşim eşleri bilinmemektedir ilgi. B'nin protein) bağlanan proteinlerin başarılı imüno teyit olmayan etkileşen protein ve başka bir western blot tükenmesi teyit bir gümüş lekeli jel etkileşen proteinlerin imunopresipitasyon teyit edebilir. Bu gümüş leke GFP ve GFP-eIF4AI/II için ed jel bantlar açık ve eIF4G için doğru boyutta göç bir bant sadece GFP-4AI/II-bound şeritlerde değil GFP kontrol şeritte mevcuttur.

Şekil 3,. Örnek sonuçlanır. Histogram (A) GFP-eIF4AI ya da (B) GFP-eIF4AII pulldown bir tekrar protein oranlarının dağılımını gösteriyor. 1.96 standart sapma kesim kesik bir çizgi ile işaretlenir. Normalde dağıtılmış kirletici maddelerin dışında kalan Etkileşimde bulunan proteinler, (B) 'de ~ 0.25 ve 1 (A) ve ~ 0.3-1.5 açıkça görülmektedir.
les/ftp_upload/51656/51656fig4.jpg "/>
Şekil 4. Bir SILAC IP deneyde tek bir kopyadan eiF karmaşık üyelerinin belirlenmesi. Proteinler yeşil proteinleri etkileşimde pulldown için kullanılan faiz protein olan kırmızı SILAC IP 2 SILAC oranını oturum beyaz göre kırmızı gölgeli edilir Analizde en bol protein olan ve beyaz 1.96 SD kesme olmak. Gri gölgeli Proteinler bu analizde tespit edilmemiştir.
Tartışmalar
Burada açıklanan SILAC açılan bir strateji, yeni proteini tespit etmek için bir çok hassas ve güçlü bir mekanizma: protein etkileşimleri ve bundan başka ilgi yakından ilgili örnekler arasında değişmiş bağlanma kalıplarının hızlı ve basit bir ayrım sağlar. EIF4AI ve eIF4AII proteinlerin protein etkileşimleri 6: Bu örnekte, bu teknik proteini incelemek için kullanılmıştır. Yazarın bilgisine, bu eIF4A bu iki izoformlannın hücresel interactome araştırmak için SILAC proteomiks programını istismar literatürdeki ilk çalışmadır.
Yukarıda tarif edildiği gibi bir yaklaşım, GFP-tag ve anti-GFP boncuk 9, 10 ve bu nedenle modifikasyonlar etiket N-ya da yer olup olmadığını, örneğin ilgi özel bir protein için, kullanılmak üzere, bu yaklaşım sağlamak için gerekli olabilir kullanır Bir proteinin C-terminus. Mümkün olan yerlerde, western blotlar ya da işlevsel tahlilleri gerektiğiBilinen bir protein etkileşim ortağının bağlayıcı tespit etmek için gerçekleştirilebilir. Bir protein, bir GFP etiketi, diğer etiketleme veya açılan stratejileri ile füzyon tahammül gereken diğer etiketler her ikisini de kullanarak SILAC Pulldowns uygulanmıştır (FLAG 11, 12 Biotin, STREP (kendi veri, yayınlanmamış)) ya da bir proteine karşı antikorlar kullanılarak, birincil ilgi konusu hedef proteinin siRNA portatif bir kontrol numunesi 13 içerir burada. Bu tür deneyler, literatürde başka yerlerde tarif edilmiştir, ama kısa olarak, basamak 1 ve adım 5,4-8 tarif edildiği gibi eşit protein girdileri ile tercih edilen bir ekspresyon / açılan sistemi için uygun şekilde modifiye adımları 2-5,3, yukarıda tanımlandığı gibi uygulanmış olur 2,4-2,5 adımları. Aşamaları miktar non-spesifik bağlanma proteinleri analiz düzeyinde çıkartılmasına olanak sağlamak üzere, düşük-protein afinite korumak için kontrol boncuk veya yüksek tuzlu yıkama ile ön-inkübasyon devre dışı bırakmak için önerilir: ilgi konusu bir protein ile bir protein etkileşimleri . Bir nukleaz may dahil veya belirli bir deney özelliklerine göre bu protokol atlanabilir. Örnek: Bu yöntemde kullanılan proteinler RNA helikazlar gibi, RNaz kokteyl RNA (adım 3.2) aracılık ettiği dolaylı etkileşimleri kaldırmak için protokol dahil edilmiştir. Bazı durumlarda bununla birlikte, nükleik asit bağımlı etkileşimlerini tespit etmek nükleazı ile ve olmadan paralel deneyler çalıştırmasını fayda var olabilir.
Bu protokol dahilinde, "orta" ve tekrarlanan deneylerde "ağır" örnekleri arasında sviçleme SILAC ortam veya hücre büyümesi farklılıklardan ortaya varyasyon kontrol etmek için önerilmektedir. Bir alternatif kontrol çoğaltma deneylerinde her üç ('hafif', 'orta' ve 'ağır') medya sıralı anahtarlama içerir. Bu yaklaşım, potansiyel olarak daha sıkı iken, en az bir tekrarında, ilgi konusu bir protein w olarak, analiz karmaşıklığını artırmak etmezhasta 'hafif' etiketli hücrelerinde üretilen ve bu nedenle sürekli olarak 'hafif' örneklerinde (örneğin keratinlerden gibi çevresel kirleticiler) ve bir proteine bağlı yalnızca 'hafif' örneklerinde zenginleştirilmiş olanlar tespit proteinler ayırt etmek gerekir faiz.
Nicel veri kullanımı sağlar iken bir eşik kullanımı yoluyla spesifik-olmayan-spesifik etkileşimleri ayrımcılık, kaçınılmaz olarak bazı gerçek etkileşimler atılabilir. Yukarıdaki yaklaşım protein belirlenmesi için basit ve hızlı bir yaklaşımdır: kolayca hiçbir önceki kütle spektrometresi ile deneyim, veya büyük protein analizi ile araştırmacılar tarafından teşebbüs edilebilir protein etkileşimleri: protein etkileşim veri setleri. Birçok kullanım için bu ilgi yeni proteinleri tanımlamak için fazlasıyla yeterli. Bu veri kaybını azaltmak için bu yaklaşım daha fazla başka modifikasyonlar literatürde tarif edilen ve bir PR kullanımını içerirDeneysel parametreler (hücre hattı, kordon matris, tampon koşulları kullanılarak) belirli bir kümesi için kirletici madde proteinlerinin bilinen otein frekans kütüphane 10, 14, iptal edilebilir. Bununla birlikte, özel deneysel parametrelere bağlı olarak bir boncuk proteomesini oluşturmak için kontrol deneyleri bir dizi çalıştırmak için gerekli olabilir ve bu nedenle, bu denemenin masraf ve karmaşıklığını hem de artırabilir. Bu teknikle ilgili ayrıntılı bilgiler www.peptracker.co.uk web sitesinden 14 edinilebilir.
Ayrıca protokol (- MAP SILAC deney saflaştırma sonra bir karıştırıcı olarak adlandırılır) immünopresipitasyon işleminin sonunda farklı işaretlenmiş numune karıştırma içerir, yukarıda tarif unutulmamalıdır. Bu protein olarak yapılır: protein etkileşimleri, belirli bir denge 15 meydana gelir. Bu, diğer gruplar örneklerin inkübe bu protokolde tarif edilen bu MAP SILAC yaklaşımı kombine unutulmamalıdırönce aşağı çekme etkisi (Karışım sonra saflaştırma - PAM SILAC) için, farklı zaman süreleri için 15, 16 (2 saat için 20 dakika literatürde kullanılmıştır). Bir protein oranı 1:1 'e doğru düşmektedir ne kadar hızlı göre, bu nitelik bağlanma afinitelerinin saptanması ve stabil veya dinamik etkileşen proteinlere 15 gibi proteinleri tanımlamak mümkündür.
Özet olarak, SILAC Pulldowns fizyolojik olarak uygun bir ortamda, ilgi konusu bir protein ile etkileşen proteinlerin belirlenmesi için bir çok güçlü bir aracı temsil eder. Bu teknik, çok kolay bir şekilde ilgi duyulan herhangi bir protein uygulanmasını sağlayan, farklı saflaştırma bir dizi strateji adapte edilebilir. Sonuç miktarının büyük ölçüde gerçek etkileşim tanımlanmasını kolaylaştırır, ve spesifik olmayan bağlayıcı kaldırmak için kullanılan tampon sıkı koşullar gevşemesine izin verir ve bu nedenle düşük afiniteli etkileşimler korur. En fazla üç numune, yukarıda karşılaştırılabilir gibistrateji, bu teknik, farklı protein izoformları, mutant proteinler, ya da farmakolojik inhibitörlerinin etkisi arasında farklılıklar bağlama proteini karşılaştırma açık güçlü sahiptir. Bütün jel dilimleri yerine bu Coomassil leke bireysel gruplarından daha incelendiğinde gibi, yüksek güven de tanımlanan proteinlerin numaraları genelde standart bir GST / TAP-pulldown belirlenen daha yüksektir, ve ilgi proteinleri ortadan kaldırılır seçiminde önyargı deneyci. Teknik, bu yüzden yeni protein-etkileşimleri (maya 2-hibrid, GST / His veya TAP PULLDOWNS) tanımlanmasında kullanılan diğer yaygın olarak kullanılan teknikler ile çok olumlu karşılaştırır.
Açıklamalar
Yazarlar, hiçbir rakip mali çıkarlarını olmadığını beyan ederim.
Teşekkürler
Bu çalışma IG Wellcome Trust ve BBSRC hibe tarafından desteklenmiştir. IG Wellcome Senior Fellow.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
Referanslar
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır