Method Article
SILAC免疫沈降定量的プロテオミクスを用いて哺乳動物細胞におけるタンパク質相互作用パートナーの同定
要約
タンパク質相互作用:SILACの免疫沈降実験は、新規タンパク質を発見するための強力な手段を表す。対照および試験試料の両方におけるタンパク質存在量の正確な相対的定量を可能にすることによって、真の相互作用は、実験的な汚染物質から容易に区別することができ、低親和性相互作用は、あまりストリンジェントな緩衝液条件を使用することによって保存した。
要約
タンパク質相互作用:免疫親和性精製、SILAC免疫沈降と組み合わせる定量的プロテオミクスは、新規タンパク質の発見のための強力な手段を表す。対照および試験試料の両方におけるタンパク質存在量の正確な相対的定量を可能にすることによって、真の相互作用は、実験的な汚染物質から容易に区別することができる。低親和性相互作用は、あまりストリンジェントな緩衝液条件を使用することによって保存され、容易に識別残ることができる。このプロトコルは、安定同位体標識アミノ酸で組織培養細胞の標識について説明し、親和性のトランスフェクションおよび免疫沈降は、質量分析施設に提出する準備に続く目的のタンパク質を、タグ付けされた。このプロトコルは、その後、目的のタンパク質と相互作用し、携帯パートナーを同定するために、質量分析装置から返されたデータを分析し、解釈する方法について説明します。一例として、この技術はiDENのに適用されるeIF4AIととeIF4AII:真核生物翻訳開始因子に結合するタンパク質をtify。
概要
タンパク質の機能を理解する上で重要なステップは、関連する相互作用するタンパク質の同定である。このようなタンパク質が知られていない場合には、利用可能な技術、自分の長所と欠点を持つそれぞれがいくつかあります。これらには、組換えタンパク質、ならびにタンデムアフィニティー精製またはTAP-タグ付け1,2を用いて、酵母ツーハイブリッドシステム、プルダウンアッセイが含まれる。
これらの技術のより最近の添加は、細胞培養(SILAC)3におけるアミノ酸の安定同位体標識を用いた定量的質量分析に続いて、関連する哺乳動物細胞株から目的のタンパク質のアフィニティー精製の 組み合わせである。これは、そのセルのローカライズおよび翻訳後修飾における酵母ツーハイブリッド手法に比べて利点が乱されていないしていだけでなく、伝統的に勝る利点それはREAにユーザーを許可する量的ではなく質的なアプローチであることをTAPは、タグ付けdily特異的に結合する宿主因子から、非特異的に相互作用するタンパク質と汚染物質を区別します。試料は、典型的には、全体ではなく、個々のタンパク質バンドとして分析されるように、さらに、目的のタンパク質は、同様に、ゲル上にタンパク質を移行することによって、マスクされていない、また彼らは、典型的には、染色後に表示されるように十分なレベルで存在する必要はない、につながる自信を持って同定されたタンパク質4の数を増加させた。
この手法を実証するために、密接に関連した真核生物の翻訳開始因子とeIF4AII eIF4AIとGFPの融合は、そのシェアの90%以上のアミノ酸同一性は、免疫沈降SILAC定量的プロテオミクスにより調べた。ヒトeIF4AIとIIとはGFPがeIF4AはのN末端に融合する融合タンパク質を形成するためのpEGFP-C1にクローニングした。一過性のトランスフェクションは、安定同位体標識された293T細胞へのこれらの構築物を送達するために使用した安定な細胞株を生成するための必要性を回避する。
細胞を、最初に、目的のタンパク質をコードするプラスミドDNAのトランスフェクションに続いてSILAC細胞培養培地中で2週間のために標識した。次いで、細胞を、溶解し、タンパク質濃度を正規化し、溶解物の親和性の等量は、抗GFPアガロースで精製した。溶出液の等量混合し、次いでLC-MS/MS分析に供した。この分析の結果は、高い信頼性タンパク質を同定するために処理される:タンパク質相互作用( 図1)。
SILAC免疫沈降は、直接相互作用だけでなく、低親和性またはタンパク質複合体4との間接的な相互作用だけではないの同定を可能にする。このシステムを使用して、eIF4AIとおよびII免疫沈降は、一次的な結合パートナーのeIF4G(アイソフォームI / IIおよびIII)5だけでなく、のeIF4Eとの間接的な相互作用、およびeIF3複合体の多数の部品の再現性があり、自信を持って同定を可能にした。
プロトコル
1。発生とSILAC標識された細胞株の継代
注意:トリプシンサンプルの不完全なラベリングにつながる標識されていないアミノ酸を含有することができるようにトリプシン-EDTAの使用は、分析のための実験サンプルの継代と準備のすべての段階で避けなければならない。
- SILACメディアのボトルを準備する(含むアルギニン/リジンを含まないDMEMの500ミリリットルボトルに適切にSILACで標識したアルギニン(84ミリグラム/ PBS中)とリジン(PBS中146 mg / ml)を0.5mlのアリコートを追加L-グルタミン)。
- 次に、500ミリリットルボトルに透析FBSを50ミリリットルおよびペニシリン/ストレプトマイシンの5ミリリットルを追加。
注:HEK 293T細胞(ATCC)を欠いているDMEM培地中で維持されるべきであるアルギニンおよびリシンおよび光(R0K0)を補充した培地(R6K4)または重鎖(R10K8)アミノ酸、透析ウシ胎児血清(10kDaのカットオフ)、およびペニシリン/ストレプトマイシン。細胞は、完全な保証するために5つの細胞分裂の最低培地中で維持されなければならないラベル。ほとんどの場合、細胞は容易≥2週間でラベル付けされている。 - 継代のために細胞を分割するには、細胞がフラスコを押すことで、単層から除去されるべきである。代替案は、セルスクレーパーまたは酵素を含まない、PBSベースの細胞解離緩衝液を使用して、使用することを含む。
注:メディアを節約するために、細胞をT25フラスコで継代されるべきであり、より大きな細胞数は、実験の直前に生成された。 - 細胞をトランスフェクションする前の24時間は、調査中の各実験条件に対して単一の10cm 2のディッシュに3.5×10 6 SILAC標識細胞を播種する。
pEGFPを融合構築物で標識された細胞の2。トランスフェクション
- 細胞から培地を除去し、抗生物質を含まないSILAC DMEM(ライト、ミディアムまたは重)メディアの9ミリリットルと交換してください。
注:これらは、リポソームベースのトランスフェクション試薬の効率を妨げる可能性があるため抗生物質を培地に添加してはならない。 - PREPA抗生物質を含まないSILAC DMEMを500μlの適当なプラスミド(epEGFP-C1、のpEGFP-eIF4Aは-I、のpEGFP-eIF4Aは-II)の10μgの混在Reおよび10を含む抗生物質を含まないSILACのDMEM500μlで混ぜるトランスフェクション試薬の液( 例えば 、リポフェクタミン2000)。上下に数回、それをピペッティングにより完全に反応を混ぜる。
- 20分間室温で反応混合物をインキュベートし、次いで滴下細胞単層を追加。左右に静かにプレートを揺する。
- 37℃でトランスフェクトされた細胞をインキュベートし、10%CO 2で24時間。
注:明らかな毒性の関心をもたらすタンパク質の発現場合、それはトランスフェクトされたプラスミドの量および/またはその後の発現期間の持続時間を減少させることが必要であり得る。
3。収穫細胞溶解物
- セルスクレーパーを用いて氷冷PBSに皿から細胞を回収する。 4、220×gでの遠心分離によって細胞を回収76; 5分間C。氷冷PBS 10mlに細胞をさらに3回洗浄します。
- 5μlの時(オプション)1×濃度およびRNaseカクテルで新たに追加されたプロテアーゼ阻害剤カクテルIIIを含有する細胞溶解緩衝液200μl(10mMのトリスCl / pH7.5で、150mMのNaCl、0.5mMのEDTA、0.5%NP40)中で細胞ペレットを再懸濁しml当たり。
注:既知の核酸とタンパク質が結合活性のためには、沈殿の前に溶解物にヌクレアーゼを追加する必要があり得る。ヌクレアーゼを添加する場合において、サンプルは、10分ごとにピペッティングして、氷上で30分間、氷上でインキュベートされるべきである。アガロースゲル電気泳動によって、試料と分析のごく一部から全核酸を抽出し、ヌクレアーゼの有効性をテストすることができます。 - 10分間13,000×gで、4℃で遠心分離試料は、可溶性の細胞溶解物として上清を保持します。
- 細胞溶解物の濃度は、製造業者の指示に従ってBCAアッセイによって評価されるべきである。
- 500μlの最終体積中のタンパク質濃度を標準化するために、プロテアーゼ阻害剤カクテルIIIを含有する溶解緩衝液を使用する。
- 希釈緩衝液500μlを加え、最終的な1X濃度でプロテアーゼ阻害剤カクテルIIIを含む(10 mMのトリスCl / PH 7.5、150mMのNaCl、0.5mMのEDTA)で1ミリリットルにボリュームを調整します。サンプルの50μlアリコートをサンプル入力として保持されるべきであり、溶解液( 例えば 、GFP-トラップ)抗GFPビーズを準備しながら、氷上に保持。
注:通常の収率は、最終的な1ミリリットル試料中のタンパク質の1〜3.5 mgの間で変動する。上記緩衝剤は、目的の多くのタンパク質に適している一方で、他のものに対しては、ベイトタンパク質を可溶化されていることを確認し、タンパク質 - タンパク質相互作用を維持するために緩衝液成分を変更する必要があり得る。可能な変化は、緩衝剤(リン酸、HEPES)、塩濃度(150〜500 mM)を、界面活性剤、または他の添加剤の選択が含まれる。
4。抗Gへの結合FPビーズ
- 簡単に言うとビーズを再懸濁し、ビーズスラリーをボルテックスする。最後に200μlのピペットチップを使用すると、切断し、新しいチューブにサンプルあたりビーズを25μlを移す。
注:ユーザーがサンプル間の変動を最小限に抑えるためにマスターミックスのような単一のSILAC実験にビーズを準備する必要があります。 - ビーズスラリーの各25μlのため、希釈緩衝液20容量(75μLスラリー1,500μl)を追加し、5分間2700×gで遠心ビーズ。次に、希釈緩衝液20容量でビーズをさらに2倍を洗う。
- 25μlのビーズスラリーあたり100μlの希釈緩衝液を追加します。最後に200μlのチップを使用することで、カットオフステップ3.6からSILAC標識されたサンプルの各々に、この再懸濁スラリー85μLを転送します。
- 2時間4℃で回転装置上でビーズでサンプルをインキュベートする。
5。洗浄、溶出、およびMS分析用サンプルの調製
- 2,700×gで遠心分離サンプル、46、5分間、C。上清50μlを捨て上清の残りは、未結合のサンプルとして保持する必要があります。
- 1ミリリットルの希釈緩衝液は、ビーズを再懸濁し、各チューブに添加されるべきであり、サンプルを5分間、4℃、2,700×gで遠心分離した。上清を捨てなければならない。これを2回実行する必要があります。
- 2×SDSローディング緩衝液50μlを添加することによってビーズからタンパク質を溶出し、10分間95℃で加熱する。 4℃で2分間、2,700×gでの遠心分離によりビーズをペレット
- 必要になるまで、それが次いで-80℃で保存することができるグリース封入管中の上清を保持する。質量分析施設に提出するため、標識サンプルを1時01分01秒( 例えば 、各10μlの)混ぜて混合試料を提出する。
注:この時点で、サンプルは、ウェスタンブロット、銀染色、または既知/未知の結合パートナーとの相互作用をテストするための他の手段によって試験することができる。例が図に示されているウェスタンブロットによるのeIF4G、および目的のタンパク質プルダウンからの試料中に存在する銀染色バンドの出現はなく、対照試料-既知の相互作用パートナーの特異的結合を示すウレア2。 - LC-MS/MS分析のためのサンプルを提出してください。
注:興味のある標識タンパク質が正常に相互作用パートナー、各標識された試料の等容量(ライト、ミディアムとヘビー)を合わせ、LC-MS/MS分析のために提出された結合されていることを一度に満足を。これは、分析のためのIPサンプルの30μlの合計を提出するのが一般的である。これは30μlの合計を与えるための媒体で標識し、10μl重標識サンプルを10μlの光で標識されたサンプルを10μlを混合伴うだろう。
6データ解析I:結果について、低信頼識別の除去
注意:プロテオームDiscovererのソフトウェアから返された列見出しの一覧を表1に示すDifを。ferentソフトウェア( 例えば 、MSQuant、MaxQuant)が異なる見出しが返されますが、これらのサブセットのみが分析に必要であり、これらは、様々なソフトウェアパッケージに共通している。データは常に特定された各タンパク質の受託番号、比率の比較、各サンプルの比(中VS光、重いなど対ヘビー、ミディアムVS光)が、ユニークなペプチドの数が特定され含まれており、偽陽性率や信頼性指標のいくつか形成すべきである。
- データを通過する前に、まず新しいスプレッドシートに生データをコピーします。このスプレッドシートからアクセス番号を与えるものを除くすべての列を削除し、独自のペプチドの数、サンプル、比率の変動、およびタンパク質の説明を比較する比率。レプリカ実験を行った場合、これらは別々のタブに表示される各実験では、単一のExcelファイルに結合する必要があります。
注:低信頼性データは、単一の固有のペプチドにより同定されたタンパク質を含み、それらの重量ここに定量化は不可能であった。 - ペプチドの数でデータを注文し、複数のペプチドを欠くタンパク質のエントリを削除するために優れてい '並べ替え」機能を使用してください。その後、比率でソートするとSILAC比(unquantifiedタンパク質)を欠くタンパク質を除去。
- 「SILAC比は'セル識別子に代入される' =ログ(SILAC比 2) '、:SILAC比はログ2の式を使用して値を変換します。
注:これはタンパク質を意味する0と1の間の値に制限される存在量の減少を示すタンパク質に対する任意のデータでSILAC比をもたらすように、それは、ログ2 SILAC比にSILAC比を変換するのが一般的であるに増加または減少の両方サンプルは、2または-2の4倍の増加または減少豊富に、および3または-3それぞれ存在量の9倍の増加または減少を表す。対数スケールで表されこの変換に続いて、データはGaのに合わせなければならない0のlog 2 SILAC比を中心ussian分布。その一例を図3に示す。 - Excelファイル内の新しい列を作成し、すべてのサンプル/モック列の2 SILAC率を記録し計算します。 「= 1/ratio ":サンプル/モック比モック/サンプル比の変換のために、式を使用
7データ解析II:さらなる研究のために精度の高い相互作用を選択する
- オープングラフパッドプリズム、ウィンドウの左側のリストから、[新規]> [データテーブルやグラフを選択... SELECT '列'、および入力/インポートデータを選択>レプリカ値を入力し、[列のオプションに積み重ねられた。を押して、「作成」。
- 新しいプリズムのファイルにExcelスプレッドシートから指定されたログ2サンプル/モックSILAC率欄を選択してコピーします。
- ドロップダウン「挿入」メニューをクリックし、「新しい分析]を選択します。列分析中」を選択します度数分布」。デフォルトのオプションを維持することは、「OK」をクリックします。
注:このステップは、指定された割合で同定されたタンパク質の数を示すヒストグラムを生成する。これはガウス分布を形成すべきである。 - 「結果」フォルダに新しい「ヒストグラム」セクションでは、生成されたでしょう。度数分布セクションを選択します。
- ドロップダウン「挿入」メニューをクリックし、新しい分析> XY>非線形回帰(カーブフィット)を選択します。 「ガウス分布」をクリックし、[OK]をクリックします。
注:このステップは、その後有意性の閾値を計算するために使用できる値が得られ、ステップ7.3で生成された頻度分布データに曲線をフィットする。 - 表示される結果ウィンドウでは、平均と標準偏差が与えられている。平均と1.96の標準偏差を追加することにより、しきい値を生成します。
注:これらの値は、平均値とガウスDISTの標準偏差を表すribution、合計ではなく、データセット。 1.96の標準偏差は95%信頼限界(0.05≤P)しきい値を配置します。 2.58 SDは99%、および3.3 SD 99.9%を与えるだろう。それは変更になる場合がありますように、しきい値は、それぞれのレプリカ実験のために、個別に決定されるべきである。
8データの分析III:レプリカデータセットのマージ
注意:一般的なSILACプルダウン実験は、理想的な結果上のメディアのいずれかの効果を制御するために、繰り返しのいずれかの切り替え中·重い標識サンプルを、3回行われるべきである相互作用パートナーを特定するのに自信を持つことができます。理想的にはこれらのいずれかを示す 'switched'メディアサンプルと、3つの実験の少なくとも二つで相互作用として現れるタンパク質は、高い信頼性の相互作用である。
- Excelファイルに戻り、「組み合わせる」という新しいタブを作成します。ラベル列A '受託'と各Oからすべてのアクセッション番号をコピーこの単一の列に、個々の実験のF。
- 「データ」タブを選択し、[オプション '重複削除」を。
- SILAC比率、及び各実験からの比の変動だけでなく、タンパク質名/説明]列のための列を作成します。
- [説明]タブでは、各アクセッション番号の説明を収集するためにVLOOKUPの数式を使用しています。タンパク質の説明を記入する列下に数式をドラッグします。 VLOOKUPの式は次のとおりです。 "= VLOOKUP(AccessionNo、WheretoLook、ColumnsAccross、FALSE)」、:。AccessionNo X は行数であると$ AXでWheretoLookは 「実験タブ名」である$ 2ドル:$ Y $ Zの場合、 Y、Zは、参照されているスプレッドシート上のデータの右下の部分です。ColumnsAccrossアク numの向かいの列の数であるBER A列に必要な値である。
注意:アクセッション番号は、列Aにあり、興味のあるデータをE列にある場合は、ここでは値は(列Aは1としてカウント)5になります。アクセッション番号は、ステップ(b)でプールしたように、VLOOKUPは、一つの実験から、必要なすべての名前が得られないという可能性が最も高い。それはN / Aが返された場合、すべての記述が取得されるまで、順番にそれぞれの実験から説明を取得する式を変更します。 - 組み合わせたデータセットの相互作用タンパク質強調表示するには、個々の比率の列を選択して>条件付き書式>ハイライトセルの規則に続く、[ホーム]タブの[詳細ルール]をクリックします。
- スタイル 'クラシック'を選択します。 「セルの値 'が含ま形式セルのみ、「より大きいか等しい」。ボックス、1.96標準偏差値を入力し、「OK」をクリックします。
- 各正の相互作用のための比率の変動を評価する。 %のVARを引いた場合iabilityが閾値以下「ヒット」をドロップします、これは慎重に扱われるべきである。
注意:これは一緒にタンパク質「SILAC比率を構成する各ペプチドの個々の比率はどのように一貫して示しており、割合として与えられる。比の変動が考慮され、タンパク質が閾値以上のSILAC比を与えると、このタンパク質はヒットを表す。比の変動がしきい値を下回る範囲を与えた場合は、「ヒット」は、汚染物質を表すことができる。 - 結果列ごとにこれを繰り返し、実験間で比較します。 2つ以上の実験で同定されたハイライトされたタンパク質は、高い信頼性の相互作用を表している。
注:タンパク質受託番号を割り当てるために使用されるデータベースに依存して、異なる実験では、異なるタンパク質、あるいは複数のアクセッション番号の下で同定されていてもよい。それは、タンパク質が誤って省略されていないことを確認するために、これを確認することが重要です。
結果
洗浄プロトコルは、細胞質汚染物質の大部分を除去する場合であっても、典型的なSILACプルダウン実験において、同定されたタンパク質(> 90%)の大部分は、汚染物質、ならびに親和性マトリックスに非特異的に結合するタンパク質を表し、これは図2Bに示されているこのような、GAPDH( 図2A)など。しかし、正規分布での非特異的に結合タンパク質のクラスタリングは、特にこれらが背景よりも高いサンプル/モック比を持っているとして区別するために、目的のタンパク質に結合するタンパク質を可能にします。汚染物質は、理論的に0のlog 2 SILAC比を中心にクラスター化する必要がありながら、これは必ずしもそうではない、この一例を図3に示す。これには考えられる理由の上に不均等なボリュームや溶解液の濃度をロードする、細胞の不完全なSILACラベルを含める抗GFPビーズ、精製または不均等なミックス中にビーズの不慮の損失精製手順3の端部に試料のING。しかし、想定したデータが正規分布し、汚染物質の平均値から標準偏差のしきい値に基づいて分析され、データのセンタリングのわずかなシフトは、結果の質に影響を与えるべきではない。
2つの関連するタンパク質間のタンパク質相互作用の差異を比較するとき有益な1つのタンパク質が、(トランスフェクション効率の変化、又はタンパク質またはmRNAの固有の特性に起因するいずれか)第よりも細胞内のより高いレベルに産生される場合、同様の状況が発生することが。いくつかの発現の変化( 例えば 、 図2A)は、これらの二つのサンプルのためのSILAC比を分析することによって補正することができる。この例では、これらは、GFP-eIF4AIとし、GFP-eIF4AIIサンプルになります。セクション7で説明したように4AI/4AII SILAC比を解析することにより、その結合アイソフォームとの間に有意に変化するタンパク質を同定することができる。
図4では= "jove_contentは">、行った実験のレプリカのいずれかで同定eIF4Aは結合タンパク質の表現は、このプロトコルが達成開始因子複合体の被覆率を示すが示されている。最高の比率は、一般的に離れてeIF4Aは結合部位から現場でのeIF4Gに結合するのeIF4E、より低い比率と、のeIF4Aに直接結合するのeIF4G、観察された。より低い比率は、eIF3複合体のメンバーについて観察された。しかし、これは明らかに実験を良好eIF4AIとIIとの直接的および間接的の両方の結合パートナーを同定することを示している。その高い配列同一6から予想されるように、タンパク質:タンパク質相互作用が大きく、この実験系7、図8には二つのアイソフォーム間で保存現れた。相互作用するタンパク質のいくつかの選択は、データ·フォーマットを示す、 表2に示す。| 列見出し | 説明 |
| 加盟 | シーケンスのアクセッション番号を表示 |
| カバレッジ | 同定されたペプチドでカバーされたタンパク質配列の割合 |
| PSM♯ | ペプチドのスペクトルが一致 |
| ♯ペプチド | ユニークなペプチドの総数は、タンパク質を同定し |
| ♯のAA | アミノ酸はタンパク質の長さ |
| MW(ダルトン) | ダルトンでのタンパク質の分子量。変更を除外します |
| CALC。 PI | タンパク質の理論上の等電点 |
| スコア | 個人ラージヒルの合計を表し、タンパク質(の合計得点idualペプチドスコア)。重要性のために必要な正確なスコアは、実験の間で変化する。 MS施設は、通常5%の偽発見率のカットオフが適用されます。 |
| シーケンス | タンパク質を構成するアミノ酸の配列 |
| 比 | 第二の標識されたサンプルと比較したという標識された試料中のペプチドの相対強度、 |
| 比カウント | 所与のタンパク質の比を計算するために使用したペプチド比の数 |
| 比率変動(%) | 所与のタンパク質の比を計算するために使用される個々のペプチドの比の変動 |
| 説明 | タンパク質の名前 |
表1。プロテオームDiscovererレポートから標準の列見出し。有用な情報は、これらの列のすべてから得ることができる一方で、この分析のための重要なものは太字で示しています。
| 加盟 | ペプチド | 4AI/Mock | 4AII/Mock | 名前 | SILAC分析 |
| A8K7F6 | 21 | 100 | 1 | eIF4AIと | 「ベイト」タンパク質 |
| Q14240 | 22 | 0.01 | 90.855 | eIF4AII | |
| G5E9S1 | 25 | 47.575 | 30.53 | eIF4GI | 相互作用するタンパク質 |
| Q59GJ0 | 5 | 11.778 | 10.619 | eIF4GII60; | |
| P06730 | 3 | 7.22 | 7.57 | のeIF4E | |
| Q5T6W5 | 4 | 0.685 | 0.646 | hnRNPK | 非特異的に結合汚染物質 |
| P62805 | 1 | 0.531 | 0.498 | ヒストンH4 | |
| H6VRG2 | 18 | - | - | ケラチン1 | 環境汚染物質 |
| P35527 | 11 | 0.01 | 0.01 | ケラチン1細胞骨格9 |
表2。SILACの免疫沈降実験からの典型的なデータ。タンパク質は、目的のタンパク質と相互作用し、関心/餌(高いペプチド、高比)のタンパク質のためのサンプルデータを与える(ハイ/ローペプチド、高比)、非特異的に結合タンパク質(高/低PEPT)この実験0.96で、環境汚染物質(しばしば高いペプチド、閾値を下回るネガティブ率/) - のIDE、比率がカットオフを下回った。

図1。実験計画。まず、細胞を2週間アルギニン、リジンを欠く培地中で増殖させ、安定同位体標識したアルギニン、リジンで置換されている(1)。 (2)細胞を、10cm 2の皿に播種し、一過性(試料)(モック)またはGFP融合タンパク質GFPをコードするプラスミドでトランスフェクトされる。 (3)細胞を溶解し、GFPまたはGFP融合タンパク質は、細胞溶解物から免疫沈降される。 (4)試料を1:1の比率で混合し、LC-MS/MS分析のために提出される。データは、次に低い信頼タンパク質IDENTを除去するために分析されるificationsと本物の相互作用するタンパク質に対応するタンパク質濃縮のレベルを選択する。
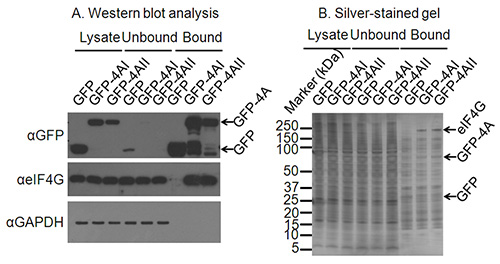
図2。に適した免疫沈降状態を確認。 A)免疫沈降からの細胞溶解物、並びに非結合および結合した画分のウェスタンブロット分析は、目的のタンパク質の発現および免疫沈降を確認する。 GAPDHに対するウェスタンブロットは、非相互作用タンパク質の枯渇、さらにウェスタンブロットのeIF4Aの既知の相互作用パートナーは、目的のタンパク質に結合するタンパク質の正常な免疫沈降を確認する確認する。B)、目的のタンパク質の相互作用パートナーは、知られていない場合銀染色ゲルは、相互作用するタンパク質の免疫沈降を確認することができる。この銀染色に GFP及びGFP-eIF4AI/IIのEDゲルバンドは明確であり、のeIF4Gの正しいサイズで移動するバンドのみGFP-4AI/II-boundレーンではなく、GFPコントロールレーンに存在している。

図3の代表的な結果。ヒストグラム(A)GFP-又はeIF4AIと(B)GFP-eIF4AIIプルダウンの一反復からのタンパク質の比率の分布を示す。 1.96標準偏差カットは破線でマークされている。正規分布の汚染物質から外れた相互作用タンパク質は、(B)中の0.25〜1(A)において、および約0.3〜1.5から明らかである。
les/ftp_upload/51656/51656fig4.jpg "/>
図4。SILACのIP実験の一つのレプリカからのeIFの同定、複雑なメンバー。緑の中のタンパク質は、タンパク質相互作用のプルダウンに使用する目的のタンパク質は、赤とSILAC IPでは2 SILAC率を記録するように、白記載赤からシェーディングされた分析の中で最も豊富なタンパク質であり、白は1.96、SDカットオフさ。灰色の影付きタンパク質は、この分析で同定されなかった。
ディスカッション
ここで説明するSILACプルダウン戦略は、新規タンパク質を検出することが非常に敏感でパワフルな手段である:タンパク質相互作用を、さらに興味のある密接に関連し、サンプル間の変化した結合パターンの迅速かつ簡単な識別を可能にする。 eIF4AIとしeIF4AII 6タンパク質のタンパク質相互作用:この例では、この技術は、タンパク質を調べるために使用した。筆者の知る限り、これはeIF4Aは、これら二つのアイソフォームの細胞インタラクトームを調査するSILACプロテオミクスの有用性を悪用する文献で最初の研究である。
上述したようなアプローチは、GFP-タグおよび抗-GFPビーズ9を使用して、10及びそれ故に修飾はタグがN-もしくはに配置されているかどうかを、例えば、関心対象の特定のタンパク質に使用するこのアプローチを有効にするために必要とされ得るタンパク質のC末端。可能な場合には、ウエスタンブロットまたは機能アッセイすべき既知のタンパク質相互作用パートナーの結合を検出するために実行することができる。タンパク質は、GFPタグ、他のタグ付けまたはプルダウン戦略は両方他のタグを用いてSILACのプルダウンに適用された(FLAG 11、ビオチン12、STREP(自身のデータ、未発表))、又はタンパク質に対する一次抗体を用いて、との融合を許容してはならない興味の標的タンパク質のsiRNAノックダウンは、対照試料13を提供する場合。このような実験は、文献に他の場所に記載されているが、簡単に、ステップ1およびステップは5.4から8にはで説明したように等しいタンパク質の入力を使用して、選択した表現/プルダウンシステムに応じて変更したステップ2から5.3で、上記のように適用される2.4-2.5を繰り返します。定量化段階は非特異的結合タンパク質は、分析レベルで除去することを可能にするように、低親和性タンパク質を保持するために、対照ビーズ、又は高塩洗浄でプレインキュベーションを省略することが推奨される:関心対象のタンパク質とのタンパク質相互作用。ヌクレアーゼMAyが含まれているか、または特定の実験の詳細に応じて、このプロトコルを省略してもよい。次に例を示します。この方法で使用されるタンパク質はRNAヘリカーゼであるとして、RNアーゼカクテルは、RNA(ステップ3.2)を介して媒介間接相互作用を除去するために、プロトコルに含まれていた。場合によっては、しかし、核酸依存性相互作用を識別するために、ヌクレアーゼでとすることなく、平行実験を実行してそこから利益を得ることができた。
このプロトコルの中では、「中」、および反復実験での「重い」サンプルの切り替えは、SILACメディアまたは細胞増殖の違いによって導入変動を制御することが推奨されます。代替コントロールは、レプリカ実験のすべての3(「光」、「中」、および「重い」)メディアの順次切り替えを必要とする。このアプローチは、潜在的に、より厳格であるが、それは、少なくとも一つの複製、目的のタンパク質重量のように、分析の複雑さを増大しない病気「光」標識された細胞で産生されそしてそれは一貫して「光」のサンプル(たとえば、ケラチンなどの環境汚染物質)とのタンパク質に結合した場合にのみ、「光」のサンプルが濃縮されているもので同定されたタンパク質を区別する必要があるインタレスト。
定量データを用いながら、必然的にいくつかの本物の相互作用が破棄されてもよい、閾値の使用を介して特定の非特異的相互作用の区別が可能になる。タンパク質相互作用データセット:簡単に以前の質量分析と経験、または大規模なタンパク質の分析と研究者が試みることができるタンパク質の相互作用:上記のアプローチは、タンパク質を同定する簡単かつ迅速なアプローチです。ほとんどの用途では、これは、目的の新規なタンパク質を同定するために十分以上である。このデータ損失を削減するために、このアプローチのさらなる修飾は他の文献で記載され、広報の使用が含まれている実験パラメータ(細胞株、ビーズマトリックス、緩衝液条件)の特定のセットのための夾雑タンパク質を公知otein周波数ライブラリーは、10、14を除外してもよい。しかし、特定の実験パラメータに依存し、それはビードプロテオームを生成するために、対照実験の数を実行する必要があり、これは、したがって、実験の費用と複雑さの両方を増加させることができる。この手法の詳細については、www.peptracker.co.ukウェブサイト14から提供されています。
また、プロトコルは( - MAPのSILAC実験を精製した後、ミキシングと呼ばれる)免疫沈降プロセスの最後に異なって標識されたサンプルを混合することを含む前述のことに留意すべきである。これは、タンパク質として行われます。タンパク質相互作用は、指定された平衡15で発生。それは、他のグループはサンプルのインキュベーションで、このプロトコルで説明このマップSILACのアプローチを組み合わせていることに留意すべきである異なる長さの時間(2時間まで20分文献で 使用されている)15、16 -プルダウン(PAM SILACミキシングした後、精製して、)の前に。タンパク質比が1:1に向かって急速に低下する方法に基づいて、定性的に結合親和性を調査するために、安定したまたは動的な相互作用タンパク質15として蛋白質を定義することが可能である。
要約すると、SILACのプルダウンは、生理学的に適切な設定では、対象とする特定のタンパク質と相互作用するタンパク質を同定するのに非常に強力な手段を表している。技術は、非常に簡単に目的の任意の所定のタンパク質への応用を可能にする、異なる精製戦略の数に適合させることができる。結果の定量化は非常に本物の相互作用の同定を単純化し、非特異的結合剤を除去するために使用されるストリンジェントな緩衝条件の緩和を可能にし、従って低親和性相互作用を維持する。最大3つのサンプルを、上記に比較することができるように戦略は、技術が異なるタンパク質アイソフォーム、変異体タンパク質、または薬理学的阻害剤の効果の間の結合蛋白質の違いを比較することで明らかに強みを持っています。全体のゲルスライスではなく個々のバンドクマシーによって染色よりも分析されるように、高い信頼度で同定されたタンパク質の数は、典型的には、標準的なGST / TAP-プルダウンで同定されたものよりも高い、目的のタンパク質を選択する際にバイアスを実験者に除去される。技術は、したがって、新規タンパク質 - 相互作用(酵母2 - ハイブリッド、GST / HisまたはTAPプルダウン)の識別に使用される他の一般的に使用される技術では非常に好意的に比較します。
開示事項
著者は、彼らが競合する経済的利益を持っていないことを宣言します。
謝辞
この作品は、IGのウェルカムトラストとBBSRCからの補助金によって支えられている。 IGがウェルカムシニアフェローである。
資料
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
参考文献
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved