Method Article
הקמת שורות תאי גזע pluripotent האדם בעריכה-הגנום: ממיקוד לבידוד
In This Article
Summary
Genome editing of human pluripotent stem cells (hPSCs) can be done quickly and efficiently. Presented here is a robust experimental procedure to genetically engineer hPSCs as exemplified by editing the AAVS1 safe harbor locus to express EGFP and introduce antibiotic resistance.
Abstract
הגנום עריכה של תאי גזע אנושיים pluripotent (hPSCs) מספקת פלטפורמה גנטית מבוקרת ורלוונטית מבחינה קלינית שממנו ניתן להבין את התפתחות אדם ולחקור את הפתופיזיולוגיה של מחלה. על ידי שימוש בnucleases אתר ספציפי (SSNs) לעריכת הגנום, הגזירה המהירה של קווים החדשים hPSC מחסה שינויים גנטיים ספציפיים בהגדרה אחרת isogenic הופכת אפשרית. אצבע האבץ nucleases (ZFNs), nucleases שעתוק כמו activator-מפעיל (TALENs) וחוזר palindromic הקצר interspaced באופן קבוע התקבצו (CRISPR) / Cas9 הם SSNs הנפוץ ביותר. כל nucleases אלה לתפקד על ידי החדרת DNA הפסקת פעמיים תקועה באתר שצוין, ובכך לקדם עריכת גן מדויקת במוקד הגנומי. עריכת הגנום מדיטציה-SSN מנצלת שני מנגנונים של התא אנדוגני תיקון DNA, הסוף שאינו הומולוגי שהצטרף (NHEJ) ותיקון מכוון הומולוגיה (HDR), או להציג הכנסה / מוטציות מחיקה או altאה הגנום באמצעות תבנית תיקון הומולוגית באתר של הפסקת גדילים הכפולה. Electroporation של hPSCs הוא אמצעי יעיל של transfecting SSNs ותבניות תיקון המשלבים transgenes כגון כתבי ניאון וקלטות עמידות לאנטיביוטיקה. לאחר electroporation, אפשר לבודד רק את אלה ששולב hPSCs מבנה התיקון על ידי בחירה לעמידות לאנטיביוטיקה. מכאני הפרדת מושבות hPSC ומאשר שילוב ראוי באתר היעד דרך genotyping מאפשרת הבידוד של שורות תאים בצורה נכונה וממוקדות גנטיים הומוגני. תוקפו של פרוטוקול זה בא לידי הביטוי כאן על ידי שימוש בכל שלוש פלטפורמות SSN לשלב EGFP והתנגדות puromycin לבנות למוקד הנמל הבטוח AAVS1 בתאי גזע pluripotent אנושיים.
Introduction
טכנולוגיות עריכת הגנום במהירות מתפתחות לכלים סטנדרטיים לביולוגיה מולקולרית של תא 1. הנדסה גנטית של תאים האנושיים pluripotent גזע (hPSCs) היא עניין מיוחד כhPSCs מהווה מקור עצמי חידוש של תאים אנושיים עיקריים הגנטית בשלמותה. יכול להיות מובחן hPSCs לסוגי תאים שונים למידול מחלה או כמקור לטיפולי השתלה 2,3. הפגין כאן הוא פרוטוקול שמנצל שלושה סוגים שונים של nucleases אתר ספציפי (SSNs) בשיתוף עם מנגנוני תיקון DNA אנדוגני לשילוב ממוקד של מבנה כתב במוקד AAVS1. לאחר transfection של SSNs לhPSCs, אנו מדגימים כיצד לבודד אוכלוסיות תאי isogenic מחסה הכתב.
היכולת לתפעל את הגנום, במיוחד pluripotent גזע הגנום של תאים, SSNs באמצעות אינה תופעה חדשה ככלי של nucleases אצבע אבץ (ZFNs) וactivat השעתוקnucleases כמו-או מפעיל (TALENs) לעריכת גן הודגם לפני כמה שנות 4-10. עם זאת, עם כניסתו של CRISPR pyogenes ס / טכנולוגית Cas9 11-13, עריכת הגן הפכה לנגישה 14 נרחב. כל SSNs להציג הפסקת DNA גדילים כפולה (DSB) באתר היעד שצוין 1,4,5,11 שתוקן על ידי מנגנונים תאיים אנדוגני או באמצעות תיקון שאינו הומולוגי (NHEJ) מכוון שהצטרף סוף או הומולוגיה (HDR) 15. NHEJ הוא מועדת לטעויות ויכול להציג את מוטציות שינוי מסגרת ותוצאה הוא אובדן של תפקוד גן, בעוד HDR מאפשר להיות הציגו אלמנטי רומן דרך שיתוף transfection של תבנית תיקון עם SSN. בעוד עקרונות היסוד של תיקון DNA המאפשרים עריכת גן נחשבים במידה רבה את אותו הדבר עבור כל SSN, כמה הבדלים בין הפלטפורמות ניתן לציין. דה נובו עיצוב של ZFNs מאפשר גמישות וnuclease אופטימיזציה 16, עם זאת שימוש בבפומביספריות זמינות הרכבה וכלים הקרנה לעצב ZFNs פרט יכולים להיות זמן רב. ברגע שהמוקד הרצוי למיקוד בתיווך ZFN נקבע, יכולים להיות מתוכננים זוגות ZFN עם הכלי המקוון ZiFit 17. לאחר עיצוב, ZFNs ניתן להרכיב באופן מודולארי דרך כמה סיבובים של שיבוט פלסמיד 18. לחלופין, יש ZFNs רב זמין מסחרי,-תוקף לפני 19. nucleases האגדה גם יכול להיות מתוכנן תוך שימוש בכלים מקוונים ורכיבים זמינים בפומבי 17,20. לדוגמא, TALENs ניתן להרכיב במהירות מלוקים של חמש חזרות סיפור, דרך ההרכבה FLASH 21 או באמצעות PCR הרכבה שער זהב היררכי מבוסס 22. קלות עיצוב SSN ומהירות של הבנייה באמצעות CRISPR / Cas9 עשו הגנום עריכת כלי נגיש לציבור רחב. המיקוד בתיווך RNA מדריך הקצר של CRISPR / Cas9 גם מאפשר ריבוב של RNAs המדריך למקד כמה לוקוסים עם מבנה יחיד 14. העיצובשל Cas9 לעריכת גן דורש רק זיהוי של מוטיב protospacer סמוך (PAM; trinucleotide NGG לס pyrogenes Cas9) הפרוקסימלי למוקד היעד. על ידי הוספת oligonucleotide המתאים עד 20 הזוגות הבסיסים 5 'של PAM לפלסמיד px330 14, המבנה ניתן להרכיב בצעד שיבוט אחד. בנוסף לס pyogenes Cas9, Cas9 מנ meningitidis (NmCas9) שמכיר 5'-NNNNGATT-3 "(PAM) הוכח כדי לאפשר לגן-עריכה יעילה בhPSCs 23.
בנוסף להבדלים בקלות עיצוב SSN, לכל פלטפורמה מאפיינים ספציפיים. לדוגמא, ZFNs וTALENs לנצל תחום nuclease FokI, אשר מייצר 5 הסככה ארבעה נוקליאוטידים '24 בזמן שהוא חשב Cas9 לייצר בעיקר להקהות DSBs הסתיימה. ZFNs, TALENs, וCas9 שונה בstabilities החלבון שלהם, שיעור ב- off על DNA היעד, ומצב של סריקת ה- DNA, שכולן גולד באופן תיאורטי לגרום להבדלים קטנים בתוצאת 1 העריכה. בעוד שמחקרים נוספים יידרשו כדי להבין את ההשלכות של הבדלים אלה, אנו מתארים כאן פרוטוקול כי הוא חזק מאוד בכל שלוש פלטפורמות וניתן להשתמש בו כדי ליצור בקלות hPSCs המהונדסת גנטי.
ללא קשר לבחירת SSN, electroporation הוא הליך חזק כדי transfect SSNs ותבניות תיקון ההומולוגיה לhPSCs 25. מספר שרד מושבות לאחר בחירה לעמידות לאנטיביוטיקה תלויה בפרמטרי מוקד ספציפי ואסטרטגית העריכה (למשל, גודל של הכנס ומצב של בחירה מהונדס). הפרוטוקול המתואר כאן בדרך כלל תוצאות בכ 150-400 מושבות תא בודד נגזרות.
במוקד AAVS1 באמצעות פרוטוקול זה עריכה-הגן כבר בעבר בשימוש כדי להדגים את היעילות של SSNs 4,5. תבנית תיקון AAV-CAGGS-EGFP משתמשת str מלכודת גןategy להקנות עמידות לpuromycin באופן ספציפי מוקד. בקצרה, תבנית התיקון מכילה אתר acceptor אחוי במעלה הזרם של קלטת התנגדות puromycin promoterless. עם שילוב נכון לאינטרון הראשון של גן PPP1R12C במוקד AAVS1, קלטת ההתנגדות באה לידי ביטוי מאמרגנו של הגן בעריכה. החוסן של assay AAVS1 הספציפי הזה מאפשר לנו להשוות את היעילות של כל פלטפורמת SSN.
עריכת גן באמצעות SSNs היא חזקה נתנה את היכולת לשבש ו / או לשנות באופן תיאורטי כל גן. יישום אסטרטגיה זו לhPSCs מספק צדדיות כhPSCs יכול להיות מובחן בהמשך למספר רב של סוגי תאים אנושיים כגון נוירונים 26, hepatocytes 27, ושריר לב 28. בנוסף, השימוש בתאי גזע pluripotent מושרה נגזר מטופל מאפשר תיקון או הקדמה של מוטציות הגורמות למחלות ידועות ברקע גנטי מטופל ספציפי 29 , מספק פלטפורמה שממנו ניתן לחקור מנגנוני מחלה ותרופות מבחן שימוש בתאים של חולה 30. לסיכום, עריכת הגן בhPSCs היא גישה יעילה ותכליתית לחקירת הביולוגיה הבסיסית של פיתוח ומחלה 31 בני אדם.
Protocol
ההליכים המתוארים בכתב היד הזה היו נבדקו ואושרו על ידי תאי גזע האוניברסיטה ברקלי ועדת ביקורת מחקר.
1. הכינו בתאי גזע לעריכה
- לגדול ותאי גזע pluripotent האנושי התרבות (hPSCs) על צלחת 6 היטב המכילה 2.4 × 10 6 תאים / צלחת של פיברובלסטים העובריים של עכבר מומת-C mitomycin מתקני האכלה (MEF) גדלו על ג'לטין 32. לשמור hPSCs ב 3 מיליליטר של תקשורת בתאי גזע העוברית אנושית בכל טוב (מדיה hESC) ולגדול ב37 חממת ° C עם 3% O CO 2/5% 2.
הערה: ההצלחה של פרוטוקול זה, זה לא הכרחי כדי לשמור על hPSCs בחממה דל בחמצן; עם זאת, חשוב לציין כי hPSCs להתרבות מהר יותר בסביבת O 2 גבוהה, כך פעמים ומספרי תא צריכים להיות בהתאם. צריך גם לציין שיש לי hPSCs נשמר בחמצן נמוך שיעורים נמוכים יותר של התמיינות ספונטנית 33.- To להפוך את תקשורת 500 מיליליטר hESC לשלב 380 מיליליטר DMEM / F12, 75 מיליליטר סרום שור עוברי (FBS), 25 מיליליטר סרום בנוקאאוט החלפה (KSR). להוסיף גלוטמין (ריכוז סופי 1 מ"מ), 5 מיליליטר 100x חומצות אמינו שאינם חיוניות, 100 יחידות / מיליליטר פניצילין, סטרפטומיצין (P / S), פיברובלסטים Growth Factor הבסיסי (bFGF) (4 ng / ריכוז סופי מיליליטר), ו2- mercaptoethanol (5.5 מיקרומטר ריכוז סופי).
- שינוי תקשורת על ידי הסרת נפח שלם של תקשורת (3 מיליליטר) בעזרת פיפטה זכוכית ואבק. החלף עם תקשורת hESC 3 מיליליטר חמה בעזרת פיפטה סרולוגיות. תקשורת חוזרת לשנות כל יום עד hPSCs כ 50% ומחוברות (יום -1).
- יום אחד לפני המיקוד (יום -1), לשנות את תקשורת hESC, הסרת המדיה הישנה והוספת מדיה hESC חמה בתוספת 10 מיקרומטר Y-27632.
- גם ביום -1, להכין 01:59 6-גם צלחות או 10 סנטימטר של תאי תרופה עמידים MEF מזין מעכברי DR4 (2.4 × 10 6 תאים / צלחת) 34.
הערה: באופן כללי, 6 צלחות גם הן מודעהvantageous מעל 10 צלחות סנטימטר, כפי שהם להכיל יותר תקשורת. 6 צלחות גם גם להבטיח כי שיבוטים שונים מבארות שונות אינם תלויים. עם זאת, צלחת 10 סנטימטרים תאפשר קטיף קל יותר, בהתאם למיקרוסקופ זמין עבור תהליך זה.
2. תאי גזע עריכת pluripotent
- הכן את פתרונות transfection ידי pipetting 5 מיקרוגרם כל אחד מZFN 1 ו -2, Talen 1 ו -2, או 15 מיקרוגרם של CRISPR / פלסמיד קידוד px330 Cas9 (איור 1) לתוך צינור 1.5 מיליליטר. 30 פיפטה מיקרוגרם של פלסמיד התיקון לצינור זה גם כן. לבסוף, פיפטה 1x בופר פוספט (PBS) לתוך הצינור להביא את הנפח של עד 300 μl.
הערה: הכן פלסמידים כmidiprep (כל ערכה מתאימה) עם ריכוז מינימאלי של 300 ng / μl כדי לשמור על הנפח הכולל של פתרון transfection תחת 300 μl. אין צורך לבצע חילוץ פנול / כלורופורם, כדי linearize פלסמיד, או להשתמש endotoערכת הכנת פלסמיד החופשי שין. - הסר תקשורת מhPSCs בעזרת פיפטה זכוכית ואבק. פיפטה 2 מיליליטר החם 1x PBS לכל גם לשטוף את התאים.
- הסר את PBS באמצעות פיפטה מזכוכית וואקום מייד. הוסף 0.5 מיליליטר 0.25% פתרון טריפסין- EDTA ישירות על תאים בכל טוב. מקום בחממה (37 ° C / 5% CO 2/3% O 2) לכ -10 דקות. או עד שכבת מזין מתחילה להרים את הצלחת.
- הוסף 2 מיליליטר תקשורת חמה esWash (470 מיליליטר DMEM / F12, 25 מיליליטר FBS, 100 יחידות / P / S מיליליטר) היטב כל אחד כדי לעצור את תגובת טריפסין.
- ודא שתאים מזינים יורדים כמו סדין. פיפטה את התוכן של כל אחד גם לתוך צינור חרוטי 50 מיליליטר אחת, המשלבת את כל הבארות. תאי Triturate בעזרת פיפטה 10 מיליליטר סרולוגית. אין צורך לשבור את שכבת המזין; hPSCs יבוא משל שכבת המזין על ידי טחינה דקה עדינה. לא צריך להיות כ -15 מיליליטר של ההשעיה התא.
- הוסף 25 מיליליטר של תקשורת esWash לצינור להביא voluשלי עד 40 מיליליטר כולל. לאפשר נתחי מזין גדולים להתיישב בחלק התחתון של צינור במשך 1-2 דקות. הסר את supernatant (~ 38 מיליליטר) מהצינור בעזרת פיפטה והפקדה סרולוגיות לתוך צינור חרוטי 50 מיליליטר חדש.
- ספין למטה 5 דקות ב 190 x גרם. הסר את supernatant מהצינור בעזרת פיפטה זכוכית ואבק. הקפד שלא להפריע תא גלולה. Resuspend התאים 500 μl 1x PBS. לשלב עם פתרון transfection פלסמיד שהוכן קודם לכן. ספירת התאים בשלב זה. השתמש 5-10 מיליון תאים לכל electroporation.
- פיפטה ההשעיה 800 μl כולו לקובט electroporation 4 מ"מ, מניח על קרח במשך 3-5 דקות. תאי Electroporate באמצעות תכנית מעריכי במערכת electroporation עם הפרמטרים הבאים: 250 V, 500 μF, ∞ התנגדות, וגודל קובט 4 מ"מ. לאחר electroporation, מקום קובט חזרה על קרח במשך 3 דקות.
הערה: שים לב לזמן הקבוע של electroporation על מערכת electroporation. קבוע הזמןמשתנה עם מספר תאים וטוהר DNA. בדרך כלל יש לי transfections המוצלח זמן קבוע בין 10-14 msec בעת שימוש בתנאים המפורטים וPulser גן השני. יעילות electroporation נמוכה יכולה להתרחש כאשר קבועי זמן משתנים מהערכים הללו. - Resuspend תאי electroporated ב -18 מיליליטר hESC מדיה חמה בתוספת 10 מיקרומטר Y-27632. צלחת 3 מיליליטר של השעיה תא בודדת זה לבאר כל צלחת 6 היטב עם תאים מזינים DR4. חזור אל חממה (37 ° C / 5% CO 2/3 O% 2).
3. בחירה של מושבות חיוביות
- יום 2, להסיר את כל התקשורת באמצעות פיפטה זכוכית ואבק. החלף עם 3 מיליליטר של תקשורת hESC החמה בתוספת 10 מיקרומטר Y-27632. יום 3, להסיר את כל התקשורת באמצעות פיפטה זכוכית ואבק. החלף עם 3 מיליליטר של תקשורת hESC החמה ללא כולל Y-27632. יום 4, להסיר את כל התקשורת באמצעות פיפטה זכוכית ואבק. החלף עם 3 מיליליטר תקשורת hESC החמה בתוספת אנטיביוטיקה לselectiעַל. חזור אל חממה (37 ° C / 5% CO 2/3 O% 2).
הערה: הסוג של אנטיביוטיקה המשמשת יהיו תלויים בקלטת ההתנגדות כלולה בתבנית התיקון. כאשר עובד עם # WIBR 3 hPSCs (רישום NIH 0079), 0.5 מיקרוגרם / מיליליטר puromycin, 70 מיקרוגרם / מיליליטר G418 (geneticin), ו -35 מיקרוגרם / מיליליטר hygromycin שימש בהצלחה. ריכוזים לבחירה צריכים להיקבע באופן אמפירי על ידי הקמת הריכוז המינימלי של אנטיביוטיקה צריכה להרוג תאים פרא-סוג בתוך כשבוע. - ימים 5-12, לשנות תקשורת יומית, החלפת המדיה ישנה בכל פעם עם תקשורת hESC החמה בתוספת אנטיביוטיקה.
הערה: צפה לכמות גדולה של מוות של תאים. מושבות בודדות תתברר סביב היום 8-10. תקשורת hESC רגילה ללא אנטיביוטיקה יכולה לשמש לאחר 12-14 ימים של בחירה מתמדת. אם צפיפות תאים היא גבוהה או המוות של תאים הוא איטי, החמצה של התקשורת יש להימנע וייתכן שיהיה צורך להגדיל את volu התקשורתשלי (עד 4-5 מיליליטר) במהלך הימים הראשונים של בחירה.
4. מושבות נבחרות קטיף (יום 12-14)
- שים לב מושבות ביום 12-14. שים לב למושבות כי הם מוכנים לקחת על מיקרוסקופ לנתיחה ולוודא שהם אינם מכילים תאים שמתחילים להבחין. הגודל המשוער צריך להיות 800-1,200 מיקרומטר. אם מושבות להגיע לגודל הזה לפני היום 12, מומלץ כי הם צריכים להיות הרים אז.
הערה: כל ניסוי מיקוד, לבחור מושבות רבות כנדרש כדי להבטיח את הגנוטיפ הרצוי מבודד. כדוגמא, עריכת AAVS1 עם תבנית תיקון AAV-CAGGS-EGFP הראתה אינטגרציה חזקה באופן עקבי, ודורשת רק 12-24 מושבות להיות נטלו לקבל כ 5-10 שיבוטים ממוקדים heterozygously. ניסויי מיקוד אחרים עשויים לדרוש יותר מושבות להיות נטלו (טבלה 1). התדירות של אירועי מיקוד נכונים תלויה בגורמים כגון היעילות של האס-אסN להציג DSB, בגודל של הכנס, ואסטרטגית הבחירה. במקרה של גישת מלכודת הגן למוקד AAVS1 שהוצג כאן, סמן הבחירה מתבטא רק כאשר משולבים בצורה נכונה באתר היעד, צמצום מספר המושבות הנדרשות להשגת שיבוט ממוקד כראוי. - יום אחד לפני הבחירה, להכין 12 גם צלחות של MEFs (2.4 × 10 6 תאים / צלחת, גם אחד ישמשו לכל מושבה הרימה).
- ביום הקטיף, להסיר את כל התקשורת מהצלחות גם 12 MEF בעזרת פיפטה זכוכית ואבק ולהחליף עם 1 מיליליטר תקשורת hESC. גם ביום של קטיף, לשנות מדיה hESC על 6 הצלחות גם כי הם הולכים להיות הרים.
- משוך טפטפות זכוכית לניתוק מכאני של מושבות בודדות משיכבת המזין. מנמיכים את טפטפות מעל מבער בונזן ב- מכסה המנוע והתרכך בנקודת ההרחבה עד הזכוכית היא נזיל. להסיר במהירות מלהבה ולמשוך את פיפטה מלבד יצירת נקודה זוויתי. לשבור אתלהצביע כ 2 סנטימטר מהציר הכפוף, עוזב ערוץ צר. פולני סוף הערוץ על ידי חשיפה לאש במשך 1-2 שניות.
הערה: לחלופין לבחור מושבות קצה pipet P20, לעומת זאת, זה עשוי להפחית את clonality של התרבות, כמו הקצה המשעמם יכול לסלק כמויות גדולות יותר של תאים מכל מושבה לתקשורת, שעלול לזהם לקט שלאחר מכן. - להרכיב לקטוף מכשיר על ידי לקיחת קצה פיפטה מסנן P1000 ומצרף הנורה יניקה לסוף הצר. הכנס את פיפטה זכוכית משך בקצה הרחב.
- מניחים צלחת 6-גם של hPSCs להיות נטלה על הבמה של מיקרוסקופ לנתיחה רכוב במכסת מנוע בתרבית רקמה. לדחוס את הנורה של מכשיר הקטיף ובעדינות הבלו ולחתוך מושבה בודדת לתוך 10-20 חתיכות שווה בגודלן, נזהר שלא לשחרר חתיכות לתקשורת. קח חתיכות hPSC נכרת של המושבה לתוך פיפטה ידי שחרור הנורה. נסה לקחת תקשורת קטנה ככל האפשר תוך העברה.
- Transfאה המושבה הפרט גם אחד של צלחת MEF 12 גם, דחיסת הנורה שוב ישירות לתוך הבאר, משחררת את המושבה עכשיו נשברה. כל תווית היטב כדי לאפשר זיהוי ייחודי של שיבוטים נגזרים מתא בודד. חזור למושבות לפי צורך, לשנות את פיפטה הזכוכית בכל פעם.
הערה: מושבות בחירה עשויות לקחת קצת זמן כדי ללמוד. מומלץ שבפועל הניסוי בכמה תאי שליטה לפני שתנסה לבודד את המושבות ממוקדות. - חזור 12 צלחות גם בחממה (37 ° / 3% O CO 2/5% C 2), בעדינות הנדנדה הצלחת ראשונה, כדי למנוע הצטברות של תאים באמצע כל הטוב.
- למחרת ובכל יום שלאחר מכן, להסיר את הנפח מלא של מדיה באמצעות פיפטה זכוכית ואבק ולהחליף עם 1.5 מיליליטר של תקשורת hESC החמה עד תאים כ -50% ומחוברות (זה בדרך כלל לוקח 10-12 ימים).
- לאחר 10-12 ימים, לבחור 1-2 מושבות מכל טוב ולהעביר ל12-גם צלחות MEF החדשות מחדשpeating צעדים 4.1-4.8 כדי ליצור העתק צלחת.
- תמצית ה- DNA (ראה להלן) מהמושבות שנותרו בכל טוב של צלחות MEF המקוריות.
- הסר תקשורת מכל הבארות באמצעות פיפטה זכוכית ואבק. מיליליטר פיפטה 1 1x PBS על כל הטוב לשטוף תאים. הסר PBS באמצעות פיפטה זכוכית ואבק. פיפטה 250 μl של חיץ תמוגה תא (ריכוזים סופיים בH 2 O: 10 מ"מ טריס HCl, 5 מ"מ EDTA, 0.2% SDS, 200 מ"מ NaCl, 0.08 מ"ג / מיליליטר proteinase-K) על כל טוב. מקום בחממת O (37/3% CO 2/5% O ° C 2) / N.
- למחרת, תוכן פיפטה של כל אחד גם לתוך צינורות 1.5 מיליליטר פרט. פיפטה 250 μl של אלכוהול איזופרופיל לתוך צינור אחד ללהאיץ את ה- DNA. Shake צינור במרץ. משקע לבן צריך להיות גלוי.
- ספין צינור אחד למטה 3 דקות ב 13,000 סל"ד בצנטריפוגה בראש הטבלה. אחרי הספין למטה, להשליך supernatant באמצעות אחסון לפסולת נוזלית. גלולה DNA קטנה צריכה להישאר דבוקה לתחתיתהצינור. פיפטה 250 μl של 70% אתנול לצינור אחד לשטוף גלולה DNA. לנער במרץ. ספין צינור אחד למטה 3 דקות ב 13,000 סל"ד בצנטריפוגה בראש הטבלה.
- אחרי שצנח, להשליך supernatant באמצעות אחסון לפסולת נוזלית. גלולה DNA קטנה צריכה להישאר דבוקה לתחתית של התחתית. פיפטה את שארית supernatant כך שאין נוזל שנשאר בצינור. השאר צינור פתוח לייבוש על benchtop למשך 5-10 דקות. לאחר ייבוש, resuspend DNA במאגר TE 250 μl. מניחים על 37 מעלות צלזיוס במשך 6 שעות כדי לאפשר DNA לפזר.
- גנוטיפ כל דגימה באמצעות אסטרטגית כתם מראש מותאם PCR או דרומי.
הערה:.. לAAVS1 מיקוד באמצעות תבנית תיקון AAV-CAGGS-EGFP, ניתן למצוא את אסטרטגית כתם הדרום בHockemeyer et al, ניתן למצוא 2009 4 פרוטוקול סופג דרום מקיף בדרום, 2006 35 לזמנית genotyping PCR של. באותו הניסוי, יחל ותנאים יכולים להיותמצא בטבלה 2 36. - בטל בארות שאינם ממוקדות כראוי ולהמשיך שינוי מדיה hESC על תאים ממוקדים כראוי בכל יום. להקפיא את התאים כאשר הם כ -50% ומחוברות, ראה להלן להקפאת פרוטוקול.
- יום אחד לפני ההקפאה, לשנות מדיה hESC, הסרת המדיה הישנה והוספת מדיה hESC חמה בתוספת 10 מיקרומטר Y-27632.
- ביום של הקפאה להכין 0.5 מיליליטר כל אחד מהפתרון ופתרון B בכל טוב של צלחת 12 גם כי הוא קפוא מטה בשני 15 מיליליטר צינורות חרוטי. פתרונות מקום על קרח. פתרון: 50% תקשורת hESC ו -50% FBS; פתרון ב ': FBS 80% ו -20% דימתיל sulfoxide (DMSO).
- לנתק את מושבות hPSC לתוך תאים בודדים לפני ההקפאה. בצע את השלבים 2.2-2.8 לעשות את זה, קנה מידה כרכים בהתאם. תא גלולה באופן מלא גלול ב0.5 מיליליטר של א 'הפתרון
- הוסף 0.5 מיליליטר הפתרון B להשעיה תא, פיפטה למעלה ולמטה כדי להפוך את ההשעיה תא הומוגנית. טאקדואר הנפח הכולל של ההשעיה התא (~ 1 מיליליטר) והפקדה לcryotube 2 מיליליטר. בורג כובע בחוזקה.
- cryotube מקום במקפיא O -80 ° C / N. היום לאחר ההקפאה, להסיר תאים קפואים מהמקפיא -80 ° C ומניח מייד למכל חנקן נוזלי לאחסון לטווח ארוך.
תוצאות
כאן אנו מדגימים פרוטוקול תואם עם שלוש פלטפורמות SSN שונות כדי ליצור קווי hPSC מהונדסים גנטי. אנו ממוקדים # WIBR 3 תאי גזע עובריים אנושיים במוקד AAVS1 באמצעות ZFNs שפורסם בעבר 4, 5 וTALENs CRISPR / Cas9s 37 באמצעות תבנית תיקון שמציגה כתב EGFP וקלטת התנגדות puromycin 4.
אנו תרבית hPSCs על MEFs לעבודת תרבית תאים המאפשרת תחזוקה והרחבת hPSCs מובחן (איור 2), שהוא גם חסכוני וניתן להרחבה. אם תאים גדלים יותר מהנחוץ קיים סיכון המוגבר של בידול, צמצום מספר תאי pluripotent כי הם transfected וכתוצאה מכך מספר מושבות hPSC ממוקדות בצורה נכונה שהושגו.
אנו electropצטט 5.0 x 10 6 תאים לכל פלטפורמת SSN ומצופה בתאים מכל מיקוד על אחד צלחת גם 6 של MEFs DR4. לאחר בחירה, כל פלטפורמה הביאה מושבות EGFP-חיובי (איור 3) ושילוב של שיבוטים לא ממוקדים, ממוקדים homozygously וממוקד heterozygously (איור 4 א ', ב'; טבלה 3). בתנאים שהוצגו כאן, אנו מוצאים כי TALENs AAVS1 הביא השיבוטים EGFP החיוביים ביותר. התבנית המשמשת לתיקון ניסוי זה מורכב מאתר acceptor אחוי במעלה הזרם של קלטת הכתב והתנגדות puromycin EGFP. באמצעות אסטרטגיה זו "גן-מלכודת" (איור 1), המבנה צריך להכניס לתוך אינטרון הראשון של מוקד AAVS1, באמצעות אמרגן אנדוגני לנהוג ביטוי של קלטת puromycin ההתנגדות. חוסר אמרגן נהיגה הביטוי של גן התנגדות puromycin בתוך תבנית התיקון צריך למנוע ביטוי ב מקרה של שילוב אקראי.
לכן, אנו מצפים שכל השיבוטים puromycin עמיד יהיו ממוקדים באתר AAVS1. יש לציין כי קבוצת משנה של שיבוטים לבצע אינטגרציות חריגות במוקד AAVS1 שלזיהוי על ידי כתם דרום באמצעות בדיקה פנימית, אך לא על ידי רוב אסטרטגיות PCR (איור 1; איור 4C) 4. אירועי אינטגרציה אלה הם ככל הנראה התוצאה של אירועים שהובילו למיקוד Heterologous אינטגרציות מרובות של פלסמיד התורם 4. אספנו 24 מושבות מכל ניסוי SSN ומצאתי כי כל פלטפורמות יעילות מיקוד גבוהה מאוד והראו הבדלים מזעריים בלבד. כנחקר על ידי ה- PCR, CRISPR / Cas9 הניב שיבוטים הממוקדים ביותר בצורה הנכונה ואילו פלטפורמת Talen השיבוטים הממוקדים ביותר homozygously (לוח 3).
"Width =" 700 /files/ftp_upload/53583/53583fig1highres.jpg "/>
סכמטי באיור 1. של הגן ערך מוקד AAVS1 באמצעות תבנית תיקון AAV-CAGGS-EGFP. השתנה מHockemeyer et al., 2009. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
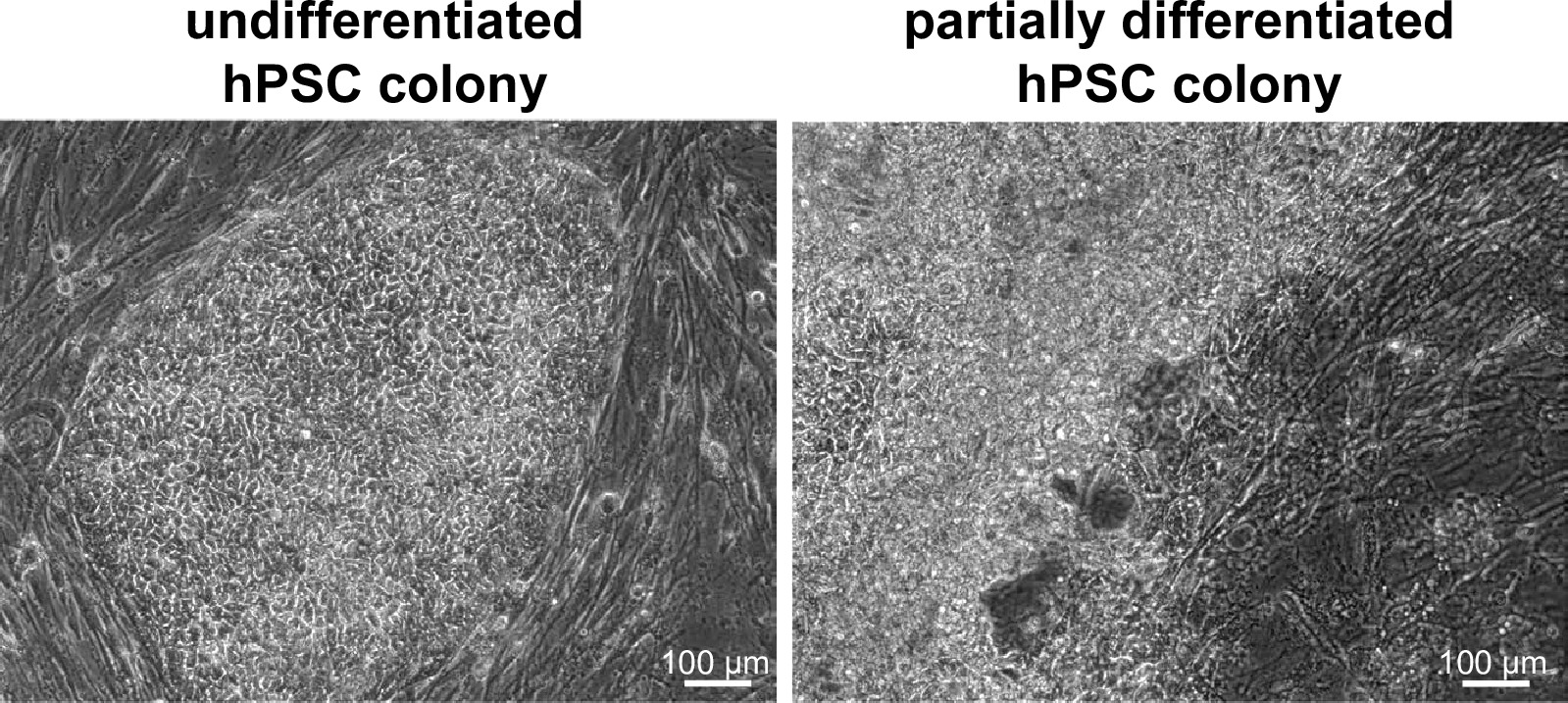
2. מושבות איור של # 3 תאי WIBR. נציג תמונות בהירות שדה של מושבות של # WIBR 3 תאי גזע עובריים אנושיים לפני המיקוד. שים לב לחוסר הבחנה וההפרדה ברורה משיכבת המזין במושבה אידיאלית (משמאל), בניגוד לאחד שאינו אידיאלי (מימין). אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
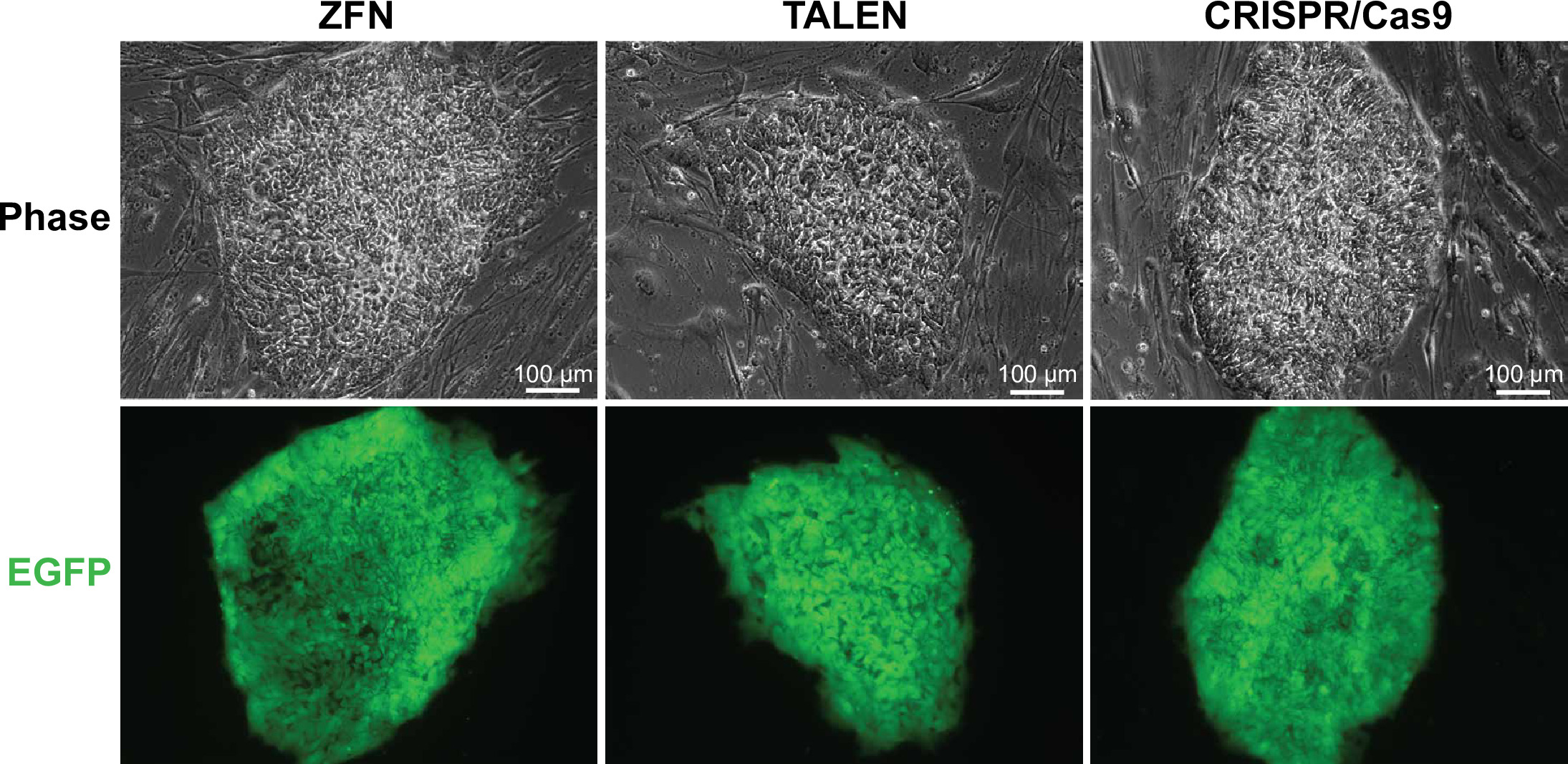
איור 3. # WIBR EGFP חיובי 3 תאים. נציג תמונות של # WIBR 3 תאים ממוקדים עם תבנית תיקון להביע EGFP במוקד AAVS1. תמונות של מושבות נציג נערכו עם ZFNs, TALENs וCRISPR / Cas9 מוצגים. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
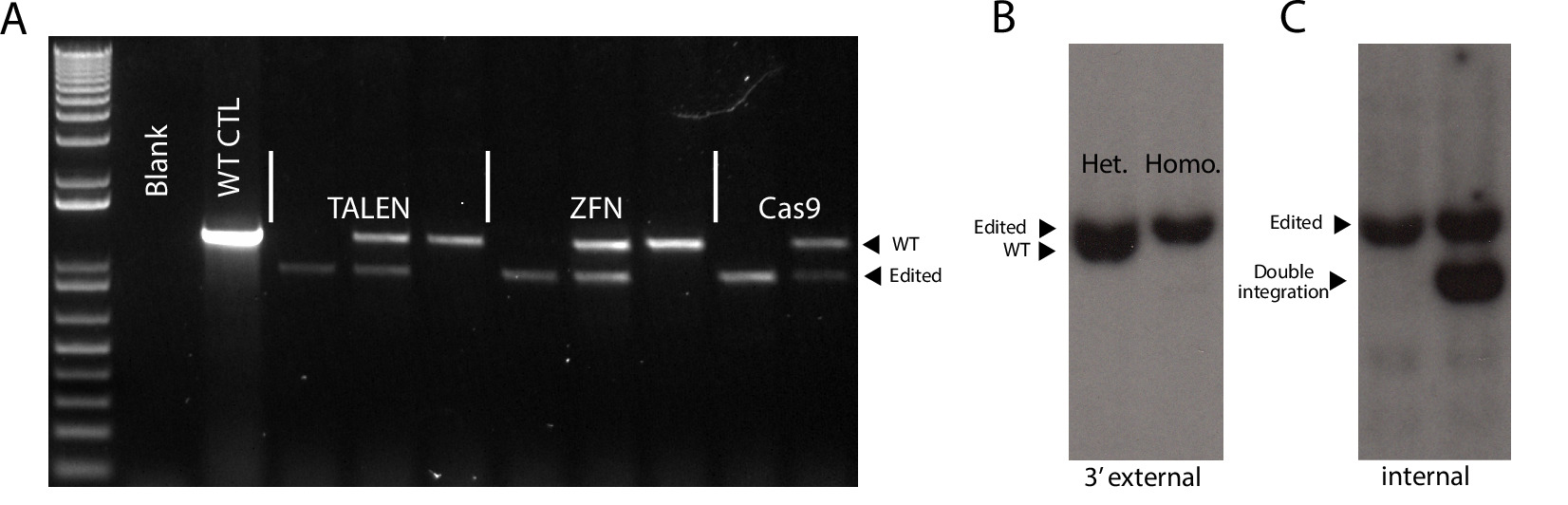
אסטרטגיות Genotyping איור 4. כדי לאשר מיקוד נכון. () תוצאות genotyping הנציג PCR מראים שיבוטים ממוקדים ממוקדים, הטרוזיגוטיים והומוזיגוטים על פני שלוש פלטפורמות SSN. WT CTL = שליטת wild-type. Repre (ב)תוצאות sentative דרום הכתם מראים שיבוט הטרוזיגוטיים ממוקדת ושיבוט הומוזיגוטים ממוקד מזוהה עם בדיקה החיצונית '3. גדלים שבר: WT-6.5 KB, בעריכת-6.9 kb. (ג) תוצאות כתם דרום נציג מראים שיבוט נערך כראוי ושיבוט הטרוזיגוטיים עם דאבל-אינטגרציה אינה אקראית. גדלים שבר:. עריכה-6.9 כראוי kb, kb אינטגרציה-5 נוסף הסוטה אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| לימוד | גן ממוקד | פּלַטפוֹרמָה | סוג תבנית תיקון | # של שיבוטים הרים | יעילות מיקוד |
| סקסטון et al., 2014 | TPP1 | ZFN | GFP-Puro | לא דווח | לא דווח |
| סקסטון et al., 2014 | טרט | ZFN | Hygromycin | לא דווח | לא דווח |
| Hockemeyer et al., 2009 | POU5F1 | ZFN | GFP-Puro | 31 | 39.0% |
| Hockemeyer et al., 2009 | PITX3 | ZFN | GFP-Puro | 74 | 14.9% |
| Hockemeyer et al., 2011 | POU5F1 | Talen | GFP-Puro | 68 | 91.0% |
| Hockemeyer et al., 2011 | PITX3 | Talen | GFP-Puro | 96 | 13.0% |
| מרקל et al., 2015 | VASA | Cas9 | כתב-Geneticin | 139 | 94.0% |
| מרקל et al. </ Em> 2015 | CRH | Cas9 | כתב-Geneticin | 30 | 93.0% |
| מרקל et al., 2015 | HCRT | Cas9 | כתב-Geneticin | 154 | 92.0% |
| מרקל et al., 2015 | HMX2 | Cas9 | כתב-Geneticin | 11 | 45.0% |
| פורסטר et al., 2014 | LRG5-Nterm | ZFN | GFP-Puro | לא דווח | 30.0% |
| פורסטר et al., 2014 | LRG5-Cterm | ZFN | GFP-Puro | לא דווח | 14.0% |
| Soldner et al., 2011 | סנכ"א | ZFN | Puro | 96 | 1.0% |
טבלת 1. תיאור של גנים אחרים ממוקד בשיטה זו, עם קורsponding מיקוד יעילות נלקחה ממחקרים שפורסמו בעבר. 4,5,38-41
| לוקוס | סדר פעולות | הערות |
| פריימר AAVS1-F | CTCTAACGCTGCCGTCTCTC | תנאי PCR: T מ '= 57 מעלות צלזיוס, 35 מחזורים |
| פריימר AAVS1-WT-R | GCTTCTCCTCTTGGGAAGTG | להקת WT: 1,273 נקודות בסיס |
| AAVS1 ממוקד-R פריימר | CGTCACCGCATGTTAGAAGA | להקה ממוקדת: 992 נ"ב |
| T2-Cas9-מדריך | GGGCCACTAGGGACAGGAT | ממאלי et al., 2013 |
| AAVS1-ZFN ימני | TAGGGACAGGAT | מHockemeyer et al., 2009 |
| AAVS1-ZFN-שמאל | TGGGGTGTCACC | מHockemeyer et al., 2009 |
| AAVS1-Talen ימני | TCCTAACCACTGTCTTT | מHockemeyer et al., 2011 |
| AAVS1-Talen-שמאל | CCCCTCCACCCCACAGT | מHockemeyer et al., 2011 |
רשימת 2. טבלה של פריימרים ורצפי מיקוד SSN.
| מיקוד לבנות | מספר EGFP + המושבות | ממוקד שיבוטים הרימו (PCR מאומת) | יעיל קודם מדווח תיקון מיקוד (על ידי כתם דרום) |
| ZFN | 150 | 86.9% (73.9% Het / 13.0% הומו) | 56% (50% Het / 6% הומו) |
| Talen | 412 | 91.3% (47.8% Het / 39.1% הומו) | 47% (Het 37.5% / 9.3% הומו) |
| CRISPR-Cas9 | 235 | 95.7% (69.5% Het / 26.3% הומו) | לא דווח |
3. מספרים השוואתיים השולחן של EGFP חיובי וממוקד Talen, ZFN וCRISPR / מושבות תאי גזע אנושיות Cas9. PCR אומת אינטגרציות במוקד AAVS1 לניסוי זה הם בהשוואה לכתם דרום מאומת אינטגרציות יחידים נכונים בניסויים קודמים 4,5.
Discussion
השיטה המוצגת כאן לבידוד אוכלוסיות הומוגניות של תאי pluripotent אדם בעריכת גן הגזע היא גישה רבת עוצמה ליצירת קווי hPSC isogenic השונה רק במוקד הממוקד. תאים אלה הם מערכת אידיאלית לחיטוט מנגנוני התמיינות תאים אנושי ופיתוח, כמו גם להבנת הפתופיזיולוגיה של מחלות monogenic בהגדרה גנטית בשליטה. כפי שהודגם כאן, אפשר להשתמש בשלוש אסטרטגיות עיצוב SSN עצמאיות (ZFNs, TALENs, וCRISPR / Cas9) כדי להשיג אינטגרציה ממוקדת במוקד AAVS1. לכל אחד משיטות אלו יתרונות משלה וחסרונות. יתרון פוטנציאלי של ZFNs ו, במידה מסוימת, TALENs הוא הגמישות בעיצוב שלהם, המאפשר הנדסת איטרטיבי כדי לשפר את ה- DNA מחייב תחומים של nucleases פרט 42. אופטימיזציה nuclease זה יכול להגדיל את הספציפיות של ZFNs וTALENs מעבר למה שהוא בר השגה עם CRIמערכת SPR / Cas9. סלקטיביות כזו עשויה להיות חשובה עבור יישומים קליניים הדורשים רמה גבוהה של היעד ספציפי. היתרון העיקרי של המערכת / Cas9 CRISPR הוא קלות שימוש. למרות ערכות בניית Talen וZFN נעשו זמינות לציבור (כלומר, דרך Addgene 21), CRISPR SSNs / מבוסס Cas9 קל יותר באופן משמעותי לבנייה, כהתאמה ההכרחית היחידה הוא oligonucleotide זוג 20 בסיס (בעת השימוש בפלסמיד px330 עיצוב 14). פשטות זה מוכיחה להיות יתרון עבור מעבדות מחקר מחפשים כולל עריכת הגנום בלימודיהם.
קיימות טכניקות transfection חלופיות, כוללים nucleofection 43, כדי ליצור קווי hPSC-גן בעריכה; עם זאת electroporation הוכח להיות יעילים 4,5,25 עקביים ועלות. Nucleofection יכול לשמש כדי transfect ישירות מתחמי ribonucleoprotein RNA Cas9-מדריך לגרעין, הגדלת יעילות SSNנאמנות ד 44. גידול hPSCs על MEFs הוא שיטה חזקה ולא יקרה כדי לשמור על hPSCs במדינת pluripotent ללא הבחנה מוגזמת. בנוסף, היא מאפשרת לבידוד קל של מושבות זהות מבחינה גנטית. לחלופין, ניתן hPSCs התרבות ללא MEFs, אולם תנאי התרבות אלה יכולים להיות יקרים יותר מתרבויות מבוססות מזין. יתר על כן, כל התהליך הוא מדרגי, המאפשר לבידוד של אירועי עריכה מאוד נדירים או הדור של שורות תאים שונות רבות בניסויי עריכה מקבילים.
הפרוטוקול המתואר כאן הוא חזק; עם זאת יש כמה שלבים עיקריים המשפיעים על היעילות שבה ניתן להשיג שיבוטים נערכו כראוי. המרכיב הקריטי ביותר לשיטה זו הוא שיש MEFs באיכות גבוהה וMEFs DR4 עמיד סמים. ההישרדות של hPSCs היחיד היא קלושה, וMEFs באיכות נמוכה יהיה לעכב את הבידוד של קווי hPSC מובחנים. שנית, השימוש של Y-27632 הוא גםמפתח להישרדות לאפשר תא בודד מבלי ליצור לחץ סלקטיבי לתאים עם קריוטיפ לא נורמלי 45. שלישית, לקטוף מושבות היטב במרווחים מבטיח הומוגניות גנטית של שורות תאים נגזרו. לבסוף, חשוב להכין טפטפות זכוכית, כך שהפתיחה היא קטנה מספיק כדי לשבור את המושבה לרבות חתיכות קטנות יותר, להבטיח כי מושבות מרובות תגדל בבאר החדשה. זה מאפשר הקטיף של subclones היטב במרווחים לצלחת העתק שיש בתרבות, שעזבה את הצלחת המקורית של מושבות מבודדות לgenotyping. חלק מאתגר בפרוטוקול זה הוא המדריך למשייכת טפטפות הזכוכית; זה צריך להיות מתורגל מראש. יש לציין שישנן טכניקות רבות של קטיף שיבוט שאינו דורשות פיפטה זכוכית. הנסיינים מוזמנים למצוא אחד שעובד הכי טוב בשבילם.
יש מגבלות לפרוטוקול זה, שניתן להתגבר על ידי שינויים פשוטים. ניסוי נועד only לשבש את המוקד של עניין ללא תיקון או אחד תיקון שתבנית אינו מכיל קלטת בחירה, חייב להשתמש בשיטה אחרת של העשרה לתאים שנערכו. שים לב שמספר מושבות שחייבות להיות נטלו למצוא שיבוט חיובי מגביר באופן משמעותי בהיעדר הבחירה. אסטרטגיה אחת כדי לשפר את היעילות היא לשתף transfect פלסמיד שאינו שילוב שמבטא חלבון פלואורסצנטי. לאחר מאפשר לתאים להתאושש במשך יומיים, ניתן למיין תאים ממוקדים על הקרינה חיובית באמצעות מיון תא הקרינה המופעל וreplated 29. תהליך זה מעשיר את תאים שכבר transfected עם פלסמידים ידי electroporation ובכך מגדיל את ההסתברות לאירוע עריכה במקביל בתא.
הטכניקות המתוארות כאן ניתן להרחיב להשתמש RNAs מדריך מרובה למקד כמה 14,46,47 לוקוסים בו זמנית. פרוטוקולים רבים הוקמו כדי לבדל hPSCsלסוגים שונים של תאים, ומאפשר מניפולציות גנטיות שונות בסוגי תאים של עניין 30. בסך הכל, יש לנו הפגנו הפוטנציאל לעריכת הגנום יעיל בhPSCs ללא קשר לבחירת SSN. אנו מציעים כי טכניקה זו יכולה להיות מותאמת ליצירת קווי hPSC isogenic שנערכו-גן בכל מוקד הגנומי.
Disclosures
המחברים מצהירים שום אינטרסים כלכליים מתחרים.
Acknowledgements
עבודה זו נתמכה על ידי גרנט זרע מוח קרן המחקר (BRFSG-2014-02) להלן Bateup. דירק Hockemeyer הוא חדש Scholar בהזדקנות של הקרן הרפואית אליסון ונתמך על ידי קרן גלן כמו גם Shurl וקיי קורצ'י הקרן. DH נתמך גם על ידי NIH 1R01CA196884-01.
Materials
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Life Technologies | 11320082 | |
| Fetal Bovine Serum (HI) | Life Technologies | 10082-147 | |
| Knockout Serum | Life Technologies | 10828-028 | |
| Fibroblast Growth Factor - basic | Life Technologies | PHG0261 | |
| Pen/Strep | Life Technologies | 15140-122 | |
| Glutamine | Life Technologies | 25030-081 | |
| MEM NEAA | Life Technologies | 11140-050 | |
| 2-mercaptoethanol | Life Technologies | 21985-023 | |
| Y-27632 | Calbiochem | 688000 | |
| 6-well plates | Corning | 3506 | |
| 12-well plates | Corning | 3512 | |
| 4 mm Electroporation cuvettes | Bio-rad | 165-2081 | |
| X-cel gene pulser II | Bio-rad | 165-2661 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| 10× Phosphate buffered saline (PBS) pH7.4 | Life Technologies | 70011-044 | |
| Puromycin | Life Technologies | A11138-02 | |
| Pasteur pipettes, plugged | VWR | 14672-412 | |
| Tris-HCl | Sigma-Aldrich | S5941 | |
| NaCl | Sigma-Aldrich | S9888 | |
| SDS | Sigma-Aldrich | L3771 | |
| EDTA | Sigma-Aldrich | E5134 | |
| Proteinase-K | Life Technologies | AM2544 | |
| Ethanol | VWR | TX89125-172SFU | |
| Isopropyl Alcohol | VWR | MK303216 | |
| TE Buffer | Life Technologies | 12090-015 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| 1.8 ml Cryotubes | ThermoScientific | 377267 |
References
- Carroll, D. Genome engineering with targetable nucleases. Annu Rev Biochem. 83, 409-439 (2014).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Matsa, E., Burridge, P. W., Wu, J. C. Human stem cells for modeling heart disease and for drug discovery. Sci Transl Med. 6 (239), 239ps6 (2014).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat Biotechnol. 27 (9), 851-857 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat Biotechnol. 29 (8), 731-734 (2011).
- Bozas, A., Beumer, K. J., Trautman, J. K., Carroll, D. Genetic analysis of zinc-finger nuclease-induced gene targeting in drosophila. Genetics. 182 (3), 641-651 (2009).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Urnov, F. D., et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature. 435 (7042), 646-651 (2005).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., Charpentier, E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jinek, M., East, A., Cheng, A., Lin, S., Ma, E., Doudna, J. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Doudna, J. A., Charpentier, E. Genome editing. the new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/cas systems. Science. 339 (6121), 819-823 (2013).
- Jasin, M. Genetic manipulation of genomes with rare-cutting endonucleases. Trends Genet. 12 (6), 224-228 (1996).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat Rev Genet. 11 (9), 636-646 (2010).
- Sander, J. D., Maeder, M. L., Reyon, D., Voytas, D. F., Joung, J. K., Dobbs, D. ZiFiT (zinc finger targeter): An updated zinc finger engineering tool. Nucleic Acids Res. 38, W462-W468 (2010).
- Carroll, D., Morton, J. J., Beumer, K. J., Segal, D. J. Design, construction and in vitro testing of zinc finger nucleases. Nat Protoc. 1 (3), 1329-1341 (2006).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39 (12), e82 (2011).
- Reyon, D., Tsai, S. Q., Khayter, C., Foden, J. A., Sander, J. D., Joung, J. K. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol. 30 (5), 460-465 (2012).
- Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng, G., Zhang, F. A transcription activator-like effector toolbox for genome engineering. Nat Protoc. 7 (1), 171-192 (2012).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from neisseria meningitidis. Proc Natl Acad Sci U S A. 110 (39), 15644-15649 (2013).
- Podhajska, A. J., Szybalski, W. Conversion of the FokI endonuclease to a universal restriction enzyme: Cleavage of phage M13mp7 DNA at predetermined sites. Gene. 40 (2-3), 175-182 (1985).
- Costa, M., et al. A method for genetic modification of human embryonic stem cells using electroporation. Nat Protoc. 2 (4), 792-796 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Cai, J., et al. Directed differentiation of human embryonic stem cells into functional hepatic cells. Hepatology. 45 (5), 1229-1239 (2007).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Soldner, F., et al. Parkinson's disease patient-derived induced pluripotent stem cells free of viral reprogramming factors. Cell. 136 (5), 964-977 (2009).
- Wen, Z., et al. Synaptic dysregulation in a human iPS cell model of mental disorders. Nature. 515 (7527), 414-418 (2014).
- Chiba, K., Hockemeyer, D. Genome editing in human pluripotent stem cells using site-specific nucleases. Methods Mol Biol. 1239, 267-280 (2015).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. J Vis Exp. , (2012).
- Ezashi, T., Das, P., Roberts, R. M. Low O2 tensions and the prevention of differentiation of hES cells. Proc Natl Acad Sci U S A. 102 (13), 4783-4788 (2005).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25 (18), 3745-3746 (1997).
- Southern, E. Southern blotting. Nat Protoc. 1 (2), 518-525 (2006).
- Henegariu, O., Heerema, N. A., Dlouhy, S. R., Vance, G. H., Vogt, P. H. Multiplex PCR: Critical parameters and step-by-step protocol. BioTechniques. 23 (3), 504-511 (1997).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Sexton, A. N., et al. Genetic and molecular identification of three human TPP1 functions in telomerase action: Recruitment, activation, and homeostasis set point regulation. Genes Dev. 28 (17), 1885-1899 (2014).
- Merkle, F. T., et al. Efficient CRISPR-Cas9-mediated generation of knockin human pluripotent stem cells lacking undesired mutations at the targeted locus. Cell Rep. 11 (6), 875-883 (2015).
- Soldner, F., et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset parkinson point mutations. Cell. 146 (2), 318-331 (2011).
- Forster, R., et al. Human intestinal tissue with adult stem cell properties derived from pluripotent stem cells. Stem Cell Reports. 2 (6), 838-852 (2014).
- Miller, J. C., et al. Improved specificity of TALE-based genome editing using an expanded RVD repertoire. Nat Methods. , (2015).
- Hohenstein, K. A., Pyle, A. D., Chern, J. Y., Lock, L. F., Donovan, P. J. Nucleofection mediates high-efficiency stable gene knockdown and transgene expression in human embryonic stem cells. Stem Cells. 26 (6), 1436-1443 (2008).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nat Biotechnol. 25 (6), 681-686 (2007).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4, 5400 (2014).
- Ousterout, D. G., Kabadi, A. M., Thakore, P. I., Majoros, W. H., Reddy, T. E., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome editing for correction of dystrophin mutations that cause duchenne muscular dystrophy. Nat Commun. 6, 6244 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved