Method Article
Istituzione di Genome-cura pluripotenti umane Linee di cellule staminali: Da Targeting per isolamento
In questo articolo
Riepilogo
Genome editing of human pluripotent stem cells (hPSCs) can be done quickly and efficiently. Presented here is a robust experimental procedure to genetically engineer hPSCs as exemplified by editing the AAVS1 safe harbor locus to express EGFP and introduce antibiotic resistance.
Abstract
Genome-editing di cellule staminali umane pluripotenti (hPSCs) fornisce una piattaforma geneticamente controllata e clinicamente rilevante da cui partire per comprendere lo sviluppo umano e indagare la fisiopatologia della malattia. Utilizzando nucleasi site-specific (SSN) per la modifica del genoma, la rapida derivazione di nuove linee HPSC ospitano specifiche alterazioni genetiche in un ambiente altrimenti isogenico diventa possibile. Zinc finger nucleasi (ZFNs), trascrizione nucleasi attivatore-come effettrici (Talens) e cluster brevi ripetizioni palindromiche regolarmente intervallati (CRISPR) / Cas9 sono i SSNs più comunemente utilizzati. Tutti questi nucleasi funzionano con l'introduzione di una doppia interruzione di DNA incagliato in un sito specificato, promuovendo in tal modo preciso l'editing gene ad un locus genomico. SSN-meditato editing genoma sfrutta due dei meccanismi endogeni della cellula di riparazione del DNA, che unisce non omologhe fine (NHEJ) e omologia riparazione diretta (HDR), a uno introdurre inserzione / delezione mutazioni o alter il genoma utilizzando un modello di riparazione omologa al sito della doppia interruzione filamento. L'elettroporazione di hPSCs è un mezzo efficace per trasfezione SSNs e modelli di riparazione che incorporano i transgeni come reporter fluorescenti e cassette di resistenza agli antibiotici. Dopo l'elettroporazione, è possibile isolare solo le hPSCs che incorporavano il costrutto riparazione selezionando per la resistenza agli antibiotici. Meccanicamente separando colonie HPSC e confermando la corretta integrazione al sito bersaglio attraverso genotipizzazione consente l'isolamento di linee cellulari sono correttamente orientati e geneticamente omogenee. La validità di questo protocollo è dimostrato qui utilizzando tutte e tre le piattaforme del SSN per incorporare EGFP e una resistenza puromicina costruire nel AAVS1 sicuro locus porto di cellule staminali pluripotenti umane.
Introduzione
Tecnologie di modifica del genoma stanno rapidamente evolvendo in strumenti standard per la biologia molecolare e cellulare 1. L'ingegneria genetica di cellule umane staminali pluripotenti (hPSCs) è di particolare interesse come hPSCs rappresentano una fonte di auto-rinnovamento delle cellule umane primarie geneticamente intatte. hPSCs possono essere differenziati in vari tipi di cellule per la modellazione della malattia o come fonte per le terapie di trapianto 2,3. Ha dimostrato qui è un protocollo che utilizza tre diversi tipi di nucleasi site-specific (SSN) in combinazione con i meccanismi endogeni di riparazione del DNA per l'integrazione mirata di un costrutto giornalista nel locus AAVS1. Dopo trasfezione di SSNs in hPSCs, dimostriamo come isolare popolazioni di cellule isogeniche ospitare il giornalista.
La capacità di manipolare il genoma, in particolare pluripotenti staminali genomi di cellule, utilizzando SSN non è un fenomeno nuovo, come l'utilità di nucleasi dita di zinco (ZFNs) e trascrizione attivata sianucleasi effettrici o simili (Talens) per la modifica del gene è stata dimostrata diversi anni fa 4-10. Tuttavia, con l'avvento di S. pyogenes CRISPR / tecnologia Cas9 11-13, editing gene è diventato ampiamente accessibili 14. Tutti SSNs introducono una doppia interruzione di DNA filamento (DSB) presso il sito di destinazione specificata 1,4,5,11 che viene riparato da meccanismi cellulari endogeni utilizzando riparazione non omologa end-joining (NHEJ) o omologia diretto (HDR) 15. NHEJ è soggetto a errori e può introdurre mutazioni frame-shift con conseguente perdita della funzione del gene, mentre HDR permette di nuovi elementi da introdurre attraverso la co-trasfezione di un modello di riparazione con il SSN. Mentre i principi alla base di riparazione del DNA che facilitano l'editing gene sono pensati per essere in gran parte la stessa per ogni SSN, alcune differenze tra le piattaforme possono essere osservato. De novo design ZFNs consente flessibilità e ottimizzazione nucleasi 16, tuttavia l'uso di pubblicamentebiblioteche di montaggio disponibili e strumenti di screening per progettare i singoli ZFNs può richiedere molto tempo. Una volta che il luogo desiderato per il targeting ZFN-mediata è determinato, coppie ZFN possono essere progettati con lo strumento online ZiFit 17. Dopo la progettazione, ZFNs può essere modulare assemblato attraverso vari cicli di plasmide clonazione 18. In alternativa, ci sono molti disponibili in commercio, ZFNs preconvalidati 19. Nucleasi TALE possono anche essere progettati utilizzando strumenti online e componenti disponibili pubblicamente 17,20. Ad esempio, Talens può essere rapidamente assemblato da blocchi di cinque ripetizioni racconto, attraverso il montaggio FLASH 21 o mediante PCR basato gerarchica Golden Gate complesso 22. Facilità di progettazione SSN e la velocità di costruzione utilizzando CRISPR / Cas9 hanno fatto genoma modifica di uno strumento ampiamente accessibile. La breve guida di RNA-mediata di mira CRISPR / Cas9 consente anche di multiplazione di RNA guida per indirizzare diversi loci con un unico costrutto 14. Il designdi Cas9 per la modifica genetica richiede solo l'individuazione di un motivo adiacente protospacer (PAM, un trinucleotide NGG per S. Pirogeni Cas9) prossimale al locus bersaglio. Inserendo un oligonucleotide corrispondente ai 20 coppie di basi 5 'del PAM nel plasmide px330 14, il costrutto può essere assemblato in un passo clonazione. Oltre a S. pyogenes Cas9, Cas9 da N. meningitidis (NmCas9) che riconosce un 5'-NNNNGATT-3 '(PAM) ha dimostrato di consentire efficiente gene-editing in hPSCs 23.
Oltre alle differenze nella facilità di progettazione SSN, ogni piattaforma ha proprietà specifiche. Ad esempio, ZFNs e Talens utilizzano il dominio nucleasi FokI, che genera un quattro nucleotidi 5 'sbalzo 24 mentre Cas9 è pensato per generare la maggior parte smussare DSBs indeterminato. ZFNs, Talens e Cas9 differiscono nella loro stabilità della proteina, tasso on-off sul DNA bersaglio, e la modalità di scansione DNA, i quali could teoricamente comportare piccole differenze nei risultati editing 1. Mentre saranno necessari ulteriori studi per comprendere appieno le conseguenze di queste differenze, si descrive qui un protocollo che sia molto robusto in tutte e tre le piattaforme e può essere utilizzato per generare facilmente hPSCs geneticamente modificati.
Indipendentemente dalla scelta SSN, elettroporazione è una procedura affidabile per trasfettare SSNs e modelli di riparazione omologia in hPSCs 25. Il numero di sopravvivere colonie dopo la selezione per la resistenza agli antibiotici dipende da parametri specifici del locus e la strategia di modifica (ad esempio, le dimensioni di inserto transgenico e le modalità di selezione). Il protocollo descritto qui di solito si traduce in circa 150-400 colonie cella singola derivati.
-Gene editing al locus AAVS1 utilizzo di questo protocollo è stato precedentemente utilizzato per dimostrare l'efficacia di SSNs 4,5. Il modello di riparazione AAV-CAGGS-EGFP utilizza una str trappola geneategy conferire resistenza puromicina in modo specifico locus. In breve, il modello di riparazione contiene un sito splice accettore monte della cassetta promoterless resistenza puromicina. Su corretta integrazione nel primo introne del gene PPP1R12C al locus AAVS1, la cassetta di resistenza è espresso dal promotore del gene modificato. La robustezza di questo test AAVS1 specifica ci permette di confrontare l'efficienza di ogni piattaforma SSN.
Editing gene utilizzando SSNs è potente data la capacità di interrompere e / o modificare teoricamente qualsiasi gene. Applicando questa strategia hPSCs fornisce versatilità hPSCs possono essere successivamente differenziate in una moltitudine di tipi di cellule umane come i neuroni 26, epatociti 27 e 28 cardiomiociti. Inoltre, l'uso di cellule staminali pluripotenti indotte derivati da pazienti consente la riparazione o l'introduzione di mutazioni patogeni noti in un background genetico specifico paziente 29 , Fornendo una piattaforma da cui partire per indagare i meccanismi della malattia e terapie di prova che utilizzano le proprie cellule del paziente 30. In sintesi, la modifica gene in hPSCs è un approccio efficiente e versatile per studiare la biologia di base dello sviluppo umano e della malattia 31.
Protocollo
Le procedure descritte in questo manoscritto sono stati esaminati e approvati dal Comitato di Sorveglianza di ricerca dell'Università di Berkeley Stem Cell.
1. Preparare cellule staminali per la modifica
- Crescere e cellule staminali pluripotenti cultura umana (hPSCs) su un 6-pozzetti contenenti 2,4 × 10 6 cellule / piastra di topo mitomicina C-inattivato fibroblasti embrionali (MEF) alimentatori coltivate su gelatina 32. Mantenere hPSCs in 3 ml di mezzi cellule staminali embrionali umane per bene (i media hESC) e crescere in un 37 ° C incubatore con il 3% O 2/5% di CO 2.
Nota: Per il successo di questo protocollo, non è necessario mantenere hPSCs in un incubatore a basso ossigeno; tuttavia è importante notare che hPSCs proliferano più velocemente in un ambiente di alta O 2, così tempi e cellulari devono essere regolati di conseguenza. Va inoltre notato che hPSCs mantenuto in ossigeno basso hanno tassi più bassi di differenziazione spontanea 33.- To fanno dei media da 500 ml hESC combinano 380 ml DMEM / F12, 75 ml di siero fetale bovino (FBS), 25 ml di siero sostituzione KnockOut (KSR). Aggiungere glutammina (1 mM concentrazione finale), 5 ml di 100x non essenziale aminoacidi, 100 unità / ml penicillina-streptomicina (P / S), fondamentale fattore di crescita dei fibroblasti (bFGF) (4 ng / ml concentrazione finale), e 2- mercaptoetanolo (5.5 micron concentrazione finale).
- Cambiare mezzi rimuovendo intero volume di supporti (3 ml) usando una pipetta di vetro e di vuoto. Sostituire con materiale hESC 3 ml caldo con una pipetta sierologica. Ripetere i media cambiano ogni giorno fino a quando hPSCs sono circa il 50% confluenti (Giorno -1).
- Un giorno prima di targeting (Giorno -1), cambiare i media hESC, rimuovendo i vecchi media e l'aggiunta di supporti hESC caldi integrato con 10 micron Y-27632.
- Anche il giorno -1, preparare uno o due 6 pozzetti o 10 cm di resistenti ai farmaci cellule di alimentazione MEF da DR4 topi (2,4 × 10 6 celle / piastra) 34.
Nota: In generale, 6 pozzetti sono annuncivantageous oltre 10 piatti cm, come essi ospitare più mezzi di comunicazione. 6 pozzetti anche assicurare che i cloni diverse da diversi pozzi sono indipendenti. Tuttavia, una piastra 10 cm consentirà più facile raccolta, a seconda del microscopio per questo processo.
2. Modifica pluripotenti cellule staminali
- Preparare soluzioni di trasfezione pipettando 5 mg ciascuno di ZFN 1 e 2, TALEN 1 e 2, o 15 ug del CRISPR / Cas9 codifica px330 plasmide (Figura 1) in una provetta da 1,5 ml. Dispensare 30 microgrammi di plasmide riparazione in questo tubo pure. Infine, pipetta 1x tampone fosfato (PBS) nel tubo per portare il volume fino a 300 ml.
Nota: Preparare plasmidi come midiprep (qualsiasi kit è adatto) con una concentrazione minima di 300 ng / ml per mantenere il volume totale della soluzione di trasfezione meno di 300 microlitri. Non è necessario eseguire estrazione con fenolo / cloroformio, per linearizzare il plasmide, o utilizzare un endotoxin kit di preparazione plasmide gratuito. - Rimuovere i supporti da hPSCs utilizzando una pipetta di vetro e di vuoto. Pipettare 2 ml caldo 1x PBS in ciascun pozzetto per lavare le cellule.
- Rimuovere la PBS immediatamente utilizzando una pipetta di vetro e di vuoto. Aggiungere 0,5 ml di soluzione di 0,25% tripsina-EDTA direttamente su cellule in ogni pozzetto. Mettere in incubatrice (37 ° C / 5% di CO 2/3% O 2) per circa 10 minuti. o fino a quando feeder layer inizia a sollevare la piastra.
- Aggiungere 2 ml di mezzi caldo esWash (470 ml DMEM / F12, 25 ml FBS, 100 unità / ml P / S) a ciascun pozzetto per arrestare la reazione tripsina.
- Assicurarsi che le cellule di alimentazione staccarsi come un lenzuolo. Pipettare il contenuto di ciascun pozzetto in una singola provetta conica da 50 ml, che unisce tutti i pozzetti. Triturare le cellule con un 10 ml pipetta sierologica. Non è necessario rompere lo strato alimentatore; hPSCs verrà via dello strato dell'alimentatore delicatamente triturazione. Ci dovrebbe essere di circa 15 ml di sospensione cellulare.
- Aggiungere 25 ml di media esWash al tubo per portare il volumi fino a 40 ml totale. Consentire grossi pezzi di alimentazione di stabilirsi sul fondo della provetta per 1-2 min. Rimuovere il surnatante (~ 38 ml) dal tubo usando una pipetta sierologica e depositare in una nuova provetta conica da 50 ml.
- Centrifugare per 5 min a 190 x g. Rimuovere il surnatante dal tubo usando una pipetta di vetro e di vuoto. Assicurarsi di non disturbare il pellet. Risospendere le cellule in 500 microlitri 1x PBS. Combinate con la soluzione plasmide trasfezione preparato in precedenza. Contare le cellule di questo passo. Utilizzare 5-10 milioni di cellule per elettroporazione.
- Pipetta la sospensione intero 800 ml in un elettroporazione cuvetta 4 millimetri, immettere sul ghiaccio per 3-5 minuti. Cellule elettroporazione utilizzando il programma esponenziale sul sistema elettroporazione con i seguenti parametri: 250 V, 500 mF, ∞ resistenza, e 4 mm dimensioni cuvetta. Dopo elettroporazione, posizionare cuvetta di nuovo in ghiaccio per 3 min.
Nota: Osservare la costante di tempo del elettroporazione sul sistema elettroporazione. La costante di tempovaria con numero di cellulare e la purezza del DNA. Trasfezioni di successo di solito hanno un tempo costante tra 10-14 msec quando si utilizzano le condizioni elencate e un generatore di impulsi gene II. Efficienza elettroporazione inferiori possono verificarsi quando costanti di tempo varia da questi valori. - Risospendere le cellule elettroporate in 18 ml di caldo hESC multimediale integrato, con 10 micron Y-27632. Tavola 3 ml di questa sospensione di cellule singole in ciascun pozzetto di una piastra 6 bene con cellule feeder DR4. Ritorno a incubatore (37 ° C / 5% di CO 2/3% O 2).
3. Selezione delle colonie positive
- Giorno 2, rimuovere tutti i media utilizzando una pipetta di vetro e di vuoto. Sostituire con 3 ml di mezzi hESC caldi integrato con 10 micron Y-27632. Giorno 3, rimuovere tutti i media utilizzando una pipetta di vetro e di vuoto. Sostituire con 3 ml di media hESC caldo senza includere Y-27632. Giorno 4, rimuovere tutti i media utilizzando una pipetta di vetro e di vuoto. Sostituire con 3 ml i media hESC caldi integrati con antibiotici per selectiin data. Ritorno a incubatore (37 ° C / 5% di CO 2/3% O 2).
Nota: Il tipo di antibiotico utilizzato dipenderà dalla cassetta di resistenza inclusa nel modello di riparazione. Quando si lavora con WIBR # 3 hPSCs (NIH di registro 0079), 0,5 mg / ml puromicina, 70 mg / ml di G418 (geneticina), e 35 mg / ml igromicina sono stati utilizzati con successo. Le concentrazioni di selezione dovrebbero essere determinati empiricamente che istituisce la concentrazione minima di antibiotico necessario per uccidere le cellule wild-type entro una settimana circa. - Giorni 5-12, cambiano i media giornaliera, in sostituzione di vecchi media ogni volta con i media hESC caldi integrati con gli antibiotici.
Nota: Aspettatevi una grande quantità di morte cellulare. Colonie individuali diventeranno evidenti intorno al giorno 8-10. Supporti regolare hESC senza antibiotici può essere utilizzato dopo 12-14 giorni di selezione continua. Se la densità cellulare è alto o morte cellulare è lento, acidificazione dei media dovrebbe essere evitata e può essere necessario aumentare le volu multimedialime (fino a 4-5 ml) durante i primi giorni di selezione.
4. Picking Colonie selezionati (giorno 12-14)
- Osservare le colonie il giorno 12-14. Osservate le colonie che sono pronti a raccogliere su un microscopio dissezione e di garantire che essi non contengono cellule che iniziano a differenziarsi. La dimensione approssimativa dovrebbe essere 800-1.200 micron. Se colonie raggiungono queste dimensioni prima del giorno 12, si raccomanda che essi dovrebbero essere raccolti poi.
Nota: Per ogni esperimento di targeting, scegliere come molte colonie necessaria per garantire il genotipo desiderato viene isolato. A titolo di esempio, la modifica AAVS1 con il modello di riparazione AAV-CAGGS-EGFP ha dimostrato costantemente robusta integrazione, e richiede solo 12-24 colonie per essere raccolti per ottenere circa 5-10 cloni heterozygously mirati. Altri esperimenti mirati potrebbero richiedere più colonie da prelevare (Tabella 1). La frequenza degli eventi di targeting corretti dipende da fattori quali l'efficienza del SSN introdurre un DSB, la dimensione dell'inserto, e la strategia di selezione. Nel caso dell'approccio trap gene per il locus AAVS1 qui presentata, il marcatore di selezione è espressa solo quando correttamente integrato al sito bersaglio, riducendo il numero di colonie necessarie per ottenere un clone correttamente mirato. - Un giorno prima della raccolta, preparare piastre da 12 pozzetti di MEF (2,4 x 10 6 cellule / piastra, saranno utilizzati un pozzetto per ogni colonia scelto).
- Il giorno della raccolta, rimuovere tutti i supporti dalle 12 piatti ben MEF utilizzando una pipetta di vetro e vuoto e sostituirlo con 1 ml di mezzi hESC. Anche il giorno di raccolta, cambiare hESC media sulle piastre 6 e che stanno per essere raccolti.
- Tirare pipette di vetro per la dissociazione meccanica delle colonie individuali dallo strato alimentatore. Abbassare le pipette su un bruciatore Bunsen-cappuccio e ammorbidito al punto di espansione fino vetro è malleabile. Eliminare rapidamente dal fuoco e tirare la pipetta a parte la creazione di un punto angolare. Rompere ilpunto circa 2 cm dall'asse piegata, lasciando uno stretto canale. Lucidare la fine del canale esponendo alla fiamma per 1-2 sec.
Nota: se si desidera scegliere colonie un puntale p20, tuttavia, questo può ridurre la clonalità della cultura, come la punta sordo può rimuovere una maggiore quantità di cellule da ciascuna colonia in media, potenzialmente contaminanti guadagni successivi. - Montare dispositivo raccolta prendendo un puntale di filtro p1000 e allegando un bulbo di aspirazione per l'estremità più stretta. Inserire la pipetta di vetro tirato in la parte più larga.
- Posizionare un 6-pozzetti di hPSCs da prelevare sul palco di un microscopio dissezione montato in una cappa di coltura di tessuti. Comprimere il bulbo del dispositivo di prelievo e accise delicatamente e tagliare un individuo colonia in 10-20 pezzi uguali dimensioni, facendo attenzione a non rilasciare pezzi nei media. Prendere asportati pezzi HPSC della colonia nella pipetta rilasciando la lampadina. Provate a prendere il meno media come possibile durante il trasferimento.
- Trasfer la colonia individuo ad un singolo pozzetto di una piastra di MEF 12 pozzetti, comprimendo il bulbo di nuovo direttamente nel pozzo, rilasciando la colonia ormai spezzata. Etichettare ogni bene per consentire l'identificazione univoca di unicellulari cloni derivati. Ripetere l'operazione per il numero di colonie, se necessario, cambiando la pipetta di vetro ogni volta.
Nota: le colonie di raccolto potrebbe richiedere del tempo per imparare. Si raccomanda che la pratica sperimentatore su alcune cellule di controllo prima di tentare di isolare colonie mirati. - Ritorno piastre da 12 pozzetti alla incubatore (37 ° C / 3% O 2/5% CO 2), dondolo delicatamente la prima piastra per evitare l'accumulo di cellule nel mezzo di ciascun pozzetto.
- Il giorno successivo e ogni giorno successivo, rimuovono tutto volume dei mezzi di comunicazione con una pipetta di vetro e di vuoto e sostituire con 1,5 ml di mezzi hESC caldo finché cellule sono circa il 50% confluenti (solitamente ciò richiede 10-12 giorni).
- Dopo 10-12 giorni, scegliere uno di due colonie di ogni bene e trasferire ai nuovi 12-pozzetti MEF ricui ripetendo i passaggi 4,1-4,8 per generare un piatto replica.
- Estrarre DNA (vedi sotto) dai restanti colonie in ciascun pozzetto delle piastre MEF originali.
- Rimuovere i supporti da tutti i pozzetti con una pipetta di vetro e di vuoto. Pipettare 1 ml 1x PBS viene eseguito su ciascun bene per lavare le cellule. Rimuovere PBS usando pipetta di vetro e di vuoto. Pipettare 250 microlitri di tampone di lisi cellulare (concentrazioni finali in H 2 O: 10 mM Tris HCl, 5 mM EDTA, 0,2% SDS, NaCl 200 mM, 0,08 mg / ml proteinasi-K) su ciascun pozzetto. Posto in incubatrice (37 ° C / 3% O 2/5% di CO 2) O / N.
- Il giorno successivo, il contenuto della pipetta di ogni pozzetto in singole provette da 1,5 ml. Pipettare 250 ml di alcool isopropilico in ciascun tubo per precipitare il DNA. Agitare vigorosamente tubo. Un precipitato bianco deve essere visibile.
- Spin ogni tubo obliquo per 3 min a 13.000 rpm in una centrifuga da tavolo. Dopo spin down, scartare il surnatante per decantazione in rifiuti liquidi. Un piccolo pellet di DNA deve rimanere attaccata al fondo diil tubo. Pipettare 250 ml di etanolo al 70% in ciascuna provetta per lavare il pellet di DNA. Agitare energicamente. Spin ogni tubo obliquo per 3 min a 13.000 rpm in una centrifuga da tavolo.
- Dopo la filatura giù, scartare il surnatante per decantazione in rifiuti liquidi. Un piccolo pellet di DNA deve rimanere attaccato al fondo della provetta. Pipettare il resto del surnatante quindi non c'è liquido rimasto nel tubo. Lasciare tubo aperto ad asciugare sul banco per 5-10 min. Dopo l'essiccazione, risospendere il DNA in 250 microlitri di buffer TE. Posizionare a 37 ° C per 6 ore per consentire il DNA a sciogliere.
- Genotipo ogni campione utilizzando una strategia di pre-ottimizzato PCR o sud macchia.
Nota:.. Per AAVS1 mira utilizzando il modello di riparazione AAV-CAGGS-EGFP, la strategia del sud macchia può essere trovato in Hockemeyer et al, 2009 4 Un protocollo assorbente del sud globale si trova nel sud, 2006 35 Per PCR multiplex genotipizzazione di. lo stesso esperimento, primer e le condizioni possono esseretrovati nella tabella 2 36. - Scartare pozzi che non sono adeguatamente mirati e continuare a cambiare i media hESC in cellule opportunamente mirati ogni giorno. Congelare le cellule quando sono circa il 50% confluenti, vedi sotto per il congelamento protocollo.
- Un giorno prima del congelamento, cambiare i media hESC, rimuovendo i vecchi media e l'aggiunta di supporti hESC caldi integrato con 10 micron Y-27632.
- Il giorno di congelamento preparare 0,5 ml ciascuna di soluzione A e la soluzione B per pozzetto di una piastra 12 pozzetti che viene congelata giù in due provette da 15 ml coniche. Geolocalizzazione su ghiaccio. Soluzione A: 50% dei media hESC e il 50% FBS; Soluzione B: 80% FBS e 20% dimetilsolfossido (DMSO).
- Dissociarsi colonie HPSC nelle singole cellule prima del congelamento. Seguire i passaggi 2.2-2.8 per fare questo, il ridimensionamento dei volumi di conseguenza. Pellet cellulare Completamente risospendere in 0,5 ml di soluzione A.
- Aggiungere 0,5 ml di soluzione B di sospensione cellulare, pipetta su e giù per fare sospensione cellulare omogenea. Take il volume totale della sospensione cellulare (~ 1 ml) e deposito in cryotube 2 ml. Tappo a vite saldamente.
- Posto cryotube in un congelatore ° -80 C O / N. Il giorno dopo il congelamento, rimuovere le cellule congelate da -80 ° C freezer e subito mettere in serbatoio di azoto liquido per la conservazione a lungo termine.
Risultati
Qui mostriamo un protocollo compatibile con tre diverse piattaforme SSN per creare linee HPSC geneticamente modificati. Abbiamo mirato WIBR # 3 cellule staminali embrionali umane a livello del locus AAVS1 utilizzando ZFNs precedentemente pubblicati 4, 5 e Talens CRISPR / Cas9s 37 utilizzando un modello di riparazione che introduce un reporter EGFP e una cassetta di resistenza puromicina 4.
Abbiamo coltivato i nostri hPSCs su MEF per un flusso di lavoro di coltura cellulare che consente il mantenimento e l'espansione delle hPSCs indifferenziate (Figura 2), che è anche conveniente e scalabile. Se le cellule sono coltivate più del necessario vi è il rischio di maggiore differenziazione, riducendo il numero di cellule pluripotenti che sono trasfettate e quindi il numero di colonie HPSC mirati correttamente ottenuti.
Noi electroporated 5.0 x 10 6 cellule per piattaforma SSN e placcato le cellule di ogni mira su un singolo piatto ben 6 di DR4 MEF. Dopo la selezione, ogni piattaforma ha provocato colonie EGFP-positivi (Figura 3) e una combinazione di non mirati, omozigosicamente mirate e heterozygously mirate cloni (Figura 4A, B, tabella 3). Nelle condizioni qui presentati, troviamo che la AAVS1 Talens provocato la maggior parte dei cloni EGFP-positivi. Il modello di riparazione utilizzato per questo esperimento consiste di un sito accettore splice monte del reporter e resistenza puromicina cassette EGFP. Usando questa strategia "gene-trap" (Figura 1), il costrutto deve inserire nel primo introne del locus AAVS1, utilizzando il promotore endogeno per guidare l'espressione della cassetta puromicina-resistenza. La mancanza di un promotore guida l'espressione del gene di resistenza puromicina all'interno del modello di riparazione dovrebbe impedire espressione nella evento di integrazione casuale.
Pertanto, ci aspettiamo che tutti i cloni puromicina-resistenti dovrebbero essere mirate presso il sito AAVS1. Va notato che un sottogruppo di cloni trasportare integrazioni aberranti nel locus AAVS1 che sono rilevabili da Southern blot utilizzando una sonda interna, ma non dalla maggior parte delle strategie di PCR (Figura 1; Figura 4C) 4. Questi eventi di integrazione sono più probabilmente il risultato di eventi rivolti eterologhi che portano a più integrazioni del plasmide donatore 4. Abbiamo scelto 24 colonie da ogni esperimento SSN e ha scoperto che tutte le piattaforme avevano efficienze molto alte di targeting e ha mostrato differenze solo minime. Come interrogato mediante PCR, CRISPR / Cas9 dato i cloni più correttamente mirata, mentre la piattaforma TALEN aveva i cloni più omozigosicamente mirati (Tabella 3).
/files/ftp_upload/53583/53583fig1highres.jpg "width =" 700 "/>
Figura 1. Schema del gene modificato AAVS1 locus utilizzando il modello di riparazione AAV-CAGGS-EGFP. Modificato da Hockemeyer et al., 2009. Clicca qui per vedere una versione più grande di questa figura.
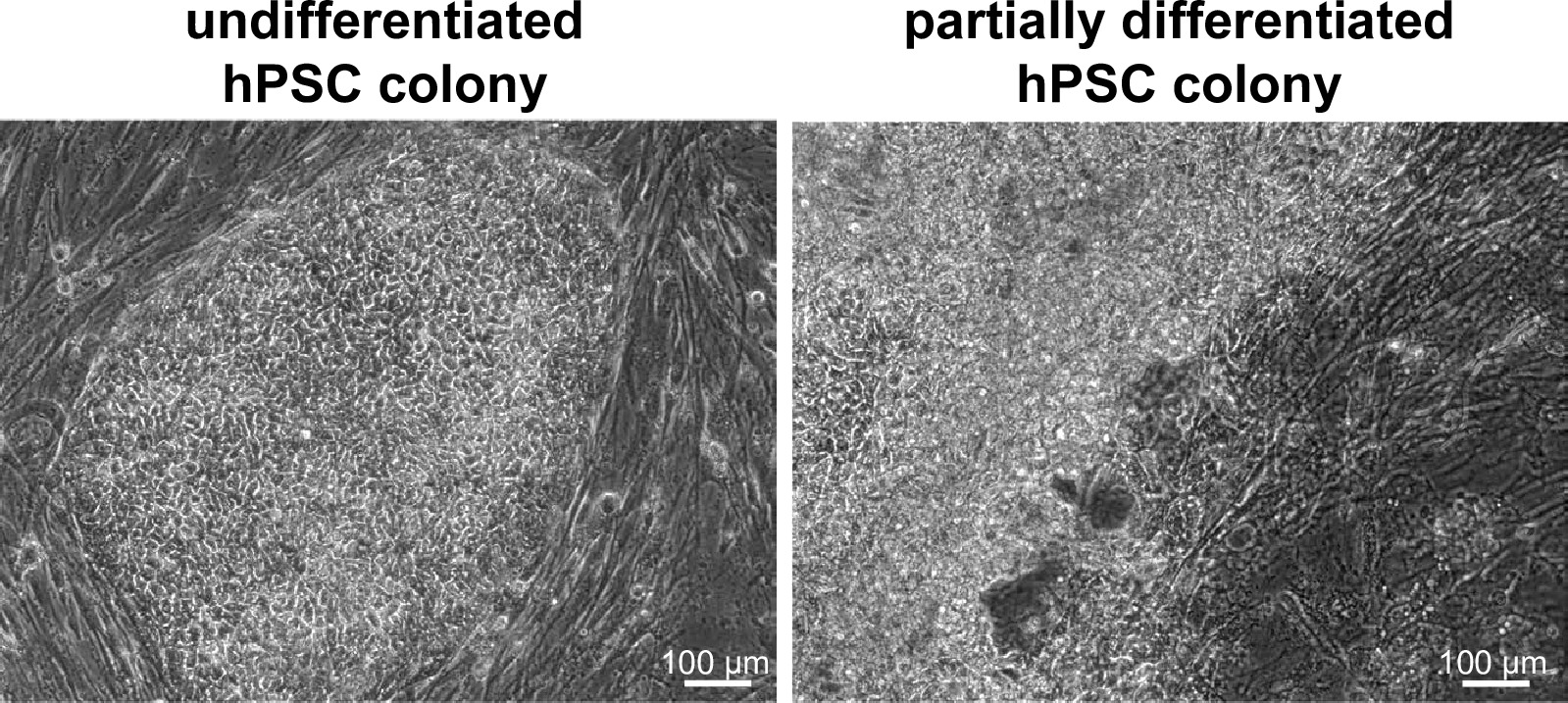
Figura 2. Le colonie di WIBR # 3 celle. Immagini rappresentative in campo chiaro di colonie di WIBR # 3 cellule staminali embrionali umane prima di targeting. Si noti la mancanza di differenziazione e la chiara separazione dal feeder layer in una colonia ideale (a sinistra) al contrario di una non ideale (a destra). Clicca qui per vedere una versione più grande di questa figura.
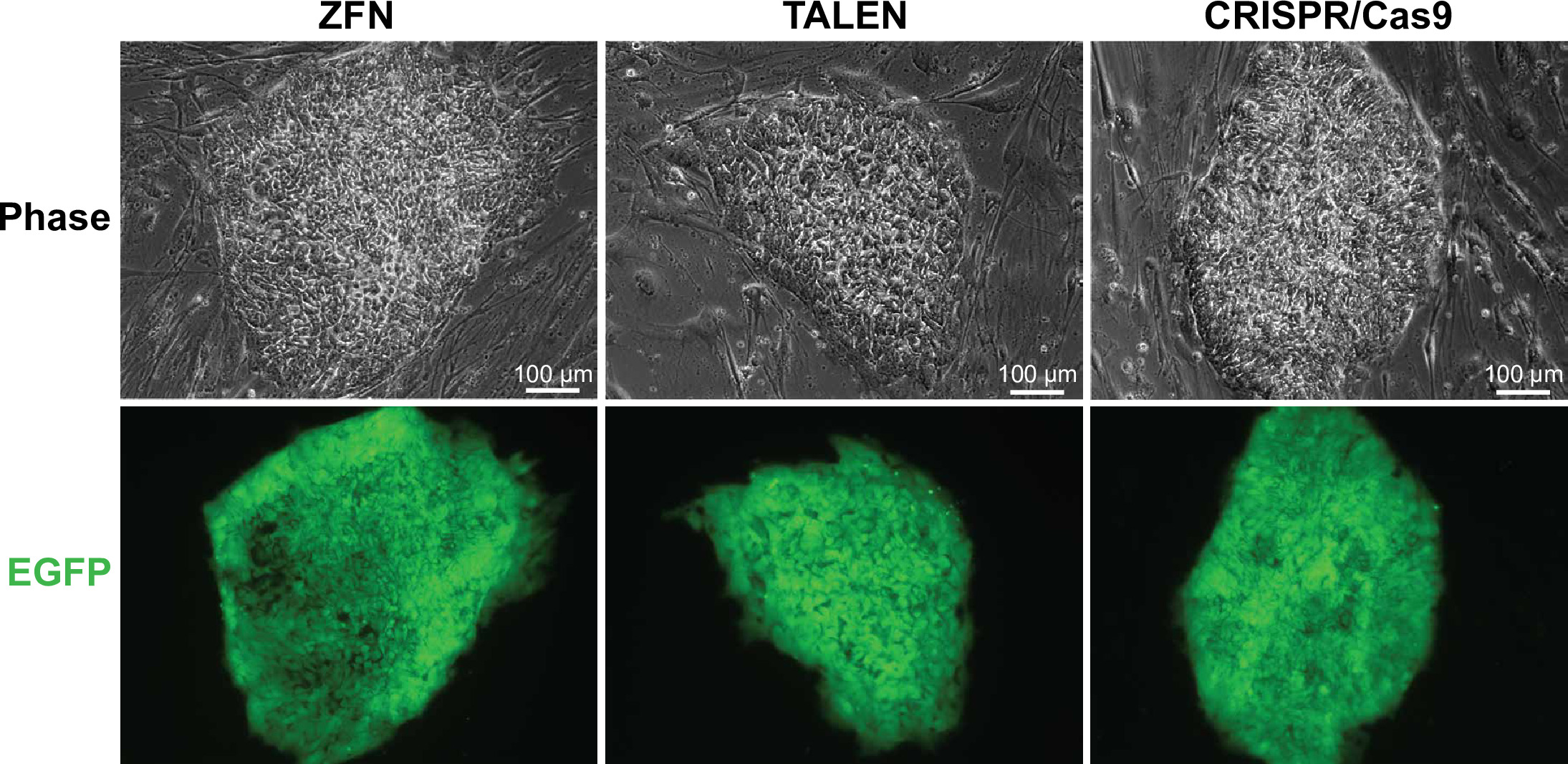
Figura 3. EGFP-positivi WIBR # 3 celle. Immagini rappresentative della WIBR # 3 cellule bersaglio con un modello di riparazione-EGFP esprimere al locus AAVS1. Immagini di colonie rappresentative modificati con ZFNs, Talens e CRISPR / Cas9 vengono mostrati. Cliccate qui per vedere una versione più grande di questa figura.
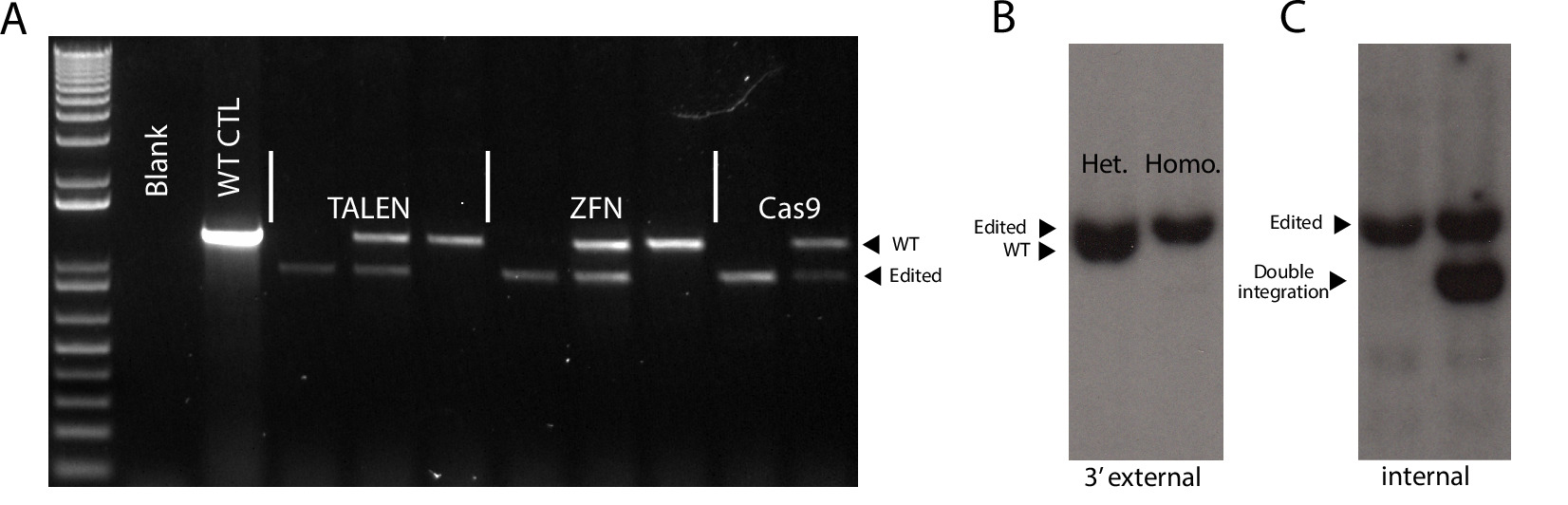
Figura 4. strategie di genotipizzazione per confermare il corretto il targeting. (A) Rappresentante PCR risultati genotipizzazione mostrano non mirati, eterozigoti ed omozigoti cloni mirati attraverso tre piattaforme SSN. WT CTL = controllo wild-type. (B) Reprerappresentative risultati macchia del sud mostrano una eterozigoti mirato clone di un clone e mirata omozigote rilevata con sonda esterna a 3 '. Dimensioni frammento: WT-6.5 kb, a cura-6.9 kb. (C) Risultati rappresentativi Southern blot mostrano un clone adeguatamente modificato e un clone eterozigote con un non casuale doppio integrazione. Dimensioni: Frammento. Correttamente modificato-6.9 kb, aberranti ulteriore integrazione a 5 kb Cliccate qui per vedere una versione più grande di questa figura.
| Studio | Gene mirata | Piattaforma | Riparazione tipo di modello | # Di cloni raccolse | Targeting efficienza |
| Sexton et al., 2014 | TPP1 | ZFN | GFP-Puro | non dichiarata | non dichiarata |
| Sexton et al., 2014 | TERT | ZFN | Hygromycin | non dichiarata | non dichiarata |
| Hockemeyer et al., 2009 | POU5F1 | ZFN | GFP-Puro | 31 | 39,0% |
| Hockemeyer et al., 2009 | PITX3 | ZFN | GFP-Puro | 74 | 14,9% |
| Hockemeyer et al., 2011 | POU5F1 | TALEN | GFP-Puro | 68 | 91.0% |
| Hockemeyer et al., 2011 | PITX3 | TALEN | GFP-Puro | 96 | 13,0% |
| Merkle et al., 2015 | VASA | Cas9 | Reporter-geneticina | 139 | 94,0% |
| Merkle et al. </ em>, 2015 | CRH | Cas9 | Reporter-geneticina | 30 | 93,0% |
| Merkle et al., 2015 | HCRT | Cas9 | Reporter-geneticina | 154 | 92.0% |
| Merkle et al., 2015 | HMX2 | Cas9 | Reporter-geneticina | 11 | 45,0% |
| Forster et al., 2014 | LRG5-Nterm | ZFN | GFP-Puro | non dichiarata | 30,0% |
| Forster et al., 2014 | LRG5-Cterm | ZFN | GFP-Puro | non dichiarata | 14,0% |
| Soldner et al., 2011 | SNCA | ZFN | Puro | 96 | 1,0% |
Tabella 1. Descrizione di altri geni mirati utilizzando questo metodo, con correcor- mira efficienze tratti da studi pubblicati in precedenza. 4,5,38-41
| Locus | Sequenza | Gli appunti |
| AAVS1-F Primer | CTCTAACGCTGCCGTCTCTC | Condizioni di PCR: T m = 57 ° C, 35 cicli |
| AAVS1-WT-R Primer | GCTTCTCCTCTTGGGAAGTG | Banda WT: 1273 bp |
| AAVS1-mirata-R Primer | CGTCACCGCATGTTAGAAGA | Banda mirata: 992 bp |
| T2-Cas9-guida | GGGCCACTAGGGACAGGAT | Dal Mali et al., 2013 |
| AAVS1-ZFN-destra | TAGGGACAGGAT | Da Hockemeyer et al., 2009 |
| AAVS1-ZFN-sinistra | TGGGGTGTCACC | Da Hockemeyer et al., 2009 |
| AAVS1-TALEN-destra | TCCTAACCACTGTCTTT | Da Hockemeyer et al., 2011 |
| AAVS1-TALEN-sinistra | CCCCTCCACCCCACAGT | Da Hockemeyer et al., 2011 |
Tabella 2. Elenco dei primer e SSN sequenze di targeting.
| Targeting Construct | Numero di EGFP + Colonie | Cloni raccolte mirate (PCR verificato) | Precedente Segnalato corretta efficienza Targeting (da Southern blot) |
| ZFN | 150 | 86,9% (73,9% het / 13,0% omo) | 56% (50% het / 6% omo) |
| TALEN | 412 | 91,3% (47,8% het / 39,1% omo) | 47% (37,5% het / 9,3% omo) |
| CRISPR-Cas9 | 235 | 95,7% (69,5% het / 26,3% omo) | non dichiarata |
Tabella 3. numeri comparativi di EGFP-positivi e mirati TALEN, ZFN e CRISPR / Cas9 colonie di cellule staminali umane. PCR verificate integrazioni a livello del locus AAVS1 per questo esperimento sono rispetto al Southern blot verificato adeguate singole integrazioni in esperimenti precedenti 4,5.
Discussione
Il metodo presentato qui per isolare popolazioni omogenee di cellule staminali umane pluripotenti geni modificati è un approccio potente per la generazione di linee isogeniche HPSC che differiscono solo a livello del locus mirato. Queste cellule sono un sistema ideale per sondare i meccanismi di differenziazione cellulare umana e sviluppo, nonché per comprendere la fisiopatologia di malattie monogeniche in un ambiente controllato genetica. Come dimostrato qui, è possibile utilizzare tre strategie progettuali SSN indipendenti (ZFNs, Talens, e CRISPR / Cas9) per realizzare l'integrazione mirata al locus AAVS1. Ciascuno di questi metodi ha i suoi vantaggi e svantaggi. Un vantaggio potenziale di ZFNs e, in una certa misura, Talens è la flessibilità del disegno, che consente ingegneria iterativo per migliorare la dominii singoli nucleasi 42 legame al DNA. Questa ottimizzazione nucleasi potrebbe aumentare la specificità della ZFNs e Talens là di ciò che è realizzabile con la CRISistema SPR / Cas9. Tale selettività può essere importante per applicazioni cliniche che richiedono un alto grado di specificità di destinazione. Il vantaggio principale del sistema / Cas9 CRISPR è la facilità d'uso. Sebbene kit di costruzione Talen e ZFN sono stati messi a disposizione del pubblico (ad esempio, attraverso Addgene 21), CRISPR / SSNs basate Cas9 sono notevolmente più facili da costruire, come l'unica personalizzazione necessaria è una coppia di oligonucleotidi 20 di base (quando si utilizza il plasmide px330 disegno 14). Questa semplicità si rivela vantaggiosa per laboratori di ricerca che desiderano includere la modifica del genoma nei loro studi.
Ci sono tecniche di trasfezione alternative, tra cui nucleofection 43, per creare linee HPSC geni modificati; tuttavia elettroporazione ha dimostrato di essere efficace 4,5,25 coerenti ed economicamente. Nucleofection può essere utilizzato per trasfettare direttamente Cas9-guida i complessi RNA ribonucleoproteina nel nucleo, aumentando l'efficienza di un SSNd fedeltà 44. Crescere hPSCs su MEF è un metodo affidabile e poco costoso da mantenere hPSCs in uno stato pluripotente, senza differenziazione eccessiva. Inoltre, esso permette una facile isolamento di colonie geneticamente identici. In alternativa, è possibile hPSCs cultura senza MEF, tuttavia queste condizioni di coltura può essere più costoso di culture fondate alimentazione. Inoltre, l'intero processo è scalabile, consentendo l'isolamento di modifica degli eventi molto rari o la generazione di molte linee cellulari distinti esperimenti montaggio parallelo.
Il protocollo qui descritto è robusto; tuttavia ci sono diversi passaggi chiave che influenzano l'efficienza con cui si possono ottenere cloni modificati correttamente. La componente più critico per questo metodo è avere MEF di alta qualità e resistenti ai farmaci MEF DR4. La sopravvivenza di singoli hPSCs è tenue, e MEFs bassa qualità impedirà l'isolamento di linee HPSC indifferenziate. In secondo luogo, l'uso di Y-27632 è anchechiave per consentire la sopravvivenza delle cellule unico senza generare una pressione selettiva per le cellule con un cariotipo anormale 45. In terzo luogo, la raccolta colonie ben distanziati assicura omogeneità genetica delle linee cellulari derivate. Infine, è importante preparare pipette di vetro in modo che l'apertura è abbastanza piccolo per rompere la colonia in molti pezzi più piccoli, in modo che più colonie crescere nel nuovo pozzo. Questo permette la raccolta di sottocloni ben distanziati per un piatto replica di avere nella cultura, lasciando il piatto originale di colonie isolate per la genotipizzazione. Una parte difficile in questo protocollo è il manuale di trazione delle pipette di vetro; questo dovrebbe essere praticata in anticipo. Va notato che ci sono più tecniche di raccolta clone che non richiedono una pipetta di vetro. Gli sperimentatori sono incoraggiati a trovare quello che funziona meglio per loro.
Ci sono limitazioni a questo protocollo che può essere superato con semplici modifiche. Un esperimento destinato a oolo interrompere il luogo di interesse, senza riparazione o uno il cui modello di riparazione non contiene una cassetta di selezione, deve utilizzare un altro metodo di arricchimento per le cellule che sono state modificate. Si noti che il numero di colonie che devono essere raccolti per trovare un clone positivo aumenta notevolmente in assenza di selezione. Una strategia per migliorare l'efficienza è co-trasfezione un plasmide non integranti che esprime una proteina fluorescente. Dopo permettendo alle cellule di recuperare per due giorni, le cellule mirate possono essere ordinati su fluorescenza positiva utilizzando la fluorescenza-attivato l'ordinamento delle cellule e ripiastrate 29. Questo processo arricchisce di cellule che sono state trasfettate con i plasmidi di elettroporazione e quindi aumenta la probabilità di un evento di editing concomitante nella cellula.
Le tecniche qui descritte possono essere estesi ad utilizzare più RNA guida per indirizzare simultaneamente diversi loci 14,46,47. Sono stati stabiliti molti protocolli di differenziare hPSCsin tipi di cellule diverse, consentendo varie manipolazioni genetiche in tipi cellulari di interesse 30. Nel complesso, abbiamo dimostrato il potenziale per un efficiente modifica del genoma in hPSCs indipendentemente dalla scelta SSN. Si propone che questa tecnica può essere adattato per creare linee isogeniche HPSC che sono state gene-modificati in qualsiasi locus genomico.
Divulgazioni
Gli autori dichiarano interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto da un seme di Grant cervello Research Foundation (BRFSG-2014-02) per Helen Bateup. Dirk Hockemeyer è un nuovo Scholar in invecchiamento della Ellison Medical Foundation ed è supportato dalla Fondazione Glenn così come The Shurl e Kay Curci Foundation. DH è supportato anche dal NIH concedere 1R01CA196884-01.
Materiali
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Life Technologies | 11320082 | |
| Fetal Bovine Serum (HI) | Life Technologies | 10082-147 | |
| Knockout Serum | Life Technologies | 10828-028 | |
| Fibroblast Growth Factor - basic | Life Technologies | PHG0261 | |
| Pen/Strep | Life Technologies | 15140-122 | |
| Glutamine | Life Technologies | 25030-081 | |
| MEM NEAA | Life Technologies | 11140-050 | |
| 2-mercaptoethanol | Life Technologies | 21985-023 | |
| Y-27632 | Calbiochem | 688000 | |
| 6-well plates | Corning | 3506 | |
| 12-well plates | Corning | 3512 | |
| 4 mm Electroporation cuvettes | Bio-rad | 165-2081 | |
| X-cel gene pulser II | Bio-rad | 165-2661 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| 10× Phosphate buffered saline (PBS) pH7.4 | Life Technologies | 70011-044 | |
| Puromycin | Life Technologies | A11138-02 | |
| Pasteur pipettes, plugged | VWR | 14672-412 | |
| Tris-HCl | Sigma-Aldrich | S5941 | |
| NaCl | Sigma-Aldrich | S9888 | |
| SDS | Sigma-Aldrich | L3771 | |
| EDTA | Sigma-Aldrich | E5134 | |
| Proteinase-K | Life Technologies | AM2544 | |
| Ethanol | VWR | TX89125-172SFU | |
| Isopropyl Alcohol | VWR | MK303216 | |
| TE Buffer | Life Technologies | 12090-015 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| 1.8 ml Cryotubes | ThermoScientific | 377267 |
Riferimenti
- Carroll, D. Genome engineering with targetable nucleases. Annu Rev Biochem. 83, 409-439 (2014).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Matsa, E., Burridge, P. W., Wu, J. C. Human stem cells for modeling heart disease and for drug discovery. Sci Transl Med. 6 (239), 239ps6 (2014).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat Biotechnol. 27 (9), 851-857 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat Biotechnol. 29 (8), 731-734 (2011).
- Bozas, A., Beumer, K. J., Trautman, J. K., Carroll, D. Genetic analysis of zinc-finger nuclease-induced gene targeting in drosophila. Genetics. 182 (3), 641-651 (2009).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Urnov, F. D., et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature. 435 (7042), 646-651 (2005).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., Charpentier, E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jinek, M., East, A., Cheng, A., Lin, S., Ma, E., Doudna, J. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Doudna, J. A., Charpentier, E. Genome editing. the new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/cas systems. Science. 339 (6121), 819-823 (2013).
- Jasin, M. Genetic manipulation of genomes with rare-cutting endonucleases. Trends Genet. 12 (6), 224-228 (1996).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat Rev Genet. 11 (9), 636-646 (2010).
- Sander, J. D., Maeder, M. L., Reyon, D., Voytas, D. F., Joung, J. K., Dobbs, D. ZiFiT (zinc finger targeter): An updated zinc finger engineering tool. Nucleic Acids Res. 38, W462-W468 (2010).
- Carroll, D., Morton, J. J., Beumer, K. J., Segal, D. J. Design, construction and in vitro testing of zinc finger nucleases. Nat Protoc. 1 (3), 1329-1341 (2006).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39 (12), e82 (2011).
- Reyon, D., Tsai, S. Q., Khayter, C., Foden, J. A., Sander, J. D., Joung, J. K. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol. 30 (5), 460-465 (2012).
- Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng, G., Zhang, F. A transcription activator-like effector toolbox for genome engineering. Nat Protoc. 7 (1), 171-192 (2012).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from neisseria meningitidis. Proc Natl Acad Sci U S A. 110 (39), 15644-15649 (2013).
- Podhajska, A. J., Szybalski, W. Conversion of the FokI endonuclease to a universal restriction enzyme: Cleavage of phage M13mp7 DNA at predetermined sites. Gene. 40 (2-3), 175-182 (1985).
- Costa, M., et al. A method for genetic modification of human embryonic stem cells using electroporation. Nat Protoc. 2 (4), 792-796 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Cai, J., et al. Directed differentiation of human embryonic stem cells into functional hepatic cells. Hepatology. 45 (5), 1229-1239 (2007).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Soldner, F., et al. Parkinson's disease patient-derived induced pluripotent stem cells free of viral reprogramming factors. Cell. 136 (5), 964-977 (2009).
- Wen, Z., et al. Synaptic dysregulation in a human iPS cell model of mental disorders. Nature. 515 (7527), 414-418 (2014).
- Chiba, K., Hockemeyer, D. Genome editing in human pluripotent stem cells using site-specific nucleases. Methods Mol Biol. 1239, 267-280 (2015).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. J Vis Exp. , (2012).
- Ezashi, T., Das, P., Roberts, R. M. Low O2 tensions and the prevention of differentiation of hES cells. Proc Natl Acad Sci U S A. 102 (13), 4783-4788 (2005).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25 (18), 3745-3746 (1997).
- Southern, E. Southern blotting. Nat Protoc. 1 (2), 518-525 (2006).
- Henegariu, O., Heerema, N. A., Dlouhy, S. R., Vance, G. H., Vogt, P. H. Multiplex PCR: Critical parameters and step-by-step protocol. BioTechniques. 23 (3), 504-511 (1997).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Sexton, A. N., et al. Genetic and molecular identification of three human TPP1 functions in telomerase action: Recruitment, activation, and homeostasis set point regulation. Genes Dev. 28 (17), 1885-1899 (2014).
- Merkle, F. T., et al. Efficient CRISPR-Cas9-mediated generation of knockin human pluripotent stem cells lacking undesired mutations at the targeted locus. Cell Rep. 11 (6), 875-883 (2015).
- Soldner, F., et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset parkinson point mutations. Cell. 146 (2), 318-331 (2011).
- Forster, R., et al. Human intestinal tissue with adult stem cell properties derived from pluripotent stem cells. Stem Cell Reports. 2 (6), 838-852 (2014).
- Miller, J. C., et al. Improved specificity of TALE-based genome editing using an expanded RVD repertoire. Nat Methods. , (2015).
- Hohenstein, K. A., Pyle, A. D., Chern, J. Y., Lock, L. F., Donovan, P. J. Nucleofection mediates high-efficiency stable gene knockdown and transgene expression in human embryonic stem cells. Stem Cells. 26 (6), 1436-1443 (2008).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nat Biotechnol. 25 (6), 681-686 (2007).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4, 5400 (2014).
- Ousterout, D. G., Kabadi, A. M., Thakore, P. I., Majoros, W. H., Reddy, T. E., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome editing for correction of dystrophin mutations that cause duchenne muscular dystrophy. Nat Commun. 6, 6244 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon