A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הגדרת מצע ספציפי עבור מועמדי lipase ו פוספוליפאז
In This Article
Summary
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
Abstract
מיקרואורגניזמים לייצר קשת רחבה של (phospho) lipases כי מופרשים על מנת להפוך מצעים חיצוניים זמינים עבור האורגניזם. לחלופין, אחרים (phospho) lipases עשויה להיות קשורה פיזי עם האורגניזם הפקת גרימת מחזור של שומנים מהותיים ולעתים קרובות והוליד שיפוץ של קרום התא. למרות lipases פוטנציאל (phospho) ניתן לחזות עם מספר אלגוריתמים כאשר רצף הגן / חלבון זמין, הוכחה ניסיונית של פעילויות האנזים, ספציפיות המצע, תפקודים פיזיולוגיים פוטנציאל שתואר פעמים רבות לא ניתנה. כתב יד זה מתאר את אופטימיזציה של תנאי assay עבור פוטנציאליים (phospho) lipases עם ספציפיות מצע ידוע ואיך להעסיק בתנאים אופטימיזציה אלה בחיפוש אחר המצע הטבעי של lipase בהתאמה (phospho). באמצעות מצעים chromogenic מלאכותיים, כגון נגזרות -nitrophenyl p, עשוי לסייע לזהות קטיןהפעילות האנזימטית עבור lipase חזה (phospho) בתנאים סטנדרטיים. שנפגש כזה פעילות האנזימטית קטין, הפרמטרים הברורים של assay אנזים יכולים להיות מגוונים על מנת להשיג הידרוליזה יעיל יותר של המצע המלאכותי. לאחר משקבע את התנאים שבם אנזים עובד היטב, מגוון מצעים טבעיים פוטנציאליים צריך להיות assayed השפלה שלהם, תהליך שיכול להיות מלווה בשיטות chromatographic ברורות. ההגדרה ספציפית מצע אנזימים חדשים, לעתים קרובות מספקת השערות עבור תפקיד פיסיולוגי פוטנציאל של אנזימים אלה, אשר לאחר מכן ניתן לבדוק באופן ניסויי. בעקבות הנחיות אלו, הצלחנו לזהות C פוספוליפאז (SMc00171) כי מדרדרת phosphatidylcholine כדי phosphocholine ו diacylglycerol, בצעד מכריע על עיצובו מחדש של ממברנות בתוך meliloti חיידק Sinorhizobium בתנאים מגבילים-זרחן של צמיחה. במשך שני ניבאו patatin-כמו phospholipases (SMc00930 ו SMc01003) מאותו אורגניזם, נוכל להגדיר מחדש ספציפיות המצע שלהם ולהבהיר כי SMc01003 הוא lipase diacylglycerol.
Introduction
גליצרול מבוסס שומנים כגון triacylglycerols ו (glycero) פוספוליפידים מהווים חשובים וכנראה מעמדות השומנים הידועות ביותר -1. Triacylglycerols (תגיות) הם שומנים או שמנים, אשר בדרך כלל לתפקד כמו שומני אחסון, ולכן כמקורות אנרגיה ופחמן פוטנציאליים. תגיות יכולות להיות מושפלות על ידי lipases, אשר מופרשים לעתים קרובות על ידי האורגניזם הפיק לעכל תגיות חיצוניות ולהפוך אותם לזמינים כמקורות פחמן. כמו כן, lipases נחקר בהרחבה לאורך השנים בשל היישומים ביוטכנולוגיים החשובים שלהם 2.
בשל אופייה amphiphilic שלהם וצורה כמעט cylindric שלהם, (glycero) התערוכה פוספוליפידים נכסים מייצרי קרום מהווים בדרך כלל את הרכיבים lipidic העיקריים של קרום bilayered 3. בשנת מיקרואורגניזמים פשוטים, כגון coli Escherichia החיידק, רק שלוש גרסות קבוצת הראש גדולות, phosphatidylglycerol (PG), cardiolipin (CL), ו phosphatidylethanolamine (PE) הם נתקלו, אף אחד לא צריך להיות מודעים לכך כל אחד מהם ניתן להחליף עם מספר לא מבוטל של רשתות acyl השומן שונות בבית SN -1 או עלייה נותנת מיקום -2 SN למספר רב של מינים מולקולריים שונים 4 . חיידקים אחרים שאולי יש פוספוליפידים אחרים בנוסף או במקום. לדוגמה, Sinorhizobium meliloti, חיידק אדמה, אשר מסוגל ליצור סימביוזה גולה שורש תיקון-חנקן עם אספסת קטניות (Sativa Medicago), מכיל בנוסף PE A (PC) השני zwitterionic פוספוליפידים, phosphatidylcholine 5. כמו כן, שומנים לא המכילים זרחן או גליצרול יכול להיות חלק amphiphilic וצורה של הממברנה התאית. לדוגמה, על תנאי גידול הגבלת זרחן, ב ס meliloti, (glycero) פוספוליפידים מוחלפים בעיקר על ידי שומנים קרום שאינם מכילים זרחן, כלומר, sulfolipids, שומנים ornithine, ו diacylglyceryl trimethylhomoserine (DGTS) -6. אצל חיידקים, DGTS נוצר מן diacylglycerol (DAG) בתוך מסלול דו-שלבי 7 אבל המקור עבור הדור DAG לא היה ברור. ניסויים Pulse-מרדף הציע PC עשוי להיות סימן מקדים עבור 8 DGTS ושימוש המתודולוגיה המתוארת בכתב היד הזה נוכל לזהות C פוספוליפאז (PlcP, SMc00171) כי נוצר בתנאים מגבילים-זרחן אשר יכול להמיר המחשב לתוך דאג phosphocholine 8.
במחקר נפרד, גילינו כי synthetase acyl-CoA (FadD) מוטציה -deficient של ס meliloti או של Escherichia coli שנצבר חומצות שומן חופשיות כאשר נכנס לשלב נייח הצמיחה 9. למרות חומצות שומן אלה נראות להיגזר ליפידים קרום, את המקור המדויק של חומצות שומן חופשיות או האנזים (ים) משחרר אותם לא היו ידוע. שוב, העסקת האסטרטגיה שמתווה בכתב היד הזה, שתי 10 patatin הדמוי (phospho) lipases (SMc00930 ואת SMc01003) שתרם להיווצרות של חומצות שומן חופשיות ב ס meliloti 11 נחזה. באופן מפתיע, SMc01003 בשימוש DAG כמו המצע להפוך אותו monoacylglycerol ולבסוף גליצרול וחומצות שומן חופשיות 11. לכן SMc01003 הוא lipase DAG (DglA).
למרות מספר אלגוריתמים קיימים לניבוי פוטנציאל (phospho) lipases 12,13, תפקידו המדויק שלהם תפקיד פיזיולוגי הוא בדרך כלל לא ידוע. כאן אנו מתארים פרוטוקול, לשבט overexpress חזה או פוטנציאל (phospho) lipases. כתב יד זה מסביר כיצד מבחני אנזים ניתן לפתח והותאמו במיוחד lipase (phospho) overexpressed באמצעות מצעי chromogenic מלאכותיים. אנו מספקים דוגמאות כיצד עם assay האנזים אופטימיזציה המצע (phospho) lipase אמיתי ניתן נתקל ואיך ממצאים אלו יכולים להיות להעשיר את הבנתנו בפיזיולוגיה של חיידקים.
Protocol
Clone 1. overexpress מבנית ג'ין עבור חזוי lipase
- באמצעות תגובת שרשרת של פולימראז (PCR) 14 ו oligonucleotides הספציפי (טבלה 1) 15, להגביר את הגן של עניין (smc01003, smc00930, או smc00171), חזה קוד עבור lipase או פוספוליפאז, מן הדנ"א הגנומי של האורגניזם המארח (כלומר , ס meliloti).
- להציג אתרי הגבלה ספציפיים (עם הרצף המתוכנן של oligonucleotides). לעכל את קטע DNA המוגבר עם אנזימי הגבלה המקביל ולשכפל אותו לתוך וקטור ביטוי כגון פלסמידים של סדרת PET 16.
- לאחר וידוי רצף ה- DNA הנכון עבור גנים משובטים, להפוך את וקטור זן ביטוי כגון BL21 Escherichia coli (DE3) pLysS 16.
- כן-תרבות מראש לילה של מארח הביטוי E. coli BL21 (DE3) PLysS, מחסה וקטור PET בהתאמה עם הגן המשובט או הווקטור הריק, ב 100 מ"ל צלוחיות תרבות המכילות 20 מיליליטר של מרק לוריא Bertani (LB) 17 בתוספת האנטיביוטיקה הנדרשת. התרבות התאים על 30 מעלות צלזיוס (או בטמפרטורה הצמיחה הרגיל של החיידק שממנו lipase מקורו).
- שימוש-תרבויות קדם לילה, לחסן 500 מ"ל של מדיום LB prewarmed (בתוספת אנטיביוטיקה חובה) צלוחיות תרבות 2 L להשיג צפיפות אופטית הראשונית ב 620 ננומטר (OD 620) 0.05 =. עקוב צמיחה של תרבויות ותמורת OD 620 = 0.3, להוסיף איזופרופיל- β-D-thiogalactoside (IPTG) לריכוז סופי של 100 מיקרומטר, דגירה תחת תסיסה על 30 מעלות צלזיוס למשך תקופה של 4 שעות.
- בסוף תקופת הדגירה, להעביר כל תרבות לצינור צנטריפוגות 500 מ"ל ו צנטריפוגות ב 5000 XG ב 4 מעלות צלזיוס למשך 30 דקות. Resuspend כדורי תא החיידק ב 5 מ"ל של חיץ ההשעיה (למשל, SMc00930- ואת SMc01003 לבטא בתאי ב 50 mM Tris-HCl pH 8.0 ו SMc00171 לבטא בתאי ב 50 מ"מ-Diethanolamine HCl pH 9.8). אחסן את המתלים תא ב -80 ° C עד השימוש.
2. כן תמציות חלבון ללא סלולריים לקבוע ריכוז חלבון
- הפשר השעיות תא חיידק ולאחסן על קרח. Pass השעיות תא שלוש פעמים באמצעות תא לחץ קר ב -20,000 ליברות לכל ב 2. הסר תאים שלמים ופסולת התא על ידי צנטריפוגה ב 5000 XG במשך 30 דקות ב 4 ° C.
- לאחר צנטריפוגה, להכין aliquots של 100 ו 500 μl מן supernatant לניתוח שלאחר מכן ולאחסן אותם ב -80 ° C עד השימוש.
- השתמש באחת aliquot 100 μl כדי לקבוע ריכוז חלבון של תמציות תא ללא מובחנת על ידי שיטת הבחירה או כמתואר 18.
3. מצעים מלאכותיים השתמש עבור אנזים אופטימיזציהפעילויות של (phospho) lipases
- לקבלת כיסוי ראשוני של פעילויות אנזים ברורות, להשתמש מצעים מלאכותיים להניב מוצר בצבע על הידרוליזה, כגון p -nitrophenol (-NP p).
- עבור מבחני אנזים מותאמים כבר עם מצעי אסתר -nitrophenyl הנדסאית (מזדקרים על פוספוליפאז C PlcP (SMc00171), כמו גם עבור phospholipases patatin דמוי חזה SMc00930 ו SMc01003), תוכניות pipetting שימוש מתוארות בטבלה 2.
- במהלך סיור lipase פוטנציאליים חדשים (phospho), להכין assay האנזים תחילה רגיל המכיל 50 mM Tris-HCl, pH 8.5, 100 מ"מ NaCl, 0.05% Triton X-100, 0.5 מ"מ p -nitrophenyl המכילים תרכובת (פוספט -nitrophenyl p , פוספט p -nitrophenyl בשדרה, decanoate p -nitrophenyl, או palmitate p -nitrophenyl), ותמצית חלבון ללא תאים (לבדוק 1, 3, 10, 30, 100, 300, ו -1,000 מיקרוגרם) בהיקף כולל של 1 מ"ל ב 1 מ"ל cuve פלסטיקttes.
הערה: pH בסיסי השתמש (איור 1) כאשר בעקבות הידרוליזה אסתר p -nitrophenyl ב assay רציפה. לחלופין, להשתמש מבחני זמן-נקודה אחת עבור מגוון של ערכי ה- pH, הוספת NaOH בסוף תקופת הדגירה כדי לסיים את התגובה אנזים ועל מנת להבטיח כי כל -NP p הוא להציג בצורה phenolate. - בצע את מהלך הזמן עבור גידול של ספיגה ב 405 ננומטר, עקב ההיווצרות של -NP p, ספקטרופוטומטר על 30 מעלות צלזיוס במשך 5 דקות. לכמת את למבנה הישר בהתחלה של p -NP ידי קביעת המדרון הראשוני של גידול של ספיגה לכל זמן.
- חשב את השינוי של ריכוז (Δc) עבור -NP p באמצעות חוק למברט-באר (ΔA = ε Δc ד) 1.
הערה: ΔA הוא שינוי לינארי של ספיגת נקבע, ε הוא מקדם הכחדה טוחנת על אורך גל, בהתאמה (ביחידות של M -1 -1 ס"מ), d הוא אורכו של נתיב האור (1 ס"מ), ו Δc הוא השינוי של ריכוז (ביחידות של M) שייקבע.- בהתחשב בכך נפח assay הוא 1 מ"ל, לחשב את כמות p -NP נוצר.
הערה: סכום = ריכוז נפח x. - חשב את פעילות האנזים על ידי חלוקת סכום p -NP נוצר על ידי הזמן שבו הוא נוצר. קבע את פעילות אנזים מסוים על ידי חלוקת פעילות האנזים על ידי כמות חלבון (במ"ג) שהייתה אחראית על יצירת פעילות זו.
- בהתחשב בכך נפח assay הוא 1 מ"ל, לחשב את כמות p -NP נוצר.
- השווה את השינויים ספיגים שעוררו תמציות חלבון בו גן מועמד (smc00171, smc00930, או smc01003) כבר הובע עם תמציות כי הנמל רק וקטור ריק.
הערה: על מנת להמשיך בביצוע השלבים הבאים, הפעילויות הספציפיות, הנגרמות על ידי תמציות חלבון בו גן מועמד כבר הובע, צריכות להיות לפחות פעמים או מ 'בצר מ הערכים שהתקבלו עבור פעילויות ספציפיות הנובעות מכך תמציות חלבון כי הנמל רק וקטור ריק. - עבור ניסויים נוספים, לבחור תנאים בם הידרוליזה של המתחם p -nitrophenyl המכיל הוא מינימאלי עם תמציות תא חינם (כלומר, וקטור ריק) ואשר ההיווצרות הבולטת של -NP p ואת אניון p -nitrophenolate (איור 1) ניתן להבחין כאשר תמציות חלבון מועסקים, בו גן מועמד כבר הובע.
- לאחר קביעת פעילות אנזים הראשונית 3.1, לייעל את התנאים assay עבור האנזים בהתאמה על ידי שינוי pH, סוג של חיץ, חיץ כוח, ריכוזים של NaCl, דטרגנטים כגון טריטון X-100, והעדר או נוכחות של קטיונים דו ערכי שונה.
- עבור ריכוזים שונים של כל משתנה, קבע את פעילות אנזים מסוים (ראה 3.1.4.2) (המספר הגבוה ביותר שהושג מגדיר את התנאיפעילות האנזים מקסימלי). השתמש שילוב של התנאים האופטימליים נתקלו לכל משתנה להגדיר assay אנזים אופטימיזציה שבו כל משתנה הוא מצוי בריכוז האופטימלי שלה.
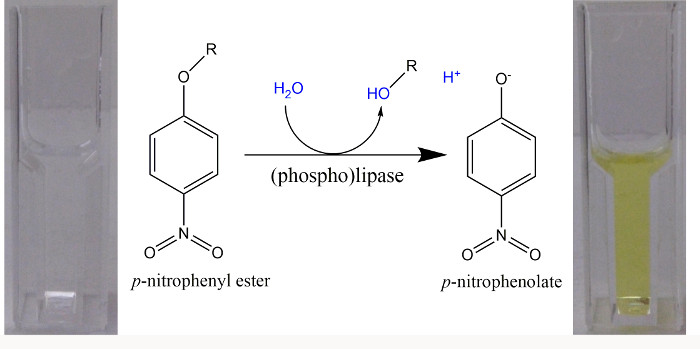
איור 1. אסטרים -Nitrophenyl p כמו מצעים מלאכותיים (phospho) lipases ב assay spectrophotometric. לאחר הידרוליזה של אסטרים p -nitrophenyl, חומצת (R-OH) ו- p -nitrophenol (-NP p) נוצרות. בשל א PK = 7.2 עבור ניתוק של + פנוליות H מ- p -NP, ב- pH> 9.2 יותר מ -99% הם בצורת p -nitrophenolate צהוב בהיר מקדם הכחדה טוחנת של 18,000 M -1 ס"מ - 1 ניתן להשתמש בכל אורך גל של 405 ננומטר עבור כימות של -nitrophenolate p חינם 22. כאשר מאגרים עם pH של 8.5 שימשו, ספיגת נקבע ב 400 ננומטר ו מקדם הכחדה טוחנת של 14,500 M -1 cm -1 הועסק 23. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
הערה: לאחר לאחר שהגדיר את התנאים האופטימליים עבור הפעילות של האנזים של עניין, לצאת בחיפוש אחר המצע האמיתי / פיסיולוגי של lipase זה. באופן עקרוני, לקחת שני, לעתים קרובות משלימים, גישות להשגת מטרה זו, גישה vivo או בגישה חוץ גופית.
4. זיהוי Vivo של המצע הפיזיולוגי של lipase
ove_content "> הערה:. בגישת in vivo, לבטא את lipase של עניין פונדקאי 8,11 כדי לרשום לאורך זמן אם ביטוי של lipase משנה את פרופיל שומני host's בעוד גישת in vivo, ליצור מוטציה הלקויה של הגן של עניין 8,11 וללמוד אם פרופיל השומנים שלה נבדל מגרסת סוג בר 6,8,11. על מנת לקבל הערכה כמותית של פרופיל השומנים של האורגניזם, שיטה פשוטה מורכבת radiolabeling תרכובות הסלולר , חילוץ שומנים, תוך הפרדתם ידי כרומטוגרפיה, וכימות שומנים מופרדים שכותרתו רדיואקטיבית.- Radiolabeling של שומנים.
- הכן-תרבות מראש לילה של אורגניזם של עניין (E. coli או ס meliloti) ב 5 מ"ל של מדיום תרבות הרצוי (בינוני מורכב או בינוני מינימלית מוגדרת) ולצמוח בקצב של 30 ° C.
- מתוך תרבות מראש, לחסן לתוך 20 מ"ל של saלי בינוני טרי בבקבוק תרבות 100 מ"ל להשיג OD הראשונית 620 = 0.3 עבור התרבות.
- קח aliquot (1 מ"ל) של התרבות בתנאים סטריליים ולהעביר לצינור מסביב לתחתית פוליסטירן 14 מ"ל סטרילי.
- הוסף 1 μCi של [1- 14 C] אצטט (60 MCI לכל מילימול) לתרבות 1 מ"ל.
- דגירת ההתרבות הנוזלת תחת תסיסה על 30 מעלות צלזיוס למשך תקופה של 24 שעות.
- בסוף תקופת הדגירה, ולהעביר את התרבות לצינור microcentrifuge 1.5 מ"ל ו צנטריפוגות ב XG 12,000 בטמפרטורת החדר למשך 5 דקות.
- Resuspend גלולה ב 100 μl של מים. בשלב זה, לאחסן את ההשעיה תא ב -20 ° C או מיד להמשיך עם החילוץ של שומנים קוטביים (סעיף 4.2).
- הפקת שומנים קוטביים.
הערה: השיטה המתוארת כאן בעצם מלווה את ההליך שדווח על ידי בליי דאייר 19.- אל 100 μl של תליוני תא מימייםension, להוסיף 375 μl של מתנול: פתרון כלורופורם (2: 1; כרך / כרך).
- וורטקס למשך 30 שניות ו דגירה במשך 5 דקות בטמפרטורת החדר.
- צנטריפוגה 5 דקות ב XG 12,000 בטמפרטורת החדר.
- מעבירים את supernatant לצינור microcentrifuge 1.5 מ"ל חדש.
- להוסיף 125 μl של כלורופורם 125 μl של מים, מערבולת 30 שניות.
- צנטריפוגה 1 דקות ב XG 12,000 בטמפרטורת החדר.
- מעביר את שלב כלורופורם התחתון לצינור טרי ויבש עם זרם של גז חנקן.
- ממסי שומנים מיובשים 100 μl של כלורופורם: פתרון מתנול (1: 1; כרך / כרך).
הערה: בשלב זה, aliquot של 5 μl של פתרון השומנים ניתן לכמת ידי ספירת נצנץ נוזלית. - לקבלת chromatographic-שכבה דקה (TLC) ניתוח, לייבש את 95 μl הנותרים עם זרם של גז חנקן redissolve שומנים מיובש 20 μl של כלורופורם: פתרון מתנול (1: 1; כרך / כרך). השתמש aliquot 3 μlלניתוח TLC.
- הפרדת שומנים קוטביים ידי כרומטוגרפיה בשכבה דקה (TLC).
הערה: בהתאם מעמדות השומנים להיות מנותח, שילובים שונים של שלבים מוצקים ניידים עשויים להיות מועסקים על הפרדה. כאן הפרדה אופיינית שומנים קוטביים טעונה אחרת, מתאימים יותר שומני קוטב ניטראליים, באמצעות כרומטוגרפיה בשכבה דקה ביצועים גבוהים (HPTLC) פחי אלומיניום ג'ל סיליקה כשלב המוצק, מתוארות.- הפרדת שומנים קוטבי שגובים דו מימדי TLC (2D-TLC).
- למרוח aliquot 3 μl של מדגם השומנים באחת הפינות של גיליון אלומיניום ג'ל סיליקה HPTLC (10 x 10 ס"מ), 2 ס"מ מהקצה של הצלחת.
- כן ומערבבים בשלב הנייד (כלורופורם 140 מיליליטר, 60 מיליליטר מתנול, ו -10 מיליליטר מים) להפרדה בממד הראשון.
- מעיל בתא בפיתוח TLC פנימי עם נייר כרומטוגרפיה.
הערה: זו היא להבטיח כי שלב הגז של התאיהיה רוויים במהירות (תוך 30 דקות) לאחר השלב הנייד עבור הממד הראשון נוסף התא והתא נסגר עם צלחת זכוכית. - כן ומערבבים בשלב הנייד (כלורופורם 130 מיליליטר, 50 מיליליטר מתנול, ו -20 מיליליטר קרחוני חומצה אצטית) להפרדה בממד והעברת השני אל חדר בפיתוח שני TLC מצופה פנימי עם נייר כרומטוגרפיה ולתת הרווי הקאמרי.
- להעביר בזהירות את סדין ג'ל אלומיניום HPTLC סיליקה עם מדגם שומנים היבש לתא הראשון ולפתח (כלומר, לבצע כרומטוגרפיה) את הצלחת במשך 60 דקות בחדר הסגור בממד הראשון 5.
- הסר את הצלחת מן החדר ולתת ממסים להתנגב במנדף זרימה למשך 30 דקות.
- לאחר סיבוב צלחת ב -90 מעלות ביחס כרומטוגרפיה הקודמת, להעביר את גיליון אלומיניום ג'ל סיליקה HPTLC, שעליו שומנים שהופרדו במימד אחד, אל SECONתא ד ולפתח את הצלחת במשך 60 דקות בממד השני 5.
- הסר את הגיליון מהאולם ולתת ממיסים להתנגב במנדף זרימה במשך שעה לפחות 2.
- הפרדת שומנים קוטב נייטרלי.
- החל 3 aliquots μl של דגימות השומנים על גיליון אלומיניום ג'ל סיליקה HPTLC החל 2 ס"מ מקצות הצלחת. אם דוגמאות רבות מנותחות בתוך כרומטוגרפיה חד ממדי, לשמור על מרחק של 1.5 סנטימטרים לפחות בין כתמי יישום מדגם השונים.
- הכן ומערבבים בשלב ניידים (140 מ"ל הקסאן, 60 מ"ל דיאתיל אתר, ו -8 מ"ל חומצה אצטית) והעברה בתא בפיתוח TLC פנימי מצופה נייר כרומטוגרפיה מכוסה בצלחת זכוכית לתת רווי קאמרית (30 דק ').
- מעביר את גיליון אלומיניום סיליקה ג'ל HPTLC עם דגימות שומנים היבשות לתא ולפתח את הצלחת למשך 30 דקות בחדר הסגור.
- הסר את הצלחת מן החדרולתת ממיסים להתנגב במנדף זרימה עבור שעה 2.
- הפרדת שומנים קוטבי שגובים דו מימדי TLC (2D-TLC).
- כימות וויזואליזציה של שומנים קוטביים מופרדים.
- לאחר גיליון TLC פתח יבש, דגירה אותו עם הארת photostimulable (PSL) מסך קסטה סגורה במשך 3 ימים.
- לחשוף את המסך וטופח על סורק PSL ולרכוש תמונה וירטואלית של שומני radiolabeled המופרדים.
- בצע כימות באמצעות PSL תוכנה 20.
- ויזואליזציה ובידוד של כיתות שומנים קוטביים פרט.
- דגירת גיליון TLC מפותח במשך 10 דקות בתא כרומטוגרפיה בנוכחות 1 גרם של גבישי יוד.
הערה: תרכובות lipidic פרוד יהיה לפזר את יוד מופיעים כתמים חומים. - הקף את הנקודות בעיפרון, ולהשוות אותם ניידות יחסית (f R) של תרכובות סטנדרטי (כלומר, 1,2-dipalmitoyl- SN -glycerol, dipalmitoyl-L-α-phosphatidylcholine, DL-α-monopalmitin, או חומצה פלמיטית), ולזהות שאליו בכיתת שומנים הוא שייך.
- במנדף, לתת יוד להתאדות מגיליון TLC.
- בעזרת מרית, לגרד את ג'ל סיליקה המכיל את המתחם של עניין מן הגיליון, ולחלץ את המתחם מן הג'ל סיליקה בתערובת של 100 μl של מים ו 375 μl של מתנול: פתרון כלורופורם (2: 1; כרך / כרך).
- המשך עם מיצוי פי בליי דייר כפי שמתואר (4.2.2 ואילך).
- חנות בכיתה השומנים מטוהרים 100 μl של כלורופורם: פתרון מתנול (1: 1; כרך / כרך) ב -20 ° C עד השימוש.
- דגירת גיליון TLC מפותח במשך 10 דקות בתא כרומטוגרפיה בנוכחות 1 גרם של גבישי יוד.
5. זיהוי חוץ-גופייה של המצע הפיזיולוגי של lipase
הערה: גישה במבחנה, לחקור האם lipase של עניין ניתן להמיר תערובת של ליפידים מבודד או שומנים טהורים פרט אל hydrol המקבילמוצרים יסיס בתנאים המוגדרים אופטימלי 3.2.
- להשתמש בצירופי pipetting עבור מבחני אנזים לפי טבלה 3 עבור פעילות המחשב הספציפי פוספוליפאז C SMc00171 (ראה 5.2), פוספוליפאז A (ראה 5.3), ואת דאג lipase SMc01003 (ראה 5.4).
- קביעת פעילות C פוספוליפאז PC ספציפי (לוח 3).
- כדי צינור 1.5 מ"ל microcentrifuge, להוסיף 5,000 ספירות לדקה (CPM) בסך הכל 14 PC C שכותרתו ופתרון של טריטון X-100.
- מערבבים ויבש תחת זרם של חנקן.
- להוסיף Diethanolamine HCl, pH 9.8 חיץ, כמו גם NaCl ו MnCl 2 פתרונות ומים bidistilled להשיג נפח סופי של 99.5 μl. וורטקס למשך 5 שניות.
- הוסף 0.5 μl של אנזים (5 מיקרוגרם חלבון) (כלומר, תמצית חינם תאים בהם ביתר SMc00171 קיים) כדי ליזום את התגובה. מערבבים בקצרה.
- לדגור על 30 מעלות צלזיוס למשך 4 שעות.
- עצור את התגובה על ידיתוספת של 250 μl של מתנול 125 μl של כלורופורם.
- חלץ ליפידים כפי שתואר לעיל (ראה 4.2).
- שומנים נפרדים על ידי חד-ממדי (1D) -TLC (ראו 4.3.2 ו -4.4), ולנתח אותם על ידי הדמית PSL.
- קביעת פעילות פוספוליפאז (לוח 3).
- כדי צינור 1.5 מ"ל microcentrifuge, להוסיף 5,000 עותקים לדקה בסך הכל 14 פוספוליפידים C שכותרתו ופתרון של טריטון X-100.
- מערבבים ויבש תחת זרם של חנקן.
- עבור assay 100 μl הסופי, להוסיף Tris-HCl, חיץ pH 8.5, פתרון NaCl ומים. וורטקס למשך 5 שניות.
- הוסף 5 μl של אנזים (50 מיקרוגרם חלבון) (כלומר, תמצית חינם תאים בהם ביתר SMc00930 או SMc01003 קיים).
- לדגור על 30 מעלות צלזיוס למשך 5 שעות.
- עצור את התגובה על ידי תוספת של 250 μl של מתנול 125 μl של כלורופורם.
- חלץ ליפידים כפי שתואר לעיל (ראה 4.2), נפרדאותם על ידי 1D-TLC באמצעות כלורופורם 130 מ"ל, 50 מ"ל מתנול, וחומצה אצטית קרחונית 20 מ"ל כשלב ניידים, ולנתח אותם על ידי הדמיה PSL.
- קביעת diacylglycerol (DAG) פעילות lipase.
- הכנת 14 C שכותרתו DAG.
- Radiolabel ס תרבויות meliloti (ראו 4.1) ולחלץ שומנים קוטביים (ראה 4.2) כמתואר. ס נפרד תמציות שומנים הכוללות meliloti ידי 1D-TLC כלורופורם: מתנול: חומצה אצטית (130: 50: 20; כרך / כרך) באמצעות בתנאים המתוארים להפרדה בממד השני ב 4.3.1.
- דמיין PC ידי מכתים יוד להשתמש בעיפרון כדי לסמן את הלוקליזציה של phosphatidylcholine (PC).
- לבודד PC radiolabeled כמתואר 4.5.
- לכמת PC שחולץ על ידי ספירת נצנץ.
הערה: על 320,000 PC לדקה צפויה. - פנקו את המחשב (250,000 עותקים לדקה) עם 0.1 U של C פוספוליפאז מ perfringens Clostridium ב 50 mM Tris-HCl, pH 7.2, 0.5טריטון% X-100 לבין 10 מ"מ CaCl 2 עבור שעה 2 בנפח כולל של 100 μl ולעצור את התגובה על ידי הוספת 250 μl של מתנול 125 μl של כלורופורם.
- חלץ ליפידים כפי שתואר לעיל ונפרדת על ידם על ידי 1D-TLC (ראה 4.3.2).
- לבודד diacylglycerol מצלחת סיליקה ולכמת ידי ספירת נצנץ (כמתואר 4.2)
- Assay lipase Diacylglycerol (לוח 3).
- כדי צינור 1.5 מ"ל microcentrifuge, להוסיף 5,000 עותקים לדקה של 14 C-שכותרתו DAG ופתרון של טריטון X-100.
- מערבבים ויבש תחת זרם של חנקן.
- עבור assay 100 μl הסופי, להוסיף Tris-HCl (9.0 pH) חיץ, פתרון NaCl ומים bidistilled. וורטקס למשך 5 שניות.
- ליזום את התגובה על ידי הוספת 5 μl של אנזים (50 מיקרוגרם חלבון של תמצית התא ללא).
- לדגור על 30 מעלות צלזיוס למשך 4 שעות.
- עצור את התגובה על ידי תוספת של 250 μl של מתנולד 125 μl של שומנים כלורופורם לחלץ כפי שתואר לעיל (ראה 4.2).
- לנתח שומנים קוטב נייטרלי ידי 1D-TLC (ראה 4.1.3.2) והדמיה PSL שלאחר מכן.
- הכנת 14 C שכותרתו DAG.
תוצאות
פעילות של פוספוליפאז PC ספציפי C SMc00171 עם בשדרת p -nitrophenyl פוספט
תמציות תא ללא המתקבל E. BL21 coli (DE3) x pLysS, אשר הביעו smc00171, נחקרו על יכולתם hydrolyze אסטרים פוספ?...
Discussion
במהלך 20 השנים האחרונות, גנומים של אורגניזמים רבים היו רצף ועל אף נתוני רצף עושר של הגנום נוצר, פירוש פונקציונלי מפגר אחרי ולכן מעכב את הבנתנו הפונקציה הגנום. ג'ין פונקציות הגנומים לעתים קרובות מוקצות בהתבסס על דמיון לגנים של תפקוד או תופעה הידועות של מוטיבים נשמרים...
Disclosures
החוקרים אין לי מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי מענקי Consejo Nacional de Ciencias y Tecnología-מקסיקו (CONACyT-מקסיקו) (82,614, 153,998, 253,549, ו 178,359 ב Investigación Científica BASICA וכן 118 Investigación en Fronteras de la Ciencia) ומן Dirección הגנרל דה Asuntos דה אישי Académico-Universidad הלאומית האוטונומית דה מקסיקו (DGAPA-UNAM; PAPIIT IN202616, IN203612).
Materials
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
References
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved