このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
リパーゼ及びホスホリパーゼ候補者のための基質特異性を定義します
要約
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
要約
微生物は、生物のための外部の基板を利用できるようにするために分泌される(ホスホ)リパーゼの広いスペクトルを生成します。代替的に、他の(ホスホ)リパーゼは、物理的に産生する生物は、内因性の脂質の代謝回転を引き起こし、しばしば細胞膜のリモデリングを生じると関連付けることができます。電位(ホスホ)リパーゼは、遺伝子/タンパク質配列が利用可能である多くのアルゴリズムを用いて予測することができますが、酵素活性、基質特異性、および潜在的な生理機能の実験的証明は頻繁に得られていません。本稿では、未知の基質特異性とどのようにそれぞれの(ホスホ)リパーゼの天然基質の検索でこれらの最適化された条件を採用することで将来の(ホスホ)リパーゼのためのアッセイ条件の最適化について説明します。このようなp個の -nitrophenyl誘導体のような人工的な発色基質を、使用して、マイナーを検出するのを助けることができます標準的な条件下での予測(ホスホ)リパーゼのための酵素活性。このようなマイナーな酵素活性を検出しましたが、酵素アッセイの異なるパラメータは、人工基質のより効率的な加水分解を得るために変えることができます。酵素がうまく機能する条件を決定した後、潜在自然種々の基材は、その分解、明確なクロマトグラフィー法を用いて追跡することができるプロセスについてアッセイされるべきです。新しい酵素の基質特異性の定義は、頻繁にして実験的に試験することができ、これらの酵素の潜在的な生理的役割のための仮説を提供します。これらのガイドラインに続いて、我々は成長のリン制限条件に菌シノリゾビウム・メリロッティ内膜のリモデリングのための重要なステップで、ホスホコリンおよびジアシルグリセロールするホスファチジルコリンを分解するホスホリパーゼC(SMc00171)を識別することができました。 2についてpatatin-を予測同じ生物のホスホリパーゼ(SMc00930とSMc01003)のように、我々はそれらの基質特異性を再定義し、SMc01003は、ジアシルグリセロールリパーゼであることを明らかにすることができました。
概要
このようなトリアシルグリセロールおよび(グリセロ)リン脂質のグリセロールベースの脂質は、重要な、おそらく最もよく知られている脂質クラス1を構成しています。トリアシルグリセロール(タグは)したがって、ポテンシャルエネルギーや炭素源として油脂、通常、貯蔵脂質として機能する、としています。タグは、頻繁に外部タグを消化し、炭素源としてそれらを使用できるようにするために産生する生物によって分泌されるリパーゼによって分解することができます。また、リパーゼは広く、それらの重要なバイオテクノロジー用途2に長年にわたって研究されてきました。
それらの両親媒性の性質とその近円筒形状、(グリセロ)リン脂質を呈する膜形成特性と、通常二層膜3の主要な脂質成分を構成します。このような細菌、大腸菌 、3つだけの主要な頭部基変種、ホスファチジルグリセロール(PG)、カルジオリピン(CL)、およびphosphatidのような単純な微生物では、一つは、それらの一つ一つがかなりのSNで異なる脂肪酸アシル鎖の数-1またはSNで置換することができることに注意する必要がありますが、ylethanolamine(PE)は、検出された-2位置が異なる分子種4の多数を生じさせます。他の細菌は、他のリン脂質に加えて、またはその代わりを持っている可能性があります。例えば、マメ科植物アルファルファ( アルファルファ )と窒素固定根粒共生を形成することが可能であるシノリゾビウム・メリロティ 、土壌細菌は、第二の両性イオン性リン脂質をPEに加えて含んで、ホスファチジルコリン(PC)5。また、リンまたはグリセロールを含まない脂質は両親媒性であると細胞膜の一部を形成するかもしれません。例えば、リン制限増殖条件時、S.でメリロティは 、(グリセロ)リン脂質は、主としてリンを含まない膜脂質、 すなわち、スルホ脂質、オルニチン脂質、およびdiacylglyceryl trimethylhoによって置き換えられていますmoserine(DGTS)6。細菌では、DGTSは2段階経路7にジアシルグリセロール(DAG)から形成されたが、DAGの生成のためのソースが明確ではなかったされています。パルスチェイス実験は、PCは、我々はリン制限条件下で形成され、DAGとホスホコリンにPCを変換できるC(PLCP、SMc00171)ホスホリパーゼを識別することができDGTS 8の前駆体であることと、この原稿に記載の方法を用いて、可能性を示唆しました8。
別の研究では、我々はそのアシルCoAシンテターゼ(FADD)Sの欠損変異体を発見しました成長9の定常期に入るとき根粒かの大腸菌は、遊離脂肪酸を蓄積しました。これらの脂肪酸は、膜脂質に由来すると思われたが、遊離脂肪酸またはそれらの遊離酵素(群)の正確な供給源が知られていませんでした。ここでも、(、2パタチンのような10(ホスホ)リパーゼを、この原稿で概説した戦略を採用S.中の遊離脂肪酸の形成に寄与しSMc00930とSMc01003) 根粒 11は、予測されました。驚くべきことに、SMc01003は、モノアシルグリセロール、最終的にはグリセロールと遊離脂肪酸11に変換する基板としてDAGを使用しました。したがって、SMc01003はDAGリパーゼ(DGLA)です。
アルゴリズムの数を潜在的(リン)を予測するために存在するが12,13は 、リパーゼ、それらの正確な機能と生理的役割は、通常知られていません。ここでは、クローンを作成する、プロトコルの概要を説明し、予測又は電位(ホスホ)リパーゼを過剰発現します。この原稿は、酵素アッセイを開発し、人工的な発色基質を使用することにより過剰発現(ホスホ)リパーゼのために最適化する方法について説明します。私たちは、最適化された酵素アッセイで、実際の(ホスホ)リパーゼ基質に遭遇することができ、どのようにこれらの知見は、微生物生理学の我々の理解を豊かにする方法の例を提供します。
Access restricted. Please log in or start a trial to view this content.
プロトコル
予測リパーゼ1.クローンおよび過剰発現する構造遺伝子
- ポリメラーゼ連鎖反応(PCR)14と特異的オリゴヌクレオチド( 表1)15 を用いて、目的の遺伝子(smc01003、smc00930、またはsmc00171)を増幅、宿主生物( すなわち、ゲノムDNAから、リパーゼ又はホスホリパーゼをコードすると予測さ、S.メリロティ )。
- (オリゴヌクレオチドの設計された配列を持つ)特定の制限部位を導入します。対応する制限酵素で増幅されたDNA断片を消化し、例えば、PET系16のプラスミドのような発現ベクター中にクローニングします。
- クローニングされた遺伝子の正しいDNA配列を確認した後、このような大腸菌 BL21(DE3)pLysSを16として発現株にベクトルを変換します。
- 発現宿主大腸菌の一晩前培養を準備大腸菌 BL21(DE3)ピコリットルYSSは、100ミリリットル培養ルリア・ベルターニブロス(LB)17の20ミリリットルを含むフラスコに加え、必要な抗生物質には、クローニングされた遺伝子または空ベクターを有するそれぞれのpETベクターを保有します。文化30℃で細胞(またはリパーゼは、元となる菌の通常の成長温度で)。
- 一晩前培養物を使用して、620nmで(OD 620)での最初の光学濃度を得るために2 L培養フラスコ中で予熱LB培地(プラス必要な抗生物質)を500mlに接種= 0.05。 、文化のとOD 620 = 0.3での成長に従って、100μMの最終濃度になるようにイソプロピルβ-D-チオガラクトシド(IPTG)を追加し、4時間の期間にわたって30℃で撹拌下でインキュベートします。
- インキュベーション期間の終わりに、30分間、4℃で5,000×gで500mLの遠心管遠心各培養物を移します。懸濁緩衝液5ml中の細菌細胞ペレットを再懸濁し( 例えば 、SMc00930-及び50mMジエタノールアミン塩酸pHは9.8)で50mMのトリス-HCl 8.0 SMc00171発現細胞で細胞をSMc01003が発現します。使用するまで-80℃で細胞懸濁液を保管してください。
2.無細胞タンパク質抽出物を調製し、タンパク質濃度を決定します
- 細菌の細胞懸濁液を解凍し、氷上で保存します。 2で20,000ポンドで冷間圧力セルを介して細胞懸濁液を3回渡します。 4℃で30分間5,000×gで遠心分離することによって、無傷細胞及び細胞片を除去します。
- 遠心分離した後、その後の分析のために上清から100と500μlのアリコートを準備し、使用するまで-80℃で保管してください。
- 選択の方法により、または18に記載のように別個の無細胞抽出物のタンパク質濃度を決定するために、100μlのアリコートのいずれかを使用します。
最適化酵素のため3.人工基質(リン酸化)リパーゼの活動
- 明確な酵素活性の初期の報道について、(P -NP)などのpニトロフェノールなどの加水分解により着色生成物を得た人工基質を、使用しています。
- 人工のp -nitrophenylエステル基質で既に最適化された酵素アッセイ(ホスホリパーゼC PLCP(SMc00171)のためだけでなく、予測パタチンのようなホスホリパーゼSMc00930とSMc01003について概説)は、 表2に記載のピペットスキームを使用します。
- 新たな可能性(ホスホ)リパーゼを探索する際に、50mMのトリス-HCl、pH8.5で、100mMのNaCl、0.05%トリトンX-100、0.5mMのpが -nitrophenyl含有化合物(p個の -nitrophenylリン酸を含有する第一の標準酵素アッセイを準備、p個の -nitrophenylリン酸、Pの -nitrophenylデカン酸、 またはP -nitrophenylパルミチン酸)ビス- 、および無細胞タンパク質抽出物(1、3、10、30、100、300をチェックし、1,000μgの1)の全量1ミリリットル中ミリリットルプラスチック醸造おけTTES。
注:連続検定におけるpの -nitrophenylエステル加水分解後の使用にアルカリ性のpH( 図1)。あるいは、酵素反応を停止し、すべてのp -NPは、フェノラート形で存在することを確認するために、インキュベーション期間の終了時にNaOHを添加、pH値の範囲に対して単一時点のアッセイを使用。 - 5分間かけて30℃で分光光度計では、p個の -NPの形成に起因する405 nmでの吸光度の増加のための時間経過に従ってください。時間当たりの吸光度の増加の初期勾配を決定することにより、p個の -NPの最初に線形形成を定量化します。
- ランベルト・ベールの法則(ΔA=εのΔCd)を1使用したp -NPのための濃度(量Δc)の変化を計算します。
注:ΔAを求めた吸光度の線形変化で、εはMの単位で(それぞれの波長でのモル吸光係数である-1 -1)まで、dは光路(1センチメートル)の長さであり、量Δcを測定するM単位の濃度の変化()です。- アッセイ容量1 mlであることを考慮すると、 形成されたp -NPの量を計算します。
注:金額=濃度xのボリューム。 - それが形成された時点で形成されたp -NPの量を割ることにより、酵素活性を算出します。この活動を生成するための責任があった(mg単位)タンパク質の量によって酵素活性を分割することにより、特定の酵素活性を決定します。
- アッセイ容量1 mlであることを考慮すると、 形成されたp -NPの量を計算します。
- 候補遺伝子(smc00171、smc00930、またはsmc01003)が唯一の空のベクターを保有する抽出物で表現されていた中でのタンパク質抽出物によって誘発される吸光度の変化を比較してください。
注:以下の手順を続行するためには、候補遺伝子が発現されていた中でのタンパク質抽出物に起因する特定の活動は、少なくとも2倍またはmでなければなりません唯一の空のベクターを保有するタンパク質抽出物によって引き起こされる特定の活動で得られた値よりも鉱石。 - さらなる実験のために、p個の -nitrophenyl含有化合物の加水分解は、無細胞抽出物( すなわち、空ベクター) とp -NPおよびp -nitrophenolateアニオン( 図のための最も顕著な形成で最小であるような条件を選択しますタンパク質抽出物を用いた場合1)候補遺伝子が発現されていた、観察することができます。
- 3.1の初期酵素活性を測定した後、pHを、緩衝液のタイプ、緩衝強度のNaClの濃度が、例えば、トリトンX-100などの界面活性剤、及び異なる二価陽イオンの有無を変えることによって、それぞれの酵素のためのアッセイ条件を最適化します。
- 各変数の異なる濃度について、特定の酵素活性を決定する(3.1.4.2参照)(得られた最大数は、条件を定義します最大の酵素活性)。各変数は、その最適濃度で存在する最適化された酵素アッセイを定義する変数ごとに遭遇した最適な条件を組み合わせて使用してください。
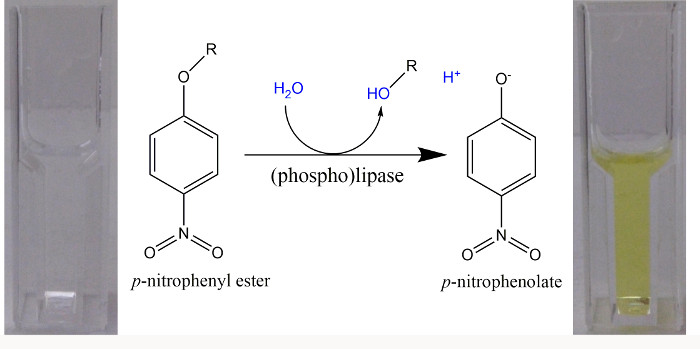
図1 のp -Nitrophenylエステル分光光度アッセイで(ホスホ)リパーゼなどの人工基質。p 個の -nitrophenylエステルの加水分解の際に、酸(R-OH) とpニトロフェノール(p個の -NP)が形成されています。原因のp -NPからフェノールのH +の解離のためのpKa = 7.2に、pHで> 9.2以上の99%は明るい黄色のp -nitrophenolateフォームと18,000 M -1 cmでのモル吸光係数であります-図1は、波長で使用することができ無料のp -nitrophenolate 22の定量化のための405nmの。 8.5のpHを有する緩衝液を用いた場合、吸光度を400 nmで測定し、14500 Mのモル吸光係数-1 cm -1 で 23を採用した。 この図の拡大版をご覧になるにはこちらをクリックしてください。
注:目的の酵素の活性のための最適な条件を定義された後、このリパーゼの実数/生理学的基質の探索に乗り出します。原理的には、多くの場合、相補2をとり、この目標、in vivoでのアプローチまたはin vitroでのアプローチを達成するために近づきます。
リパーゼの生理的基質のインビボ同定4.
"ove_content>注:他のインビボのアプローチでは、生成するインビボのアプローチでは、リパーゼの発現はhost's脂質プロフィールを変化させるかどうかを経時的に登録するために宿主生物8,11の関心のリパーゼを発現します。関心8,11の遺伝子の欠損変異体およびその脂質プロファイルは、野生型バージョン6,8,11とは異なるかどうかを研究する。生物の脂質プロフィールの定量的評価を得るために、簡単な方法は、細胞の化合物を放射性標識することからなります、脂質を抽出クロマトグラフィーによってそれらを分離し、放射性標識された分離脂質を定量化します。- 脂質の放射能標識。
- 希望培地(複合培地または規定の最小培地)5mlに関心の生物( 大腸菌またはS.メリロッティ )の一晩前培養を調製し、30℃で成長します。
- 前培養から、SAの20ミリリットルに接種培養のための初期OD 620 = 0.3を得るために、100ミリリットルの培養フラスコ中で私の新鮮な培地。
- 無菌条件下で培養物のアリコート(1ミリリットル)に乗り、14ミリリットルの滅菌ポリスチレン丸底チューブに移します。
- 1ミリリットルの培養液に[1- 14 C]酢酸(1mmol当たり60 MCI)1μCiのを追加します。
- 24時間の期間、30℃で撹拌下で液体培養をインキュベートします。
- インキュベーション期間の終了時に、室温で5分間12,000×gで1.5ml微量チューブと遠心分離機に培養物を移します。
- 100μlの水にペレットを再懸濁。この時点で、-20℃で細胞懸濁液を保存するか、すぐ極性脂質(セクション4.2)の抽出を続行します。
- 極性脂質の抽出。
注:ここで説明する方法は、本質的にブライとダイアー19によって報告された手順に従います。- 水性細胞SUSP100μlに(:;体積/体積1 2)クロロホルム溶液:ensionは、メタノールの375μlを添加します。
- 30秒間ボルテックスし、室温で5分間インキュベートします。
- 室温で12,000×gで5分間遠心。
- 新しい1.5ミリリットルのマイクロチューブに上清を移します。
- クロロホルム125μlを添加して、水125μlの、ボルテックス30秒。
- 室温で12,000×gで1分間遠心。
- 新鮮なチューブに低いクロロホルム相を移し、窒素ガス流で乾燥しました。
- クロロホルムを100μlの乾燥脂質を溶解:メタノール溶液(1:1、体積/体積)。
注:この時点で、脂質溶液を5μlのアリコートを液体シンチレーション計数により定量することができます。 - 薄層クロマトグラフィー(TLC)分析のために、窒素ガス流と残りの95μLの下乾燥し、20μlのクロロホルム中で乾燥させた脂質を再溶解:メタノール溶液(1:1、体積/体積)。 3μlのアリコートを使用しますTLC分析のために。
- 薄層クロマトグラフィー(TLC)により、極性脂質を分離します。
注:分析すべき脂質クラスに応じて、固相と移動相の異なる組み合わせは、分離のために使用される可能性があります。ここで、固相として、高性能薄層クロマトグラフィー(HPTLC)シリカゲルアルミシートを用いて、中性極性脂質に対してより適した荷電極性脂質と他、のための典型的な分離は、概説されています。- 2次元TLC(2D-TLC)による荷電極性脂質の分離。
- HPTLCシリカゲルアルミシート(10×10センチ)の一角、プレートの端から2センチメートルに脂質サンプルの3μlのアリコートを適用します。
- 最初の次元での分離のための移動相(140ミリリットルクロロホルム、60ミリリットルのメタノール、および10ミリリットルの水)を準備し、混合します。
- コートクロマトグラフィーペーパーで、内部TLC現像室。
注:これは、チャンバの気相を確保することです第一次元の移動相は、チャンバーに添加され、チャンバがガラス板で閉鎖された後(30分以内)に急速に飽和します。 - 準備とミックス分離のための移動相(130ミリリットルクロロホルム、メタノール50ml、20mlの氷酢酸を)二次元で、内部クロマトグラフィーペーパーで被覆された第二TLC現像室に転送し、チャンバー飽和をしましょう。
- 慎重に一次元5閉鎖チャンバー内に60分間( すなわち 、クロマトグラフィーを行って)、プレートを第一のチャンバに乾燥した脂質サンプルとHPTLCシリカゲルアルミニウムシートを転送し、開発します。
- チャンバーからプレートを取り外し、溶媒を30分間流フードで乾かしましょう。
- 前のクロマトグラフィーに関して90度板を入れた後、seconに、脂質は一次元で分離されたHPTLCシリカゲルアルミニウム板を転送D室と第二の次元5で60分間プレートを開発。
- チャンバーからシートを外し、溶媒は、少なくとも2時間流フードで乾かしましょう。
- 中性の極性脂質の分離。
- プレートの縁から2センチメートルを開始HPTLCシリカゲルアルミシート上の脂質サンプルの3μlのアリコートを適用します。複数のサンプルを1次元クロマトグラフィーで分析している場合は、別のサンプル・アプリケーション・スポット間1.5センチ以上の距離を保ちます。
- 移動相を準備し、ミックス(140ミリリットルヘキサン、60ミリリットルのジエチルエーテル、および8ミリリットル酢酸)内部クロマトグラフィーペーパーで被覆し、チャンバー飽和(30分)させるためのガラス板で覆われたTLC現像室へと移転。
- チャンバに乾燥した脂質サンプルとHPTLCシリカゲルアルミニウムシートを転送し、閉じたチャンバー内で30分間、プレートを開発。
- チャンバーからプレートを取り外しますそして溶媒は2時間流フードで乾かしましょう。
- 2次元TLC(2D-TLC)による荷電極性脂質の分離。
- 定量化と分離極性脂質の可視化。
- 開発したTLCシートが乾燥したら、3日間、閉鎖カセットに尽性発光(PSL)の画面でそれをインキュベートします。
- PSLスキャナにインキュベートし、画面を公開し、分離し、放射性標識脂質の仮想イメージを取得します。
- PSLソフトウェア20を使用して定量化を行います。
- 個々の極性脂質クラスの可視化と分離。
- インキュベートは、ヨウ素結晶を1gの存在下でのクロマトグラフィーチャンバー中で10分間TLCシートを開発しました。
注:分離された脂質化合物は、ヨウ素を溶解し、茶色がかったスポットとして表示されます。 - 鉛筆でサークルスポットは、 すなわち、1,2- dipalmitoyl- のsn -グリセロール、ジパルミトイルL-α-PHOS(標準化合物の相対移動度(式中、R f)にそれらを比較しますホスファチジルコリン、DL-α-モノパルミチン、またはパルミチン酸)、およびそれらが属する可能性のある脂質クラスに識別します。
- ヒュームフードでは、ヨウ素はTLCシートから蒸発してみましょう。
- クロロホルム溶液(2:スパチュラの助けを借りて、シートから目的の化合物を含有するシリカゲルをこすり、そして100μlの水及びメタノールの375μLの混合物を用いてシリカゲルから化合物を抽出1、体積を/容量)。
- (以降4.2.2)で概説したようブライとダイアーに応じて抽出を続行します。
- 使用するまで-20℃;:メタノール溶液(容量/容量1 1:)ストアは100μlのクロロホルム中の脂質クラスを精製しました。
- インキュベートは、ヨウ素結晶を1gの存在下でのクロマトグラフィーチャンバー中で10分間TLCシートを開発しました。
リパーゼの生理的基質のインビトロ同定5.
注:in vitroでのアプローチでは、関心のリパーゼは、対応するハイドロールに分離された脂質または個々の純粋な脂質の混合物を変換できるかどうかを検討3.2に最適なように定義された条件の下でysis製品。
- PC-特異的ホスホリパーゼC SMc00171(5.2を参照)、ホスホリパーゼA(5.3を参照)、およびDAGリパーゼSMc01003については、 表3のとおり酵素アッセイのためのピペッティングスキームを使用して、アクティビティ(5.4を参照)。
- PC-特異的ホスホリパーゼC活性の決意( 表3)。
- 1.5ミリリットルのマイクロチューブに、合計14 C標識PCおよびTriton X-100の溶液を毎分5000カウント(CPM)を追加します。
- 窒素気流下で混合し、乾燥しました。
- 99.5μlの最終容量を得るために、ジエタノールアミン-塩酸、pHが9.8バッファ、ならびにNaClおよびのMnCl 2ソリューションおよび再蒸留水を追加します。 5秒間ボルテックス。
- 反応を開始するために酵素0.5μlの(5μgタンパク質)( すなわち、SMc00171が存在する過剰発現した無細胞抽出液)を追加します。簡単に混ぜます。
- 4時間30℃でインキュベートします。
- によって反応を停止させますメタノール250μlのクロロホルム125μlに加えて。
- 前述したように、脂質を抽出し(4.2を参照)。
- 1次元(1D)-TLCで区切り脂質は(4.3.2および4.4を参照)、およびPSLイメージングによってそれらを分析します。
- ホスホリパーゼA活性を決意した( 表3)。
- 1.5ミリリットルのマイクロチューブに、5000合計14 C標識リン脂質のCPMおよびTriton X-100の溶液を加えます。
- 窒素気流下で混合し、乾燥しました。
- 最終100μlのアッセイのために、トリス-HCl、pH8.5の緩衝剤、NaCl溶液と水を追加します。 5秒間ボルテックス。
- 酵素の5μL(50μgタンパク質)( すなわち 、SMc00930またはSMc01003が存在する過剰発現した無細胞抽出液)を追加します。
- 5時間30℃でインキュベートします。
- メタノール250μlのクロロホルム125μlに添加することにより反応を停止させます。
- 前述したように、脂質を抽出し(4.2を参照)、独立しました1D-TLCによってそれらの移動相130がmlのクロロホルム、メタノール50ml、20mlの氷酢酸を使用し、PSLイメージングによってそれらを分析します。
- ジアシルグリセロール(DAG)リパーゼ活性の決意。
- 14 C標識DAGの調製。
- 放射性標識S.根粒培養物は、(4.1を参照)と記載されているように(4.2を参照)極性脂質を抽出します。独立したS.クロロホルム中1D-TLCによる根粒総脂質抽出物:メタノール:酢酸(130:50:20;体積/体積)4.3.1における第二の次元での分離のために記載された条件を使用して。
- ヨウ素染色によりPCを視覚化し、ホスファチジルコリン(PC)の局在をマークするために鉛筆を使用しています。
- 4.5で説明したように、放射性標識のPCを隔離します。
- シンチレーション計数によって抽出されたPCを定量化します。
注:約32万cpmのPCが期待されています。 - 50 mMトリス-塩酸、pHが7.2、0.5にウェルシュ菌からのホスホリパーゼCの0.1 Uを搭載したPC(25万CPM)の治療全量100μlで2時間%トリトンX-100及び10mMのCaCl 2を 、メタノールとクロロホルム125μlの250μlのを加えて反応を停止します。
- 1D-TLC(4.3.2を参照)ことによって、それらによって、以前、別の記載されているように脂質を抽出します。
- 石英プレートからジアシルグリセロールを分離し、シンチレーション計数によって定量(4.2に記載されるように)
- ジアシルグリセロールリパーゼアッセイ( 表3)。
- 1.5ミリリットルのマイクロチューブに、14 C標識DAGおよびTriton X-100の溶液の5000 CPMを追加します。
- 窒素気流下で混合し、乾燥しました。
- 最終100μlのアッセイのために、トリス塩酸(pHは9.0)緩衝液、NaCl溶液および再蒸留水を追加します。 5秒間ボルテックス。
- 酵素5μlの(無細胞抽出物の50μgのタンパク質)を加えることにより反応を開始。
- 4時間30℃でインキュベートします。
- メタノールANの250μlに添加することによって反応を停止させます前述のようにクロロホルム抽出物の脂質のD 125μL(4.2を参照)。
- 1D-TLC(4.1.3.2を参照)、その後のPSLイメージングによって、中性極性脂質を分析します。
- 14 C標識DAGの調製。
Access restricted. Please log in or start a trial to view this content.
結果
ビス のp -nitrophenylリン酸 とPC固有ホスホリパーゼC SMc00171の活動
E.から得られた無細胞抽出物大腸菌 BL21(DE3)をsmc00171が発現していたpLysSをし、xは、 形成されたp -NPを測定し、分光光度酵素アッセイを用いて、ビスのp
Access restricted. Please log in or start a trial to view this content.
ディスカッション
過去20年間、多くの生物のゲノムが配列決定されており、ゲノム配列データの富が生成されているが、機能的な解釈が遅れ、したがって、ゲノム機能の理解を妨げています。ゲノム中の遺伝子の機能は、多くの場合、既知の機能または保存されたモチーフの発生の遺伝子との類似性に基づいて割り当てられます。しかし、所定の遺伝子の正確な機能はしばしば知られていません。特に、酵素?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは、開示することは何もありません。
謝辞
この作品は、(InvestigaciónエンFronterasのデ・ラ・CienciaでInvestigaciónCientíficaBASICAで82614、153998、253549、および178359だけでなく、118)とDirección一般デからConsejoナショナルデCienciasのyTecnología・メキシコ(CONACyT-メキシコ)からの助成金によってサポートされていましたAsuntosデパーソナルAcadémico-ナショナル大学自治・デ・メヒコ(DGAPA-UNAM; PAPIIT IN202616、IN203612)。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
参考文献
- Nelson, D. L., Cox, M. M. Lehninger, Principles of Biochemistry. , W. H. Freeman and Company. New York. (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 5th, Elsevier. Amsterdam, The Netherlands. 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids? Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria? Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998(2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115(2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. Experiments in Molecular Genetics. , Cold Spring Harbor Laboratory Press. Plainview, NY, USA. (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610(2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. Phosphorimager SI User´s Guide. , http://www.camd.lsu.edu/SAXS/Files/PIManual.pdf (1994).
- Dixon, M., Webb, E. Enzymes: Third Edition. , Longman, USA. (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , Article ID 306798 (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. Protein Purification, Principles and Practice. , Springer. New York. (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved