Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Lipaz ve Fosfolipaz Adayları için substrat özellikleri tanımlama
Bu Makalede
Özet
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
Özet
Mikroorganizmalar organizma için dış yüzeyler kullanılabilir hale getirmek için salgılanan (fosfo) lipaz çok geniş bir yelpaze oluşturur. Alternatif olarak, diğer (fosfo) lipazlar fiziksel üreten organizma iç lipidlerin ciro sebep olması ve sıklıkla, hücre membranlarının bir biçimlenme sebebiyet veren ile ilişkili olabilir. potansiyeli (fosfor) lipazlar gen / protein dizisi mevcuttur algoritmaları bir dizi ile tahmin edilebilir, ancak, enzim aktiviteleri, alt-tabaka özgüllükleri ve potansiyel fizyolojik fonksiyonları deneysel kanıtı sıklıkla elde edilmemiştir. Bu yazıda bilinmiyor alt-tabaka özgüllükleri ve nasıl bir ilgili (fosfo) lipaz doğal alt-tabaka için arama, bu optimum koşulların kullanılması ile potansiyel (fosfo) lipazlar için Tayin koşullarının optimize edilmesi açıklanmaktadır. Küçük tespit etmek için yardımcı, p-nitrofenil türevleri gibi yapay kromojenik substratlar, olabilir kullanmaStandart koşullar altında bir tahmin (fosfo) lipaz için enzimatik aktivite. Böyle küçük bir enzimatik aktivite karşılaşmıştır, bir enzim tahlili ayrı parametre ile yapay alt tabaka daha verimli bir hidrolizi elde etmek için değiştiirlebilir. bir enzim çalışır koşulları tespit ettikten sonra, potansiyel, doğal substratlar, çeşitli tenzillerinden tat kromatografik yöntemler kullanılarak takip edilebilir bir işlem test edilmelidir. Yeni enzimler için alt-tabaka özgüllükleri tanımı, genellikle daha sonra deneysel olarak test edilebilir, bu enzimlerin, potansiyel bir fizyolojik rolü için hipotez içerir. Bu yönergeleri izleyerek, biz büyüme fosfor sınırlayıcı koşullara bağlı bakteri Sinorhizobium meliloti membranların remodeling için önemli bir adım olarak, fosfokoline ve diaçilgliserol için fosfatidilkolin bozan bir fosfolipaz C (SMc00171) tespit başardık. İki patatin- tahmin içinAynı organizmanın fosfolipaz (SMc00930 ve SMc01003) gibi, biz onların alt tabaka özgüllükleri yeniden tanımlamak ve SMc01003 bir diaçilgliserol lipaz olduğunu açıklamak olabilir.
Giriş
Bu tür triaçilgliserol ve (glisero) fosfolipidler gibi gliserol tabanlı lipidler önemli oluşturmaktadır ve muhtemelen en iyi bilinen lipit sınıfları 1. Triaçilgliseroller (TAG) yağlar ya da genellikle depolama lipidler olarak işlev yağlar, ve bu nedenle potansiyel enerji ve karbon kaynağı olarak bulunmaktadır. TAG'lar sık sık dış TAG sindirmek ve karbon kaynağı olarak kullanılabilir hale getirmek için üreten organizma tarafından salgılanan lipaz tarafından bozulmuş olabilir. Ayrıca, lipazlar yaygın nedeniyle önemli biyo teknolojik uygulamalarda 2 yıl içinde incelenmiştir.
Nedeniyle amfifilik doğası ve onların yakın silindirik şekil, (glisero) fosfolipidler sergi zar oluşturan özellikleri ve genellikle iki katmanlı membranın 3 ana lipidik bileşenlerini teşkil edecek. Bu bakteri Escherichia coli, sadece üç ana kafa grubu varyantları, fosfatidilgliserol (PG), kardiyolipin (CL) ve phosphatid basit mikroorganizmalardabunlardan biri, her biri farklı moleküler türler 4 çok sayıda bir sn farklı yağlı asil zincirleri önemli sayıda -1 ya da sn -2 pozisyonu veren artış ile ikame edilebilir bilmelidir, ancak ylethanolamine (PE), karşılaşılan . Diğer bakteriler diğer fosfolipidler ek olarak veya bunun yerine sahip olabilir. Örneğin, Sinorhizobium meliloti, baklagil yonca (Medicago sativa), bir azot bağlayıcı kök nodül simbiyoz oluşturmak için güçlü bir toprak bakterisi, ikinci bir zitteriyonik fosfolipid, fosfatidilkolin (PC), PE ilave olarak ihtiva eden 5 ile. Ayrıca, lipit içermeyen, fosfor veya gliserol, hücre zarının amfifilik ve form parçası olabilir. Örneğin, fosfor sınırlayıcı büyüme koşullarına bağlı olarak, S. meliloti (glisero) Fosfolipidler, büyük ölçüde, yani sulfolipidlerin, ornitin lipidler ve diacylglyceryl trimethylho fosfor içermeyen membran lipidleri ile ikame edilmektedirmoserine (DGTS) 6. Bakterilerde DGTS iki aşamalı yolun 7 diasilgliserole (DAG) teşekkül eden, fakat DAG üretimi için bir kaynak belli değildi edilir. Darbe kovalayıcı deneyler bilgisayar fosfor sınırlayıcı koşullar altında meydana geldiği ve DAG ve fosfokolin haline PC dönüştürmek olan DGTS 8 için bir ön-madde olarak ve bir fosfolipaz C (PLCP, SMc00171) tanımlayabilir Bu yazıda tarif edilen metodoloji kullanılarak düşündürmektedir 8.
Ayrı bir çalışmada, biz keşfettik bir açil-KoA sentetaz (fadd) S. -açıklı mutant büyüme 9 sabit faz girerken melılotı veya Escherichia coli serbest yağ asitleri birikmiş. Bu yağlı asitler, membran lipitleri elde edilecek gibi görünse de, serbest yağ asitleri ya da bunların serbest bırakılması enzim (ler) için kesin bir kaynak bilinmemektedir. Yine, bu stratejiyi uygulayan bu yazının ana hatlarıyla, iki patatin gibi 10 (fosfo) lipazlar (S. serbest yağ asitleri oluşmasına katkıda SMc00930 ve SMc01003) meliloti 11 tahmin edildi. Şaşırtıcı bir şekilde, SMc01003 o gliserol ve serbest yağ asitleri 11 nihayet monoaçilgliserol ve dönüştürme substrat olarak DAG kullanıldı. Bu nedenle, SMc01003 DAG lipazı (DGLA) 'dir.
Algoritmaları bir dizi potansiyel (fosforo) tahmin etmek için mevcut olmakla beraber 12,13, lipazlar onların hassas fonksiyon ve fizyolojik rolü genellikle bilinmemektedir. Burada klonlamak, bir protokol anahat ve tahmin veya potansiyel (fosfo) lipazlar aşın. Bu el yazması enzim tahlilleri geliştirilmiş ve yapay kromojenik maddeleri kullanarak aşırı (fosfo) lipaz için optimize edilebilir açıklar. Bu bulguların mikrobiyal fizyoloji anlayışımızı zenginleştirecek nasıl optimize edilmiş bir enzim testi ile gerçek (fosfo) lipaz substrat karşılaştı olabilir ve nasıl örnekler sağlar.
Access restricted. Please log in or start a trial to view this content.
Protokol
Öngörülen lipaz 1. Klonlama ve aşın Yapısal gen
- Polimeraz zincir reaksiyonu (PCR) 14 ve özel oligonükleotidler (Tablo 1) 15 kullanarak, ilgi (smc01003, smc00930 veya smc00171) geni amplifiye konakçı organizmanın genomik DNA'dan, bir lipaz için kodlama veya fosfolipaz tahmin (örn S. meliloti).
- (Oligonükleotid tasarlanmıştır dizisi ile birlikte) bir kısıtlama yerleri sokacak. Karşılık gelen kısıtlama enzimleri ile büyütülmüş DNA fragmanı sindirmek ve pET serisi 16 plazmid gibi bir ifade vektörü içine klonlamak.
- Klonlanmış genin doğru DNA dizisinin doğrulanması sonra Escherichia coli BL21 (DE3) pLysS, 16 gibi bir ifade suşu vektörü dönüşümü.
- Ifadesi ev sahibi E. bir gecede öncesi kültür hazırlayın E. coli BL21 (DE3) 'e uLYss, 100 ml kültür Luria Bertani etsuyu (LB) 17 20 ihtiva eden şişelere, artı gerekli antibiyotikler, klonlanmış genin veya boş vektör ile ilgili pET vektörü barındıran. Kültür, 30 ° C'de hücreler (veya bir lipaz kaynaklandığı bakterinin normal büyüme sıcaklıkta).
- Gece boyunca ön kültürleri kullanılarak, 620 nm (OD 620) = 0.05 başlangıçtaki optik yoğunluk elde etmek için 2 L'lik kültür şişelerinde 500 önceden ısıtılmış LB ortamı ml (artı gerekli antibiyotik) inoküle. Kültürlerin ve OD 620 = 0.3, büyümeyi takip 100 uM nihai konsantrasyona kadar izopropil-β-D-tiyogalaktozid (IPTG) ekleyin ve 4 saatlik bir süre için 30 ° C'de çalkalama altında inkübe edilir.
- İnkübasyon süresinin sonunda, 30 dakika boyunca 4 ° C'de 5,000 x g'de 500 ml'lik bir santrifüj tüpüne ve santrifüj her kültür aktarın. Süspansiyon tamponu, 5 ml (ör S bakteriyel hücre pelletleri yeniden süspanseMc00930- ve 50 mM dietanolamin-HCI pH 9.8), 50 mM Tris-HCI, pH 8.0 ve SMc00171-ifade eden hücrelerde hücre SMc01003-sentezleyen. Kullanılana kadar -80 ° C'de hücre süspansiyonları saklayın.
2. Hücre içermeyen protein ekstreleri hazırlamak ve protein konsantrasyonu belirlemek
- Bakteriyel hücre süspansiyonları Defrost ve buz üzerinde saklayın. 2'de 20.000 lb hücre süspansiyonları soğuk basınç hücresi ile üç kez geçmektedir. 4 ° C'de 30 dakika boyunca 5000 x g'de santrifüj ile sağlam hücreler ve hücre kalıntıları uzaklaştırılmıştır.
- Santrifüj işleminden sonra, bir sonraki analiz için süpernatanından 100 ve 500 ul alikotları hazırlamak ve kullanılana kadar -80 ° C'de saklayın.
- Tercih edilen bir yöntem ile ya da 18 tarif edildiği gibi farklı hücre içermeyen ekstreler protein konsantrasyonunu belirlemek için, 100 ul bölüntüde birini kullanabilirsiniz.
Optimize enzimini 3. Yapay Elyaf(Fosfo) lipaz aktiviteleri
- Tat enzim faaliyetlerinin bir başlangıç içerisinde için, p-nitrofenolu (s -np) halinde hidroliz üzerine, renkli bir ürün elde yapay substratlar kullanır.
- Yapay p-nitrofenil ester, alt-tabakalar ile zaten optimize Enzim tahlilleri için (fosfolipaz C PLCP (SMc00171) tarif edilen, hem de tahmin edilen patatin benzeri fosfolipazlar SMc00930 ve SMc01003 için) kullanımı pipetleme şeması Tablo 2'de tarif edilen.
- Yeni bir potansiyel (fosfo) lipaz gezinirken 50 mM Tris-HCl, pH 8.5, 100 mM NaCl,% 0.05 Triton X-100, 0.5 mM p-nitrofenil-içeren bileşik (p-nitrofenil fosfat içeren bir birinci standart enzim deneyi hazırlanması , bis- p-nitrofenil fosfat, p-nitrofenil dekanoat, ya da p-nitrofenil palmitat) ve hücresiz protein ekstresi (kontrol 1, 3, 10, 30, 100, 300 ve 1 toplam hacim içinde 1000 ug) ml 1 ml'lik plastik CUVETTES.
Not: sürekli bir deneyde p-nitrofenil ester hidrolizi, aşağıdaki kullanımlar alkali pH (Şekil 1). Alternatif olarak, enzim reaksiyonu sonlandırmak için tüm p -np fenolat şeklinde mevcut olduğundan emin olmak için kuluçka süresi sonunda NaOH eklenerek, bir pH değerleri aralığı için tek bir zaman noktası tahlilleri kullanabilir. - 5 dakikalık bir süre boyunca 30 ° C'de bir spektrofotometre p -np oluşumuna bağlı 405 nm'de absorbans bir artış, zaman takip edin. Zaman başına absorbans artışı, ilk eğrisinin eğimi P -np başlangıçtaki düz oluşumunu ölçmek.
- Lambert-Beer yasası (ΔA = ε Δc d) 1 kullanılarak p -np için konsantrasyon (Δc) değişimi hesaplayın.
NOT: ΔA belirlenen absorbans doğrusal değişim, ε M birimlerinde ilgili dalga boyunda molar tükenme katsayısı (-1 cm-1), d ışık yolu (1 cm) uzunluğu ve Δc belirlenecek M birimleri konsantrasyon değişim () yukarı.- Deney hacmi 1 ml olduğu göz önüne alındığında, oluşan p -np miktarını hesaplayın.
NOT: Tutar = konsantrasyon x hacim. - Bu oluşturulduğu zaman oluşturduğu s -np miktarını bölerek enzim aktivitesinin hesaplanması. Bu aktiviteyi üretilmesi için sorumlu olduğu (mg olarak) protein miktarına göre, enzim aktivitesi bölünmesiyle özel enzim aktivitesi belirlenir.
- Deney hacmi 1 ml olduğu göz önüne alındığında, oluşan p -np miktarını hesaplayın.
- Bir aday gen (smc00171, smc00930 veya smc01003) Sadece boş bir vektör liman özleri ile ifade edilmişti protein özleri kışkırttığı absorbans değişimleri karşılaştırın.
Not: Aşağıdaki adımlara devam etmek için bir aday gen olarak ifade edilmiş olan protein ekstrelerinin kaynaklanan belirli faaliyetleri, en az iki veya m olmalıdırSadece boş bir vektör liman protein ekstrelerinin neden belirli faaliyetleri için elde edilen değerlerden daha cevher. - Daha fazla deneyler için, p-nitrofenil-içeren bileşiğin hidrolizi hücre içermeyen özütler (yani, boş vektör) ile p -np en belirgin bir şekilde oluşması ve p -nitrophenolate anyondur (Şekil minimal olduğu gruplardır koşulları seçmek protein özleri, bir aday gen olarak ifade edilmiş olan, kullanıldıklarında, 1) görülmektedir.
- 3.1 ilk enzim etkinliğinin belirlenmesi sonra, NaCI konsantrasyonu, pH, tampon türü değiştirilerek ilgili enzim için deney koşulları optimize gücü tampon, örneğin Triton X-100 gibi deterjanlar ve yokluğunda ya da farklı bivalent katyonlar varlığında.
- Her değişkenin farklı konsantrasyonu için, (3.1.4.2 bakınız) özel enzim aktivitesinin belirlenmesi (elde edilen en yüksek sayısı koşulu tanımlarmaksimal enzim etkinliğinin). Her bir değişken optimum bir konsantrasyonda mevcut olduğu, optimum enzim tahlili tanımlamak için her bir değişken için karşılaşılan en iyi şartlarda bir arada kullanarak.
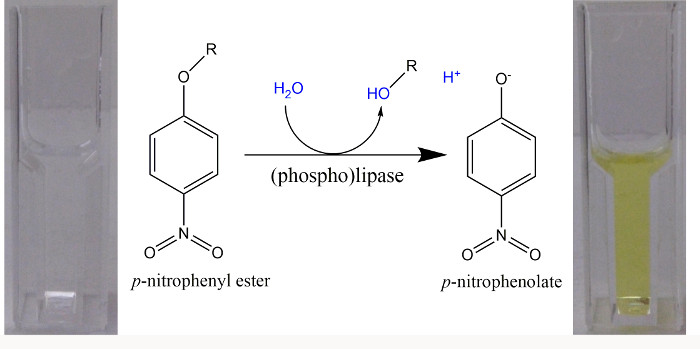
Şekil 1. p-Nitrofenil esterler bir spektrofotometrik tahlilinde (fosfo) lipazlar gibi yapay substratlar. S-nitrofenil esterlerin hidrolizi üzerine, bir asit (R = OH) ve p-nitrofenolu (s -np) oluşturulur. Nedeniyle pKa s -np fenolik H + ayrılması için = 7.2, ile> 9,2 fazla% 99, parlak san bir p -nitrophenolate formu ve 18,000 M-1 sm bir molar sönüm katsayısı olan bir pH değerinde - 1, bir dalga boyu kullanılabilir serbest p -nitrophenolate 22 miktarının 405 nm. 8.5 pH ile tamponlar kullanıldığı zaman, absorbans -1 400 nm ve 14.500 M -1 cm molar sönüm katsayısı belirlenmiştir 23 kullanılmıştır. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
NOT: ilgi enzim aktivitesi için en uygun koşulları tanımlanmış ettikten sonra, bu lipaz fizyolojik / gerçek substrat için arama atılmak. Prensip olarak, genellikle tamamlayıcı iki almak, bu amaca, bir in vivo yaklaşımı veya bir in vitro yaklaşım elde etmek için yaklaşımlar.
Bir lipaz Fizyolojik Yüzey 4. İn Vivo Kimlik
"ove_content> Not: Başka bir in vivo yaklaşımda, bir oluşturmak in vivo yaklaşımda, lipaz ifade host's lipid profili değiştirip değiştirmediğini zamanla kaydetmek için bir konak organizmada 8,11 ilgi lipaz ifade eder. Çevrede 8,11 konusu genin eksikli mutant ve lipid profili, vahşi tip sürüm 6,8,11 farklı olup olmadığını incelemek. organizmanın lipid profili kantitatif bir değerlendirmesini elde etmek için, basit bir yöntem hücresel bileşikler, radyo-etiketleme oluşur , lipidler ayıklanması kromatografi ile ayırarak ve radyoaktif işaretli ayrıldı lipidlerin ölçülmesidir.- Lipidlerin radyo-etiketlenmesi.
- İstenen kültür ortamında (karmaşık, orta ya da tanımlanan minimal ortam) 5 ml daki (E. coli veya S. melılotı) bir organizmanın bir gece boyunca kültür öncesi hazırlamak ve 30 ° C'de büyür.
- Ön-kültür, sa 20 ml inoküle100 ml'lik bir kültür şişesi içinde me taze ortam ilk OD 620 = kültür 0.3 elde edildi.
- steril koşullar altında kültür bir kısım (1 mi) al ve 14 ml steril polistiren yuvarlak tabanlı bir tüpe aktarılır.
- 1 ml kültüre [1- 14C] asetat (mmol başına 60 mCi) 1 uCi ekleyin.
- 24 saatlik bir süre için 30 ° C'de çalkalama altında sıvı bir kültürün inkübe edin.
- İnkübasyon süresinin sonunda, 5 dakika boyunca oda sıcaklığında 12.000 xg'de 1.5 ml mikrosantrifüj tüpü ve santrifüj kültürünün aktarın.
- su, 100 ul pelletini. Bu noktada, -20 ° C'de bir hücre süspansiyonu depolar ya da hemen polar lipidler (Bölüm 4.2) ekstraksiyonu ile devam edin.
- polar lipidler çıkarımı.
NOT: esasen burada anlatılan yöntem Bligh ve Dyer 19 tarafından bildirilen prosedürü takip eder.- Sulu hücre asmalar 100 ul için(:; Vol / vol 1 2) kloroform çözüm: ension, metanol 375 ul ekleyin.
- 30 saniye vorteksleyin ve oda sıcaklığında 5 dakika inkübe edilir.
- Santrifüj 5 dakika oda sıcaklığında 12.000 xg'de.
- yeni bir 1.5 ml mikrosantrifüj tüpüne süpernatant aktarın.
- kloroform 125 ul su ve 125 ul, girdap 30 sn ekleyin.
- Santrifüj 1 dak oda sıcaklığında 12.000 xg'de.
- bir azot gazı akımı ile, yeni bir tüpe ve kuru alt kloroform fazı aktarın.
- (:; Hacim / hacim 1: 1), metanol çözeltisi: kuru kloroform 100 ul lipidleri çözülür.
NOT: Bu noktada, yağ çözeltisi, 5 ul bir alikosu, sıvı sintilasyon sayımı ile tayin edilebilir. - ince tabaka kromatografisi (TLC) analizi için, bir azot gazı akımı, geri kalan 95 ul aşağı kurutun ve 20 kloroform ul kurutulmuştur lipitleri çözülür: metanol çözeltisi (1: 1; hac / hac). 3 ul tablet kullanınTLC analizi için.
- İnce tabakalı kromatografi (TLC) ile bir polar lipidler ayrılması.
NOT: lipit sınıfları bağlı olarak analiz edilmesi, bir katı ve mobil fazların farklı kombinasyon ayrılması için kullanılabilen. Burada katı faz yüksek performanslı ince tabaka kromatografisi (HPTLC) silis jel, alüminyum levhalar kullanılarak nötr polar lipidler için daha uygun şarj polar lipidler ve başka, tipik bir ayırma özetlenmiştir.- iki boyutlu TLC (2D-TLC) tarafından tahsil polar lipidler ayrılması.
- HPTLC silika jel alüminyum levha (10 x 10 cm), levhanın kenarı 2 cm bir köşesinde lipid Numunenin 3 ul tablet uygulanır.
- Hazırlama ve birinci boyutta ayrılması için mobil faz (140 mi kloroform, 60 mi metanol ve 10 ml su) karıştırınız.
- Coat kromatografi kağıdı ile içten bir TLC gelişmekte odası.
Not: Bu sağlamak olduğunu odasının gaz fazıilk boyut mobil aşamasından sonra (30 dakika içinde) hızlı bir şekilde doyacak bölmeye eklendi ve oda bir cam plaka ile kapatıldı. - Hazırlama ve mobil faz karışımı (130 mi kloroform, 50 mL metanol ve asetik asit 20 ml buzlu) dahili kromatografi kağıdı ile kaplanmış bir ikinci TLC gelişmekte olan bölmeye, ikinci boyutta ayırma ve transfer ve oda doyurulması sağlar.
- Dikkatle ilk boyut 5 kapalı bir odada, 60 dakika boyunca (yani, kromatografi yerine) plaka birinci bölme kurutularak lipit numune ile HPTLC silika jel alüminyum levha transferi ve geliştirilmesi.
- odasından plaka çıkarın ve çözücüler 30 dakika boyunca bir akış kaputu kapalı kurumaya bırakın.
- Önceki kromatografi ile ilgili olarak 90 derece plaka açtıktan sonra, secon için, lipitler bir boyutta ayrılmıştır üzerinde HPTLC silika jel alüminyum levha transferD odası ve ikinci boyut 5 60 dakika boyunca plaka geliştirir.
- odasından sayfasını çıkarın ve çözücüler en az 2 saat süreyle bir akış kaputu kapalı kurumaya bırakın.
- nötral polar lipidler ayrılması.
- levhanın kenarlarından 2 cm başlayarak HPTLC silika jel alüminyum levha lipid örnekleri 3 ul alikotları uygulanır. Birden çok numune tek boyutlu kromatografi analiz ise, farklı örnek uygulama noktaları arasında en az 1.5 cm bir mesafe bırakmak.
- mobil faz hazırlayın ve karıştırılır (140 mi heksan, 60 mL dietileter, ve 8 ml asetik asit) dahili kromatografi kağıdı ile kaplanmış ve oda doyurulması (30 dakika) sağlamak için bir cam levha ile kaplanmış bir ince tabaka kromatografisi, gelişmekte odasına transfer.
- bölme kurutularak lipit örnekleri ile HPTLC silika jel alüminyum levha transfer ve kapalı bölme 30 dakika boyunca plaka geliştirir.
- odasından plaka çıkarınve çözücüler 2 saat için bir akış kaputu kapalı kurumaya bırakın.
- iki boyutlu TLC (2D-TLC) tarafından tahsil polar lipidler ayrılması.
- Sayısallaştırılması ve ayrılan polar lipidler görselleştirme.
- geliştirilmiş TLC tabaka kuruduktan sonra, 3 gün süre ile kapalı bir kaset bir ışığa duyarlı lüminesans (PSL) ekran ile inkübe edin.
- Bir PSL tarayıcıya kuluçkaya ekranı Açığa ve ayrılan radyoaktif işaretli lipidlerin bir sanal görüntü kazanır.
- PSL yazılımı 20 kullanılarak ölçümü gerçekleştirin.
- Görselleştirme ve bireysel polar lipid sınıfların izolasyon.
- iyot kristalleri 1 g mevcudiyetinde bir kromatografi odası içinde 10 dakika boyunca, gelişmiş TLC levha inkübe edin.
NOT: Ayrı lipidik bileşikler iyot çözülür ve kahverengimsi lekeler olarak görünür. - Çember bir kalemle noktalar, standart bileşikler (yani, 1,2-dipalmitoyl- sn-gliserol, dipalmitoil-L-α-Fos nispi hareketlilik (R f) bunları karşılaştırmakphatidylcholine, DL-α-monopalmitin veya palmitik asit) ve onlar ait olabilir hangi lipit sınıfı tanımlamak.
- Davlumbaz, iyot TLC sacdan buharlaşmasına izin verin.
- Kloroform çözeltisi (2: Bir spatula yardımıyla tabakadan ilgi konusu bileşiği ihtiva eden silika jel kazımak ve suyun 100 ul metanol 375 ul bir karışımı ile, silika jel den bileşik elde 1; Vol / hacim).
- (Itibaren 4.2.2) belirtildiği gibi Bligh ve Dyer göre ekstraksiyon ile devam edin.
- Kullanılana kadar -20 ° C'de, (hac / hac 1: 1), metanol çözeltisi: kloroform 100 ul saklayın saflaştırılmıştır lipit sınıfı.
- iyot kristalleri 1 g mevcudiyetinde bir kromatografi odası içinde 10 dakika boyunca, gelişmiş TLC levha inkübe edin.
Bir lipaz Fizyolojik Yüzey 5. İn Vitro Kimlik
NOT: Bir in vitro yöntemde, ilgi konusu lipaz mukabil Hidrolojik izole lipitler veya bağımsız olarak saf bir lipit karışımı dönüştürmek olup olmadığını incelemek3.2 optimum olarak tanımlanan koşullar altında Salman kararı ürünleri.
- PC-spesifik fosfolipaz C SMc00171 (5.2), fosfolipaz A (5.3), ve DAG lipaz SMc01003 için Tablo 3'de göre enzim testleri için pipet şemalarını kullanın aktivitesini (5.4).
- PC özgü fosfolipaz C aktivitesinin (Tablo 3) belirlenmesi.
- 1.5 ml mikrosantrifüj tüpü için, dakika başına 5000 sayımları toplam 14 C-etiketli PC (CPM) ve Triton X-100 içeren bir çözelti ilave edin.
- bir azot akımı altında karıştırın ve kurutulur.
- 99.5 ul bir son hacim elde etmek için dietanolamin-HCl, pH 9.8 tampon, hem de NaCI ve MnCl2 çözümleri ve bidistile su ilave edilir. 5 saniye vorteksleyin.
- Enzim (5 ug protein), 0.5 ul ekle reaksiyonun başlatılması için (örneğin, bir hücre barındırmayan özü içinde SMc00171 mevcut aşırı eksprese). kısaca karıştırın.
- 4 saat 30 ° C'de inkübe edin.
- tarafından reaksiyonu durdurmakmetanol, 250 | il kloroform 125 ul eklenmesi.
- daha önce açıklandığı gibi lipidler (4.2) ayıklayın.
- tek boyutlu (1D) -TLC ile ayırın lipidler (4.3.2 ve 4.4) ve PSL görüntüleme ile bunları analiz.
- Fosfolipaz A aktivitesinin belirlenmesi (Tablo 3).
- 1.5 ml mikrosantrifüj tüpü için, 5000 toplam 14 C-etiketli fosfolipidlerin CPM ve Triton X-100 ihtiva eden bir çözelti ilave edin.
- bir azot akımı altında karıştırın ve kurutulur.
- bir son 100 ul tahlil için, Tris-HCI, pH 8.5 tampon, NaCl çözeltisi ve su ilave edilir. 5 saniye vorteksleyin.
- Enzim 5 ul (50 ug protein), ekleme (örneğin, bir hücre barındırmayan özü içinde SMc00930 ya SMc01003 mevcut aşırı eksprese).
- 5 saat boyunca 30 ° C'de inkübe edin.
- metanol, 250 | il kloroform 125 ul ilave ederek reaksiyonu durdurun.
- Ayrı daha önce açıklandığı gibi lipidler (4.2) Özü1D-TLC 130 mi kloroform, 50 mL metanol, ve mobil faz olarak 20 mL buzlu asetik asit kullanılarak PSL görüntüleme ile analiz ile bunları.
- diaçilgliserol (DAG) lipaz aktivitesinin belirlenmesi.
- 14C-etiketli DAG hazırlanması.
- Radyoetiket S. meliloti kültürleri (4.1) ve açıklandığı gibi (bkz: 4.2) kutup lipitleri ayıklayın. Ayrı S. metanol: kloroform 1D-TLC ile meliloti toplam lipid özler, asetik asit (130: 50: 20; hacim / hacim) 4.3.1 ikinci boyutta ayrılması için tarif edilen koşullar kullanılarak.
- iyot boyama ile PC görselleştirmek ve fosfatidilkolin (PC) lokalizasyonu işaretlemek için bir kalem kullanın.
- 4,5 de tarif edildiği gibi radyoaktif olarak işaretlenmiş bir PC izole edin.
- sintilasyon sayımı ile çıkarılan PC ölçmek.
NOT: Yaklaşık 320.000 cpm PC bekleniyor. - 50 mM Tris-HCI, pH 7.2, 0.5 Clostridium perfringens elde edilen fosfolipaz C 0.1 U PC (250,000 cpm) tedavisinde% Triton X-100 ve 100 ul toplam hacim içinde 2 saat süreyle, 10 mM CaCl2 ve metanol 250 ul kloroform 125 ul ilave etmek suretiyle reaksiyonu durdurun.
- 1D-TLC (4.3.2 bakınız) onlar tarafından daha önce ve ayrı açıklandığı gibi lipidler ayıklayın.
- Silika plakası diasilgliserol izole etmek ve (4.2 de tarif edildiği gibi), sintilasyon sayımı ile ölçmek
- Diaçilgliserol lipaz deneyi (Tablo 3).
- 1.5 ml mikrosantrifüj tüpü için, 5000 14C etiketli DAG CPM ve Triton X-100 ihtiva eden bir çözelti ilave edin.
- bir azot akımı altında karıştırın ve kurutulur.
- bir son 100 ul tahlil için, Tris-HCl (pH 9.0) tampon maddesi, bir NaCI solüsyonu ve bidistile su ilave edilir. 5 saniye vorteksleyin.
- enzim 5 ul (hücresiz ekstraktı 50 ug protein) eklenerek reaksiyon başlatılır.
- 4 saat 30 ° C'de inkübe edin.
- Metanol 250 ul ilave ederek reaksiyonu durdurunDaha önce tarif edildiği gibi kloroform ve ekstre lipidlerin D 125 ul (4.2).
- 1D-TLC (4.1.3.2) ve daha sonraki PSL görüntüleme nötr kutup lipidlerin analiz edin.
- 14C-etiketli DAG hazırlanması.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Bis p-nitrofenil Fosfat PC özgü Fosfolipaz C SMc00171 Faaliyet
E. elde edilen hücre içermeyen özütler E. coli BL21 (DE3) 'smc00171 oluşan s -np ölçme bir spektrofotometrik enzimatik analiz kullanılarak, bis- p-nitrofenil fosfat esterleri hidrolize etme kabiliyetleri açısından incelendi ifad...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Son 20 yılda, birçok organizmanın genomu dizilimi edilmiş ve genom dizisi verilerin bir zenginlik üretilen olmasına rağmen, fonksiyonel yorumlama geride ve bu nedenle genom fonksiyonunun anlayışımızı engellemektedir edilir. genomları Gen fonksiyonları genellikle bilinen işlev veya korunmuş motiflerinin oluşum genlerine benzerliğine dayalı atanır. Bununla birlikte, belirli bir genin tam işlevi genellikle bilinmez. Özellikle, enzimler için yapısal genler nedeniyle kolaylıkla en enzimler iki yüzey...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarlar ifşa hiçbir şey yok.
Teşekkürler
Bu eser (Investigación en Fronteras de la Ciencia de Investigación Científica BasicA 82614, 153998, 253549, ve 178359 yanı sıra 118) ve Dirección Genel de Consejo Nacional de Ciencias y Tecnología-México (CONACyT-Meksika) hibe ile desteklenmiştir (DGAPA-UNAM; PAPIIT IN202616, IN203612) asuntos de Kişisel Académico-Universidad Nacional Autonoma de México.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
Referanslar
- Nelson, D. L., Cox, M. M. Lehninger, Principles of Biochemistry. , W. H. Freeman and Company. New York. (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 5th, Elsevier. Amsterdam, The Netherlands. 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids? Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria? Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998(2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115(2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. Experiments in Molecular Genetics. , Cold Spring Harbor Laboratory Press. Plainview, NY, USA. (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610(2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. Phosphorimager SI User´s Guide. , http://www.camd.lsu.edu/SAXS/Files/PIManual.pdf (1994).
- Dixon, M., Webb, E. Enzymes: Third Edition. , Longman, USA. (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , Article ID 306798 (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. Protein Purification, Principles and Practice. , Springer. New York. (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır