A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
באמצעות תלת-צבעי קשת/קשתית מולקולה בודדת ללמוד הקורלציה של אינטראקציות חלבון
In This Article
Summary
כאן, אנו מציגים פרוטוקול כדי לקבל נתונים smFRET תלת-צבעי וניתוח שלה עם אנסמבל תלת-ממד מודל מרקוב חבוי. מתוך תפיסה זו, מדענים יכולים לחלץ מידע קינטי מערכות חלבונים מורכבים, כולל cooperativity או אינטראקציות מתואם.
Abstract
העברת אנרגיה תהודה של מולקולה בודדת פורסטר (smFRET) הפך טכניקה biophysical בשימוש נרחב כדי ללמוד את הדינמיקה של מולקולות. עבור רבים למכונות המולקולריות בחלבונים התא יש לפעול יחד עם שותפים אינטראקציה במחזור תפקודית כדי למלא את המשימה שלהם. ההרחבה של שני צבעים לצבע מרובה smFRET מאפשר לחקור בו זמנית אינטראקציה אחד או יותר או לשינוי הסתגלותי. זה לא רק מוסיף מימד חדש ניסויים smFRET אך גם מציעה את האפשרות ייחודי ללמוד ישירות רצף האירועים כדי לזהות אינטראקציות מתואם כאשר באמצעות דוגמה קיבוע פלורסצנטיות החזרה גמורה מיקרוסקופ (TIRFM). לכן, צבע רב smFRET הוא כלי רב-תכליתי ללמוד למערכות ביולוגיות מתחמי באופן כמותי, בפירוט שעצם בעבר.
כאן, אנחנו מדגימים כיצד להתגבר על האתגרים המיוחדים של צבע רב smFRET ניסויים על חלבונים. אנו מציגים נתונים היסטוריים פרוטוקולים להשגת הנתונים, לחילוץ מידע קינטי. זה כולל מעקב קריטריוני הבחירה מהמדינה, ההתאוששות של המדינה מסלולים מתוך נתוני רעש באמצעות אנסמבל תלת-ממד של מודל מרקוב חבוי (הממ). לעומת שיטות אחרות, המידע קינטי לא החלים להתעכב זמן היסטוגרמות אלא ישירות מאת הממ. המסגרת נראות מקסימלית מאפשר לנו להעריך באופן ביקורתי את מודל קינטי וכדי לספק אי ודאות משמעותיים על המחירים.
על-ידי החלת שיטת שלנו החלבון הלם חום 90 (Hsp90), אנו מסוגלים disentangle האיגוד נוקלאוטיד והשינויים הסתגלותי הכללית של החלבון. זה מאפשר לנו לצפות ישירות את cooperativity בין איגוד נוקלאוטיד שני הכיסים של דיימר Hsp90.
Introduction
חלבונים רבים ממלאים תפקידם במתחמי דינמי עם מולקולות אחרות, מתווכת על-ידי שינויים הסתגלותי ואגודות ארעי על מגוון רחב של צירי זמן-1,-2,-3. מצמידים חיצוני כמקור אנרגיה (למשל, ATP) אינטראקציות דינמי אלה יכולים להוביל כיוון במחזור פונקציונלי ולתחזק בסופו של דבר ללא שיווי משקל היציב-המדינה בתא, תנאי מוקדם לכל החיים.
כדי להבין באופן מלא את מכונות מולקולריות אלה, תיאור סטטי מודרכת על ידי מחקרים מבניים אינה מספיקה. בנוסף, זה חיוני לא ידע קינטי מהמודל המשמש כבסיס וכדי לקבוע את קבועי קצב קינטי. קיימות מספר שיטות לאפשר לחוקרים ללמוד את הדינמיקה של האינטראקציות בינארית בין שתי מולקולות של עניין, למשל, פני השטח פלזמון תהודה, שיטות הרפיה עם הבדיקה ספקטרוסקופיות (למשל, קפיצה או זרימה עצר טכניקות), ותהודה מגנטית גרעינית. אולם, ישימות שלהם הוא ברוב המקרים מוגבל שתי המדינות מערכות פשוטות (למשל, אחד מאוגד, מדינה לא מאוגד) עקב בממוצע הטמון בניסויים בצובר. במקרים שבהם מעורבים יותר מדינות או intermediates, הן מפיקות רק תערובת מורכבת של הקבועים קצב. מולקולה בודדת שיטות כגון פינצטה אופטית או מגנטי או smFRET שני צבעים, קרי, תורם אחד אחד מקבל fluorophore, עם דגימת משטח. מרותק למיטה יכול לשחזר את קבועי קצב לכל נצפו שינויים הסתגלותי. אולם, כשמדובר אינטראקציות המשפיעים על אתר קישור אחד או יותר, שיטות אלה נשארים מוגבל, המידע לאינטראקציות מתאם אפשרי של השניים (או יותר) רק יהיה נגיש דרך עקיפה מסקנות ערכה של ניסויים.
צבע רב smFRET4,5,6,7,8,9 מציע הזדמנות ללמוד את האינטראקציה בין רכיבים אלה באופן ישיר, בזמן אמת, תחת תנאים ליד-פיזיולוגיים10. זה מתיר אחד לחקור לדוגמה, הכריכה תלויי-קונפורמציה של ליגנד או חלבון אחר8,9,11. הגישה הכללית המובאת כאן היא לסמן את protein(s) של עניין-עמדות מסוימות, כדי לצרף חלבון אחד על פני השטח של התא מדידה, וכדי לעקוב אחר עוצמת קרינה פלואורסצנטית לאורך זמן על TIRFM פריזמה-סוג (עבור פרטים ראה 9 , 12). הקרבה המרחבי של צבעים שונים ואז יכול להיקבע מתוך העברת האנרגיה ביניהם. תיוג אסטרטגיות עשויים להשתנות מחלבון לחלבון (נבדקה 13), קווים מנחים כדי להימנע חפצים במדידות smFRET קיימים14.
מאז צבע התורם יכול להעביר אנרגיה שונה מקבל צבע בניסוי צבע רב smFRET, המיקום היחסי של כל צבעי אינו נגיש של עירור של צבע אחד לבד15,16. אבל בשילוב עם מתחלפים עירור לייזר (אלכס17, ו שנסקרה ב 18) שיטה זו מספקת את כל המידע-עתיים-פחות משנייה והרזולוציה תת ננומטר.
המנהל, נתוני מבנה יכולה להיות מושגת על ידי שימוש של המרחקים בין צבע ברזולוציה גבוהה מחושב משילוב של כל עוצמות קרינה פלואורסצנטית בניסוי צבע רב smFRET עם אלכס. עם זאת, כאן אנו מתמקדים זיהוי מצב, הפרדה, כמו גם הפקת מודלים קינטי, שבו צבע רב smFRET היא הכרחית. כאשר "רק" מבנה נחישות על ידי טריאנגולציה היא הרצויה, ערכת ניסויים פשוטים smFRET שני צבעים עם יחס אות לרעש גבוה יכול להיות בביצוע12,19.
אנו משתמשים על ידי קרינה פלואורסצנטית חלקית ( ) כמדד להעברת אנרגיה בין שני fluorophores7. PF מחושבת על פי עוצמת קרינה פלואורסצנטית מקביל היעילות סריג של ניסוי שני צבעים:
) כמדד להעברת אנרגיה בין שני fluorophores7. PF מחושבת על פי עוצמת קרינה פלואורסצנטית מקביל היעילות סריג של ניסוי שני צבעים:

איפה, 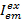 הוא עוצמת ב פליטה ערוץ em לאחר עירור עם צבע לשעברו- c הוא מקבל עם אורך הגל הארוך ביותר. זיהוי ערוצי מייצגים באותה התנוחה לדוגמה קאמרית, אבל הרשומה טווחי האור פלורסצנטיות ספקטרלי שונים. אותו מזהה עירור, פליטה נמצאים בשימוש פרוטוקול זה (קרי, "כחול", "ירוק" או "אדום").
הוא עוצמת ב פליטה ערוץ em לאחר עירור עם צבע לשעברו- c הוא מקבל עם אורך הגל הארוך ביותר. זיהוי ערוצי מייצגים באותה התנוחה לדוגמה קאמרית, אבל הרשומה טווחי האור פלורסצנטיות ספקטרלי שונים. אותו מזהה עירור, פליטה נמצאים בשימוש פרוטוקול זה (קרי, "כחול", "ירוק" או "אדום").
בגלל חסרונות ניסיוני עוצמות קרינה פלואורסצנטית נמדד תלויים לא רק על העברת אנרגיה, אלא גם על מאפייני fluorophore וההתקנה. על מנת להשיג את יעילות העברת אנרגיה אמיתית בין שתי fluorophores, עוצמות נמדד צריך לתקן. ההליך הבא מבוסס על התייחסות9. גורמים תיקון דליפה נראית לעין (lk, קרי, הגילוי של פוטונים מ fluorophore בערוץ המיועד צבע אחר), גאמה נראית לעין (ag, קרי, התשואה קוונטית זריחה של לצבוע את זיהוי יעילות של הערוץ) מתקבלים מן עקבות מולקולה בודדת המציגים מקבל של הלבנת אירוע.
דליפת לצבוע התורם לתוך בכל ערוץ אפשרי מקבל מחושבת על פי כל נקודות נתונים עקבות קרינה פלואורסצנטית מוקלט שבו מקבל לצבוע מולבן אך התורם הוא עדיין פלורסנט (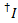 ):
):
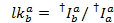
החציון של ההיסטוגרמה זליגת משמש כגורם זליגת נראית לעין. לאחר תיקון עבור דליפה, הגורם גמא לכאורה נקבע מתוך אותה קבוצה של עקבות. הוא מחושב על ידי חלוקת השינוי של קרינה פלואורסצנטית בערוץ מקבל על ידי השינוי של קרינה פלואורסצנטית בערוץ התורם על הלבנה של לצבוע מקבל:

כאשר c הוא שוב בערוץ זיהוי עבור מקבל עם אורך הגל הארוך ביותר. החציון של ההתפלגות וכתוצאה מכך משמש פקטור התיקון נראית לעין.
עוצמות המתוקן בכל אחד מהערוצים מתקבלים על ידי:
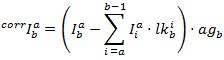
PF לאחר מכן מחושב על פי:

ניתן להפריד בין אוכלוסיות שונות במרחב רב ממדית שמקיפות את s PF. המיקום ואת הרוחב של כל מדינה נקבע על ידי התאמת הנתונים עם פונקציות גאוסיאנית רב-מימדי. אופטימיזציה עוקבות של אחד הממ גלובלית המבוססת על כל עקבות PF מספק תיאור כמותי של קינטיקה התצפיות. אפילו שינויים קטנים של התעריפים הן לזיהוי.
HMMs מספקים דרך של אומדן משוער מודל המדינה מאוסף של עקבות הזמן רועש. המערכת נחשבת באחד ערכה של בדידה, הברית מוסתרים בכל זמן נתון, התצפית בפועל (קרי, הפליטה) היא פונקציה הסתברותית של המדינה מוסתרים20. במקרה של נתונים smFRET TIRFM, פליטת הסתברויות bאני לכל המדינה אני שניתן למדל באמצעות פונקציות צפיפות ההסתברות גאוסיאנית רציפה. בנקודות זמן בדידים מפוזרים באופן קבוע, מעברים מאחד למדינה אחרת יכולה להתרחש לפי ההסתברות מעבר הזמן-שמורה, רק תלויה במצב הנוכחי. המטריקס המעבר A מכיל אלה הסתברות המעבר ij בין כל המדינות מוסתרים. התפלגות המצב ההתחלתי של  נותן ההסתברויות ספציפיים-
נותן ההסתברויות ספציפיים- 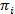 לנקודה בפעם הראשונה של עקבות הזמן. שימוש בגישה מרבי-הסבירות, פרמטרים אלה ניתן למטב בצורה הטובה ביותר לתאר את הנתונים עם קדימה-אחורה, באום-ולש אלגוריתמים20,21. זה מניב את estimators נראות מקסימלית (MLE). לבסוף, שניתן להסיק על רצף מצב סביר הפיק את המסלול של תצפיות עם אלגוריתם ויטרבי. בניגוד אחרים ניתוחים הממ של smFRET נתונים24,25,26 אנחנו לא משתמשים את הממ כמו גרידא "החלקה" של נתונים אבל תמצית המודל המדינה קינטי של ערכת הנתונים ללא הצורך עבור התאמת זמן להתעכב היסטוגרמות27. ניתוח הממ מתבצעת באמצעות קבצי script ללא צורך במיקור חוץ באמצעות איגור Pro. הטמעת הקוד מבוסס על התייחסות21. אנו מספקים ערכת תוכנה ונתונים למופת בדף שלנו לעקוב סעיפים 5 ו- 6 של פרוטוקול זה, (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). תוכנה מלא הינו זמין על פי בקשה.
לנקודה בפעם הראשונה של עקבות הזמן. שימוש בגישה מרבי-הסבירות, פרמטרים אלה ניתן למטב בצורה הטובה ביותר לתאר את הנתונים עם קדימה-אחורה, באום-ולש אלגוריתמים20,21. זה מניב את estimators נראות מקסימלית (MLE). לבסוף, שניתן להסיק על רצף מצב סביר הפיק את המסלול של תצפיות עם אלגוריתם ויטרבי. בניגוד אחרים ניתוחים הממ של smFRET נתונים24,25,26 אנחנו לא משתמשים את הממ כמו גרידא "החלקה" של נתונים אבל תמצית המודל המדינה קינטי של ערכת הנתונים ללא הצורך עבור התאמת זמן להתעכב היסטוגרמות27. ניתוח הממ מתבצעת באמצעות קבצי script ללא צורך במיקור חוץ באמצעות איגור Pro. הטמעת הקוד מבוסס על התייחסות21. אנו מספקים ערכת תוכנה ונתונים למופת בדף שלנו לעקוב סעיפים 5 ו- 6 של פרוטוקול זה, (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). תוכנה מלא הינו זמין על פי בקשה.
נקודות הנתונים עם PF זמן <-1 או PF > 2 בערוץ כל זיהוי מוקצים ההסתברות פליטה מינימלית על כל המדינות (10-200). פעולה זו מונעת מעברים מלאכותי בנקודות אלו נתונים.
הפרמטרים עבור ההסתברויות פליטה מתקבלים מן ההתאמה של ההיסטוגרמה PF 3D עם פונקציות לפי עקומת גאוס, כפי שמתואר בשלב 5.7. פרמטרים אלה נשמרים קבועים במהלך אופטימיזציה של הממ.
הגישה שהוצגו, הווקטור הפצה המצב ההתחלתי של המטריקס המעבר משמשים באופן גלובלי לתאר את ההרכב כולו עקבות. והם מתעדכנים בהתבסס על כל N מולקולות של ערכת הנתונים על פי הפניה27.
התחל פרמטרים עבור התפלגות המצב ההתחלתי נקבעים מתוך תחזיות דו-מימדית של ההיסטוגרמה PF (שלב 5.3) ולא ההסתברויות המעבר מוגדרות כ 0.05 למעט ההסתברויות כדי להישאר באותו המצב, אשר נבחרו כך כי ההסתברות לעזוב את מצב מסוים מנורמל לאחדות.
שיטה פרופיל הסבירות משמש כדי לתת מרווחי ביטחון (CIs) עבור כל המעבר המחירים21,22, אשר לשמש הערכות משמעותי עבור חוסר הבהירות. לחישוב גבולות של המודיע על שיעור מסוים, ההסתברות המעבר של ריבית קבוע בערך השונה MLE. זה מניב את λ דגם המבחן '. מבחן הסבירות יחס (LR) הסבירות 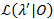 בהתחשב ערכת הנתונים 0 מתבצע על פי:
בהתחשב ערכת הנתונים 0 מתבצע על פי:
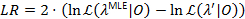
ביטחון 95% המאוגד עבור הפרמטר הוא הגיע כאשר LR חורג 3.841, quantile 95% של x2-התפלגות עם דרגת חופש אחת22,23.
כוחה של השיטה הוא הפגין באמצעות של Hsp90. החלבון בשפע זה נמצא ב חיידקים וב פרוקריוטים והוא חלק תגובת לחץ הסלולר28. זה מטרה סמים מבטיח טיפול בסרטן29. Hsp90 הוא homodimer עם כיס כריכה נוקלאוטיד בתחום N-מסוף של כל יחידה משנית30. זה יכול לעבור במעברים בין לפחות שני הייצורים החיים באופן גלובלי מובהק, אחד סגור,31,19,פתוח, בצורת V קונפורמציה N-מסוף אחד32. הטבע dimeric ישירות מעלה את השאלה של יחסי הגומלין בין האתרים מחייב נוקלאוטיד שני ב- Hsp90.
בחלק זה, אנו מספקים פרוטוקול צעד אחר צעד עבור רכישת נתונים וניתוח של ניסוי smFRET תלת-צבעי על שמרים Hsp90, נוקלאוטיד. הכריכה תלויי-קונפורמציה של המגבר-PNP שכותרתו fluorescently (AMP-PNP *, אנלוגים הלא-hydrolyzable ATP) ניתוח. היישום של ההליך המתואר היתרים המחקר של האיגוד נוקלאוטיד ו באותו הזמן את השינויים הסתגלותי של Hsp90, ובכך חושף את cooperativity בין איגוד נוקלאוטיד שני הכיסים של Hsp90.
Protocol
1. התקנה ותנאים מוקדמים
- מבצע המדידות צבע רב smFRET TIRFM מנסרה-סוג. תיאור של מלכודת שני צבעים כמו פרסום יופיטר ניתנת התייחסות12.
- בנו של TIRFM צבע רב. פריסה כללי מפורט ב 9.
- השימוש להחלפה, דיודה שאוב לייזרים רציפה wave מצב מוצק, ההופכים את השימוש תריסים מכני בנתיבים עירור מיותרים.
- מעסיקים פריזמה אסימטרי, מוארך, המונעת את ההשתקפות האחורי של הקרן עירור מן הצד האחורי כדי להזין את המטרה.
- להשתמש עדשות 2 אינץ אכרומטי אספריים fused סיליקה בנתיבים זיהוי זה לאסוף כמה שיותר אור ככל האפשר ולמנוע אוטומטי-זריחה, סטיות, למשל, עיוותים באזורים מחוץ למרכז של התמונה.
- מתמקדים כל נתיב זיהוי השבב של EMCCD עם עדשות נפרדות. פעולה זו מאפשרת התמקדות אופטימלית של כל ערוץ איתור.
התראה: Class 3B לייזרים משמשים את TIRFM. כלומר, שהם מסוכנים אם העין חשוף ישירות, אך השתקפויות ' מאטום לשקוף ' אינם מזיקים. להבטיח תאימות עם לייזר זהירות לפי תקנות הרשות המקומית לפני המערכת מופעלת.
- לקבוע מקדמי תיקון הגדרת ומאפייני fluorophore מראש באמצעות דגימות dsDNA.
- להשתמש במדגם גבוהה-סריג dsDNA אחת עבור כל צבע בשילוב עם מקבל בעל אורך הגל הארוך ביותר של עירור (עבור ההתקנה שהוצגו: Atto488-Atto647N, Atto550-Atto647N, Atto594-Atto647N). ודא כי ה-DNA בנוסף משתנה עם ביוטין.
- לדלל את הדגימה כדי 5 nM עם מאגר TNM (5 מ מ טריס pH 7.5, 5 מ מ NaCl, 20 מ מ MgCl2) ו- 2 מ מ Trolox (השתמש מאגר זה גם למדידה).
- לשתק את dsDNA כמתואר עבור Hsp90 בשלבים 2.5 ו- 2.7.
- חישוב מקדמי תיקון דליפה נראית לעין (lk), גאמה נראית לעין (ag) של מולקולה בודדת עקבות המראים אירוע הלבנת מקבל.
- לבנות תא תזרים זה כריך של שקופית קוורץ passivated פג/ביוטין-יתד, דקה בסרט כי הוא דבק על שני הצדדים, ולטובת מכסה. לקבלת פירוט פרוטוקולים עבור ניקוי של קוורץ שקופיות, פסיבציה ראה הפניה9.
- שימוש עבה (3 מ"מ) קוורץ שקופיות גאומטרית למנוע את האוסף של אור לייזר מפוזרים על הממשק קוורץ-גליצרול-קוורץ בין מנסרתיות השקופית קוורץ functionalized.
- להשתמש דק (מיקרומטר 40) איטום הסרט מרוסס עם דבק בצד ללא דבק. הסרט דק מפחית את המרחק בין מולקולות צמוד-השטח לבין המטרה. מחממים עד 80 ° C ולחץ על.
- במקום מכסה על החלק העליון. מחממים עד 80 ° C ולחץ על. השתמש טיפה של גליצרול בעת הצבת תא זרימה מעל המנסרה.
הערה: החומרים את המנסרה, השקופיות קוורץ, כמו גם של גליצרול הינם מתאימים-אינדקס.
- אקספרס Hsp90 מ האפייה בצורה של שתי מוטציות נקודה ציסטאין יחיד על המיקומים D61 או Q385. הוסף מוטיב של סליל מפותל C-מסוף כדי למנוע דיימר דיסוציאציה בריכוזים picomolar. תווית בנפרד את החלבונים המוטנטים ולהחליף את מונומרים כדי לקבל heterodimers עם Atto488 במיקום חומצת אמינו 61 ו- Atto550-חומצת אמינו עמדה 3859תוויות.
2. מדידה
- הפעל את תוכנת המצלמה והגדר את הפרמטרים הדמיה כמצוין להלן:
- הגדר את הטמפרטורה על החיישן קירור יהיה נמוך ככל האפשר (-95 מעלות צלזיוס עם קירור מים חיצוני) כדי להקטין את הרעש הנוכחי כהה.
- השתמש בהגדרות המצלמה ממוטבים עבור הקלטת מולקולה בודדת: 3.3 µs אנכי shift מהירות, השעון אנכי נורמלי מתח, 17 מגה-הרץ 16 סיביות אופקי הקריא, קדם הגברה הגברה 3, רווח של אלקטרון מכפיל 1,000.
- הגדר מפעילה של רכישת "חיצוניות", את זמן החשיפה לסרטים שיא 70 גב' באורך 750 רכישה מחזורים.
הערה: לכבות את האור בחדר כאשר המצלמה היא רכישת למניעת רוויה של החיישן EMCCD.
- צור תיקייה על SSD מקומי למדידה. בתוכנה הגדרות ללכת הרוכב < שמירה אוטומטית > הפעל < שמירה אוטומטית >, בחר את תבנית הקובץ "Tiff" עבור רכישת הסרט. בחר את התיקיה על SSD כמיקום שמירה אוטומטית.
- להתחיל את תוכנת סינון tunable פקדים אקוסטו אופטי (AOTF), התוכנה השולט על פעולת של הלייזרים, התוכנה ההדק המסנכרנת הלייזרים, AOTF, תריסי גילוי הנתיב, ואת המצלמות. להתאים את עוצמת הלייזר עם AOTF (ca. 3 mW לפני הכניסה את המנסרה) ולטעון את התבנית המניעים הנכונים.
- הר בעל הדגימה את המנסרה, תא זרימה לצרף אבובים, למקם את צינור כניסת בכוס microcentrifuge, להתחבר לאורך צינור עודפים משאבת מזרק. לרוקן את המיכל עם 150 מאגר µL, ליישר את עירור קרן, ולהתרכז. אקונומיקה כל מזהמים פלורסנט על פני השטח על-ידי הזזת באיטיות לאורך טווח זיהוי מוחלט השקופית עם כוח לייזר של בערך 10 mW עבור כל לייזרים. זה לוקח בערך 1 h.
הערה: אם לא צוין אחרת, המאגר משמש מכיל 40 מ מ HEPES pH 7.5, 150 מ מ. אשלגן כלורי, ו- 10 מ מ MgCl2. - ועצירה כ 300 µL של פתרון NeutrAvidin (0.25 mg/mL במאגר) החדרה של תקופת דגירה של 1 מינימלית ריקון החוצה NeutrAvidin לא מאוגד עם µL כ-300 מאגר, סומק של פתרון BSA (0.5 mg/mL במאגר) דרך החדר.
הערה: בלוק זה שנותר functionalization משטח פגמים על-ידי ספיחה לא ספציפי של BSA אל פני השטח. - לשתק את הדגימה על-ידי טעינת תא זרימה עם 150 µL של biotinylated ומתויגים Hsp90 בריכוזים העולה (מדולל מאגר + 0.5 מ"ג/מ"ל BSA) עד צפיפות המשטח מספיק, שהוא בדרך כלל המקרה-ריכוז של 5-10 בלילה. לשטוף חלבון לא מאוגד עם מאגר µL כ 300 + 0.5 מ"ג/מ"ל BSA.
- וחלילה µL 150 של 25 ננומטר AMP-PNP * מאגר + 0.5 מ"ג/מ"ל BSA. . תן לזה תקופת דגירה של 5 דקות, חזור על שלב זה פעם אחת כדי להבטיח ריכוז נוקלאוטיד הנכון.
הערה: לניסויים בנוכחות נוספים ללא תווית AMP-PNP, גם להוסיף מיקרומטר 250 אמפר-PNP. - מתחילים עם רכישת נתונים. עבור הכמות המתאימה של נתונים לרכוש כ 20 סרטים אשר לוקח בערך 1.5 h.
- העבר את המיקום של תא הדגימה בניצב קרן עירור עם צעד piezo לשנות את שדה הראייה.
- התאם את המוקד עם z-piezo השולטת על גובה המטרה במידת הצורך. זה לא צריך להיות נחוץ לעתים קרובות מדי כאשר התא מדידה נטענה ללא נטייה.
- להכין את ההקלטה של המצלמות על ידי לחיצה על < לקחת אות > תוכנה המצלמה והתחל את מחזורי עירור/רכישה באמצעות לחצן < התחל > בתוכנה על ההדק. זה מתחיל הרכישה של עוצמת קרינה פלואורסצנטית.
- לבצע רישום הערוץ על ידי הראשון הקלטת סרט עם חרוזים פלורסנט המציגים פליטת קרינה פלואורסצנטית בטווח ספקטרלי של כל הערוצים זיהוי של ההתקנה. לאחר מכן, לזהות עמדות חרוז בסרט כיול על-ידי חיפוש המקומות המבריקים, לקבוע את שהמיקום המרכזי החל Gaussian להתאים לפרופיל בעוצמה. שמור את הקואורדינטות של חרוזים מצויים כל הערוצים, להתאים הן את ההיסט מיפוי ב x - y-כיוון עם פולינום 2D של שלוש מעלות9.
3. מבחר של מולקולה בודדת עקבות
- ניתוח הנתונים נעשה באמצעות קבצי script ללא צורך במיקור חוץ באמצעות איגור Pro. לטעון את כל פריטי script הכרחי על-ידי פתיחת "iniTIRF.ipf" ובחר בסוג הנכון של הניסוי.
הערה: בחלק זה, < לחצן > מציין אלמנטים לחיץ בתפריט או בממשק המשתמש. קריאות לפונקציות מסומנים בתוך גרשיים, למשל, "דפוס"שלום עולם"". פקודות אלה יכולים להדביק לשורת הפקודה של איגור Pro (ללא המרכאות תוחם). - ודא שהפרמטרים עבור הרישום הערוץ זיהוי נטענים.
- התחל GUI על-ידי לחיצה על < smFRET חדש | ניתוח GUI >.
- לטעון את הסרטים (קרי, רצף מסגרות עם 512 x 512 פיקסלים המאוחסנים כ- 16 סיביות TIFF ערימות) להחזיק את עוצמת בערוצים המתאימים. לעשות זאת על ידי לחיצה על לחצן < טעינת הסרט > בחירת הקבצים ממצלמות כביכול "מאסטר", "העבד" אחד אחרי השני.
- לזהות את העמדות של פוטנציאל מולקולות יחיד על-ידי חיפוש המקומות המבריקים בסכום של המסגרות חמישה בערוץ זיהוי מסוים. לחשב את עמדות כל הערוצים זיהוי אחרים מהמיפוי הערוץ המקביל. כדי להשיג את האיתור עוצמת קרינה פלואורסצנטית, לסכם את עוצמת ריבוע פיקסל סביב המיקום המרכזי לכל מסגרת. לעשות זאת על ידי לחיצה על לחצן < למצוא עקבות > GUI.
הערה: האורך לצד הכיכר (פיקסל) ניתנת על ידי: 2 * < סכום Pxs > + 1. - עבור כל מולקולה, לחשב מעקב משותף בעוצמה raw כסכום של כל עקבות של המקום הזה עם אותו צבע עירור. להעריך את הפרופיל בעוצמה של המולקולה של כל הערוצים עבור הקריטריונים הבאים:
- מישור שטוח בערך האינטנסיביות raw משותפת, הלבנת יחיד צעד כל עירור צבעים, התנהגות anticorrelated בערוצים המתאימים זיהוי, איתור של פלואורסצנטי אדום (המציין סריג המגבר-PNP מאוגד *) לפחות פעם אחת בתוך מעקב, אין מספר השלבים ב פלואורסצנטי אדום, מה שמצביע על הנוכחות של המגבר-PNP שני * מאוגדים אחד Hsp90 דימר.
- שמור את עקבות קרינה פלואורסצנטית המקום עבור ניתוח נוסף אם קריטריונים אלה מתקיימים. לעשות זאת באמצעות בחירת המעקב עם הסמן ולאחר מכן הקשה על לחצן < שמור > בגרף "צירי זמן". באופן ידני לבדוק את העקבות בעוצמה של כל הערוצים זיהוי שלושה לאחר עירור כחול כ-200 מולקולות לכל סרט.
4. חישוב של עקבות קרינה פלואורסצנטית חלקית
- הצגת כל עקבות עוצמת קרינה פלואורסצנטית מאחת המולקולות שנשמרו. השתמש הסמנים בגרף כדי לבחור טווחי הזמן.
- בחר מרווח זמן שבו כל fluorophores הם מולבן כבר. עוצמת רקע רשע שמחושבים לטווח זה, להחסירו המעקב בעוצמה בכל אחד מהערוצים. לעשות זאת באמצעות לחיצה על לחצן < רקע >.
- בחר את הטווח יעילות סריג, שבו לפחות בשני הצבעים המצורפת Hsp90 (Atto488 ו Atto550) נוכחים. הקפד לכלול עקבות המכילים אירוע מהבהב (ראה איור 3B). אירועים אלה מאופיינים של עוצמת קרינה פלואורסצנטית פתע בכל אחד מהערוצים ללא עלייה הנלווה לאף ערוץ אחר.
- לחשב את עקבות PF . לעשות זאת באמצעות לחיצה על לחצן < PF Calc >. מקדמי תיקון מראש לכאורה דליפה (lk), גאמה נראית לעין (ag) חלות על עוצמת raw על מנת לתקן צילום-גופנית, מאפייני תוכנית ההתקנה.
5. האוכלוסייה בחירה, התאמה היסטוגרמה תלת-ממד
- הסר את מולקולות הצג את יחס אות לרעש נמוך ב- PF העקבות. מולקולות החורגות את מרווח הזמן [-1; 2] עקבות PF עבור יותר מ-10% של המסגרות יוסרו ערכת הנתונים. לעשות זאת על ידי הפעלת "RemoveTracesLowSNR()" מתוך חלון הפקודה.
- לחשב תחזיות 2D נשברתי הנתונים PF . מגרש
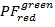 על
על 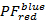 ,
, 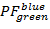 על
על 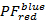 בטווח [-0.5; 1.5] עם רזולוציה של 100 100x סלים. לשם כך, בצע:
בטווח [-0.5; 1.5] עם רזולוציה של 100 100x סלים. לשם כך, בצע:- "HistFret2D ("r_b","r_g", binHist = 100)"
- "HistFret2D ("r_b","g_b", binHist = 100); MoveWindow 553.5, 42.5, 1055.25, 508.25"
- לקבוע את האוכלוסייה היחסי של כל מדינה ניתן להבחנה ב- 2D ההקרנות.
- להציג את הגרף המתאים בחזית ובצע "panelHist2DCount()".
- לחץ על לחצן < Init >, לצייר מצולע סגנון חופשי סביב הפסגה.
- לחץ על לחצן < ספירת >. את מספר נקודות הנתונים בתוך המצולע ואת המספר הכולל של נקודות נתונים בההקרנה מודפסות בחלון הפקודה.
- הכן היסטוגרמה תלת-ממדית של הנתונים PF על ידי ביצוע "HistFret3D ("g_b","r_b","r_g")".
- לנרמל את ההיסטוגרמה 3D אינטגרל של 1. בצע את הפעולות הבאות:
- "NewDataFolder/S fit0"
- "שכפול/O:: סריג: Hist3D, Hist3D"
- "Div משתנה /G = sum(Hist3D)*(DimDelta(Hist3D,0)) ^ 3"
- "Hist3D / = div; Div הדפסה"
- לספק ראשונית פרמטרים עבור גאוסיאן תלת-ממד מתאים ולהכין מבני נתונים הדרושים.
- לבצע "Gauss3D_initParam(); ערוך W_coef_old."
- להוסיף את אוכלוסיית המדינה לסוף של וקטור פרמטר.
- לבצע "Gauss3D_prepareFit()."
הערה: W_coef_old הוא וקטור שמכיל את הפרמטרים הראשוני עבור התאם. לכל מדינה זה אומר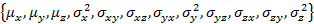 ואת האוכלוסייה המדינה משורשר בקצה של הווקטור. ודא כי מטריצת השונות המשותפת היא סימטרית.
ואת האוכלוסייה המדינה משורשר בקצה של הווקטור. ודא כי מטריצת השונות המשותפת היא סימטרית.
- מתאים הסכום של פונקציות גאוסיאנית 3D S כדי ההיסטוגרמה PF תלת-ממד, S להיות מספר מדינות ניתן להבחנה.
- לבצע "do3D()." פעולה זו עשויה להימשך שעה או יותר ב- office נורמלי PC, בהתאם לאיכות של הפרמטרים הראשונית.
- לבצע "postprocessFitMultiGauss3D(); evalFitMultiGauss3D(); ערוך W_coef."
- הצגת התוצאה מתאים. לכל אחד שני הקרנות 2D, השתמש בפקודות הבאות:
- "contourPF3D_new(0); contourPF3D_new(1); contourPF3D_new(2); contourPF3D_new(3); contourPF3D_new(4)"
- "contourPF3D_colorize()"
6. קינטי ניתוח עם אנסמבל תלת-ממד?
- להכין את אנסמבל הממ לרוץ כדי לחלץ את המידע קינטי. הממ אחד ממוטבת ממולקולות כל בערכת הנתונים. השתמש במידע המתקבל בשלב הקודם כדי להגדיר את המיקום ואת הרוחב של כל מדינה בשטח PF תלת-ממד.
- אתחל את ממשק המשתמש הממ (< הממ | Init הממ >) ובחר מספר מתאים של מדינות (במקרה של הנתונים Hsp90 פירוש < NumStates > = 5), מספר הממדים של אותות קלט (< NumDims > = 3), ואת סוג הקלט (< סוג הקלט > = "bgr תלת סריג").
- למטב את הפרמטרים של הממ בכך שהוא מאפשר את התוכנה מתכנסת את הסבירות הממ (ביצוע "prepENS_CONVERGE_gB(GetDataFolder(1),-14)") עד לשינוי מטריצת המעבר בהשוואה האיטראציה הקודמת יורד מתחת לסף מסוים (10-14 סכום השינוי המוחלט על כל הסתברות המעבר). זה מניב את MLE עבור המעבר ההסתברויות כשעה על סניף רגיל PC.
- חזור על הבחירה האוכלוסייה התאמה לפי עקומת גאוס, אופטימיזציה הממ עבור קבוצות משנה של הנתונים (למשל, 75% של ערכת הנתונים המלא). אם ערכת הנתונים המלא הושלם מיזוגו של ניסויים שונים, חזור על אופטימיזציה גם עבור כל אחד הניסויים יחיד. ניתוח של קבוצות משנה מאפשרת להעריך את הוודאות של בחירת האוכלוסייה ידנית, ההשתנות בתוך ערכת הנתונים.
- לחשב את המודיע על ההסתברויות המעבר, אשר הדו ח על הטרוגניות ערכת נתונים של דיוק הממ.
- לקבל הערכה גסה של גבולות המודיע על ידי הפעלת "תקליטור $(הבסיס: path3Dimport +"הממ"); loop_getCI_estimate_limits()."
- לחשב את הגבולות המדויקים של המודיע על ידי הפעלת:
- "loop_getCI_HMM_converge(1)"
- "CIresults_conv_new()"
- "תקליטור:: HMM_CIresult; reportCI_conv()"
- "תקליטור:: cmp_CI_conv; CI_plot2 ("הממ", doAppend = 0) "
- לדחוס את המידע קינטי זמין כדי לפשט את הפרשנות.
- אוספים מידע על הזמן נוקלאוטיד שכותרתו נשאר אל Hsp90. עושים זאת על ידי כיווץ המדינות המאוגדות לשדות AMP-PNP * (קרי, S1, S2 ו- S3, ראה גם באיור 2) ולעבד ההיסטוגרמה זמן להתעכב. לבצע "תקליטור $(הבסיס: path3Dimport +"הממ"); collapse_states_get_DT({0,1,1,1,0}) ", המשלב שוכנת של מדינות S0 ו- S4 וכן S1, S2ו- S3.
- לחלץ את הזמנים להתעכב מהנתיב ויטרבי לכל מדינה עניין והשווה ההיסטוגרמה זמן להתעכב וכתוצאה מכך בתנאי ניסוי שונים. לבצע "plot_collapsed_DT_Hist (wDTo_01110_record1)."
- עבור תמונה מפורטת יותר, התמוטטות מדינות נפרדות PF אך זהים מבחינה תפקודית כדי להקל את נתוני ניתוח נוסף, למשל, במקרה של ניסוי תלת-צבעי עם שכותרתו Hsp90, נוקלאוטיד, הברית S2 ו- S 3 יכולה להתכווץ. לבצע "collapse_states_get_DT({0,1,2,2,4})."
תוצאות
צבע רב smFRET המידות לאפשר זיהוי ישיר קורלציה בין שניים או יותר אתרים אינטראקציה ברורים. זה הופך את הטכניקה הייחודית לחקור מערכות רכיבים מרובים, כגון חלבון מתחמי. אנו מתמקדים המצגת של smFRET תלת-צבעי ניסוי כאן, אשר משמש דוגמה להמחשה.
תהליך העבוד...
Discussion
אנו מציגים בהליך ניסיוני להשיג smFRET תלת-צבעי נתונים עבור מערכת חלבונים מורכבים, תיאור שלב אחר שלב של הניתוח של מדידות אלה. גישה זו מציעה את האפשרות ייחודי להעריך ישירות המתאם בין מספר אתרים אינטראקציה או שינויים הסתגלותי.
על מנת לקבל נתונים יחיד מולקולה צבע רב מתאימים חלבוני...
Disclosures
המחברים מצהירים שאין ניגודי אינטרסים.
Acknowledgements
עבודה זו ממומן על ידי קרן מחקר גרמני (מוסד 39/969-1), המועצה האירופית למחקר באמצעות הסכם גרנט ERC 681891 (ש. ע.)..
Materials
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
References
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved