Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
À l’aide de trois couleurs frette de molécules simples pour étudier la corrélation des Interactions de protéine
Dans cet article
Résumé
Nous présentons ici un protocole afin d’obtenir des données de trois couleurs smFRET et son analyse avec un ensemble 3D modèle de Markov caché. Avec cette approche, les scientifiques peuvent extraire informations cinétiques systèmes complexe des protéines, y compris coopérativité ou interactions corrélées.
Résumé
Transfert d’énergie unique molécules Förster (smFRET) est devenu une technique largement utilisée biophysique pour étudier la dynamique des biomolécules. Pour beaucoup de machines moléculaires dans une protéines cellulaires ont à agir conjointement avec des partenaires d’interaction dans un cycle fonctionnel pour remplir leur mission. L’extension de deux couleurs à multicolore smFRET permet de sonder simultanément plus d’une interaction ou changement conformationnel. Cela non seulement ajoute une nouvelle dimension à des expériences de smFRET, mais il offre aussi la possibilité unique d’étudier directement la séquence des événements et de détecter des interactions corrélées lorsque vous utilisez un échantillon immobilisé et une fluorescence de la réflexion totale interne microscope (TIRFM). SmFRET multi-color est donc un outil polyvalent pour l’étude biomoléculaire complexes de manière quantitative et dans un détail auparavant irréalisable.
Ici, nous démontrons comment surmonter les défis particuliers de multicolore smFRET expériences sur les protéines. Nous présentons des protocoles détaillés pour obtenir les données et d’extraction d’informations cinétiques. Cela comprend les critères de sélection de trace, la séparation de l’État et la récupération des trajectoires de l’État dans les données bruitées en utilisant un ensemble 3D modèle de Markov cachés (HMM). Par rapport aux autres méthodes, l’information cinétique n’est pas récupérée des histogrammes de temps de s’attarder, mais directement à partir de la HMM. Le cadre du maximum de vraisemblance nous permet d’évaluer de façon critique le modèle cinétique et d’incertitudes significatives pour les tarifs.
En appliquant notre méthode à la protéine de choc thermique 90 (Hsp90), nous sommes en mesure de démêler la liaison des nucléotides et les globales changements de conformation de la protéine. Cela nous permet d’observer directement la coopération entre les deux poches de liaison de nucléotides du dimère Hsp90.
Introduction
Beaucoup de protéines remplissent leur fonction dans les dynamiques complexes avec d’autres molécules, médiées par des changements de conformation et associations transitoires sur une large gamme d’échelles de temps1,2,3. Couplé à une source d’énergie externe (p. ex., ATP) ces interactions dynamiques peuvent conduire à la directionnalité dans un cycle fonctionnel et en fin de compte maintenir l’état d’équilibre hors de l’équilibre dans une cellule, la condition sine qua non pour la vie.
Pour bien comprendre ces machines moléculaires, une description statique, guidée par des études structurales n’est pas suffisante. En outre, il est essentiel d’avoir une connaissance du modèle cinétique sous-jacent et de déterminer les constantes cinétiques. Plusieurs méthodes existantes aux chercheurs d’étudier la dynamique des interactions binaires entre deux molécules d’intérêt, par exemple, la résonance plasmonique de surface, des méthodes de relaxation avec une lecture spectroscopique (p. ex., saut ou arrêt d’écoulement techniques) et la résonance magnétique nucléaire. Cependant, leur applicabilité est le plus souvent limitée à des systèmes simples de deux États (par exemple, une liaison et un État indépendant) en raison de l’étalement inhérente à l’expérimentation en vrac. Dans le cas où plusieurs États ou intermédiaires sont impliqués, ils donnent seulement un mélange complexe des constantes de vitesse. Méthodes de molécules simples tels que des pinces optiques ou magnétiques ou deux couleurs smFRET, c.-à-d. un donneur et un accepteur fluorophore, auprès d’un échantillon de surface immobilisée peuvent récupérer les constantes de vitesse pour tous observé des changements de conformation. Cependant, quand il s’agit d’interactions qui touchent plus d’un accepteur, ces méthodes restent limitées et les informations sur les interactions de la corrélation possible entre les deux (ou plus) ne seront accessibles par l’intermédiaire de conclusions indirectes d’un ensemble d’expériences.
Multi-color smFRET4,5,6,7,8,9 offre la possibilité d’étudier l’interaction entre ces éléments directement, en temps réel et sous des conditions physiologiques près de10. Cela permet d’étudier par exemple, la liaison de conformation dépendant d’un ligand ou d’une autre protéine8,9,11. L’approche générale présentée ici est d’étiqueter les protéines d’intérêt aux positions spécifiques, d’attacher une protéine à la surface de la chambre de mesure et de suivre l’intensité de la fluorescence au fil du temps sur un TIRFM type de prisme (pour détails voir 9 , 12). la proximité spatiale des différents colorants peut décider du transfert d’énergie entre eux. Stratégies d’étiquetage peut varier de protéine à la protéine (évaluée à 13) et il existait des directives pour éviter les artefacts dans les mesures smFRET14.
Puisqu’un colorant donneur peut transférer l’énergie aux colorants différents accepteurs dans une expérience multi-color smFRET, la position relative de tous les colorants n’est pas accessible depuis l’excitation d’un colorant seul15,16. Mais en combinaison avec une alternance d’excitation de laser (ALEX17et examiné dans 18), cette méthode fournit toutes les informations spatio-temporelles à la seconde et résolution sous nanomètre.
En principe, haute résolution informations structurelles peuvent être réalisées en utilisant les distances inter-colorants calculée à partir de la combinaison de toutes les intensités de fluorescence dans une expérience multi-color smFRET avec ALEX. Cependant, ici, nous nous concentrons sur l’identification de l’État et de séparation, mais aussi l’extraction des modèles cinétiques, où smFRET multicolore est indispensable. Lorsque vous souhaitez « seulement » détermination de structure par triangulation, une série d’expériences de deux couleurs smFRET plus simples avec rapport signal-bruit élevé peut être effectué12,19.
Nous utilisons la fluorescence partielle ( ) comme un proxy pour le transfert d’énergie entre deux fluorophores7. Le PF est calculée à partir de l’intensité de fluorescence analogue à l’efficacité FRET d’une expérience de deux couleurs :
) comme un proxy pour le transfert d’énergie entre deux fluorophores7. Le PF est calculée à partir de l’intensité de fluorescence analogue à l’efficacité FRET d’une expérience de deux couleurs :

Où, 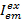 est l’intensité d’émission canal em après excitation avec couleur ex, et c est l’accepteur avec la longueur d’onde plus longue. Canaux de détection représente la même position dans le compartiment de mesure mais enregistrer différentes gammes spectrales de la lumière de fluorescence. Le même identificateur pour l’excitation et d’émission sont utilisés dans le présent protocole (c'est-à-dire, « bleu », « vert » et « rouge »).
est l’intensité d’émission canal em après excitation avec couleur ex, et c est l’accepteur avec la longueur d’onde plus longue. Canaux de détection représente la même position dans le compartiment de mesure mais enregistrer différentes gammes spectrales de la lumière de fluorescence. Le même identificateur pour l’excitation et d’émission sont utilisés dans le présent protocole (c'est-à-dire, « bleu », « vert » et « rouge »).
En raison de lacunes expérimentales, les intensités de fluorescence mesurée dépendent non seulement sur le transfert d’énergie, mais aussi sur les propriétés de configuration / fluorophore. Afin d’obtenir l’efficacité de transfert d’énergie véritable entre deux fluorophores, les intensités mesurées doivent être corrigées. La procédure suivante repose sur la référence9. Facteurs de correction pour la fuite apparente (lk, c'est-à-dire, la détection de photons d’un fluorophore dans un canal nommée pour un autre colorant) et gamma apparent (ag, c.-à-d. le rendement quantique de fluorescence du colorant et la efficacité de la détection du chenal) sont obtenus à partir des traces de molécules simples qui montrent un accepteur d’événement de blanchiment.
La fuite du colorant donneur dans tous les canaux possibles accepteur est calculée à partir de tous les points de données dans les traces de fluorescence enregistré où le colorant accepteur blanchi, mais le donateur est toujours fluorescent (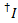 ) :
) :
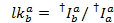
La médiane de l’histogramme de fuite est utilisée comme le facteur de fuite apparente. Après correction des fuites, le facteur gamma apparent est déterminé de la même série de traces. Il est calculé en divisant le changement de la fluorescence dans le canal de l’accepteur par le changement de fluorescence dans le chenal de donneur sur le blanchiment de la teinture de l’accepteur :

Où c est encore une fois le canal de détection pour l’accepteur avec la longueur d’onde plus longue. La médiane de la distribution qui en résulte est utilisée comme le facteur de correction apparente.
Les intensités corrigées dans chaque canal sont obtenues par :
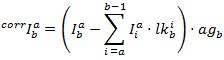
Le PF est ensuite calculé selon :

Des populations différentes peuvent être séparées dans l’espace multidimensionnel, enjambé par le s PF. La position et la largeur de chaque État est déterminé en ajustant les données avec des fonctions gaussiennes multidimensionnelles. Optimisation ultérieure d’un HMM global basé sur toutes les traces PF fournit une description quantitative de la cinétique observée. Même les petits changements des tarifs sont détectables.
HMMs permettent de déduire un modèle d’état d’une collection de tracés chronologiques bruyant. Le système est considéré comme en partie d’une série de discret, États cachés à un moment donné et l’observation réelle (c'est-à-dire l’émission) est une fonction probabiliste de cet état masqué20. Dans le cas des données TIRFM de smFRET, l’émission probabilités bje par état j’ai peuvent être modélisés par des fonctions de densité de probabilité gaussienne continue. Aux points régulièrement espacés de temps discret, transitions à partir d’un autre État peuvent se produire selon la probabilité de transition qui est invariant et dépend seulement de l’état actuel. La matrice de transition A contient ces probabilités de transition unij entre tous les États cachés. La répartition de l’état initial  donne les probabilités de spécifiques à l’état
donne les probabilités de spécifiques à l’état 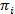 pour le premier point de temps d’une trace du temps. En utilisant une approche du maximum de vraisemblance, ces paramètres peuvent être optimisés pour décrire au mieux les données avec l’avant-arrière et algorithmes de Baum-Welch20,21. Cela donne les estimateurs du maximum de vraisemblance (MLE). Enfin, la séquence de l’État qui a probablement produit la trajectoire des observations peut être déduite avec l’algorithme de Viterbi. Contrairement à d’autres analyses HMM des smFRET données24,25,26 , nous n’utilisons pas le HMM comme un simple « lissage » des données, mais le modèle cinétique état extrait le jeu de données sans avoir besoin pour ajuster les temps de pause histogrammes27. HMM analyse se fait avec les scripts internes à l’aide de Igor Pro. Mise en œuvre du code repose sur21de référence. Nous fournissons un kit logiciel et données exemplaires sur notre site Web afin de suivre les sections 5 et 6 du présent protocole (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Logiciel complet est disponible sur demande.
pour le premier point de temps d’une trace du temps. En utilisant une approche du maximum de vraisemblance, ces paramètres peuvent être optimisés pour décrire au mieux les données avec l’avant-arrière et algorithmes de Baum-Welch20,21. Cela donne les estimateurs du maximum de vraisemblance (MLE). Enfin, la séquence de l’État qui a probablement produit la trajectoire des observations peut être déduite avec l’algorithme de Viterbi. Contrairement à d’autres analyses HMM des smFRET données24,25,26 , nous n’utilisons pas le HMM comme un simple « lissage » des données, mais le modèle cinétique état extrait le jeu de données sans avoir besoin pour ajuster les temps de pause histogrammes27. HMM analyse se fait avec les scripts internes à l’aide de Igor Pro. Mise en œuvre du code repose sur21de référence. Nous fournissons un kit logiciel et données exemplaires sur notre site Web afin de suivre les sections 5 et 6 du présent protocole (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Logiciel complet est disponible sur demande.
Fois points dans les données avec PF < -1 ou PF > 2 dans n’importe quel canal de détection sont assignés à la probabilité d’émission minimal pour tous les États (10-200). Vous éviterez les transitions artificielles à ces points de données.
Les paramètres pour les probabilités des émissions proviennent de l’ajustement de l’histogramme 3D de PF avec fonctions gaussiennes comme indiqué au point 5.7. Ces paramètres sont maintenus fixes durant l’optimisation de la HMM.
Dans l’approche présentée, le vecteur de distribution état initial et la matrice de transition sont utilisés dans le monde pour décrire l’ensemble entier de traces. Elles sont mises à jour basées sur toutes les molécules de N de l’ensemble des données selon la référence27.
Les paramètres de démarrage pour la distribution de l’état initial sont déterminés à partir des projections 2D de l’histogramme PF (étape 5.3) et les probabilités de transition sont définies à 0,05 à l’exception des probabilités de rester dans le même État, qui sont choisis de telle que la probabilité de quitter un certain état est normalisée à l’unité.
Une méthode de profilage de probabilité est utilisée pour donner des intervalles de confiance (IC) pour toute transition taux21,22, qui servent des estimations significatives pour leur incertitude. Pour calculer les limites de la CI pour un taux spécifique, la probabilité de transition d’intérêt est fixée à une valeur autre que le MLE. Cela donne le test modèle λ'. Un test du rapport (LR) de probabilité du risque 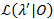 compte tenu de l’ensemble de données 0 est effectuée conformément à :
compte tenu de l’ensemble de données 0 est effectuée conformément à :
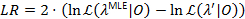
La confiance de 95 % liée pour le paramètre est atteinte lorsque LR dépasse 3.841, la quantile de 95 % d’un x2-distribution avec un seul degré de liberté22,23.
La puissance de la méthode est démontrée à l’aide de la Hsp90. Cette protéine abondante se trouve chez les bactéries et des eucaryotes et fait partie de la réponse de stress cellulaire28. C’est une cible de médicament prometteur dans le traitement de cancer du29. HSP90 est un homodimère avec une poche de liaison de nucléotides dans le domaine N-terminal de chaque sous-unité30. Il peut subir une transition entre au moins deux conformations globalement distinctes, l’un fermé et un N-terminal conformation ouverte en forme de V,19,31,32. La nature dimère pose directement la question de l’interaction entre les deux sites de liaison de nucléotides dans Hsp90.
Par la suite, nous fournissons un protocole étape par étape d’acquisition de données et d’analyse d’une expérience de trois couleurs smFRET sur levure Hsp90 et nucléotides. La liaison de conformation dépendant du fluorescent étiqueté AMP-PNP (AMP-PNP *, un analogue non hydrolysable de l’ATP) est analysé. L’application de la procédure décrite permet l’étude de la liaison de nucléotides et en même temps les changements de conformation de la Hsp90 et révèle ainsi la coopération entre les deux poches de liaison de nucléotides de Hsp90.
Protocole
1. installation et configuration requise
- Effectuer les mesures multicolore smFRET sur un TIRFM type de prisme. Une description d’une installation de deux couleurs comme une publication JoVE est donnée référence à12.
- Construire un TIRFM multicolore. Une présentation générale est détaillée dans 9.
- Utilisation commutable, diode pompé onde entretenue lasers à état solide, qui rendent l’utilisation des volets mécaniques dans les chemins de l’excitation inutile.
- Employer un prisme asymétrique, allongé qui empêche la réflexion arrière de la poutre de l’excitation de l’arrière pour entrer dans l’objectif.
- Utiliser des lentilles achromatiques pouces 2 asphériques en silice fondue dans les chemins de détection qui recueillent autant de lumière que possible et éviter l’auto-fluorescence et aberrations, par exemple, des distorsions dans les régions excentrées de l’image.
- Chaque chemin d’accès détection l’accent sur la puce de l’EMCCD avec un objectif distinct. Cela permet une mise au point optimale de chaque canal de détection.
Mise en garde : Les lasers de classe 3 b sont utilisés dans le TIRFM. Cela signifie qu’ils sont dangereux si le œil est exposé directement, mais les réflexions diffuses ne sont pas nuisibles. Assurer la conformité avec laser précautions de sécurité conformément à la réglementation de l’administration locale avant que le système est exploité.
- Déterminer les facteurs de correction pour les propriétés de configuration et fluorophore au préalable à l’aide d’échantillons d’ADN double brin.
- Utiliser un échantillon d’ADN double brin haut-FRET pour chaque colorant en combinaison avec l’accepteur ayant la plus longue longueur d’onde d’excitation (pour la configuration présentée : Atto488-Atto647N, Atto550-Atto647N, Atto594-Atto647N). Assurez-vous que l’ADN est en outre modifié avec un biotine.
- Diluer l’échantillon à 5 nM avec un tampon TNM (5 mM Tris pH 7.5, 5 mM NaCl, 20 mM MgCl2) et 2 mM Trolox (aussi utiliser cette mémoire tampon pour la mesure).
- Immobiliser le dsDNA comme décrit pour Hsp90 aux points 2.5 et 2.7.
- Calculer les facteurs de correction pour la fuite apparente (lk) et gamma apparent (ag) des traces de molécules simples qui montrent un événement blanchiment accepteur.
- Construire une chambre à flux qui est un "sandwich" d’une lame de quartz passivé PEG/biotine-PEG, une couche mince qui est adhésif sur les deux côtés et une lamelle couvre-objet. Protocoles de détail pour le nettoyage du quartz diapositives et passivation voir référence9.
- Utilisation (3 mm) d’épaisseur quartz glisse à géométriquement empêcher la collecte de la lumière laser dispersée à l’interface de quartz-glycérol-quartz entre le prisme et la lame de quartz fonctionnalisés.
- Utiliser un mince (40 µm) film qui est pulvérisé avec de la colle sur le côté non adhésif d’étanchéité. La couche mince permet de réduire la distance entre l’objectif et les molécules attachées à la surface. Chauffer à 80 ° C et appuyez sur.
- Placez une lamelle couvre-objet sur le dessus. Chauffer à 80 ° C et appuyez sur. Utiliser une goutte de glycérol lorsque vous placez la chambre flow sur le prisme.
Remarque : Les matériaux de prisme et les lames de quartz ainsi que le glycérol sont appariés selon l’indice.
- Exprimer la Hsp90 chez Saccharomyces cerevisiae , sous la forme de deux mutants de point unique de cystéine aux positions D61 ou Q385. Ajouter un motif C-terminal de coiled-coil pour éviter la dissociation dimère picomoles concentrations. Étiqueter les protéines mutantes séparément et échanger les monomères pour obtenir des hétérodimères marquée à Atto488 à la position de l’acide aminé 61 et Atto550 à l’acide aminé poste 3859.
2. mesure
- Lancez le logiciel de la caméra et régler les paramètres d’imagerie tel que précisé ci-dessous :
- Régler la température pour le capteur de refroidissement aussi bas que possible (-95 ° C à refroidissement par eau externe) pour diminuer le bruit courant sombre.
- Utiliser les réglages de l’appareil qui sont optimisés pour l’enregistrement de la molécule unique : 3,3 µs décalage vertical Vitesse, tension normale horloge verticale, 17 MHz 16-bit horizontal donne lecture, gain de pré-amplification 3, gain d’électrons multiplicateur 1 000.
- Définissez le déclenchement de l’acquisition à le « Extérieur » et le temps d’exposition à 70 films Record de Mme avec une longueur de 750 cycles d’acquisition.
NOTE : Éteindre la lumière lorsque la caméra fait l’acquisition pour éviter la saturation du capteur EMCCD.
- Créez un dossier sur le SSD local pour la mesure. Dans le logiciel paramètres aller au coureur < Auto-Save > Activer < Auto-save > et choisissent le format de fichier « Tiff » pour l’acquisition de film. Sélectionnez le dossier sur le SSD comme emplacement de sauvegarde automatique.
- Démarrez le logiciel qui filtre accordable contrôles acousto-optique (AOTF), le logiciel qui contrôle le fonctionnement des lasers et le logiciel de déclencheur qui synchronise les lasers, AOTF, volets dans le chemin d’accès détection et des caméras. Ajuster la puissance du laser avec le AOTF (ca. 3 mW avant d’entrer dans le prisme) et charger le modèle correct de déclenchement.
- Monter le porte-échantillon avec le prisme et la chambre de flux, raccorder le tube, placer le tuyau d’arrivée dans une tasse de microcentrifuge et raccordez le tuyau de sortie d’une pompe à seringue. Rincer la chambre avec environ 150 µL de tampon, alignez l’excitation faisceau et se concentrer. Tout contaminant fluorescent sur la surface d’eau de Javel en déplaçant lentement le long de la plage de détection complète de la lame avec une puissance de laser d’environ 10 mW pour tous les lasers. Cela prend environ 1 h.
Remarque : si pas d’indication contraire, le tampon utilisé contient 40 mM HEPES pH 7.5, KCl, de 150 mM et 10 mM MgCl2. - Rincer environ 300 µL d’une solution de neutravidine (0,25 mg/mL dans le tampon) dans la chambre et incuber pendant 1 min. Flush out neutravidine indépendant avec tampon et chasse environ 300 µL d’une solution de BSA (0,5 mg/mL dans le tampon) dans la chambre.
Remarque : Les défauts de ce bloc restant la fonctionnalisation de surface par adsorption non spécifique de BSA à la surface. - Immobiliser l’échantillon en chargeant la chambre flow avec environ 150 µL de la biotinylé et étiqueté Hsp90 à la hausse des concentrations (diluées dans le tampon + 0,5 mg/mL de BSA) jusqu'à atteindre une densité de surface suffisante, qui est généralement le cas à une concentration de 5-22:00. Laver des protéines non lié avec environ 300 µL de tampon + BSA de 0,5 mg/mL.
- RAS dans 150 µL de 25 nM AMP-PNP * tampon + 0,5 mg/mL de BSA. Laisser incuber pendant 5 minutes et répétez cette étape une fois afin d’assurer la concentration correcte nucléotides.
NOTE : Pour des expériences en présence de l’AMP-PNP supplémentaire, sans étiquette, il faut ajouter également 250 µM AMP-PNP. - Commencer par l’acquisition de données. Pour une quantité appropriée de données acquérir environ 20 films, ce qui prend environ 1,5 h.
- Modifie la position de la chambre de mesure perpendiculairement à la poutre d’excitation avec un stepper piezo pour modifier le champ de vision.
- Ajuster la mise au point avec le z-piezo qui contrôle la hauteur de l’objectif si nécessaire. Cela ne devrait pas être nécessaire trop souvent lorsque la chambre de mesure est montée sans inclinaison.
- Préparer l’enregistrement des caméras en appuyant sur < prendre Signal > dans le logiciel de la caméra et de commencer les cycles d’excitation/acquisition en utilisant < Démarrer > dans le logiciel de déclencheur. Cette opération démarre l’acquisition de l’intensité de la fluorescence.
- Effectuez un enregistrement canal par le premier enregistrement d’un film avec des perles fluorescentes qui montrent l’émission de fluorescence dans le domaine spectral de tous les canaux de détection de l’installation. Puis, détecter les positions de perle dans le film de calibrage en recherchant les endroits les plus brillants et déterminer que la position centrale d’une gaussienne correspondait au profil d’intensité. Enregistrez les coordonnées de perles qui sont trouvent dans tous les canaux et s’adapter tant le décalage de la cartographie dans x - et y-direction avec un 2D polynôme de degré trois9.
3. sélection des Traces de molécules simples
- Analyse des données se fait avec les scripts internes à l’aide de Igor Pro. Chargez tous les scripts nécessaires en ouvrant « iniTIRF.ipf » et sélectionnez le type d’expérience.
Remarque : dans ce qui suit, < Button > spécifie les éléments cliquables dans le menu ou l’interface utilisateur. Appels de fonction sont indiqués entre guillemets, par exemple, « Print « Bonjour tout le monde » ». Ces commandes peuvent être collés à la ligne de commande de Igor Pro (sans les guillemets englobants). - Assurez-vous que les paramètres pour l’enregistrement de canaux de détection sont chargés.
- Démarrer l’interface graphique en cliquant sur < smFRET nouveau | Analyse GUI >.
- Charger les films (c'est-à-dire, la séquence d’images avec 512 x 512 pixels, stockées sous forme de piles TIFF 16 bits) qui détiennent l’intensité dans les canaux. Le faire en appuyant sur le bouton < charge Film > et en sélectionnant les fichiers dans les caméras dites de « maîtres » et « esclave » un après l’autre.
- Identifier les positions des molécules simples possibles en recherchant les endroits les plus brillants dans la somme des cinq premiers cadres dans un certain canal de détection. Calculer les positions correspondantes à tous les autres canaux de détection de la cartographie de la chaîne. Pour obtenir la trace d’intensité de fluorescence, somme l’intensité d’un carré de pixel autour de la position centrale de chaque image. Le faire en appuyant sur le bouton < trouver Traces > dans l’interface GUI.
Remarque : La longueur du côté du carré (en pixel) est donnée par : 2 * < Pxs somme > + 1. - Pour chaque molécule, calculer une trace commune d’intensité brute comme la somme de toutes les traces de cet endroit avec la même couleur d’excitation. Évaluer le profil d’intensité de la molécule dans tous les canaux pour les critères suivants :
- Un plateau à peu près plat à l’intensité brute mixte et un blanchiment simple étape pour toutes les couleurs de l’excitation, anthropogéniques comportement dans les canaux appropriés de détection, détection de fluorescence rouge (indiquant la frette pour une AMP lié-PNP *) au moins une fois dans la trace et pas plusieurs étapes dans la fluorescence rouge, ce qui laisse supposer la présence de deux AMP-PNP * lié à un dimère de Hsp90.
- Enregistrer les traces de la fluorescence de la place pour une analyse ultérieure si ces critères sont remplis. Cela en sélectionnant la trace avec le curseur et en appuyant sur le bouton < enregistrer > dans le graphique « échéancier ». Inspecter manuellement les traces d’intensité sur les trois canaux de détection après excitation bleue pour environ 200 molécules par film.
4. calcul des Traces Fluorescence partielle
- Afficher toutes les traces d’intensité de fluorescence pour l’une des molécules enregistrées. Utilisez les curseurs dans le graphique pour sélectionner des plages horaires.
- Sélectionnez un intervalle de temps où les fluorochromes sont déjà blanchies. L’intensité moyenne de fond est calculée à partir de cette gamme et soustrait de la trace de l’intensité de chaque canal. Cela en appuyant sur le bouton < arrière-plan >.
- Sélectionnez la plage de rendement frette, où au moins deux les colorants attachés à Hsp90 (Atto488 et Atto550) sont présents. N’oubliez pas d’exclure les traces qui contiennent un événement clignotant (voir la Figure 3 b). Ces événements sont caractérisés par une intensité de fluorescence encastrée dans un seul canal sans accompagnement augmentation dans n’importe quel autre canal.
- Calculer les traces PF . Cela en appuyant sur le bouton < PF Calc >. Les facteurs de correction prédéfinie pour fuite apparente (lk) et gamma apparent (ag) sont appliquées à l’intensité brute afin de corriger photo-physiques et les propriétés de configuration.
5. population sélection et ajustement de l’histogramme 3D
- Enlever les molécules qui montrent un faible rapport signal-bruit dans les traces PF . Les molécules qui dépassent l’intervalle [-1 ; 2] dans n’importe quelle trace PF pour plus de 10 % des cadres sont supprimés de l’ensemble de données. Cela en exécutant « RemoveTracesLowSNR() » dans la fenêtre commande.
- Calculer des projections 2D des données PF . Terrain
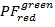 sur
sur 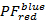 et
et 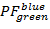 sur
sur 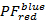 dans l’intervalle [-0.5 ; 1,5] avec une résolution de 100 x 100 emplacements. Pour ce faire, exécutez :
dans l’intervalle [-0.5 ; 1,5] avec une résolution de 100 x 100 emplacements. Pour ce faire, exécutez :- « HistFret2D ("r_b", "sofiene", binHist = 100) »
- « HistFret2D ("r_b", "g_b", binHist = 100) ; MoveWindow 553,5, 42,5, 1055.25, 508,25"
- Déterminer la population relative de chaque État distingue dans les projections en 2D.
- Le graphique approprié de porter à l’avant et exécuter « panelHist2DCount() ».
- Appuyez sur le bouton < Init > et dessiner un polygone à main levée autour du pic.
- Cliquez sur le bouton < nombre >. Le nombre de points de données dans le polygone et le nombre total de points de données dans la projection sont imprimés dans la fenêtre commande.
- Préparer un histogramme 3D des données PF en exécutant « HistFret3D ("g_b", "r_b", "sofiene") ».
- Normaliser l’histogramme 3D à une intégrale de 1. Exécutez ce qui suit :
- « NewDataFolder/S fit0 »
- « Duplicata/O :: frette : Hist3D, Hist3D »
- « Div variable/g = sum(Hist3D)*(DimDelta(Hist3D,0)) ^ 3 »
- « Hist3D / = div ; Impression div »
- Contient les paramètres initiaux de la gaussienne 3D s’adapter et préparent les structures de données nécessaires.
- Exécutez « Gauss3D_initParam() ; modifier W_coef_old. »
- Ajouter les populations de l’État à la fin du vecteur paramètre.
- Exécuter « Gauss3D_prepareFit(). »
Remarque : W_coef_old est un vecteur qui contient les paramètres initiaux pour l’ajustement. Cela signifie que chaque état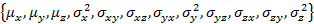 et la population de l’État, qui est concaténée à la fin du vecteur. Assurez-vous que la matrice de covariance est symétrique.
et la population de l’État, qui est concaténée à la fin du vecteur. Assurez-vous que la matrice de covariance est symétrique.
- La somme des fonctions gaussiennes 3D de S à l’histogramme 3D de PF , équipés de S étant le nombre d’États distinguables.
- Exécuter « do3D(). » Cela peut prendre une heure ou plus sur un PC, de bureau normal selon la qualité des paramètres initiaux.
- Exécutez « postprocessFitMultiGauss3D() ; evalFitMultiGauss3D() ; modifier W_coef. »
- Afficher le bon résultat. Pour chacun des deux projections 2D, utilisez les commandes suivantes :
- « contourPF3D_new(0) ; contourPF3D_new(1) ; contourPF3D_new(2) ; contourPF3D_new(3) ; contourPF3D_new(4) »
- « contourPF3D_colorize() »
6. analyse cinétique avec Ensemble 3D HMM
- Préparer un ensemble HMM exécuter pour extraire les informations cinétiques. Un HMM est optimisé de toutes les molécules dans le jeu de données. Utiliser les informations obtenues à l’étape précédente pour définir la position et la largeur de chaque État dans l’espace 3D de PF .
- Initialiser l’interface utilisateur HMM (< HMM | Init HMM >) et choisissez le nombre approprié d’États (dans le cas des données de Hsp90, cela signifie < NumStates > = 5), le nombre de dimensions du signal d’entrée (< NumDims > = 3) et le type de l’entrée (< Input Type > = « Frette bgr 3D »).
- Optimiser les paramètres de la HMM en laissant le logiciel convergent la probabilité de la HMM (exécuter « prepENS_CONVERGE_gB(GetDataFolder(1), -14) ») jusqu'à ce que le changement de la matrice de transition par rapport à l’itération précédente tombe au-dessous d’un seuil (10-14 pour la somme de la variation absolue pour chaque probabilité de transition). Cela donne l’EMV pour les probabilités de transition en environ une heure sur un PC de bureau normal.
- Répétez la sélection de la population, raccord gaussien et l’optimisation HMM pour des sous-ensembles des données (par exemple, 75 % de l’ensemble de données complet). Si l’ensemble de données complet a été fusionné à partir de différentes expériences, répétez l’optimisation également pour chacune des expériences unique. Analyse des sous-ensembles permet d’estimer l’incertitude de la sélection manuelle de la population et de la variabilité au sein de l’ensemble de données.
- Calculer le CI pour les probabilités de transition, qui compte sur l’hétérogénéité de l’ensemble des données et la précision de la HMM.
- Obtenir une estimation approximative des limites CI en exécutant « cd $(racine : path3Dimport + "HMM") ; loop_getCI_estimate_limits(). »
- Calculer les limites exactes de la CI en exécutant :
- « loop_getCI_HMM_converge(1) »
- « CIresults_conv_new() »
- « cd :: HMM_CIresult ; reportCI_conv() »
- « cd :: cmp_CI_conv ; CI_plot2 ("HMM", doAppend = 0) »
- Condenser les informations cinétiques disponibles pour simplifier l’interprétation.
- Collecter des informations sur le temps, un nucléotide marqué reste obligé de Hsp90. Pour ce faire s’effondrer les Etats qui sont liés à l’AMP-PNP * (c.-à-d., S1, S2 et S3, voir aussi la Figure 2) et compilez l’histogramme de temps de s’attarder. Exécuter « cd $(racine : path3Dimport + "HMM") ; collapse_states_get_DT({0,1,1,1,0}) «, qui combine la demeure des États S0 et S4 ainsi que S1, S2et S3.
- Extrait le chemin d’accès de Viterbi pour chaque État de l’intérêt des temps de pause et de comparer l’histogramme résultant du temps s’attarder pour différentes conditions expérimentales. Exécuter « plot_collapsed_DT_Hist (wDTo_01110_record1). »
- Pour un aperçu plus détaillé, s’effondrer les États qui ont distincts PF mais sont fonctionnellement identiques afin de faciliter l’analyse des données supplémentaire, par exemple, dans le cas d’une expérience de trois couleurs avec marqué Hsp90 et nucléotides, États S2 et S 3 peut être réduit. Exécuter « collapse_states_get_DT({0,1,2,2,4}). »
Résultats
Multi-color smFRET mesures permettent la détection directe de la corrélation entre deux ou plusieurs sites distincts d’interaction. Cela rend la technique unique pour étudier les systèmes à composants multiples, tels que des complexes protéiques. Nous nous concentrons sur la présentation d’une expérience de trois couleurs smFRET ici, qui sert d’illustration.
Le déroulement général de la méthode est montré dans...
Discussion
Nous présentons la procédure expérimentale pour obtenir des données de trois couleurs smFRET pour un système complexe des protéines ainsi qu’une description étape par étape de l’analyse de ces mesures. Cette approche offre la possibilité unique pour évaluer directement la corrélation entre plusieurs sites d’interaction ou de changements de conformation.
Pour obtenir des données de molécules simples multicolores adaptées sur les protéines, il est important d’effectuer des...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêt.
Remerciements
Ce travail est financé par la Fondation de recherche allemande (INST 39/969-1) et le Conseil européen de la recherche par le biais de la Convention de subvention ERC n. 681891.
matériels
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
Références
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon