Method Article
3'-Termini הקובע ורצפים של מולקולות Nascent חד גדילי ה-DNA הנגיפי במהלך HIV-1 הפוך שעתוק בתאים נגועים
In This Article
Summary
כאן אנו מציגים בגישה רצף עמוק המספקת של קביעה לא משוחדת של nascent-3' טרמיני, כמו גם פרופילים mutational של מולקולות דנ א חד גדילי. היישום העיקרי הוא אפיון nascent retroviral משלימים DNAs (cDNAs), intermediates שנוצר במהלך התהליך של retroviral שעתוק במהופך.
Abstract
ניטור של חומצת גרעין intermediates במהלך שכפול הוירוס מספק תובנות לגבי ההשפעות של מנגנוני הפעולה של תרכובות אנטי ויראליים וחלבונים התא המארח לסינתזת DNA נגיפי. כאן אנחנו כתובת חוסר assay מבוססת תא, גבוהה-סיקור ו ברזולוציה גבוהה המסוגלת הגדרת intermediates retroviral שעתוק במהופך בתוך ההקשר פיזיולוגיים להידבקות בווירוס. השיטה המתוארת לוכדת את 3'-טרמיני (termini) של מולקולות DNA (cDNA) משלימים המתהווה בתוך תאים נגועים ב- HIV-1 ברזולוציה נוקלאוטיד יחיד. הפרוטוקול כרוך קציר של התא כולו ה-DNA, העשרת יישוב של ה-DNA נגיפי באמצעות לכידת היברידית, מתאם מצדו, גודל fractionation על ידי ג'ל לטיהור, PCR הגברה, רצף עמוק, ניתוח נתונים. צעד המפתח הוא מצדו רציונלית ובלתי יעיל של מולקולות מתאם לפתוח 3'-DNA טרמיני (termini). היישום של השיטה המתוארת קובע את השפע של הפרוטוקולים הפוכה של כל אורך מסוים במדגם נתון. הוא גם מספק מידע על וריאציית רצף (פנימי) תעתיקים הפוכה, ובכך יש מוטציות פוטנציאליים. באופן כללי, וזמינותו מתאים שאלות הנוגעות דנ א 3'-סיומת, ובלבד הרצף תבנית ידועה.
Introduction
על מנת לנתח ולהבין שכפול ויראלי לחלוטין, יותר ויותר מעודן טכניקות ללכוד שכפול intermediates נדרשים. בפרט, ההגדרה המדויקת של מינים חומצת גרעין ויראלי בתוך ההקשר של תאים נגועים יכולה לספק תובנות חדשות, שכן מנגנוני שכפול ויראלי רבים יש לתאריך כבר בדק בתגובות מבודד במבחנה . דוגמה הוא התהליך של שעתוק במהופך בתוך רטרווירוסים, כגון וירוס הכשל החיסוני האנושי (HIV-1) 1. השלבים השונים של שעתוק במהופך HIV-1, במהלכו רוורס טרנסקריפטאז אנזים ויראלי (RT) מעתיק הגנום RNA חד-גדילי לתוך שני גדילי ה-DNA, היו למד בעיקר בתחום מבחני הארכת תחל עם חלבונים מטוהרים ולא nucleic חומצות1,2,3,4,5. בעוד עקרונות היסוד הוקמו, מבחני כזה לא לשלב את כל הרכיבים ויראלי וסלולריות, אינן משקפות בהכרח רלוונטי מבחינה ביולוגית stoichiometries של גורמים המעורבים. לכן, עיצבנו טכניקה חזקה כדי לקבוע את הספקטרום של שעתוק במהופך intermediates עם cDNA מדויק 3'-termini (קרי, קביעת אורך המדויק שלהם) נוקלאוטיד רצפים בהקשר של זיהומים של החיים שלהם תאים6. אוסף של נתונים בזמן הקורס ניסויים יכול להיות מנוצל כדי להשוות את הפרופיל של הפרוטוקולים תחת תנאים שונים, כגון הנוכחות של מולקולות אנטי ויראליים או חלבונים, עשוי להשפיע את היעילות ואת processivity לסינתזת DNA ו הצטברות. דבר זה מאפשר הבנה מפורטת יותר של מחזור החיים של פתוגן טבעי, אשר לעיתים קרובות בסיס עיצוב שיגור תרופות, התערבות טיפולית מוצלחת.
שעתוק במהופך HIV-1 כוללת סדרה של אירועים רצופים שיזם חישול של פריימר tRNA לתבנית RNA הגנומי, אשר לאחר מכן מורחב על ידי RT לייצר תעתיק קצר cDNA חד גדילי שנקרא חיסור-strand חזק-עצירה (-sss) (ראה איור 1). לאחר מכן, cDNA - בועז מועבר מטרמינל 5' באורך חוזר (משמאל לימין) 3'-LTR של RNA הגנומי, איפה זה anneals ואני מגישה למתחילים המשך RT המתווך התארכות של המינוס לנטישה דנ א (ראה את הדעת על שעתוק במהופך1 , 2 , 3 , 4). זו העברה סטרנד הראשון הוא אחד הצעדים הגבלת קצב של שעתוק במהופך; לפיכך, cDNA - בועז ידוע לצבור. העיצוב הבסיסי של זרימת וספריית כדי ללכוד את המוצרים שעתוק במהופך תאים נגועים המותווה איור2 א. תחל ספציפי וניתוח הגדרות אשר נמצאים בשימוש בפרוטוקול ולרשום טבלה 1 . המטרה כל מוקדם הפוך שעתוק intermediates cDNA בטווח באורך של 23 ל 650 ~ nt, הכולל את 180-182 nt - בועז הדנ א. עם זאת, עיבודים קלים המתאים את האסטרטגיה יאפשר ליישום לא רק מוצרים שעתוק במהופך מאוחר אבל גם וירוסים אחרים ומערכות, שבו המטרה היא לזהות את הקצוות DNA המכיל 3'-הו. מגבלות חשוב לשקול לכלול את טווח אורך של המוצר הסופי PCR הספריה; בפרט, תבניות שבו המרחק בין את המתאם-פתוח 3'-הסופית באתר upstream פריימר לחרוג nt ~ 1000 סביר היה פחות ביעילות לסדרם, פוטנציאל היכרות עם מטעה הטיות טכני במהלך ספריית הכנה ( ראה דיון לקבלת פרטים נוספים והצעות הסתגלות).
בעבר דווח על טכניקות מצפני שיטתי 3'-טרמיני (termini) של חומצת גרעין קווצות התמקדו RNA DNA, מולקולות. דוגמה אחת היא 3' המירוץ (מהירה הגברה קצות cDNA)7, אשר נשענת על פוליאדנילציה של mRNA. בנוסף, מתאם מבוססי מצדו אסטרטגיות העסקת RNA ligases פותחו, אשר כללו RLM-מרוץ (RNA בתיווך ליגאז גזע)8 או תחרה (הגברה מבוססת מצדו של הקצוות cDNA)9. חשוב להדגיש כי amplifications מבוססי מצדו רגישים לכל הטיה שהציג את התגובה מצדו עצמו. לדוגמה, מצדו עשוי להיות יעיל יותר או פחות בהתאם נוקלאוטיד מסוים המיקום 3', רצף, מולקולה סה כ אורך, או מבנה מקומי. העדפות ליגאז כזה להוביל לכידת לא שלם של מולקולות, סילוף של המדידה, אשר אנחנו ואחרים הבחינו9,10. כדי לצמצם הטיה מצדו במהלך השלבים בנוסף מתאם בפרוטוקול כפי שיתואר בהמשך, אנחנו מספר אסטרטגיות מצדו נבדק ונמצא את השימוש T4 DNA ליגאז עם מתאם דנ א חד גדילי סיכת ראש (כפי שתואר על ידי קווק et al. 11) להיות ההליך היחיד עם יד מצדו כמותית זה לא לגרום הבדלים משמעותיים מצדו יעילות כאשר העריך עם קבוצת הבקרה oligonucleotides6שנבחרו במיוחד. הבחירה של אסטרטגיה זו מצדו הוא, לכן, תכונה מפתח להצלחת פרוטוקול זה.
עד כה, ניטור של התקדמות RT HIV-1 תאים נגועים בעיקר, הושג על ידי מדידת מוצרים שעתוק במהופך של אורך שונים עם ה-PCR כמותי (qPCR) באמצעות ערכות פריימר-בדיקה המודדים באופן ייחודי ארוכה או קצרה יותר (בראשית ו מאוחר, בהתאמה) cDNA מוצרים12,13,14. בעוד גישה qPCR זו הוא המתאים לקביעת היעילות מהותי מתהליך שעתוק במהופך בתוך מערכות סלולריות, הפלט הוא של רזולוציה נמוכה יחסית, ללא מידע רצף להיות נגזר. הגישה החדשה שלנו, בהתבסס על מתאם ממוטבת מצדו, ספריית בתיווך PCR הדור, רצף עמוק כתובות הפער טכנולוגיה, מציע הזדמנות לעקוב אחר שעתוק במהופך במהלך זיהום ב- HIV-1 באופן כמותי, נוקלאוטיד יחיד רזולוציה.
לנו יש מאויר השירות של שיטה זו במחקר זה להבחין בין שני מודלים מוצעים עבור הקיבולת של הגורם הגבלת HIV-1 APOBEC3G (אפוליפופרוטאין B mRNA העריכה אנזים קטליטי מפוליפפטיד כמו דור 3) להתערב עם הפקה של נגיפי תעתיקים הפוכה6.
Protocol
הערה: נא עיין טבלה של חומרים עבור ריאגנטים ספציפיים וציוד המשמשים פרוטוקול זה.
1. וירוס ייצור ולדלקת תא
התראה: זיהומיות HIV-1 צריך רק יטופלו במעבדות הבלימה אבטחה מאושרות.
הערה: הפקה של חלקיקים HIV-1 על ידי תרביות תאים ארעית של האדם מתחלקים כליות (HEK) 293T תאים, כפי שמתואר בספר צעד 1.1, זה נוהל שגרתי, היה שתואר לעיל15,16. תרבית תאים כללי שגרות הן שתוארה לעיל17.
- ייצור וירוס HIV-1.
- לשמור על תאים 293T של Dulbecco ששינה נשר בינוני (DMEM) בתוספת 10% סרום שור עוברית (FBS) ו- 1% פניצילין/סטרפטומיצין (DMEM מלא) בחממה תרבות תא סטנדרטי-37 מעלות צלזיוס ו-5% CO2 כפי שתואר לעיל17.
- ב תא סטנדרטי תרביות רקמה למינארי מסיר את המדיה צמיחה ולהוסיף 3 מ"ל של טריפסין ומחוממת מראש (37 מעלות צלזיוס) ליד-confluent 10 ס מ התא לצלחת תרבות (~1.2 x 107 תאים) של תאים 293T. להחזיר את המנה החממה למשך 2-3 דקות.
- קח את הצלחת מן החממה בחזרה לתוך המנוע תרביות רקמה ולהוסיף mL 7 בינוני מלא. פיפטה למעלה ולמטה בתוך קערה מספר פעמים כדי resuspend את התאים. פיצול תאים 1:4 על-ידי הוספת 2.5 מ של התליה תא תבשיל 10 ס מ חדש ולמלא אותו 7.5 mL בינוני מלא.
- למחרת, לערבב µg 10 של פלסמיד HIV-1 proviral דנ א (כגון pNL4.3) עם 1 מ"ל של מדיום חיוני ללא סרום מינימלי ולהוסיף polyethylenimine (פיי) פתרון (25,000 mW, 1 מ"ג/מ"ל pH 7) ב- 4.5 µL לכל 1 µg DNA. תקופת דגירה של 10 דקות ב RT ולהוסיף dropwise תאי 293T.
- 24 שעות לאחר תרביות תאים, להסיר את המדיום ולהחליף אותו עם 6 מ של DMEM מלא המכיל נטולת RNase DNase 20 U/mL בינוניים. לאחר 6 שעות, החלף את המדיום 10 מ"ל של DMEM מלא.
- 48 שעות לאחר תרביות תאים, לקצור את תגובת שיקוע ולסנן אותו דרך מסנן מיקרומטר 0.22, באמצעות מזרק 10 מ"ל, לתוך צינור פוליפרופילן 15 מ"ל.
- להוסיף 2 מ של סוכרוז סטרילי 20% 1 x buffered פוספט תמיסת מלח (PBS) צינור פתוח עם קירות דקים ultracentrifuge. לאט לאט שכבת-על סוכרוז עם supernatant תא מסוננים.
- צנטריפוגה עבור h 1 15 דקות ב g x 134,000 ב 4 ° C באמצעות של ultracentrifuge.
- הסר את החצוצרות שלה ultracentrifuge בזהירות. לאט לאט את תגובת שיקוע והן באמצעות יניקה או פיפטה של סוכרוז. פיפטה קטנים יותר ולהשתמש להטות את הצינור כשהוא מוציא את הפתרון האחרון סוכרוז. להשאיר את הוירוס pelleted בחלק התחתון של הצינור.
הערה: בגדר לא יהיה גלוי. - להוסיף 200 µL ל- 1 x PBS להשאיר במקרר במשך 4 עד 12 h, resuspend, להקפיא במצב aliquots 20 µL ב-80 מעלות צלזיוס.
- לקבוע p24מחסום פה תוכן משתמש אנטיגן HIV-1 p24 אליסה קיט (הוראות היצרן הבא).
- תא T קו זיהום.
- תרבות קו T-cell immortalized (למשל., תאים CEM-SS) במדיום רוזוול פארק אנדרטת המכון (RPMI) 1640 בתוספת 10% FBS ו 1% פניצילין/סטרפטומיצין (RPMI מלא). לספור את התאים באמצעות hemocytometer18 זרע 1 טוב עבור דגימה עם 1 מ"ל של RPMI מלא עם 2 x 106 תאים למ"ל בצלחת התרבות תאים בפורמט 12-. טוב.
- להוסיף חלקיקים HIV-1 צנטריפוגהמחסום פה , המקום הצלחת לתוך מתנדנד שקול ל- 150 ng p24 דלי biocontainment בשלמותם כדי להדביק את-ספין על ידי צנטריפוגה ומפרידה benchtop עבור 2 h ב g 2,000 x-30 ° C.
- הסר לוחית של צנטריפוגה ולתת לו לנוח 1 ש ח בחודש חממה תרביות רקמה סטנדרטי-CO 37 ° C ו- 5%2.
- לשטוף את וירוס קלט, לאסוף תאים על ידי העברת את המתלים תא microcentrifuge צינורות צריך שתוציאו ב microcentrifuge ב RT (RT) ב 500 g x למשך 2 דקות. . תוריד את תגובת שיקוע מבלי להפריע בגדר תא
- Resuspend תא כדורי ב 1 מ"ל של טרום ומחוממת (37 מעלות צלזיוס), עקר 1 x PBS. חזור על צנטריפוגה, הסרת supernatant, צעדים resuspension פעמיים נוספות.
- צנטריפוגה שוב, הסר את תגובת שיקוע ולאחר resuspend כדורי תא ב 1 מ"ל של RPMI מלא. להוסיף כל השעיה באר אחת בצלחת טוב 12 חדשים.
- ב- 6-אייץ ' תוספת שלאחר הראשונית של וירוס (צנטריפוגה שלאחר 4 שעות), לקצור את התאים על ידי צנטריפוגה כפי שנעשה צעד 1.2.4. להסיר ולמחוק את תגובת שיקוע. כדורי תא יכול להיות קפוא ב-80 מעלות צלזיוס או מעובד ישירות להפקת DNA.
2. DNA החילוץ, HIV-1 DNA כימות, העשרה על ידי לכידת היברידית
- תמצית DNA התא כולו עם דם ורקמות הכולל DNA החילוץ ערכת על-ידי ביצוע במדריך קיט של תרביות רקמה תאים. הדבר היחיד שהשתנה זה • תנאי ב 200 µL של נוקלאז ללא H2O במקום המאגר שסופק • תנאי.
הערה: לאחר תוספת של מאגר פירוק chaotropic ("AL מאגר של ערכת"), proteinase, יכול להיות הסרת במעבדה הבלימה אבטחה ודוגמאות טופל במעבדה רמת הבטיחות הסטנדרטי למשך שארית בפרוטוקול. - לקבוע את עותק מספר cDNA HIV-1 על ידי qPCR.
- לקחת µL 17 של eluate מ שלב 2.1 ולהוסיף µL 2 x 10 אנזים הגבלה מאגר יחד עם µL 1 של אנזים הגבלה DpnI. תקופת דגירה של h 1 ב 37 ° C כדי להסיר כל פוטנציאל פלסמיד קלט שרידי דנ א של תרביות תאים.
- לבצע את qPCR עבור חיסור-strand חזק-stop cDNA שימוש בערכת המכשיר תחל הבאה: oHC64 (יונקות-taactagggaacccactgc-3′), oHC65 (יונקות-gctagagattttccacactg-3′), בדיקה oHC66 (יונקות-FAM-acacaacagacgggcacacacta-טמרה-3′). qPCR התקנה ותנאים המדויקים ניתן למצוא את הפניות6,13. נושאים לאורך דגימות עם דילול טורי פלסמיד proviral pNL4.3 כמו עיקול רגיל כדי לקבוע מספרים עותק של cDNA מולקולות.
הערה: ראה דיון עבור כמויות הצפוי.
- HIV-1 DNA העשרה על ידי לכידת היברידית.
הערה: מזה צעד קדימה, עדיף להשתמש microcentrifuge צינורות עם מאפייני האיגוד חומצת גרעין נמוך, כמו גם תרסיס מסנן פיפטה טיפים עבור כל דגימות די אן איי. במידת האפשר, עובד תחנת עבודה-PCR. כל המדרגות ריאגנטים נמצאים ב RT (RT) אלא אם צויין אחרת.- כדי להכין את תערובת הבסיס של חרוזים streptavidin מגנטי, פיפטה 100 חרוזים µL עבור דגימה לתוך צינור microcentrifuge יחיד. מניחים את הצינורית על מגנט מתאים microcentrifuge צינורות.
- לאחר החרוזים התיישבו לכיוון הצד מגנט של הצינור (~ 1 דקות), להוריד את המאגר אחסון, להסיר את הצינורית של המגנט, resuspend חרוזים ב 500 µL של bind ולשטוף מאגר (מאגר BW, 5 מ מ טריס-HCL pH 7.5, 0.5 מ מ EDTA 1 M NaCl) לשטוף.
- מקם את הצינור בחזרה על מגנט, להסיר את תגובת שיקוע והוסף 500 µL קזאין פתרון. קח של המגנט, resuspend, תקופת דגירה של 10 דקות ב RT ולאחר מכן לשטוף עם מאגר BW.
הערה: שטיפה מתייחס הנחת הצינור על המגנט, מורידה את תגובת שיקוע, לוקח את הצינור את המגנט, הוספת המאגר, ו resuspending. - במקום הצינור בחזרה על המגנט, תוריד את תגובת שיקוע, resuspend חרוזים ב 500 µL BW המאגר. הוספת pmol 50 של כל oligonucleotides biotinylated לכידת (ראה טבלה 1, שלוש oligos במקרה זה) לפי הדוגמה. (לדוגמה, אם דגימות די אן איי 5 להיות מעובד, השתמש µL 500 של beads מגנטי של pmol 2.3.1 ו- 250 שלב של כל oligonucleotide).
- תקופת דגירה של 30 דקות ב- RT תוך נדנדה במיקסר מעל קצה-.
- רחץ חרוזים עם oligonucleotides קיבוע פעמיים עם µL 500 x 1 10 מאגר (10 מ מ טריס HCl pH 8.0, 1 מ"מ EDTA, 100 מ מ NaCl).
- Resuspend חרוזים ב µL 10 x 1 10 מאגר עבור דגימה.
- עבור כל דגימה תווית שפופרת microcentrifuge אחת ולהוסיף 10 µL של חרוזים השעיה, 170 µL של ה-DNA (מתוך שלב 2.1), µL 90 של 3 x 10 מאגר. דגירה בתוך גוש חום יבש ב 92 מעלות צלזיוס למשך 2 דקות denature את ה-DNA.
- להזיז את הצינורות בלוק חום יבש שונים, אשר מוגדר כ- 52 ° C, וכן תקופת דגירה של 1 ח' ' היפוך ' כדי לערבב באופן קבוע (~ כל 10 דקות) במהלך הדגירה הזה.
- לשטוף פעם עם µL 500 1 x מאגר עשר ו resuspend ב 35 µL ללא נוקלאז H2O.
- כדי elute, דגירה הצינורות ב 92 מעלות צלזיוס בתוך גוש חום יבש למשך 2 דקות. לאחר מכן, להעביר במהירות את הצינורות על המגנט (שפופרת אחת בכל פעם). ברגע החרוזים קשורים לצד של הצינור, להעביר את תגובת שיקוע המכיל את ה-HIV-1 DNA לרכבת התחתית טריים.
- אופציונלי: חזור על qPCR (כפי שנעשה צעד 2.2.2) כדי לקבוע את cDNA HIV-1 התאושש.
הערה: ראה דיון עבור כמויות הצפוי.
3. מתאם מצדו
- הכנת את מתאם
- Resuspend מתאם lyophilized (ראה טבלה 1 "מלא קווק + MiSeq") ב- 100 מיקרומטר ב H נטולת נוקלאז2O.
- לכל דגימה פלוס פקד אחד לדוגמה, לשלב 0.45 µL של 10 x T4 DNA ליגאז מאגר µL 4 של מתאם, µL 0.05 של נוקלאז ללא H2O. חום עד 92 º C למשך 2 דקות ולתת כל להתקרר לאט.
הערה: אם האפשרות זו זמינה להשתמש מכשיר PCR עם קצב הקירור מתכווננת (שימוש 2% שיעור). זה לוקח בערך 30 דקות מ 92 º C ל-16 ° C. לחלופין, השתמש גוש חום יבש ב 92 מעלות צלזיוס, לכבות. מוציא את mastermix מתאם הבלוק החום היא חזרה ב RT. זה נועד להראות את מתאם יוצרים מבנה סיכת ראש (ראה איור 2b).
- להכין תגובה שליטה עם קבוצת מסונתז oligonucleotides (ראה טבלה 2) במקום דנ א שחולצו מן התאים.
- להפוך את המניות של 100 מיקרומטר של כל oligonucleotide. לערבב 1 µL של כל אחד oligonucelotides 17, להוסיף µL 8 H2O עבור יחס equimolar נפח סופי של 25 µL.
- לדלל את 1:2,500 מיקס ב H נטולת נוקלאז2O לדילול טורי. לשלב 1 µL של השילוב עם 17.3 µL של נוקלאז ללא H2O כדי להשתמש ב- מצדו דגימת הבקרה בשלב 3.3.1 כך כל oligonucleotide הוא נוכח 1.6 fmol (שקול ל- 0.026 nM, התגובה 60 µL).
- הגדרת ligations
- עבור 60 µL נפח סופי בתגובות המבחנות לשלב 6 µL של 10 x T4 DNA ליגאז מאגר, 24 µL של 40% פג, µL 6 של 5 מ' betaine, µL 4.5 (400 pmol) של מתאם (pre-annelead כמו שלב 3.1.2), µL 1.2 של T4 DNA ליגאז (יחידות 2,000,000/mL) ו- 18.3 µL של ה-DNA (מ שלב 2.3.11)
הערה: לקחת טיפול מיוחד עם פתרונות צמיגה כמו 40% יתד כדי לשמור על אמצעי אחסון מדויק. אל תבצע את mastermix. - להגדיר את אותה התגובה כפי שבוצע בשלב 3.3.1 אבל עם השילוב oligonucleotide שליטה מוכן בשלב 3.2.2.
- מערבבים היטב את התגובות, דגירה במכונת ה-PCR ב 16 מעלות צלזיוס למשך הלילה.
- עבור 60 µL נפח סופי בתגובות המבחנות לשלב 6 µL של 10 x T4 DNA ליגאז מאגר, 24 µL של 40% פג, µL 6 של 5 מ' betaine, µL 4.5 (400 pmol) של מתאם (pre-annelead כמו שלב 3.1.2), µL 1.2 של T4 DNA ליגאז (יחידות 2,000,000/mL) ו- 18.3 µL של ה-DNA (מ שלב 2.3.11)
4. מתאם להסרת והפרדה גודל
- Denaturing בג'ל
- להוסיף 30 µL של DNA המכיל formamide ג'ל טעינת מאגר על כל תגובה מצדו. מערבבים היטב בעזרת pipetting.
- חום למשך 2 דקות ב 94 ° C ב- PCR מכונת ולאחר מכן מיד לשים על קרח.
- הצב precast 6% טריס/בוראט/EDTA (TBE) denaturing אוריאה לזיהוי ג'ל (10-ובכן המסרק), במיכל ג'ל המתאים. להוסיף 1 x TBE (89 מ"מ טריס-בסיס, 89 מ מ פנמיות, 2 מ מ EDTA) פועל מאגר ולהפעיל מראש את הג'ל במשך 20 דקות-קבוע V/מקס 250.
- לשטוף את הכיסים ג'ל עם הפעלת מאגר באמצעות מזרק ואת המחט 21G.
- עומס כל מדגם 90 µL משלוש בארות (30 µL לכל טוב), המנוהל במשך 20 דקות (250 V max) עד לחזית צבען כחול נמצא בחצי הדרך דרך הג'ל.
- צביעת ו חומצות גרעין חיתוך של הג'ל
- הכן microcentrifuge קטן 3 צינורות (0.5 מ"ל) עבור דגימה באמצעות פוערים חורים לתוך החלק התחתון באמצעות מחט מזרק 21 G (קח זהירות בעת עבודה עם שרפ). הכנס כל אחד מן הצינורות מוכן לתוך צינור microcentrifuge 2.0 mL, התווית אותם עם שם דגימה פלוס "נמוך", "אמצע" או "גבוהה".
- להוציא ופתח לחטט בקלטת ג'ל. חותכים את הג'ל באופן אנכי עם סכין גילוח לסלק בנדיבות את הרצועה עם הבארות 3 דוגמאות טעון. להוסיף רצועת ג'ל מיכל עם 1-TBE (כ- 30 מ"ל) ו- 5 µL של cyanine חומצת גרעין כתם. תקופת דגירה של 3-5 דקות.
הערה: השלב החילוץ ג'ל הוא רגישות גבוהה במיוחד זיהום צולב. מומלץ לפעול רק דוגמה 1 לכל ג'ל באמצעות מיכל נפרדת, נקי עבור כל ג'ל מכתים. כפפות צריכה להשתנות אם חלקיקי הג'ל קשר עם האצבעות בכפפה - לנקות את השטח של transilluminator אור כחול ביסודיות עם ddH2O. . קח את פיסת ג'ל מתוך המכולה מכתימים והוסף אותו אל תיבת אור.
- להפעיל את תיבת אור ולבדוק חומצות גרעין מוכתמים דרך המסנן כתום.
הערה: המתאם בדרך כלל מופיע עמוס ופועל כמו גדול "בועה" עם ה-DNA HIV-1 מחוברים פועל מעל פס. - שימוש חדש סכין גילוח, לחתוך משם את הצדדים של הג'ל אם ישנם אזורים עם דגימת אין טעון עדיין נוכחים. בשלב הבא, לחתוך בדיוק מעל מתאם כדי להסיר את המתאם נמוך ג'ל חלקים. לבסוף, לחתוך ראש הג'ל כולל כ- 1 מ מ של הג'ל כיסים, אשר לעיתים קרובות יש אות עז חדה של משקל מולקולרי גבוה יותר דנ א.
- לחלק את הפיסה האחרונה ג'ל המכיל את הדגימה, שהוא בדרך כלל ~ 2 x 3 ס"מ בגודל, בצורה אופקית לתוך שלוש חתיכות אפילו:"נמוך","אמצע"ואזורי משקל מולקולרי 'גבוהה'.
הערה: כל חתיכה עכשיו יטופלו בנפרד [כלומר., יהיו שלושה צינורות (נמוך, אמצע, וגבוה)] לדגימה המקורי. - לחתוך כל אחד השברים ג'ל שלושה לחתיכות קטנות יותר (~ 2 x 2 מ מ חלקיקים), להעביר אותם לתוך הצינורות microcentrifuge יהיה 0.5 mL (שלב 4.2.1).
- ספין בשיא המהירות בשלמותם פתוח עבור 1 דקות לסחוט את החלקים ג'ל דרך החור לתוך הצינור 2 mL כדי ליצור את הרפש ג'ל. אם כל חלקיקי הג'ל נשארים בתחתית של הצינור 0.5 mL, להעביר אותם הצינור 2 מ"ל באופן ידני באמצעות טיפ מחט או פיפטה.
- הפקת דנ א
- להוסיף 1 מ"ל של שתנן ג'ל החילוץ מאגר (0.5 M NH4CH3CO2, 1 מ מ EDTA, 0.2% מרחביות) הרפש ג'ל. לסובב את הצינורות לקבוצות של 3 שעות (בן לילה הוא מקובל) ב RT במערבל מעל קצה-.
- השתמש קבוצה נקייה של פינצטה כדי להוסיף מסנן סיבי זכוכית עגול קטן אחד לעמודות צנטריפוגה עם מסננים ממברנה אצטט תאית (0.2 µm) אשר מונע סתימת ממברנה. לשים את המסנן במקום עם קצה פיפטה הפוכה.
- בקצרה לסובב את צינורות 2 מ"ל עם ג'ל מאגר הרפש, חילוץ microcentrifuge, להעביר µL 700 של תגובת שיקוע העמודות מסנן מוכן. שמור את הרפש ג'ל ואת תגובת שיקוע הנותרים.
- Centrifuge הטורים מסנן microcentrifuge בשיא המהירות עבור 1 הגבלת העברת flowthrough לתוך צינור microcentrifuge החדש של 2.0 mL.
- לטעון מחדש את העמודות עם תגובת שיקוע הנותרים. נסו להשיג כמה שיותר נוזלים ככל האפשר של החילוץ הרפש. העברה של ג'ל חתיכות היא לא דאגה. ספין שוב ומשלבים flowthroughs של הדגימות החילוץ אותו.
- משקעים דנ א
- להוסיף 3 µL של תיקים עשויים RNA (µg 1/µL; כנשא), µL 1 של גליקוגן, 0.7 מ ל אלכוהול איזופרופיל כדי flowthrough מ שלב 4.3.5. מערבולת בקצרה, הקפאת ב-80 מעלות צלזיוס למשך הלילה.
- לקחת דגימות מהמקפיא-80 ° C ולתת להם להפשיר בקצרה. . שים אותם microcentrifuge מקורר (4 ° C) ו ספין למשך 30 דקות בשיא המהירות.
- להסיר ולמחוק את תגובת שיקוע. להיות זהיר מאוד לא להסיר את גלולה. השאירו 30-50 µL של נוזל אם זה לא בטוח גלולה זו תוסר אחרת.
הערה: בדרך כלל כל דוגמאות 'גבוהה' הצג גלולה גלוי יותר מאשר דגימות "אמצע", "נמוך". - הוסף µL 800 של 80% אתנול. היפוך צינורות, ספין שוב בשביל 1 דקות בשיא המהירות. להסיר את הרוב המכריע של אתנול עם פיפטה בקצרה לסובב את הצינורות שוב, להסיר אתנול נוספים עם פיפטה קטנים יותר נפח.
- תן לכל אתנול הנותרים להתאדות על ידי הנחת הצינורות עם מכסה פתוח לתוך גוש חום יבש 55 ° צלזיוס. מתי הם דוגמאות יבש (2-4 דקות) להוסיף 20 µL של נוקלאז ללא H2O, התפשטה סביב החלק התחתון של צינור כדי להבטיח בגדר DNA התפרקה. לדנ שניתן לאחסן ב-20 ° C.
5. PCR הגברה והכנת ספריית
- להגדיר את תגובת ה-PCR 40 µL עם 20 µL של DNA פולימראז מיקס מראש, µL 18 של זירז, redissolved ה-DNA מ שלב 4.4.5, µL 1 של פריימר לפנים "MP1.0 + 22HIV" (10µM) (ראה טבלה 1), ו- 1 µL של מולטיפלקס oligo תחל (אינדקס תחל 1 עד 24) (ראה טבלה של אמא terials).
הערה: הפעל התגובות שלוש (נמוך, בינוני, גבוה) של כל מדגם תגובות PCR נפרד, אלא עם אותם באינדקס פריימר. השתמש בעל אינדקס שונה עבור כל אחת הדגימות הזיהום המקורי.- הפעל את תגובות PCR בתנאים הבאים: 2 דקות-דנטורציה 94 ° C, אז 18 מחזורים של 3-צעד PCR; 15 s-דנטורציה 94 ° C, s 15 חישול 55 ° C, ואת הסיומת s 30-68 מעלות צלזיוס.
- כאפשרות בקרת איכות, ניתוח תגובות PCR עם רגישות גבוהה אוטומטית ג'ל אלקטרופורזה מערכת. לקחת 2 µL של מדגם נמוך, בינוני וגבוה לפעול לפי הוראות היצרן.
הערה: שני צבעי יסוד צריך להיות גלוי ומנוהל לעיתים קרובות על אורך מחושב על nt 45 ו 95 (אורך בפועל שונה). בנוסף, ה-DNA שיזוהו בין 150-500 nt. אם אין קליטה, רצוי להוסיף מחזורי PCR נוספים, בין 2 ל- 10 מחזורים נוספים. אל תוסיף מחזורים נוספים עבור דגימות הבקרה oligonucleotide שנוצרו בשלב 3.3.2. - כדי להסיר תחל השתמש פאראמגנטיים חרוז PCR לנקות מערכת מבוססת.
- קח 20 µL של כל תגובה PCR, מאגר הדגימות ביחד (לערבב כל הדגימות בשלב זה). להקפיא את התגובות µL 20 הנותרים כמו גיבויים ב-20 ° C.
- תן את החרוזים פאראמגנטיים לבוא RT ומערבבים את תגובות PCR במאגר עם 1.8 x הנפח של הפתרון חרוז. לערבב על-ידי pipetting, תקופת דגירה של 5 דקות.
הערה: כמו דוגמה, אם 4 דגימות הוכנו וכל אחד יש תגובות נמוך, בינוני וגבוה, אמצעי האחסון יהיה 4 x 3 x 20 µL = 240 תגובות PCR µL עם פתרון חרוז 432 µL. - הצינורות על מגנט צינור microcentrifuge, תן את החרוזים לאגד ~ 1 דקות, להוריד את תגובת שיקוע כדי למחוק. להשאיר את הצינורות על המגנט ולהוסיף µL 500 של 80% אתנול.
- להשאיר האתנול ב-30 s, אז תוריד ביסודיות. ותני את airdry חרוזים עבור µL להוסיף 40 ~ 5 מינימלית של נוקלאז ללא H2O, קח את הצינורות את המגנט, פיפטה לאורך מספר פעמים.
- להשאיר את ההשעיה עבור 5 דק. לשים את הצינור בחזרה על המגנט, תן את החרוזים להתיישב לצד, ולהעביר את תגובת שיקוע צינור חדש. זוהי הספרייה. . קח את µL 10 aliquot עבור פקדים איכות ומקפיאים את השאר ב-20 ° C.
6. הערכת הספרייה
- לקבוע את איכות ספריה, ריכוז, molarity.
- להשתמש בשיטה fluorometric כמת. למדוד 1 µL ו- 3 µL של הספרייה עם ערכת assay dsDNA רגישות גבוהה לפי הוראות היצרן.
הערה: ריכוזי טיפוסי הם בין 1 ל 10 ng/µL. - מדד הקשת משקל מולקולרי ספריית ה-DNA על ידי רגישות גבוהה אוטומטית בג'ל כמתואר לעיל (שלב 5.2).
- שימוש ג'ל אלקטרופורזה ניתוח כדי לקבוע את המשקל המולקולרי הממוצע של הספרייה ולחשב אוטומטית לדלל בספריה נטול נוקלאז H2O 4 nM. ניתן לשלב מספר ספריות כל עוד כל המדדים הם ייחודיים.
- להשתמש בשיטה fluorometric כמת. למדוד 1 µL ו- 3 µL של הספרייה עם ערכת assay dsDNA רגישות גבוהה לפי הוראות היצרן.
- בקרת איכות תפוקה נמוכה אופציונלי
- נושא את ספריית ה-DNA TA שיבוט19 להכניס ספריה מולקולות וקטורים עבור הגברה. בצע את ההוראות של הערכה, לגדול ~ 10-20 מושבות ופרוטוקולים תמצית הדנ א באמצעות miniprep, כפי שמתואר כאן20.
- וקטור רצף באמצעות שירותי רצף המקומית ולבדוק כי התוספות מכילים את הרצוי של HIV-1 נגזר רצפים ומתאמים הספציפיים של ספריית.
7. תפוקה גבוהה רצף הפעלה
- צור גליון מדגם רצף עם התוכנה המסחרית מסופק עם פלטפורמת רצף.
- ציין את ערכת רצף שנבחרו. בדרך כלל, לבחור ערכת 150-מחזור, אבל אחרים אינם מתאימים בהתאם לאורך קריאה הרצוי.
- בחר "Fastq רק" זרימת העבודה של היישום. בחר אחת מהתבניות המכיל את מדדי 24 נוכח ערכות oligonucleotide מולטיפלקס (המצוין במדריך קיט).
- בחר "25 nt" עבור Read1 ו "125 nt" עבור Read2. שמור 6 nt עבור אינדקס אחד לקרוא.
הערה: במהלך הניתוח ללא צורך במיקור חוץ רק Read2 משמש בניתוח. לשמור Read1 לכל הפחות של 25 nt עבור קביעת רצף פלטפורמה אלגוריתם למטרות.
- בצע את הוראות היצרן במדויק ספריית מבחו והכנה ההתקנה. באופציית ה-20 מרבית הריכוז pM ושימוש יתד PhiX 15%, כמו הספרייה הוא המורכבות נמוך מאוד.
8. ניתוח נתונים
- בדוק אם עוברים סינון באחוזים בין הציון הממוצע של איכות Q30 מקובלים לפי רצף ההנחיות של יצרן הפלטפורמה.
הערה: מסנן לעבור בדרך כלל > 90%, Q30 הציונים בדרך כלל > 80%. - להוריד. fastq.gz קבצים מרכזת רצף של היצרן.
- הגדרת קובץ ה-script רצף
- ליצור ספריה חדשה (תיקיה) בשם "AnalysisXYZ" וללכת https://github.com/malimlab/seqparse כדי להוריד את כל קבצי קוד המקור (parse_sam.pl, rc_extract.pl, parse.sh) לתוך ספריה זו.
- הורד את aligner קצר-קריאה עניבת הפרפר, החל מגרסא 1.1.2, http://bowtie-bio.sourceforge.net/index.shtml לתוך באותה הספריה.
- ההורדה יוצר ספריית משנה בתוך "AnalysisXYZ" בשם "עניבת הפרפר-1.1.2". בתוך ספריה זו תפתח ספריית המשנה "אינדקסים" ולהוריד את רצפי התבנית שסופק בהיקף של 6 קבצים עם הסיומות .ebwt.
- הורד את קריאות FASTQ/A קצר מראש עיבוד toolkit fastx-0.0.13 מ http://hannonlab.cshl.edu/fastx_toolkit/download.html לתוך הספריה "AnalysisXYZ".
- להוריד Samtools (https://sourceforge.net/projects/samtools/files/) והן bam-readcount (https://github.com/genome/bam-readcount) לתוך הספריה "מסמכים".
- להזיז. fastq.gz קבצים, להוריד בשלב 8.2, כל קרא 2s (המסתיימת ב-. _R2_001.fastq.gz) לתוך הספריה "AnalysisXYZ".
- פתח את המסוף הפקודה/מסוף. להעביר "AnalysisXYZ" כמו הספריה הנוכחית באמצעות פקודות cd. הקלד "./parse.sh." כדי להפעיל את קבצי ה-script.
- למצוא את קבצי ה-csv עם סיכומים עבור כל הדגימות באשמות קריאה הכולל, אורך מותאם לקריאה ספירות, מווסת את ספירת קריאה, וכן קבצים עם וריאציה בסיס, עבור כל דגימה בספריה בשם parse_results הספריה "ניתוח XYZ".
הערה: ראה הדיון לקבלת מידע נוסף אודות תהליך ניתוח. קובץ ה-script מחזיר קבצי csv עם קריאות הכולל עבור כל נוקלאוטיד לאורך רצפי חזק-stop HIV-1NL4.3 , העברת סטרנד הראשון עד נוקלאוטיד 635. כמו הדרכה, קריאות ייחודי 50,000 ל 100,000 הם נצפו בדרך כלל דגימות זיהומים עם מספרי הטלפון הנייד המצוין ואת inocula ויראלי וללא חלבונים ויראליים או תרכובות. המדגם שליטה oligonucleotide בדרך כלל מניבה קריאות 100,000 עד 200,000.
תוצאות
בטכניקה המתוארת במאמר זה היה מוחל על מחקר רחב יותר כדי לטפל המנגנונים עיכוב של HIV-1 שעתוק במהופך על ידי החלבון האנושי תרופתי APOBEC3G (A3G)6. איור 3 מראה נציג התוצאות המתקבלות לאחר העסקת פרוטוקול דגימות CEM-טי אס-תאים נגועים vif-HIV-1 לקוי היעדרות או נוכחות של A3G. המספר הכולל של קריאות ייחודי המתקבל כל מדגם לאחר סינון הכפולות PCR יש 6 באותו ברקוד nt ו באותו האורך (המבוצעת על ידי תוכנת ניתוח שסופק) מותוות בתרשימי איור 3. רמות הולכות וגדלות של A3G להפחית את המספר הכולל קריאה המשקף את ההשפעה המעכבת של A3G על סינתזה cDNA RT בתיווך מצרי קודם לכן תיאר, נמדדת qPCR6,13,21,22. איור 3b, השבר של מולקולות על כל אורך אפשרי בתוך 182 הראשון nt מוצגים. זיהום ב- HIV-1 בהיעדרו של A3G, המין הנפוץ ביותר הוא 180 הראשי nt חזקה-. תפסיק המולקולה עצמה, עם כמה הצטברות הקריאות בטווח קצר (23-40 nt) (top היסטוגרמות גרף, כחול). התוספת של A3G שינויים שבפרופיל זה כמו עלייה חדה של קצר יותר, מעוגל cDNA מולקולות ב כמה תפקידים ספציפיים מאוד, לשחזור הוא זיהה (האמצעי והתחתון תרשימים). מאחר A3G הוא cytidine deaminase, ציטוזין-אל-uridine (המזוהה כג-ל-T) מוטציות cDNA להתרחש כאשר A3G קיים המזהם virions21,23,24. באמצעות המידע שהושג רצף, האחוז של מוטציות ג-ל-T היה התווייתו באותו התרשים (קו מקווקו אדום). יצוין כי הפרופיל mutational נגזרת קריאות כולם ייחודיים בשילוב, כיסוי של כל נוקלאוטיד ישתנו. עם זאת, אם נדרש רצף מידע יכול להיות הקשורות בחזרה כל מולקולה ו בקורלציה עם מסוים 3'-terminus. הנתונים שסופקו נלקחו Pollpeter. et al. 6 והמתאם בין mutational ו פרופילי אורך cDNA הדגימו להיות בגלל המחשוף של cDNA deaminated על ידי הטלפון הסלולרי וזיהוי DNA תיקון מכונות.
פקד חיובי עבור הגישה 3'-מיפוי בקלות יכול להיות מיוצר על-ידי עיבוד מאגר של oligonucleotides סינתטי של רצף ידוע, אורך ריכוז. פקד זה להוסיף את מצדו מתאם בשלב 3.3.2, מומלץ להיכלל בספריות מרובבת כל. והנתונים שהתקבלו דגימת הבקרה צריך את כל oligonucleotides-היחס הצפוי קלט, עם קריאות רקע מאוד קלה בלבד. איור 4 מציג תוצאות של קבוצת בקרה חיובית 17 oligonucleotides מסונתז כימית (עבור רצפים, עיין בטבלה 2), אשר היו מעורבים ב יחסי equimolar. כצפוי, וכל המולקולות מופיעים קרוב שפע שווה עם וריאציות קטנות בלבד (הגרף העליון). בעוד רוב עמדות בתוך רצף הדנ א - בועז היו מיוצגים על ידי oligonucleotide להחזיר אפס ספירת קריאה, הבחנו מינים קטנים כי הם קצרים יותר מאשר oligonucleotides שליטה בפועל nt 1 או 2. אנחנו עוד לא חקרו את המינים הללו קטין אבל להניח שהן מייצגות מוצרים מפורק שגויה או שעשויות להיות נוכח המניות oligonucleotide שסופק בעת הרכישה (oligonucleotides הורו כפי HPLC טהור, אשר היצרן מציין > 80% טוהר). הגרף התחתון מציג את המדגם שליטה מספריית שונים להפעיל, איפה וריאציה מעט גבוה יותר בין oligonucleotides 17, עולה בקנה אחד עם אורך כללי בכך יותר שליטה מולקולות מזוהים בצורה יעילה יותר ואז אלה שורטר. זה יכול להיות בגלל דעה קדומה משניות ב- PCR תגובות או בקיבוץ באשכולות במהלך רצף MiSeq, שבו יש אופטימום בגודל של הוספה, עלולה להתרחש עם ספריות נושאת טווחים הוספה רחבה במיוחד. דרך בסיסית כדי לטפל הנטיה הזו הוא היישום של פקטור נרמול המבוסס על המדרון המציין את הטיית התאמת אורך המולקולה (קו ורוד). החישובים הדרושים כלולים בתוכנית ניתוח (ראה שלב 8.3 בפרוטוקול).

איור 1: דיאגרמה המציגה הצעדים הראשונים של HIV-1 הפוך שעתוק. התהליך מתחיל עם חישול של tRNA(Lys,3) (כתום) לאתר איגוד פריימר (PBS) RNA נגיפי גנומית (שלב 1), אשר מאפשר החניכה, התארכות של נגיפי cDNA (כחול, שלב 2). / ת, ה-RNA הגנומי תבנית נפגמים בשל פעילות RNaseH של RT (שלב 3). ביניים המלא הראשון בתהליך של שעתוק במהופך היא מינוס-strand חזק-התחנה (-) sss cDNA, אשר תושלם כאשר הפילמור RT מזורז מגיע 5'-הסופית של gRNA באזור (R) חוזר (שלב 3). בועז (-) ביניים מועבר על ידי חישול משלימים טרמינל 3' באורך חוזרת באזור (משמאל לימין) R 3'-הסופית של התבנית RNA הגנומי. מכאן, ממשיך הפילמור (שלב 4). השיטה המתוארת התקדמות שעתוק במהופך נקבעת על-ידי מיפוי את האורך המדויק של nascent cDNA ויראלי (כחול). PPT, בדרכי polypurine; U5, 5'-רצף ייחודי; U3, 3'-רצף ייחודי. איור זה פרסום מחדש של הפרסום הקודם6. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
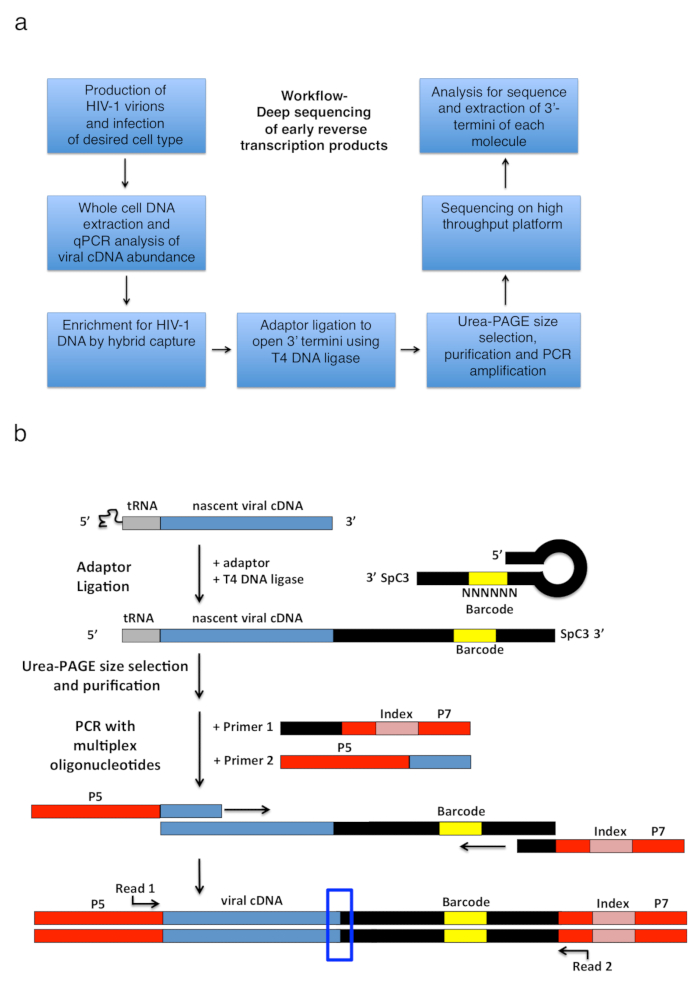
איור 2 : חלוקה לרמות זרימת עבודה ובתרשימים מתאם מצדו ואסטרטגיה הגברה PCR. (א) לזרימת בהתוויית שלבים עיקריים של הטכניקה המתוארת לקבוע 3'-טרמיני (termini) של HIV-1 הפוך תעתיקים תאים נגועים. הדמות היא ממאמרו של הפרסום הקודם6. (ב) תיאור סכמטי של מתאם מצדו ואסטרטגיה הגברה PCR. מולקולות cDNA המתהווה של אורך משתנה מטוהר השלבים הקודמים מאתרים את מתאם דנ א חד גדילי באמצעות T4 DNA ליגאז. מתאם סיכת ראש (בשם "מלאה קווק + MiSeq", ראה טבלה 1) עיצוב בהשראת קווק ואח. 11. נושאת המתאם אקראי 6 רצף ברקוד nt, אשר מאפשר בשיוך בסיס כדי להקל על מצדו, במקביל משמש כמזהה ייחודי קריאות. 3'-טרמיני של מתאם נושא כרווח (SpC3) כדי למנוע מצדו עצמית. המוצרים מחוברים מופרדים מתאם עודף על-ידי denaturing לזיהוי בג'ל (עמוד). חומצות גרעין הג'ל צבעונית, חותכים לשלושה חלקים נפרדים, שוויון-גודל ג'ל באזור מלמעלה המתאם לבאר כפי שנעשה25. אחרי • תנאי, משקעים, ו resuspension, המוצרים הם מוגבר עם תחל חישול רצף ידוע של המתאם ה-PCR (פריימר 1, קיט oligonucleotide מולטיפלקס, ראה טבלה של חומרים), פריימר נושאת 22 הראשון nt של HIV-1 רצף 5'-LTR מיד לאחר של tRNA (פריימר 2, MP1.0 + 22HIV). 5'-טרמיני של תחל שבחרת נושא מתאמים עבור פלטפורמת רצף שבחרת (P5 ו- P7) כמו גם רצף אינדקס כדי להבחין בין דוגמאות בודדות לרוץ באותה ספריה. נקודות המוצא של הרצף לקרוא תחל מסומנים. התיבה הכחולה מציינת אזור עניין כדי לקבוע את המקורי 3'-טרמיני (termini) של מולקולת שנתפסו. נתון זה ממאמרו של הפרסום הקודם6. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
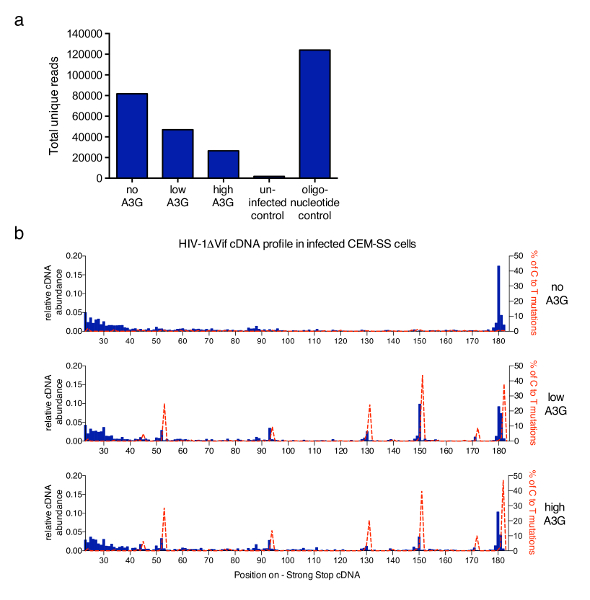
איור 3 : תוצאות נציג. (א) קרא הספירה הכוללת של מדגמים מייצגים מעובד עם פרוטוקול המתואר. זה כולל את כל רצפי שמצבן הייחודי קריאות של מולקולות HIV-1 עם שלהם 3'-טרמיני (termini) בתוך 635 הראשון nt של המינוס לנטישה cDNA (עד PPT, ראה איור 1). זיהום עם HIV-1 לא נושאת A3G מניב המספר הגבוה ביותר של קריאות, בעוד A3G מעכב סינתזה cDNA ובכך מפחיתה את הסכום הכולל ספירת קריאה. תאים נגוע שימש פקד שליליים, בעוד קבוצה של oligonucleotides סינתטי מספק פקד חיובי. b) שפע יחסי של cDNAs עבור כל אורך בין nt עמדות 23 ו- 182 (באורך מלא - בועז cDNA הוא 180 עד 182 nt) של HIV-1NL4.3 רצף (ציר x) מוצג היסטוגרמות כחול (סולם על ציר-y השמאלי). שפע יחסי של cDNA מחושב לפי מספר רצפי הפסקת ב נוקלאוטיד נתון בתוך הרצף cDNA - בועז לחלק את הסכום של כל קורא מדידה 182nt או פחות מוחלט. בקווים אדום מקווקו מוצגות האחוזים של קריאות נושאת ג-ל-T/U מוטציות במיקום המתאים (סולם על הצירים y נכון–) על כל שטח התקליטור. איור 3 b פרסום מחדש של הפרסום הקודם6. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
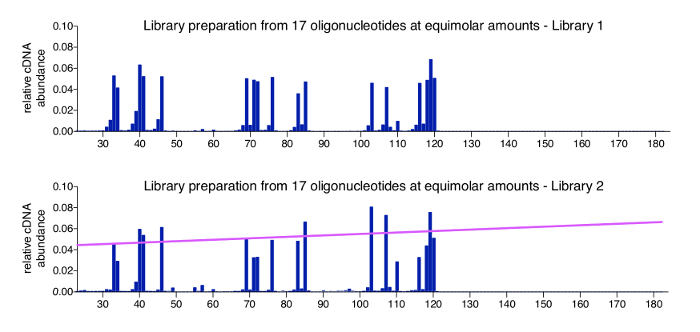
איור 4 : תוצאות נציג של דגימות הבקרה. מוצגות שני פרופילים עבור בריכות המכיל כמויות equimolar 17 oligonucleotides סינטטי אורך שונים. Oligonucleotides אלה יש רצפים של HIV-1NL4.3 , נבחרו כדי לכסות באורכים שונים ולהציג כל 4 בסיסים כמו 3'-נוקלאוטיד (ראה טבלה 2). הגרף העליון מציג את דגימת בקרה חיובית של איור 3. הטייה משמעותית כלפי אורך המולקולה או פתוח 3'-טרמיני מזוהה. הגרף התחתון מציג בספרייה אחרת להפעיל, אשר הופק דעה קדומה אורך משניות ברצף. במקרה זה, מומלץ להחיל גורם נרמול, אשר נגזר מן המדרון (מוצגת בוורוד) המייצג הטיה בגודל. איור זה פרסום מחדש של הפרסום הקודם6. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| Oligo שם | אורך ב- nt | רצף | מטרה | יצרן (טיהור) | ||||||||||||||
| מלא קווק + MiSeq | 61 | 5'-PHO-tgaagagcctagtcgctgttcannnnnnctgcccatagagagatcggaagagcacacgtct-SpC3-3' | מתאם | טכנולוגיות IDT DNA (HPLC) | ||||||||||||||
| 2xBiotin SS פיתיון | 40 | 5'-ביוטין-cagtgtggaaaatctctagcagtggcgcccgaacagggac-ביוטין-3' | לכידת היברידית | Mwg ב Eurofins (HPLC) | ||||||||||||||
| ביוטין 1-16 ss | 22 | 5'-cagtgtggaaaatctctagcag-BiTEG-3' | לכידת היברידית | Mwg ב Eurofins (hplc, קורס | ||||||||||||||
| ביוטין tRNA + CTG | 16 | 5'-cagtggcgcccgaaca-BITEG-3' | לכידת היברידית | Mwg ב Eurofins (HPLC) | ||||||||||||||
| MP1.0 + 22HIV | 82 | 5'-aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatctcactgctagagattttccacactg-3' | PCR-הגברה | Mwg ב Eurofins (hplc, קורס | ||||||||||||||
טבלה 1: טבלה של oligonucleotides כולל אורך, רצפים של שינויים שאינם מנוצלים בפרוטוקול מתואר. הטבלה היא ממאמרו של הפרסום הקודם6. אנא לחץ כאן כדי להוריד את השולחן הזה כקובץ excel.
| Oligo שם | אורך ב- nt | רצף | יצרן (טיהור) | |||||||||||||
| HTP קון סי ארוך | 120 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggcttaagc-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP לרמות G ארוך | 119 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggcttaag-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP לרמות לא ארוך | 116 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggctt-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP con A ארוכה | 118 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagctttattgaggcttaa-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| קון HTP באמצע C | 76 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcac-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| קון HTP אמצע G (א) | 71 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacg-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| קון HTP אמצע G (b) | 72 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgg-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| קון HTP מאמצע א | 69 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacaga-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| קון HTP אמצע T | 85 | 5'-ctgctagagattttccacactgactaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactt-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP con A קצרה | 40 | 5'-ctgctagagattttccacactgactaaaagggtctgaggga-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP לרמות T קצר | 33 | 5'-ctgctagagattttccacactgactaaaagggt-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP לרמות G קצר | 41 | 5'-ctgctagagattttccacactgactaaaagggtctgaggg-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP קון סי קצר | 34 | 5'-ctgctagagattttccacactgactaaaagggtc-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| קון HTP 46 (T) | 46 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctct-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP Con 83 (C) | 83 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactac-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| HTP Con 103 (C) | 103 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagc-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
| קון HTP 107 (א) | 107 | 5'-ctgctagagattttccacactg actaaaagggtctgagggatctctagttaccagagtcacacaacagacgggcacacactactttgagcactcaaggcaagcttta-3' | Mwg ב Eurofins (HPLC) | |||||||||||||
בטבלה 2: עניינים 17 oligonucleotides שליטה סינתטי המשמש כמדגם בקרה חיובית. Oligonucleotides ראש 13 נבחרו בהתאם לגודל [ארוך (116 עד 120 nt), באמצע (69 כדי 85 nt), קצר (33: 41 nt)] כמו גם שלהם 3'-טרמיני (termini). הטבלה היא ממאמרו של הפרסום הקודם6. אנא לחץ כאן כדי להוריד את השולחן הזה כקובץ excel.
Discussion
הזמינות של רצף עמוק מהירה, אמינה וחסכונית יש מהפכה היבטים רבים בתחום מדעי החיים, המאפשר עומק רב ניתוחים המבוססת על רצף. אתגר הנותרים טמון חדשני לעיצוב ויצירה של נציג רצף ספריות. כאן נתאר פרוטוקול כדי ללכוד nascent ויראלי cDNA מולקולות, במיוחד intermediates של תהליך שעתוק במהופך HIV-1.
השלב הקריטי ביותר באסטרטגיה זו הוא מצדו של מתאם ל פתוח 3'-טרמיני באופן כמותי לא משוחדת. היעילות של ligations בין שני ssDNA טרמיני (termini), שניהם אינטר- ואת התפלגות, חקר, אופטימיזציה עבור מגוון יישומים11,26,27,28,29. הבחירה של באמצעות מתאם סיכת ראש עם T4 DNA ליגאז בתנאים המתוארים שלב 3.3 הוא התוצאה של אופטימיזציה אמפירי שבו אנו מוערכים ligases שונים, מתאמים, ריאגנטים עבור מצדו של oligonucleotides סינתטיים המייצגים רצפים HIV-1 (טבלה 2) (נתונים לא מוצג). בתגובות מבחן אלה במבחנה , אישרנו T4 DNA ליגאז מתווכת מצדו של מתאם סיכת ראש, כפי שתואר על ידי קווק ואח. 11, יש דעה קדומה נמוכה מאוד, והוא משיג ליד מוחלטת מצדו של מולקולות מקבל כאשר המתאם משמש עודף. היעילות מצדו לא הושפע התוספת של נוקלאוטיד ברצף כדי להבהיר את המתאם תואם למערכת פריימר מולטיפלקס (ראה איור 4). לשם השוואה, מצאנו כי thermostable 5' DNA/RNA ליגאז (ראה "ליגאז A", בטבלה של חומרים עבור המדויק ligases בהשוואה כאן), שהינו מהונדסים RNA ליגאז שפותחה באופן חלקי כדי לשפר את יעילות מצדו עם ssDNA כמו מקבל 27, היה אכן יותר יעיל נבדק שתי מולקולות ssDNA מאשר רנ א ליגאז (ליגאז B") אבל נאלץ דעה קדומה משמעותי, עם הבדלים חריפים יעילות מצדו אפילו בין oligonucleotides עם הבדלים אורך בסיס יחיד [בטבלה 2 ; HTP con אמצע G (א) ו- (ב)]. יתר על כן, מצאנו רק דעה קדומה מינימלי בתגובות עם "ליגאז C" בשילוב עם מתאם נושא של אקראי 5'-termini (אסטרטגיה להשתמש כדי לקזז נוקלאוטיד ידוע הטייה של "ליגאז C"; ר' למשל דינג et al. 30). עם זאת, ליגאז C-ligations הבין-מולקולרי בתיווך לא היו שלמים, עיבוד T4 DNA ליגאז המערכת הבחירה מעולה.
מספר פעולות בקרת איכות על הקורס של הפרוטוקול והכללה של פקדים חיוביים ושליליים לאפשר איתור בעיות פוטנציאליות לפני המשך assay ולספק הדרכה לפתרון מאמצים. Quantifications qPCR בשלבים 2.2.2 ו 2.3.12 להבטיח כי כמות החומר קלט מספיקה. CDNA טיפוסי עותק המספרים בטווח • תנאי (מתוך שלב 2.1) 200 µL מ בסביבות 10,000 ל 300 אלף µL. הצעד לכידת היברידית יכול לגרום לאובדן של HIV-1 הכוללת חלק cDNA כמות אך צריך לגרום לכך העשרת חזקה של cDNA HIV-1 ספציפית על הסלולר הדנ א, אשר יכול להיקבע על-ידי שימוש תחל המתאים כדי לכמת את הדנ א לפני ואחרי העשרה על-ידי qPCR או על ידי מדידת ריכוז ה-DNA מוחלט. התאושש cDNA HIV-1 לאחר ההיברידית ללכוד צעדים צריך להיות לפחות 10% של הקלט. נמוך החל חומר עשוי להסביר אחרת פקד חיובי oligonucleotide מוצלח (ראה שלב 3.3.2) אבל רק מוגבלת קריאות מושגת הדגימות. נמוך לקרוא מספרים הכוללת גם יכול להיות מוסבר על ידי מופרזת של הריכוז ספריית בשל נוכחותם של מינים לא רלוונטי DNA ללא מתאמים MiSeq. זה יגרום בצפיפות נמוכה אשכול, ניתן לשפר באמצעות קביעת הריכוז של HIV-1 רצפים בספרייה מאת qPCR בנוסף הסכום הכולל של ה-DNA על ידי מבחני fluorometric. בשל אופיו רגישה מאוד של השיטה, טיפול מיוחד יש לנקוט כדי למנוע זיהום אפילו ברמה נמוכה, שניהם מדגימות אחרים (בפרט, מתוך המניות שליטה ריכוז גבוה oligonucleotide) כמו גם כמו של ציוד מעבדה. עובד באולטרה לחיטוי PCR תחנת העבודה הוא מועיל בהקשר זה. בג'ל אוטומטיות של הספרייה הסופי (שלב 6.1.2) הוא מדד בקרת איכות נוספים. הטווח גודל חומצת גרעין בדרך כלל שנצפה הוא בין 150-500 nt. תחל שניתן לאתר בפקד אופציונלי לאחר את ה-PCR, ולפני טיהור (ראה הערה בשלב 5.2) אמור הנעדר. תוצאה נציג, עקומת העוצמה המדגם כולל לשיא בסביבות 160 170 nt וחד יותר השני של שיא בסביבות 320 ל 350 nt. זה משקף ככל הנראה לעתים קרובות-ראה שפע גבוה יותר קצר יחסית תעתיקים הפוכה (1-20 nt הוספה אורך) והן באורך מלא חזק הפסקה (180 עד 182 nt הוספה אורך) (איור 3ב').
בעוד הפרוטוקול הציג לבין תחל שנבחרו הן ספציפיות עבור בונה מוקדם של שעתוק במהופך HIV-1, השיטה זו חלים באופן כללי על כל מחקר במטרה לקבוע פתוח 3'-טרמיני (termini) של ה-DNA. השינויים העיקריים הנדרשים בהקשרים אחרים תהיה השיטה עבור לכידת היברידית ואסטרטגיה עיצוב של פריימר. לדוגמה, אם המטרה היא להיות מותאם מאוחר תעתיקים HIV-1, מספר גדול יותר של שונים oligonucleotides biotinylated לכידת חישול על פני אורך cDNA יהיה מומלץ, יקטן ככל הנראה את ההפסד בשלב לכידת היברידית. כאמור במבוא, חשוב לקחת בחשבון את מגבלות בעת עיצוב הטווח שבו 3'-termini הם יזוהו להימנע מקורות שונים של הטיה. ראשית, ייתכן דעה קדומה תגובות PCR אם התבניות עם מתאם אורך משתנה במידה רבה. שנית, רצף פלטפורמת המשמש כאן (למשל, MiSeq) יש טווח אורך הוספה המועדפת עבור קיבוץ באשכולות אופטימלית, ולא באופן משמעותי מוצרים קצר וארוך עשוי לא להיות וסודרו עם יעילות זהה. בחלקה זו ניתן לטפל שהמפתחות, כפי שנעשה על-ידי חישוב מקדם תיקון עבור אורך קדומה (ראה איור 4, הגרף התחתון). עם זאת, אם האזור שבו רצוי 3'-termini מיפוי ארוך (nt > 1000), רצוי יותר לפצל את התגובות עם התרשימים מחוברים ולהשתמש primers נגד הזרם מרובים כדי להעריך 3'-termini בסעיפים.
התוכנית ניתוח נכתב בתוך הבית עבור מטרה ספציפית של ניתוח שני נוקלאוטיד האחרון את רצף HIV-1 סמוכים רצף מתאם קבוע, כמו גם את הווריאציה הבסיס של כל בסיסים לזהות מוטציות כל. השלבים הבודדים המרכיבים הבאים: ראשית, רצפי מתאם נסגרים באמצעות ערכת הכלים fastx-0.0.13; לאחר מכן, יוסרו כל רצפי אשר משוכפלות (כלומר רצפים זהים כולל הברקוד). כל קורא ייחודי הנותרים מיושרים ואז הרצף HIV-1 באמצעות עניבת הפרפר (http://bowtie-bio.sourceforge.net/index.shtml) עם ההתאמה המרבית שוכן בגובה שלושה בסיסים. הרצף תבנית מורכבת של 635 הראשון nt של HIV-1 cDNA (זן NL4.3), הכוללת את הרצף - בועז, המוצר הראשון של העברת סטרנד עד המסלול polypurine (U5-R-U3-PPT; ראה איור 1). ובכך, התוכנה שסופקה ואת תבניות מתאימים רק ישירות אם השיטה משמשת אותו יישום (זיהוי מוקדם הפוכה תעתיקים של HIV-1NL4.3). התאמות תצטרך להתבצע עבור רצפי יעד אחרים. העמדות של 3'-טרמיני עבור כל קריאה נקבעו על ידי העמדה היישור. השיחות הבסיס לכל תפקיד מוקלטות, מוטציה המחירים מחושבים מתוך סה כ סיקור כל בסיס, אשר משתנה, כמו קורא באורכים שונים, מוסיף הרבה זמן אולי לא לגמרי יכוסה על ידי רצף 125-בסיס ב- Read2.
לסיכום, אנו מאמינים השיטה המתוארת כדי להיות כלי רב ערך עבור סוגים רבים של מחקרים. ברור יישומים כוללים חקירות של המנגנונים עיכוב שעתוק במהופך דרך תרופות תרופתי או הגבלת הסלולר גורמים. אולם, התאמות קלות יחסית צריך שיהיה צורך להתאים את המערכת כדי 3'-termini מיפוי בתוך אחרים חד גדילי DNA נגיפי intermediates, אשר נמצאים, לדוגמה, בשכפול הפרוו. יתר על כן, העיקרון של השיטה, במיוחד שלה צעד מצדו ממוטבת, באפשרותך לספק חלק הליבה של הספרייה הכנה עיצוב עבור אפיון כל הרחבות 3'-DNA, כולל elongations על ידי תאי DNA גדילי כפול polymerases.
Disclosures
המחברים מצהירים כי יש להם כלום לחשוף.
Acknowledgements
המחברים להכיר את התמיכה של חברי של מלים מעבדה, לואיס אפולוניה, אולה Jernej, רבקה אוקי. המחברים תודה מאט ארנו במרכז של המלך קולג בלונדון גנומית, דבי יוז ב לונדון מכללת אוניברסיטת (UCL), מנהלת המכון לנוירולוגיה הבא הדור רצף מתקן, על העזרה עם רצף MiSeq. העבודה נתמך על ידי בריטניה הרפואי המועצה למחקר (G1000196 ומר/M001199/1 כדי מ'מ), טרסט (106223/ת/14/Z כדי מ'מ), תכנית המסגרת השביעית של הנציבות האירופית (האיחוד FP7/2007-2013) בהסכם גרנט לא. PIIF-GA-2012-329679 (כדי D.P.), ועל את משרד הבריאות באמצעות המכונים הלאומיים פרס הבריאות מחקר מקיף ביו-רפואית מרכז מחקר לקרן NHS של האיש, סנט תומס אמון בשותפות בקינגס קולג '-לונדון, המלך מכללת בית החולים NHS קרן נאמנות.
Materials
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | |
| Dulbecco's Modified Eagle's Medium | Gibco | 31966-021 | |
| Penicillin/Streptomycin | Gibco | 15150-122 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| HeraCell Vios 250i CO2 Incubator | Thermo Scientific | 51030966 | |
| Laminar flow hood - CAS BioMAT2 | Wolflabs | CAS001-C2R-1800 | |
| 10mm TC-treated culture dish | Corning | 430167 | |
| TrypLE™ Express (1x), Stable Trypsin Replacement Enzyme | Gibco | 12605-010 | |
| OptiMEM® (Minimal Essential Medium) | Gibco | 31985-047 | |
| HIV-1 NL4-3 Infectious Molecular Clone (pNL4-3) | NIH Aids reagent program | 114 | |
| Polyethylenimine (PEI) - MW:25000 | PolySciences Inc | 23966-2 | dissolved at 1mg/ml and adjusted to pH7 |
| RQ1- Rnase free Dnase | Promega | M6101 | |
| Filter 0.22 μm | Triple Red Limited | FPE404025 | |
| 15 mL polypropylene tubes | Corning | CLS430791 | |
| Sucrose | Calbiochem | 573113 | |
| Phosphate Buffered Saline (1x) | Gibco | 14190-094 | |
| Ultracentrifuge tubes | Beckman Coulter | 344060 | |
| Ultracentrifuge | Sorval | WX Ultra Series | Th-641 Rotor |
| Alliance HIV-1 p24 antigen ELISA kit | Perkin Elmer | NEK050001KT | |
| CEM-SS cells | NIH Aids reagent program | 776 | |
| Roswell Park Memorial Institute Medium | Gibco | 31870-025 | |
| CoStar® TC treated multiple well plates | Corning | CLS3513-50EA | |
| Benchtop centrifuge: Heraus™ Multifuge™ X3 FR | Thermo Scientific | 75004536 | |
| TX-1000 Swinging Bucket Rotor | Thermo Scientific | 75003017 | |
| Microcentrifuge: 5424R | Eppendorf | 5404000060 | |
| Total DNA extraction kit (DNeasy Blood and Tissue kit) | Qiagen | 69504 | |
| Nuclease free H2O | Ambion | AM9937 | |
| Cutsmart buffer | New England Biolabs (part of DpnI enzyme) | R0176S | |
| DpnI restriction enzyme | New England Biolabs | R0176S | |
| Oligonucleotides for qPCR | MWG Eurofins | N/A | HPSF purification |
| TaqMan PCR Universal Mastermix | Thermo | 4304437 | |
| LoBind Eppendorf® tubes | Eppendorf | 30108078 | |
| Axygen™ aerosol filter pipette tips, 1000 μL | Fisher Scientific | TF-000-R-S | |
| Axygen™ aerosol filter pipette tips, 200 μL | Fisher Scientific | TF-200-R-S | |
| Axygen™ aerosol filter pipette tips, 20 μL | Fisher Scientific | TF-20-R-S | |
| Axygen™ aerosol filter pipette tips, 10 μL | Fisher Scientific | TF-10-R-S | |
| PCR clean hood | LabCaire | Model PCR-62 | |
| DynaMag™2-magnet | Thermo | 12321D | |
| Streptavidin MagneSphere® paramagnetic particles | Promega | Z5481 | |
| Casein | Thermo Scientific | 37582 | |
| End over end rotator, Revolver™ 360° | Labnet | H5600 | |
| Tris-Base | Fisher Scientific | BP152-5 | |
| Hydrochloric Acid | Sigma | H1758-100ML | |
| EDTA disodium salt dihydrate | Electran (VWR) | 443885J | |
| Sodium Chloride | Sigma | S3014 | |
| Dri-Block® Analog Block Heater | Techne | UY-36620-13 | |
| PCR tubes and domed caps | Thermo Scientific | AB0266 | |
| PCR machine | Eppendorf | Mastercycler® series | |
| T4 DNA ligase | New England Biolabs | M0202M | |
| 40% Polyethylene glycol solution (PEG) in H2O, MW: 8000 | Sigma | P1458-25ML | |
| Betaine solution, 5M | Sigma | B0300-1VL | |
| Gel loading buffer II (formamide buffer) | Thermo Scientific | AM8546G | |
| Precast 6% TBE urea gels | Invitrogen | EC6865BOX | |
| Mini cell electrophoresis system | Invitrogen, Novex | XCell SureLock™ | |
| Tris/Borate/EDTA solution (10x) | Fisher Scientific | 10031223 | |
| Needle 21 G x1 1/2 | VWR | 613-2022 | |
| SYBR Gold nucleic acid stain (10000x) | Life Technologies | S11494 | |
| Dark Reader DR46B transilluminator | Fisher Scientific | NC9800797 | |
| Ammonium acetate | Merck | 101116 | |
| SDS solution 20% (w/v) | Biorad | 161-0418 | |
| Centrifuge tube filter | Appleton Woods | BC591 | |
| Filter Glass Fibre Gf/D 10mm | Whatman (VWR) | 512-0427 | |
| polyadenylic acid (polyA) RNA | Sigma | 10108626001 | |
| Glycogen, molecular biology grade | Thermo Scientific | R0561 | |
| Isopropanol (2-propanol) | Fisher Scientific | 15809665 | |
| Ethanol, molecular biology grade | Fisher Scientific | 10041814 | |
| Accuprime™ Supermix I (DNA polymerase premix) | Life Technologies | 12342-010 | |
| NEBNext® Multiplex Oligo for Illumina (Index Primer Set 1 and 2) | New England Biolabs | E7335S; E7500S | |
| Tapestation D1000 Screentape High sensitivity | Agilent Technologies | 5067- 5584 | |
| Tapestation D1000 Reagents | Agilent Technologies | 5067- 5585 | |
| 2200 Tapestation - automated gel electrophoresis system | Agilent Technologies | G2965AA | |
| Agencourt® AMPure® beads XP | Beckman Coulter | A63880 | |
| Qubit™ dsDNA HS Assay Kit | Invitrogen | Q32851 | |
| Qubit™ 2.0 Fluorometer | Invitrogen | Q32866 | |
| Topo™ TA cloning Kit | Invitrogen | 450071 | |
| Sequencing platform: MiSeq System | Illumina | ||
| Experiment Manager (Sample sheet software) | Illumina | Note: Use TruSeq LT as a template | |
| Miseq™ Reagent kit V3 (150 cycle) | Illumina | MS-102-3001 | |
| Sequencing hub: Basespace | Illumina | https://basespace.illumina.com | |
| Ligase A: Thermostable 5’ App DNA/RNA ligase | NEB | M0319S | Not used in this protocol, but tested in optimization process with results described in the discussion. |
| Ligase B: T4 RNA ligase 1 | NEB | M0204 | Not used in this protocol, but tested in optimization process with results described in the discussion. |
| Ligase C: CircLigase | Epicentre | CL4111K | Not used in this protocol, but tested in optimization process with results described in the discussion. |
References
- Herschhorn, A., Hizi, A. Retroviral reverse transcriptases. Cellular and Molecular Life Sciences. 67 (16), 2717-2747 (2010).
- Hu, W. S., Hughes, S. H. HIV-1 reverse transcription. Cold Spring Harbor Perspectives in Medicine. 2 (10), (2012).
- Levin, J. G., Mitra, M., Mascarenhas, A., Musier-Forsyth, K. Role of HIV-1 nucleocapsid protein in HIV-1 reverse transcription. RNA Biology. 7 (6), 754-774 (2010).
- Menendez-Arias, L., Sebastian-Martin, A., Alvarez, M. Viral reverse transcriptases. Virus Research. , (2016).
- Telesnitsky, A., Goff, S. P., Coffin, J. M., Hughes, S. H., Varmus, H. E. . Retroviruses. , (1997).
- Pollpeter, D., et al. Deep sequencing of HIV-1 reverse transcripts reveals the multifaceted antiviral functions of APOBEC3G. Nature Microbiology. 3 (2), 220-233 (2018).
- Frohman, M. A., Dush, M. K., Martin, G. R. Rapid production of full-length cDNAs from rare transcripts: amplification using a single gene-specific oligonucleotide primer. Proceedings of the National Academy of Sciences of the United States of America. 85 (23), 8998-9002 (1988).
- Liu, X., Gorovsky, M. A. Mapping the 5' and 3' ends of Tetrahymena thermophila mRNAs using RNA ligase mediated amplification of cDNA ends (RLM-RACE). Nucleic Acids Research. 21 (21), 4954-4960 (1993).
- Ince, I. A., Ozcan, K., Vlak, J. M., van Oers, M. M. Temporal classification and mapping of non-polyadenylated transcripts of an invertebrate iridovirus. Journal of General Virology. 94, 187-192 (2013).
- Hafner, M., et al. RNA-ligase-dependent biases in miRNA representation in deep-sequenced small RNA cDNA libraries. RNA. 17 (9), 1697-1712 (2011).
- Kwok, C. K., Ding, Y., Sherlock, M. E., Assmann, S. M., Bevilacqua, P. C. A hybridization-based approach for quantitative and low-bias single-stranded DNA ligation. Analytical Biochemistry. 435 (2), 181-186 (2013).
- Abram, M. E., Tsiang, M., White, K. L., Callebaut, C., Miller, M. D. A cell-based strategy to assess intrinsic inhibition efficiencies of HIV-1 reverse transcriptase inhibitors. Antimicrobial Agents and Chemotherapy. 59 (2), 838-848 (2015).
- Bishop, K. N., Verma, M., Kim, E. Y., Wolinsky, S. M., Malim, M. H. APOBEC3G inhibits elongation of HIV-1 reverse transcripts. PLoS Pathogens. 4 (12), 1000231 (2008).
- Zack, J. A., Haislip, A. M., Krogstad, P., Chen, I. S. Incompletely reverse-transcribed human immunodeficiency virus type 1 genomes in quiescent cells can function as intermediates in the retroviral life cycle. Journal of Virology. 66 (3), 1717-1725 (1992).
- Adachi, A., et al. Production of acquired immunodeficiency syndrome-associated retrovirus in human and nonhuman cells transfected with an infectious molecular clone. Journal of Virology. 59 (2), 284-291 (1986).
- Shah, V. B., Aiken, C. In vitro uncoating of HIV-1 cores. Journal of Visualized Experiments. (57), (2011).
- JoVE Science Education Database. Science Education Database: Basic Methods in Cellular and Molecular Biology: Passaging Cells. Journal of Visualized Experiments. , (2018).
- JoVE Science Education Database. JoVE Science Education Database: Basic Methods in Cellular and Molecular Biology: Using a Hemocytometer to Count Cells. Journal of Visualized Experiments. , (2018).
- Zhou, M. Y., Gomez-Sanchez, C. E. Universal TA cloning. Current Issues in Molecular Biology. 2 (1), 1-7 (2000).
- Zhang, S., Cahalan, M. D. Purifying plasmid DNA from bacterial colonies using the QIAGEN Miniprep Kit. Journal of Visualized Experiments. (6), 247 (2007).
- Mangeat, B., et al. Broad antiretroviral defence by human APOBEC3G through lethal editing of nascent reverse transcripts. Nature. 424 (6944), 99-103 (2003).
- Gillick, K., et al. Suppression of HIV-1 infection by APOBEC3 proteins in primary human CD4(+) T cells is associated with inhibition of processive reverse transcription as well as excessive cytidine deamination. Journal of Virology. 87 (3), 1508-1517 (2013).
- Harris, R. S., et al. DNA deamination mediates innate immunity to retroviral infection. Cell. 113 (6), 803-809 (2003).
- Zhang, H., et al. The cytidine deaminase CEM15 induces hypermutation in newly synthesized HIV-1 DNA. Nature. 424 (6944), 94-98 (2003).
- Konig, J., et al. iCLIP--transcriptome-wide mapping of protein-RNA interactions with individual nucleotide resolution. Journal of Visualized Experiments. (50), (2011).
- Troutt, A. B., McHeyzer-Williams, M. G., Pulendran, B., Nossal, G. J. Ligation-anchored PCR: a simple amplification technique with single-sided specificity. Proceedings of the National Academy of Sciences of the United States of America. 89 (20), 9823-9825 (1992).
- Zhelkovsky, A. M., McReynolds, L. A. Structure-function analysis of Methanobacterium thermoautotrophicum RNA ligase - engineering a thermostable ATP independent enzyme. BMC Molecular Biology. 13 (24), (2012).
- Li, T. W., Weeks, K. M. Structure-independent and quantitative ligation of single-stranded DNA. Analytical Biochemistry. 349 (2), 242-246 (2006).
- Gansauge, M. T., et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase. Nucleic Acids Research. 45 (10), 79 (2017).
- Ding, Y., Kwok, C. K., Tang, Y., Bevilacqua, P. C., Assmann, S. M. Genome-wide profiling of in vivo RNA structure at single-nucleotide resolution using structure-seq. Nature Protocols. 10 (7), 1050-1066 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved