A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ספקטרוסקופיה של תקליטור לחקר אינטראקציות בין DNA לחלבון
In This Article
Summary
האינטראקציה של שיפוץ כרומטין תלוי ATP עם ליגנד DNA מתוארת באמצעות ספקטרוסקופיה CD. ניתן להשתמש בשינויים הקונפורמציה המושרים על מקדם גנים שניתח על ידי הפסגות שנוצרו כדי להבין את מנגנון הרגולציה התמלולית.
Abstract
ספקטרוסקופיה של דיכרויזם מעגלי (CD) היא שיטה פשוטה ונוחה לחקור את המבנה המשני ואת האינטראקציות של ביומולקולים. ההתקדמות האחרונה בספקטרוסקופיית תקליטורים אפשרה לחקור אינטראקציות בין חלבון DNA ודינמיקה קונפורמציה של DNA במיקרו-סביבה שונה בפירוט להבנה טובה יותר של ויסות התמלול ב- vivo. האזור סביב אזור תמלול פוטנציאלי צריך להיות לא אחיד כדי שתעתיק יתרחש. זהו תהליך מורכב הדורש תיאום של שינויים histone, כריכה של גורם שעתוק ל- DNA, ופעילויות אחרות שיפוץ כרומטין. באמצעות ספקטרוסקופיה CD, ניתן ללמוד שינויים קונפורמציה באזור המקדם הנגרמים על ידי חלבונים רגולטוריים, כגון שיפוץ כרומטין תלוי ATP, כדי לקדם שעתוק. ניתן לעקוב אחר השינויים הקונפורמציה המתרחשים בחלבון. בנוסף, ניתן לטפל בשאילתות לגבי הזיקה של החלבון לדנ"א היעד שלו ולייחודיות הרצף שלו על ידי שילוב מוטציות בדנ"א היעד. בקיצור, ההבנה הייחודית של שיטה רגישה וזולה זו יכולה לחזות שינויים בדינמיקה הכרומטית, ובכך לשפר את ההבנה של רגולציה תמלול.
Introduction
דיכרויזם מעגלי (CD) היא טכניקה ספקטרוסקופית הנשענת על כיראליות אינהרנטית של מקרומולקולים ביולוגיים המובילה לספיגה דיפרנציאלית של אור מקוטב ימני ושמאלי. ספיגה דיפרנציאלית זו ידועה כדיכרואיזם מעגלי. הטכניקה, אם כן, יכולה לשמש כדי לתאר את הקונפורמציה של מקרומולקולים ביולוגיים, כגון חלבונים ו- DNA, שניהם מכילים מרכזי כיראליים1,2.
גלים אלקטרומגנטיים מכילים רכיבים חשמליים ומגנטיים כאחד. הן השדות החשמליים והן השדות המגנטיים מתנדנדים בניצב לכיוון התפשטות הגלים. במקרה של אור לא קוטבי, שדות אלה מתנדנדים בכיוונים רבים. כאשר האור מקוטב מעגלית, שני שדות אלקטרומגנטיים מתקבלים בהפרש פאזה של 90° זה לזה. מולקולות כיראליות מראות סיבוב אופטי מעגלי (birefringence) כך שהן יספגו את האור המקוטב המעגלי הימני ואת האור המקוטב העגול השמאלי בהיקפים שונים3. השדה החשמלי המתקבל יעקוב כאליפסה, פונקציה של אורך הגל. ספקטרום התקליטורים, אם כן, נרשם כאליפטיות (q), והנתונים מוצגים כאליפטיות שאריות ממוצעת כפונקציה של אורך גל.
במקרה של חלבונים, Cα של חומצות אמינו (למעט גליצין) הוא chiral, וזה מנוצל על ידי ספקטרוסקופיה CD כדי לקבוע את המבנה המשני של מאקרומולקול זה4. ספקטרום התקליטור של מולקולות חלבון נרשם בדרך כלל בטווח ה- UV הרחוק. לחלבונים α-הליליים יש שתי רצועות שליליות ב-222 ננומטר ו-208 ננומטר ושיא חיובי אחד ב-193 ננומטר4. חלבונים עם מבנה משני אנטי-מקבילי β יריעות מראים שיא שלילי ב 218 ננומטר ושיא חיובי ב 195 nm4. חלבונים עם מבנים פרועים מראים אליפטיות נמוכה ליד 210 ננומטר ושיא שלילי ב 195 nm4. לכן, שיא מוגדר היטב / רצועות עבור מבנים משניים שונים להפוך CD כלי נוח כדי להבהיר את השינויים הקונפורמציה המתרחשים במבנה המשני של החלבונים במהלך denaturation, כמו גם כריכת ליגנד.
לחומצות גרעין יש שלושה מקורות של כיראליות: מולקולת הסוכר, ההוליות של המבנה המשני, והסדר של שלשה ארוכת טווח של DNA בסביבה5,6. ספקטרום התקליטור של חומצות גרעין נרשם בדרך כלל בטווח של 190 עד 300 ננומטר5,6. כל התאמה של DNA, בדיוק כמו חלבונים, נותן ספקטרום אופייני, אם כי הפסגות / רצועות יכול להשתנות במעלות מסוימות עקב תנאי ממס והבדלים ברצפי DNA7. B-DNA, הצורה הנפוצה ביותר, מאופיין בשיא חיובי סביב 260-280 ננומטר ושיא שלילי סביב 245 nm6. הפסגות/רצועות הדנ"א בצורת B הן בדרך כלל קטנות מכיוון שזוגות הבסיס מאונכים לסליל הכפול, ומעניקים כיראליות חלשה למולקולה. A-DNA נותן שיא חיובי דומיננטי ב 260 ננומטר ושיא שלילי סביב 210 nm6. Z-DNA,סל שמאלי, נותן רצועה שלילית ב 290 ננומטר ושיא חיובי סביב 260 nm6. DNA זה גם נותן שיא שלילי מאוד ב 205 nm6.
בנוסף לקונפורמציות אלה, DNA יכול גם ליצור טריפלקסים, מרובעים וסיכות ראש, שכולם ניתן להבחין על ידי ספקטרוסקופיה CD. G-quadruplex המקביל לתת להקה חיובית דומיננטית ב 260 ננומטר, בעוד G-quadruplex אנטי מקביל נותן רצועה שלילית ב 260 ננומטר ושיא חיובי ב 290 ננומטר, מה שהופך אותו קל להבחין בין שתי צורות של מבנים quadruplex6. טריפלקסים לא נותנים ספקטרום אופייני8. לדוגמה, הספקטרום של DNA באורך 36 נוקלאוטיד עם פוטנציאל ליצור סליל משולש תוך-עיני המכיל זוגות בסיס G.G.C ו- T.A.T בנוכחות Na+ מראה רצועה שלילית חזקה ב 240 ננומטר ושיא חיובי רחב. השיא החיובי הרחב מראה תרומות של 266, 273 ו-286 ננומטר. אותו אוליגונוקלאוטיד בנוכחות Na+ ו- Zn+ מציג ארבע להקות שליליות (213, 238, 266 ו-282 ננומטר) ושיא חיובי של 258 ננומטר. לכן, הספקטרום של DNA טריפלקס יכול להשתנות בהתאם לתנאי מלח8.
בנוסף לקונפורציות אלה, ספקטרום תקליטורים אפשר זיהוי של צורה אחרת של DNA הנקרא X-DNA. X-DNA נוצר כאשר רצף ה- DNA מכיל שאריות אדנין ותימין חלופיות. ספקטרום התקליטור של X-DNA מכיל שתי פסגות שליליות ב 250 ו 280 ננומטר. מעט מאוד מידע זמין על X-DNA, אם כי זה כבר ספקולציות לתפקד כמו כיור עבור supercoiling חיובי6,9. שינויים בספקטרום התקליטורים יכולים גם לחשוף פרטים על אינטראקציות חלבון ליגנד, ולכן, נוספו לארסנל של שיטות מולקולריות לאיתור אינטראקציות בין חלבון תרופתי10,11,12,13,14. ספקטרום תקליטור שימש גם כדי לפקח על השינויים במבנה המשני של חלבונים במהלך תהליך הקיפול15. באופן דומה, ספקטרום תקליטור יכול לשמש גם לבדיקת אינטראקציות ליגנד-DNA16,17.
ספקטרוסקופיה CD, אם כן, היא שיטה קלה וזולה להבחין בין צורות שונות של התאמת DNA, בתנאי שיש גישה לציוד ותוכנה לא כל כך זולים. השיטה רגישה ומהירה ביותר. זה רק דורש כמות קטנה של DNA, נותן לו יתרון על הטכניקה החלופית של ספקטרוסקופיה תהודה מגנטית גרעינית (NMR). קל גם לבצע טייטציות עם ליגנדים ומצעים. האילוץ העיקרי הוא שהדנ"א צריך להיות טהור מאוד. מומלץ להשתמש ב- DNA מטוהר של ג'ל פוליאקרילמיד (PAGE).
המידע המתקבל על ידי ספקטרום תקליטור שימש בעיקר כדי להסיק תכונות מבניות חלבון ולזהות קונפורמיות DNA ברורות. במחקר זה, ספקטרום תקליטור שימשו כדי לשלב את התוצאות שהתקבלו מניסוי in vivo Chromatin Immunoprecipitation (ChIP) כדי לתאר אם חלבון של עניין/ גורם שעתוק חזוי יכול להביא לשינוי קונפורמציה באזור האמרגן של הגנים המשפיעים שלה. שיתוף פעולה זה מסייע בהתקדמות של טכניקות ספקטרוסקופיות תקליטור מסורתיות על ידי חיזוי המנגנון של ויסות התמלול על ידי גורם התמלול החזוי באתר התחלת התמלול (TSS) של מקדם.
שיפוץ כרומטין הוא מנגנון מוגדר היטב הידוע לווסת תהליכים מטבוליים DNA על ידי הפיכת הכרומטין ארוז היטב נגיש לגורמים רגולטוריים שונים כגון גורמי שעתוק, רכיבים של שכפול DNA, או נזק חלבונים תיקון. משפצים כרומטין תלויי ATP, הידועים גם בשם משפחת החלבונים SWI/SNF, הם חלבונים מפתח לשיפוץ הנמצאים בתאים אאוקריוטים18,19. קיבוץ פילוגנטי סיווג את משפחת החלבונים SWI/SNF ל-6 תת-קבוצות20: דמוי Snf2, דמוי Swr1, דמוי SSO1653, דמוי Rad54, דמוי Rad5/16 ומרוחק. SMARCAL1, חלבון העניין במחקר זה, שייך לתת-קבוצה רחוקה20. חלבון זה שימש כדי לחקור את אופן הרגולציה שעתוק באמצעות ספקטרוסקופיה CD.
רוב החברים בחלבוני שיפוץ הכרומטין התלויים ב-ATP הוכחו כמי שממקמים מחדש או מפנים נוקלאוזומים או מחליפים וריאנטים של היסטון באופן תלוי ATP21,22. עם זאת, כמה מבני המשפחה לא הוכחו לשפץ נוקלאוזומים, למשל, SMARCAL1. למרות שמחקרים הראו כי SMARCAL1 מקשר עם כרומוזומים פוליטן, ראיות ניסיוניות לגבי יכולתו לשפץ נוקלאוזומים חסר 23. לכן, הו להניח כי SMARCAL1 עשוי לווסת את התמלול על ידי שינוי הקונפורמציה של DNA24. ספקטרוסקופיית תקליטורים סיפקה שיטה קלה ונגישה לאימות השערה זו.
SMARCAL1 הוא חלבון שיפוץ כרומטין תלוי ATP המתפקד בעיקר כהליקאז חישול25,26,27. זה כבר להניח כדי לווסת את התמלול על ידי שיפוץ קונפורמציה DNA24. כדי לבדוק השערה זו, התפקיד של SMARCAL1 בוויסות שעתוק גנים במהלך נזק DNA המושרה doxorubicin נחקר. במחקרים אלה, SMARCAL1 שימש לניתוח ויוו ו ADAAD עבור במבחנה assays28,29. מחקרים קודמים הראו כי ADAAD יכול לזהות DNA באופן תלוי מבנה אך רצף עצמאי30,31. החלבון נקשר בצורה אופטימלית למולקולות דנ"א בעלות גדיל כפול לאזורי מעבר חד-גדיליים, בדומה לדנ"א של לולאת גזע, והידרוליזים ATP 30,31.
ניסויי In vivo הראו כי SMARCAL1 מווסת את הביטוי של MYC, DROSHA, DGCR8 ו- DICER על ידי איגוד לאזורי האמרגן28,29. אזור האינטראקציה זוהה על ידי ניסויי ChIP28,29. טכניקת ChIP משמשת לניתוח האינטראקציה של חלבון עם ה- DNA הקוגניט שלו בתוך התא. מטרתו היא לקבוע אם חלבונים ספציפיים, כגון גורמי שעתוק על מקדמים או אתרי כריכת DNA אחרים, קשורים לאזורים גנומיים ספציפיים. החלבון הקשור לדנ"א מקושר לראשונה באמצעות פורמלדהיד. זה ואחריו בידוד של הכרומטין. הכרומטין המבודד מוצמד לשברים של 500 bp על ידי עיכול סוניקציה או נוקלאז, והחלבון הקשור לדנ"א הוא אימונופרציפיטאט באמצעות נוגדנים ספציפיים לחלבון. הקישור הצולב הפוך, והדנ"א מנותח באמצעות תגובת שרשרת פולימראז (PCR) או PCR כמותי בזמן אמת.
תוצאות ChIP הובילו להשערה כי SMARCAL1 אולי מתווך רגולציה תמלול על ידי גרימת שינוי קונפורמציה באזורי האמרגן של גנים אלה. ממפה QGRS ותוכנת Mfold שימשו לזיהוי הפוטנציאל של אזורי מקדם אלה ליצור מבנים משניים28,29. ממפה QGRS משמש לחיזוי G-quadruplexes32, בעוד Mfold33 מנתח את היכולת של רצף ליצור מבנים משניים כגון לולאות גזע.
לאחר ניתוח מבנה משני, ניסויי במבחנה נוספים בוצעו עם רקומביננטי 6X ATPase A דומיין פעיל תלוי DNA (ADAAD), ההומולוג בקר של SMARCAL1, מטוהר Escherichia coli34. בדיקות ATPase בוצעו באמצעות ADAAD כדי לקבוע כי רצפי DNA שזוהו יכול לשמש כמשפיעים28,29. לבסוף, ספקטרוסקופיה CD בוצעה כדי לפקח על השינויים הקונפורמציה המושרה במולקולת ה- DNA על ידי ADAAD28,29.
כדי להוכיח שפעילות ה-ATPase של החלבון חיונית לגרימת שינוי קונפורמציה במולקולת הדנ"א, נוספה חומצה אתילנדיאמין טטראאצטית (EDTA) ל-Chelate Mg+2 או ATPase פעיל תלוי DNA A מעכב תחום Neomycin (ADAADiN), מעכב ספציפי של חלבון SWI/SNF, נוספה 35,36 . טכניקה ספקטרוסקופית זו CD ניתן להשתמש עם כל חלבון מטוהר כי הוכח על ידי ChIP או כל מבחנת רלוונטית אחרת להיקשר לאזור גנומי חזוי של מקדם.
Protocol
1. ריכוז עבודה של רכיבי התגובה
- הכן את ריכוזי העבודה של מאגרים עבור תקליטור ורכיבי תגובה אחרים טריים (ראה טבלה 1) ולשמור אותם ב 4 °C (70 °F) לפני הגדרת התגובות.
הערה: עבור תגובות התקליטור המתוארות במאמר זה, ריכוזי העבודה של רכיבים הם כדלקמן: מאגר נתרן פוספט (pH 7.0) 1 mM, ATP 2 mM, DNA 500 ננומטר, חלבון 1 מיקרומטר, MgCl2 10 mM, EDTA 50 mM, ADAADiN 5 μM.
2. פעילות ATPase
- לפני ספקטרוסקופיית התקליטור, בססו את פעילות ה-ATPase של החלבון בנוכחות מולקולות ה- DNA כדי להבטיח שהחלבון המשמש בספקטרוסקופיית התקליטור פעיל ולזהות את מולקולות ה- DNA היעילות באופן אופטימלי בהפעלת הידרוליזה ATP.
- מדוד את פעילות ATPase של החלבון בנוכחות מולקולות DNA שונות על ידי בדיקת חמצון מצמיד NADH המורכבת משתי התגובות הבאות.
- ערבבו 0.1 מיקרומטר ADAAD, ATP של 2 מ"מ, 10 ננומטר DNA וחיץ REG 1x בלוח 96 בארות לנפח סופי של 250 μL.
הערה: האנזים pyruvate kinase משתמש ADP ו Pi להמיר זרחן לפירובאט, ובכך לחדש ATP. זה מבטיח כי ATP הוא תמיד בריכוז רווי בתגובה. בתגובה השנייה, הפירובאט שנוצר על ידי הפעולה של פירובט קינאז מומר על ידי לקטט דהידרוגנאז לקטט. בתגובה זו, מולקולת NADH אחת מחומצנת ל- NAD+. הצריכה של NADH נמדדת על ידי מדידת ספיגת המולקולה ב 340 ננומטר. - דגירה במשך 30 דקות ב 37 °C (50 °F) באינקובטור.
- מדוד את כמות NAD+ ב- 340 ננומטר באמצעות קורא מיקרו-לוח.
- כדי למדוד את כמות NAD+, השתמש בתוכנה המסופקת יחד עם קורא המיקרו-לוח.
- לחץ על תצודת NADH כדי למדוד את הספיגה ב 340 ננומטר.
- מניחים את הצלחת 96-well על מחזיק הצלחת בכלי. לחץ על לחצן לוח קריאה כדי להקליט את הספיגה.
הערה: הריכוז של NAD+ מחושב באמצעות מקדם הכחדת הטוחנת של NADH כ- 6.3 mM−1 באמצעות eq (1).
A = εcl (1)
כאן, A = ספיגה
ε = מקדם הכחדה טוחנת
c = ריכוז מולרי
l = אורך נתיב אופטי בס"מ
- ערבבו 0.1 מיקרומטר ADAAD, ATP של 2 מ"מ, 10 ננומטר DNA וחיץ REG 1x בלוח 96 בארות לנפח סופי של 250 μL.
3. בחירה והכנה של תקליטורים
- לאסוף ספקטרום תקליטורים בשקיפות גבוהה קוורץ cuvettes. השתמש בקובטות מלבניות או גליליות.
הערה: cuvette קוורץ תקליטור (נפח נומינלי של 0.4 מ"ל, אורך נתיב של 1 מ"מ) שימש עבור כל התגובות המתוארות במאמר זה. - השתמש בתמיסת ניקוי cuvette כדי לנקות את cuvette. הוסף 1% cuvette ניקוי פתרון במים כדי להפוך 400 μL של הפתרון, לשפוך אותו cuvette, ולדגירה אותו ב 37 °C (50 °F) במשך 1 שעה.
- לשטוף את cuvette עם מים כמה פעמים כדי לנקות את cuvette. קח סריקה של המים או חוצץ בקובט כדי לבדוק אם הוא נקי.
הערה: המים או המאגר חייבים לתת קריאה בטווח של 0 עד 1 mdeg.
4. הכנת חלבונים ואוליגונוקלאוטיד DNA
- שמור את נפח החלבון מתחת 50 μL בתגובה כדי למזער את כמויות של רכיבי המאגר כי לפעמים לגרום להיווצרות של פסגות מעורפלות. שמור את החלבון על הקרח לאורך כל הניסוי כדי למנוע כל השפלה.
- השתמש באוליגונוקלאוטידים מטוהרים על ידי עמוד בתגובות.
הערה: בתגובות המתוארות כאן, DNA שימש הן בצורות מקוריות והן בצורות מקוררות חום (מקוררות במהירות (FC) ומצוננות באיטיות (SC)). קירור מהיר מקדם מליטה תוך-עינית בדנ"א, ומניב מבנים משניים יותר. לעומת זאת, קירור איטי מקדם קשר בין-מולקולרי בדנ"א, וכתוצאה מכך פחות מבנים משניים. - לקירור מהיר, מחממים דנ"א במהירות של 94 מעלות צלזיוס למשך 3 דקות על בלוק החימום ומקררים אותו מיד על קרח. לקירור איטי, מחממים דנ"א ב-94 מעלות צלזיוס למשך 3 דקות ומאפשרים לו להתקרר לטמפרטורת החדר בקצב של 1 מעלות צלזיוס לדקה.
5. הגדרת ניסויי בקרה כדי לתעד את הספקטרום הבסיסי
- שמור את נפח התגובה ב 300 μL בכל התגובות. הגדר סך של 5 תגובות בסיסיות בצינורות צנטריפוגות 1.5 מ"ל, אחד אחד, כדלקמן: i) חוצץ + מים; ii) חוצץ + MgCl2 + ATP + מים; iii) חוצץ + MgCl2 + ATP + חלבון + מים; 4) iii + EDTA או ADAADiN; v) חוצץ + חלבון + מים.
6. הגדרת הניסויים להקלדת ספקטרום תקליטורים
- הגדר סך של 5 תגובות, אחד אחד, ב 1.5 מ"ל צינורות צנטריפוגות כדלקמן: i) חוצץ + DNA + מים; ii) חוצץ + DNA + MGCl2 + ATP + מים; iii) חוצץ + DNA + MGCl2 + ATP + חלבון + מים; 4) iii + EDTA או ADAADiN; v) חוצץ + DNA + חלבון + מים.
7. סריקת הקלטה
- תדליק את הגז ותדליק את ספקטרומטר התקליטורים.
- להפעיל את המנורה לאחר 10-15 דקות. להפעיל את אמבט המים ולהגדיר את טמפרטורת המחזיק על 37 °C (50 °F).
- פתח את תוכנת ספקטרום התקליטורים .
- הגדר את הטמפרטורה ל 37 °C (50 °F).
- הגדר את טווח אורך הגל על 180 - 300 ננומטר.
- הגדר את הזמן לנקודה ל - 0.5 s.
- הגדר את מספר הסריקה ל - 5.
- לחץ על Pro-Data Viewer, ליצור קובץ חדש, ולשנות את שמו עם פרטים על הניסוי והתאריך.
- שמור את כל רכיבי התגובה על קרח כדי למנוע כל השפלה. הפוך את קווי הבסיס ואת התגובות, אחד אחד, צינורות צנטריפוגות ולערבב אותם על ידי pipetting. מעבירים את תערובת התגובה לקובט בזהירות, ומבטיחים שאין בועות אוויר.
- אם מבצעים ניסוי בזמן, לדגור על התגובות ב 37 °C (37 °F) לזמן הנדרש ולקחת את הסריקה. הוסיפו את EDTA למאגר המכיל את הדנ"א, ה-ATP, Mg+2 והחלבון כדי לעצור הידרוליזה של ATP.
- להגדיל את הריכוז של EDTA ואת זמן הדגירה שלה כדי לעכב את פעילות ATPase לחלוטין.
- הפחת את קווי הבסיס מהתגובות המתאימות בתוכנה (למשל, הפחת את התגובה 1 מקו הבסיס 1). החלק את הנתונים בתוכנת ספקטרום התקליטורים או בתוכנת התוויית הנתונים. התווה את הנתונים בתוכנת התוויית הנתונים.
הערה: חיסור קווי הבסיס מהתגובות המתאימות ייתן לספקטרום התקליטור נטו של DNA בלבד.
8. ניתוח נתונים ופרשנות
- השתמש בנוסחה שניתנה על-ידי eq (2) כדי להמיר את הערכים המתקבלים במילידגריות כך שמשמעותם אליפטיות שאריות.
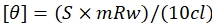 (2)
(2)
כאן, S הוא אות התקליטור במילידגרי, c הוא ריכוז ה- DNA ב- mg / mL, mRw הוא מסת השאריות הממוצעת, ואני אורך הנתיב בס"מ. - התווה גרף נגד אורך גל ואליפטיות שאריות ממוצעת באמצעות תוכנת התוויית הנתונים וניתח את הפסגות.
- כדי להתוות את הגרף, בחרו את אליפטיות השאריות הממוצעת בציר ה-Y ואורך הגל בציר ה-X והתוו גרף קו ישר.
הערה: גרף זה יספק את המאפיינים פסגות של צורות שונות של DNA. ניתן לזהות את צורות הדנ"א המתאימות לפסגות באמצעות ספרות קיימת6.
תוצאות
ADAAD מייצב מבנה דמוי לולאת גזע על מקדם MYC
ראיות ניסיוניות קודמות הראו כי SMARCAL1 הוא רגולטור שלילי של MYC29. ניתוח של אזור המקדם הארוך של 159 bp של הגן MYC על ידי ממפה QGRS הראה כי לגדיל הקדמי יש פוטנציאל ליצור G-quadruplex (טבלה 2). ספל הראה כי ש?...
Discussion
מטרת מאמר זה היא להציג את טכניקת ספקטרוסקופיית התקליטורים כגישה לחקור את השינויים הקונפורמציה המתרחשים ב- DNA בנוכחות חלבוני שיפוץ כרומטין תלויי ATP ולקשר את השינויים הקונפורמיים האלה לביטוי גנים. ספקטרוסקופיה של תקליטור מספקת שיטה מהירה ונגישה לחקר השינויים הקונפורמציה בדנ"א.
Disclosures
למחברים אין ניגוד אינטרסים להצהיר.
Acknowledgements
המחברים רוצים להודות למתקן מחקר מכשור מתקדם, JNU, על ספקטרופוטומטר התקליטורים. V.J. ו- A.D. נתמכו על ידי מלגה מ CSIR.
Materials
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Fisher scientific | O3446I-100 | |
| Adenosine 5′-triphosphate disodium salt hydrate | Sigmaaldrich | A2383 | |
| CD Quartz Cuvette | STARNA | 21-Q-1 | |
| Chirascan V100 CD spectrometer | Applied Photophysics | Not available | |
| EDTA Disodium Salt Dihydrate | SRL | 43272 | |
| Glutathione Sepharose 4B | GE Healthcare | 17-0756-01 | Glutathione affinity chromatography |
| Hellmanex III cleaning solution | Hellma | 9-307-011-4-507 | |
| L-Lactic Dehydrogenase | Sigmaaldrich | L2625 | |
| Magnesium Acetate Tetrahydrate | Fisher scientific | BP215-500 | |
| Magnesium Chloride Hexahydrate | Fisher scientific | M33-500 | |
| NADH disodium salt | Sigmaaldrich | 10107735001 | |
| Phosphoenolpyruvate Monocyclohexylammonium Salt | SRL | 40083 | |
| Potassium Acetate | Fisher scientific | P178-3 | |
| Pyruvate Kinase | Sigmaaldrich | P1506 | |
| Sodium Phosphate Dibasic Anhydrous | Fisher scientific | S374-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher scientific | S369-500 | |
| Synergy HT microplate reader | BioTek | Not available | |
| Tris Base | Fisher scientific | BP152-500 |
References
- Woody, R. W. Circular dichroism. Methods in Enzymology. 246, 34-71 (1995).
- Kelly, S., Price, N. The Use of Circular Dichroism in the Investigation of Protein Structure and Function. Current Protein & Peptide Science. 1 (4), 349-384 (2000).
- Rodger, A., Marshall, D. Beginners guide to circular dichroism. The Biochemist. 43 (2), 58-64 (2021).
- Greenfield, N. J. Using circular dichroism spectra to estimate protein secondary structure. Nature Protocols. 1 (6), 2876-2890 (2006).
- Kypr, J., Kejnovská, I., Bednářová, K., Vorlíčková, M. Circular Dichroism Spectroscopy of Nucleic Acids. Comprehensive Chiroptical Spectroscopy. , 575-586 (2012).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Miyahara, T., Nakatsuji, H., Sugiyama, H. Helical Structure and Circular Dichroism Spectra of DNA: A Theoretical Study. The Journal of Physical Chemistry A. 117 (1), 42-55 (2013).
- Khomyakova, E. B. Parallel intramolecular DNA triple helix with G and T bases in the third strand stabilized by Zn2+ ions. Nucleic Acids Research. 28 (18), 3511-3516 (2000).
- Kypr, J., et al. The unusual X-form DNA in oligodeoxynucleotides: dependence of stability on the base sequence and length. Journal of Biomolecular Structure & Dynamics. 13 (6), 999-1006 (1996).
- Zohoorian-Abootorabi, T., Sanee, H., Iranfar, H., Saberi, M. R., Chamani, J. Separate and simultaneous binding effects through a non-cooperative behavior between cyclophosphamide hydrochloride and fluoxymesterone upon interaction with human serum albumin: multi-spectroscopic and molecular modeling approaches. Spectrochimica Acta. Part A, Molecular and Biomolecular Spectroscopy. 88, 177-191 (2012).
- Sharifi-Rad, A., Mehrzad, J., Darroudi, M., Saberi, M. R., Chamani, J. Oil-in-water nanoemulsions comprising Berberine in olive oil: biological activities, binding mechanisms to human serum albumin or holo-transferrin and QMMD simulations. Journal of Biomolecular Structure & Dynamics. 39 (3), 1029-1043 (2021).
- Mokaberi, P., Babayan-Mashhadi, F., Amiri Tehrani Zadeh, Z., Saberi, M. R., Chamani, J. Analysis of the interaction behavior between Nano-Curcumin and two human serum proteins: combining spectroscopy and molecular stimulation to understand protein-protein interaction. Journal of Biomolecular Structure & Dynamics. 39 (9), 3358-3377 (2021).
- Danesh, N., et al. Determining the binding site and binding affinity of estradiol to human serum albumin and holo-transferrin: fluorescence spectroscopic, isothermal titration calorimetry and molecular modeling approaches. Journal of Biomolecular Structure & Dynamics. 36 (7), 1747-1763 (2018).
- Sadeghzadeh, F., et al. Characterizing the binding of angiotensin converting enzyme I inhibitory peptide to human hemoglobin: influence of electromagnetic fields. Protein and Peptide Letters. 27 (10), 1007-1021 (2020).
- Chamani, J., et al. Cooperative alpha-helix formation of beta-lactoglobulin induced by sodium n-alkyl sulfates. Journal of Colloid and Interface Science. 293 (1), 52-60 (2006).
- Dareini, M., et al. A novel view of the separate and simultaneous binding effects of docetaxel and anastrozole with calf thymus DNA: Experimental and in silico approaches. Spectrochimica Acta. Part A, Molecular and Biomolecular Spectroscopy. 228, 117528 (2020).
- Dehghani Sani, F., et al. Changes in binding affinity between ofloxacin and calf thymus DNA in the presence of histone H1: Spectroscopic and molecular modeling investigations. Journal of Luminescence. 203, 599-608 (2018).
- Hargreaves, D. C., Crabtree, G. R. ATP-dependent chromatin remodeling: genetics, genomics and mechanisms. Cell Research. 21 (3), 396-420 (2011).
- Morettini, S., Podhraski, V., Lusser, A. ATP-dependent chromatin remodeling enzymes and their various roles in cell cycle control. Frontiers in Bioscience: A Journal and Virtual Library. 13, 5522-5532 (2008).
- Flaus, A., Martin, D. M. A., Barton, G. J., Owen-Hughes, T. Identification of multiple distinct Snf2 subfamilies with conserved structural motifs. Nucleic Acids Research. 34 (10), 2887-2905 (2006).
- Flaus, A., Owen-Hughes, T. Mechanisms for ATP-dependent chromatin remodelling: the means to the end. The FEBS Journal. 278 (19), 3579-3595 (2011).
- Mizuguchi, G., et al. ATP-driven exchange of histone H2AZ variant catalyzed by SWR1 chromatin remodeling complex. Science. 303 (5656), 343-348 (2004).
- Elizondo, L. I., et al. Schimke immuno-osseous dysplasia: SMARCAL1 loss-of-function and phenotypic correlation. Journal of Medical Genetics. 46 (1), 49-59 (2009).
- Baradaran-Heravi, A., et al. SMARCAL1 deficiency predisposes to non-Hodgkin lymphoma and hypersensitivity to genotoxic agents in vivo. American Journal of Medical Genetics. Part A. 158 (9), 2204-2213 (2012).
- Bansal, R., et al. SMARCAL1, the annealing helicase and the transcriptional co-regulator. IUBMB life. 72 (10), 2080-2096 (2020).
- Yusufzai, T., Kadonaga, J. T. HARP is an ATP-driven annealing helicase. Science. 322 (5902), 748-750 (2008).
- Yusufzai, T., Kong, X., Yokomori, K., Kadonaga, J. T. The annealing helicase HARP is recruited to DNA repair sites via an interaction with RPA. Genes & Development. 23 (20), 2400-2404 (2009).
- Patne, K., et al. BRG1 and SMARCAL1 transcriptionally co-regulate DROSHA, DGCR8 and DICER in response to doxorubicin-induced DNA damage. Biochimica et Biophysica Acta. 1860 (9), 936-951 (2017).
- Sharma, T., Bansal, R., Haokip, D. T., Goel, I., Muthuswami, R. SMARCAL1 negatively regulates c-Myc transcription by altering the conformation of the promoter region. Scientific Reports. 5, 17910 (2015).
- Muthuswami, R., Truman, P. A., Mesner, L. D., Hockensmith, J. W. A eukaryotic SWI2/SNF2 domain, an exquisite detector of double-stranded to single-stranded DNA transition elements. The Journal of Biological Chemistry. 275 (11), 7648-7655 (2000).
- Nongkhlaw, M., Dutta, P., Hockensmith, J. W., Komath, S. S., Muthuswami, R. Elucidating the mechanism of DNA-dependent ATP hydrolysis mediated by DNA-dependent ATPase A, a member of the SWI2/SNF2 protein family. Nucleic Acids Research. 37 (10), 3332-3341 (2009).
- Kikin, O., D'Antonio, L., Bagga, P. S. QGRS Mapper: a web-based server for predicting G-quadruplexes in nucleotide sequences. Nucleic Acids Research. 34, 676-682 (2006).
- Zuker, M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Research. 31 (13), 3406-3415 (2003).
- Gupta, M., et al. Ligand-induced conformation changes drive ATP hydrolysis and function in SMARCAL1. The FEBS Journal. 282 (19), 3841-3859 (2015).
- Dutta, P., et al. Global epigenetic changes induced by SWI2/SNF2 inhibitors characterize neomycin-resistant mammalian cells. PloS One. 7 (11), 49822 (2012).
- Muthuswami, R., et al. Phosphoaminoglycosides inhibit SWI2/SNF2 family DNA-dependent molecular motor domains. Biochemistry. 39 (15), 4358-4365 (2000).
- Gondeau, C. Circular dichroism and UV melting studies on formation of an intramolecular triplex containing parallel T*A:T and G*G:C triplets: netropsin complexation with the triplex. Nucleic Acids Research. 26 (21), 4996-5003 (1998).
- Nongkhlaw, M., Gupta, M., Komath, S. S., Muthuswami, R. Motifs Q and I are required for ATP hydrolysis but not for ATP binding in SWI2/SNF2 proteins. Biochemistry. 51 (18), 3711-3722 (2012).
- Luchnik, A. N. DNA conformational transitions induced by supercoiling control transcription in chromatin. Gene Regulation and Systems Biology. 8, 89-96 (2014).
- Siddiqui-Jain, A., Grand, C. L., Bearss, D. J., Hurley, L. H. Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription. Proceedings of the National Academy of Sciences of the United States of America. 99 (18), 11593-11598 (2002).
- Uribe, D. J., Guo, K., Shin, Y. -. J., Sun, D. Heterogeneous nuclear ribonucleoprotein K and nucleolin as transcriptional activators of the vascular endothelial growth factor promoter through interaction with secondary DNA structures. Biochemistry. 50 (18), 3796-3806 (2011).
- Young, S. L., Krawczyk, S. H., Matteucci, M. D., Toole, J. J. Triple helix formation inhibits transcription elongation in vitro. Proceedings of the National Academy of Sciences of the United States of America. 88 (22), 10023-10026 (1991).
- Dürr, H., Flaus, A., Owen-Hughes, T., Hopfner, K. -. P. Snf2 family ATPases and DExx box helicases: differences and unifying concepts from high-resolution crystal structures. Nucleic Acids Research. 34 (15), 4160-4167 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved