È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Spettroscopia CD per studiare le interazioni DNA-proteina
In questo articolo
Riepilogo
L'interazione di un rimodellatore di cromatina ATP-dipendente con un ligando del DNA è descritta utilizzando la spettroscopia CD. I cambiamenti conformazionali indotti su un promotore genico analizzato dai picchi generati possono essere utilizzati per comprendere il meccanismo della regolazione trascrizionale.
Abstract
La spettroscopia del dicroismo circolare (CD) è un metodo semplice e conveniente per studiare la struttura secondaria e le interazioni delle biomolecole. I recenti progressi nella spettroscopia CD hanno permesso lo studio delle interazioni DNA-proteina e delle dinamiche conformazionali del DNA in diversi microambienti in dettaglio per una migliore comprensione della regolazione trascrizionale in vivo. L'area intorno a una potenziale zona di trascrizione deve essere srotolata affinché si verifichi la trascrizione. Questo è un processo complesso che richiede il coordinamento delle modificazioni istoniche, il legame del fattore di trascrizione al DNA e altre attività di rimodellamento della cromatina. Utilizzando la spettroscopia CD, è possibile studiare i cambiamenti conformazionali nella regione del promotore causati da proteine regolatrici, come i rimodellatori di cromatina ATP-dipendenti, per promuovere la trascrizione. Anche i cambiamenti conformazionali che si verificano nella proteina possono essere monitorati. Inoltre, le domande riguardanti l'affinità della proteina verso il suo DNA bersaglio e la specificità della sequenza possono essere affrontate incorporando mutazioni nel DNA bersaglio. In breve, la comprensione unica di questo metodo sensibile e poco costoso può prevedere i cambiamenti nella dinamica della cromatina, migliorando così la comprensione della regolazione trascrizionale.
Introduzione
Il dicroismo circolare (CD) è una tecnica spettroscopica che si basa sulla chiralità intrinseca delle macromolecole biologiche che porta all'assorbimento differenziale della luce polarizzata circolarmente destrorsa e mancina. Questo assorbimento differenziale è noto come dicroismo circolare. La tecnica, quindi, può essere utilizzata per delineare la conformazione di macromolecole biologiche, come proteine e DNA, che contengono entrambi centri chirali1,2.
Le onde elettromagnetiche contengono componenti sia elettrici che magnetici. Sia il campo elettrico che quello magnetico oscillano perpendicolarmente alla direzione di propagazione dell'onda. Nel caso della luce non polarizzata, questi campi oscillano in molte direzioni. Quando la luce è polarizzata circolarmente, si ottengono due campi elettromagnetici a 90° di differenza di fase l'uno dall'altro. Le molecole chirali mostrano una rotazione ottica circolare (birifrangenza) tale da assorbire la luce polarizzata circolarmente destrorsa e la luce polarizzata circolarmente mancina in misura diversa3. Il campo elettrico risultante sarà tracciato come un'ellisse, una funzione della lunghezza d'onda. Lo spettro CD è, quindi, registrato come ellitticità (q), e i dati sono presentati come Ellitticità media residuo in funzione della lunghezza d'onda.
Nel caso delle proteine, la Cα degli amminoacidi (eccetto la glicina) è chirale, e questo viene sfruttato dalla spettroscopia CD per determinare la struttura secondaria di questa macromolecola4. Gli spettri CD delle molecole proteiche sono tipicamente registrati nell'intervallo Far UV. α-elicoidali hanno due bande negative a 222 nm e 208 nm e un picco positivo a 193 nm4. Le proteine con struttura secondaria anti-parallelo β-foglio mostrano un picco negativo a 218 nm e un picco positivo a 195 nm4. Le proteine con strutture disordinate mostrano una bassa ellitticità vicino a 210 nm e un picco negativo a 195 nm4. Pertanto, i picchi / bande ben definiti per diverse strutture secondarie rendono il CD uno strumento conveniente per chiarire i cambiamenti conformazionali che si verificano nella struttura secondaria delle proteine durante la denaturazione e il legame del ligando.
Gli acidi nucleici hanno tre fonti di chiralità: la molecola di zucchero, l'elicità della struttura secondaria e l'ordinamento terziario a lungo raggio del DNA nell'ambiente5,6. Gli spettri CD degli acidi nucleici sono tipicamente registrati nell'intervallo da 190 a 300 nm5,6. Ogni conformazione del DNA, proprio come le proteine, fornisce uno spettro caratteristico, anche se i picchi/bande possono variare di alcuni gradi a causa delle condizioni del solvente e delle differenze nelle sequenze di DNA7. Il B-DNA, la forma più comune, è caratterizzato da un picco positivo intorno a 260-280 nm e un picco negativo intorno a 245 nm6. I picchi/bande del DNA di forma B sono generalmente piccoli perché le coppie di basi sono perpendicolari alla doppia elica, conferendo debole chiralità alla molecola. A-DNA dà un picco positivo dominante a 260 nm e un picco negativo intorno a 210 nm6. Z-DNA, l'elica mancina, dà una banda negativa a 290 nm e un picco positivo intorno a 260 nm6. Questo DNA dà anche un picco estremamente negativo a 205 nm6.
Oltre a queste conformazioni, il DNA può anche formare triplex, quadruplex e forcine, che possono essere distinte dalla spettroscopia CD. Il parallelo G-quadruplex dà una banda positiva dominante a 260 nm, mentre l'anti-parallelo G-quadruplex dà una banda negativa a 260 nm e un picco positivo a 290 nm, rendendo facile distinguere tra le due forme di strutture quadruplex6. I triplex non danno uno spettro caratteristico8. Ad esempio, gli spettri di un DNA lungo 36 nucleotidi con il potenziale di formare una tripla elica intramolecolare contenente coppie di basi G.G.C e T.A.T in presenza di Na+ mostrano una forte banda negativa a 240 nm e un ampio picco positivo. L'ampio picco positivo mostra contributi a 266, 273 e 286 nm. Lo stesso oligonucleotide in presenza di Na+ e Zn+ mostra quattro bande negative (213, 238, 266 e 282 nm) e un picco positivo a 258 nm. Pertanto, gli spettri del DNA triplex possono variare a seconda delle condizioni del sale8.
Oltre a queste conformazioni, gli spettri CD hanno permesso l'identificazione di un'altra forma di DNA chiamata X-DNA. X-DNA si forma quando la sequenza di DNA contiene residui alternativi di adenina e timina. Gli spettri CD di X-DNA contengono due picchi negativi a 250 e 280 nm. Sono disponibili pochissime informazioni su X-DNA, anche se è stato ipotizzato che funzioni come un pozzo per il supercoiling positivo6,9. I cambiamenti negli spettri CD possono anche rivelare dettagli sulle interazioni ligando-proteina e, quindi, sono stati aggiunti all'arsenale di metodi molecolari per rilevare le interazioni farmaco-proteina10,11,12,13,14. Gli spettri CD sono stati utilizzati anche per monitorare i cambiamenti nella struttura secondaria delle proteine durante il processo di ripiegamento15. Allo stesso modo, gli spettri CD possono essere utilizzati anche per sondare le interazioni ligando-DNA16,17.
La spettroscopia CD, quindi, è un metodo facile ed economico per distinguere tra le diverse forme di conformazione del DNA, a condizione che vi sia accesso ad apparecchiature e software non così economici. Il metodo è estremamente sensibile e veloce. Richiede solo una piccola quantità di DNA, dandogli un vantaggio rispetto alla tecnica alternativa della spettroscopia di risonanza magnetica nucleare (NMR). Anche le titolazioni con ligandi e substrati sono facili da eseguire. Il vincolo principale è che il DNA dovrebbe essere altamente puro. Si consiglia di utilizzare DNA purificato con elettroforesi su gel di poliacrilammide (PAGE).
Le informazioni ottenute dagli spettri CD sono state utilizzate principalmente per dedurre le caratteristiche strutturali delle proteine e per identificare distinti conformisti del DNA. In questo studio, gli spettri CD sono stati utilizzati per integrare i risultati ottenuti da un esperimento in vivo di immunoprecipitazione della cromatina (ChIP) per delineare se la proteina di interesse / fattore di trascrizione previsto può determinare un cambiamento conformazionale nella regione promotore dei suoi geni effettori. Questa collaborazione aiuta nel progresso delle tradizionali tecniche spettroscopiche CD prevedendo il meccanismo di regolazione della trascrizione da parte del fattore di trascrizione previsto su e intorno al sito di inizio della trascrizione (TSS) di un promotore.
Il rimodellamento della cromatina è un meccanismo ben definito noto per regolare i processi metabolici del DNA rendendo la cromatina strettamente imballata accessibile a vari fattori regolatori come fattori di trascrizione, componenti della replicazione del DNA o proteine di riparazione del danno. I rimodellatori di cromatina ATP-dipendenti, noti anche come famiglia di proteine SWI/SNF, sono proteine rimodellanti chiave presenti nelle cellule eucariotiche18,19. Il clustering filogenetico ha classificato la famiglia di proteine SWI/SNF in 6 sottogruppi20: Snf2-like, Swr1-like, SSO1653-like, Rad54-like, Rad5/16-like e distant. SMARCAL1, la proteina di interesse in questo studio, appartiene al sottogruppo distante20. Questa proteina è stata utilizzata per studiare la sua modalità di regolazione trascrizionale utilizzando la spettroscopia CD.
La maggior parte dei membri delle proteine rimodellanti della cromatina ATP-dipendenti hanno dimostrato di riposizionare o sfrattare i nucleosomi o mediare lo scambio di varianti istoniche in modo ATP-dipendente21,22. Tuttavia, alcuni membri di questa famiglia non hanno dimostrato di rimodellare i nucleosomi, ad esempio SMARCAL1. Anche se gli studi hanno dimostrato che SMARCAL1 si associa ai cromosomi politenici, mancano prove sperimentali riguardanti la sua capacità di rimodellare i nucleosomi23. Pertanto, è stato postulato che SMARCAL1 può regolare la trascrizione alterando la conformazione del DNA24. La spettroscopia CD ha fornito un metodo facile e accessibile per convalidare questa ipotesi.
SMARCAL1 è una proteina rimodellante della cromatina ATP-dipendente che funziona principalmente come elicasi ricottura25,26,27. È stato postulato per modulare la trascrizione rimodellando la conformazione del DNA24. Per verificare questa ipotesi, è stato studiato il ruolo di SMARCAL1 nella regolazione della trascrizione genica durante il danno al DNA indotto dalla doxorubicina. In questi studi, SMARCAL1 è stato utilizzato per l'analisi in vivo e ADAAD per i saggi in vitro28,29. Studi precedenti hanno dimostrato che l'ADAAD è in grado di riconoscere il DNA in modo struttura-dipendente ma indipendente dalla sequenza30,31. La proteina si lega in modo ottimale alle molecole di DNA che possiedono regioni di transizione a doppio filamento a singolo filamento, simili al DNA ad anello dello stelo, e idrolizza ATP 30,31.
Esperimenti in vivo hanno dimostrato che SMARCAL1 regola l'espressione di MYC, DROSHA, DGCR8 e DICER legandosi alle regioni del promotore28,29. La regione di interazione è stata identificata dagli esperimenti ChIP28,29. La tecnica ChIP viene utilizzata per analizzare l'interazione di una proteina con il suo DNA affine all'interno della cellula. Il suo obiettivo è determinare se proteine specifiche, come i fattori di trascrizione sui promotori o altri siti di legame del DNA, sono legate a specifiche aree genomiche. La proteina legata al DNA viene prima reticolata usando la formaldeide. Questo è seguito dall'isolamento della cromatina. La cromatina isolata viene tranciata a frammenti di 500 bp mediante sonicazione o digestione della nucleasi e la proteina legata al DNA viene immunoprecipita utilizzando anticorpi specifici per la proteina. Il cross-linking viene invertito e il DNA viene analizzato utilizzando la reazione a catena della polimerasi (PCR) o la PCR quantitativa in tempo reale.
I risultati di ChIP hanno portato all'ipotesi che SMARCAL1 possa mediare la regolazione trascrizionale inducendo un cambiamento conformazionale nelle regioni promotrici di questi geni. Il mappatore QGRS e il software Mfold sono stati utilizzati per identificare il potenziale di queste regioni promotrici per formare strutture secondarie28,29. Il mappatore QGRS viene utilizzato per prevedere G-quadruplexes32, mentre Mfold33 analizza la capacità di una sequenza di formare strutture secondarie come gli stem-loop.
Dopo l'analisi della struttura secondaria, sono stati eseguiti ulteriori esperimenti in vitro con il recombinant 6X His-tagged Active DNA-dependent ATPase A Domain (ADAAD), l'omologo bovino di SMARCAL1, purificato da Escherichia coli34. I saggi ATPasi sono stati eseguiti utilizzando ADAAD per stabilire che le sequenze di DNA identificate potrebbero agire come effettori28,29. Infine, è stata eseguita la spettroscopia CD per monitorare i cambiamenti conformazionali indotti nella molecola di DNA da ADAAD28,29.
Per dimostrare che l'attività ATPasi della proteina era essenziale per indurre un cambiamento conformazionale nella molecola del DNA, è stato aggiunto acido tetraacetico etilendiammina (EDTA) al chelato Mg+2 o ATPasi attiva DNA-dipendente A Domain Inhibitor Neomycin (ADAADiN), un inibitore specifico della proteina SWI/SNF35,36 . Questa tecnica spettroscopica CD può essere utilizzata con qualsiasi proteina purificata che è stata dimostrata da ChIP o qualsiasi altro test pertinente per legarsi a una regione genomica prevista di un promotore.
Protocollo
1. Concentrazione di lavoro dei componenti di reazione
- Preparare nuovamente le concentrazioni di lavoro dei tamponi per CD e altri componenti di reazione (vedere Tabella 1) e mantenerle a 4 °C prima di impostare le reazioni.
NOTA: Per le reazioni CD descritte in questo articolo, le concentrazioni di lavoro dei componenti sono le seguenti: tampone fosfato di sodio (pH 7,0) 1 mM, ATP 2 mM, DNA 500 nM, proteina 1 μM, MgCl2 10 mM, EDTA 50 mM, ADAADiN 5 μM.
2. Attività atpasi
- Prima della spettroscopia CD, stabilire l'attività ATPasi della proteina in presenza delle molecole di DNA per garantire che la proteina utilizzata nella spettroscopia CD sia attiva e per identificare le molecole di DNA che sono ottimamente efficaci nel provocare l'idrolisi dell'ATP.
- Misurare l'attività ATPasi della proteina in presenza di diverse molecole di DNA mediante un saggio di ossidazione accoppiato a NADH costituito dalle seguenti due reazioni.
- Mescolare 0,1 μM ADAAD, 2 mM ATP, 10 nM DNA e 1x buffer REG in una piastra a 96 pozzetti fino a un volume finale di 250 μL.
NOTA: L'enzima piruvato chinasi utilizza l'ADP e il Pi per convertire il fosfoenolpiruvato in piruvato, rigenerando così l'ATP. Ciò garantisce che l'ATP sia sempre in concentrazione di saturazione nella reazione. Nella seconda reazione, il piruvato formato dall'azione della piruvato chinasi viene convertito dalla lattato deidrogenasi in lattato. In questa reazione, una molecola di NADH viene ossidata a NAD+. Il consumo di NADH viene misurato misurando l'assorbanza della molecola a 340 nm. - Incubare per 30 minuti a 37 °C in un'incubatrice.
- Misurare la quantità di NAD+ a 340 nm utilizzando un lettore di micropiastre.
- Per misurare la quantità di NAD+, utilizzare il software fornito insieme al lettore di micropiastre.
- Fare clic sul test NADH per misurare l'assorbanza a 340 nm.
- Posizionare la piastra a 96 pozzetti sul portatarga dello strumento. Fare clic sul pulsante Leggi piastra per registrare l'assorbanza.
NOTA: La concentrazione di NAD+ viene calcolata utilizzando il coefficiente di estinzione molare di NADH come 6,3 mM−1 utilizzando eq (1).
A = εcl (1)
Qui, A = Assorbanza
ε = Coefficiente di estinzione molare
c = Concentrazione molare
l = Lunghezza percorso ottico in cm
- Mescolare 0,1 μM ADAAD, 2 mM ATP, 10 nM DNA e 1x buffer REG in una piastra a 96 pozzetti fino a un volume finale di 250 μL.
3. Scelta e preparazione delle cuvette CD
- Raccogli spettri CD in cuvette di quarzo ad alta trasparenza. Utilizzare cuvette rettangolari o cilindriche.
NOTA: Una cuvetta di quarzo CD (volume nominale di 0,4 mL, lunghezza del percorso di 1 mm) è stata utilizzata per tutte le reazioni descritte in questo articolo. - Utilizzare una soluzione detergente per cuvette per pulire la cuvetta. Aggiungere la soluzione detergente per cuvette all'1% in acqua per ottenere 400 μL della soluzione, versarla nella cuvetta e incubarla a 37 °C per 1 ora.
- Lavare la cuvetta con acqua più volte per pulire la cuvetta. Fai una scansione dell'acqua o del tampone nella cuvetta per verificare se è pulita.
NOTA: l'acqua o il tampone devono fornire una lettura nell'intervallo da 0 a 1 mdeg.
4. Preparazione di proteine e DNA oligonucleotido
- Mantenere il volume della proteina al di sotto di 50 μL nella reazione per ridurre al minimo le quantità dei componenti tampone che a volte causano la formazione di picchi ambigui. Mantieni la proteina sul ghiaccio durante l'esperimento per evitare qualsiasi degradazione.
- Utilizzare oligonucleotidi di DNA purificati da PAGE nelle reazioni.
NOTA: Nelle reazioni qui descritte, il DNA è stato utilizzato sia in forme native che raffreddate a caldo (raffreddato rapidamente (FC) e raffreddato lentamente (SC)). Il raffreddamento rapido promuove il legame intramolecolare nel DNA, producendo più strutture secondarie. Al contrario, il raffreddamento lento promuove il legame intermolecolare nel DNA, con conseguente minor numero di strutture secondarie. - Per un raffreddamento rapido, riscaldare il DNA a 94 °C per 3 minuti sul blocco riscaldante e raffreddarlo immediatamente sul ghiaccio. Per il raffreddamento lento, riscaldare il DNA a 94 °C per 3 minuti e lasciarlo raffreddare a temperatura ambiente ad una velocità di 1 °C al minuto.
5. Impostazione di esperimenti di controllo per registrare gli spettri di base
- Mantenere il volume di reazione a 300 μL in tutte le reazioni. Impostare un totale di 5 reazioni basali in tubi centrifuga da 1,5 ml, uno per uno, come segue: i) Tampone + Acqua; ii) Tampone + MgCl2 + ATP + Acqua; iii) Tampone + MgCl2 + ATP + Proteine + Acqua; iv) iii + EDTA o ADAADiN; v) Tampone + Proteine + Acqua.
6. Impostazione degli esperimenti per registrare spettri di CD
- Impostare un totale di 5 reazioni, una per una, in tubi centrifuga da 1,5 ml come segue: i) Tampone + DNA + Acqua; ii) Tampone + DNA + MgCl2 + ATP + Acqua; iii) Tampone + DNA + MgCl2 + ATP + Proteine + Acqua; iv) iii + EDTA o ADAADiN; v) Tampone + DNA + Proteine + Acqua.
7. Scansione della registrazione
- Accendere il gas e accendere lo spettrometro CD.
- Accendere la lampada dopo 10-15 min. Accendere il bagno d'acqua e impostare la temperatura del supporto a 37 °C.
- Aprire il software dello spettro CD .
- Impostare la temperatura a 37 °C.
- Impostare l'intervallo di lunghezze d'onda su 180 - 300 nm.
- Impostare il tempo per punto su 0,5 s.
- Impostare il numero di scansione su 5.
- Fai clic su Pro-Data Viewer, crea un nuovo file e rinominalo con i dettagli sull'esperimento e sulla data.
- Mantenere tutti i componenti di reazione sul ghiaccio per evitare qualsiasi degrado. Effettuare le linee di base e le reazioni, una per una, in tubi di centrifuga e mescolarle mediante pipettaggio. Trasferire accuratamente la miscela di reazione sulla cuvetta, assicurandosi che non vi siano bolle d'aria.
- Se si esegue un esperimento a tempo, incubare le reazioni a 37 °C per il tempo richiesto e fare la scansione. Aggiungere EDTA al tampone contenente DNA, ATP, Mg + 2 e proteine per fermare l'idrolisi dell'ATP.
- Aumentare la concentrazione di EDTA e il suo tempo di incubazione per inibire completamente l'attività dell'ATPasi.
- Sottrarre le linee di base dalle reazioni corrispondenti nel software (ad esempio, sottrarre la reazione 1 dalla linea di base 1). Smussare i dati nel software dello spettro CD o nel software di stampa dei dati. Traccia i dati nel software di stampa dei dati.
NOTA: Sottraendo le linee di base dalle reazioni corrispondenti si otterranno gli spettri NETTI CD del solo DNA.
8. Analisi e interpretazione dei dati
- Utilizzare la formula data da eq (2) per convertire i valori ottenuti in millidegrees in ellitticità dei residui medi.
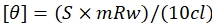 (2)
(2)
Qui, S è il segnale CD nei millidegrei, c è la concentrazione di DNA in mg / mL, mRw è la massa media dei residui e l è la lunghezza del percorso in cm. - Traccia un grafico contro la lunghezza d'onda e l'ellitticità media dei residui utilizzando il software di tracciamento dei dati e analizza i picchi.
- Per tracciare il grafico, selezionare l'ellitticità media dei residui sull'asse Y e la lunghezza d'onda sull'asse X e tracciare un grafico a linee rette.
NOTA: Questo grafico fornirà i picchi caratteristici delle diverse forme di DNA. Le forme di DNA corrispondenti ai picchi possono essere identificate utilizzando la letteratura esistente6.
Risultati
ADAAD stabilizza una struttura simile a un anello di stelo sul promotore MYC
Precedenti prove sperimentali hanno dimostrato che SMARCAL1 è un regolatore negativo di MYC29. L'analisi della regione del promotore lungo 159 bp del gene MYC da parte del mappatore QGRS ha mostrato che il filamento in avanti aveva il potenziale per formare un G-quadruplex (Tabella 2). Mfold ha mostrato che entrambi i filam...
Discussione
Lo scopo di questo articolo è quello di introdurre la tecnica di spettroscopia CD come approccio per studiare i cambiamenti conformazionali che si verificano nel DNA in presenza di proteine rimodellanti della cromatina ATP-dipendenti e per collegare questi cambiamenti conformazionali all'espressione genica. La spettroscopia CD fornisce un metodo veloce e facilmente accessibile per studiare i cambiamenti conformazionali nel DNA.
Un punto cruciale da considerare per questa tecnica è la...
Divulgazioni
Gli autori non hanno alcun conflitto di interessi da dichiarare.
Riconoscimenti
Gli autori desiderano ringraziare Advanced Instrumentation Research Facility, JNU, per lo spettrofotometro CD. V.J. e A.D. sono stati sostenuti da una borsa di studio del CSIR.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Fisher scientific | O3446I-100 | |
| Adenosine 5′-triphosphate disodium salt hydrate | Sigmaaldrich | A2383 | |
| CD Quartz Cuvette | STARNA | 21-Q-1 | |
| Chirascan V100 CD spectrometer | Applied Photophysics | Not available | |
| EDTA Disodium Salt Dihydrate | SRL | 43272 | |
| Glutathione Sepharose 4B | GE Healthcare | 17-0756-01 | Glutathione affinity chromatography |
| Hellmanex III cleaning solution | Hellma | 9-307-011-4-507 | |
| L-Lactic Dehydrogenase | Sigmaaldrich | L2625 | |
| Magnesium Acetate Tetrahydrate | Fisher scientific | BP215-500 | |
| Magnesium Chloride Hexahydrate | Fisher scientific | M33-500 | |
| NADH disodium salt | Sigmaaldrich | 10107735001 | |
| Phosphoenolpyruvate Monocyclohexylammonium Salt | SRL | 40083 | |
| Potassium Acetate | Fisher scientific | P178-3 | |
| Pyruvate Kinase | Sigmaaldrich | P1506 | |
| Sodium Phosphate Dibasic Anhydrous | Fisher scientific | S374-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher scientific | S369-500 | |
| Synergy HT microplate reader | BioTek | Not available | |
| Tris Base | Fisher scientific | BP152-500 |
Riferimenti
- Woody, R. W. Circular dichroism. Methods in Enzymology. 246, 34-71 (1995).
- Kelly, S., Price, N. The Use of Circular Dichroism in the Investigation of Protein Structure and Function. Current Protein & Peptide Science. 1 (4), 349-384 (2000).
- Rodger, A., Marshall, D. Beginners guide to circular dichroism. The Biochemist. 43 (2), 58-64 (2021).
- Greenfield, N. J. Using circular dichroism spectra to estimate protein secondary structure. Nature Protocols. 1 (6), 2876-2890 (2006).
- Kypr, J., Kejnovská, I., Bednářová, K., Vorlíčková, M. Circular Dichroism Spectroscopy of Nucleic Acids. Comprehensive Chiroptical Spectroscopy. , 575-586 (2012).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Miyahara, T., Nakatsuji, H., Sugiyama, H. Helical Structure and Circular Dichroism Spectra of DNA: A Theoretical Study. The Journal of Physical Chemistry A. 117 (1), 42-55 (2013).
- Khomyakova, E. B. Parallel intramolecular DNA triple helix with G and T bases in the third strand stabilized by Zn2+ ions. Nucleic Acids Research. 28 (18), 3511-3516 (2000).
- Kypr, J., et al. The unusual X-form DNA in oligodeoxynucleotides: dependence of stability on the base sequence and length. Journal of Biomolecular Structure & Dynamics. 13 (6), 999-1006 (1996).
- Zohoorian-Abootorabi, T., Sanee, H., Iranfar, H., Saberi, M. R., Chamani, J. Separate and simultaneous binding effects through a non-cooperative behavior between cyclophosphamide hydrochloride and fluoxymesterone upon interaction with human serum albumin: multi-spectroscopic and molecular modeling approaches. Spectrochimica Acta. Part A, Molecular and Biomolecular Spectroscopy. 88, 177-191 (2012).
- Sharifi-Rad, A., Mehrzad, J., Darroudi, M., Saberi, M. R., Chamani, J. Oil-in-water nanoemulsions comprising Berberine in olive oil: biological activities, binding mechanisms to human serum albumin or holo-transferrin and QMMD simulations. Journal of Biomolecular Structure & Dynamics. 39 (3), 1029-1043 (2021).
- Mokaberi, P., Babayan-Mashhadi, F., Amiri Tehrani Zadeh, Z., Saberi, M. R., Chamani, J. Analysis of the interaction behavior between Nano-Curcumin and two human serum proteins: combining spectroscopy and molecular stimulation to understand protein-protein interaction. Journal of Biomolecular Structure & Dynamics. 39 (9), 3358-3377 (2021).
- Danesh, N., et al. Determining the binding site and binding affinity of estradiol to human serum albumin and holo-transferrin: fluorescence spectroscopic, isothermal titration calorimetry and molecular modeling approaches. Journal of Biomolecular Structure & Dynamics. 36 (7), 1747-1763 (2018).
- Sadeghzadeh, F., et al. Characterizing the binding of angiotensin converting enzyme I inhibitory peptide to human hemoglobin: influence of electromagnetic fields. Protein and Peptide Letters. 27 (10), 1007-1021 (2020).
- Chamani, J., et al. Cooperative alpha-helix formation of beta-lactoglobulin induced by sodium n-alkyl sulfates. Journal of Colloid and Interface Science. 293 (1), 52-60 (2006).
- Dareini, M., et al. A novel view of the separate and simultaneous binding effects of docetaxel and anastrozole with calf thymus DNA: Experimental and in silico approaches. Spectrochimica Acta. Part A, Molecular and Biomolecular Spectroscopy. 228, 117528 (2020).
- Dehghani Sani, F., et al. Changes in binding affinity between ofloxacin and calf thymus DNA in the presence of histone H1: Spectroscopic and molecular modeling investigations. Journal of Luminescence. 203, 599-608 (2018).
- Hargreaves, D. C., Crabtree, G. R. ATP-dependent chromatin remodeling: genetics, genomics and mechanisms. Cell Research. 21 (3), 396-420 (2011).
- Morettini, S., Podhraski, V., Lusser, A. ATP-dependent chromatin remodeling enzymes and their various roles in cell cycle control. Frontiers in Bioscience: A Journal and Virtual Library. 13, 5522-5532 (2008).
- Flaus, A., Martin, D. M. A., Barton, G. J., Owen-Hughes, T. Identification of multiple distinct Snf2 subfamilies with conserved structural motifs. Nucleic Acids Research. 34 (10), 2887-2905 (2006).
- Flaus, A., Owen-Hughes, T. Mechanisms for ATP-dependent chromatin remodelling: the means to the end. The FEBS Journal. 278 (19), 3579-3595 (2011).
- Mizuguchi, G., et al. ATP-driven exchange of histone H2AZ variant catalyzed by SWR1 chromatin remodeling complex. Science. 303 (5656), 343-348 (2004).
- Elizondo, L. I., et al. Schimke immuno-osseous dysplasia: SMARCAL1 loss-of-function and phenotypic correlation. Journal of Medical Genetics. 46 (1), 49-59 (2009).
- Baradaran-Heravi, A., et al. SMARCAL1 deficiency predisposes to non-Hodgkin lymphoma and hypersensitivity to genotoxic agents in vivo. American Journal of Medical Genetics. Part A. 158 (9), 2204-2213 (2012).
- Bansal, R., et al. SMARCAL1, the annealing helicase and the transcriptional co-regulator. IUBMB life. 72 (10), 2080-2096 (2020).
- Yusufzai, T., Kadonaga, J. T. HARP is an ATP-driven annealing helicase. Science. 322 (5902), 748-750 (2008).
- Yusufzai, T., Kong, X., Yokomori, K., Kadonaga, J. T. The annealing helicase HARP is recruited to DNA repair sites via an interaction with RPA. Genes & Development. 23 (20), 2400-2404 (2009).
- Patne, K., et al. BRG1 and SMARCAL1 transcriptionally co-regulate DROSHA, DGCR8 and DICER in response to doxorubicin-induced DNA damage. Biochimica et Biophysica Acta. 1860 (9), 936-951 (2017).
- Sharma, T., Bansal, R., Haokip, D. T., Goel, I., Muthuswami, R. SMARCAL1 negatively regulates c-Myc transcription by altering the conformation of the promoter region. Scientific Reports. 5, 17910 (2015).
- Muthuswami, R., Truman, P. A., Mesner, L. D., Hockensmith, J. W. A eukaryotic SWI2/SNF2 domain, an exquisite detector of double-stranded to single-stranded DNA transition elements. The Journal of Biological Chemistry. 275 (11), 7648-7655 (2000).
- Nongkhlaw, M., Dutta, P., Hockensmith, J. W., Komath, S. S., Muthuswami, R. Elucidating the mechanism of DNA-dependent ATP hydrolysis mediated by DNA-dependent ATPase A, a member of the SWI2/SNF2 protein family. Nucleic Acids Research. 37 (10), 3332-3341 (2009).
- Kikin, O., D'Antonio, L., Bagga, P. S. QGRS Mapper: a web-based server for predicting G-quadruplexes in nucleotide sequences. Nucleic Acids Research. 34, 676-682 (2006).
- Zuker, M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Research. 31 (13), 3406-3415 (2003).
- Gupta, M., et al. Ligand-induced conformation changes drive ATP hydrolysis and function in SMARCAL1. The FEBS Journal. 282 (19), 3841-3859 (2015).
- Dutta, P., et al. Global epigenetic changes induced by SWI2/SNF2 inhibitors characterize neomycin-resistant mammalian cells. PloS One. 7 (11), 49822 (2012).
- Muthuswami, R., et al. Phosphoaminoglycosides inhibit SWI2/SNF2 family DNA-dependent molecular motor domains. Biochemistry. 39 (15), 4358-4365 (2000).
- Gondeau, C. Circular dichroism and UV melting studies on formation of an intramolecular triplex containing parallel T*A:T and G*G:C triplets: netropsin complexation with the triplex. Nucleic Acids Research. 26 (21), 4996-5003 (1998).
- Nongkhlaw, M., Gupta, M., Komath, S. S., Muthuswami, R. Motifs Q and I are required for ATP hydrolysis but not for ATP binding in SWI2/SNF2 proteins. Biochemistry. 51 (18), 3711-3722 (2012).
- Luchnik, A. N. DNA conformational transitions induced by supercoiling control transcription in chromatin. Gene Regulation and Systems Biology. 8, 89-96 (2014).
- Siddiqui-Jain, A., Grand, C. L., Bearss, D. J., Hurley, L. H. Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription. Proceedings of the National Academy of Sciences of the United States of America. 99 (18), 11593-11598 (2002).
- Uribe, D. J., Guo, K., Shin, Y. -. J., Sun, D. Heterogeneous nuclear ribonucleoprotein K and nucleolin as transcriptional activators of the vascular endothelial growth factor promoter through interaction with secondary DNA structures. Biochemistry. 50 (18), 3796-3806 (2011).
- Young, S. L., Krawczyk, S. H., Matteucci, M. D., Toole, J. J. Triple helix formation inhibits transcription elongation in vitro. Proceedings of the National Academy of Sciences of the United States of America. 88 (22), 10023-10026 (1991).
- Dürr, H., Flaus, A., Owen-Hughes, T., Hopfner, K. -. P. Snf2 family ATPases and DExx box helicases: differences and unifying concepts from high-resolution crystal structures. Nucleic Acids Research. 34 (15), 4160-4167 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon