È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Cella di misura Kinetics progressione del ciclo con la marcatura metabolica e citometria a flusso
In questo articolo
Riepilogo
Monitoraggio sottili cambiamenti nella progressione e cinetica di fasi del ciclo cellulare può essere realizzata mediante l'uso di una combinazione di marcatura metabolica di acidi nucleici con BrdU e DNA genomico totale colorazione tramite ioduro di propidio. Questo metodo evita la necessità di sincronizzazione chimica del ciclo cellulare, impedendo l'introduzione di non-specifico danno al DNA, che a sua volta influisce progressione del ciclo cellulare.
Abstract
Controllo preciso di inizio e la progressione successiva attraverso le varie fasi del ciclo cellulare sono di fondamentale importanza in cellule proliferanti. Ciclo di divisione cellulare è parte integrante della crescita e la riproduzione e deregolamentazione dei componenti chiave del ciclo cellulare sono stati implicati negli eventi scatenanti di 1,2 carcinogenesi. Agenti molecolari in terapie anti-cancro spesso bersaglio vie biologiche responsabili della regolamentazione e il coordinamento della divisione del ciclo cellulare 3. Sebbene cinetica del ciclo cellulare tendono a variare in base al tipo di cellula, la distribuzione delle celle tra le quattro fasi del ciclo cellulare è piuttosto consistente in una linea cellulare particolare grazie al modello di costante mitogeni e espressione di fattore di crescita. Eventi genotossici ed altri fattori di stress cellulare può risultare in un blocco temporaneo di progressione del ciclo cellulare, con conseguente arresto temporaneo o una pausa in una particolare fase del ciclo cellulare per consentire istituzionigazione del meccanismo di risposta adeguata.
La capacità di osservare sperimentalmente il comportamento di una popolazione di cellule con riferimento alla fase progressione del ciclo cellulare è un importante progresso in biologia cellulare. Procedure comuni come mitotico scrollarsi di dosso, centrifugazione differenziale o citometria a flusso basato ordinamento vengono utilizzati per isolare le cellule in specifici momenti del ciclo cellulare 4-6. Questi frazionati, ciclo cellulare fase arricchiti popolazioni vengono poi sottoposti a trattamenti sperimentali. Resa, la purezza e la vitalità delle frazioni separate possono spesso essere compromessa usando questi metodi di separazione fisici. Inoltre, l'intervallo di tempo tra la separazione delle popolazioni cellulari e l'inizio del trattamento sperimentale, la quale le cellule frazionati può passare dallo stadio selezionato ciclo cellulare, può creare problemi rilevanti il successo e l'interpretazione di questi esperimenti.
Altri approcci alla stUdy fasi del ciclo cellulare includono l'uso di prodotti chimici per sincronizzare le cellule. Il trattamento delle cellule con inibitori chimici di chiave processi metabolici per ogni fase del ciclo cellulare sono utili nel bloccare la progressione del ciclo cellulare alla fase successiva. Ad esempio, i ribonucleotide reduttasi idrossiurea cellule arresta a G1 / S frangente limitando la fornitura di deossinucleotidi, i blocchi di costruzione di DNA. Altre sostanze chimiche importanti includono il trattamento con afidicolina, un inibitore di alfa polimerasi per l'arresto G1, il trattamento con colchicina e nocodazole, entrambi interferire con la formazione mitotica mandrino per arrestare le cellule in fase M e, infine, il trattamento con il DNA catena terminatore 5-fluorodeoxyridine di avviare S arresto di fase 7-9. Il trattamento con queste sostanze è un mezzo efficace di sincronizzazione un'intera popolazione di cellule ad una fase particolare. Con la rimozione della sostanza chimica, le cellule rientrare nel ciclo cellulare all'unisono. Trattamento della seguente comunicato di prova agentedalla chimica ciclo cellulare bloccando assicura che la risposta farmaco sollecitato è da un uniforme, ciclo cellulare stadio-specifico popolazione. Tuttavia, poiché molti dei sincronizzatori chimiche sono noti composti genotossici, prendere in giro a parte la partecipazione di vari percorsi di risposta (per i sincronizzatori contro gli agenti di test) è una sfida.
Qui si descrive un metodo metabolico etichettatura per seguire una sottopopolazione di cellule attivamente in bicicletta attraverso la loro progressione dalla fase di replicazione del DNA, attraverso la divisione e la separazione delle loro cellule figlie. Accoppiato con quantificazione citometria a flusso, questo protocollo permette per la misurazione della cinetica progressione del ciclo cellulare in assenza di una meccanicamente o chimicamente cellulare indotta sottolinea comunemente associati con altre metodologie ciclo cellulare di sincronizzazione 10. Nelle sezioni seguenti si discuterà la metodologia, così come alcune delle sue applicazioni nella ricerca biomedica.
Protocollo
1. Preparazione cellulare
- Cellule piastra per raggiungere una densità di circa il 60% di confluenza. Le cellule devono essere in fase log al momento della raccolta. Per MCF7, questo viene realizzato semina a 5 x 10 5 cellule / piastra 10 centimetri nei mezzi appropriati. HT29 e LS180 cellule sono seminate a 6 x 10 5 cellule / piastra 6 cm. Abbiamo usato DMEM mezzo supplementato con 10% FBS e 1x penicillina / streptomicina. Assicurati di piastre semi per i controlli corretti positivi e negativi inOltre ai campioni di prova. Essi comprendono i seguenti:
| Io | controllo positivo | BrdU solo |
| ii | controllo positivo | PI solo |
| iii | controllo negativo | BrdU negativo, PI negativo |
(Si noti che un esperimento preliminare con vari intervalli di tempo come quello descritto in questo protocollo può essere utilizzato per restringere l'intervallo di tempo durante il quale per effettuare collezioni future.)
- Le cellule sono placcati incubate a 37 ° C, 5% CO 2 per 24-48 h, consentendo alle cellule di recuperare e collegare.
- Per pulsare cellule di etichette con bromodeossiuridina (BrdU), è sostituito wi DMEMth mezzo fresco contenente 10 pM BrdU. Le cellule vengono incubate per 1 ora a 37 ° C, 5% CO 2 per consentire l'incorporazione di BrdU nel DNA. Assicurati di lasciare una piastra non trattata per fungere da controllo negativo.
- Gli impulsi di etichettatura supporto viene rimosso e le cellule vengono risciacquate brevemente con 1X PBS.
- Mezzi di crescita fresco è aggiunto (meno il BrdU) e le cellule possono continuare ad incubare a 37 ° C, 5% CO 2 fino timepoint appropriato per la raccolta viene raggiunto.
2. Raccolta e fissaggio
- Cellule non trattate dal punto 1.3 sono considerati i timepoint zero. Questo esempio può essere raccolto insieme con la h 1 timepoint che viene raccolto subito dopo il trattamento BrdU.
- Per raccogliere le cellule, supporto viene rimosso e le piastre vengono risciacquata con 1X PBS.
- Le cellule vengono tripsinizzate, raccolti in mezzi di coltura e successivamente pelletizzata mediante centrifugazione a 1500 rpm per 5 min Supernatant viene scartato.
- Sciacquare 1-10 x 10 6 cellule in 5 ml di ghiaccio freddo 1X PBS. Centrifugare a 1500 rpm per 5 minuti Il supernatante viene scartato.
- Il pellet cellulare viene quindi risospese in 100 pl di ghiaccio freddo PBS / 1% FBS. L'aggiunta di 1% FBS in PBS aiuta a prevenire aggregazione cellulare.
- Aggiungere queste cellule goccia a goccia a 5 ml di -20 ° C, 70% etanolo per fissare le cellule.
- Incubare in ghiaccio per 30 minuti o conservare a 4 ° C per una notte. Questo è un passo ideale a cui interrompere la raccolta dei campioni delle punti temporali diversi. I campioni possono essere lasciati in etanolo per diversi giorni, consentendo loro di essere trattati simultaneamente attraverso il resto del protocollo.
3. BrdU e PI colorazione
- Celle a pellet di centrifugazione per 5 min a 1500 rpm.
- Togliere il fissativo, ma lasciare ~ 50 microlitri in cui allentare il pellet nel vortex.
- Snaturare il DNA, lentamente, aggiungere 1 ml di HCL 2N / Triton X-100goccia a goccia mentre il vortex. Incubare campioni a temperatura ambiente per 30 min.
- Celle a pellet di centrifugazione campioni per 5 min a 1500 rpm. Aspirare e scartare il surnatante.
- Risospendere il pellet di cellule in 1 ml di tetraborato di sodio 0,1 M, pH 8,5, per neutralizzare la fase di denaturazione.
- Centrifugare cellule per 5 min a 1500 rpm. Aspirare e scartare il surnatante.
- Risospendere il pellet di cellule in 75 pl di miscela di colorazione BrdU (50 pl 0,5% Tween 20/1% BSA / PBS + 20 pl coniugato con FITC anti-BrdU + 5 pl 10 mg / ml RNasi).
- Incubare a temperatura ambiente per 45 minuti al riparo dalla luce.
- Per cellule pellet, centrifugare i campioni per 5 min a 1500 rpm. Aspirare e scartare il surnatante.
- Risospendere il pellet di cellule in 1 ml di PBS contenente 5 mg / ml di ioduro di propidio.
4. Citometria a Flusso
- Per analizzare le cellule, un citofluorimetro equipaggiato con un laser 488 nm e laopportuni filtri è necessario. Il software di analisi come CellQuest è necessaria per creare le trame descritte di seguito.
- Quando si esegue il campione attraverso il citofluorimetro, creare un forward scatter (SSC-H) vs lato scatter (FSC-H) piano per garantire la distribuzione corretta dimensione delle cellule (Figura 1A). Entrambi questi parametri vengono tracciate su una scala lineare.
- Contemporaneamente visualizzare un appezzamento di FL3-H (PI macchia) vs FL2-W (Figura 1B). Questo grafico è utilizzata per creare un cancello (R3) per isolare la frazione di cellule che sono all'interno del modello di distribuzione normale del ciclo cellulare. In generale, questo modello di distribuzione è caratterizzato da due gruppi di cellule del contenuto di DNA 2N e 4N di colorazione PI rappresentare fasi G1 e G2 del ciclo cellulare, rispettivamente. Una stringa di cellule situate tra questi 2 gruppi rappresentativa della replicazione del DNA continuo che si verifica nella fase S del ciclo cellulare.
- Creare un grafico displaying le cellule gated (R3) dal punto 4.3, con FL1-H (FITC, plot log) sulla y-asse e PI (trama lineare) sul l'asse x (Figura 1C). Questa trama verrà utilizzato per impostare i parametri rimanenti utilizzando i vari controlli descritti in 1,1, nonché a raccogliere dati per l'analisi.
- Posizionare la PI solo campione colorato sul citometro. Regolare il guadagno di inserire il G1 a ~ 200 sul l'asse x. Questo sarà più facile da visualizzare in un grafico a istogramma della macchia PI.
- Un secondo controllo prevede l'esecuzione di uno BrdU solo campione sul citometro a flusso. Regolare il guadagno in modo che le 2 popolazioni di cellule BrdU (BrdU positive vs negative) appaiono sul terreno. Idealmente, le cellule BrdU negativi sono posizionati a comparire appena sotto 10 -1.
- Il controllo finale è il BrdU / PI campione negativo. Eseguire questa controllo negativo per garantire che le cellule non apparire in qualsiasi dei quadranti superiori mano o destra.
- Una volta che i parametri dei vari appezzamenti sono stati impostati, ilcitometro a flusso è calibrato e pronto per la lavorazione delle cellule BrdU e PI macchiati. Un minimo di 10.000 cellule che sono gated nella frazione corretta PI (vedi punto 4.3) deve essere letta per la raccolta.
- Per analizzare i dati raccolti, software come FlowJo o FacsDiva vengono utilizzati. Ognuno dei lotti di cui sopra può essere ricreato in questi programmi e l'analisi quantitativa e statistica effettuata.
- L'endpoint di analisi coinvolge diverse fasi successive. Creazione di un appezzamento di FITC vs PI con le cellule gated come positivo dalla trama PI FL2-W PI permette di distinguere la popolazione ciclistica. Una seconda porta viene creato da questo grafico, isolando la popolazione BrdU positive. Esprimendo questa popolazione distintivo su un terreno istogramma con PI l'asse x permette di monitorare l'andamento nel tempo della progressione del ciclo cellulare. Questo può essere ulteriormente visualizzati tracciando il numero di BrdU-positive cellule con G1 o G2 contenuto in funzione del tempo.
5. Risultati rappresentativi
Normalmente le cellule ciclismo colorate con ioduro di propidio avere picchi distinti a G1 e G2, corrispondenti a celle contenenti contenuto di DNA 2N e 4N, rispettivamente (figura 2). Pulse etichettatura con BrdU consente di etichettatura selettiva di una sotto-popolazione di cellule che sono attivamente sintetizzare DNA (cioè fase S). Poco dopo la rimozione del reagente BrdU, tutte le cellule marcate sono in fase S (Figura 3). Limitando l'etichettatura di un breve impulso si è in grado di seguire questa ora distinti sub-popolazione di cellule in tutta numerosi punti temporali che passano attraverso le successive fasi del ciclo cellulare. Questo può essere visualizzato al punto 1 volta h in figura 3 come una netta mancanza di G1 e G2 picchi.
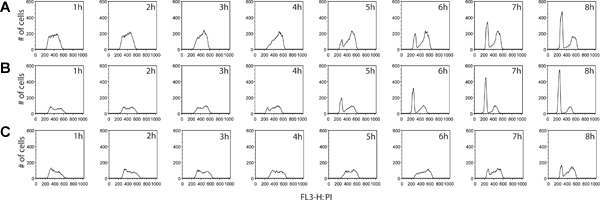
Ulteriori informazioni possono essere derivato dalla fase di marcatura BrdU. Non solo la proporzione di cellule in divisione attivamente essere misurato, ma un ESTiMate di distribuzione stadio del ciclo cellulare tra due campioni possono anche essere determinata. Con la raccolta di cellule equidistante dopo la rimozione del reagente BrdU, le cellule possono essere rintracciati mentre continuano a ciclo, progressi G2 e infine passare attraverso la divisione cellulare delle cellule originali, infine, emerge come BrdU-positive cellule figlie con G1 contenuto di DNA (Figura 4). Un esempio di quantificazione fase del ciclo cellulare e analisi cinetica viene fornita in Figura 5.
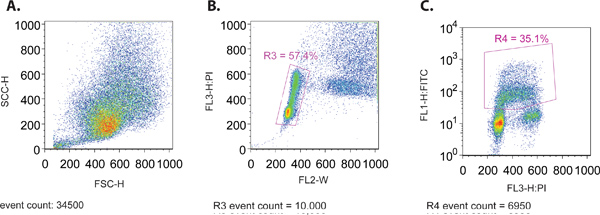
Figura 1. (A) rispetto al Forward Scatter Scatter Plot Side di un rappresentante MCF7 popolazione di cellule. (B) PI rispetto Larghezza (FL-W) Plot di un rappresentante MCF7 popolazione di cellule. Gating è mostrato escludere doppietti cellule in ultima analisi (R3). Doppietti cellulari avrà larghezza di impulso maggiore di una singola cella, che prendono più a passare attraverso il raggio laser e therefore può essere escluso dall'analisi. (C) PI rispetto FITC (BrdU) Plot di una popolazione rappresentativa MCF7 cellulare. Gating viene mostrata per includere solo i FITC (BrdU) cellule positive (R4).
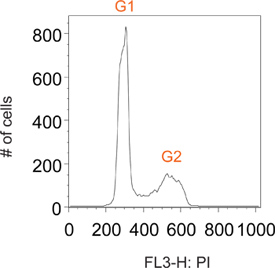
Figura 2. Istogramma grafico rappresentante di una popolazione totale di cellule normalmente in bicicletta.

Figura 3. Trama istogramma di cellule che sono stati raccolti 1h dopo la rimozione del polso BrdU, dopo gating per FITC (BrdU)-cellule positive. BrdU-cellule positive ad un tempo di valutazione precoce dopo la rimozione di etichetta mostrano profili PI che corrispondono a campioni che mostrano contenuti DNA coerente con cellule che sono nella fase S del ciclo cellulare, confermando l'etichettatura di successo di cellule soltanto durante la sintesi del DNA.
 lt = "Figura 4" />
lt = "Figura 4" />
Figura 4. Confronto cinetica di progressione del ciclo cellulare tra linee cellulari tumorali, cancri colorettali HT29 (A) e LS180 (B), così come il cancro della mammella MCF7 (C). Le cellule sono state raccolte ogni ora per 8 ore, dopo la rimozione del polso BrdU. In questo esperimento, abbiamo osservato un chiaro profilo di progressione accelerata ciclo cellulare attraverso la fase G2 del ciclo cellulare nella LS180 colorettale linea cellulare. Confrontando la cinetica tra i due profili delle cellule tumorali del colon-retto, l'emergere di un picco G1 è evidente a T = 4 ore post-BrdU nelle cellule LS180, rispetto al punto di tempo corrispondente per la linea cellulare HT-29 che manca di questo picco. Rispetto sia con una delle due linee di cellule del colon-retto, MCF7 cellule sono in bicicletta ad un tasso notevolmente ridotto. Clicca qui per ingrandire la figura .
re 5 "/>
Figura 5. (A) l'analisi del ciclo cellulare quantitativa fase di BrdU-marcati cellule tumorali. Il Preside / Jett / Fox algoritmo è stato applicato a HT29, LS180 e MCF7 (illustrata in verde). Le cellule risultanti distribuzioni fase del ciclo di ciascun campione sono espressi come% Totale BrdU-cellule positive per ogni fase. Solo selezionare i punti temporali per ciascuna linea cellulare vengono visualizzati, in quanto le analisi per alcuni dei punti temporali precedenti ha prodotto risultati validi. In questi punti temporali precedenti, tutte BrdU-cellule positive sono in fase S e non hanno quindi distinto G1 e G2 picchi, che sono necessari per l'applicazione dell'algoritmo. (B) istogrammi di cellule tumorali che mostra la cinetica di progressione attraverso G2 / M fasi del ciclo cellulare. La progressione attraverso G2 / M è il più veloce le fasi per la LS180 cellule, seguita da HT29 e MCF7. Clicca qui per ingrandire la figura .
jpg "alt =" Figura S1 "/>
S1 Figura. Citometria a flusso di controllo. MCF7 sono mostrati come (A) controllo negativo in cui le cellule non sono né PI né tinto BrdU. (B) PI solo macchiato. (C) BrdU-FITC campioni.

Figura S2. Rappresentazione schematica che mostra la relazione tra lo spettro di emissione fluorescente PI rispetto FITC (BrdU). Le finestre di raccolta spettrali fluorescente per il canale 1 (FL1, per FITC) e il canale fluorescente 3 (FL3, per PI) sono mostrati nelle caselle corrispondenti. Non vi è alcuna sovrapposizione tra gli spettri fluorofori FL1 e FL3 rilevamento. Evidentemente, compensazione della fluorescenza non è necessaria quando i dati sperimentali da PI e FITC-BrdU co-cellule marcate vengono raccolte nelle FL1/FL3 canali rispettivamente. Spettri di fluorescenza sono stati ottenuti dal sito BD BioSciences:target = "_blank"> http://www.bdbiosciences.com/research/multicolor/spectrum_viewer. Clicca qui per ingrandire la figura .
Discussione
Grazie alla combinazione di citometria a flusso con incorporazione di BrdU, abbiamo gli strumenti necessari per studiare cinetica del ciclo cellulare. La proprietà caratteristica di BrdU per funzionare come un analogo della timidina è ciò che permette di quantificare il contenuto di DNA di una cellula bicicletta. L'incorporazione di BrdU in un filamento figlia crescente DNA durante la fase di sintesi del ciclo cellulare è quello che permette di seguire una sottopopolazione di cellule bicicletta attraverso la rep...
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Ringraziamo Andy Johnson del Centro di Ricerca Biomedica presso la UBC per l'assistenza con l'analisi FACS. Finanziamento della ricerca sul cancro in laboratorio Wong è fornito dalla Canadian Cancer Society Research Institute (sovvenzione di funzionamento # 019250) e dal reinvestimento dei fondi di ricerca della Facoltà di Scienze Farmaceutiche, UBC. JMYW è supportato da sedie di ricerca del Canada e la Fondazione Michael Smith carriera di ricerca per i programmi di sviluppo della salute.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagente | Azienda | Numero di catalogo | Comments |
| bromodeossiuridina | Becton Dickinson | 55089 | |
| propidio ioduro | Sigma | 287075 | 1mg/ml magazzino |
| FITC anti-BrdU | Becton Dickinson | 347583 | |
| di sodio tetraborato | Pescatore | S80172 | 0,1 M, pH 8,5 |
| FACS Caliber | Becton Dickinson |
Riferimenti
- Musgrove, E. A., Caldon, C. E., Barraclough, J., Stone, A., Sutherland, R. L. Cyclin D as a therapeutic target in cancer. Nat. Rev. Cancer. 11, 558-572 (2011).
- Molchadsky, A., Rivlin, N., Brosh, R., Rotter, V., Sarig, R. p53 is balancing development, differentiation and de-differentiation to assure cancer prevention. Carcinogenesis. 31, 1501-1508 (2010).
- Dickson, M. A., Schwartz, G. K. Development of cell-cycle inhibitors for cancer therapy. Curr. Oncol. 16, 36-43 (2009).
- Banfalvi, G. Cell cycle synchronization of animal cells and nuclei by centrifugal elutriation. Nat. Protoc. 3, 663-673 (2008).
- van Opstal, A., Boonstra, J. Inhibitors of phosphatidylinositol 3-kinase activity prevent cell cycle progression and induce apoptosis at the M/G1 transition in CHO cells. Cell Mol. Life Sci. 63, 220-228 (2006).
- Gasnereau, I. Flow cytometry to sort mammalian cells in cytokinesis. Cytometry. A. 71, 1-7 (2007).
- Harper, J. V. Synchronization of cell populations in G1/S and G2/M phases of the cell cycle. Methods Mol. Biol. 296, 157-166 (2005).
- Pedrali-Noy, G. Synchronization of HeLa cell cultures by inhibition of DNA polymerase alpha with aphidicolin. Nucleic Acids Res. 8, 377-387 (1980).
- Merrill, G. F. Cell synchronization. Methods Cell Biol. 57, 229-249 (1998).
- Fleisig, H. B., Wong, J. M. Telomerase promotes efficient cell cycle kinetics and confers growth advantage to telomerase-negative transformed human cells. Oncogene. , (2011).
- Cai, D., Byth, K. F., Shapiro, G. I. AZ703, an imidazo[1,2-a]pyridine inhibitor of cyclin-dependent kinases 1 and 2, induces E2F-1-dependent apoptosis enhanced by depletion of cyclin-dependent kinase 9. Cancer Res. 66, 435-444 (2006).
- Pozarowski, P., Darzynkiewicz, Z. Analysis of cell cycle by flow cytometry. Methods Mol. Biol. 281, 301-311 (2004).
- Sherwood, S. W., Rush, D. F., Kung, A. L., Schimke, R. T. Cyclin B1 expression in HeLa S3 cells studied by flow cytometry. Exp. Cell Res. 211, 275-281 (1994).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon