Method Article
Multi-target Parallel Processing Approccio per Gene-to-struttura Determinazione della polimerasi Influenza PB2 subunità
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Structure-based drug design gioca un ruolo importante nello sviluppo di farmaci. Perseguire obiettivi multipli in parallelo aumenta notevolmente la probabilità di successo per la scoperta di lead. L'articolo che segue mette in evidenza come il Seattle Center Genomica strutturale di Malattie Infettive utilizza un approccio multi-obiettivo per la determinazione del gene-per-struttura della influenza PB2 A subunità.
Abstract
Pandemie influenzali di ceppi altamente virulenti possono causare morbilità e mortalità diffusa nelle popolazioni umane in tutto il mondo. Negli Stati Uniti da soli, una media di 41.400 morti e 1.860.000 ricoveri sono causati da infezione da virus influenzale ogni anno 1. Le mutazioni puntiformi nel polimerasi di base delle proteine 2 subunità (PB2) sono stati collegati all'adattamento dell'infezione virale nell'uomo 2. I risultati di tali studi hanno rivelato il significato biologico di PB2 come un fattore di virulenza, evidenziando in tal modo il suo potenziale come bersaglio di un farmaco antivirale.
Il programma di genomica strutturale messo avanti dall'Istituto Nazionale di allergie e malattie infettive (NIAID) finanzia Emerald Bio e di altre tre istituzioni Pacific Northwest che insieme costituiscono il centro di Seattle Genomica strutturale per malattie infettive (SSGCID). Il SSGCID è dedicato a fornire alla comunità scientifica di tre Tepstrutture proteiche e-dimensionali del NIAID categoria patogeni AC. Rendere tali informazioni strutturali a disposizione della comunità scientifica serve per accelerare la struttura basata su drug design.
Structure-based drug design gioca un ruolo importante nello sviluppo di farmaci. Perseguire obiettivi multipli in parallelo aumenta notevolmente la probabilità di successo per la nuova scoperta di piombo di mira una via o di una intera famiglia di proteine. Emerald Bio ha sviluppato una pipeline di elaborazione parallela ad alta velocità, multi-target (MTPP) per la determinazione del gene-per-struttura a supporto del consorzio. Qui si descrivono i protocolli utilizzati per determinare la struttura della subunità PB2 da quattro influenza diversa A ceppi.
Protocollo
Una panoramica del protocollo è presentato nella figura 1.
Biologia Molecolare
1. Costruire design
Utilizzare software Composer Gene per progettare costrutto proteine e codone ingegnerizzati sequenze di geni sintetici. L'uso di software Composer Gene è stato offerto in dettaglio altrove 3.
- Utilizza il modulo Viewer Allineamento e Costruire Design del modulo per confrontare sequenze proteiche allineamenti e definire costrutto proteine. Allineare bersaglio sequenza amminoacidica a entrambi gli elementi strutturali primari e 3D da omologhi nella Protein Data Bank (PDB), se disponibili (Figura 2).
- Utilizzare le informazioni di allineamento per fare i disegni costrutto struttura a guida scegliendo nuovi termini sulla base di conservazione della struttura primaria e strutture 3D di omologhi.
- Progettazione inserto PCR (IPCR) e vettore PCR (vPCR) amplimeri (terminale primer).
- Utilizzando Gene CAlgoritmo proteina-a-DNA di omposer, back-tradurre la sequenza amminoacidica costrutto ingegnerizzato in codone sequenza di acido nucleico.
- Utilizzare la corretta tabella codon usage (CUT) per ottimizzare sequenza per l'espressione in E. coli.
- Praticamente clonare inserto in pET28 vettore modificato per incorporare un tag N-terminale Istidina 6x e proteina di fusione Smt3/SUMO che consente una facile purificazione.
- Mettere ordine gene sintetico con DNA 2.0 e ordine primer dalle tecnologie del DNA integrati.
2. Polimerasi incompleta Primer Extension (PIPE) Clonazione
- Preparare Primer e geni
- Centrifugare le piastre forniti dal produttore contenenti primer a 1.000 rpm per 1 min.
- Portare concentrazione dei primer a 100 micron e aggiungere 50 microlitri di buffer TE.
- Diluire primer a 10 micron con deionizzata (DI) in una piastra a 96 pozzetti fondo a V.
- Centrifugare il gene fornito dal produttore in una provetta da 1,5 ml a 1.300 rpm per 1 min.
- Usando tampone TE, portare la concentrazione di DNA di ciascun tubo a 50 ng / mL.
- In provette da 1,5 ml, fare diluizioni di ciascun primer a 10 ng / ml.
- Conservare primer e geni a -20 ° C quando non in uso.
- Preparare Inserisci PCR (IPCR)
- Scongelare una fiala di Pfu Master Mix su ghiaccio; mantenere i geni e primer a temperatura ambiente.
- Creare una mappa piastra assegnazione pozzi per un set di primer e costruire.
- Aggiungere 13 ml di acqua deionizzata in ogni pozzetto di una piastra PCR da 96 pozzetti.
- Aggiungere 5 ml di avanti e 5 ml di primer reverse per ciascuna reazione nella piastra a 96 pozzetti secondo la mappa piastra, assicurando cambiare punte tra ogni pozzetto.
- Aggiungere 2 ml di ogni gene piena lunghezza adeguata al suo bene secondo la mappa piastra.
- Aggiungere 25 ml di Pfu Master Mix per ogni bene, assicurando a cambiare punte tra ogni bene.
- Ciclo le reazioni utilizzando le seguenti condizioni di PCR:
- 95 ° C 2 min
- 95 ° C 30 sec
- 50 ° C 45 sec
- 68 ° C 3 min
- 4 ° C ∞
- Ripetere i passaggi bd per 25 cicli.
- Trasferire 10 ml di ogni reazione IPCR ad una nuova piastra PCR da 96 pozzetti.
- Aggiungere 3 ml di colorante carico 6X per ogni campione.
- Separare ogni campione su una% TAE EtBr gel di agarosio 1 a 110 V, accanto a un 100-500 bps scaletta del DNA per confermare frammento di amplificazione.
- Conservare IPCR prodotto a -20 ° C, quando non in uso (evitare congelamento scongelamento per quanto possibile).
3. Preparare PCR Vector (vPCR)
- Inizio cultura durante la notte di trasformare E. coli con pET28 vettore plasmidico.
- Inoculare due provette da 5 ml di brodo 2-YT con 50 mg / ml di kanamicina.
- Grow culture durante la notte a 37 ° C in agitatore a 220 giri al minuto.
- Spin down colture dopo la crescita durante la notte per centrifugazione a 3000 rpm per 15 min.
- Utilizzare un Qiagen QIAprep Spin Miniprep Kit per estrarre pET28 vettore dal pellet batterico in base alle istruzioni del produttore.
- Setup enzimi di restrizione digestioni di estratti pET28 plasmide.
- Aggiungere 2,2 ml di tampone BamHI 10X e 1 ml di BamHI e HindIII per 20 l di pET28 vettore.
- Incubare la reazione per 1 ora a 37 ° C.
- Prodotto di digestione separato su un gel.
- Fare riferimento al punto 2.2.10.
- Tagliare banda vettore dal gel e purificarla con il QIAquick Gel Extraction Kit secondo le istruzioni del produttore.
- Utilizzando un NanoDrop, quantificare la concentrazione di DNA.
- Diluire tagliato vettore a 10 ng / ml. Conservare a -20 ° C, quando non in uso.
- Preparare primer vPCR.
- Centrifugare IDT oligonucliotides forniti per 1 min a 1.300 giri al minuto.
- Portare la concentrazione di 100 mM con acqua deionizzata.
- Preparare 10 micron diluizione di entrambi i primer forward e reverse in una provetta da 1,5 ml.
- Conservare primer e diluizioni di primer a -20 ° C.
- Scongelare Pfu Master Mix su ghiaccio e disgelo modello e primer a temperatura ambiente.
- Reazioni vPCR di installazione in una piastra PCR da 96 pozzetti:
- Nella prima riga di una piastra a 96 pozzetti combinano 60 pl di entrambi i primers vPCR avanti e inversa e 24 pl di digerito pET28 dima (10 ng / ml).
- Usando una pipetta multicanale 12-tip, trasferire 12 microlitri del primer e master mix modello a ogni residua pozzetto della piastra. Ciò dovrebbe tradursi in 12 microlitri di primer e template master mix in ciascun pozzetto della piastra.
- Aggiungere 13 ml di acqua deionizzata in ogni pozzetto.
- Aggiungere 25 ml di Pfu Master Mix in ciascun pozzetto.
- Ciclo le reazioni attraverso le condizioni di PCR utilizzati nella fase 2.2.7.
- Pool tutte le reazioni vPCR in una provetta Falcon 15.
- Verificare frammento di amplificazione separando 10 ml di prodotto in pool PCR su un gel (lunghezza prevista del digerito pET28 vettoreè di circa 6 KB).
- Fare riferimento al punto 2.2.10.
- Preparare unire le piastre.
- Aliquota 3 ml di prodotto vPCR in ciascun pozzetto di una piastra a 96 pozzetti fondo a V.
- Conservare le piastre a -20 ° C fino a fusione con il prodotto IPCR.
4. Unisci IPCR e vPCR Prodotti
- Prodotti IPCR disgelo e pre-aliquotate vPCR 96 pozzetti fondono piastra a temperatura ambiente.
- Aggiungere 3 ml di ogni prodotto IPCR al suo rispettivo pozzetto della piastra di unione.
- Trasforma unire piastra in Top Ten cellule competenti chimicamente.
- Aggiungere 2 ml di ogni reazione di fusione in un unico tubo da 50 ml di cellule chimicamente competenti del fornitore e procedere con il protocollo fornito dal produttore.
- Preparare le culture durante la notte per ogni costrutto dalla piastra di trasformazione.
- Un'aliquota 5 ml di brodo TB (con 50 pg / ml kanamicina) da un serbatoio 25 ml sterile in ciascun pozzetto di un blocco ben profondo.
- Utilizzando steriLe tecniche, scegliere una colonia isolata da ogni piatto trasformazione e inoculare il bene appropriata del blocco pozzo profondo.
- Coprite il blocco con un coperchio Airpore.
- Agitare blocco a 220 rpm a 37 ° C per una notte.
- Cellule pellet mediante centrifugazione del blocco per 30 min a 4000 rpm.
- Eliminare il sopranatante e picchiettare la parte superiore del blocco di asciugare con un tovagliolo di carta.
- Mini-prep usando un Qiagen 96 pozzetti apparato vuoto secondo le istruzioni del produttore.
5. Preparazione Glicerina Azioni di costrutti clonati con successo
- Trasformare clonato con successo sequenza di DNA validato in BL21 (DE3) cellule chimicamente competenti in base alle istruzioni del produttore.
- Per ogni costrutto, scegliere una singola colonia isolata dal (DE3) trasformazione e inoculare BL21 in 1 ml di 2-YT brodo (con 50 mg / ml kanamicina).
- Agitare culture a 220 rpm per 3-4 ore a 37 ° C.
- Etichettare un 1,5 mlprovetta con tappo a vite con il numero unico costrutto di identificazione, ceppo cellula, e la data. Aggiungere 500 ml di glicerolo al 50% e 500 ml di coltura cellulare e capovolgere varie volte. Memorizzare immediatamente magazzino glicerolo in ghiaccio secco o in un congelatore ° -80 C.
6. Expression Testing
| Lysis Buffe r | Tampone di lavaggio | Tampone di eluizione |
| 50 mM NaH 2 PO 4, pH 8.0 300 mM di NaCl 10 mM imidazolo 1% Tween 20 2 mM MgCl2 0,1 microlitri / ml Benzonase 1 mg / ml di lisozima | 50 mM NaH 2 PO 4, pH 8.0 300 mM di NaCl 20 mM imidazolo 0,05% Tween 20 | 25 mM Tris, pH 8,0 300 mM di NaCl 250 mM imidazolo 0,05% Tween 20 |
* Aggiungi Benzonase, lisozima,e inibitore di proteasi immediatamente prima lisi.
- Streak un campione dal magazzino glicerolo su kanamicina agar selettivo e incubare una notte a 37 ° C.
- Avviare un non-indurre pre-coltura in un blocco 96 pozzetti a fondo tondo; inoculare 1,2 ml TB brodo (con 50 mg / ml kanamicina) supplementato con 0,5% di glucosio con una E. freschi coltivati coli isolano. Cresce durante la notte agitazione a 220 rpm a 37 ° C.
- Dopo una crescita overnight, avviare culture induzione inoculando 1,2 ml di brodo TB (con 50 mg / ml kanamicina) supplementato con Novagen Overnight sistema Express 1 (secondo il protocollo del produttore) con 40 pl di pre-coltura.
- Far crescere le culture induzione di piccola scala a 20 ° C per 48 ore, agitazione a 220 rpm.
- Celle di raccolta per centrifugazione a 4000 rpm per 15 minuti, versare il surnatante e conservare a -20 ° C per almeno 1 ora prima della lavorazione.
- Nel blocco 96 pozzetti, risospendere il pellet di cellule in 300 microlitri tampone di lisi.
- Incubare le cellule in tampone di lisi a temperatura ambiente per 30 minuti seguita da lisi meccanica da agitando vigorosamente per 30 min a temperatura ambiente.
- Chiarire il lisato grezzo mediante centrifugazione per 30 min a 4000 rpm a 4 ° C.
- Utilizzare una pipetta multicanale per trasferire 200 microlitri di lisato chiarificato (frazione solubile) per un vassoio 96 pozzetti a fondo piatto (Qiagen). Per ciascun pozzetto contenente un campione, aggiungere 40 microlitri perline Ni-NTA (Qiagen magnetici).
- Agitare delicatamente la piastra su una sedia a dondolo per 1 ora a 16 ° C.
- Posizionare la piastra su un piatto posto magnetica (Qiagen) e rimuovere la frazione solubile non legato. Fare attenzione a non pipettare nessuna delle perline Ni-NTA.
- Rimuovere la piastra dalla piastra posta e delicatamente risospesi le perline in 200 microlitri di tampone di lavaggio. Pipettare su e giù per 30 secondi e poi mettere la piastra posteriore sulla piastra posta.
- Rimuovere il tampone di lavaggio e ripetere il punto 6.12.
- Rimuovere la piastra di lamiera posta ed eluire il Ni-NTA legato target proteina mediante lavaggio con 50 microlitri tampone di eluizione per 5 min.
- Ritorno piatto fondo piatto per piatto messaggio magnetica e trasferire l'eluizione di una nuova piastra a 96 pozzetti fondo a V.
- Trasferire 20 microlitri della eluizione di una fresca 96 pozzetti fondo a V e reagiscono con 1 microlitri ULP1 proteasi.
- Secondo il protocollo del produttore, analizzare la frazione eluita ed eluita + Ulp1 mediante elettroforesi capillare utilizzando un LabChip 90.
- In alternativa, tutte le frazioni dalla sperimentazione espressione possono essere analizzati mediante SDS-PAGE.
7. Grande Fermentazione Scala
- Utilizzare un puntale sterile per ottenere un graffio da un glicerolo magazzino, inoculare 100 ml di brodo di TB (con 50 mg / ml di kanamicina) e crescere durante la notte. Agitare a 220 rpm e 37 ° C.
- Dopo una crescita overnight, espandere precoltura inoculando 1 L di brodo TB con soluzioni dell'autoinduzione EMD (cfr. protocollo del produttore) (con 50 mg / ml kanamicina) in un pallone da 2 L confuso con 10 ml dipre-cultura (diluizione 1:100).
- Agitare le colture espanse 1 L a 37 ° C; modificare la temperatura della agitazione incubatore a 20 ° C quando viene raggiunta una densità ottica di 0,6 (OD 600).
- Dopo la crescita durante la notte, prendere un rappresentante 10 ml aliquota da ogni costrutto per la prova di espressione.
- Cella Harvest pasta per centrifugazione a 5000 rpm per 15 minuti e scartare il surnatante.
- Cella di congelamento incollare a -80 ° C.
Purificazione delle proteine
Buffer:
| Lysis Buffer | Buffer A (equilibrazione) | Buffer B (eluizione) | Dimensionamento Colonna Buffer |
| 25 mM Tris, pH 8,0 200 mM NaCl 0,5% Glicerina 0,02% CHAPS 10 mM imidazolo 1 mM TCEP 50 mM di arginina 5 Benzonase microlitri 100 mg lisozima 3 proteasi Compresse serotonina (EDTA-libero) | 25 mM Tris, pH 8,0 200 mM NaCl 10 mM imidazolo 1 mM TCEP 50 mM di arginina 0,25% Glicerina | 25 mM Tris, pH 8,0 200 mM NaCl 200 mM imidazolo 1 mM TCEP | 25 mM Tris, pH 8,0 200 mM NaCl 1% Glicerina 1 mM TCEP |
* Aggiungi Benzonase, lisozima, e compresse di inibitori della proteasi per ogni campione da 150 ml immediatamente prima di lisi.
8. Cell Lysis
- Fare 2 L di tampone di lisi, non aggiungere lisozima, compresse inibitore della proteasi o benzonasi (ogni campione saranno lisate separatamente in 150 ml di tampone di lisi).
- Disgelo e risospendere cella incolla in tampone di lisi in una massa 01:05: rapporto in volume da vigorosamente agitazione per 30 min a 4 ° C. Rompere pezzi sciolti dai lati del bicchiere con una spatola pulita. Durante questo periodo di tempo, preparare Ni e Dialysis Buffer
- Sul ghiaccio, lisare le cellule utilizzando un sonicatore Misonix (il 70% di potenza, 2 sec a / 1 sec spento impulsi per 3 min) e delicatamente contenitore girandola per evitare il surriscaldamento. Salvare un piccolo (200 ml) aliquota del lisato grezzo per le analisi future.
- Chiarire il lisato grezzo per centrifugazione a 18.000 xg per 35 min a 4 ° C, raccogliere il surnatante e salvare un piccolo (200 ml) aliquota per le analisi future. Conservare pellet a 4 ° C fino a quando si conferma la proteina è stata lisate nella frazione solubile.
9. Proteine Setup Maker Pre-run
- Con il produttore di proteina attivata e il software aperto, inizializzare lo strumento.
- Una volta inizializzato, allegare uno 5,0 ml GE Healthcare HisTrap FF colonna Nichel-chelato (Ni colonna) in una linea separata del gantry per ciascuno dei campioni.
- Esegui volumi di colonna 3-4 (CV) di tampone di equilibrazione attraverso ogni colonna.
- Primo il raggiungimento dell'equilibrio e linee tampone di eluizione.
- Equilibrate le colonne aspirando buffer A attraverso la colonna una volta.
10. Nickel 1 (NI1) Colonna
- Lavare ciascuna colonna con 20 ml di acqua Milli-Q per rimuovere tampone di conservazione. Esegui 5 ml di tampone B e 25 ml di tampone A per il raggiungimento dell'equilibrio.
- Caricare il lisato chiarificato contenente proteina solubilizzata nelle colonne a una velocità di 2 ml / min poi seguire da un lavaggio 15 ml con tampone A.
- Eluire le proteine in un gradiente passo con i buffer A e B con i seguenti rapporti rispettosamente: 5 ml 95:5, 60:40 5 ml, 10 ml di 0:100. Raccogliere separatamente ogni frazione di eluizione.
- Analizzare: frazioni eluite, lisato grezzo, lisato chiarificato e flow-through mediante SDS-PAGE. Pool frazioni contenenti la proteina e utilizzare un NanoDrop alla misura A 280 per determinare approssimativamente la quantità di proteine presenti.
11. ULP1 Decolleté
- Mantenere una piccola aliquota (250 microlitri) del pool NI1 colonna per la successiva analisi del gel. Portare il restodella piscina NI1 a 10 ml e aggiungere ubiquitina-proteasi come 1 (ULP1) a 1 ml / 5 mg di proteine totali per rimuovere il tag affinità His-Smt.
- Dializzare la piscina NI1 + ULP1 contro 2 L di tampone A per 4 ore a 4 ° C in 10 kDa cutoff peso molecolare (MWCO) su una piastra di agitazione a 4 ° C.
- Dopo la dialisi, eseguire SDS-PAGE di NI1 piscina e NI1 piscina + ULP1 per determinare se ULP1 scissione ha avuto successo.
12. Nickel 2 (Ni2) Colonna
- Caricare proteina spaccati sulla stessa colonna Ni e ripetere il passo 9,3 ad una portata ridotta di 1 ml / min. Il tag off spaccati si legherà alla colonna e la proteina bersaglio tagless sarà ora a flusso continuo. Raccogliere il flusso passante in un contenitore fresco.
- Lavare la colonna di Ni con 3 ml di tampone A seguito da 5 ml di tampone B per eluire tutto His-tagged proteine e non specifico legato. Raccogliere separatamente ogni frazione.
- Esegui SDS-PAGE di Ni2 flow-through, lavare e Ni2 frazioni di eluizione per verificare ULP1 scissione e che protein è presente nel flusso passante. Utilizzare un NanoDrop per misurare A 280 per determinare o meno la presenza di proteine.
13. Concentrando
- Concentrare il flusso passante Ni2 (e Ni2 eluizione se proteina è presente) a 5 ml con una Ultra 10 kDa MWCO provetta da centrifuga Amicon. Spin a intervalli di 10 min a 4000 rpm a 4 ° C. Mescolare con una pipetta tra ogni giro per evitare che le proteine da un'eccessiva concentrazione lungo la membrana.
14. Cromatografia ad esclusione dimensionale (SEC)
- Impostare una Sephacryl S-100 10/30 GL colonna (GE Healthcare) da equilibrare con 200 ml di tampone SEC ad una portata di 0,5 ml / min su un sistema AKTApurifier (GE Healthcare).
- Preparare 10 ml superloops per l'uso su colonna SEC in base alle istruzioni del produttore.
- Usando una siringa da 5 ml, caricare i campioni su superloops e iniziare la corsa SEC.
- Monitorare la traccia UV-assorbanza a 280 nm, mentre la raccolta piccolo volume frazioni.
- Esegui frazioni SEC tramite SDS-PAGE.
- PISCINA La SEC frazioni che mostrano le bande di intensità più elevati.
- Concentrato frazioni SEC in pool. Fare riferimento al punto 13.1.
- Proteine aliquota in 100 campioni microlitri, Flash congelare in azoto liquido e conservare a -80 ° C.
CRISTALLIZAZIONE
15. Proteine Cristallizzazione
- Pre-riempire ogni serbatoio di una piastra a 96 pozzetti cristallizzazione compatta Jr (Emerald Bio) con 80 ml di schermo cristallizzazione (Emerald Bio) di scelta.
- Diluire proteina con il dimensionamento del buffer di 2-20 mg / ml e conservare il ghiaccio.
- Dispensare 0.4 ml di proteine e 0,4 microlitri della schermata cristallizzazione in ciascuno dei 96 pozzi e coprire con cristallo trasparente nastro di tenuta (Manco).
- Conservare la piastra a 16 ° C durante il controllo per la cristallizzazione della proteina periodicamente nel corso delle prossime settimane sotto un microscopio da dissezione.
16. Cristallo raccolta
- Creare un crioprotettore dalle acque madri e glicole etilenico. Tagliare il nastro adesivo trasparente che copre il bene con il cristallo di proteina bersaglio. Per un pozzetto vuoto, aggiungere 1,6 microlitri della corrispondente condizione di cristallizzazione e si combinano con 0,4 ml di glicole etilenico producendo una concentrazione finale del 20% di glicole etilenico e 80% cristallizzando condizione. Nota: per ottimizzare diffrazione a cristallo provare diversi crioprotettori come: glicerolo, oli, glicoli di polietilene a bassa MW, e / o in percentuali variabili del crioprotettore.
- Prima della raccolta raffreddare un puck ALS-stile in un dewar pieno di azoto liquido e coprire con il coperchio.
- Raccogliere il cristallo ponendo un CryoLoop con il diametro interno adeguamento delle dimensioni del cristallo su una bacchetta di cristallo magnetico (Hampton Research) e scoop direttamente dalla soluzione bene.
- Immergere immediatamente il CryoLoop con il cristallo raccolto nel cryoprotectant poi immergere nella puck ALS-style a lampeggiare congelare il crystal. Ripetere per un numero desiderato di cristalli.
17. Collezione Screening / Dati di cristallo
- Una volta che la raccolta è completa usare una bacchetta puck per posizionare il disco magnetico crio coperchio sulla SLA puck. Con pinze piegate, capovolgere il disco a testa in giù.
- Trasferire il disco ad un Rigaku ATTORE dewar, avvitare un Pusher Puck sul disco, e perforare il coperchio lasciandolo nel dewar con perni a faccia in su.
- Utilizzando il software di JDirector, schermo ogni cristallo con i seguenti parametri: fessura fascio impostato a 0,5 gradi, la distanza rivelatore impostato a 50 mm, passo immagine a 70 gradi, e la lunghezza di esposizione impostato a 30 sec.
- Eseguire Mosflm sulle immagini di prova che hai scattato con JDirector per determinare quale sia la migliore strategia è di cristallo e per la raccolta dati.
- Raccogliere un set di dati completo, sulla base di risultati di Mosflm.
18. Elaborazione / Determinazione Struttura dati
- Esegui XDS / XSCALE 4 per elaborare il set di dati.
- Aprire il software CCP4 privato.
- Esegui Phaser 5 per calcolare una soluzione di sostituzione molecolare utilizzando un modello di ricerca ad alta omologia, se disponibile. In questo caso abbiamo usato il 3CW4 PDBID come modello di ricerca 6.
- Esegui Refmac 7 per affinare il modello molecolare contro la riflessione raccolti nel dataset. Risoluzione finale deve essere basato fuori del guscio più alta e determinata dai seguenti parametri: Fattore R> 50%, I / sigma> 2, e la completezza> 90%.
- Costruire un modello di densità elettronica 3-Dimensional con il software di grafica molecolare COOT 8.
- Prima di depositare la struttura nel PPB convalidare con un software MolProbity 9 per verificare la qualità della struttura è adatta per la deposizione.
Risultati
I seguenti risultati illustrano i risultati attesi del protocollo descritto, e nel caso delle PB2, i risultati osservati.
Utilizzando Gene compositore, cinque full-length bersaglio sequenze amminoacidiche del virus influenzale polimerasi subunità PB2 stati progettati (Figura 2). Le sequenze PB2 sono stati tradotti indietro e sottoposti a molte fasi di engineering 3, con conseguente codone sequenze armonizzate ottimizzati per l'espressione in E. coli. Dai prodotti IPCR (Figura 3b), per un totale di trentaquattro costrutti sono stati clonati con successo in un sistema modificato pET28 vettore 10 con un N-terminale 6x His-tag Smt fusione utilizzando PIPE clonazione 3 come mostrato in figura 3a. Una sintesi del flusso di lavoro clonazione è presentato in Figura 4.
Dopo la clonazione successo, l'espressione della proteina micro-scala di ciascun costrutto è stato testato in BL21 (DE3) E.coli. Le cellule sono state coltivate in terreno supplementato con TB Novagen Overnight Express 1 medio (secondo il protocollo del produttore) per 48 ore a 20 ° C in un incubatore set agitazione a 220 rpm. Dopo la crescita, le cellule sono state raccolte e testate per l'espressione della proteina solubile usando elettroforesi capillare con una pinza LabChip 90. Quattordici dei trentaquattro PB2 costrutti portato solubile proteina bersaglio ed è entrato fermentazione su larga scala. Colture su larga scala di ciascun costrutto sono state coltivate in terreno TB integrato con Overnight Express 1 media di Novagen secondo il protocollo del produttore. Dopo la crescita, le cellule sono state raccolte tramite centrifugazione e conservati a -80 ° C. Espressione proteica su larga scala di ogni coltura è stata confermata mediante analisi SDS-PAGE (Figura 5) prima di procedere con la purificazione su larga scala.
La proteina Maker è stato usato per condurre purificazione parallelo dei quattordici costrutti PB2. I lisati chiarito di all quattordici costrutti sono stati eseguiti attraverso una colonna di nichel-chelato. Dopo aver determinato quali frazioni conteneva proteina bersaglio mediante SDS-PAGE, le corrispondenti frazioni sono state miscelate per ciascun campione e la concentrazione di ciascun stato determinato da un Una lettura 280. Rimozione del tag His-Smt 6x è stato condotto con l'aggiunta di ULP1 seguita da dialisi durante la notte e una seconda colonna di nichel. La conferma della rimozione His-tag Smt stato condotto mediante SDS-PAGE (Figura 6), e ciascun campione è stato concentrato con un kDa Amicon Ultra tubo da centrifuga 10. Dopo la concentrazione utilizzando i tubi Ultra centrifuga Amicon, ogni campione è stato eseguito su una colonna dimensionamento per raggiungere la purezza cristallografica. Una seconda concentrazione è stato condotto per aumentare la concentrazione proteica al livello necessario per la cristallizzazione. Tutti i quattordici costrutti sono stati purificati con successo e sono entrati in prove di cristallizzazione.
La cristallizzazione è stata avviata da scongelamento la precedenza frproteine ozen. Cristallizzazione è stata eseguita in una camera climatica controllata a 16 ° C con piastre appositamente progettati smeraldo (Bio) per seduta goccia diffusione del vapore (Figura 7). Screening iniziale è stato condotto con quattro schermi a matrice sparsa; JCSG +, Patto, guidata completa, e CryoFull (Emerald Bio), a seguito di una strategia di Newman estesa. 0,4 ml di soluzione proteica è poi mescolato con 0,4 ml di crystallant (o soluzione serbatoio) dal serbatoio corrispondente utilizzando piastre di cristallizzazione Jr 96 pozzetti compatti (Emerald Bio). Dei quattordici campioni purificati nove di loro fruttato cristalli adatti per studi di diffrazione (Figura 8). Un in-house insieme di dati di diffrazione sono stati raccolti su cinque dei nove costrutti cristallizzati in Cu Kα lunghezza d'onda utilizzando un SuperBright FR-E generatore di raggi X + rotante-anodo Rigaku dotato Osmic VariMAX HF ottica e un 944 rilevatore di Saturno + CCD (Figura 9 ). Ogni set di dati è stato elaborato con XDS / XSCALE 4 < / Sup> e la scalata a una risoluzione definitiva. Tentativi di risolvere le strutture tramite sostituzione molecolare sono state effettuate con Phaser 5 dalla CCP4 Suite 7. I modelli finali sono stati ottenuti dopo affinamento in REFMAC 7 e ricostruzione manuale con Folaga 11. Le strutture sono stati valutati e corretti per la geometria e la forma fisica con MolProbity 9. Un totale di quattro strutture della subunità PB2 sono stati determinati (Figura 10) e depositato nel PPB. Figura 11 illustra il risultato complessivo in ogni fase della pipeline MTPP.

Figura 1. Panoramica del SSGCID via gene-per-struttura per l'elaborazione in parallelo multi-target a Emerald Bio.
Figura 2. Visualizzatore di allineamento e proteine Costruire Design del modulo nel software Composer Gene. Base di aminoacidi costrutto di destinazione viene visualizzato in verde (finestra centrale) e la struttura troncamenti guidate di costrutti alternativi sono indicati in oro (finestra in basso). L'allineamento di più sequenze virali influenzali PB2 è mostrato rispetto alla sequenza ed elementi di struttura secondaria del dominio C-terminale dal 3CW4 PDBID. La conoscenza della struttura di dominio e di elementi di struttura secondaria permette troncamenti N-terminale a scelta all'interno del Compositore Modulo Progettazione Gene facendo clic destro sul residuo desiderato aminoacido. Clicca qui per ingrandire la figura .
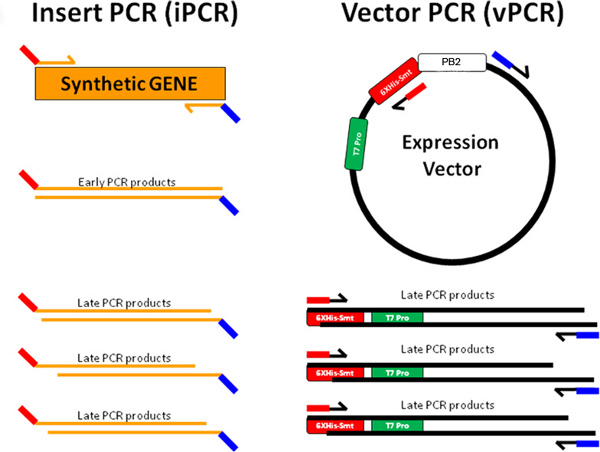
Figura 3a. PIPE clonazione è illustrato in cui l'inserto gene sintetico (arancione) è amplificata dal progettati in avanti (linee rosso-arancio) e inversa (linee arancione e blu) primer per generare inserire materiale PCR. Il vettore di espressione è amplificato con linee inverso (rosso-nero ) e in avanti (linee blu-nero) primer per generare vettore materiale PCR. Le sequenze terminali prodotti IPCR sono complementari alle sequenze terminali dei prodotti vPCR (rosso di IPCR complementi rosso di vPCR e blu di IPCR complementi blu di vPCR). In questo modo i prodotti IPCR e vPCR per temprare a formare plasmidi che vengono replicati su di trasformazione in serie BL21 (DE3) chimicamente competente E. coli.

Figura 3b. Agarosio analisi del gel di IPCR produzione ts dalla subunità PB2. fallimenti IPCR possono essere visti come bande deboli o untuosa, mentre i prodotti IPCR di successo sono rappresentati da bande robusti. IPCR qualità del prodotto può generalmente essere correlata con successo clonazione. Marcatori di peso molecolare sono in chilodalton. La figura è riprodotto da Raymond et al., 2011 12.

Figura 4. Gene fasi di engineering del bersaglio le proteine PB2 sono state eseguite utilizzando il software Composer Gene. Dopo la sequenza di acido nucleico di ingegneria è stato stabilito per ogni target, 6-7 costrutti proteici alternativi sono stati progettati per ciascuno. Elaborazione in parallelo multi-target nelle fasi iniziali di progettazione genetica e la clonazione ha provocato 34 costrutti, 14 dei quali erano gli obiettivi vitali che producevano proteine solubili in E. coli.
re 5 "src =" / files/ftp_upload/4225/4225fig5.jpg "/>
Figura 5. Rappresentante SDS-PAGE analisi di fermentazione su larga scala che mostra l'espressione della proteina robusto (dimensione prevista di 25,76 kDa), circa il 50% solubile (corsia 4) e circa il 50% scissione del tag 6x His-Smt da proteine eluite (corsia 7).
Figura 6. Risultati di SDS-PAGE per tre costrutti della polimerasi subunità PB2 corsia 1, marcatori di peso molecolare (etichettati a sinistra in kDa),. Corsie 2, 6, e 10, proteina aggregata da Nickel 1 colonna; corsie 3, 7 e 11, flusso continuo di proteine spaccati in tampone A da Nickel 2; corsie 4, 8, e 12, la rimozione del tag 6x His-Smt in tampone B da nichel 2.
d/4225/4225fig7.jpg "/>
Figura 7. Uno schema di diffusione del vapore con il metodo goccia seduta. Metodo goccia seduta per la cristallizzazione delle proteine rientra nella categoria di diffusione del vapore. Questo metodo comporta una campione purificato di proteine e precipitante equilibrare con un serbatoio più grande contenente condizioni simili in una concentrazione più alta. Come l'acqua evapora dal campione proteico e trasferisce al serbatoio, la concentrazione precipitante aumenta ad un livello ottimale per la cristallizzazione della proteina.

Figura 8. Cristallo di proteina PB2 subunità della polimerasi da un ceppo del virus influenzale.

Figura 9. Immagine di diffrazione di raggi X della polimerasi PB2 da una subunitàceppo del virus influenzale.

La figura 10. Diagrammi Ribbon delle molecole nell'unità asimmetrica cristallografica di 4 PB2 strutture. Strutture secondarie colorate nel modello arcobaleno con i corrispondenti codici PDB. (A) 3K2V (A/Yokohama/2017/2003/H3N2) (b) 3KHW (A / Messico / InDRE4487/2009/H1N1) (c) 3KC6 (A/Vietnam/1203/2004/H5N1) (d) 3L56 (A/Vietnam/1203/2004/H5N1).

Figura 11. Analisi di outcome per l'influenza PB2 obiettivi con i metodi descritti. L'structurpipeline di determinazione e è illustrato in cinque fasi: determinazione Clonazione, solubilità, purificazione, cristallizzazione e struttura.
Discussione
Multi-Target Parallel Processing
Structure-based drug design gioca un ruolo importante nella scoperta di nuovi farmaci. Il SSGCID è dedicato a fornire alla comunità scientifica con strutture proteiche a tre dimensioni da NIAID categoria patogeni AC. Rendere tali informazioni strutturali ampiamente disponibili in ultima analisi, servirà ad accelerare la struttura basata progettazione di farmaci.
Il primo passo fondamentale dell'approccio MTPP è il design costrutto. Molteplici i costrutti di ogni proteina bersaglio aumenta la probabilità di determinazione della struttura di successo e gli aumenti di turnaround. E 'inevitabile che alcuni costrutti proteici mancheranno durante fasi della pipeline. Attuazione del metodo di clonazione TUBO supporta il metodo MTPP consentendo la generazione di molti costrutti in formato a 96 pozzetti senza lavoro intensivo fasi di purificazione. TUBO clonazione abbinamento con la capacità di analizzare l'espressione della proteina nello stesso formato a 96 pozzetti (Pinza LabChip 90) accelera ulteriormente il flusso complessivo. L'accoppiamento di questi metodi consente una rapida identificazione di costrutti che producono proteina solubile che assicura il successo della produzione di proteina su larga scala e purificazione.
Un aspetto essenziale per il successo del MTPP alta produttività è la proteina Maker (US Patent No. 6.818.060, smeraldo Bio) strumento. La proteina Maker è un sistema di cromatografia liquida-parallelo 24 canali sviluppata specificamente per aumentare l'efficienza della produzione di proteine ad alta velocità e relative applicazioni strutturali genomiche di ricerca condotte. Utilizzando il protocollo precedentemente descritto per la proteina Maker, i vantaggi sono evidenti in confronto ad un unico sistema FPLC riga. Una singola persona può purificare fino a 48 bersagli in parallelo all'interno di un periodo di otto ore. Al contrario, una sola persona con un unico sistema FPLC linea può purificare solo un massimo di quattro bersagli entro il medesimo termine. Gli alti livelli di purezza per ogni targetrealizzato con la proteina Maker sono un fattore critico nel successo di seguito la crescita di cristalli di proteine per l'analisi della struttura.
Limitazioni e risoluzione dei problemi
Solving strutture tridimensionali da cristallografia a raggi x è uno sforzo plurifase con molte sfide, uno dei quali è l'impossibilità di ottenere grandi quantità di proteina solubile bersaglio. Una strategia che può essere implementato per superare il problema solubilità è l'uso di un host espressioni alternative come E. coli sono in grado di eseguire diversi importanti eucariotici modificazioni post-traduzionali. Espressione in varie lievito, linee cellulari di insetto e di mammifero, che sono in grado di eseguire queste modificazioni post-traduzionali sono spesso una valida alternativa. Proteine bersaglio a volte sono espressi, ma completamente insolubile in condizioni di lisi standard. La proteina Maker può essere una risorsa preziosa per il test rapido di condizioni alternative lisi cellularecome descritto in Smith et al. 2011 13. Questa strategia è spesso necessario per mantenere bersagli in movimento attraverso la conduttura. In ogni pipeline di genomica strutturale, protocolli standardizzati possono non essere adatti per ogni obiettivo che passa attraverso la pipeline e gli obiettivi possono avere bisogno di ottimizzazione individuale. Per esempio, abbiamo scelto di utilizzare il 20% di glicole etilenico per ogni cryoprotectant. Nei casi in cui questa condizione non è adatto, crioprotettivi alternativi o concentrazioni possono avere bisogno di essere testati.
A causa della natura unica di ogni singolo obiettivo della proteina, il fattore limitante e step imprevedibili nella determinazione di una struttura è cristallizzazione. Gli offset conduttura MTPP il tasso di successo comunemente basso di cristallizzazione delle proteine con l'ottimizzazione delle schermate iniziali matrici sparse. Ogni cristallo iniziale colpito dagli schermi matrici sparse in commercio è ulteriormente ottimizzata con un Builder E-Screen (Emerald Bio). La schermata di ottimizzazione è progettato around la condizione della hit cristallo iniziale, alterando le concentrazioni dei tamponi, sali e additivi. Schermate di ottimizzazione di successo producono cristalli adatti per studi di diffrazione e la determinazione della struttura.
Il programma di genomica strutturale messo avanti dall'Istituto Nazionale di allergie e malattie infettive (NIAID) finanzia Emerald Bio e di altri tre istituti del Nord-Ovest del Pacifico, che insieme sono la SSGCID (Emerald Bio, SeattleBiomed, l'Università di Washington e il Pacific Northwest National Laboratory) . Ciascun membro del consorzio è stato scelto per la loro esperienza nell'applicazione di tecnologie state-of-the-art necessari per realizzare gli obiettivi del programma di genomica strutturale NIAID. Ad oggi, SSGCID ha depositato 461 strutture nel ranking PDB come il settimo più grande contributore al mondo, e nel 2011, il più produttivo. I protocolli e le metodologie della SSGCID sono forniti con l'intenzione di beneficiare delcomunità scientifica e perpetuare la ricerca di malattie infettive.
Divulgazioni
Gli autori sono dipendenti di Emerald Bio, Inc.
Riconoscimenti
Gli autori desiderano ringraziare tutti i membri del consorzio SSGCID. Raggiungimento degli obiettivi del SSGCID è reso possibile dagli enormi sforzi di tutti i membri del team a Emerald Bio. Questa ricerca è stata finanziata nell'ambito federale contratto n HHSN272200700057C presso l'Istituto Nazionale di allergie e malattie infettive, il National Institutes of Health e il Dipartimento di Salute e Servizi Umani.
Materiali
| Name | Company | Catalog Number | Comments |
| Primers | IDT | ||
| Genes | DNA 2.0 | ||
| TE buffer | Qiagen | provided in kit | |
| 96-well half skirt PCR plates | VWR | 10011-248 | |
| PFU Master Mix | |||
| 6X Orange Loading Dye | Fermentas | R0631 | |
| 10X TAE | Teknova | T0280 | |
| Agarose | Sigma-Aldrich | A9414-10G | |
| pET28 vector | |||
| 2-YT Broth | VWR | 101446-848 | |
| Kanamycin | Teknova | K2151 | |
| Restriction Enzymes | Fermentas | ||
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Top 10 chemically comp cells | Invitrogen | C4040-06 | |
| Disposable Troughs (Sterile, 25 ml) | VWR | 89094-662 | |
| Airpore covers (Rayon films for bio cultures) | VWR | 60941-086 | |
| 24-well blocks | VWR | 13503-188 | |
| QIAvac 96 | Qiagen | 19504 | |
| BL21(DE3) cells chemcomp (phageR) | NEB | C2527H | |
| 50% Glycerol | VWR | 100217-622 | |
| TB Media | Teknova | T7060 | |
| IPTG | Sigma-Aldrich | ||
| 1 M Tris pH 8.0 | Mediatech | 46-031-CM | |
| 5 M NaCl | Teknova | S0251 | |
| Glycerol | Aldrich | G7893-4L | |
| CHAPS | JT Baker | 4145-01 | |
| Imidazole | Sigma | 56749-1KG | |
| TCEP | Amresco | K831-10G | |
| L-arginine | Amresco | 0877-500G | |
| Benzonase | EMD | 70746-3 | |
| Lysozyme | USB | 1864525GM | |
| 10 kDa MWCO dialysis tubing | Thermo | 68100 | |
| Amicon Ultra 10 ka MWCO concentrators | Millipore | UFC901024 | |
| HisTrap FF columns | GE | 17-5255-01 | |
| HiTrap Chelating columns | GE | 17/0408-01 | |
| Compact Jr crystallization plates | Emerald Bio | EBS-XJR | |
| Crystalization screens | Emerald Bio | ||
| Ethylene Glycol 100% | Emerald Bio | EBS-250-EGLY | |
| Crystal Wand Magnetic Straight | Hampton Research | HR4-729 | |
| Mounted CryoLoop 0.1-0.2 mm | Hampton Research | HR4-955 | |
| ALS style puck | |||
| Puck Wand | |||
| Bent Tongs | |||
| Puck Pusher |
Riferimenti
- Lowen, A. C., Mubareka, S., Steel, J., Palese, P. Influenza virus transmission is dependent on relative humidity and temperature. PLoS Pathogens. 3 (10), 1470-1476 (2007).
- Yamada, S., et al. Biological and structural characterization of a host-adapting amino acid in influenza virus. PLoS Pathog. 6, e1001034 (2010).
- Lorimer, D., Raymond, A., Walchli, J., Mixon, M., Barrow, A., Wallace, E., Grice, R., Burgin, A., Gene Stewart, L. Composer: database software for protein construct design, codon engineering, and gene synthesis. BMC Biotechnol. 9, 36 (2009).
- Kabsch, W. Integration, scaling, space-group assignment and post-refinement. Acta Cryst. D. 66, 125-132 (2010).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. J. Phaser crystallographic software. Appl. Cryst. 40, 658-674 (2007).
- Kuzuhara, T., Kise, D., et al. Structural basis of the influenza A virus RNA polymerase PB2 RNA-binding domain containing the pathogenicity-determinant lysine 627 residue. J. Biol. Chem. 284, 6855-6860 (2009).
- Murshudov, G. N., Skubàk, P., Lebedev, A. A., Pannu, N. S., Steiner, R. A., Nicholls, R. A., Winn, M. D., Long, F., Vagin, A. A. REFMAC5 for the refinement of macromolecular crystal structures. Acta Cryst. D. 67, 355-367 (2011).
- Cowtan, K. Recent developments in classical density modification. Acta Cryst. D. 66, 470-478 (2010).
- Chen, V. B., Arendall, W. B., Headd, J. J., Keedy, D. A., Immormino, R. M., Kapral, G. J., Murray, L. W., Richardson, J. S., Richardson, D. C. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Cryst. D. 66, 12-21 (2010).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Mol. Cell. 5, 865-876 (2000).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Cryst. D. 66, 486-501 (2010).
- Raymond, A. C., Haffner, T. E., Ng, N., Lorimer, D., Staker, B. L., Stewart, L. J. Gene design, cloning and protein-expression methods for high-value targets at the Seattle Structural Genomics Center for Infectious Disease. Acta Cryst. F. 67, 992-997 (2011).
- Smith, E. R., Begley, D. W., Anderson, V., Raymond, A. C., Haffner, T. E., Robinson, J. I., Edwards, T. E., Duncan, N., Gerdts, C. J., Mixon, M. B., Nollert, P., Staker, B. L., Stewart, L. J. The Protein Maker: an automated system for high-throughput parallel purification. Acta Cryst. F. 67, 1015-1021 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon