È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Cattura selettiva di 5 hydroxymethylcytosine dal DNA genomico
In questo articolo
Riepilogo
Descritto è un processo a due fasi usando etichettatura β-glucosil (β-GT) per trasferire una azide-glucosio al 5-HMC, seguita dalla chimica click per trasferire un linker biotina per l'arricchimento facile e densità-indipendenti. Questo metodo efficace di etichettatura e specifica consente l'arricchimento di 5-HMC con fondo estremamente basso e high-throughput mappatura epigenomic tramite sequenziamento di prossima generazione.
Abstract
5-metilcitosina (5-mC) costituisce ~ 2-8% dei cytosines totali in DNA genomico umano e influisce un'ampia gamma di funzioni biologiche, tra l'espressione genica, il mantenimento dell'integrità genomica, imprinting parentale, inattivazione del cromosoma X, regolazione sviluppo, invecchiamento e cancro 1. Recentemente, la presenza di un ossidato 5-mC, 5-hydroxymethylcytosine (5-HMC), è stato scoperto in cellule di mammifero, in particolare nel staminali embrionali (ES) cellule e cellule neuronali 2-4. 5-HMC è generato per ossidazione di 5-mC catalizzata da TET famiglia ferro (II) / α-chetoglutarato-dipendente diossigenasi 2, 3. 5-HMC si propone di essere coinvolto nel mantenimento di staminali embrionali (MES) delle cellule, emopoiesi normale e le neoplasie, e zigote sviluppo 2, 5-10. Per capire meglio la funzione di 5-HMC, un sistema di sequenziamento affidabile e semplice è essenziale. Sequenziamento bisolfito tradizionale non è possibile distinguere 5-HMC da 5-MC 11 12.
Qui si descrive una semplice procedura in due fasi per l'etichettatura chimica selettiva di 5-HMC. Nella prima fase di etichettatura, 5-HMC nel DNA genomico viene etichettato con un 6-catalizzata azide-glucosio da β-GT, un glucosiltransferasi dal batteriofago T4, in modo che trasferisce il 6-azide-glucosio al 5-HMC dal modificato cofattore, UDP-6-N3-Glc (6-N3UDPG). Nella seconda fase, biotinilazione, un linker biotina disolfuro è attaccato al gruppo azide dalla chimica click. Entrambe le fasi sono altamente specifici ed efficienti, portando a completare etichettatura indipendentemente l'abbondanza di 5-HMC in regioni genomiche e dando fondo estremamente bassa. Biotinilazione seguito di 5-HMC, i 5-HMC contenenti frammenti di DNA vengono selettivamente catturatiutilizzando perline streptavidina in una densità modo indipendente. La risultante 5-HMC arricchiti frammenti di DNA potrebbe essere utilizzato per analisi a valle, sequenziamento di prossima generazione.
La nostra etichettatura selettiva e il protocollo di acquisizione conferisce elevata sensibilità, applicabile a qualsiasi fonte di DNA genomico con variabili / 5-HMC diverse abbondanze. Anche se lo scopo principale di questo protocollo è la sua applicazione a valle (ad es., Sequenziamento di prossima generazione per tracciare il 5-HMC distribuzione nel genoma), è compatibile con singola molecola, in tempo reale SMRT (DNA) sequenziamento, che è in grado di erogare singola base sequenziamento risoluzione del 5-HMC.
Protocollo
1. Frammentazione del DNA genomico
Frammento di DNA genomico mediante ultrasuoni ad una vasta dimensione desiderata adatto per il genoma a livello di piattaforma di sequenziamento. (Di solito sonicazione a ~ 300 bp.) Verificare la distribuzione dimensionale del DNA genomico frammentato 1% gel (Figura 1).
2. Estrazione del DNA
Determinare le quantità di DNA di partenza in base alla abbondanza di 5-HMC nel DNA genomico. Dal 5-HMC livelli variano in modo significativo in diversi tipi di tessuto, quantità di DNA di partenza dipendono dalle 5-HMC livelli dei campioni. Si prega di fare riferimento alla Tabella 1 per gli esempi.
3. β-GT reazione catalizzata (reazione di trasferimento del glucosio)
- Mescolare pipettando la miscela come descritto in Tabella 2 e incubare a 37 ° C in bagno di acqua per 1 ora.
- Dopo l'incubazione, pulire la reazione con Kit Rimozione QIAquick Nucleotide, con 10 microgrammi di DNA percolonna. Eluire con 30 microlitri di acqua per colonna e combinare.
4. Biotinilazione Reazione (Click Chemistry)
- Aggiungere DBCO-SS-PEG3-Biotin coniugato soluzione di lavoro (1 mM) nella soluzione di DNA eluito (punto 3) ad una concentrazione finale di 150 pM (cioè 5 ml di soluzione di lavoro per 30 microlitri soluzione di DNA).
- Mescolare pipettando e incubare a 37 ° C bagno di acqua per 2 ore.
- Pulire la reazione con Kit Rimozione QIAquick Nucleotide. Il volume ideale di eluizione totale è di 100 ul.
- Quantificare la quantità di DNA recuperato mediante spettrofotometro scala microlitro (ad es., NanoDrop).
5. Cattura di 5-HMC contenente DNA
- Lavare 50 pl di Dynabeads MyOne streptavidina C1 3 volte con 1 ml di 1X B & W tampone secondo le istruzioni del produttore. Separare le perline con un supporto magnetico.
- Aggiungere il volume uguale di 2X B & W per il buffer di recupero D biotinilatoNA (100 ul) ai talloni lavate.
- Incubare per 15 minuti a temperatura ambiente con lieve rotazione su un rotatore.
- Separare le perline con un supporto magnetico e lavare le microsfere 3 volte con 1 ml di tampone 1X B & W.
- Eluire il DNA incubando le perle in 100 pl di preparati DTT 50 mM per 2 ore a temperatura ambiente con lieve rotazione su un rotatore.
- Separare le perline con un supporto magnetico. Aspirare il eluente e carico su un Micro Bio-Spin 6 Colonna secondo le istruzioni fabbricazione per rimuovere il DTT. Il DNA bersaglio è nella soluzione ora.
- Purificare il DNA eluito dal passaggio precedente Purification Kit Qiagen MinElute PCR e DNA eluire in 10 microlitri di tampone EB. Quantificare DNA utilizzando fluorimetro Qubit, NanoDrop o se la concentrazione è superiore a 20 ng / ul. Il DNA è pronto per la valle genome-wide preparazione sequenziamento biblioteca.
6. Risultati rappresentativi
Se la qualità of DNA genomico è alta, i rendimenti tipici di recupero dopo il β-GT e le reazioni sono biotinilazione ~ 60-70%. Tuttavia, l'efficienza di cattura variare significativamente diversi tipi di tessuto a seconda delle 5-HMC livelli dei campioni. Tipicamente, l'efficienza di cattura per il DNA genomico è cervello ~ 4-9%, e in alcuni casi estremi, l'efficienza può arrivare fino al 12%. Per le cellule ES, l'efficienza di cattura medio è ~ 2-4%, in contrasto con ~ 0,5% per le cellule staminali neuronali. Il più basso rendimento visto finora è stato per il DNA genomico da cellule tumorali. Tutto DNA arricchito è pronto per i protocolli della libreria standard di nuova generazione di preparazione. Inoltre, il DNA catturato può anche essere usato come stampo per PCR in tempo reale per rilevare l'arricchimento di alcuni frammenti rispetto al DNA di ingresso, se i primers correlati sono disponibili.
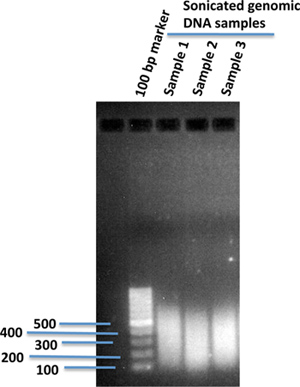
Figura 1. Sonicati umani frammenti di DNA genomico in1% gel. 10 ug di DNA genomico isolato da cellule iPS umane in 120 pl di tampone TE 1X stata sonicata utilizzando un dispositivo di sonicazione (Covaris). Dopo sonicazione, 2 microlitri del DNA sonicato viene caricata su gel di agarosio 1% usando 100 bp di DNA marcatore per confrontare le dimensioni dei frammenti di DNA sonicato.
| Componente | Volume | Concentrazione finale |
| Acqua | _ Pl | |
| 10 X β-GT Reaction Buffer | 2 microlitri | 1 X |
| Fino a 10 microgrammi di DNA genomico | _ Pl | Fino a 500 ng / pl |
| UDP-6-N 3-Glc (3 mM) | 0,67 pl | 100 pM |
| β-GT (40 pM) | 1 ml | 2 pM |
| Volume totale | 20 pl |
i) Per il DNA genomico del tessuto (alta 5-HMC contenuto> 0,1%)
| Componente | Volume | Concentrazione finale |
| Acqua | _ Pl | |
| 10 X β-GT Reaction Buffer | 10 pl | 1 X |
| Fino a 20 microgrammi di DNA genomico | _ Pl | Fino a 500 ng / pl |
| UDP-6-N3-Glc (3 mM) | 1,33 pl | 100 pM |
| β-GT (40 pM) | 2 microlitri | 2 pM |
| Volume totale | 40 ml |
ii) Per il DNA genomico di cellule staminali (mediana 5 HMC contenuti ~ 0,05%)
| Componente | Volume | Concentrazione finale |
| Acqua | _ Pl | |
| 10 X β-GT Reaction Buffer | 10 pl | 1 X |
| Fino a 50 microgrammi di DNA genomico | _ Pl | Fino a 500 ng / pl |
| UDP-6-N3-Glc (3 mM) | 3,33 pl | 100 pM |
| β-GT (40 pM) | 5 microlitri | 2 pM |
| Volume totale | 100 pl |
iii) Per il DNA genomico delle cellule del cancro (basso 5-HMC contenuti ~ 0,01%)
Tabella 1. Esempi di quantità di DNA di ingresso e di reazioni etichettatura utilizzando i campioni con diversi livelli di 5-HMC secondo il metodo chimico selettivo etichettatura.
| Campione | 5-HMC livello | DNA di partenza (mg) | Il recupero dopo l'etichettatura (ingresso a sfere) (mg) | Recupero resa | Pull-down DNA (ng) | Pull-down cedere |
| Del mouse cervelletto adulto | 0,4% | 10 | 7,5 | 75% | 236 | 3,1% |
| Postnatale giorno 7 del mouse cervelletto | 0,1% | 11 | 9 | 82% | 140 | 1,6% |
| Topo ES cellule E14 | 0,05% | 60 | 42 | 70% | 350 | 0,8% |
Tabella 2. Risultati rappresentativi di tessuti cerebrali di topo e cellule ES.
Discussione
5-hydroxymethylcytosine (5-HMC) è un regalo recentemente identificato modificazione epigenetica in quantità notevoli in alcuni tipi di cellule di mammifero. Il metodo qui presentato è per la determinazione del genoma distribuzione a livello di 5-HMC. Usiamo batteriofago T4 β-glucosil trasferire una porzione multistrato glucosio contenente un gruppo azide sul gruppo ossidrile di 5-HMC. Il gruppo azide può essere chimicamente modificato con biotina per il rilevamento, l'arricchimento di affinità, e sequenziament...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo studio è stato sostenuto in parte dal National Institutes of Health (GM071440 a CH e NS051630/MH076090/MH078972 a PJ).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome | Azienda | Catalogare # | Commento |
| Reagenti | |||
| Cloruro di sodio 5M (NaCl) | Promega | V4221 | |
| 0.5M pH8.0 Etilendiammintetraacetato (EDTA) | Promega | V4231 | |
| 1M Trizma base (Tris) pH7.5 | Invitrogen | 15567-027) | |
| HEPES 1M, pH 7,4 | Invitrogen | 15630 | |
| Cloruro di magnesio (MgCl2) 1M | Ambion | AM9530G | |
| Dimetilsolfossido (DMSO) | Sigma | D8418 | |
| Tween 20 | Fisher BioReagents | BP337-100 | |
| DBCO-SS-PEG3-biotina coniugato | Fare clic su Strumenti Chimica | A112P3 | |
| 1,4-Dithiothreitol, ultrapura (DTT) superpuro | Invitrogen | 15508-013 | |
| QIAquick Kit Rimozione Nucleotide | Qiagen | 28304 | |
| Micro Bio-Spin 6 Colonna | Bio-Rad | 732-6222 | |
| Dynabeads MyOne | Invitrogen | 650-01 | |
| Streptavidina C1 | |||
| Qiagen MinElute PCR Purification Kit | Qiagen | 28004 | |
| Ultrapura agarosio | Invitrogen | 16500500 | |
| UDP-6-N 3-glucosio | Motif attivo | 55013 | |
| Enzima | |||
| β-glucosil (β-GT) | New England Biolab | M0357 | |
| Attrezzatura | |||
| Sonicazione dispositivo | Covaris | ||
| Desktop centrifuga | |||
| Bagnomaria | Fisher Scientific | ||
| Gel apparecchi in esecuzione | Bio-Rad | ||
| NanoDrop1000 | Thermo Scientific | ||
| Labquake Tubo Shaker | Barnstead | ||
| Labquake Tubo Shaker | Thermolyne | ||
| Separazione supporto magnetico | Promega | Z5342 | |
| Qubit 2,0 fluorimetro | Invitrogen | ||
| Reagente di installazione 10 X β-GT Reaction Buffer (500 mM HEPES pH 7,9, 250 mM MgCl2) 2 X Legatura e lavaggio (B & W) buffer (10 mM Tris pH 7.5, 1 mM EDTA, 2 M NaCl, 0,02% Tween 20) . |
Riferimenti
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. , 245-254 (2003).
- Ito, S. Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification. Nature. 466, 1129-1133 (2010).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Kriaucionis, S., Heintz, N. The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain. Science. 324, 929-930 (2009).
- Ko, M. Impaired hydroxylation of 5-methylcytosine in myeloid cancers with mutant TET2. Nature. 468, 839-843 (2010).
- Koh, K. P. Tet1 and tet2 regulate 5-hydroxymethylcytosine production and cell lineage specification in mouse embryonic stem cells. Cell Stem Cell. 8, 200-213 (2011).
- Iqbal, K., Jin, S. G., Pfeifer, G. P., Szabo, P. E. Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine. Proceedings of the National Academy of Sciences of the United States of America. 108, 3642-3647 (2011).
- Wossidlo, M. 5-Hydroxymethylcytosine in the mammalian zygote is linked with epigenetic reprogramming. Nat. Commun. 2, 241 (2011).
- Gu, T. P. The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes. Nature. 477, 606-610 (2011).
- Dawlaty, M. M. Tet1 is dispensable for maintaining pluripotency and its loss is compatible with embryonic and postnatal development. Cell Stem Cell. 9, 166-175 (2011).
- Huang, Y. The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing. PLoS One. 5, e8888 (2010).
- Song, C. X. Selective chemical labeling reveals the genome-wide distribution of 5-hydroxymethylcytosine. Nat. Biotechnol. 29, 68-72 (2011).
- Pastor, W. A. Genome-wide mapping of 5-hydroxymethylcytosine in embryonic stem cells. Nature. 473, 394-397 (2011).
- Matarese, F., Pau, C. a. r. r. i. l. l. o. -. d. e. S. a. n. t. a., E, ., Stunnenberg, H. G. 5-Hydroxymethylcytosine: a new kid on the epigenetic block. Mol. Syst. Biol. 7, 562 (2011).
- Szwagierczak, A., Bultmann, S., Schmidt, C. S., Spada, F., Leonhardt, H. Sensitive enzymatic quantification of 5-hydroxymethylcytosine in genomic DNA. Nucleic Acids Res. 38, 181 (2010).
- Terragni, J., Bitinaite, J., Zheng, Y., Pradhan, S. Biochemical characterization of recombinant β-glucosyltransferase and analysis of global 5-hydroxymethylcytosine in unique genomes. Biochemistry. , (2012).
- Rusmintratip, V., Sowers, L. C. An unexpectedly high excision capacity for mispaired 5-hydroxymethyluracil in human cell extracts. Proc. Natl. Acad. Sci. U.S.A. 97, 14183-14187 (2000).
- Globisch, D. Tissue distribution of 5-hydroxymethylcytosine and search for active demethylation intermediates. PLoS One. 5, e15367 (2010).
- Yildirim, O. Mbd3/NURD Complex Regulates Expression of 5-Hydroxymethylcytosine Marked Genes in Embryonic Stem Cells. Cell. 147, 1498-1510 (2011).
- Szulwach, K. E. Integrating 5-hydroxymethylcytosine into the epigenomic landscape of human embryonic stem cells. PLoS Genet. 7, e1002154 (2011).
- Szulwach, K. E. 5-hmC-mediated epigenetic dynamics during postnatal neurodevelopment and aging. Nat. Neurosci. 14, 1607-1616 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon