Method Article
Accelerato radiazione Biodosimetry automatizzato di identificazione del cromosoma dicentrico (ADCI) e stima della Dose
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'analisi citogenetica del cromosoma dicentrico (DC) quantifica l'esposizione a radiazioni ionizzanti. Il software automatizzato di identificatore di cromosoma dicentrico e l'estimatore di Dose con precisione e rapidamente stima dose biologica da DCs in cellule di metafase. Esso distingue monocentric cromosomi e altri oggetti da DCs e le stime di dose di radiazione biologica dalla frequenza di DCs.
Abstract
Dose di radiazione biologica può essere stimato da frequenze di cromosoma dicentrico in cellule di metafase. L'esecuzione di queste analisi citogenetica del cromosoma dicentrico è tradizionalmente un processo manuale e laborioso, non adatto per gestire il volume di campioni che possono richiedere l'esame a seguito di un evento di massa dell'incidente. Software automatizzato di identificatore di cromosoma dicentrico e Dose Estimator (ADCI) automatizza questo processo esaminando insiemi di immagini di metafase utilizzando tecniche di elaborazione immagine basata su apprendimento di macchina. Il Seleziona software immagini appropriate per analisi rimuovendo immagini inadatti, classifica ogni oggetto come un centromero-contenenti del cromosoma o non-cromosoma, più ulteriormente contraddistingue i cromosomi come monocentric cromosomi (MCs) o dicentrico cromosomi (DCs), determina la frequenza DC all'interno di un campione e le stime di dose di radiazione biologica confrontando la frequenza di campionamento DC con curve di calibrazione calcolate utilizzando campioni di calibrazione. Questo protocollo viene descritto l'utilizzo del software ADCI. In genere, sia calibrazione (dose nota) e insiemi di test (sconosciuto dose) di immagini di metafase sono importati per eseguire la stima accurata dose. Immagini ottimali per l'analisi possono essere trovati automaticamente utilizzando filtri di immagine preimpostati o possono anche essere filtrati attraverso ispezione manuale. Il software elabora le immagini all'interno di ogni campione e frequenze DC vengono calcolate a diversi livelli di rigore per la chiamata a controller di dominio, utilizzando un'approccio di apprendimento della macchina. Curve di taratura lineare-quadratica vengono generate in base sulle frequenze di DC in campioni di calibrazione esposti a dosi fisiche conosciute. Dosi di campioni esposti a livelli di radiazioni incerto sono stimate da loro frequenze DC utilizzando queste curve di calibrazione. Rapporti possono essere generati su richiesta e forniscono la sintesi dei risultati di uno o più campioni, di una o più curve di calibrazione o di stima di dose.
Introduzione
Radiazione biodosimetry utilizza marcatori biologici, principalmente le aberrazioni cromosomiche quali cromosomi dicentrici (DCs) e traslocazioni cromosomiche per misurare dosi di radiazioni che gli individui sono esposti a. Una dose biologicamente assorbita potrebbe essere diversa dalla dose fisica misurata dagli strumenti a causa della variabilità tra individui. Allo stesso modo, radiazione di una certa dose di fisica può produrre diverse esposizioni biologiche dovute a sottostanti condizioni fisiologiche o ambientali. Conoscenza della dose biologica è di particolare importanza per la diagnosi ed il trattamento.
Il dosaggio di DC è il gold standard della organizzazione mondiale della sanità (OMS) e Agenzia internazionale per energia atomica (AIEA) per valutare l'esposizione a radiazioni biologico nelle persone. E ' stato il primo test consigliato dall'AIEA e che per la valutazione della dose di radiazione. Frequenza DC è relativamente stabile per circa 4 settimane dopo l'esposizione di radiazioni1 e loro correlazione quantitativa con la dose di radiazione emessa è accurata, che rendono DCs il biomarcatore ideale. Il rapporto tra la dose di radiazione (a cui fa riferimento in unità Gray [Gy]) e la frequenza di DC (indicato come numero di DCs per cella) può essere espressa come una funzione lineare-quadratica.
L'analisi citogenetica di DC è stato lo standard industriale per circa 55 anni2. È stata eseguita manualmente, che richiedono 1-2 giorni per analizzare i dati di microscopio da un campione di sangue. Diverse centinaia a diverse migliaia di immagini sono necessarie per stimare con precisione l'esposizione a radiazioni a seconda della dose3. A dosi superiori a 1 Gy, AIEA raccomanda un minimo di 100 DCs essere rilevato. Esame di 250-500 immagini di metafase è pratica comune nei laboratori di citogenetica biodosimetry. Per campioni con esposizioni < 1 Gy, 3.000-5.000 immagini sono suggeriti dovuto la probabilità inferiore di formazione DC. In entrambi i casi, è un'attività intensa di lavoro.
Laboratori di citogenetica biodosimetry creano proprio in vitro le curve di calibrazione biodosimetry di radiazione prima di valutare le dosi biologiche in campioni di prova. I campioni di sangue da individui di controllo normali, sono esposti a radiazioni e linfociti sono quindi colta e preparati per l'analisi del cromosoma di metafase. Utilizzando questi campioni, hanno ricevute le dosi biologiche sono tarate per il note fisiche dosi emesse da una sorgente di radiazione standard. Dopo immagini delle cellule di metafase sono stati registrati, esperti esaminare immagini, contano DCs e calcolare le frequenze DC per ogni campione. Una curva di calibrazione è costruita montando una curva lineare-quadratica per le frequenze di DC a tutte le dosi. Quindi, l'esposizione in esempio di test da individui possono essere dedotte abbinando le frequenze DC per le dosi calibrate sulla curva o specificandoli nella formula quadratica lineare corrispondente.
Ci hanno automatizzato sia il rilevamento dei controller di dominio e determinazione per accelerare questa procedura utilizzando il software della dose. Identificatore di cromosoma dicentrico automatizzati e Dose Estimator (ADCI) utilizza immagine di macchina basata su apprendimento tecniche di lavorazione per rilevare e discriminare i cromosomi dicentrici (DCs) da monocentric cromosomi (MCs) e altri oggetti e automatizza radiazione stima della dose. Il software mira a significativamente ridurre o eliminare la necessità per la verifica manuale dei conteggi di DC e ad accelerare la stima della dose tramite l'automazione. Esso è stato sviluppato con il coinvolgimento dei laboratori di riferimento biodosimetry a salute Canada (HC) e laboratori nucleari canadesi (CNL). Loro feedback farà in modo che le prestazioni continuerà a soddisfare criteri di AIEA per questo test.
Il software svolge le seguenti funzioni: 1) filtro DCs e selezionando immagini cellulare ottimale metafase per analisi, riconoscimento 2) cromosoma, rilevamento di DC e DC frequenza determinazione e 3) stimare la dose di radiazioni da dose-calibrato, dati relativi alla radiazione citogenetica. Questo software elabora gruppi di immagini di metafase da stesso individuo (definiti un campione), conta il numero di controller di dominio in ogni utilizzo immagine tecniche di lavorazione e restituisce la dose di radiazione stimato ricevuta da ciascun campione in unità di grigi (Gy).
Il software è stato progettato per gestire una gamma di strutture cromosomiche, conteggi e densità. Tuttavia, l'algoritmo esegue in modo ottimale nelle immagini di metafase che contiene un complemento completo nei pressi di cromosomi ben separati, lineare4. Immagini che contengono altamente sovrapposti set di cromosomi, più cellule, cellule in metafase incompleta, sorella cromatidio della separazione, i nuclei, oggetti non-cromosomici e altri difetti possono ridurre la precisione dell'algoritmo. Modelli di selezione immagine e altri segmentazione oggetto soglie possono filtrare la maggior parte delle immagini sub-ottimale e falsi positivi DCs dedicati.
Cromosoma dicentrico rilevamento viene eseguito quando viene elaborata un'immagine. L'algoritmo tenta di determinare quali oggetti in un'immagine sono i cromosomi e quindi individua le due regioni più probabile di essere centromeri su ogni cromosoma. Quindi, una serie di supporto differenti Vector Machine (SVM) modelli di apprendimento distinguere cromosomi come DCs o normale, monocentric cromosomi. I modelli SVM differiscono nella sensibilità e specificità di rilevazione DC (Vedi punto 3.1.4 qui sotto), che può interessare le frequenze di DC che sono determinate in un campione.
ADCI elabora set di Giemsa - (o DAPI-) tinto immagini digitali di metafase (in formato TIFF o JPG) per uno o più campioni. Il software analizza DCs in entrambi esempi di taratura e prova. Le dosi di fisiche (in Gy) dei campioni di calibrazione sono noti e sono utilizzate nella generazione di una curva di calibrazione. Le dosi fisiche e biologiche degli individui con le esposizioni sconosciute vengono dedotti dal software dalla curva di calibrazione generato dal computer. Anche se laboratori utilizzano tecniche comparabili, le curve di calibrazione da diversi laboratori variano spesso3. Entrambi esempi di curva e test di calibrazione dal laboratorio stesso devono essere elaborati per la stima della dose esatta in campioni di prova.
Questo software offre velocità, precisione e scalabilità quali indirizzi la produttività necessaria per gestire un evento in cui molti individui contemporaneamente devono essere testati. E ' stato sviluppato dal 2008-20174,5,6,7,8,9,10,11,12 ,13. Utilizzando hardware computer recente, questo desktopSoftware per PC in grado di elaborare e dose di radiazioni di stima in un campione di 500 equivalenti di genoma di metafase in 10-20 min 4. Il codice è basato su un insieme di segmentazione di immagini proprietarie e machine learning algoritmi per l'analisi del cromosoma. Esperto di analisi di ogni cromosoma 3 Gy radiazioni ha dato esattezze paragonabili a ADCI. In una serie di 6 campioni di esposizioni sconosciute (precedentemente utilizzate in un esercizio di competenza internazionale), il software stimato dosi entro 0,5 Gy dei valori ottenuti da revisione manuale dei dati stessi di HC e CNL, che soddisfi i requisiti dell'AIEA per triage biodosimetry. Inoltre, standardizzazione interlaboratorio e, infine, la riproducibilità della dose stima beneficio dall'avere un controller di dominio comune, automatizzato, segnando algoritmo. Tuttavia, il software permette personalizzazione dell'immagine di filtraggio e criteri di selezione, che consente differenze nei metodi di preparazione del cromosoma e sorgenti di taratura di radiazione per essere presi in considerazione.
Questo software è un'interfaccia di utente grafica (GUI) - basati su sistema che analizza set di immagini di cromosoma contenente Giemsa (o DAPI) - macchiato cellule in metafase per le anomalie che derivano dall'esposizione a radiazioni ionizzanti. I set di immagine sono fotografati digitalmente con un sistema di microscopio luce (o epifluorescente) e ogni insieme corrisponde a un campione diverso. Il software utilizza tecniche per rilevare e discriminare le DCs da MCs e altri oggetti di elaborazione delle immagini. Empiricamente derivati segmentazione filtri eliminano quindi automaticamente i falsi positivi DCs senza intaccare il vero DCs. Infine, il software automaticamente filtra indesiderati immagini basati su varie strutture di immagine trovate immagini di metafase di scarsa qualità con modelli di selezione immagine pre-calcolate (o specificato dall'utente). Queste immagini includono quelli contenenti eccessiva o insufficiente numero di oggetti "rumorosi", più cromosomi sovrapposti, immagini mancanti cromosomi in metafase, un numero eccessivo di sorella cromatidi4. I dati di immagine automaticamente a cura vengono utilizzati per generare la curva di calibrazione di dose da campioni della dose di radiazione noti e sono utilizzati per stimare le esposizioni di prova campioni esposti a dosi sconosciute.
Output del software possono essere visualizzati e salvati come: 1) basato su testo output visualizzato nella console, 2) trame che possono essere salvati come immagini e 3) di report in formato HTML. Molti aspetti del software sono personalizzabili per soddisfare le esigenze specifiche dei diversi laboratori. Laboratori individuali forniscono solitamente i campioni di test e calibrazione preparato e raccolti basata sul protocollo citogenetico convalidato in quel laboratorio. Questo mantiene l'uniformità della preparazione del campione e permette curve di calibrazione generate dai campioni di calibrazione chiaramente da applicare per testare campioni derivati usando lo stesso protocollo. Curve di calibrazione possono essere create anche da coefficienti di curva o frequenze DC alle dosi definite. Le stime più accurate di dose sono ottenute filtrando immagini di qualità inferiore e falsi positivi DCs (FPs). Selezione dei sottoinsiemi di immagine ottimale all'interno di ogni campione viene eseguita utilizzando 'Modelli di immagine selezione' che eliminano immagini scadente che tendono a introdurre FPs. Una serie di modelli pre-convalidate sono inclusi con il software, tuttavia ulteriori modelli con soglie personalizzate e filtri possono essere creati e salvati dall'utente.
Una volta che il software viene caricato correttamente, la principale interfaccia grafica (GUI) è presentato (Vedi Figura 1). Da questa interfaccia, campioni, ciascuno composto da una cartella di file immagine di metafase cella, possono essere selezionate e lavorate per identificare controller di dominio, curve di calibrazione possono essere create e confrontate e dose di esposizione di radiazioni di campioni può essere determinata.
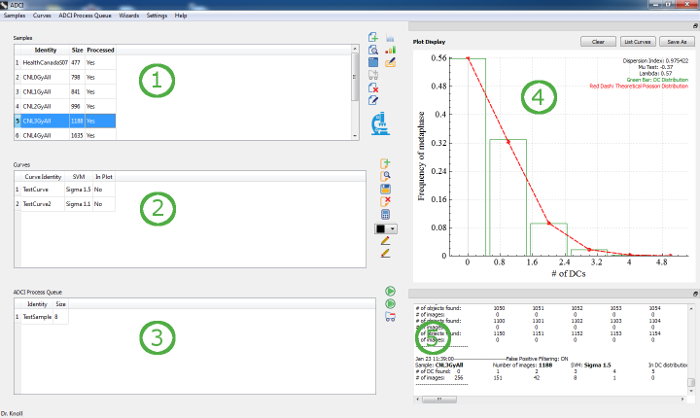
Figura 1: The settori principali dell'interfaccia di utente grafica includono: un elenco di campioni (1), un elenco di calibrazione delle curve (2), il processo di coda (3), che controlla lo stato del rilevamento di DC in ogni set di immagini di ogni campione, una trama display (4), che riassume le statistiche o altre proprietà quantitativa di un set di immagini nei campioni o curve di calibrazione e una console (5) che contiene un testo descrittivo come uscite di ogni operazione eseguita dal programma. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. importazione e campioni di processo
- clic ' campioni ' nella barra dei menu e selezionare ' nuovo campione '. Sfoglia in una directory appropriata contenente un gruppo di metafase immagini e fare clic su ' selezionare la cartella '.
- Digitare un ID univoco per il campione all'interno del ' specifica un ID univoco per il nuovo campione ' campo di testo. Questo ID identificherà il campione nell'area di lavoro. ID campione deve contenere alfanumerico, ' _ ', o '-' solo caratteri. È noto l'inclusione del laboratorio sorgenti e dose fisica (per campioni di calibrazione) l'ID campione.
- (Facoltativo) fornire una descrizione del campione se desiderato all'interno della ' Descrizione del campione (opzionale) ' area di testo.
- Clic ' OK ' per aggiungere il nuovo campione per l'area di lavoro.
- Ripetere passaggi da 1.1 a 1.4 per aggiungere ulteriori campioni. Creare un minimo di 3 campioni di taratura (sette o più sono raccomandati 3 di diverse esposizioni) e almeno un test campione per eseguire la stima di dose.
- Evidenziare tutti i campioni creati nei passaggi da 1.1 a 1.5 alla ' campioni ' elenco e fare clic su ' campione aggiungere alla coda processi ' (
 ) icona.
) icona. - Clic ' tutti i campioni nella coda di processo ' (
 ) icona per elaborare tutti i campioni in sequenza all'interno della coda - un ' ADCI elaborazione ' finestra di dialogo contenente tutti i campioni nella coda con una barra di avanzamento.
Fare clic su
) icona per elaborare tutti i campioni in sequenza all'interno della coda - un ' ADCI elaborazione ' finestra di dialogo contenente tutti i campioni nella coda con una barra di avanzamento.
Fare clic su - quando tutti i campioni hanno completato l'elaborazione, la
 . Salvare i campioni ora, oppure fare clic su ' salvare un campione trasformato in un file di esempio ADCI ' (
. Salvare i campioni ora, oppure fare clic su ' salvare un campione trasformato in un file di esempio ADCI ' ( ) icona per salvare un campione preparato dopo.
) icona per salvare un campione preparato dopo.
2. visualizzazione e selezione di immagini (facoltativo, passaggio consigliato)
Nota: questo passaggio viene descritto l'utilizzo del Visualizzatore di immagine di metafase e la creazione di un modello di selezione immagine. Alcuni modelli di selezione immagine convalidati sono inclusi con il software che può essere utilizzato nella generazione della curva di calibrazione e stima della dose. Così, questo passaggio non è obbligatorio, tuttavia può essere utilizzato come una guida che descrive i passaggi necessari per eseguire questa operazione se si desidera.
- Evidenziare un campione entro il ' campioni ', fare clic ' campioni ' nel menu bar e selezionare ' Image View ' per aprire la ' metafase Image Viewer ' . Navigare tra le immagini
- selezionare un'immagine dalla casella a discesa per visualizzare un'immagine specifica. Clicca sulle icone freccia sinistra e destra per scorrere le immagini.
- Selezionare un valore di Sigma SVM dalla casella a discesa per visualizzare i risultati di rilevamento DC a quel valore di Sigma. Selezionare " Unprocessed " dalla casella a discesa per visualizzare immagini raw senza cromosoma contorni.
- Verifica la ' invertire ' casella di controllo per invertire i valori di luminosità e colore per ogni pixel nell'immagine.
- Controllare la ' immagine nella Watch List ' casella di controllo per aggiungere l'immagine visibile di una ' Watch list '. Fare clic ' salvare la Watch List in un file di testo ' (
 ) icona per salvare i nomi di tutte le immagini nell'elenco di controllo per un file di testo.
) icona per salvare i nomi di tutte le immagini nell'elenco di controllo per un file di testo. - Modelli di selezione immagine
- clic ' Mostra tutte le immagini ' (selezione predefinita) per includere tutte le immagini nella casella a discesa di selezione immagine. Osservare il testo adiacente al ' immagini incluse ' per scoprire la frazione di immagini selezionate dal modello di selezione immagine attualmente applicata.
- Clic ' Vista incluso immagini ' per includere solo queste immagini che non sono stati esclusi dal modello di selezione di immagine nella casella a discesa.
- Clic ' vista escluso immagini ' per includere le immagini che sono state escluse dal modello di selezione immagine applicata nella casella a discesa.
- Controllo del ' escludere ' casella di controllo per escludere manualmente una singola immagine.
Nota: Immagini manualmente esclusi vengono ripristinate l'immagine selezionata, impostata se successivamente viene applicato un modello di selezione immagine. - Salvare una selezione di immagini cliccando il ' Salva selezione ' pulsante. Immettere un nome di file per la selezione salvata quando richiesto. Fare clic ' carica selezione ' di applicare una selezione salvata in precedenza.
- Fare clic ' applica filtri di immagine ' per aprire la ' modello di selezione immagine basato su Applica filtro per esempio corrente ' finestra di dialogo, che crea, Salva, o applica criteri per la selezione di immagini di metafase in un campione.
- Selezionare un modello di selezione immagine dall'elenco precompilato. Fare clic ' OK ' di applicare il modello attuale.
- Immettere una descrizione per un nuovo modello desiderato, definire ' filtri di esclusione immagine ', definire ' immagine Ranking e inclusione ' e fare clic su ' Salva selezione modello ' per creare un'immagine Selezione modello.
Nota: Le definizioni di ' immagine Ranking e inclusione ' metodi e ognuno ' filtro di esclusione immagine ' può essere trovato nella documentazione in linea di software 14.
3. Generazione della curva di
- (Recommended optional step) curva di taratura guidata
- garantire un minimo di tre campioni di calibrazione sono presenti nell'area di lavoro prima di procedere. Fare clic ' maghi ' nella barra dei menu e selezionare ' curva di calibrazione ' per aprire la curva di calibrazione guidata.
Nota: Anche se solo tre campioni sono matematicamente necessari per adattarsi e calcolare una curva di calibrazione, sette o più campioni che abbracciano una gamma delle esposizioni compreso tra 0 e 5 Gy sono raccomandati. I campioni supplementari sono necessari per adattare la curva di taratura per una lineare-quadratica dose risposta, tuttavia i valori ottimali di Sigma possono essere inferiori al fine di ottenere curve che possono essere utilizzate per la stima della dose bassa (< 1 Gy); i valori ottimali di Sigma per dosi superiori a questa soglia sono diversi (fare riferimento al punto 3.1.4). - Procedi attraverso la schermata introduttiva e posto un segno di spunta accanto a ciascun campione di calibrazione desiderata. Per ogni campione di calibrazione selezionati in questo modo, specificare la dose fisica (in Gy) il campione è stato esposto all'interno del relativo campo di testo adiacente. Continuare alla schermata successiva della procedura guidata.
- Selezionare un modello di selezione immagine se desiderato dall'elenco dei modelli contenenti modelli di selezione immagine preimpostata in bundle con il software oltre a tutti i modelli creati manualmente. Continuare alla schermata successiva della procedura guidata.
- Selezionare un valore di Sigma SVM dalla casella a discesa. Continuare alla schermata successiva della procedura guidata .
Nota: È consigliabile un valore SVM Sigma 1.4 o 1.5 per stime di dose > 1 Gy e un valore di 1.0 per stime sotto 1 Gy ( Figura 2). - Esaminare tutte le precedenti selezioni la schermata di riepilogo e fare clic su ' fine ' per completare la procedura guidata, causando un prepopulated ' creare una curva ' finestra di dialogo venga visualizzato.
- garantire un minimo di tre campioni di calibrazione sono presenti nell'area di lavoro prima di procedere. Fare clic ' maghi ' nella barra dei menu e selezionare ' curva di calibrazione ' per aprire la curva di calibrazione guidata.
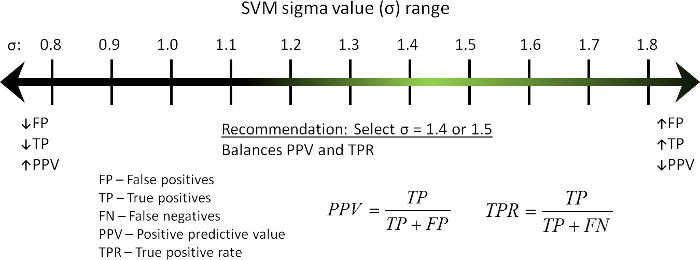
Figura 2: visualizzazione dell'effetto di modifica del valore di Sigma SVM dall'algoritmo sul vero positivo (TP) e falsi positivi (FP) DC conteggi, il Calue predittivo positivo (PPV) e vero positivo tassi (TPR). Clicca qui per visualizzare una versione più grande di questa figura.
- creare una finestra di dialogo curva.
- (Salta questo passaggio se è stato utilizzato la procedura guidata) fare clic su ' curve ' nella barra dei menu e selezionare ' nuova curva '. Scegliere ' curva di raccordo ai dati Dose-risposta ' dalla casella a discesa presentato all'interno della finestra di dialogo e fare clic su ' OK '.
- Specificare un'identità univoca per la curva nella ' specificare un'identità univoca per la nuova curva ' casella di testo all'interno del ' creare una curva ' finestra di dialogo.
- (Facoltativo) digitare una descrizione per la nuova curva all'interno della ' aggiungere una breve descrizione per la curva deve essere creato ' casella di testo.
- (Skip i seguenti passaggi se la creazione guidata curve è stata utilizzata per creare una curva di calibrazione) impostare i valori della curva.
Nota: La procedura guidata di calibrazione curva descritta al punto 3.1 vengono precompilate da campi alla ' creare una curva ' finestra di dialogo. I seguenti passaggi descrivono come inserire manualmente questi campi. Se la procedura guidata è stata utilizzata, alcuni passaggi qui sotto possono essere seguiti ancora se lo si desidera, aggiungere o rimuovere dati aggiuntivi.- Selezionare un valore di Sigma SVM opzioni nella ' SVM ' casella di riepilogo a discesa - è altamente raccomandato che il valore di Sigma scelto qui corrisponde al valore di Sigma scelto quando si utilizza questa curva per eseguire la stima di dose.
- (Facoltativo) specificare un modello di selezione immagine cliccando il ' specificare il File di ' pulsante.
- Clic ' Input ' per aggiungere una nuova voce vuota nell'elenco di dose-risposta sotto l'intestazione ' risposta-Dose di Input dati per creare una curva di '.
- Inserire la dose di un campione di calibrazione in Gy sotto il titolo ' Dose '.
- INVIO ' risposta (DC/cella) ' disegnata da output di esempio all'interno della console quando un campione viene evidenziato. Individuare il valore appropriato di DC/cella per il valore di SVM Sigma precedentemente selezionato all'interno della console o dal report di esempio corrispondente (punto 5.1, se disponibile) e inseriscilo in questo campo.
- Ripetere i tre passaggi precedenti fino a quando tutti i campioni di taratura sono stati aggiunti.
- Stampa ' convalidare dati ' affinché il contenuto della risposta-Dose elenco è formattato correttamente – verificare tutti i campi nell'elenco risposta-Dose sono evidenziati in verde che indica dati valido.
- Press ' OK ' per completare la creazione della curva. Per salvare la nuova curva nella ' Salva curva? ' finestra di dialogo che viene visualizzata dopo aver premuto ' OK '. Oppure fare clic su ' Salva curva in un file di curva ADCI ' (
 ) icona evidenziata all'interno del ' curve ' riportate di seguito.
) icona evidenziata all'interno del ' curve ' riportate di seguito.
4. Stima della dose
- (Recommended optional step) Dose stima guidata
- clic ' maghi ' nella barra dei menu e selezionare ' stima di Dose '.
- Procedi attraverso la schermata introduttiva della procedura guidata e selezionare una curva di calibrazione precedentemente creato dalla casella a discesa - sue proprietà apparirà sotto. Continuare alla schermata successiva della procedura guidata.
- Inserire un segno di spunta accanto a campioni di prova di esposizione sconosciuta di includerli nella stima di dose. Continuare alla schermata successiva della procedura guidata.
- Osservare la descrizione e le proprietà del modello di selezione immagine applicate durante la generazione della curva di calibrazione. Osservare che lo stesso modello di selezione immagine viene applicato ai campioni test selezionato. Continuare alla schermata successiva della procedura guidata.
Nota: Sotto la descrizione del modello di selezione immagine, lo stesso modello è già compilato e verrà applicato per i campioni di prova di selezione. Applicare lo stesso modello di selezione immagine alla calibrazione e campioni di prova. Mentre è possibile applicare modelli di selezione immagine diversa selezionando un modello diverso dal menu a discesa, questo non è consigliato. - Selezionare un valore di SVM Sigma dal menu a discesa. Continuare alla schermata successiva della procedura guidata.
Nota: Il valore di Sigma SVM utilizzato durante la generazione della curva di calibrazione è precompilato. È consigliabile che questo valore resta immutato. - Rivedere le precedenti selezioni nella schermata di riepilogo e fare clic su ' finire ' per completare la procedura guidata - un precompilato ' Dose calcolatrice ' finestra di dialogo apparirà.
- Dose calcolatrice
- (ignorare questo passaggio se è stato utilizzato la procedura guidata) evidenziare una curva di calibrazione dall'elenco delle curve sotto l'intestazione ' curve ', fare clic ' curve ' nel menu bar e selezionare ' Calcolare Dose ' per aprire la ' Dose calcolatrice ' finestra di dialogo.
- (Skip procedura se guidata è stato utilizzato) impostare i valori per la stima della dose.
Nota: La procedura guidata di stima di Dose descritta al punto 4.1 vengono precompilate da campi alla ' Dose calcolatrice ' finestra di dialogo. I seguenti passaggi descrivono come inserire manualmente questi campi. Se la procedura guidata è stata utilizzata, alcuni passaggi qui sotto possono essere seguiti ancora se lo si desidera, aggiungere o rimuovere dati aggiuntivi.- Clic ' Uso dei campioni nell'area di lavoro per riempire le frequenze DC ' (
 ) icona e punto culminante testare campioni entro il ' campioni trattati in ADCI Area di lavoro ' elenco per aggiungere i campioni selezionati per la ' DC frequenze per la stima della Dose ' elenco.
) icona e punto culminante testare campioni entro il ' campioni trattati in ADCI Area di lavoro ' elenco per aggiungere i campioni selezionati per la ' DC frequenze per la stima della Dose ' elenco. - Selezionare un modello di selezione valore e l'immagine del Sigma SVM per questi campioni dalle caselle a discesa.
Nota: È necessario per la stima della dose precisa un valore di Sigma SVM corrispondente al valore di Sigma utilizzato nella generazione della curva di calibrazione. Il valore di Sigma associato con la curva di taratura è elencato nella parte inferiore della ' Dose calcolatrice ' finestra di dialogo. - (opzionale) Aggiungi ulteriori campioni di ripetere i due passaggi precedenti. In alternativa, aggiungere campioni multipli simultaneamente evidenziando campioni multipli nella ' campioni elaborati nell'area di lavoro ' elenco.
- (facoltativo) Clicca la ' un valore di frequenza DC Input ' (
 ) icona per inserire manualmente un DC frequenza non associato a qualsiasi campione se lo si desidera - la nuova frequenza DC verrà aggiunto alla ' DC aberrazioni per la stima della Dose ' elenco.
) icona per inserire manualmente un DC frequenza non associato a qualsiasi campione se lo si desidera - la nuova frequenza DC verrà aggiunto alla ' DC aberrazioni per la stima della Dose ' elenco. - (opzionale) fare doppio clic il ' nome ' campo di frequenza DC immessa manualmente per modificare il nome.
- (opzionale) evidenziare appropriati campioni e fare clic su ' frequenza DC rimuovere ' (
 ) icona per rimuovere campioni che sono stati aggiunti per la ' DC Le aberrazioni per la stima della Dose ' elenco errore.
) icona per rimuovere campioni che sono stati aggiunti per la ' DC Le aberrazioni per la stima della Dose ' elenco errore.
- Clic ' Uso dei campioni nell'area di lavoro per riempire le frequenze DC ' (
- Fare clic ' OK ' per chiudere il ' Dose calcolatrice ' ed eseguire la stima di dose - risultati sono output di console.
- Come dose stima risultati vengono visualizzati nella console in formato tabulare per ogni campione, osservare ' frequenza DC ', ' SVM ', ' Dose stimata ' (contiene la stima dose biologica del campione da analizzare in Gy), e ' modello di selezione immagine applicata ' campi.
5. Segnalazione
Nota: il metodo utilizzato per denominare un report e selezionare una directory all'interno del quale viene salvato è comune a tutti i tipi di report. A ' nome Report ' devono essere fornite. Quando viene generato un report, verrà creata una directory contenente i file di report utilizzando questo nome automaticamente. Questa directory verrà inserita all'interno è il ' cartella Report '. Per impostazione predefinita, il ' cartella Report ' è una directory denominata ' report ' trovato nella directory dei dati specificata durante l'installazione.
- Report di esempio
- fare clic ' Report ' nella barra dei menu e selezionare ' Report di esempio ' per aprire la ' generare report di esempio ' finestra di dialogo.
- Immettere un nome per il rapporto nella ' nome Report ' campo di testo. Fare clic su ' Sfoglia ' per modificare il ' cartella Report ' se lo si desidera.
- Selezionare almeno un campione preparato da includere nel report inserendo un segno di spunta accanto a campioni adeguati al ' selezionare campioni ' elenco.
- Specificare una gamma di SVM Sigma i valori per cui generare diagrammi distribuzione DC selezionando i valori in ' Min ' e ' Max ' caselle a discesa all'interno della ' distribuzione di controller di dominio nel campione ' zona. Escludere DC distribuzione trame dal report se si desidera deselezionando la ' includono ' casella di controllo nella ' distribuzione di controller di dominio nel campione ' zona.
- Specificare quali trame contenente statistiche di filtraggio da includere nel report inserendo segni di spunta accanto a parcelle appropriati nella ' selezionare trame ' zona. Fare clic ' OK ' per generare il report.
- Sulla curva
- fare clic ' Report ' nella barra dei menu e selezionare ' sulla curva ' per aprire il ' generare report di curva ' finestra di dialogo.
- Immettere un nome per il rapporto nella ' nome Report ' campo di testo. Fare clic ' Sfoglia ' per modificare il ' cartella Report ' se lo si desidera.
- Selezionare almeno una curva da includere nel report inserendo un segno di spunta accanto appropriate curve nella ' selezionare le curve per essere inclusi nel report ' elenco. Fare clic ' OK ' per generare il report.
- Relazione di stima di dose
- eseguire le operazioni di stima dose descritte nella sezione 4.
Nota: Rapporti di stima di Dose sono generati dai risultati indicati in aree del tracciato e console. Così, una trama di stima della dose deve essere presente nell'area del tracciato nel momento in cui viene generato un report. - Fare clic ' Report ' nella barra dei menu e selezionare ' relazione di stima di Dose ' per aprire la ' Genera rapporto di stima dose ' finestra di dialogo.
- Immettere un nome per il rapporto nella ' nome Report ' campo di testo. Fare clic ' Sfoglia ' per modificare il ' cartella Report ' se lo si desidera.
- Clic ' OK ' per generare il report.
- eseguire le operazioni di stima dose descritte nella sezione 4.
6. Funzionalità di controllo
Nota: il software registra tutte le operazioni eseguite durante una sessione in un file di log. Il programma fornisce un'applicazione software accessorio che consente i file di registro essere guardato, cercato, utilizzata per valutare l'integrità di un'analisi e in alcuni casi, di recuperare i dati di esempio da incompleta o prematuramente terminata sessioni.
- Clic ' aiutare ' nella barra dei menu e selezionare ' visualizzare i registri ' per aprire il software del Visualizzatore di file registro supplementare.
- Assicurarsi che i file di registro sono elencati nella barra laterale sul lato sinistro della finestra. Se nessun file sono visibile, fare clic ' File ', scegliere ' directory file registro selezionare ' e passare a una directory contenente i file di log.
- Fare doppio clic sul nome di un file di registro nella barra laterale per visualizzare il contenuto del file di log nel ' Viewer ' Tab.
- Seleziona il ' ricerca ' termini per la ricerca di uno o più file di log di ricerca scheda e input.
I parametri di ricerca
- input se desiderato nella ' da ', ' a ', ' utente ', ' licenza ', ' operazione ', e ' Parametri ' campi.
- Usare il cursore per selezionare il ' Max ricerca risultati per ciascun file '.
Nota: Alcuni parametri di ricerca, ad esempio username, restituirà molti risultati in ogni file di log corrispondente. Questo parametro limita il numero di risultati di ricerca visualizzati in ogni file di log. - Un segno di spunta nella ' ricerca evidenziato solo file ' casella di controllo (tutti i log file vengono cercati per impostazione predefinita) ed evidenziare i file di registro nella barra laterale per cercare solo un sottoinsieme dei file di registro.
- Verifica la ' eseguire controllo di integrità ' casella di controllo (impostazione predefinita) per esaminare ogni file di log idonei a essere cercato per errori correlati a una chiusura inaspettata software.
- Clic ' ricerca ' per cercare i file di registro e cerca di osservare i risultati sul lato destro della finestra.
- Scegliere la ' Visualizza file di log ' pulsante adiacente a un risultato di ricerca per evidenziare e Mostra la linea indicata nella ' Viewer ' Tab.
- Problemi di integrità dei file di log
- scegliere la ' integrità ' scheda per visualizzare gli errori rilevati durante il controllo di integrità (se è stato richiesto il check).
Nota: Deve essere eseguita una ricerca per esaminare i file di registro per problemi di integrità. Per eseguire un controllo di integrità senza dover cercare i file di registro per uny di termini di ricerca, è sufficiente lasciare tutti i campi di parametro di ricerca nero nella ' ricerca ' scheda, garantire il ' eseguire controllo di integrità ' sia selezionata, quindi fare clic su ' ricerca '. Se vengono rilevati problemi di integrità, il ' integrità ' scheda sfondo colore diventerà rosso - Integrità di risolvere problemi (uscita è raggruppato in file di log) dove possibile.
Nota: Per ulteriori informazioni sulla procedura per risolvere problemi di integrità, consultare la documentazione in linea 14.
- scegliere la ' integrità ' scheda per visualizzare gli errori rilevati durante il controllo di integrità (se è stato richiesto il check).
7. Curva e Dose stima statistiche opzioni
- fare clic ' impostazioni ' nella barra dei menu e selezionare ' statistiche opzioni ' per aprire la ' statistiche opzioni ' finestra di dialogo.
- Selezionare una curva di calibrazione montaggio metodo (minimi quadrati o massima verosimiglianza) dalla casella a discesa.
- Un segno di spunta accanto a ' visualizzare CI 95% curva di calibrazione, se applicabile ' per visualizzare gli intervalli di confidenza di 95% quando si tracciano una curva di calibrazione.
- Un segno di spunta accanto a ' stima di Dose calcola CI di 95% a causa di Poisson ' per calcolare i limiti di confidenza del 95% sulle stime delle dosi in base alla natura di Poisson di rendimento DC.
- Un segno di spunta accanto a ' stima di Dose calcola CI di 95% a causa della curva, se applicabile ' per calcolare i limiti di confidenza del 95% sulla dose stime basate sull'incertezza legata alla curva di calibrazione.
Risultati
Test del software è stato effettuato con i dati immagine del cromosoma di metafase ottenuti da HC e CNL. I campioni di sangue sono stati irradiati da un'unità di XRAD-320 (250 kV i raggi x, 12,5 mA, 2mm Al filtraggio, rateo di dose: 0.92 o 1,7 Gy/min) calibrato con una camera a ioni a HC e lavorate ad entrambi i laboratori. Campioni di sangue periferico del linfocita erano coltivati, corretti e macchiati presso ogni impianto secondo protocolli stabiliti3,15. Immagini di metafase da vetrini colorati Giemsa furono catturati in modo indipendente da ogni laboratorio utilizzando un sistema di microscopia automatizzata. Esperti in ciascun laboratorio segnato DCs in molti di questi campioni manualmente, costruito le proprie curve di calibrazione e stimato dosi di campioni di prova di esposizioni sconosciute. Nella tabella 1 viene fornita una descrizione dettagliata di questi set di dati.
| Dose fisica | Scopo | Preparazione di HC | Preparazione di CNL | ||
| Cui nome | n # di immagini | Cui nome | n # di immagini | ||
| 0 Gy | Calibrazione | HC0Gy | 731 | CNL0Gy | 798 |
| 0.1 Gy | Calibrazione | HC01Gy | 2162 | NA | NA |
| 0,25 Gy | Calibrazione | HC025Gy | 1826 | NA | NA |
| 0,5 Gy | Calibrazione | HC05Gy | 1054 | CNL05Gy | 1532 |
| 0.75 Gy | Calibrazione | HC075Gy | 1233 | NA | NA |
| 1 Gy | Calibrazione | HC1Gy | 1566 | CNL1Gy | 841 |
| 2 Gy | Calibrazione | HC2Gy | 1147 | CNL2Gy | 996 |
| 3 Gy | Calibrazione | HC3Gy | 1212 | CNL3Gy | 1188 |
| 4 Gy | Calibrazione | HC4Gy | 909 | CNL4Gy | 1635 |
| 5 Gy | Calibrazione | HC5Gy | 1019 | NA | NA |
| 3.1 Gy | Prova | HCS01 | 540 | CNLS01 | 500 |
| 2.3 Gy | Prova | HCS08 | 637 | CNLS08 | 500 |
| 1.4 Gy | Prova | HCS10 | 708 | NA | NA |
| 1.8 Gy | Prova | HCS04 | 600 | CNLS04 | 957 |
| 2.8 Gy | Prova | HCS05 | 1136 | CNLS05 | 1527 |
| 3.4 Gy | Prova | HCS07 | 477 | CNLS07 | 735 |
Tabella 1: Fonti di dati di immagine forniti da HC e CNL per la valutazione del Software.
Nota: Modificata dalla tabella 1 in Rogan et al., 20164. Solo immagini manualmente preselezionati erano precedentemente disponibili a noi da CNL. Immagini non filtrate sono diventato disponibili e immagine conteggi vengono aggiornati di conseguenza. Inoltre, recentemente acquisito campioni HC (0.25Gy, 0.75Gy e 5Gy) sono presentati qui.
Selezione automatica dell'immagine nei campioni
Qualità dell'immagine è fondamentale per la corretta rilevazione di DC in analisi DC. Selezione delle immagini da specialisti citogenetici viene solitamente eseguita manualmente in analisi DC convenzionale. ADCI utilizza criteri quantitativi immagine per selezionare automaticamente le immagini prima di calcolo frequenza DC16. Gli utenti possono o filtro immagini basato su morfologie di cromosoma specifico e/o celle di ordinamento secondo proporzioni relative delle lunghezze degli oggetti secondo note lunghezze di citogenetica definiti gruppi di cromosomi in un cariotipo umano normale (definito il Metodo di distanza di gruppo-bin). I filtri morfologici disponibili utilizzano soglie invariante di scala per rifiutare immagini delle cellule con cromosoma incompleta di moda o con più metafasi, con i cromosomi di prometaphase, con prominente sorella dissociazione tra cromatidi fratelli, con altamente piegato e torto cromosomi, con oggetti che hanno contorni lisci caratteristici dei nuclei intatti e quelli in cui meno oggetti sono riconosciuti come cromosomi. Figura 3 (a) e (b) mostrare esempi di immagini selezionate, mentre nella figura 3 (c) e (d) sono esempi di immagini che vengono filtrati dal software. Queste immagini sono derivate dall'esempio HCS05 (descritto nella tabella 1) e vengono selezionate per il modello di selezione di immagini predefinito che truppa tutte le immagini di distanza del gruppo di bin, quindi seleziona le migliori 250 immagini. Cromosomi in Figura 3 (A), (b) sono ben separati e morfologia soddisfacente per mostre. Figura 3 (c) contiene un numero eccessivo di cluster di cromosoma sovrapposte. Figura 3 (d) spettacoli severa sorella separazione tra cromatidi fratelli. Sorella cromatidi sono completamente separati per almeno 8 dei cromosomi e le costrizioni centromeriche sono ambigue nella maggior parte dei altri cromosomi.
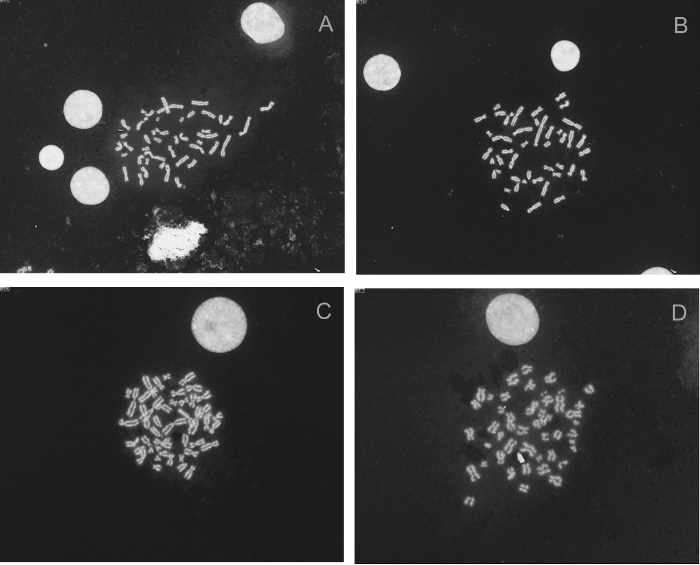
Figura 3: esempi di immagini di metafase nel campione HCS05 (ingrandimento: 63 X), Unselected sia selezionato dal modello 'Gruppo Bin distanza, Top 250 immagini'. Immagini selezionate (A) e (B) . (C) e (D) sono immagini che sono state eliminate dal modello. (C) è stato escluso perché conteneva troppi cromosomi sovrapposti e (D) aveva un numero eccessivo di separati sorella cromatidi. Clicca qui per visualizzare una versione più grande di questa figura.
Gli effetti dell'applicazione di questi modelli di selezione immagine è evidente esaminando il livello di probabilità di rilevazione di DC in un campione. Occorrenze di DCs in una popolazione delle cellule da un campione irradiato seguano una distribuzione di Poisson. Il test di bontà di adattamento di chi-quadrato confronta la distribuzione di frequenza osservata di DC per la misura prevista per la distribuzione di Poisson. Modelli che correttamente i dati di esempio di filtro presentano frequenze DC non significativamente differenti dalle Poisson previsto valori derivati (in genere il livello di significatività > 0,01). Figura 4 Visualizza le occorrenze DC e il corrispondente si adatta alla distribuzione di Poisson per il campione di HC4Gy di tutte le immagini vs solo immagini selezionate dal modello "distanza del gruppo di bin, top 250 immagini". Figura 4 (b) dimostra una migliore vestibilità per la distribuzione di Poisson. Il p-valore (0,36) di set filtrato di immagini supera notevolmente quello della distribuzione DC non filtrata in Figura 4 (a). Al 5% o 1% i livelli di significato, il campione non filtrato in Figura 4 (a) è meno affidabile, poiché contiene dati di DC qualità inferiori, come l'ipotesi di una distribuzione di Poisson di DCs viene rifiutata.
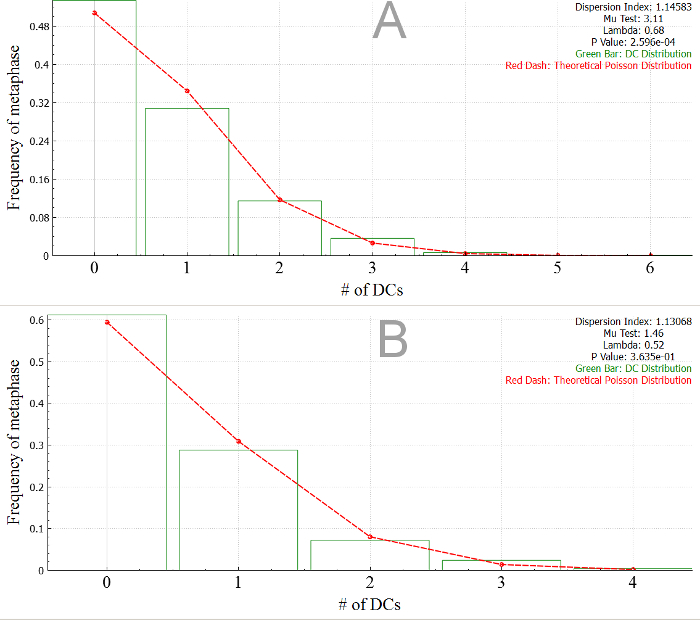
Figura 4: Screenshots delle frequenze proporzionate DC adatta a Poisson Dstributions di HC4Gy di esempio nel Software. (A) tutte le immagini sono inclusi, (B) solo immagini selezionate dal modello (distanza del gruppo di bin, top 250 immagini) sono inclusi. La leggenda (in alto a destra) indica le statistiche della fit per la distribuzione di Poisson (indice di dispersione, test di Mu e Lambda) e la bontà di chi-quadrato di prova adatta (valore p) Clicca qui per visualizzare una versione più grande di questa figura.
Rilevamento di cromosoma dicentrico (DC)
Rilevazione accurata di DC è il requisito fondamentale dei prerequisiti dell'ADCI. Correttamente rilevato DCs e quelli persi dal software sono rispettivamente definiti come veri positivi (TPs) e falsi negativi (FNs). Oggetti che non sono controller di dominio, ma erroneamente rilevato come controller di dominio, sono definiti come falsi positivi (FPs). FPs sono monocentric cromosomi, frammenti dei cromosomi, separata sorella cromatidi, cromosoma sovrapposte cluster e gli oggetti non-cromosomici. La figura 5 Mostra i risultati di rilevamenti di DC in due immagini di metafase. Oggetti 1 e 3 sono TPs, mentre oggetto 4 è un FP che comprende due cromosomi distinti monocentric congiunti lungo loro bracci corti. In Figura 5 (a), oggetto 2 era originariamente un FP, ma successivamente rettificato da FP filtri nel software. Oggetto 5 e 6 di oggetto in Figura 5 (b) sono esempi di probabili del FNs.
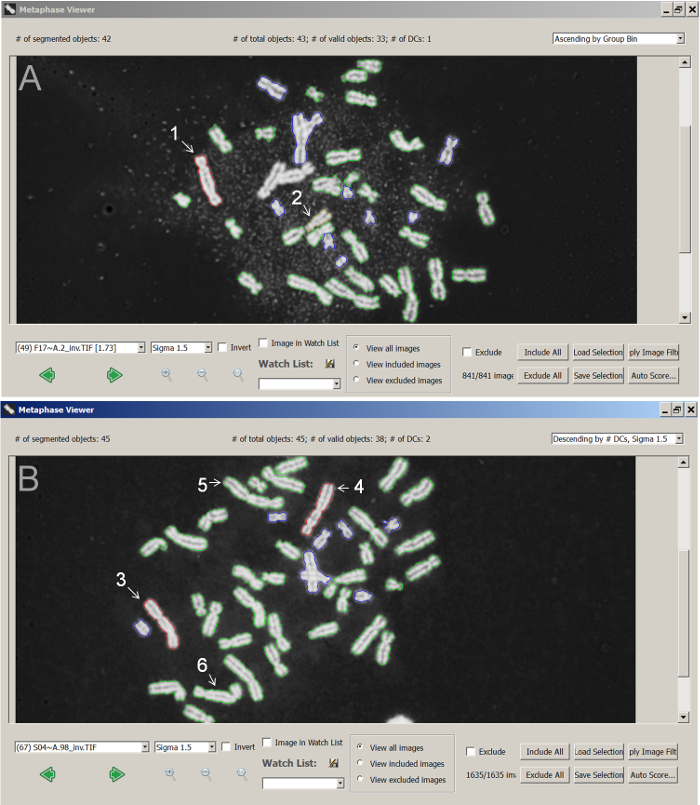
Figura 5: Screenshot per indicare la classificazione di cromosoma di metafase di potenziali DCs. (A) Un'immagine campione CNL1Gy (ingrandimento: 63 X) Risultati 1 TP, oggetto "1"; e 1 corretto FP, oggetto "2". (B) un'immagine campione CNL4Gy (ingrandimento: 63 X) Risultati 1 TP, oggetto "3"; 1 FP, oggetto "4"; e 2 potenziali FNs, oggetti "5" e "6". TPs, FPs corretto, normale monocentric e cromosomi non classificati sono descritte rispettivamente con contorni rossi, gialli, verdi e blu. Clicca qui per visualizzare una versione più grande di questa figura.
Stima della dose dei campioni di prova
Il risultato finale dell'analisi ADCI sono la dose stima dei campioni dedotti dalle curve di calibrazione. Le valutazioni di dose apportate dal software per i campioni di prova nella tabella 1 sono indicate nelle tabelle 2 e 3. Per confronto, la dose di radiazione fisica emessa e sono indicate le dosi manuale ha ottenute dagli esperti di HC per i campioni HCS01, HCS08 e HCS10. Allo stesso modo, la fisica e manuale segnato le dosi da esperti CNL sono mostrati per CNLS04, CNLS05 e CNLS07.
Figura 6 illustra le curve di calibrazione con le stime delle dosi di radiazione per campioni di laboratorio di Health Canada biodosimetry HCS01, HCS08, HCS10, HCS04, HCS05 e HCS07. La curva di taratura viene generata utilizzando campioni HC0Gy, HC1Gy, HC2Gy, HC3Gy e HC4Gy. Il modello di selezione immagine contenente 3 filtri basati su Z-punteggio + "gruppo bin distanza, immagini top 250" viene applicato a tutti i campioni. Le stime delle dosi insieme ad analisi statistiche associate sono riportati nella tabella 2.
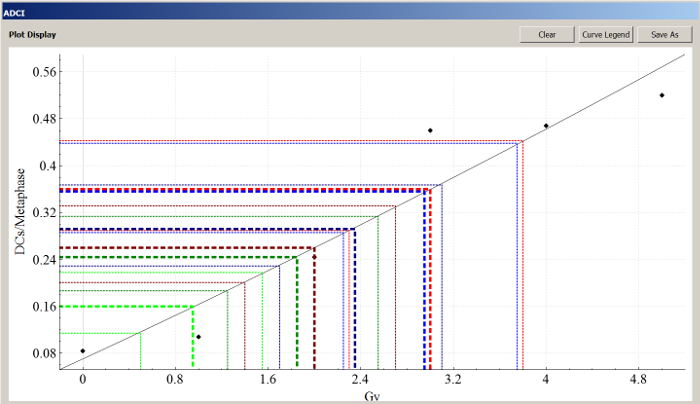
Figura 6: Screenshot di stima di Dose di campioni di prova HC. Quadrati neri rappresentano campioni di calibrazione. Immagini in esempi di prova e taratura vengono selezionati dal modello (3 filtri FP + distanza del gruppo di bin, top 250 immagini). Linee spesse tratteggiate rappresentano il mapping di DCs/metafase tramite la curva di taratura per dose stimata. Sottili linee punteggiate indicano superiore e abbassare i limiti di confidenza del 95% di DCs/metafase. I codici dei campioni di prova colore: rosso brillante, HC S01 (dose fisica: 3.1Gy, dose HC dedotto: 3.4Gy, ADCI: 3Gy); verde scuro, HC S04 (dose fisica: 1.8Gy, ADCI: 1.85Gy); blu brillante, HC S05 (dose fisica: 2.8Gy, ADCI: 2.95Gy); blu scuro, HC S07 (dose fisica: 3.4Gy, ADCI: 2.35Gy); rosso scuro, HC S08 (dose fisica: 2.3Gy, dose HC dedotto: 2.5Gy, ADCI: 2Gy); verde brillante, HC S10 (dose fisica: 1.4Gy, dose HC dedotto: 1.4Gy, ADCI: 0.95Gy). Clicca qui per visualizzare una versione più grande di questa figura.
| Campioni | Dose fisica | HC dedotto Dose | ADCI stimato Dose | Dose stimata LCL | Dose stimata UCL | P-valore * |
| HCS01 | 3.1 | 3.4 | 3 | 2.3 | 3.8 | 0,117 |
| HCS08 | 2.3 | 2.5 | 2 | 1.4 | 2.7 | 0,815 |
| HCS10 | 1.4 | 1.4 | 0.95 | 0,5 | 1,55 | 0,211 |
| HCS04 | 1.8 | NA | 1.85 | 1.25 | 2,55 | 0,0293 |
| HCS05 | 2.8 | NA | 2.95 | 2.25 | 3.75 | 0.00354 |
| HCS07 | 3.4 | NA | 2.35 | 1.7 | 3.1 | 0,0002 |
Tabella 2: Dose stima risultati di campioni di prova HC.
Nota: Modificata dalla tabella 3 in Rogan et al., 20164. Le stime delle dosi ADCI precedentemente segnalati sono stati basati su immagini non filtrate e adattamento della curva è stata effettuata usando il metodo dei minimi quadrati. Qui, la curva di taratura era adatta utilizzando il metodo di massima verosimiglianza e un modello di selezione immagine contenente 3 filtri FP + "gruppo bin distanza, immagini top 250" è stato applicato prima stima di dose. Dose stimata UCLe LCL si riferiscono all'alta stima della dose e abbassare i limiti di confidenza del 95% sulla base della natura di Poisson di rendimento DC. * Il bontà quadrato chi di adattamento per distribuzione di Poisson teorica; NA: Risultati della dose manualmente dedotto non erano forniti.
Stime di dose di radiazione per campioni da Canadian Nuclear CNLS04 di laboratori, CNLS05, CNLS07, CNLS01 e CNLS08 sono illustrati nella Figura 7. La curva di taratura viene generata utilizzando campioni CNL0Gy, CNL0.5Gy, CNL1Gy, CNL2Gy, CNL3Gy e CNL4Gy. Abbiamo applicato un modello di selezione immagine composto da 6 FP filtri per tutti i campioni. I risultati con analisi statistiche sono mostrati nella tabella 3.
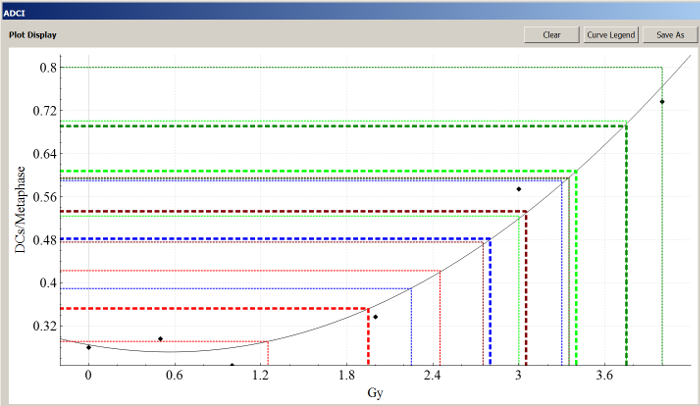
Figura 7: Screenshot di stima di Dose di campioni CNL. Quadrati neri rappresentano campioni di calibrazione. Immagini in esempi di prova e taratura vengono selezionati con 6 filtri FP. Linee spesse tratteggiate rappresentano il mapping di DCs/metafase tramite la curva di taratura per dose stimata. Sottili linee punteggiate indicano superiore e abbassare i limiti di confidenza del 95% di DCs/metafase. I codici dei campioni di prova colore: rosso brillante, CNL S04 (dose fisica: 1.8Gy, dose CNL dedotto: 1.7Gy, ADCI: 1.95Gy); rosso scuro, S05 CNL (dose fisica: 2.8Gy, dose CNL dedotto: 2.7Gy, ADCI: 3.05Gy); verde brillante, CNL S07 (dose fisica: 3.4Gy, dose CNL dedotto: 3.1Gy, ADCI: 3.4Gy); verde scuro, CNL S01 (dose fisica: 3.1Gy, ADCI: 3.75Gy); blu, CNL S08 (dose fisica: 2.3Gy, ADCI: 2.8Gy). Clicca qui per visualizzare una versione più grande di questa figura.
| Campioni | Dose fisica | CNL dedotto Dose | ADCI stimato Dose | Dose stimata LCL | Dose stimata UCL | P-valore * |
| CNLS04 | 1.8 | 1.7 | 1.95 | 1.25 | 2.45 | 0,0545 |
| CNLS05 | 2.8 | 2.7 | 3,05 | 2,75 | 3,35 | 0.325 |
| CNLS07 | 3.4 | 3.1 | 3.4 | 3 | 3.75 | 0.473 |
| CNLS01 | 3.1 | NA | 3.75 | 3,35 | > 4 | 7.63E-11 |
| CNLS08 | 2.3 | NA | 2.8 | 2.25 | 3.3 | 0,777 |
Tabella 3: Risultati di stima di dose di CNL testare campioni.
Nota: Modificata dalla tabella 3, Rogan et al., 20164. Le stime delle dosi ADCI precedentemente segnalati sono stati basati su non filtrato (HC) o manualmente selezionato (CNL) immagini e adattamento della curva è stata effettuata usando il metodo dei minimi quadrati. Qui, la curva di taratura era adatta utilizzando il metodo di massima verosimiglianza e un modello di selezione immagine contenente 3 filtri FP + "gruppo bin distanza, immagini top 250" è stato applicato prima stima di dose. Dose stimata UCL e LCL, rispettivamente, si riferiscono per dose stimata superiore e abbassare i limiti di confidenza del 95% sulla base della natura di Poisson di rendimento DC.
* Il bontà quadrato chi di adattamento per distribuzione di Poisson teorica; NA: Risultati della dose manualmente dedotto non erano disponibili.
Stima della dose di radiazione all'interno della gamma lineare della curva di calibrazione (< 1 Gy) possono essere effettuati con il software, tuttavia un valore di 1.0 è consigliato di Sigma è quello di ridurre ulteriormente la frequenza di segnalarli DCs (Figura 8).
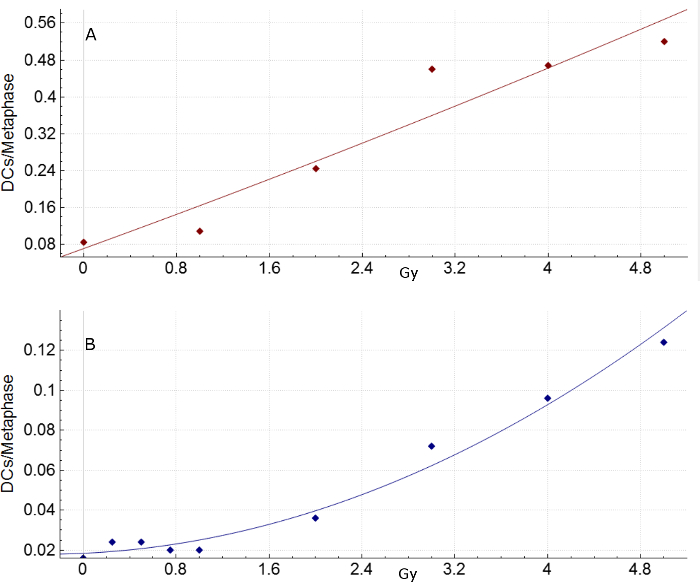
Figura 8: Screenshots delle due curve di taratura derivato da HC campioni di taratura presso Sigma diversi valori. (A) campioni di calibrazione HC: 0Gy, 2Gy, 3Gy, 4Gy e 5Gy presso Sigma = 1.5. Campioni di calibrazione di HC (B) : 0Gy, 0.25Gy, 0.5Gy, 0.75Gy, 1Gy, 2Gy, 3Gy, 4Gy, e 5Gy utilizzando SVM Sigma = 1.0. Clicca qui per visualizzare una versione più grande di questa figura.
Queste analisi indicano che ci sono piccole, ma accettabili differenze tra fisica e biologicamente inferita dose interpretate dagli esperti e dal software. La differenza tra manuale o software stima dalla dose fisica si riferisce a come "errore". L'errore nelle dosi dedotte di campioni manualmente segnati da HC e CNL è ≤ 0.3 Gy. Trattamento automatizzato dal software è meno preciso di quelli esperti, ma generalmente entro i limiti di triage AIEA-specificato di ± 0,5 Gy3. Per la maggior parte dei campioni nelle tabelle 2 e 3, il software prodotto risultati corretti all'interno di questa soglia. Tuttavia, HCS07 e CNLS01 presentano una scarsa bontà di adattamento per la distribuzione di Poisson, suggerendo che c'erano problemi potenziali in immagine e qualità DC in questi campioni che non sono stati risolti dall'applicazione dell'immagine e modelli di selezione FP. La soglia di rilevanza del valore p sembra essere eccessivamente severe in caso di HCS05, dove il software determinato con precisione la dose corretta.
Discussione
Funzionalità e limitazioni del software
Il protocollo descritto in questo documento introduce la procedura graduale tipica usata in ADCI per importare ed elaborare immagini di metafase citogenetica, creare radiazioni curve di calibrazione e stimare la dose biologica nelle persone o nei campioni esposti a sconosciuto livelli di radiazione. Tuttavia, non è necessario eseguire queste istruzioni in sequenza. Ad esempio, molti campioni di prova della dose sconosciuta possono essere elaborati e analizzati utilizzando la stessa curva di taratura pre-calcolate. Inoltre, una volta completata l'elaborazione, la selezione delle immagini e DC modelli di filtraggio può essere iterata da parte dell'utente. Applicazione di un modello di selezione immagine appropriata dipende dalle caratteristiche e qualità dei dati dell'immagine di metafase, che a sua volta si basa sia sul protocollo di laboratorio utilizzato per preparare le cellule e i criteri di rigore utilizzati per selezionare le celle con automatizzato sistemi di cattura di metafase. Morfologie di cromosoma saranno diverso tra biodosimetry e laboratori di citogenetiche, e così, i modelli di selezione immagine dovrebbero essere valutati dall'utente per determinare se i modelli di selezione immagine predefiniti forniti con il software sarà sufficienti per produrre le stime delle dosi precise, o se personalizzato modelli definiti dall'utente soglie devono essere creati. Basandoci sulla nostra esperienza, l'efficacia di modelli di selezione immagine risente l'origine e la qualità delle immagini delle cellule. Gli utenti possono progettare i propri criteri di selezione immagine utilizzando diverse combinazioni di filtri per eliminare il falso positivo DCs e modelli di selezione immagine e i valori di soglia corrispondenti per selezionare le immagini desiderate. C'è flessibilità in ingresso di curve di calibrazione e stima della dose, come coefficienti della curva lineare-quadratica e frequenze DC possono essere modificati o inseriti manualmente.
Anche se il software è completamente automatizzato, immagini possono essere recensione e selezionati manualmente. Questa funzionalità è disponibile per includere o rimuovere immagini singolarmente elaborate attraverso la funzione di Visualizzatore di microscopio nella GUI principale. Tuttavia, grazie all'automazione, il software è molto più efficiente rispetto con il manuale notante di immagini di metafase e contando DCs. Un campione costituito da 1000 immagini possa essere elaborato in 20 (tiff) per 40 min (jpg) su una workstation di prestazioni multi-core. Questo software sarà particolarmente utile in situazioni critiche o laboriosi, come eventi in cui individui multipli sono stati esposti o sono stati ritenuti sospetto di essere stati esposti a radiazioni, o dove il tempo-sensibile diagnosi e trattamento le decisioni sono fondamentali.
Rilevamento preciso e accurato elevato throughput di DCs, come pure la stima di dose sono necessari per la valutazione automatica di radiazione. Altre alternative disponibili per il software non soddisfare entrambi questi requisiti. Un'analisi citogenetica utente-assistita, basata sulle immagini (DCScore, Metasystems17) sistema richiede la verifica manuale del candidato DCs, a causa di un errore elevato tasso attribuibile alla non corretta si sovrappone tra cromosomi e il sistema non determina dose di radiazioni. DCScore non sarebbero altrettanto efficaci come ADCI in un evento di radiazioni che coinvolgono un gran numero di soggetti potenzialmente esposti. Sistemi di microscopia di grande apertura in grado di raccogliere immagini di più cellule di metafase18, tuttavia, non analizzarli. "CABAS"19 e "Stima di Dose"20 software può generare calibrazione curve e stima della dose, ma non segnano DCs. Altri saggi di biodosimetry che non sono basati su analisi DC includono H2AX fluorescenza, sonde di ibridazione in situ di fluorescenza con DNA mirati a specifici cromosomi, espressione genica, micronucleo e l'urina e biomarcatori respiratorie. Questi metodi sono meno specifici e sensibili per le radiazioni ionizzanti, possono essere più costosi, in alcuni casi, sono più che richiede tempo e generalmente non sono stati standardizzati attraverso molteplici laboratori di riferimento. La maggior parte di queste tecniche non rilevano radiazioni stabile risposte, quindi non possono essere utilizzati per la valutazione a lungo termine (> post-esposizione di 7 giorni) della dose di radiazione. Al contrario, questo può valutare gli individui fino a 90 giorni di post-esposizione e in grado di elaborare dati da qualsiasi microscopio del laboratorio di citogenetica sistema di imaging. Tuttavia, se un campione è disegnato > 4 settimane post-esposizione, sensibilità è ridotta a causa del decadimento di aberrazioni dicentrico1,2,3 e il software non risolve attualmente frequenze DC per ritardi nel prelievo individui esposti.
Questo software ha alcune limitazioni. Modelli di selezione immagine esistente selezionare immagini di metafase accettabile per la maggior parte, ma in alcuni casi, non riescono a eliminare immagini insoddisfacente, che possono ridurre la precisione del rilevamento di DC. È ancora una questione aperta come progettare un modello di selezione immagine soddisfacente che elimina tutte le cellule in metafase inadatto. Il software fornisce una stima accurata per campioni esposti a dosi di radiazioni elevate (≥ 2 Gy). Nonostante i notevoli progressi nel ridurre il numero di falsi positivi DCs16, questi oggetti non sono stati eliminati. Abbassare le cellule di metafase di qualità a bassa dose di radiazioni (soprattutto < 1 Gy) sono più inclini a rilevamento del falso positivo DC. Di conseguenza, campioni di basso dosaggio non sono stati inclusi quando si genera la curva di taratura utilizzata per la stima della dose dei campioni di prova HC. Tuttavia, se una curva contenente campioni di basso dosaggio è desiderata, un valore inferiore di SVM Sigma riduce la conta falsi positivi nei campioni di basso dosaggio ma può provocare calo dei rendimenti DC in campioni di dose elevata. Figura 8 confronta la curva HC utilizzata per la stima della dose (Sigma = 1.5) con una curva di calibrazione misura con campioni di dose aggiuntiva di basso valore inferiore del sigma SVM (1.0). Nei campioni con un numero insufficiente di cellule in metafase e/o immagini di metafase di scarsa qualità, non è possibile stimare le esposizioni biologiche a basso dosaggio, potenzialmente con conseguente deviazioni da fisici dose superiore a 0,5 Gy.
Il software non può valutare con precisione tipi di radiazioni se le loro curve dose-risposta migliore adattano un modello lineare o quasi lineare. Finora, è stato testato solo con campioni esposti a X e raggi Gamma. Se un'altra fonte di radiazione è esaminata, gli utenti devono verificare i campioni di test e calibrazione sono esposti allo stesso tipo di radiazioni. Il software utilizza la massima verosimiglianza o minimi quadrati raccordo per costruire una curva dose-risposta utilizzando un modello lineare-quadratica. Attualmente non esiste alcuna opzione per imporre una rigorosa curva lineare, appropriato per le esposizioni di particelle ad alta energia, tuttavia tale funzionalità sarà disponibile in futuro.
Sviluppo futuro
Nostri sforzi sono concentrati sul miglioramento di modelli di selezione immagine e misura della dose precisa, in particolare dei campioni esposti a dosi di radiazione bassa. Versioni successive del software fornirà errore standard misure le stime delle dosi e intervalli di confidenza sulle curve di calibrazione. Inoltre, una versione high-performance computing del software per il supercomputer Blue Gene (BG/Q, IBM) è in fase di sviluppo per la tempestiva valutazione di individui esposti in un evento di massa-incidente radiazione. Alcuni componenti del software sono già stati testati e distribuiti su questa piattaformaLass = "xrif" > 11.
Divulgazioni
PKR e JHMK stato cofondatore di CytoGnomix, che è commercializzando ADCI e detiene brevetti correlati. YL e BCS sono dipendenti di CytoGnomix. ADCI è protetto da copyright, e il metodo di localizzazione del centromero in ADCI è brevettato (Pat. N. 8.605.981; Pat tedesca. N. 112011103687).
Riconoscimenti
Siamo grati al Dr. Ruth Wilkins, radiobiologia e protezione Division presso Health Canada e Farrah Flegal, Canadian Nuclear Laboratories e il loro personale di laboratorio per l'accesso ai dati immagine metafase dai loro laboratori di citogenetica biodosimetry. Questa carta è stata sostenuta da un contratto da costruire in Canada Innovation Program a CytoGnomix (numero di serie. EN579-172270/001/SC). La versione iniziale di ADCI e sviluppo di algoritmi sono stati sostenuti dal fondo di innovazione occidentale; Scienze naturali e ingegneria Research Council of Canada (NSERC Discovery Grant 371758-2009); U.S. Public Health Service (DART-DOSE CMCR, 5U01AI091173-0); la Fondazione Canada per l'innovazione; Canada ricerca sedie e CytoGnomix Inc.
Materiali
| Name | Company | Catalog Number | Comments |
| Automated Dicentric Chromosome Identifier and Dose Estimator (ADCI) | CytoGnomix | NA | ADCI software is released in a binary installation package file for Microsoft Windows 7, 8, 8.1 and 10; 235 Mb of disk storage are required for a typical installation. The software has been tested with Intel or AMD x86-64 processors; at least 1 Gb RAM is recommended. Analyses have been benchmarked on a computer configured with an Intel I7 processor and 16 Gb RAM. Operation of ADCI requires an active license and a USB-based hardware dongle, which must remain plugged in while the software is executing. The dongle encodes the software expiry date. Each time the software is started, this date is read. The software will allow access to the program if the current date and time precedes the expiration time-date stamp. Extending an expired software license can be accomplished by obtaining a new dongle or by renewing the license with an updated key at startup. |
| Digital images of metaphase cell nuclei | Examples: Metasystems, Leica Microsystems | M-Search (Metasystems), Cytovision (Leica) software | High resolution TIFF format; typically >250 digital images generated with a microscope imaging capture system (minimum 63X magnification objective, 10X magnification ocular). |
| MSI Leopard Pro (recommended, optional) | Micro-Star International | MSI GP62 6QF 480CA Leopard Pro | Multi-core performance workstation. |
Riferimenti
- Brewen, J. G., Preston, R. J., Littlefield, L. G. Radiation-Induced Human Chromosome Aberration Yields Following an Accidental Whole-Body Exposure to60 Co γ-Rays. Radiat Res. 49 (3), 647-656 (1972).
- Bender, M. A., Gooch, P. C. Persistent Chromosome Aberrations in Irradiated Human Subjects. Radiat Res. 16 (1), 44-53 (1962).
- INTERNATIONAL ATOMIC ENERGY AGENCY. Cytogenetic Dosimetry: Applications in Preparedness for and Response to Radiation Emergencies. , IAEA: Vienna. (2011).
- Rogan, P. K., Li, Y., Wilkins, R. C., Flegal, F. N., Knoll, J. H. M. Radiation Dose Estimation by Automated Cytogenetic Biodosimetry. Radiat Prot Dosimetry. 172 (1-3), 207-217 (2016).
- Arachchige, A. S., Samarabandu, J., Knoll, J., Khan, W., Rogan, P. An image processing algorithm for accurate extraction of the centerline from human metaphase chromosomes. 2010 IEEE International Conference on Image Processing. , 3613-3616 (2010).
- Arachchige, A. S., Samarabandu, J., Knoll, J., Khan, W., Rogan, P. An Accurate Image Processing Algorithm for Detecting FISH Probe Locations Relative to Chromosome Landmarks on DAPI Stained Metaphase Chromosome Images. 2010 Canadian Conference on Computer and Robot Vision. , 223-230 (2010).
- Arachchige, A. S., Samarabandu, J., Rogan, P. K., Knoll, J. H. M. Intensity integrated Laplacian algorithm for human metaphase chromosome centromere detection. 2012 25th IEEE Canadian Conference on Electrical and Computer Engineering (CCECE). , 1-4 (2012).
- Li, Y., et al. Towards large scale automated interpretation of cytogenetic biodosimetry data. 2012 IEEE 6th International Conference on Information and Automation for Sustainability. , 30-35 (2012).
- Ranjan, R., Arachchige, A. S., Samarabandu, J., Knoll, J. H. M., Rogan, P. K. Automatic Detection of Pale Path and Overlaps in Chromosome Images using Adaptive Search Technique and Re-thresholding. International Conference on Computer Vision Theory and Applications. , 462-466 (2017).
- Arachchige, A. S., Samarabandu, J., Knoll, J. H. M., Rogan, P. K. Intensity Integrated Laplacian-Based Thickness Measurement for Detecting Human Metaphase Chromosome Centromere Location. IEEE Trans Biomed Eng. 60 (7), 2005-2013 (2013).
- Rogan, P. K., et al. Automating dicentric chromosome detection from cytogenetic biodosimetry data. Radiat Prot Dosimetry. 159 (1-4), 95-104 (2014).
- Li, Y., Knoll, J. H., Wilkins, R. C., Flegal, F. N., Rogan, P. K. Automated discrimination of dicentric and monocentric chromosomes by machine learning-based image processing. Microsc Res Tech. 79 (5), 393-402 (2016).
- Subasinghe, A., et al. Centromere detection of human metaphase chromosome images using a candidate based method. F1000Res. 5, 1565(2016).
- Shirley, B. C., Li, Y., Knoll, J. H. M., Rogan, P. K. Online manual for the Automated Dicentric Chromosome Identifier and Dose Estimator, Version 1.2. , Available from: http://adciwiki.cytognomix.com (2017).
- Wilkins, R. C., et al. Evaluation of the annual Canadian biodosimetry network intercomparisons. Int J Radiat Biol. 91 (5), 443-451 (2015).
- Liu, J., Li, Y., Wilkins, R., Flegal, F., Knoll, J. H. M., Rogan, P. K. Accurate Cytogenetic Biodosimetry Through Automation Of Dicentric Chromosome Curation And Metaphase Cell Selection. F1000Research. 6, 1396(2017).
- Schunck, C., Johannes, T., Varga, D., Lörch, T., Plesch, A. New developments in automated cytogenetic imaging: unattended scoring of dicentric chromosomes, micronuclei, single cell gel electrophoresis, and fluorescence signals. Cytogenet Genome Res. 104 (1-4), 383-389 (2004).
- Ramakumar, A., Subramanian, U., Prasanna, P. G. S. High-throughput sample processing and sample management; the functional evolution of classical cytogenetic assay towards automation. Mutat Res Genet Toxicol Environ Mutagen. 2015, 132-141 (2015).
- Deperas, J., et al. CABAS: a freely available PC program for fitting calibration curves in chromosome aberration dosimetry. Radiat Prot Dosimetry. 124 (2), 115-123 (2007).
- Ainsbury, E. A., Lloyd, D. C. Dose estimation software for radiation biodosimetry. Health Phys. 98 (2), 290-295 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon