È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
High throughput sequenziamento parallelo al Fitness misura di Leptospira interrogans trasposone inserimento mutanti durante dorato siriano criceto infezione
In questo articolo
Riepilogo
Qui descriviamo una tecnica che combina la mutagenesi transposon con sequenziamento ad alta produttività per identificare e quantificare il trasposone leptospirale mutanti in tessuti dopo una sfida di criceti. Questo protocollo può essere utilizzato per mutanti di schermo per la sopravvivenza e la diffusione negli animali e può essere applicato anche agli studi in vitro .
Abstract
In questo manoscritto, descriviamo un trasposone sequenziamento (Tn-Seq) tecnica per identificare e quantificare i mutanti di Leptospira interrogans alterati in palestra durante l'infezione di criceti dorati siriani. TN-Seq combina mutagenesi casuale trasposone con la potenza della tecnologia di sequenziamento ad alta velocità. Gli animali sono sfidati con un pool di mutanti trasposone (ingresso piscina), seguito dalla raccolta del sangue e nei tessuti un paio di giorni più tardi per identificare e quantificare il numero di mutanti in ogni organo (uscita piscine). Le piscine di uscita vengono confrontate con la piscina ingresso per valutare l'idoneità in vivo di ogni mutante. Questo approccio consente la proiezione di una grande piscina di mutanti in un numero limitato di animali. Con modifiche minori, il presente protocollo può essere eseguito con qualsiasi modello animale di leptospirosi, modelli di host di serbatoio come i ratti e i modelli di infezione acuta quali criceti, come pure gli studi in vitro . TN-Seq fornisce un potente strumento per schermo per mutanti con difetti di fitness in vivo e in vitro .
Introduzione
Identificazione di geni di virulenza per alcuni batteri, come ad esempio Leptospira spp., è difficile a causa del numero limitato di strumenti genetici disponibili. Un approccio comunemente utilizzato è la creazione di una collezione di mutanti di mutagenesi casuale trasposone seguita mediante l'identificazione del sito di inserimento in ogni mutante e virulenza prova di mutanti singoli trasposone in un modello animale. Questo approccio è che richiede tempo, costoso e richiede un gran numero di animali.
Quando mutagenesi casuale è stata sviluppata per l'agente patogeno Leptospira interrogans, sono stati identificati geni coinvolti nella virulenza di test singoli mutanti in un modello animale1. Mutanti sono stati selezionati in base a criteri quali i loro ruoli potenziali nella segnalazione o motilità o loro membrana esterna prevista o posizione superficiale. Come la maggior parte dei lipopolosaccaridi geni codificano proteine ipotetiche di funzione sconosciuta2, selezione di mutanti basato su questi limiti di criteri la possibilità di scoprire i geni di virulenza leptospirale romanzo.
Più recentemente, piscine di mutanti di L. interrogans trasposoni sono state schermate per infettività nei modelli criceto e mouse3. Ogni animale è stato sfidato con un pool di fino a 10 mutanti. Infettività di un mutante è stato segnato come positivo se è stato rilevato dalla PCR di colture ottenute da sangue e reni. Prova di PCR era laboriosa perché richiedeva una reazione di PCR individuale per ogni mutante in piscina. Perché la frequenza di ogni mutante nelle culture non è stata quantificata, l'approccio era prevenuto verso identificazione di mutanti altamente attenuati.
Descriviamo un trasposone sequenziamento (Tn-Seq) tecnica, come strategia per schermo in modo più efficiente per i geni di virulenza. TN-seq è costituito dalla creazione di una libreria di mutanti mediante mutagenesi transposon seguita da sequenziamento massivo parallelo4,5,6. Brevemente, trasposoni mutanti sono riuniti, inoculati in animali e poi recuperati da diversi organi (uscita piscine). Il DNA dalle piscine uscita è estratta e digerito con enzimi di restrizione o Tosato di sonicazione. Vengono eseguiti due cicli di PCR targeting per le giunzioni dei siti inserimento trasposoni. Questo passaggio consente di aggiungere gli adattatori necessari per il sequenziamento. I prodotti PCR ottenuti sono analizzati dall'ordinamento ad alta produttività per identificare il sito di inserzione del trasposone di ogni mutante della piscina insieme a loro abbondanza relativa, che è rispetto alla composizione iniziale della piscina del mutante.
Il vantaggio principale di questo approccio è la capacità di schermo simultaneamente un gran numero di mutanti con un numero limitato di animali. TN-Seq non richiede la conoscenza dei siti inserimento trasposone che aumenta le possibilità di scoprire nuove Leptospira-geni specifici coinvolti nella virulenza con meno tempo e una maggiore efficienza. Perché leptospirale onere nei tessuti è relativamente alto in roditore modelli suscettibili di letale infezione (tipicamente 104 -108 batteri/g di tessuto)7,8,9 pure come padroni di casa serbatoio 10,11, tessuti possono essere analizzati direttamente senza la necessità di cultura, riducendo le distorsioni a causa della crescita in vitro .
Negli studi di Tn-Seq con maggior parte dei batteri patogeni descritti fin qui, l'alta frequenza di mutagenesi inserzionale ammessi infezione con grandi vasche contenenti mutanti collettivamente avendo più inserimenti trasposone ravvicinate all'interno di ogni gene4 ,12,13,14. TN-Seq è stato sviluppato anche per un batterio per cui la frequenza di mutagenesi è molto inferiore6. Con Leptospira, una libreria di mutanti trasposone può essere generata introducendo il trasposone su un plasmide che possono essere mobilitato da coniugazione come descritto da Slamti et al.15. Tuttavia, la frequenza di mutagenesi transposon di L. interrogans è bassa. Quando il trasposone Himar1 è stato introdotto su un plasmide coniugativi, la frequenza di transconjugant è stata segnalata per essere solo 8,5 x 10-8 per ogni destinatario delle cellule con il ceppo di Lai di L. interrogans16 e rischia di essere allo stesso modo scarsa con la maggior parte altri ceppi di L. interrogans. Il protocollo descritto qui è in parte basato su quello sviluppato per Borrelia burgdorferi, in cui la frequenza di mutagenesi inserzionale trasposone è anche basso6.
Per il nostro esperimento pilota con il protocollo17, abbiamo condotto la mutagenesi transposon con L. interrogans serovar Manilae ceppo L495 a causa del successo di altri gruppi ad isolare mutanti di inserimento trasposone nel ceppo insieme al suo basso LD50 (dose letale) per virulenza1. Abbiamo proiettato 42 mutanti di Tn-Seq e identificato parecchi candidati mutanti difettosi nella virulenza, compreso due con inserimenti in un gene di adenilato ciclasi del candidato. Prova individuale dei due mutanti in criceti ha confermato che erano carenti in virulenza17.
Protocollo
Attenzione: Ceppi patogeni di Leptospira spp devono essere gestiti secondo le procedure di contenimento del livello 2 di biosicurezza (BSL-2). Devono essere indossati appropriati dispositivi di protezione individuale (PPE). Una cappa di biosicurezza classe II deve essere utilizzato per tutte le manipolazioni di patogeni Leptospira spp.
1. creazione del trasposone mutante biblioteca15
-
Trasferimento del trasposone in Leptospira spp. di coniugazione (Figura 1)
- Inoculare un volume della fase esponenziale cultura di patogeni Leptospira spp, corrispondente a 107 cellule in 10 mL di Ellinghausen-McCullough-Johnson-Harris medie (terreno EMJH)18,19. Incubare a 30 ° C con 150 giri/min agitazione fino a quando la densità raggiunge 2-8 x 108 cellule/mL.
Nota: Il tempo di raddoppiamento di leptospire patogene è 12 a 24 h, a seconda del ceppo. - Inoculare 50 µ l del donatore Escherichia coli ceppo β216320 portando il trasposone mobilizzabile plasmide (pCjTKS2)16 in 5 mL di Luria brodo (LB) completati con 0,3 mM di 2,6-diaminopimelicacid (DAP), 50 µ g/mL di kanamicina (Km) e 50 µ g/mL di spectinomicina (Spc) e posto durante la notte in un incubatore a 37 ° C a 255 giri/min.
- Inoculare 60 µ l di cellule di Escherichia coli in 3 mL di terreno EMJH completati con 0,3 mM di DAP (terreno EMJH + DAP). Incubare a 37 ° C in agitazione 255 giri/min per 3-4 h fino a un OD600nm≈ 0,3.
- Per montare l'unità di filtrazione (Figura 1), posizionare la base su un braccio laterale 125 mL flacone erlenmeyer, posizionare un filtro di acetato di cellulosa (dimensione dei pori 0,1 mm; diametro 25 mm) sulla base e morsetto l'imbuto sulla base. Collegare l'unità di filtrazione ad un sistema di vuoto.
- Aggiungere 5 mL di Leptospira spp. cultura e 0,5 mL di coltura di Escherichia coli nell'imbuto. Aspirare il liquido attraverso il filtro.
- Trasferire il filtro con la superficie dei batteri rivolto verso l'alto su un terreno EMJH + DAPplate. Incubare a 30 ° C durante la notte con il filtro verso l'alto.
- Collocare il filtro in una provetta da 15 mL contenente 1 mL di terreno EMJH e vortexare per 10 s a rilasciare i batteri nella media. Sviluppa 200 µ l della sospensione sul 5 terreno EMJH platescontaining 50 µ g/mL di Km utilizzando perline di vetro sterile di 10-15 mm 1 o una spatola sterile monouso. Avvolgere le piastre con parafilm e incubare loro testa in giù a 30 ° C per 3-4 settimane fino a quando le colonie sono visibili.
- Trasferire colonie singolarmente in 3 mL di terreno EMJH contenente 50 µ g/mL di Km (terreno EMJH + Km) a 30° C sotto agitazione 150 giri/min per 7-10 giorni fino a quando la cultura raggiunge una densità di ≈ 10/ml8.
Nota: Culture possono essere conservate a-80 ° C o in azoto liquido (con 4% glicerolo).
- Inoculare un volume della fase esponenziale cultura di patogeni Leptospira spp, corrispondente a 107 cellule in 10 mL di Ellinghausen-McCullough-Johnson-Harris medie (terreno EMJH)18,19. Incubare a 30 ° C con 150 giri/min agitazione fino a quando la densità raggiunge 2-8 x 108 cellule/mL.
-
Identificazione del trasposone inserimento sito dalla PCR annidata (Figura 2)
- Lyse 50 µ l di ogni mutante trasposone in provette PCR mediante incubazione a 95 ° C per 15 min.
Nota: Il DNA può essere purificato invece, utilizzando un kit di estrazione del DNA. - Preparare la miscela PCR con primer Deg1 e Tnk1 (tabella 3) secondo la tabella 1. Trasferire 23,7 µ l della miscela in ciascuna provetta PCR e aggiungere 1,3 µ l di cellule lisate. Eseguire il programma: 95° C per 5 min; 40 cicli: 95 ° C per 15 s, 40 ° C per 1 min, 72 ° C per 2 min; 72 ° C per 10 min.
- Rendere il mix PCR con primer Tag e TnkN1 (tabella 3) secondo la tabella 2. Trasferire 24,2 µ l della miscela in ciascuna provetta PCR e aggiungere 0,8 µ l della reazione di PCR n. 1. Eseguire il programma: 95 ° C per 5 min; 35 cicli: 95 ° C per 15 s, 55 ° C per 30 s, 72 ° C per 2 min; 72 ° C per 10 min.
- Eseguire 3 µ l dei prodotti di PCR su un gel di agarosio 1% con 1 tampone X Tris-acetato-EDTA (TAE) a 10-15 V/cm (Figura 2B).
- Purificare i prodotti di PCR dei campioni positivi utilizzando un kit di purificazione di PCR. Eluire il DNA con il volume più basso consentito dal kit per massimizzare la concentrazione del DNA recuperato.
- Invia i prodotti di PCR purificati per Sanger sequenziamento utilizzando il primer TnkN1 (tabella 3).
- Identificare i siti di inserimento confrontando la sequenza risultante con la sequenza del genoma del ceppo parentale di analisi BLASTN (http://blast.ncbi.nlm.nih.gov/) o utilizzando il SpiroScope database (http://www.genoscope.cns.fr/agc/mage)21.
- Confermare il sito di inserzione del trasposone mediante PCR utilizzando primers ricottura per le sequenze fiancheggianti di host.
Nota: Il trasposone aumenta la dimensione del selvaggio-tipo sequenza di ≈ 2 kb.
- Lyse 50 µ l di ogni mutante trasposone in provette PCR mediante incubazione a 95 ° C per 15 min.
2. esperimento animale (Figura 3)
-
Cultura di Leptospira mutanti
- Crescere individualmente ogni mutante trasposone selezionati in 10 mL di terreno EMJH + Km a 30 ° C in agitazione 150 giri/min per una densità di 107-108 le leptospire/mL.
- Contare le leptospire da microscopia del campo scuro con un contatore di Petroff-Hausser o come descritto da Miller23.
- Diluire ogni cultura nel terreno EMJH alla stessa densità, per esempio 106 cellule/mL.
- Per assemblare l'ingresso piscina, mescolare insieme le culture diluite in volumi uguali.
Nota: Includere controlli in ingresso piscina aggiungendo mutanti con difetti di fitness noto come loa2217,24 e mutanti con fitness inalterato come ligB17,25, rispettivamente. Piscine di mutanti possono essere memorizzate con 4% glicerolo a-80 ° C o in azoto liquido.
-
Sfida
Nota: I metodi descritti qui sono stati approvati dal comitato di uso (protocollo #09018-14) e Veterans Affairs Greater Los Angeles istituzionale Animal Care.- Iniettare per via intraperitoneale 1 mL di ingresso piscina per ogni animale con una siringa da insulina U-100 con ago 26 x ½".
Nota: Nell'esperimento pilota, 8 animali sono stati sfidati con 1 mL di ingresso piscina, vale a dire, 106 batteri totale17. L'infezione è stato permesso di procedere per 4 giorni prima dell'eutanasia. - Raccogliere 10 mL di ingresso piscina e girare per 20 min a 3.220 x g. Rimuovere attentamente il sopranatante senza disturbare il pellet. Conservare il pellet cellulare a - 80 ° c fino all'utilizzo (punto 3.1.3).
- Monitorare gli animali ogni giorno fino a che vengano terminati all'endpoint pre-determinato. Pesare i criceti ogni giorno e cercare i criteri dell'endpoint: perdita di appetito, andatura o respirazione di difficoltà, prostrazione, pelo arruffato, o perdita di > 10% del peso massimo raggiunto.
- Iniettare per via intraperitoneale 1 mL di ingresso piscina per ogni animale con una siringa da insulina U-100 con ago 26 x ½".
-
Esperimento in vitro
- Il giorno della sfida, inoculare 3 boccette di 25 mL di terreno EMJH + Km con 5 mL di ingresso piscina. Coltivare culture a 30 ° C sotto agitazione 150 giri/min.
- Contare le leptospire quotidianamente utilizzando uno dei metodi della sezione 2.1.2. Quando la densità raggiunge ≈ 1 x 108/mL, rotazione verso il basso ogni sospensione per 20 min a 3.220 x g.
- Conservare il pellet di cellule a-80 ° C fino all'utilizzo.
-
Raccolta e conservazione dei tessuti
- Eutanasia degli animali per inalazione isoflurane seguita da toracotomia bilaterale26.
- Raccogliere immediatamente 1-2 mL di sangue cardiaca puntura con una siringa da 3 mL e un ago 25 x 5/8". Trasferire il sangue nella provetta contenente EDTA. Mescolare per inversione, 5 o 6 volte.
- Raccogliere un rene e ⅓ a ½ del lobo mediano ventrale del fegato in provette per criogenia.
- Negozio di tessuti a-80 ° C fino all'utilizzo.
3. costruzione delle librerie Genomic per High-throughput sequenziamento (Figura 4)
-
Estrazione del DNA
- Estrazione di DNA da sangue.
- Trasferire 100 µ l di sangue dalla provetta EDTA ad un tubo del microcentrifuge.
- Purificare il DNA utilizzando un kit di estrazione del DNA. Seguire le istruzioni del produttore.
- Estrazione del DNA dai tessuti.
- Utilizzo di bisturi e forbici, tagliare a dadini tra 50-80 mg di ogni organo in piccoli pezzi (1 x 1 mm) e trasferirli in una provetta asciutta sterile con tappo a vite. Misurare il peso del tessuto con una bilancia di precisione.
- Aggiungere 500 µ l di PBS sterile nel tubo.
- Omogeneizzare campioni utilizzando il disgregatore per 1 min a 5 movimenti al secondo.
- Calcolare il volume corrispondente a 25 mg di tessuto usando la seguente equazione:
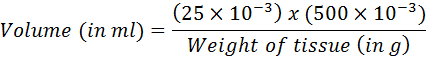
- Trasferire il volume calcolato nel kit di estrazione del DNA. Procedere con la purificazione del DNA seguendo le istruzioni del produttore.
- Estrazione del DNA dalle culture ingresso piscina e in vitro .
- Scongelare il pellet batterico a temperatura ambiente per 5-10 min.
- Procedere con l'estrazione di DNA seguendo le istruzioni che accompagnano il kit.
- Archivio DNA a-80 ° C fino all'utilizzo.
- Estrazione di DNA da sangue.
-
Il DNA di taglio (Figura 5)
- Trasferire 50 µ l di DNA estratto in una provetta da microcentrifuga da 1,5 mL.
- Disporre le provette nel rack del sonicatore Coppa corno riempito con acqua fredda (4 ° C).
- Far funzionare il sonicatore per 3 min a 80% intensità con 10 s su impulso e 5 s off impulso.
Attenzione: Indossare cuffie o tappi per le orecchie per proteggere l'udito. - Eseguire 2,5 µ l del tranciato DNA su un gel di agarosio al 2% per confermare che la maggior parte del DNA è < 600 bp in dimensione.
-
Aggiunta della C-coda
- Misurare la concentrazione di DNA con uno spettrofotometro di piccolo volume.
- Calcolare il volume corrispondente a 500 ng di DNA utilizzando la seguente equazione:
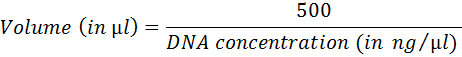
- Preparare tailing reazione (tabella 4). Incubare 1h a 37 ° C; inattivare a 75 ° C per 20 min.
Nota: Per i campioni che necessitano di regolazione per un volume superiore a 14,5 µ l, aumentare il volume finale della reazione a 40 µ l e di conseguenza scala i componenti rimanenti. - Pulire i campioni con un kit di purificazione di PCR. Eluire il DNA con 12 µ l di tampone di eluizione.
-
PCR annidata
- PCR #1
- Preparare miscele PCR secondo tabelle 3 e 5. Trasferire 22 µ l del mix biblioteca in ciascuna provetta PCR e aggiungere 3 µ l di DNA purificato. Procedere allo stesso modo con il mix di controllo.
Nota: Primer TnkN317 è specifico per il trasposone e primer olj3766 è specifico per la C-coda. Il mix di controllo manca il primer di TnkN3, che si rivolge specificamente il trasposone. - Eseguire il seguente programma: 95 ° C per 2 min; 24 cicli: 95 ° C per 30 s, 60 ° C per 30 s, 72 ° C per 2 min; 72 ° C per 2 min.
- Preparare miscele PCR secondo tabelle 3 e 5. Trasferire 22 µ l del mix biblioteca in ciascuna provetta PCR e aggiungere 3 µ l di DNA purificato. Procedere allo stesso modo con il mix di controllo.
- PCR #2.
- Preparare le miscele PCR seconda secondo tabelle 3 e 6. Trasferire µ l 49 del mix biblioteca in ciascuna provetta PCR e aggiungere 1 µ l di reazione di PCR n. 1 biblioteca. Trasferire 24,5 µ l del controllo mix in ciascuna provetta PCR e aggiungere 0,5 µ l del controllo PCR n. 1.
Nota: Primer pMargent2 è specifico per il trasposone, e l'IP primer6 contengono sei-base-accoppiamenti barcode e sequenze specifiche riconosciuti dalla piattaforma di sequenziamento di nuova generazione. - Eseguire il seguente programma: 95 ° C per 2 min; 18 cicli: 95 ° C per 30 s, 60 ° C per 30 s, 72 ° C per 2 min; 72 ° C per 2 min.
- Preparare le miscele PCR seconda secondo tabelle 3 e 6. Trasferire µ l 49 del mix biblioteca in ciascuna provetta PCR e aggiungere 1 µ l di reazione di PCR n. 1 biblioteca. Trasferire 24,5 µ l del controllo mix in ciascuna provetta PCR e aggiungere 0,5 µ l del controllo PCR n. 1.
- Eseguire 3 µ l su un gel di agarosio al 2%. La biblioteca dovrebbe mostrare una sbavatura con la maggior parte del segnale tra 200 a 600 bp (Figura 6) e nessuna amplificazione per la reazione di controllo.
- PCR #1
-
Purificazione dei prodotti di PCR.
Nota: Pulire le librerie genomiche con un kit di purificazione di PCR seguendo le istruzioni del produttore. Eluire il DNA con 30 µ l di tampone di eluizione. - Misurare la concentrazione di DNA usando un fluorimetro. Media 2 o 3 letture.
- Calcolare il volume di ogni libreria equivalente a 15 o 20 ng utilizzando l'equazione seguente:
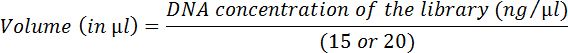
-
Mescolare tutte le librerie insieme secondo i calcoli precedenti. Determinare la concentrazione molare del DNA con equazione del seguente sito Web:
http://www.MOLBIOL.edu.ru/eng/scripts/01_07.html
Attenzione: I requisiti di volume e concentrazione di DNA dipendono la piattaforma di sequenziamento.
4. high-throughput sequenziamento e analisi dei dati
- Sequenziamento
- Librerie di sequenza come 64 bp singolo fine letture utilizzando il pMargent3 di primer di sequenziamento personalizzato e il primer di sequenziamento commerciale standard. La piattaforma di sequenziamento vi fornirà i file FastQ con tutte le letture di sequenziamento.
- Analisi con software Galaxy
- Scaricare il file di sequenza del genoma
- Nella homepage del database SpiroScope (Vedi punto 1.2.7 per link), l'organismo utilizzato per l'esperimento di selezionare e fare clic su "LOAD in GENOME BROWSER".
- Nella barra degli strumenti (nella parte superiore della homepage), selezionare "ricerca/Export > Scarica dati", nella riga "Sequenza (fasta)", fare clic su "Genoma" per scaricare la sequenza.
- Aprire il file con blocco note (PC) o TextEdit (Mac) e rinominare il cromosoma (ad esempio "ChrI"). Mantenere il formato fasta.
- Seguire la stessa procedura per scaricare la sequenza del cromosoma II. Combinare entrambe le sequenze del cromosoma in un file singolo con estensione txt copiando e incollando.
Nota: Sequenze del cromosoma possono anche essere scaricati dal sito Web di NCBI (https://www.ncbi.nlm.nih.gov) e combinati in un file singolo con estensione txt.
- Caricare file sul server Galaxy
Nota: Galaxy è un open source, la piattaforma web-based per la gestione dei dati bioinformatica intensivo i flussi di lavoro27,28,29,30 e può essere letta in https://usegalaxy.org/.- Nel menu Strumenti, selezionare "Ottieni dati > Carica File dal tuo computer". Trascinare i file. fastq generati dalla piattaforma di sequenziamento nella finestra poi cliccare "Start".
- Seguire la stessa procedura per caricare il file di txt Leptospira sequenza del genoma.
- Legge dello sposo
- Selezionare "NGS: QC e manipolazione > FASTQ Groomer" dal menu strumenti.
- Accanto a File di pulirsi, selezionare le librerie caricate al punto 4.2.2. In Input FASTQ qualità Spartiti tipo, selezionare il sistema di sequenziamento appropriato. Per le opzioni avanzate, lasciare Hide Advanced Office selezionati.
- Fare clic su "Esegui".
- Rimuovere gli artefatti di sequenziamento
- Selezionare "NGS: QC e manipolazione > rimuovere gli artefatti di sequenziamento". Accanto alla biblioteca per filtrare, selezionare i file curati generati nel passaggio 4.2.3. Fare clic su "Esegui".
- Rimuove le sequenze di C-coda
Nota: Ripetere questi passaggi una volta o due volte per assicurare tutti i C-code vengono rimossi.- Selezionare "NGS: QC e manipolazione > Clip sequenze adattatore".
- Selezionare o immettere quanto segue:
Biblioteca a clip: selezionare i file generati nel passaggio 4.2.4.
Lunghezza di sequenza minima: 15
Fonte: Inserisci sequenza personalizzata
Immettere la sequenza di ritaglio personalizzato: CICCARINO
Immettere il valore diverso da zero per mantenere le sequenze di adattatore e basi di x che lo seguono: 0
Scartare le sequenze con sconosciuti (N) basi: Sì
Opzioni di output: uscita sequenze tagliate e non smussato - Fare clic su "Esegui".
- Rimuove le sequenze di adattatore
- Selezionare "NGS: QC e manipolazione > Clip sequenze adattatore".
- Selezionare o immettere quanto segue:
- Biblioteca a clip: selezionare file generati al punto 4.2.5.
- Lunghezza di sequenza minima: 15
- Fonte: Inserisci sequenza personalizzata
- Immettere la sequenza di ritaglio personalizzato: CGTATGCCGTCTTCTGCTTG
- Immettere il valore diverso da zero per mantenere le sequenze di adattatore e basi di x che lo seguono: 0
- Scartare le sequenze con sconosciuti (N) basi: Sì
- Opzioni di output: uscita sequenze tagliate e non smussato
- Fare clic su "Esegui".
- Letture di filtro basate sulla loro qualità
- Selezionare "NGS: QC e manipolazione > filtro di qualità".
Nota: Questo strumento consente di selezionare letture basate su punteggi di qualità. - Selezionare le opzioni seguenti:
Libreria per filtrare: selezionare i file generati nel passaggio 4.2.6.
Valore di cut-off di qualità: 20
Per cento delle basi nella sequenza che deve avere qualità uguale o superiore al valore di cut-off: 95 - Fare clic su "Esegui".
Nota: Con queste impostazioni, vengono scartate letture più brevi rispetto a 20 nucleotidi o con un punteggio di qualità di 20 o meno per il 95% dei cicli. Adattare le impostazioni di rigore all'esperimento.
- Selezionare "NGS: QC e manipolazione > filtro di qualità".
- Mappa legge32
- Selezionare "NGS: mappatura > Bowtie2"32.
- I campi nella finestra principale, selezionare le opzioni seguenti:
È questa libreria singola o accoppiata: monolato
File FASTQ: selezionare la libreria di filtrato per qualità dal passaggio 4.2.7.
Scrivere non allineate (in formato fastq) letture per separare file: no
Scrivere allineate (in formato fastq) letture per separare file: no
Vi si seleziona un genoma di riferimento dalla cronologia o utilizzare un indice incorporato?: utilizzare un genoma dalla storia e costruire indice
Selezionare il genoma di riferimento: selezionare il file di genome.txt di Leptospira caricato al punto 4.2.2.
Set letto informazioni gruppi?: non impostato
Modalità scegliere analisi: 1: impostazione predefinita solo
Si desidera utilizzare le impostazioni predefinite?: No, basta utilizzare impostazioni predefinite
Salvare le statistiche di mappatura bowtie2 alla storia: No
I parametri delle risorse di lavoro: utilizzare i parametri predefiniti lavoro risorsa - Fare clic su "Esegui" per allineare letture al genoma.
- Convertire i file
- Selezionare "NGS: SAMtools > BAM-al-SAM convertire BAM a SAM".
- Selezionare le opzioni seguenti:
File BAM per convertire: selezionare mappato biblioteca dal passaggio 4.2.8.
Opzioni di intestazione: includere intestazione nell'output di SAM (-h) - Fare clic su "Esegui".
- Convertire i file
- Selezionare "NGS: SAMtools > convertire SAM ad intervallo".
- Selezionare le opzioni seguenti:
Selezionare dataset da convertire: selezionare il file di libreria mappata SAM generato nel passaggio 4.2.9.
Stampa tutto?: Sì - Fare clic su "Esegui".
- Legge di ordinamento
- Selezionare "Ordina e filtra > ordinare i dati in ordine crescente o decrescente".
- Selezionare le opzioni seguenti:
Dataset di sorta: selezionare il file di intervallo generato nel passaggio 4.2.10.
colonna: 2
sapore: ordine numerico
tutto in: ordine crescente - Fare clic su "Esegui".
- Selezionare letture del cromosoma di corrispondenza
- Selezionare "Ordina e filtra > selezionare le righe che corrispondono a un'espressione".
- Selezionare le opzioni seguenti:
Selezionare le righe da: selezionare il file con letture ordinate generato nel passaggio 4.2.11.
che: corrispondenza
il modello: immettere il nome del cromosoma come determinato al punto 4.2.1.3 (ad es., "ChrI"). - Fare clic su "Esegui".
- Selezionare letture corrispondenti cromosoma II
Procedere seguendo passo 4.2.12. - Gruppo di legge secondo i siti di inserzioni nel cromosoma I
- Selezionare "Join, sottrarre e gruppo > raggruppare dati da una colonna ed eseguire operazione di aggregazione su altre colonne".
- Selezionare le opzioni seguenti:
Selezionare i dati: selezionare il file risultante dal passaggio 4.2.12.
Gruppo di colonna: 2
Ignora maiuscole/minuscole mentre raggruppamento?: No
Ignorare le righe che iniziano con tali caratteri: Ø
Funzionamento > + operazione di inserimento
Tipo: conteggio
Colonna: 2
Arrotondare il risultato al numero intero più vicino: NO - Fare clic su "Esegui".
- Gruppo legge secondo i siti di inserzione nel cromosoma II
Procedere seguendo passo 4.2.14. - Siti d'inserzione sorta sul cromosoma ho
- Selezionare "Ordina e filtra > ordinare i dati in ordine crescente o decrescente".
- Selezionare le opzioni seguenti:
Dataset di sorta: Selezionare file dal passaggio 4.2.14.
su colonna: 1
sapore: ordine numerico
tutto in: ordine crescente - Fare clic su "Esegui".
- Sorta di inserimento siti sul cromosoma II
Procedere seguendo passo 4.2.16.
- Scaricare il file di sequenza del genoma
- Analisi statistica
- Trasferire i dati di Galaxy in fogli di calcolo copiando e incollando le due colonne da passaggi 4.2.16. e 4.2.17. in un file di Excel.
Nota: La prima colonna è la coordinata del nucleotide del sito di inserzione del trasposone, e la seconda colonna è il numero di letture in ogni sito di inserimento. - Identificare il gene che harboring il trasposone utilizzando la coordinata del nucleotide fornita nella tabella.
Nota: ad esempio, un trasposone inserito a nucleotide #30718 del cromosoma I è situato nel gene LIC10024 , che si estende su nucleotidi 29263-31539 in cromosoma I. - Calcolare frequenze relative (F) per ogni mutante in ogni tessuto e in ingresso piscina seguendo l'equazione seguente:
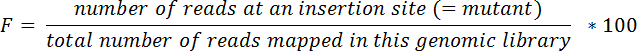
- Calcolare i rapporti di ingresso/uscita (R) per ogni mutante e tessuto utilizzando l'equazione seguente:
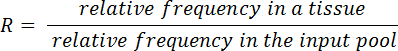
- Wilcoxon firmare-allinea la prova
- Normalizzare tutti i rapporti di input/output impostando il rapporto mediano per ogni tessuto in ogni animale a 1.0. Un rapporto di 1.0 è neutro, > 1.0 è svantaggioso, e < 1.0 è vantaggioso33.
- Confronta rapporti di input/output a 1.0 (neutro fitness) utilizzando il rango di Wilcoxon con valori di P < 0.05 considerato statisticamente significativo.
- Trasferire i dati di Galaxy in fogli di calcolo copiando e incollando le due colonne da passaggi 4.2.16. e 4.2.17. in un file di Excel.
Risultati
Creazione di una libreria di mutanti trasposone in L. interrogans mediante coniugazione richiede un'unità di filtrazione, come mostrato nella Figura 1. Abbiamo recuperato 100-200 transconjugants da ogni accoppiamento.
Il sito di inserzione del trasposone è identificato in ogni mutante ordinando il prodotto PCR generato da PCR semi-casuale che gli obiettivi alla fine del trasposone e accog...
Discussione
Anche se il nostro esperimento pilota per criceto sfidato intraperitonealmente con 42 L. interrogans mutanti i risultati sono presentati17, ci aspettiamo che piscine più grandi di mutanti possono essere schermate da Tn-segg. Perché la frequenza di transconjugants è bassa (100-200 transconjugants/accoppiamento), parecchi accoppiamenti sono necessari per generare un numero sufficiente di mutanti per grandi esperimenti di Tn-Seq. Mantenere un gran numero di mutanti in colture liquide pres...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da un premio di merito di affari di veterani (a D.A.H.) e un istituto superiore di sanità concede R01 AI 034431 (per D.A.H.).
Materiali
| Name | Company | Catalog Number | Comments |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K4000 | |
| 2,6-diaminopimelic acid | Sigma-Aldrich | D1377 | |
| Spectinomycin dihydrochloride pentahydrate | Sigma-Aldrich | S4014 | |
| Axio Lab A1 microscope with a darkfieldcondenser | Zeiss | 490950-001-000 | |
| DNeasy blood and tissue kit | Qiagen | 69504/69506 | |
| MinElute PCR Purification | Qiagen | 28004/28006 | |
| QIAquick PCR purification kit | Qiagen | 28104/28106 | |
| Model 505 Sonic Dismembrator | Fisher Scientific | FB-505 | |
| 2.5" Cup horn | Fisher Scientific | FB-4625 | |
| Bead Ruptor 24 | Omni International | 19-010 | Step 3.1.2.4 |
| Terminal deoxynucleotidyl transferase | Promega | M828C | |
| Master mix Phusion | Thermo Scientific | F531 | Preparation of genomic libraries, step 3.4. |
| DreamTaq Master Mix | Thermo Scientific | K9011/K9012 | Identification of the transposon insertion site, step 1.2. |
| dCTP | Thermo Scientific | R0151 | |
| ddCTP | Affymetrix/ USBProducts | 77112 | |
| T100 Thermal cycler | BioRad | 1861096 | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | step 3.6. |
| Qubit dsDNA HS assay kit | Invitrogen | Q32851/Q32854 | step 3.6. |
| Qubit assay tubes | life technologies | Q32856 | step 3.6. |
| PBS pH 7.2 (1X) | Gibco | 20012-027 20012-050 | |
| Disposable scalpel No10 | Feather | 2975#10 | |
| Plastic K2 EDTA 2 ml tubes | BD vacutainer | 367841 | |
| syringe U-100 with 26G x ½” needle | BD vacutainer | 329652 | IP challenge, step 2.2.1. |
| 3 mL Luer-Lok tip syringe | BD vacutainer | 309657 | Cardiac puncture, step 2.4.2. |
| 25G X 5/8” needle | BD vacutainer | 305901 | Cardiac puncture, step 2.4.2. |
| 25 mm fritted glass base with stopper | EMD Millipore | XX1002502 | Filtration unit system, step 1.1.7. |
| 25 mm aluminum spring clamp | EMD Millipore | XX1002503 | Filtration unit system, step 1.1.7. |
| 15 ml borosilcate glass funnel | EMD Millipore | XX1002514 | Filtration unit system, step 1.1.7. |
| 125 ml side-arm Erlenmeyer flask | EMD Millipore | XX1002505 | Filtration unit system, step 1.1.7. |
| Acetate-cellulose filter VVPP (pore size 0.1 mm; diameter 25 mm) | EMD Millipore | VVLP02500 |
Riferimenti
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infect Immun. 77 (2), 810-816 (2009).
- Adler, B., Lo, M., Seemann, T., Murray, G. L. Pathogenesis of leptospirosis: the influence of genomics. Vet Microbiol. 153 (1-2), 73-81 (2011).
- Marcsisin, R. A., et al. Use of a high-throughput screen to identify Leptospira mutants unable to colonize the carrier host or cause disease in the acute model of infection. J Med Microbiol. 62, 1601-1608 (2013).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat Methods. 6 (10), 767-772 (2009).
- van Opijnen, T., Lazinski, D. W., Camilli, A. Genome-wide fitness and genetic interactions determined by Tn-seq, a high-throughput massively parallel sequencing method for microorganisms. Curr Protoc Microbiol. 36, 1E.3.1-1E.3.24 (2015).
- Troy, E. B., et al. Understanding barriers to Borrelia burgdorferi dissemination during infection using massively parallel sequencing. Infect Immun. 81 (7), 2347-2357 (2013).
- Wunder, E. A., et al. Real-time PCR reveals rapid dissemination of Leptospira interrogans after intraperitoneal and conjunctival inoculation of hamsters. Infect Immun. 84 (7), 2105-2115 (2016).
- Lourdault, K., Aviat, F., Picardeau, M. Use of quantitative real-time PCR for studying the dissemination of Leptospira interrogans in the guinea pig infection model of leptospirosis. J Med Microbiol. 58, 648-655 (2009).
- Coutinho, M. L., et al. Kinetics of Leptospira interrogans infection in hamsters after intradermal and subcutaneous challenge. PLoS Negl Trop Dis. 8 (11), e3307 (2014).
- Athanazio, D. A., et al. Rattus norvegicus as a model for persistent renal colonization by pathogenic Leptospira interrogans. Acta Trop. 105 (2), 176-180 (2008).
- Ratet, G., et al. Live imaging of bioluminescent Leptospira interrogans in mice reveals renal colonization as a stealth escape from the blood defenses and antibiotics. PLoS Negl Trop Dis. 8 (12), e3359 (2014).
- Gutierrez, M. G., Yoder-Himes, D. R., Warawa, J. M. Comprehensive identification of virulence factors required for respiratory melioidosis using Tn-seq mutagenesis. Front Cell Infect Microbiol. 5 (78), (2015).
- Gawronski, J. D., Wong, S. M., Giannoukos, G., Ward, D. V., Akerley, B. J. Tracking insertion mutants within libraries by deep sequencing and a genome-wide screen for Haemophilus genes required in the lung. Proc Natl Acad Sci U S A. 106 (38), 16422-16427 (2009).
- Gallagher, L. A., Shendure, J., Manoil, C. Genome-scale identification of resistance functions in Pseudomonas aeruginosa using Tn-seq. MBio. 2 (1), (2011).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods Mol Biol. 859, (2012).
- Picardeau, M. Conjugative transfer between Escherichia coli and Leptospira spp. as a new genetic tool. Appl Environ Microbiol. 74 (1), 319-322 (2008).
- Lourdault, K., Matsunaga, J., Haake, D. A. High-throughput parallel sequencing to measure fitness of Leptospira interrogans transposon insertion mutants during acute infection. PLoS Negl Trop Dis. 10 (11), e0005117 (2016).
- Ellinghausen, H. C., McCullough, W. G. Nutrition of Leptospirapomona and growth of 13 other serotypes: Fractionation of oleic albumin complex and a medium of bovine albumin and polysorbate 80. Am J Vet Res. 26, 45-51 (1965).
- Johnson, R. C., Harris, V. G. Differentiation of pathogenic and saprophytic letospires. I. Growth at low temperatures. J Bacteriol. 94 (1), 27-31 (1967).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPa) conjugative machineries and their cognate Escherichia coli host strains. Res Microbiol. 156 (2), 245-255 (2005).
- Vallenet, D., et al. MicroScope: a platform for microbial genome annotation and comparative genomics. Database (Oxford). , bap021 (2009).
- Goodman, A. L., Wu, M., Gordon, J. I. Identifying microbial fitness determinants by insertion sequencing using genome-wide transposon mutant libraries. Nat. Protocols. 6 (12), 1969-1980 (2011).
- Miller, J. . Spirochetes in body fluids and tissues manual of investigative methods. , 22-23 (1971).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathog. 3 (7), (2007).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infect Immun. 76 (12), 5826-5833 (2008).
- Haake, D. A. Hamster model of leptospirosis. Curr Protoc Microbiol. 12, 2 (2006).
- Giardine, B., et al. Galaxy: a platform for interactive large-scale genome analysis. Genome Res. 15 (10), 1451-1455 (2005).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86 (2010).
- Blankenberg, D., et al. Galaxy: a web-based genome analysis tool for experimentalists. Curr Protoc Mol Biol. (Chapter 19), 11-21 (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Res. 8 (44), (2016).
- McCoy, K. M., Antonio, M. L., van Opijnen, T. MAGenTA; a Galaxy implemented tool for complete Tn-Seq analysis and data visualization. Bioinformatics. , 1367 (2017).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Kamp, H. D., Patimalla-Dipali, B., Lazinski, D. W., Wallace-Gadsden, F., Camilli, A. Gene fitness landscapes of Vibrio cholerae at important stages of its life cycle. PLoS Pathog. 9 (12), e1003800 (2013).
- Poggi, D., Oliveira de Giuseppe, P., Picardeau, M. Antibiotic resistance markers for genetic manipulations of Leptospira spp. Appl Environ Microbiol. 76 (14), 4882-4885 (2010).
- van Opijnen, T., Camilli, A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms. Nat Rev Microbiol. 11 (7), 435-442 (2013).
- Pappas, C. J., Picardeau, M. Control of gene expression in Leptospira spp. by Transcription activator-like effectors demonstrates a potential role for LigA and LigB in Leptospira interrogans virulence. Appl Environ Microbiol. 81 (22), 7888-7892 (2015).
- Koskiniemi, S., Sun, S., Berg, O. G., Andersson, D. I. Selection-driven gene loss in bacteria. PLoS Genet. 8 (6), e1002787 (2012).
- Troy, E. B., et al. Global Tn-seq analysis of carbohydrate utilization and vertebrate infectivity of Borrelia burgdorferi. Mol Microbiol. 101 (6), 1003-1023 (2016).
- Ramsey, M. E., et al. A high-throughput genetic screen identifies previously uncharacterized Borrelia burgdorferi genes important for resistance against reactive oxygen and nitrogen species. PLoS Negl Trop Dis. 13 (2), e1006225 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon