È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutazione In Vitro del DNA danni utilizzando test della cometa
In questo articolo
Riepilogo
Il test della cometa è un metodo efficace per rilevare danni al DNA compreso singolo e doppio filamento di DNA si rompe. Descriviamo la cometa alcalino e neutro saggi per misurare il danno del DNA in cellule di cancro per valutare l'effetto terapeutico della chemioterapia.
Abstract
Danno del DNA è un fenomeno comune per ogni cella durante la sua esistenza ed è definito come un'alterazione della struttura chimica del DNA genomico. Terapie del cancro, come la radio e la chemioterapia, introducono una quantità enorme di ulteriori danni al DNA, che conduce all'arresto del ciclo cellulare e apoptosi per limitare la progressione del cancro. Valutazione quantitativa del danno del DNA durante la terapia sperimentale del cancro è un passo fondamentale per giustificare l'efficacia di un agente genotossico. In questo studio, ci concentriamo su un'analisi di elettroforesi unicellulare, noto anche come il test della cometa, che si può quantificare singolo e doppio filamento DNA si rompe in vitro. Il test della cometa è un metodo di quantificazione del danno del DNA che è efficiente e facile da eseguire, e ha basse esigenze di budget di tempo e un'elevata riproducibilità. Qui, si evidenzia l'utilità di analisi della cometa per uno studio preclinico valutando l'effetto genotossico olaparib/temozolomide della terapia di combinazione di cellule di glioma U251.
Introduzione
Il test della cometa è stato sviluppato da Ostling e Johanson nel 1984 dimostrando che la migrazione del DNA frammenti dai nuclei in un neutro condizione1. La tecnica venne successivamente sviluppata da Singh et al., mostrando che una condizione alcalina notevolmente aumentata la specificità e la riproducibilità del test2. Da allora, l'analisi della cometa neutro principalmente è usato per rilevare le rotture del doppio filamento del DNA, mentre l'analisi alcalina della cometa è più sensibile per piccoli importi di danno del DNA, compreso il singolo e doppio filamento del DNA si rompe, siti alcali-labili, DNA-DNA o Cross-linking del DNA-proteina e rotture del singolo filamento del DNA connesse con l'asportazione incompleta ripristino siti3,4. Entrambe le analisi permettono la visualizzazione di DNA frammentato e forniscono un modo semplice per valutare quantitativamente il danno del DNA. Il test della cometa è considerato come un metodo sensibile per gli studi tossicologici genetici in vitro e in vivo ed è applicabile ad aree di ricerca diversi, quali la selezione precoce di droga-candidato, monitoraggio ambientale, biomonitoraggio umano, e fondamentale ricercare in danno del DNA e riparazione5.
Il principio del test è che, in un campo elettrico, DNA frammentato migra fuori dal corpo di nucleoide (conosciuto anche come la "testa della cometa") e forma una macchia di DNA in gel di agarosio (noto anche come "coda di cometa"). Con la colorazione del nucleotide nella misura di danno del DNA può essere quantificata analizzando "comete" formate da questo elettroforesi unicellulare. Calcolo del momento di coda può aiutare ulteriormente a confrontare il danno del DNA fra differenti gruppi sperimentali. Rispetto ai tradizionali metodi di rilevamento dei danni del DNA, il test della cometa è diretto, sensibile, poco costoso e relativamente semplice.
La radioterapia e la chemioterapia è strategie comuni per il trattamento del cancro generando filo singolo e doppio filamento DNA si rompe in cromosomi6. Il recente progresso nella inibitori di riparazione del DNA permette un più efficace effetto genotossico di chemioterapia di combinazione e quindi, potenzialmente riduce effetti collaterali sistemici quali l'anemia, infezioni e midollo osseo soppressione7, 8. in questo studio, abbiamo dimostrato l'inchiesta di un inibitore di poli (ADP-ribosio) polimerasi (PARP), olaparib (Ola)9. PARP è una proteina nucleare abbondante ed è responsabile della riparazione bassa di asportazione del DNA formando un polimero di poli (ADP-ribosio)10. Temozolomide (TMZ) è un agente alchilante orale disponibile ed è stato ampiamente usato per il trattamento di pazienti di glioma. Utilizzando l'analisi della cometa per quantificare il danno del DNA, dimostriamo che combinando olaparib con temozolomide profondamente migliora il danno del DNA in cellule del glioma, che suggerisce la terapia di combinazione di olaparib/temozolomide è una strategia efficace per il trattamento di gliomi, confrontato con temozolomide solo11.
Protocollo
1. preparare i reagenti
- 1X PBS
- diluire 100 mL di PBS 10X con 900 mL dH 2 O e regolare il pH a 7,4 utilizzando un pH-metro. Conservare a temperatura ambiente.
- Soluzione di lisi (LS)
- preparare 2,5 M NaCl, EDTA disodico 100 mM, 10 mM Tris base e 200mm NaOH in 900 mL dH 2 O; prende comunemente circa 20 min per consentire la miscela dissolvere completamente. Regolare il pH a 10 utilizzando un pH-metro. Aggiungere 1% sodio lauryl sarcosinate e 1% Triton X-100 e regolare il volume finale a 1.000 mL. Raffreddare a 4 ° C per almeno 30 min prima dell'uso.
- Soluzione l'elettroforesi alcalina (AES), pH > 13
- preparare 200mm NaOH e 1 mM EDTA disodico in 800 mL dH 2 O. regolare il pH e assicurarsi che è pH > 13. Regolare il volume finale a 1.000 mL. Fare fresco prima dell'uso e raffreddare a 4 ° C per almeno 30 min prima dell'uso.
- Neutro elettroforesi sollution (NES)
- preparare 1.000 mL di tampone di elettroforesi neutro miscelando 100 mM Tris base e 300 mM sodio acetato a 1.000 mL dH 2 O. regolare il pH a 9.0 con acido acetico glaciale. Raffreddare a 4 ° C per almeno 30 min prima dell'uso.
- Soluzione di precipitazione del DNA (DPS)
- preparazione di 10 mL brodo di 7.5 M ammonio acetato. Per 50 mL di soluzione di precipitazione del DNA, mescolare 6,7 mL 7,5 M di acetato di ammonio con 43,3 mL di etanolo al 95%. Conservare a temperatura ambiente.
- Soluzione di colorazione
- aggiungere 1 µ l 10.000 x macchia verde fluorescente dell'acido nucleico (ad es., SYBR Green) in tampone di 30 mL di Tris-EDTA (10 mM Tris-HCl, 1 mM EDTA bisodico, pH 7,4) e conservare a 4 ° C. Protect da luce.
- 1% bassa fusione dell'agarosi
- sciogliere 1% bassa fusione punto agarosio (1 g in 100 mL dH 2 O) in un forno a microonde. Agitare l'agarosio ogni 15-20 s per assicurarsi che l'agarosio è completamente fusa. Posizionare l'agarosio in bagnomaria a 37 ° C per almeno 20 minuti prima dell'uso.
- Pre-riscaldare puntali per
- tagliato le estremità strette di puntali per pipette P200 da 3 mm e calda a 37 ° C prima di pipettaggio agarosio.
2. Preparare la cometa scivola
- Slide rivestimento
- Melt agarosio all'1% (1 g in 100 mL dH 2 O) in un forno a microonde per 2-3 minuti o fino a quando l'agarosio è completamente fusa. Immergere i vetrini in agarosio e strofinare un lato della diapositiva servendosi di un panno privo di lanugine.
- Disporre i vetrini su una superficie piana per asciugare all'aria o scaldare a 50 ° C per asciugatura più rapida; un film trasparente dell'agarosi dovrebbe essere formato dopo l'essiccazione. Porre i vetrini rivestiti a 37 ° C prima dell'uso.
- Preparazione delle sospensioni di cellule singole
- cultura e trattare le cellule di glioma
- cultura le cellule U251 MG in medium DMEM-Ham F-12 supplementato con 10% FBS, 100 U/mL di penicillina e 10 µ g/mL di streptomicina a 37 & n. 176; C con 5% CO 2.
- Digerire le cellule con tripsina 1 mL per 3 min e neutralizzare la tripsina usando mezzo DMEM-Ham F-12 con FBS. Raccogliere in provetta da 15 mL, girare a 300 x g per 4 min, aspirare il mezzo e sospendere le cellule a 2 x 10 5 cellule/mL in PBS 1X.
Nota: Il campione di cellule dovrebbe essere preparato immediatamente prima di iniziare l'analisi e tutti i campioni devono essere manipolati in un ambiente scuro o grigio per impedire il danno del DNA dalla luce. - Unire la sospensione cellulare con agarosio all'1% fuso basso punto di fusione (a 37 ° C) con un rapporto 01:10 (v/v), mescolare delicatamente pipettando su e giù e immediatamente una pipetta 30 µ l in una diapositiva. Usare il lato della la punta della pipetta di diffondere la miscela di agarosio/cella per garantire la formazione di un sottile strato.
- Mettere il vetrino piatta a 4 ° C al buio per 10 minuti, aumentando il tempo di gelificazione a 30 min migliora l'aderenza dei campioni in ambienti con elevata umidità.
- Immergere il vetrino in 4 ° C LS al buio per 1 h a tutta la notte.
- cultura e trattare le cellule di glioma
3. Singola cella elettroforesi
- Vai al alcalina (passaggio 3.2) o neutro (punto 3.3) cometa dosaggio
- per analisi alcalina della cometa
- delicatamente togliere i vetrini dalla LS, scarico tampone in eccesso e immergere delicatamente in AES per 1 h a 4 ° C per consentire la rimozione del DNA. Mantenere le diapositive al buio.
- Aggiungi AES pre-refrigerati nel vassoio scorrevole elettroforesi, non superare 0,5 cm di sopra le diapositive (questo dipende dalle dimensioni delle unità elettroforesi), posizionare le diapositive all'interno e coprire con un coperchio. Impostare la tensione di alimentazione a 1 V/cm (la lunghezza tra gli elettrodi) ed eseguire per 30 min a 4 ° C.
- Soluzione di elettroforesi in eccesso di scarico da diapositiva. Immergere delicatamente i vetrini due volte in dH 2 O per 5 minuti ciascuno, a temperatura ambiente.
- Delicatamente immergere i vetrini in etanolo al 70% per 5 min a temperatura ambiente. Procedere al passaggio 4.
- Per analisi neutrale della cometa
- rimuovere delicatamente i vetrini dalla LS, scarico tampone in eccesso e immergere delicatamente a NES per 30 min a 4 ° C. Keep la diapositiva al buio. Buffer di pre-refrigerati elettroforesi neutro
- Aggiungi nella barra delle applicazioni di elettroforesi diapositiva, non superare 0,5 cm sopra diapositive (questo dipende dalle dimensioni delle unità elettroforesi), posizionare le diapositive all'interno e coprire con un coperchio. Impostare la tensione di alimentazione a 1 V/cm (la lunghezza tra gli elettrodi) ed eseguire per 45 min a 4 ° C.
- Scarico tampone in eccesso dalle diapositive. Immergere delicatamente i vetrini in DPS per 30 min a temperatura ambiente.
- Delicatamente immergere i vetrini in etanolo al 70% per 30 min a temperatura ambiente. Procedere al passaggio 4.
4. Colorare vetrini cometa
- a secco diapositive a 37 ° C per 10-15 min al buio.
- Posto 50-100 µ l verde fluorescente dell'acido nucleico macchiatura soluzione su ogni secchi agarosio e macchia per 15 min a temperatura ambiente al buio.
- Sciacquare brevemente le diapositive in dH 2 O e asciugare completamente a 37 ° C al buio. Procedere all'acquisizione di immagini e analisi.
5. Acquisizione e analisi di immagine
Nota: la visualizzazione e la quantificazione delle rotture del DNA si basano su epifluorescenza e il software di dosaggio di cometa (Vedi Tabella materiali) 12 .
- Porre i vetrini al microscopio con un supporto per diapositive. Accertarsi che il gel dell'agarosi è rivolta alla lente dell'obiettivo. In modo casuale e catturare immagini dalle diapositive cometa macchiato con un microscopio a fluorescenza con una lente di obiettivo 10x. Evitare i bordi e le zone intorno a bolle d'aria.
- Assicurarsi ogni coda di cometa è distribuito in orizzontale. Teste di cometa devono provenire da sinistra e la coda da destra.
- Salvare ogni immagine in un formato binario TIF con brillante macchia di DNA e sfondo scuro. Caricare immagini per il software utilizzando il " selezionare i file da analizzare " pulsante, che si trova sulla sinistra della barra degli strumenti. Una finestra di visualizzazione immagine dovrebbe apparire ( Figura 1).
- Disegnare un quadro di misura sullo schermo e regolarne le dimensioni secondo la cometa della cella. Clicca la " regolare " pulsante per impostare la soglia della testa, cometa e coda secondo l'immagine, quindi fare clic sui " iniziare la misurazione " pulsante ( Figura 1).
- Selezionare una cella utilizzando il telaio e attivare la misura cliccando con il mouse sul " la cometa di analisi " pulsante; un'intensità immagine si presenta sul " profili " finestra con i parametri di misura selezionata. I risultati possono essere salvati cliccando il " memorizzare il risultato " pulsante ( Figura 1).
Nota: Il software calcola i parametri tra cui la lunghezza della coda di cometa, la percentuale di DNA dalla coda, il momento di coda (TM) e il momento di coda d'oliva (OTM). I momenti di coda sono calcolati mediante le formule come segue:

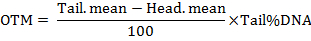
- analizzare almeno 50 cellule per ogni trattamento.
Risultati
Il presente protocollo viene descritto un flusso di lavoro dettagliato per l'esecuzione di analisi di cometa e analisi dei dati (Figura 1). Risultati dall'analisi della cometa alcalino e neutro ha mostrato che la coda della cometa di cellule di U251 trattati con doxorubicina (1 µM, 20 h) era più lungo e aveva una maggiore intensità di DNA, suggerendo un notevole accumulo di DNA frammentato a causa della chemioterapia (Figura 2...
Discussione
Il test della cometa è uno strumento efficace per misurare le rotture di singolo e doppio filamento del DNA a livello cellulare. Il dosaggio è stato ampiamente applicato come un "golden standard" negli studi per quanto riguarda la genotossicità e biomonitoraggio13, che vanno dalle lesioni della base, legami incrociati del DNA, lo sviluppo di farmaci e siti sensibili dell'alcali. Nello studio presente, abbiamo mostrato due distinti protocolli passo-passo per i dosaggi cometa alcalino e neutro, r...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questa ricerca è stata sostenuta dal programma di ricerca intramurale del NIH, NCI e CCR. Tutti gli autori ricevevano assegno di ricerca intramurale da NIH, NCI e CCR.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 10x PBS(Ca++, Mg++ free) | TEKnova | P0196 | |
| NaCl | Sigma | S5886 | |
| EDTA | TEKnova | E0308 | |
| Trizma base | Sigma | T1503 | |
| NaOH | Sigma | 72068 | |
| Sodium lauryl sarcosinate | Sigma | L7414 | |
| Triton X-100 | Sigma | 93443 | |
| Sodium acetate | Sigma | 32318 | |
| Glacial acetic acid | Sigma | 695092 | |
| Ammonium acetate | Sigma | A1542 | |
| SYBR Green | Invitrogen | S33102 | |
| Low melting point agarose | Invitrogen | 16520 | |
| Agarose | Invitrogen | 16500 | |
| 95% ethanol | WARNER-GRAHAM | #64-17-5 | |
| Trypsin | GIBICO | 25300-054 | |
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| Glass tissue slides | ELECTRON MICROSCOPY SCIENCES | 63422-11 | |
| Kimwipes | KIMberly-Clark | ||
| 1.5 mL Microcentrifuge Tubes | DENVILLE | ||
| Pipette Tips | SHARP | ||
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Microwave | Avanti | ||
| Waterbath | PRECISION | ||
| Horizontal electrophoresis chamber | TREVIGEN | Cometassay ES II | |
| Power supply | Bio-Rad | ||
| Incubator | Quincy Lab | Model 12-140E | |
| Fluorescent microscope | Zeiss | LSM700 | |
| Micropipettor | Eppendorf |
Riferimenti
- Ostling, O., Johanson, K. J. Microelectrophoretic study of radiation-induced DNA damages in individual mammalian cells. Biochem Biophys Res Commun. 123 (1), 291-298 (1984).
- Singh, N. P., McCoy, M. T., Tice, R. R., Schneider, E. L. A simple technique for quantitation of low levels of DNA damage in individual cells. Exp Cell Res. 175 (1), 184-191 (1988).
- Tice, R. R., et al. Single cell gel/comet assay: guidelines for in vitro and in vivo genetic toxicology testing. Environ Mol Mutagen. 35 (3), 206-221 (2000).
- Shah, A. J., Lakkad, B. C., Rao, M. V. Genotoxicity in lead treated human lymphocytes evaluated by micronucleus and comet assays. Indian J Exp Biol. 54 (8), 502-508 (2016).
- Azqueta, A., Collins, A. R. The essential comet assay: a comprehensive guide to measuring DNA damage and repair. Arch Toxicol. 87 (6), 949-968 (2013).
- Goldstein, M., Kastan, M. B. The DNA damage response: implications for tumor responses to radiation and chemotherapy. Annu Rev Med. 66, 129-143 (2015).
- Gavande, N. S., et al. DNA repair targeted therapy: The past or future of cancer treatment?. Pharmacol Ther. 160, 65-83 (2016).
- Torgovnick, A., Schumacher, B. DNA repair mechanisms in cancer development and therapy. Front Genet. 6, 157 (2015).
- Weston, V. J., et al. The PARP inhibitor olaparib induces significant killing of ATM-deficient lymphoid tumor cells in vitro and in vivo. Blood. 116 (22), 4578-4587 (2010).
- Brown, J. S., O'Carrigan, B., Jackson, S. P., Yap, T. A. Targeting DNA Repair in Cancer: Beyond PARP Inhibitors. Cancer Discov. 7 (1), 20-37 (2017).
- Lu, Y., et al. Chemosensitivity of IDH1-Mutated Gliomas Due to an Impairment in PARP1-Mediated DNA Repair. Cancer Res. 77 (7), 1709-1718 (2017).
- Konca, K., et al. A cross-platform public domain PC image-analysis program for the comet assay. Mutat Res. 534 (1-2), 15-20 (2003).
- Valverde, M., Rojas, E. Environmental and occupational biomonitoring using the Comet assay. Mutat Res. 681 (1), 93-109 (2009).
- Collins, A. R. The comet assay for DNA damage and repair: principles, applications, and limitations. Mol Biotechnol. 26 (3), 249-261 (2004).
- Karbaschi, M., Cooke, M. S. Novel method for the high-throughput processing of slides for the comet assay. Sci Rep. 4, 7200 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon