Method Article
Ottimizzazione di un'analisi di espressione di RNA-ha basato multisala usando materiale archivistico di cancro al seno
In questo articolo
Riepilogo
Degradazione dell'acido nucleico in tessuto archivistico, eterogeneità del tumore e una mancanza di campioni di tessuto fresco congelato può influire negativamente sulle servizi di diagnostica del cancro nei laboratori di patologia in tutto il mondo. Questo manoscritto descrive l'ottimizzazione di un pannello di biomarcatori usando un'analisi multiplex Beads magnetiche per classificare i tumori al seno.
Abstract
Degradazione dell'acido nucleico in tessuto archivistico, eterogeneità del tumore e una mancanza di campioni di tessuto fresco congelato può influire negativamente sulle servizi di diagnostica del cancro nei laboratori di patologia in tutto il mondo. L'amplificazione del gene e l'espressione diagnostica test utilizzando materiale d'archivio o materiale che necessita di trasporto al servizio dei laboratori, ha bisogno di un più robusto e preciso test adattato per flussi di lavoro clinici attuali. Il nostro team di ricerca ottimizzato l'uso di Invitrogen™ QuantiGene™ Plex analisi (Thermo Fisher Scientific) per quantificare il RNA a materiale d'archivio utilizzando la tecnologia DNA ramificato (bDNA) su Luminex xMAP® biglie magnetiche. L'analisi di espressione genica descritto in questo manoscritto è un metodo rapido e multisala di romanzo, che consentono di classificare con precisione il cancro al seno nei diversi sottotipi molecolari, omettendo la soggettività di interpretazione inerente a tecniche di imaging. Inoltre, a causa del basso input di materiale richiesto, tumori eterogenei possono essere laser microdissected con ematossilina ed eosina (H & E) sezioni macchiate. Questo metodo ha una vasta gamma di applicazioni possibili, compresa la classificazione del tumore con potenziale diagnostico e misurazione dei biomarcatori nelle biopsie liquide, che permettono una migliore gestione paziente e monitoraggio delle malattie. Inoltre, la misurazione quantitativa di biomarcatori in materiale archivistico è utile nella ricerca oncologica con accesso alle librerie di materiale clinicamente con annotazioni, in cui gli studi retrospettivi possono convalidare potenziali biomarcatori ed il loro risultato clinico correlazione.
Introduzione
Ottimizzazione dei dosaggi di RNA basato utilizzando materiale di (FFPE) di paraffina-incastonato formalina-fisso d'archivio è impegnativo a causa della variabilità nel trattamento chirurgico del tessuto e degradazione del RNA causato da formalina usato per tessuto integrità conservazione1, 2. Per superare le limitazioni di esecuzione di studi di espressione genica accurata da materiale d'archivio, il nostro gruppo usato l'analisi di biglie magnetiche multisala bDNA. Invece di amplificazione enzimatica di un modello di destinazione, la tecnologia di bDNA utilizza l'ibridazione di sonde specifiche e l'amplificazione di un reporter segnale3. Le sequenze di riconoscimento breve delle sonde di rilevamento e acquisizione sono progettate per ibridare ad brevi frammenti di RNA obiettivo4. Inoltre, l'uso degli omogeneati del tessuto come il materiale di partenza diretto in questa analisi, sormonta l'inevitabile perdita di RNA risultante dall'analisi che richiede purificazione e previa estrazione del RNA. Amplificazione del segnale, l'utilizzo di sequenze di riconoscimento breve e l'esclusione di una fase di purificazione, contribuiscono a ridurre in tecnica variazione del dosaggio. La tecnologia fornisce la possibilità di multiplex il dosaggio (fino a 80 obiettivi di RNA) e misurare l'espressione di un panel di bersagli da basso materiale in ingresso. Questo protocollo descrive la preparazione e colorazione dei campioni di tessuto per il laser microdissection. Di colorazione sui vetrini membrana facilitare l'imaging del tumore e dell'architettura istologica per fornire accurata selezione e profilatura di: (1) il tumore e dotti normali nel tessuto del seno e (2) la cellula maligna cloni all'interno dei tumori eterogenei.
Classificazione molecolare del tumore della mammella è un processo che interroga marcatori molecolari per classificare i pazienti tumori in tre classi molecolari, cioè, ricevitore di fattore di crescita epidermico luminal, umano 2 (HER2)-sottotipo arricchito e basale. Il sottotipo di HER2-arricchito è ben definito, con alta espressione del recettore HER2, per l'amplificazione del gene ERBB2, combinato con basso o assente del ricevitore dell'estrogeno (ER) e il ricevitore del progesterone (PgR). Il sottotipo di luminal è generalmente positivo per ER e il sottotipo basale sono in generale negativo per i tre ricevitori (HER2, ER, PgR) e significativamente sovrapposizioni con al seno triplo negativo (TNBC) cancro diagnostica sottotipo5,6. Altri marcatori vengono utilizzati per determinare le caratteristiche epiteliali e mesenchimali. Fibronectina (FN1) è un componente principale del vano mesenchimali del tessuto del seno. Espressione aumentata di FN1 è accompagnata da alta Ki67 colorazione e Mostra una firma per un più invasiva del tumore7,8 ed è associato con metastasi9. È interessante notare che, FN1 è stato trovato per essere presenti in microvescicole che proviene dalle cellule del tumore, che ha indotto l'attivazione di segnali mitogeni in fibroblasti destinatario10. Quindi, circolanti microvescicole come esosomi sono un potenziale marker di diagnosi precoce o metastasi e ricadono11.
Selezione dell'area di tumore per seno cancro trascrizionale sottotipizzazione ricorrentemente è stata eseguita da macrodissection12,13. Per superare l'eterogeneità del tessuto e aumentare la sensibilità, abbiamo combinato in modo affidabile tessuto classico colorazione con metodi di analisi molecolari multiplex. Come prova di principio, due cloni di cancro al seno distinti sono stati definiti da loro firma mesenchymal epiteliale ed il potenziale metastatico. Il flusso di lavoro del protocollo descritto può essere facilmente tradotto all'impostazione clinica corrente e utilizzato selettivamente isolare e caratterizzare i sottotipi di tessuto realizzati con profilatura di mRNA mirati.
Protocollo
Approvazione etica per l'uso di materiale di tessuto del seno in questo studio è stato ottenuto da università ricerca etica Comitato (UREC) dell'Università di Malta (Ref: 22/2012).
1. tessuto preparazione
- Utilizzando appropriati H & E procedure14, preparare riferimento macchiato le sezioni di tessuto e scansione digitale le diapositive per riferimento durante microdissection.
- Con un microtomo, tagliare 20 µm sezioni di tessuti su un bagno di acqua RNAsi-libera a 40 ° C. Raccogliere le sezioni sulle diapositive di membrana per il laser microdissection utilizzando materiali e attrezzature pulite, RNasi-decontaminati.
- Asciugare i vetrini di membrana a 37 ° C in un incubatore di essiccazione durante la notte. Macchia usando l'appropriata procedura H & E15 con soluzioni di grado di biologia molecolare e aumentando il tempo di colorazione di ematossilina a 6 min. Se necessario, macchia le diapositive di membrana con i protocolli appropriati immunohistochemical16.
2. laser Microdissection
- Impostare i limiti di diapositiva e calibrare il movimento scenico.
- Spostare la vista laser in un'area vuota di una diapositiva di membrana. Tarare il laser fuoco sparando un laser sparato ad aumentare gli intervalli di messa a fuoco (5%) fino a quando il laser inizia a perforare attraverso la membrana. Draw e microdissect qualsiasi forma e finetune il fuoco del laser per massimizzare l'efficienza del laser durante la dissezione laser.
- Aumentare la velocità di movimento di fase in un punto dove l'efficienza di dissezione laser non è compromessa. Tarare il laser alla lente dell'obiettivo selezionato e ri-calibrare se cambiando gli obiettivi (laser velocità: 1 – 15%), messa a fuoco al 70-75% e potenza al 90 – 100% quando si utilizza l'obiettivo 4x.
- Scansione le diapositive di membrana macchiato usando l'obiettivo 4x.
- Selezionare e circondare le zone di destinazione per microdissezione sulla diapositiva della membrana. Laser sezionare una superficie minima di 42 mm2. Record della zona sezionato per determinare il volume di tampone omogeneizzazione del campione da utilizzare.
- Utilizzare il meccanismo di sollevamento tappo per recuperare la sezione dissecata sui tappi di diffusore di provette attaccato all'annesso appropriato. Se la sezione dissecata non viene recuperata sul cappuccio, ripetere la dissezione laser della zona.
3. tessuto Lisi
Nota: Diverse soluzioni e materiali sono forniti insieme al kit menzionato nella Tabella materiali.
- Preparare la quantità necessaria di miscela d'omogeneizzazione per lisi del campione mediante omogeneizzazione soluzione e proteinasi K con un rapporto di 60: 1.
- 2.4 µ l di soluzione di omogeneizzazione in ciascuna provetta per ogni 1 mm2 di area di tessuto, vortexare per 10 s a velocità massima e centrifuga a temperatura ambiente per 5 s a 2.500 x g.
- Incubare in un blocco di riscaldamento a 65 ° C per 12 – 18 h con agitazione a 600 giri/min.
- Centrifugare i lisati a 21.000 x g per 5 min a temperatura ambiente.
- Utilizzando una pipetta, attentamente aspirare il supernatante chiaro e pipettare in una provetta etichettata fresca. Evitare di aspirare i frammenti di tessuto/membrana, in quanto ciò potrebbe ridurre la qualità dei risultati.
- Conservare il supernatante chiaro in un congelatore a-80 ° C.
4. test di ibridazione
- Eseguire le seguenti operazioni preparatorie.
- Pre-riscaldare la miscela di lisi inserendo il flacone di reagente in un incubatore a 37 ° C per 30 min e mescolare delicatamente per inversione.
- Se gli omogenati sono congelati, scongelare a temperatura ambiente e incubare a 37 ° C per 30 min. Vortex i campioni alla massima velocità dopo l'incubazione.
- Impostare l'incubatore agitazione a 54 ° C e posizionare la sonda del kit temperatura convalida in un pozzetto vuoto di una piastra a 96 pozzetti. Dopo 2 h controllare la temperatura sul termometro digitale e impostare la temperatura di delta di agitazione incubatore per correggere qualsiasi differenza di temperatura.
- Disgelo e vortice la sonda impostata e bloccando il reagente per 10 s, centrifuga a temperatura ambiente per 5 s a 2.500 x g e tenere sul ghiaccio.
- Tenere il flaconcino di proteinasi K sul ghiaccio durante l'uso.
- Sonicare le perline di cattura a 46 KHz per 3 min, poi vortice alla massima velocità per un altro 3 min.
- Preparare un 25 mix di perle di lavoro µ l per pozzetto di scalabilità di conseguenza i seguenti reagenti: 4,25 µ l di acqua priva di nucleasi, 16.65 µ l di miscela di Lisi, 1.00 µ l di blocco 0.10 µ l di proteinasi K, 0,50 µ l di cattura perline, reagente, 2.50 µ l di sonda insieme.
- Vortice il mix di perle di lavoro per 10 s a velocità massima, quindi Pipettare 25 µ l/pozzetto nella piastra di separazione magnetica.
- Dispensare 25 µ l del tessuto omogeneizzato per una piastra a 96 pozzetti separazione magnetica e 25 µ l di soluzione di 3 pozzi come gli spazii in bianco di omogeneizzazione.
- Sigillare la piastra di separazione magnetica utilizzando un sigillo di plastica trasparente pressione e posizionare la piastra nell'incubatore a 54 ± 1 ° C per 18 – 22 h mentre si stringono a 600 giri/min.
- Preparare una soluzione tampone di lavaggio di 1x per 24 pozzetti mescolando 31,5 mL di RNAsi-libera acqua con 0,1 mL di wash buffer componente 1 e 1,67 mL del componente di tampone di lavaggio 2.
- Rimuovere la piastra dall'incubatore. Impostare la temperatura dell'incubatore d'agitazione a 50 ± 1 ° C.
- Montare la piastra di separazione magnetica sulla rondella piastra magnetica portatile e bloccarlo in posizione. Rimuovere il sigillo di pressione e attendere 1 min per i branelli magnetici a stabilirsi.
- Fase di lavaggio: Capovolgere la piastra di separazione magnetica montata sulla piastra sopra il lavandino e la macchia delicatamente su una carta velina per rimuovere i residui della soluzione. Usando una pipetta multicanale aggiungere 100 µ l di soluzione tampone di lavaggio: 1x in ciascun pozzetto e aspettare 15 s. Ripetere questo passaggio per lavare la piastra 3 volte.
- Pipettare 50 µ l del reagente pre-amplificatore in ciascun pozzetto. Sigillare la piastra con un sigillo di piastra in alluminio e scuotere la piastra a 800 rpm per 1 min a temperatura ambiente. Incubare per 1 h a 50 ± 1 ° C mentre si stringono a 600 giri/min.
- Ripetere il passaggio 4.9 (fase di lavaggio).
- Pipettare 50 µ l del reagente amplificatore in ciascun pozzetto. Sigillare la piastra con un sigillo di piastra in alluminio e scuotere la piastra come descritto al punto 4.10.
- Ripetere il passaggio 4.9 (fase di lavaggio).
- Vortice della sonda di etichetta per 10 s a velocità massima. Aggiungere 50 µ l di sonda etichetta ad ogni pozzetto. Sigillare e scuotere la piastra (passo 4,10).
- Ripetere i passaggio 4.9 (fase di lavaggio).
- Vortice la ficoeritrina streptavidina (SAPE) per 10 s a velocità massima. Aggiungere 50 µ l di SAPE in ciascun pozzetto. Sigillare e scuotere la piastra (passo 4,10).
- Ripetere il passo 4.9 (fase di lavaggio) l'eccezione di usare il tampone di lavaggio SAPE invece la soluzione tampone di lavaggio.
- Aggiungere 130 µ l di tampone di lavaggio SAPE per ogni pozzo e guarnizione piastra con un sigillo di piastra in alluminio. Agitare la piastra a 800 giri/min a temperatura ambiente per 3 min.
- Start-up l'analizzatore di biglie magnetiche. Eseguire la routine di calibrazione e verifica seguendo le istruzioni del produttore.
- Istituire un protocollo sull'analizzatore Beads magnetiche utilizzando i seguenti parametri: dimensione del campione: 100 µ l; Cancello DD: 5.000-25.000; Timeout: 90 s; Regione di evento/tallone tallone: 100; e fare clic su Avanti. Selezionare i numeri rispettivi tallone nel pannello e definire le regioni di perlina con geni rispettivi target come indicato dal produttore.
- Aggiungere una nuova analisi selezionando l'opzione "creare Batch utilizzando un protocollo esistente" dalla scheda batch . Nel layout di piastra designare i pozzi come sconosciuto o vuoto, rispettivamente e fornire un'etichetta per ogni campione nel pannello campioni .
- Rimuovere la guarnizione del piatto di alluminio dalla piastra 96 pozzetti di reazione. L'espulsione del piatto facendo clic sul pulsante di espulsione , la piastra di carico nell'analizzatore biglie magnetiche. Fare clic su ritrarre| Esecuzione Batch per avviare l'analizzatore. Dopo la lettura, espellere e rimuovere la piastra a 96 pozzetti. Pulire e arrestare l'analizzatore di biglie magnetiche, come raccomandato dal produttore.
5. analisi dei dati
- Nel Batch| Scheda risultati , selezionare il file di analisi corrispondente e fare clic sul pulsante Esporta to.csv . File CSV aperto utilizzando un software di foglio di calcolo.
- Osservare i conteggi di perlina e omettere alcun risultato con < 40 perline/regione.
- La media di valori dei pozzetti bianchi e lavorare fuori la deviazione standard (SD) e il limite di rilevamento (LOD) (blank media + (3xSD)) al gene bersaglio. Impostare i valori inferiori al LOD come inosservato.
Nota: Se uno dei valori dell'espressione normalizzante inosservato, considerare i dati di esempio come inadeguata. Prendere in considerazione i valori dell'espressione oltre 20.000 come oltre il limite di sensibilità superiore. I lisati di questi campioni devono essere diluiti e ri-analizzati. - Sottrarre la media vuota dai dati grezzi a gene.
- Calcolare la media geometrica di geni selezionati normalizzante per campione. Normalizzare i dati campione individuale per la rispettiva media geometrica.
- Analizzare i dati su piattaforme di scienza di dati o utilizzando algoritmi definiti.
- Importare i dati nella piattaforma di scienza di dati facendo clic su Aggiungi dati. Trascinare il file di dati nel Processo di area di lavoro e connettersi ad un operatore [Seleziona attributi] , quindi a un principali analisi dei componenti (PCA) operatore e quindi alla porta t resul.
- Nell' Attributo selezionare operatore selezionare il sottoinsieme di dati normalizzati genica e una variabile nominale come sottotipo di cancro al seno. Eseguire il processo facendo clic su Play. Nella scheda tabelle selezionare la "dispersione 3D colore" nello stile grafico e la variabile nominale nel campo del colore . Valutare la variabilità di dati PCA su un terreno di 3-dimensionale.
Risultati
Il metodo descritto è stato applicato per la misura simultanea di 40 trascrizioni in H & E macchiato (Figura 1), microdissected (Figura 2) altamente degradato materiale FFPE. Utilizzando questo metodo, vi mostriamo la caratterizzazione accurata di stato del ricevitore (Figura 3A), classificazione dei tumori in sottotipi molecolari luminal e basale17e l'espressione differenziale del marcatore mesenchymal, FN1, durante il confronto di tumore e tessuto del controllo abbinato (Figura 3B), i vari sottotipi di recettori positivi e negativi.

Figura 1: espressione genica facendo uso di H & E macchiato materiale. Correlazione tra un profilo di espressione derivata da una sezione di tumore non marcate rispetto a una sezione di tumore macchiato. [Di correlazione di Pearson p-valore = 5.34E-26]. Riprodotto con permesso17. Clicca qui per visualizzare una versione più grande di questa figura.
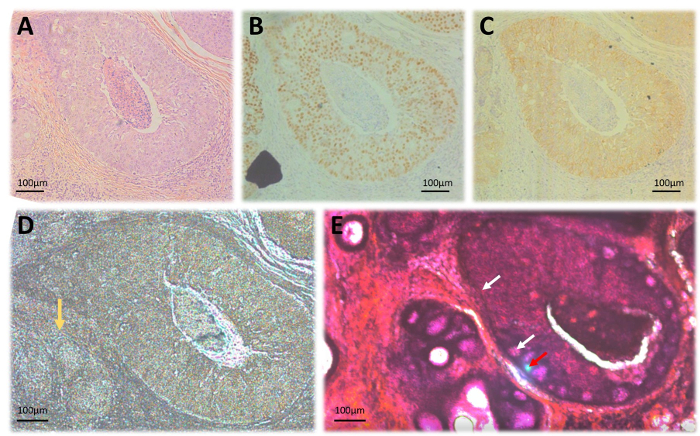
Figura 2: Laser microdissection dei tessuti FFPE. (A) H & E vetrino colorato; (B) macchiatura Immunohistochemical per l'espressione di ER; (C) la macchiatura immunohistochemical di HER2; (D) non macchiate sezione 20 µm su diapositive di membrana microdissezione laser. La freccia gialla indica un'area di tumore invasivo che non è chiaramente delimitata dovuta alla mancanza di colorazione. (E) A 20 µm sezione macchiato con H & E per migliore delineazione delle aree di interesse. Frecce bianche indicano il sentiero di dissezione laser mentre la freccia rossa indica il fuoco del laser durante la dissezione. Tutte le illustrazioni vennero catturate a 10 ingrandimenti. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: espressione di stato (A) del recettore e indicatore (B) mesenchymal FN1, nel tumore del seno rispetto alla corrispondenza normale. I sottotipi di cancro al seno diagnostica classica sono definiti secondo il risultato diagnostico usando immunohistochemistry e fluorescenza in situ di ibridazione (pesce) per la macchiatura di immunohistochemical equivoca di HER2. L'espressione normalizzato di HER2 (A) ed ER, FN1 (B) misurato mediante l'analisi di ibridazione è illustrato in HER2 positivo, ER positivo e casi TNBC rispetto al paziente abbinato tessuto normale del seno. La statistica Test di Kruskal Wallis (K-W χ2) dimostra che l'espressione di HER2 è significativamente (p < 0,05) più elevati nel tessuto del tumore rispetto al tessuto normale abbinato nella coorte HER2 positivo e significativamente più bassa nel gruppo TNBC. Espressione di ER non è stato trovato per essere significativamente più alto nel tessuto del tumore in contrasto con il normale abbinato in ER positivo e coorti TNBC. FN1 è significativamente più alto nel tessuto del tumore nei sottotipi positivi HER2 ed ER e Mostra una tendenza ad essere significativamente elevati nel tessuto del tumore anche nella coorte TNBC. Clicca qui per visualizzare una versione più grande di questa figura.
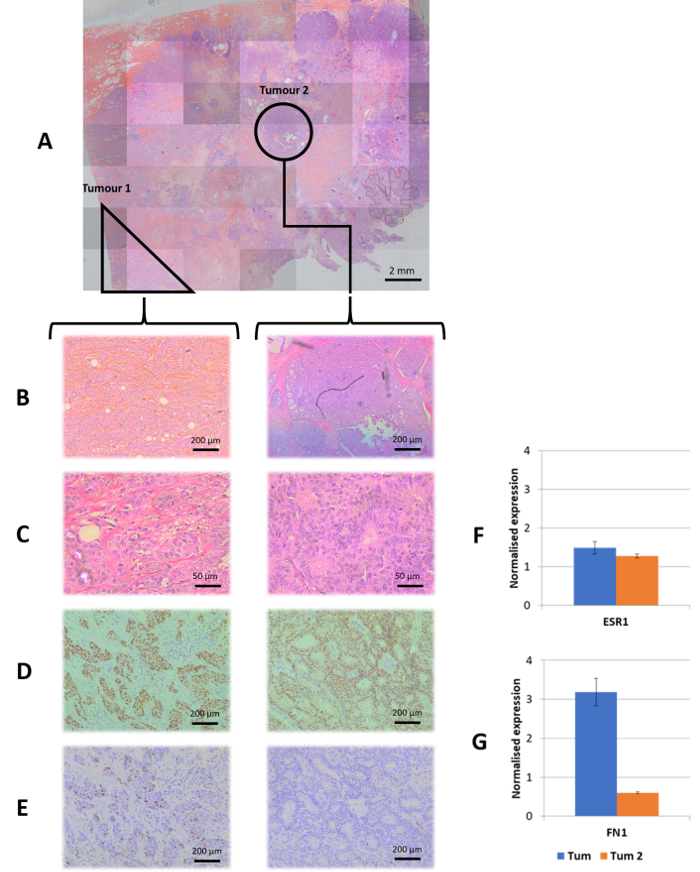
Figura 4: caso di studio: eterogeneità tumorale. Tumori morfologicamente distinti erano microdissected e trattati come campioni distinti. (A), il Maestro scansione della sezione H & E. (B, C) Un 10x e 40x ingrandimento, rispettivamente per ogni morfologia del tumore identificato. (D) la macchiatura Immunohistochemical per ER a 10 ingrandimenti. (E) la macchiatura Immunohistochemical per Ki67 10 ingrandimenti, mostrando un' più alta attività mitotica in tumore 1. (F) ha normalizzato i livelli di espressione del gene ESR1 in ogni tumore mostrano espressione relativamente alta e uguale fra i tumori come previsto dal risultato immunohistochemical. (G) ha normalizzato i livelli di espressione di FN1, un indicatore mesenchymal, dove l'espressione aumentata di FN1 è accompagnata da alta Ki67 macchiatura mostrando una firma per un tumore più invasiva. L'inverso è osservata nel tumore 2, che sembra essere un tumore più lenta proliferazione con un più basso potenziale maligno rappresentato da ridotta FN1 espressione. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Un'analisi basata su perlina multiplex bDNA è stata ottimizzata per quantificare l'espressione genica su RNA degradato derivato dal tessuto del cancro di seno FFPE e dotti seno normale. Ottimizzare il dosaggio, coinvolto lo sviluppo di un algoritmo per classificare i tumori del cancro al seno in sottotipi luminal e basale che utilizzano 8 ben noti biomarcatori e 5 potenziali normalizzante geni. Normalizzazione dei dati è stato fatto utilizzando permutazioni dei geni normalizzanti. La selezione dei geni normalizzante è basata sulla migliore previsione di stato del ricevitore utilizzando i geni di classificatore di Luminal/basali. Per classificare i sottotipi di Luminal/basali da tessuti FFPE, normalizzanti geni selezionati erano Beta-actina (ACTB), gliceraldeide 3-fosfato deidrogenasi (GAPDH) e ipoxantina fosforibosiltransferasi 1 (HPRT1).
Il metodo può essere adattato per l'uso in altri diagnostica e aree di ricerca seguendo un'adeguata selezione del set gene normalizzante. Un'importante applicazione di questo metodo nel settore della ricerca è la misura di biomarcatori in materiale archivistico che ben è annotato con gli esiti clinici. Questo potrebbe convalidare potenziali marcatori predittivi in studi retrospettivi, rapidamente e con precisione ed evitare gli studi prospettivi a lungo termine in attesa di sopravvivenza libera da malattia e dati di sopravvivenza complessiva. Attualmente il nostro gruppo sta studiando l'uso del test per rilevare gli esosomi ricevitore-positivi, che richiede lo sviluppo di un nuovo algoritmo utilizzando alternativo normalizzante geni per la normalizzazione dei dati. L'uso di liquide biopsie e analisi di espressione genica robusto permetterà analisi multiplex elevato throughput adattate per la gestione del paziente durante il trattamento e forniscono un mezzo per seguire l'efficacia del trattamento, potenziale ricaduta a causa di resistenza alla terapia e la capacità metastatica del tumore.
Questo metodo inoltre ha una vasta gamma di possibili applicazioni nella diagnosi dei tumori ed è adattato all'iter diagnostico corrente. I principali vantaggi di questo metodo nel campo diagnostico includono: (1) implementazione di saggi di throughput elevato, (2) escluse le soggettività e risultati ambigui provenienti da misure basate su immagine, (3) rilevazione accurata di bersagli multipli contemporaneamente, che aumentano la precisione e non minimizzare l'uso di preziosi campioni di pazienti e (4) nessun requisito per strutture altamente specializzate e risorse umane. Il processo di campionamento ottimizzata, insieme con il basso input di materiale richiesto per il dosaggio di multiplex basati su tallone, permette ulteriore indagine di eterogeneità del tumore; mediante microdissezione laser per separare accuratamente i fuochi multipli del tessuto maligno dalla sezione del paziente stesso, è possibile confrontare più espressione genica tra di loro nonché con tessuto normale corrispondente (Figura 4). Materiale a basso input è di vitale importanza per l'applicazione diagnostica sulle biopsie del tumore che forniscono il tessuto del tumore limitato. La capacità del test per misurare l'espressione genica ottenuti da campioni di RNA degradati permette facile trasporto dei campioni per l'analisi all'interno di un'istituzione o a laboratori di manutenzione. Inoltre, tutta la sezione analisi era anche possibile utilizzando H & E macchiato del materiale (Figura 1).
Per il successo di questo protocollo, è imperativo per: (1) assicurare il corretto campionamento del tumore sito/s che vengono lisate per il dosaggio e (2) sviluppare ben ottimizzato e convalidati gli algoritmi di normalizzazione dei dati, per ogni pannello di espressione genica e/o prognostici individuali o biomarcatori predittivi. Il primo dipende dall'esperienza tecnica dello scienziato/tecnico eseguendo il campionamento. Si consiglia di prendere un core aggiuntivo e preparare un tissue microarray (TMA) nello stesso formato del dosaggio multisala Beads magnetiche (formato da 96 pozzetti). Ciò fornirà un archivio di siti del tumore come una replica di campioni utilizzati per il dosaggio di RNA-ha basato. TMAs può essere valutato anche con altre tecniche per la ricerca di follow-up o la convalida dei risultati. Lo sviluppo di algoritmi di normalizzazione è dipenda del materiale indagato e normalizzanti geni selezionati per la normalizzazione. Diversi pannelli di normalizzazione dei geni sono selezionati basati sul livello e sulla variabilità di espressione nel campione analizzato e questo varia tra tessuti tumorali di diversa origine, esosomi da plasma o cellule tumorali circolanti. Validazione del test include trattamento del campione poiché varie preparazioni comporterà anche algoritmi di normalizzazione differente.
Per riassumere, l'uso della tecnologia bDNA in combinazione con la tecnologia di biglie magnetiche e la selezione del corretto pannello di geni bersaglio, fornirà il vantaggio aggiunto di misurare l'espressione genica direttamente nei lisati di tessuto derivato da piccole quantità di materiale paziente, compreso materiale, esosomi microdissected e cellule tumorali circolanti. Oltre al rilevamento di eterogeneità del tumore, l'uso corretto dei pannelli ha il potenziale per rilevare il tumore derivato esosomi per la diagnostica precoce e diagnosi precoce delle recidive. Poiché non vi è alcuna necessità di un passaggio di amplificazione dell'acido nucleico, l'amplificazione del segnale utilizzando la tecnologia bDNA, combinata con il multiplex basati su tallone, misure più espressione genica in materiale archivistico clinicamente con annotazioni e fornire una risorsa per convalida di biomarcatore.
Divulgazioni
The Invitrogen™ QuantiGene™ Plex analisi è di proprietà di Thermo Fisher Scientific.
Riconoscimenti
Il lavoro è stato supportato da (1) una borsa di progetto del cancro al seno (2014-2016) finanziato dall'azione per Breast Cancer Foundation e ALIVE 2013 attraverso la ricerca, l'innovazione & sviluppo Trust (RIDT) dell'Università di Malta, (2) la facoltà di medicina E chirurgia, Università di Malta e (3) progetto ACT finanziato dal Consiglio Malta for Science & Technology attraverso fusione: The R & I tecnologia sviluppo programma 2016. La pubblicazione di questo manoscritto è supportata attraverso la concessione di Jove-Luminex.
Materiali
| Name | Company | Catalog Number | Comments |
| Microtome | Leica | RM2235 | |
| Heamatoxylin Mayer's | Sigma | MHS16-500mL | |
| Eosin Y Aquaeous solution | Sigma | HT110216-500mL | |
| Normal Rabbit Serum | Monosan | MONX10963 | Working dilution: 1/40 |
| Biotinylated Rabbit anti-mouse | Dako | E0354 | Working dilution: 1/200 |
| ER antibody (6F11) | Vector Laboratories | VPE614 | Working dilution: 1/45 |
| HER2 antibody (CB11) | Novocastra | CB11-L-CE | Working dilution: 1/325 |
| Ki67 antibody (MIB-1) | Dako | M7240 | Working dilution: 1/500 |
| Avidin Biotin Complex kit | Vector Laboratories | PK-6100 | |
| Nikon Eclipse Ti-E Inverted microscope | Nikon | Ti-E | 4x, 10x, 20x and 40x objectives |
| Laser Microdissection membranes | Molecular Machines &Industries | S0103 | |
| mmi CellCamera 1.4 | Molecular Machines &Industries | MX4285c-ACK07 | |
| mmi Cellcut Plus | Molecular Machines &Industries | ||
| Diffuser caps | Molecular Machines &Industries | 50210 | |
| mmi Celltools Software v.4.01rcl | Molecular Machines &Industries | ||
| Eppendorf Thermomixer comfort | Eppendorf | 5355000038 | |
| 1.5mL heating block for Eppendorf Thermomixer | Eppendorf | 22670522 | |
| 96-well plate heating block for Eppendorf Thermomixer | Eppendorf | 22670565 | |
| Labnet Vortemp 56 Shaking incubator | Labnet | 52056A-220 | |
| LX200 100/200 | Luminex | Magnetic bead analyser | |
| Invitrogen QuantiGene Sample Processing Kit - FFPE Tissues | ThermoFisher Scientific | QS0109 | |
| Invitrogen QuantiGene Plex 2.0 Assay Kit (Magnetic Separation) | ThermoFisher Scientific | QP1011 | |
| Thermaseal RTS Sealing Film | Thermaseal | 765246 | |
| Hand-Held Magnetic Plate Washer | ThermoFisher Scientific | QP1011 | |
| Invitrogen QuantiGene Incubator Temperature Validation Kit | Affymetrix/Panomics | QS0517 | |
| Proteinase K (50µg/µL) | ThermoFisher Scientific | 14622 | |
| Invitrogen QuantiGene Plex 2.0 Sets | ThermoFisher Scientific | Various | |
| Multi Speed Vortex | Kisker Biotech | MSV-3500 | |
| Sonicator | Silvercrest | ||
| RNASEZAP | Sigma | R2020-250ML | |
| Aluminium 96-well plate seal | Sigma | Z721549-100EA | |
| Temperature Validation Kit | ThermoFisher Scientific | QS0517 | |
| RapidMiner Studio Community 7.1.001 | RapidMiner | Data Science Platform | |
| Hybridisation oven | Hybaid (Thermo Scientific) |
Riferimenti
- Abrahamsen, H. N., Steiniche, T., Nexo, E., Hamilton-Dutoit, S. J., Sorensen, B. S. Towards quantitative mRNA analysis in paraffin-embedded tissues using real-time reverse transcriptase-polymerase chain reaction: a methodological study on lymph nodes from melanoma patients. J Mol Diagn. 5 (1), 34-41 (2003).
- Macabeo-Ong, M., et al. Effect of duration of fixation on quantitative reverse transcription polymerase chain reaction analyses. Mod Pathol. 15 (9), 979-987 (2002).
- Yang, W., et al. Direct quantification of gene expression in homogenates of formalin-fixed, paraffin-embedded tissues. Biotechniques. 40 (4), 481-486 (2006).
- Flagella, M., et al. A multiplex branched DNA assay for parallel quantitative gene expression profiling. Anal Biochem. 352 (1), 50-60 (2006).
- Goldhirsch, A., et al. Strategies for subtypes--dealing with the diversity of breast cancer: highlights of the St. Gallen International Expert Consensus on the Primary Therapy of Early Breast Cancer 2011. Ann Oncol. 22 (8), 1736-1747 (2011).
- Schnitt, S. J. Classification and prognosis of invasive breast cancer: from morphology to molecular taxonomy. Mod Pathol. 23, S60-S64 (2010).
- Bae, Y. K., et al. Fibronectin expression in carcinoma cells correlates with tumor aggressiveness and poor clinical outcome in patients with invasive breast cancer. Hum Pathol. 44 (10), 2028-2037 (2013).
- Park, J., Schwarzbauer, J. E. Mammary epithelial cell interactions with fibronectin stimulate epithelial-mesenchymal transition. Oncogene. 33 (13), 1649-1657 (2014).
- Zhou, Z., et al. MRI detection of breast cancer micrometastases with a fibronectin-targeting contrast agent. Nat Commun. 6, 7984 (2015).
- Antonyak, M. A., et al. Cancer cell-derived microvesicles induce transformation by transferring tissue transglutaminase and fibronectin to recipient cells. Proc Natl Acad Sci U S A. 108 (12), 4852-4857 (2011).
- Moon, P. G., et al. Fibronectin on circulating extracellular vesicles as a liquid biopsy to detect breast cancer. Oncotarget. 7 (26), 40189-40199 (2016).
- Parker, J. S., et al. Supervised risk predictor of breast cancer based on intrinsic subtypes. J Clin Oncol. 27, (2009).
- Nielsen, T., et al. Analytical validation of the PAM50-based Prosigna Breast Cancer Prognostic Gene Signature Assay and nCounter Analysis System using formalin-fixed paraffin-embedded breast tumor specimens. BMC Cancer. 14, 177-177 (2014).
- Fischer, A. H., Jacobson, K. A., Rose, J., Zeller, R. Hematoxylin and Eosin Staining of Tissue and Cell Sections. Cold Spring Harbor Protocols. (5), (2008).
- Lin, F., Prichard, J. . Handbook of Practical Immunohistochemistry: Frequently asked questions. , (2011).
- Grech, G., et al. Molecular Classification of Breast Cancer Patients Using Formalin-fixed Paraffin-embedded Derived RNA Samples. Journal of Molecular Biomarkers & Diagnosis. 7 (S8), (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon