È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Liposoma singole misurazioni per lo studio degli enzimi di membrana Proton-pompaggio usando microscopia fluorescente ed elettrochimica
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per studiare il meccanismo molecolare della traslocazione di protoni attraverso le membrane del lipido di liposomi singoli, utilizzando citocromo bo3 come esempio. Combinando elettrochimica e microscopia a fluorescenza, le variazioni del pH in lumen del singole vescicole, contenenti singole o multiple enzima, possono essere rilevati e analizzati singolarmente.
Abstract
Protone-pompaggio enzimi dell'elettrone trasferimento reazioni di ossidoriduzione Coppia catene alla traslocazione di protoni attraverso la membrana, creando una Forza Protonomotrice utilizzata per la produzione di ATP. La natura anfifilica delle proteine di membrana richiede particolare attenzione al loro trattamento, e la ricostituzione nell'ambiente naturale del lipido è indispensabile quando si studia i processi di trasporto di membrana come protone traslocazione. Qui, abbiamo dettaglio un metodo che è stato utilizzato per l'indagine sul meccanismo di pompaggio di protoni di enzimi redox della membrana, prendendo citocromo bo3 da Escherichia coli come un esempio. Una combinazione di microscopia di fluorescenza ed elettrochimica viene utilizzata per controllare lo stato redox dei cambiamenti di pH piscina e monitor del chinone in lumen. A causa della risoluzione spaziale di microscopia fluorescente, centinaia di liposomi possa essere misurati simultaneamente mentre il contenuto degli enzimi può essere scalato verso il basso per un singolo enzima o trasportatore a liposoma. L'analisi di rispettivo singolo enzima può rivelare modelli nella dinamica funzionale degli enzimi che potrebbe altrimenti essere nascosti dal comportamento dell'intera popolazione. Includiamo una descrizione di uno script per l'analisi di immagine automatizzato.
Introduzione
Informazioni sui meccanismi degli enzimi e cinetica sono ottenuti solitamente al livello ensemble o macroscala con popolazione di enzima in migliaia a milioni di molecole, dove misure rappresentano una media statistica. È noto, tuttavia, che le macromolecole complesse quali enzimi possono dimostrare eterogeneità nel loro comportamento e osservati a livello di insieme di meccanismi molecolari non sono necessariamente validi per ogni molecola. Tali deviazioni sulla scala singola molecola sono stati ampiamente confermati da studi di singoli enzimi con una varietà di metodi emergenti durante l'ultimo ventennio1. In particolare, rilevazione della fluorescenza di attività enzimatica individuale è stato utilizzato per indagare eterogeneità di enzimi attività2,3 o scoprire l'effetto della cosiddetta memoria (periodi di attività di enzimi elevati è riuscito da periodi di bassa attività e viceversa)4,5.
Molti studi di singolo enzima richiedono che gli enzimi sono immobilizzati sulla superficie o nello spazio fisso in un altro modo per rimanere sufficientemente lunga nel campo visivo per osservazione continua. Incapsulamento di enzima nei liposomi è stato indicato per attivare immobilizzazione degli enzimi, evitando qualsiasi impatto negativo dovuto la superficie-enzima o proteina-proteina, interazioni6,7. Inoltre, liposomi offrono una possibilità unica per lo studio di proteine di membrana singola nel loro naturale dei lipidi bilayer ambiente8,9,10.
Una classe di proteine di membrana, trasportatori, esercita uno spostamento direzionale di sostanze attraverso la membrana cellulare, un comportamento che può essere studiato solo quando proteine sono ricostituite in lipidi bilayer (ad es., liposomi)11, 12,13. Ad esempio, traslocazione protonica, esibito da parecchi enzimi delle catene di trasporto dell'elettrone procariote ed eucariote, svolge un ruolo importante nella respirazione cellulare, creando una Forza Protonomotrice utilizzata per la sintesi di ATP. In questo caso, il protone attività di pompaggio è accoppiato per il trasferimento di elettroni, anche se il meccanismo dettagliato di questo processo spesso resta sfuggente.
Recentemente, abbiamo dimostrato la possibilità di rilevazione fluorescente coppia con elettrochimica per studiare protone pompaggio attività di singoli enzimi dell'ossidasi terminale ubiquinol di Escherichia coli (citocromo bo3) ricostituiti nei liposomi14. Questo è stato ottenuto mediante incapsulamento di un colorante fluorescente membrana impermeabile pH sensibili nel lumen di liposomi preparato da E. coli lipidi polari (Figura 1A). La quantità di proteina è stata ottimizzata in modo che maggior parte dei liposomi o contenuto nessuna o solo una molecola di enzima ricostituito (secondo la distribuzione di Poisson). I due substrati del citocromo bo3 sono stati forniti da aggiungere alla miscela di lipidi che formavano i liposomi e ossigeno (ambientale) in soluzione ubichinone. I liposomi sono quindi scarsamente adsorbiti su elettrodo d'oro ultra-liscio semi-trasparente, coperto con un monostrato auto-assemblato di 6-mercaptohexanol. Infine, l'elettrodo è montato sulla parte inferiore di una cella di Spettroelettrochimica semplice (Figura 1B). Elettrochimico controllo dello stato redox chinone piscina permette uno per attivare in modo flessibile o per fermare la reazione enzimatica in qualsiasi momento, mentre la tintura di pH sensibili viene utilizzata per monitorare le variazioni del pH all'interno del lume dei liposomi come conseguenza di spostamento di protoni dalla enzimi. Utilizzando che l'intensità di fluorescenza di un colorante fluorescente in secondo luogo, associati a lipidi, la dimensione e il volume dei liposomi individuali può essere determinato e così la quantificazione del protone di enzima attività di pompaggio. Utilizzando questa tecnica, abbiamo trovato in particolare che il citocromo bo3 molecole sono in grado di entrare in uno stato di perdita spontanea che si dissolve rapidamente la forza motrice protonica. L'obiettivo di questo articolo è quello di introdurre la tecnica delle misurazioni singole liposoma in dettaglio.
Protocollo
1. preparazione di una miscela di lipidi/UQ-10/FDLL
Nota: E. coli lipidi utilizzati per la preparazione dei liposomi dovrebbero essere aliquotati e accuratamente miscelati con ubiquinone-10 (enzima-substrato) e di lunghezza d'onda fluorescenti ha tingere-contrassegnati i lipidi (per determinare la dimensione liposomi) prima dei ricostituzione.
- Usando una siringa di vetro, trasferimento 200 µ l di brodo di cloroformio di lipidi polari estrarre da Escherichia coli (25 mg/mL) in fiale di vetro per rendere aliquote di 5 mg.
- Aggiungere 50 µ l di 1 mg/mL ubiquinone-10 (UQ-10; in cloroformio) per i lipidi per rendere il rapporto finale di UQ-10: 1: 100 di lipidi (1% p/p).
- Aggiungere 20 µ l di 1 mg/mL (0,4% w/w) di un lunghezza d'onda fluorescenti tingere-contrassegnati lipidici (FDLL) per il mix di lipidi/UQ-10.
- Omogeneizzare la soluzione di cloroformio nel Vortex breve ed evaporare la maggior parte del cloroformio sotto un delicato flusso di azoto o argon. Rimuovere le tracce di cloroformio interamente da ulteriore evaporazione sotto vuoto per almeno 1 h.
Nota: Le aliquote dei lipidi possono essere memorizzate sotto atmosfera inerte a-20 ° C per diversi mesi.
2. ricostituzione del citocromo bo3
Nota: Per la purificazione di citocromo bo3 da e. coli, seguire il protocollo da Rumbley et al. 15 per garantire elevata purezza dei campioni di enzima nativo-piegato, aggiungere cromatografia di esclusione dimensionale dopo la fase di purificazione di affinità descritta da Rumbley et al. 15
- Aggiungere 312.5 µ l di 40 mM MOPS-KOH/60 mM K2modo4, pH 7.4, ad un'aliquota di lipidi/UQ-10/FDLL asciutto mix (5 mg, passo 1.4) e miscelare con vortice fino a quando il film lipidico è completamente risospeso, seguiti da 2 min di trattamento in un bagno ad ultrasuoni.
- Aggiungere 125 µ l di 25 mM 8-hydroxypyrene-1,3,6-trisulphonic acido (HPTS), una tintura fluorescente pH sensibili che deve essere incapsulato in liposomi.
- 137,5 µ l di tensioattivo di 250 mM n-ottile β-D-glucopyranoside (OGP), mescolare con vortex e Sonicare in un bagno d'acqua ad ultrasuoni per 10 min garantire che tutti i lipidi sono solubilizzati in micelle di tensioattivo. Trasferire la dispersione in un tubo di plastica da 1,5 mL.
Nota: Sospensione torbida di lipidi dovrebbe diventare trasparente dopo la solubilizzazione con surfattante. - Aggiungere la quantità necessaria di citocromo bo3 (vedere la nota qui sotto) e aggiungere acqua ultrapura per fare un volume totale di 50 µ l ( citocromo bo3 soluzione più acqua). Incubare a 4 ° C per 10 minuti in un mixer di rullo.
Nota: Quantità tipica per le condizioni di singolo enzima è 0,1 – 0,2% (w/w-a-lipidico, cioè. 5 – 10 µ g di proteine), anche se può essere aumentato fino a 1 – 2% (50 – 100 µ g di proteine) se l'obiettivo è quello di osservare solo l'attività elettrochimica (Vedi sotto). Come controllo negativo, liposomi senza citocromo bo3 possono essere preparati. - Pesare 2 x 50 mg e 2 x 100 mg di microsfere di polistirolo in quadrato quattro 1,5 mL-tubo e chiudere con la pellicola di paraffina per evitare l'essiccazione.
Nota: Prima dell'uso, microsfere di polistirolo devono essere lavati con metanolo, acqua e conservati in acqua secondo il manuale del produttore.
Nota: Se viene utilizzata una miscela di acqua/della microperla, microsfere di polistirolo può convenientemente essere trasferiti al tappo con una micropipetta con una punta larga. Acqua quindi può essere rimosso dalle microsfere utilizzando una micropipetta con una punta sottile. - Aggiungere l'1st50 mg di microsfere di polistirolo nella miscela di ricostituzione (punto 2.4) mettere il tappo con microsfere di polistirolo sul tubo 1,5 mL-plastica con dispersione ed eseguendo un breve giro per un paio di secondi. Incubare a 4 ° C su un mixer a rulli per microsfere di polistirolo adsorbire il tensioattivo per 30 min.
- Ripetere le aggiunte di microsfere di polistirolo e le incubazioni come segue: aggiungere 50 mg di microsfere per 60 min di incubazione; aggiungere 100 mg di microsfere per 60 min di incubazione; e aggiungere 100 mg di microsfere per 120 min di incubazione.
Nota: La soluzione sopra le microsfere si stabilirono diventerà traslucida durante passaggio 2.6 come proteoliposomi sono formati.
- Ripetere le aggiunte di microsfere di polistirolo e le incubazioni come segue: aggiungere 50 mg di microsfere per 60 min di incubazione; aggiungere 100 mg di microsfere per 60 min di incubazione; e aggiungere 100 mg di microsfere per 120 min di incubazione.
- Separare la soluzione proteoliposome da microsfere di polistirolo con una micropipetta con una punta sottile. Diluire la dispersione in 90 mL di 20mm MOPS-KOH/30 mM K2così4, pH 7,4 (tampone MOPS) e trasferimento in un tubo di Ti45 ultracentrifuga.
- Ultracentrifuga la dispersione utilizzando tipo 45 Ti rotore a 125.000 x g (a rmax) per 1 h a pellet il proteoliposomi.
Nota: Piccole provette da centrifuga possono essere utilizzati anche se la diluizione nel volume di buffer di grandi dimensioni consente di ridurre la concentrazione dei non-incapsulato HPTS la sospensione finale. - Eliminare il supernatante, sciacquare i pellet con 20 mM MOPS-KOH/30 mM K2modo4, pH 7.4 del buffer (senza risospendere il pellet). Dopo aver scartato il buffer di risciacquo, risospendere il proteoliposomi in 500 µ l di tampone MOPS pipettando avanti e indietro con una micropipetta punta sottile. Poi il trasferimento a un tubo di plastica da 1,5 mL.
- Centrifugare la sospensione per 5 min a 12.000 x g per rimuovere i detriti. Trasferire il surnatante (ricostituita proteoliposomi) in un nuovo flacone.
- Memorizzare la dispersione proteoliposomi ricostituiti a 4 ° C durante la notte e utilizzare entro 2 giorni.
3. fabbricazione di elettrodi d'oro semi-trasparenti
Nota: La superficie liscia di oro si ottiene un metodo di "stripping" modello di un 30 nm-spessore strato d'oro del 99,99% da un wafer di silicio atomicamente liscia. Il piccolo spessore dello strato d'oro è importante poiché esso deve essere semi-trasparente per consentire l'osservazione della fluorescenza. Dettagli di oro evaporazione (deposizione fisica da vapore, PVD) si possono trovare altrove16 e unico modello-stripping è coperto qui. In alternativa, ultra-lisce gettoni d'oro possono essere acquistati altrove (Vedi Tabella materiali).
- Colla fino a 9 vetro vetrini coprioggetto (0,17 mm di spessore) sulla superficie dell'oro evaporata utilizzando resina epossidica bi-componente basso-fluorescenza. Curare la colla a 80 ° C per 4 h.
- Appena prima di modifica con un monostrato auto-assemblato (passaggio 4), staccare il vetro vetrini coprioggetto dai wafer di silicio con una lama. A causa della magrezza di vetrini coprioggetto, prendersi cura prima di scollegare il coprioggetto per non crepa o rompere i vetrini.
4. modifica della superficie dell'oro con Self-Assembled Monolayer (SAM)
- Preparare 5 mL di soluzione acquosa di 1 mM 6-mercaptohexanol (6MH).
- Immergere appena staccato rivestito in oro vetrini coprioggetto (punto 3.1) nella soluzione 6MH e lasciare a 20 – 25 ° C durante la notte (> 16 h) per formare il SAM.
Nota: Tiolo soluzioni hanno un odore sgradevole, quindi un vaso chiuso deve essere utilizzato quando incubando il vetrino coprioggetto ricoperto in oro. - Il giorno successivo, rimuovere il vetrino coprioggetto ricoperto in oro dalla soluzione 6MH, lavare rapidamente con acqua o metanolo e poi con isopropanolo. A secco sotto un flusso di gas dolce.
5. test elettrochimica dei proteoliposomi attività
Nota: L'attività elettrochimica dell'enzima prima viene verificata su un livello molto attentamente imballate liposomi (passo 5) e i riempimenti di vescicola inferiori utilizzati in vescicole singolo esperimento per misurare le variazioni di pH nel lume dei liposomi (passaggio 6).
- Montare il vetrino coprioggetto ricoperto in oro in una cella elettrochimica spectro (Vedi Figura 1). Fare contatto all'oro con una piattina all'esterno di un'area definita da una guarnizione in gomma.
- Aggiungere 2 mL di soluzione tampone dell'elettrolito e posizionare il riferimento ed elettrodi ausiliari nella cella.
Nota: Come ulteriormente discusso nella sezione discussione, elettrodi di riferimenti senza cloruro sono preferiti per evitare formazione di Au (I) Cl durante elettrochimica. Qui, un Hg/Hg2SO4 (sab. K2SO4) elettrodo di riferimento è utilizzato e i potenziali sono dato contro elettrodo Standard di idrogeno (SHE) utilizzando 0,658 V vs lei per Hg/Hg2SO4 (sab. K2così4). - Eseguire spettroscopia di impedenza elettrochimica (0.1 Hz-100 kHz) al potenziale (OCP) potenziale della cellula aperta a valutare la qualità di SAM. Convertire i valori di impedenza in ingresso e dividere per 2πω per tracciare una trama di Cole-Cole, dove ω è la frequenza (vedere i seguenti riferimenti16,17,18 per ulteriori informazioni sulla conversione e interpretariato impedenza spettri).
Nota: SAMs compatta e densa di 6MH dovrebbe dare una stretta a semi-cerchio trama Cole-Cole e dare valori di capacità nell'intervallo 2.5-3.0 µF/cm2 (Figura 2A). Se si ottengono deviazione significativa dalla forma semicircolare o valori di capacitanza di out-of-range, cambiare l'elettrodo. - Eseguire vuoto voltammograms ciclico (CVs) con scansione tariffe 100 e 10 mV/s nella regione potenziale -0,3 – 0,8 V. Un CV tipico è mostrato su (Figura 2B, linea tratteggiata).
Nota: Questo dovrebbe dimostrare quasi puro comportamento capacitivo e un'assenza di significative corrente faradica, anche in condizioni di ossigeno ambientale come usato qui. - Aggiungere proteoliposomi (concentrazione di lipidi finale di 0,5 mg/mL, 1-2% (w/w) rapporto di citocromo bo3 al lipido) a cella elettrochimica e mescolare leggermente con una pipetta. Attendere che l'adsorbimento dei proteoliposomi sulla superficie dell'elettrodo è finito (30-60 min a temperatura ambiente).
Nota: Voltammograms ciclico (CVs) a 10 mV/s può essere eseguito durante l'adsorbimento proteoliposomi di seguire il processo. L'adsorbimento è finito quando consecutivi CVs smettere di cambiare. Informazioni sulle tecniche di CV possono essere trovati nei libri di testo seguente. 19 , 20 - Lavare la cella modificando la soluzione tampone almeno 10 volte, ma evitare di lasciare completamente asciutta la superficie dell'elettrodo.
- Eseguire la spettroscopia di impedenza elettrochimica a OCP (Figura 2A, linea blu) per confermare che il SAM sull'elettrodo d'oro rimane invariato e CVs con scansione Tariffe 10 e 100 mV/s per osservare l'ossidazione catalitica ubiquinol (e riduzione dell'ossigeno) dal citocromo Bo 3 a potenziali di insorgenza della riduzione elettrochimica chinone su 0 V vs SHE) (Figura 2B).
6. rilevazione del protone enzimatica di pompaggio mediante microscopia a fluorescenza
- Modificare l'elettrodo d'oro come nel passaggio 5, ma usando 100 x meno proteoliposomi rispetto al passaggio 5.5 (cioè, 5 µ g/mL). Per gli studi di singolo enzima, ridurre il citocromo bo3 al rapporto del lipido a 0,1-0,2% (w/w).
Nota: Liposomi saranno scarsamente adsorbono sulla superficie dell'elettrodo, l'attivazione del singola vescicola monitoraggio mediante microscopia a fluorescenza. In queste condizioni di singolo-enzima, la quantità di enzima immobilizzato sulla superficie dell'elettrodo è insufficiente per l'osservazione di una corrente catalitica. - Posizionare la cella elettrochimica sull'obiettivo di olio (60 X) di un microscopio a fluorescenza invertito con una goccia di olio per immersione. Utilizzando gli appositi filtri per fluorescenza FDLL, concentrarsi sulla superficie dell'elettrodo. Liposomi singoli dovrebbero apparire come punti luminosi al limite di diffrazione dell'obiettivo del microscopio. Prendere un'immagine di fluorescenza FDLL.
- Passare a quella di set di filtri di fluorescenza HPTS il microscopio per verificare che HPTS fluorescenza è chiaramente visibili e distinguibili dallo sfondo (presso il tempo di esposizione selezionate, vedi punto 6.4). Se non è il caso, aumentare l'intensità della luce.
- Programmare il software microscopio per eseguire un'acquisizione di immagini temporizzata alternando due set di filtri HPTS (menu applicazioni | Define/Run ND acquisizione). Impostare il ritardo tra acquisizioni di immagine al minimo. In questo esperimento, utilizzare un'esposizione di s 1 e 0,3 s ritardo (dovuto il movimento di torretta).
Nota: Il rapporto tra intensità di fluorescenza a questi due canali si più tardi convertirà al pH all'interno di liposomi in ogni momento. La durata dell'acquisizione può variare secondo il tasso di cambiamento di pH previsto; 5 min viene utilizzato in questo articolo. - Regolare le impostazioni del potenziostato per modificare il potenziale durante l'acquisizione dell'immagine. Ad esempio, in questo esperimento, utilizzare la seguente sequenza: 0 – 60 s: nessun potenziale applicato (cioè OCP); 60-s: 180 -0,2 V (vs SHE); 180 – 300 s: 0,4 V (vs SHE). In questo caso, solo il potenziale applicato nella seconda fase (-0,2 V vs SHE) è sufficiente a ridurre in modo efficiente la piscina di chinone.
- Eseguire contemporaneamente l'acquisizione di immagini programmate (microscopio) e la sequenza potenziale (potenziostato) avviando manualmente entrambe le misurazioni allo stesso tempo.
Nota: L'esperimento può essere ripetuto sull'elettrodo stesso tempo diversi spostando il tavolino del microscopio in un'area diversa sulla superficie. Il ritardo tra le acquisizioni deve essere almeno 5-10 min per assicurare l'equilibrio di pH completo di liposomi sulla superficie. Durate diverse e modelli potenziali possono essere applicati a seconda delle necessità, anche se tempo di imaging è limitato dovuto photobleaching di HPTS durante l'acquisizione.
7. analisi di fluorescenza immagini
Nota: Un tipico esperimento produce una serie di immagini con un punto di tempo (ad es., 2.6 s) per ciascuno dei due canali, vale a dire, (durata * 2 / 2.6) immagini. Un esempio di tale immagine impostato registrato durante un esperimento di singolo enzima sono accessibili tramite la repository di ricerca dati Leeds21. Un trattamento di immagine è costituito da diversi passaggi utilizzando Fiji (ImageJ) e l'analisi matematica di alto livello programmazione lingua software (d'ora in poi denominati come software di scripting, Vedi Tabella materiali per dettagli).
- Utilizzare Fiji per separare i file lasso di tempo, allineare le immagini e salvare come canali separati e le tempistiche.
- File di lasso di tempo aperto utilizzando l'importatore Bioformats fornito all'interno di Fiji (comando macro: eseguire ("Bio-Formats importatore")).
- Utilizzare il plugin StackReg per allineare ogni fotogramma al primo per contabilizzare i movimenti possibili fase o deriva termica si è verificato durante l'acquisizione (comando macro: eseguire ("StackReg", "trasformazione = traduzione")).
- Salvare il file di immagine non compresso separati per ogni canale e ogni passo in formato TIFF in un'unica cartella.
- Estrarre i francobolli di ora esatta per ogni fotogramma dai metadati del file lasso di tempo e salvarli come file CSV nella stessa cartella delle immagini. In alternativa, estrarre data e ora utilizzando il software di acquisizione e salvare manualmente in un file CSV utilizzando un software di foglio di calcolo.
Nota: La cartella con le immagini e i timestamp è ora pronta per essere analizzati dal software di scripting. A pochi passi 7.1.1. – 7.1.4. può essere automatizzato utilizzando uno script di Fiji (utilizzare uno script scritto in Python per l'elaborazione batch dei file lasso di tempo è fornito, "Ctrl-Shift-N" (in Windows), quindi File | Aperto per caricare lo script).
- Utilizzo software per l'elaborazione automatizzata delle immagini di scripting. Caricare il codice fornito per il software e fare clic su Esegui alla fine. Quando richiesto, selezionare la cartella contenente le immagini dal punto 7.1.
Nota: Uno script per l'analisi è fornito come script live mlx-formato, con una serie di commenti per identificare singoli liposomi, adattarle alle funzione 2D-gaussiana, filtrarli e quantificare i valori di pH in ogni momento. I seguenti sotto-fasi vengono eseguite automaticamente dallo script.- Caricare tutti i time frame immagini per un singolo esperimento nella memoria.
- Media di tutte le immagini per un singolo canale (selezionare il canale con la massima intensità di fluorescenza) e utilizzare l'immagine di una media per identificare tutti i valori massimi che possono corrispondere ai liposomi singoli.
- Parametri per ogni liposoma in forma fit i massimi individuati sull'immagine media per il 2D-gaussiana funzionano e salvare il risultato (per esempio componibile liposoma, vedere Figura 4A ).
- Filtrare i valori massimi in base ai criteri di liposomi singolo previsto, quali dimensioni, circolarità e intensità. Rifiutare di liposomi che vengono montati male a causa di basso segnale/rumore, strettamente vicine ai liposomi o essere troppo vicini al bordo immagine.
- Caricare il timestamp del file esterno e montare separatamente ogni liposoma filtrato alla funzione 2D-gaussiana su ogni immagine di lasso di tempo.
Nota: L'uso della programmazione parallela e CPU multicore consente di migliorare notevolmente la velocità di calcolo in questa fase. - Filtrare i liposomi nuovamente secondo i criteri simili come descritto al punto 7.2.4 ma applicata per ogni periodo di tempo.
- Su ogni passo, è possibile definire il rapporto di intensità di fluorescenza di un liposoma come il rapporto tra volumi racchiusi componibile 2D-Gaussiana funzione a due canali. Calcolare i valori di pH dal rapporto di intensità determinata dai due canali HPTS e utilizzando una curva di calibrazione (passaggio 8). Tracciare curve di pH-tempo risultante per i liposomi e osservare il loro pH cambia quando viene applicato il potenziale.
8. esecuzione di una curva di calibrazione della fluorescenza HPTS
Nota: Per convertire HPTS fluorescenza rapporto ad un pH di intravesicular, una curva di calibrazione è necessario prima stabilire che prenderebbe in considerazione di condizioni particolari di esperimenti come trasmittanza oro, filtri di qualità, ecc. Questo passaggio deve essere eseguita solo una volta o due volte e i dati di calibrazione possono essere utilizzati fino a quando i parametri di configurazione e misura rimangono invariati nel passaggio 6.
- Preparare un elettrodo con liposomi scarsamente adsorbiti (senza citocromo bo3 come descritto nei passaggi 5 e 6.1).
- Aggiungere 2 µ l di soluzione Gramicidina 0,1 mg/mL in etanolo a 2 mL di tampone per creare la concentrazione 100 ng/mL.
- Catturare due immagini di fluorescenza per i due canali HPTS.
- Modificare il pH della cellula tramite l'aggiunta di piccole aliquote di 1 M HCl o 1 M H2così4. Misurare il pH del buffer con un misuratore di pH standard e catturare due immagini di fluorescenza per i due canali HPTS.
- Ripetere i passaggi da 8,4 per l'intervallo di pH da 6 a 9.
- Utilizzando l'algoritmo da 7.2, adattarsi, filtrare e calcolare il rapporto medio di fluorescenza HPTS di liposomi individuali a ogni pH. Prendere il rapporto HPTS medio sopra tutti i liposomi.
- Adattare la dipendenza di pH-rapporto risultante per la seguente equazione:
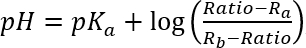 , dove pKun, Ra e Rbsono raccordo parametri.
, dove pKun, Ra e Rbsono raccordo parametri. - Utilizzare pKun, Ra e Rba convertire il rapporto HPTS di liposomi individuali nel passaggio 7.2.7. per valori di pH.
Risultati
La qualità del coprioggetto oro-modificato (l'elettrodo) con una SAM di 6MH è controllata prima di ogni esperimento con la spettroscopia di impedenza elettrochimica. La Figura 2A Mostra rappresentative trame di Cole-Cole misurati mediante spettroscopia di impedenza elettrochimica prima e dopo i liposomi sono adsorbiti. Se la qualità di SAM è sufficiente, spettroscopia di impedenza dovrebbe dimostrare un comportamento capacitivo quasi puro risultante in un...
Discussione
Il metodo descritto è adatto per studiare protone pompaggio di proteine di membrana respiratoria che possono essere ricostituite in liposomi e sono in grado di scambiare elettroni con lo stagno del chinone. Protone attività di pompaggio può essere monitorato a livello di singolo-enzima utilizzando coloranti sensibili al pH (raziometrici) incapsulati nel lume liposoma (Figura 1A).
Il metodo si basa sulla capacità di ubiquinone (o altri chinoni), incorporato nel...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori riconoscono la BBSRC (BB/P005454/1) per il sostegno finanziario. NH è stato finanziato dal programma VILLUM Foundation Young Investigator.
Materiali
| Name | Company | Catalog Number | Comments |
| 6-Mercapto-1-hexanol (6MH) | Sigma | 451088 | 97% |
| 8-hydroxypyrene-1,3,6-trisulphonic acid (HPTS) | BioChemika | 56360 | |
| Aluminium holder (Electrochemical cell) | Custom-made | 30x30x7 mm; inner diameter: 26 mm; hole diameter: 15 mm | |
| Auxiliary electrode | platinum wire | ||
| Chloroform | VWR Chemicals | 83627 | |
| E.coli polar lipids | Avanti | 100600C | 25 mg/mL in chloroform |
| Epoxy | EPO-TEK | 301-2FL | low fluorescence epoxy |
| Fiji (ImageJ 1.52d) | Required plugins: StackReg and TurboReg (http://bigwww.epfl.ch/thevenaz/stackreg/) | ||
| Filter cube ("ATTO633") | Chroma Technology Corporation | Ex: 620/60 nm; DM: 660 nm; Em: 700/75 nm | |
| Filter cube ("HPTS1") | Chroma Technology Corporation | Ex: 470/20 nm; DM: 500 nm; Em: 535/48 nm | |
| Filter cube ("HPTS2") | Chroma Technology Corporation | Ex: 410/300 nm; DM: 500 nm; Em: 535/48 nm | |
| Filter cube ("Texas Red") | Chroma Technology Corporation | Ex: 560/55 nm; DM: 595 nm; Em: 645/75 nm | |
| Fluorescent dye-labelled lipids (FDLL) | ThermoFisher Scientific | T1395MP | TexasRed-DHPC was used in this work (λexc 595 nm; λem 615 nm) |
| Fluorescent dye-labelled lipids (FDLL) (alternative) | ATTO-TEC | AD 633-161 | ATTO633-DOPE can be used as alternative (λexc 630 nm; λem 651 nm) |
| Gel filtration column | GE Healthcare | 28-9893-33 | HiLoad 16/600 Superdex 75 pg, for additional protein purification |
| Glass coverslips | VWR International | 631-0172 | No 1.5 |
| Glass syringe | Hamilton | 1725 RNR | 250 µL |
| Glass vials | Scientific Glass Laboratories Ltd | T101/V1 | 1.75 mL capacity |
| Gold | GoodFellow | 99.99% | |
| Gramacidin | Sigma | G5002 | |
| Mercury sulfate reference electrode | Radiometer (Hash) | E21M012 | |
| Microcentrifuge | Eppendorf | Minispin NL040 | |
| Microscope | Nikon | Eclipse Ti | |
| Microscope Camera | Andor | Zyla 5.5 sCMOS | |
| Microscope Lamp | Nikon | Intensilight C-HGFI | |
| NIS-Elements AR 5.0.2 | Nikon | Microscope acquisition software | |
| n-Octyl β-D-glucopyranoside | Melford Laboratories | B2007 | |
| Nova 1.10 | Metrohm | Potentiostat control software | |
| Objective | Nikon | Plan Apo λ 60x/1.4 oil | |
| OriginPro 2017 | OriginLab | Plotting software | |
| O-ring (Electrochemical cell) | Orinoko | Inner diameter: 16 mm; cross section: 1.5 mm | |
| Plastic tubes | Eppendorf | 3810X | 1.5 mL |
| Polystyrene microbeads | Bio-RAD | 152-3920 | Biobeads, 20-50 mesh |
| Potentiostat | Metrohm Autolab | PGSTAT 128N | |
| Potentiostat | CH Instruments | CHI604C | |
| Scripting software | Matlab | R2017a | Required toolboxes: 'Image Processing Toolbox', 'Parallel Computing Toolbox', 'Curve Fitting Toolbox', 'System Identification Toolbox', 'Optimization Toolbox' |
| Silicon wafers | IDB Technologies LTD | Si-C2 (N<100>P) | Ø 25 mm, 525 um thick |
| Teflon cell (Electrochemical cell) | Custom-made | Outer diameter: 26 mm; inner diameter: 13.5 mm | |
| Temple-Stripped Ultra-Flat Gold Surfaces | Platypus Technologies | AU.1000.SWTSG | Alternative ready-to-use ultra-flat gold surfaces (Thickness below 100 nm on demand) |
| Thin micropipette tips | Sarstedt | 70.1190.100 | or similar gelloader tips 200 µL |
| Ubiquinone-10 | Sigma | C-9538 | |
| Ultracentrifuge | Beckman-Coulter | L-80XP | with Ti 45 rotor |
| Ultrasonic bath | Fisher Scientific | FB15063 |
Riferimenti
- Claessen, V. I., et al. Single-Biomolecule Kinetics: The Art of Studying a Single Enzyme. Annual Review of Analytical Chemistry. 3 (1), 319-340 (2010).
- Rojek, M. J., Walt, D. R. Observing Single Enzyme Molecules Interconvert between Activity States upon Heating. PLoS ONE. 9 (1), e86224 (2014).
- Velonia, K., et al. Single-Enzyme Kinetics of CALB-Catalyzed Hydrolysis. Angewandte Chemie International Edition. 44 (4), 560-564 (2005).
- Lu, H. P., Xun, L., Xie, X. S. . Single-molecule enzymatic dynamics. 282 (5395), 1877-1882 (1998).
- Engelkamp, H., Hatzakis, N. S., Hofkens, J., De Schryver, F. C., Nolte, R. J. M., Rowan, A. E. Do enzymes sleep and work?. Chemical Communications. 9 (9), 935-940 (2006).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. The Journal of Physical Chemistry B. 105 (48), 12165-12170 (2001).
- Hsin, T. -. M., Yeung, E. S. Single-Molecule Reactions in Liposomes. Angewandte Chemie International Edition. 46 (42), 8032-8035 (2007).
- García-Sáez, A. J., Schwille, P. Single molecule techniques for the study of membrane proteins. Applied Microbiology and Biotechnology. 76 (2), 257-266 (2007).
- Onoue, Y., et al. A giant liposome for single-molecule observation of conformational changes in membrane proteins. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (6), 1332-1340 (2009).
- Jefferson, R. E., Min, D., Corin, K., Wang, J. Y., Bowie, J. U. Applications of Single-Molecule Methods to Membrane Protein Folding Studies. Journal of Molecular Biology. 430 (4), 424-437 (2018).
- Rigaud, J. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. BBA - Bioenergetics. 1231 (3), 223-246 (1995).
- Jesorka, A., Orwar, O. Liposomes: Technologies and Analytical Applications. Annual Review of Analytical Chemistry. 1 (1), 801-832 (2008).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: Not just a soap opera. Biochimica et Biophysica Acta - Biomembranes. 1666 (1-2), 105-117 (2004).
- Li, M., et al. Single Enzyme Experiments Reveal a Long-Lifetime Proton Leak State in a Heme-Copper Oxidase. Journal of the American Chemical Society. 137 (51), 16055-16063 (2015).
- Rumbley, J. N., Furlong Nickels, E., Gennis, R. B. One-step purification of histidine-tagged cytochrome bo3 from Escherichia coli and demonstration that associated quinone is not required for the structural integrity of the oxidase. Biochimica et Biophysica Acta (BBA) - Protein Structure and Molecular Enzymology. 1340 (1), 131-142 (1997).
- Jeuken, L. J. C., et al. Phase separation in mixed self-assembled monolayers and its effect on biomimetic membranes. Sensors and Actuators, B: Chemical. 124 (2), 501-509 (2007).
- Barsoukov, E., Macdonald, J. R. . Impedance Spectroscopy Theory, Experiment, and Applications. , (2005).
- Jeuken, L. J. C., Connell, S. D., Henderson, P. J. F., Gennis, R. B., Evans, S. D., Bushby, R. J. Redox Enzymes in Tethered Membranes. Journal of the American Chemical Society. 128 (5), 1711-1716 (2006).
- Compton, R. G., Banks, C. E. Chapter 4. Understanding voltammetry. , (2018).
- Girault, H. H. Chapter 10. Analytical and Physical Electrochemistry. , (2004).
- Mazurenko, I., Jeuken, L. J. C. . Timelapse (movie) of single vesicles fluorescence change upon potential application. , (2018).
- Li, M., Khan, S., Rong, H., Tuma, R., Hatzakis, N. S., Jeuken, L. J. C. Effects of membrane curvature and pH on proton pumping activity of single cytochrome bo3enzymes. Biochimica et Biophysica Acta - Bioenergetics. 1858 (9), 763-770 (2017).
- Li, M., Tuma, R., Jeuken, L. J. C. . MATLAB code for the analysis of proton transport activity in single liposomes. , (2017).
- Li, M., Tuma, R., Jeuken, L. J. C. . Time-resolved fluorescence microscopic data (traces) of individual lipid vesicles with proton transport activity. , (2017).
- Paula, S., Volkov, A. G., Van Hoek, A. N., Haines, T. H., Deamer, D. W. Permeation of protons, potassium ions, and small polar molecules through phospholipid bilayers as a function of membrane thickness. Biophysical Journal. 70 (1), 339-348 (1996).
- Brookes, P. S., Hulbert, A. J., Brand, M. D. The proton permeability of liposomes made from mitochondria) inner membrane phospholipids: No effect of fatty acid composition. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1330 (2), 157-164 (1997).
- Eriksson, E. K., Agmo Hernández, V., Edwards, K. Effect of ubiquinone-10 on the stability of biomimetic membranes of relevance for the inner mitochondrial membrane. Biochimica et Biophysica Acta - Biomembranes. 1860 (5), 1205-1215 (2018).
- Seneviratne, R., et al. A reconstitution method for integral membrane proteins in hybrid lipid-polymer vesicles for enhanced functional durability. Methods. , 1-8 (2018).
- Veshaguri, S., et al. Direct observation of proton pumping by a eukaryote P-type ATPase. Science. 351 (6280), 1-6 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon