Method Article
Uso di un microarray antigeno influenzale per misurare l'ampiezza degli anticorpi del siero attraverso i sottotipi di virus
In questo articolo
Riepilogo
Presentiamo un protocollo per l'utilizzo di un microarray proteico costruito stampando antigeni influenzali su vetrini rivestiti di nitrocellulosa per sondare simultaneamente il siero per più isotipi anticorpi contro oltre 250 antigeni provenienti da diversi ceppi di virus, quindi consentendo la misurazione dell'ampiezza degli anticorpi del siero attraverso i sottotipi di virus.
Abstract
Il virus dell'influenza rimane una causa significativa di mortalità in tutto il mondo a causa della limitata efficacia dei vaccini attualmente disponibili. Una sfida fondamentale per lo sviluppo di vaccini universali per l'influenza è l'elevata diversità antigenica derivante dalla deriva antigenica. Il superamento di questa sfida richiede nuovi strumenti di ricerca per misurare l'ampiezza degli anticorpi del siero diretti contro molti ceppi di virus in diversi sottotipi antigenici. Qui presentiamo un protocollo per analizzare l'ampiezza degli anticorpi del siero contro diversi ceppi di virus influenzali utilizzando una microarray proteica di antigeni influenzali.
Questo microarray antigene influenzale è costruito stampando antigeni di emagglutinina e neuraminidasi purificati su una membrana rivestita di nitrocellulosa utilizzando una stampante microarray. Le sera umane vengono incubate nel microarray per legare anticorpi contro gli antigeni influenzali. Gli anticorpi secondari coniugati quantici vengono utilizzati per rilevare simultaneamente gli anticorpi IgG e IgA che si legano a ciascun antigene sul microarray. Il legame quantitativo degli anticorpi viene misurato come intensità di fluorescenza utilizzando un imager portatile. I risultati rappresentativi sono mostrati per dimostrare la riproducibilità del saggio nella misurazione degli anticorpi influenzali specifici del sottotipo e intergovernativi nella sera umana.
Rispetto ai metodi tradizionali come ELISA, il microarray di antigeni influenzali fornisce un approccio multiplexed ad alta produttività in grado di testare centinaia di marini per isotipi anticorpi multipli contro centinaia di antigeni in un breve lasso di tempo, e quindi ha applicazioni nello sviluppo di sero-sorveglianza e vaccini. Una limitazione è l'incapacità di distinguere gli anticorpi leganti dagli anticorpi neutralizzanti.
Introduzione
Il virus dell'influenza è responsabile di una perdita di 20 milioni di anni di vita all'anno a cause di morte o disabilità, tra cui l'1% di tutti i decessi in tutto il mondo ogni anno, con impatti sproporzionati sugli anziani e sulle popolazioni dei tropici e del mondo in via di sviluppo1, 2,3. Oltre al carico di malattie delle epidemie stagionali, l'emergere di nuovi ceppi influenzali attraverso il riassortimento genetico sia naturalmente negli ospiti comuni che artificialmente per il bioterrorismo potrebbe portare a pandemie in tutto il mondo con rapida diffusione e elevata letalità 4 DEL psu' , 5. Mentre sono attualmente disponibili numerosi vaccini antinfluenzali,la loro efficacia è limitata dalla specificità del sottotipo 6, creando la necessità di sviluppare vaccini antinfluenzali universali che conferiscono un'immunità duratura contro più virus ceppi7.
Una sfida fondamentale per lo sviluppo di vaccini universali per l'influenza è l'elevata diversità antigenica tra i ceppi. La specificità antigenica degli attuali vaccini combinata con la variazione antigenica dei virus circolanti crea un disallineamento tra ceppi di vaccino e ceppi circolanti. Questo conferisce un vantaggio evolutivo che favorisce un'ulteriore deriva genetica dai ceppi di vaccino durante un'epidemia, limitando l'efficacia del vaccino spesso a meno del 50%8,9. Un'ulteriore fonte di mancata corrispondenza antigenica sono le mutazioni virali adattive alle uova generate durante la produzione di vaccini, che portano ad anticorpi che si legano male ai virus circolanti10,11.
Superare questa sfida di elevata diversità antigenica richiederà nuovi strumenti di ricerca per caratterizzare l'ampiezza delle risposte immunitarie preesistenti e suscitate attraverso varianti antigeniche clinicamente rilevanti in campioni di siero e mucosali. Attualmente i metodi disponibili, tra cui l'inibizione dell'emagglutinazione (HAI), la microneutralizzazione (MN) e la tradizionale ELISA, si limitano a rilevare anticorpi contro un singolo ceppo di virus alla volta, quindi il loro uso per il rilevamento di più isotipi di anticorpi contro più ceppi di virus esaurisce rapidamente le risorse cliniche e di laboratorio disponibili. Inoltre, HAI e MN richiedono una coltura di virus dal vivo disponibile solo in laboratori specializzati.
Microarray proteici, potenzialmente costituiti da migliaia di antigeni stampati su vetrini rivestiti di nitrocellulosa come mostrato nella Figura 1, possono soddisfare questo bisogno12. Questi microarray possono essere prodotti e sondati in modo ad alta produttività consumando piccole quantità di campioni clinici per determinare i livelli quantitativi di isotipo/sottotipo degli anticorpi rispetto a ogni singolo antigene sulla matrice. Questo approccio alla scoperta di antigeni è stato applicato allo sviluppo diagnostico e del vaccino contro più patogeni infettivi13. Ad oggi, abbiamo prodotto microarray proteici per oltre 35 patogeni, tra cui oltre 60.000 proteine espresse totali, e li abbiamo usati per sondare oltre 30.000 sera umani da individui infetti e di controllo. Una piattaforma di imaging portatile recentemente sviluppata per diapositive microarray ha reso questa metodologia più accessibile all'utente finale14.
Sulla base di un ampio lavoro precedente di più contributori nel campo15,16,17 ,18,19, è stato recentemente sviluppato un microarray di proteine influenzali che contiene oltre 250 varianti antigeniche emagglutinina purificata (HA) con rappresentazione di tutti i 18 sottotipi12,20. Utilizzando questa metodologia, è stata dimostrata un'infezione influenzale naturale per generare anticorpi IgG e IgA ampiamente reattivi contro sottotipi di HA correlati ai filogeneticamente, mentre una vaccinazione all'influenza intramuscolare ha generato solo IgG specifico per sottotipo anticorpi21. Tuttavia, è stato dimostrato che l'aggiunta di un adiuvante che attiva i recettori simili a i pedaggio ai vaccini antinfluenzali amplia la risposta degli anticorpi IgG suscitata attraverso i sottotipi di HA negli studi sugli animali22.
Questo microarray è attualmente utilizzato per sondare sera raccolti da un prospettiva di studio di coorte di studenti universitari che sono stati seguiti per l'infezione influenzale. Qui, viene riportata la metodologia del microarray di antigene influenzale con dimostrazione della riproducibilità del saggio per rilevare anticorpi specifici e cross-reattivi specifici del sottotipo in un sottoinsieme di campioni di questo studio.
Protocollo
Tutti i sera umani sono trattati e disposti secondo protocolli istituzionali approvati per la biosicurezza con l'uso di dispositivi personali protettivi. Tutto il personale di laboratorio che partecipa a questo protocollo ha ricevuto una formazione in materia di biosicurezza ed etica della ricerca.
1. Produrre microarray antigeni influenzali
-
Progettare e ottenere l'antigene set per microarray
- Ottenere antigeni proteici espressi e purificati come polvere liofilizzata.
-
Stampa di antigeni su diapositive microarray
- Ricostituire ogni antigene lofilfato a una concentrazione di 0,1 mg/mL nella salina con buffer fosfato (PBS) con 0,001% Tween-20 (T-PBS). Trasferire 10 l di ogni antigene ricostituito in singoli pozze di una piastra piatta di 384 pozzetto non trattata.
- Stampare antigeni utilizzando una stampante microarray (la stampante microarray utilizzata in questo studio non è più disponibile in commercio, vedere Discussione) con perni di avvistamento microarray a basso volume che aspirano l'antigene nel canale campione e il deposito tramite contatto e azione capillare su scivoli in vetro rivestito di nitrocellulosa da 16 pad.
- Programmare il software di stampa (ad esempio, Gridder) con la configurazione della lastra di origine e i parametri di stampa.
- Utilizzare il menu a discesa per selezionare il nome del tipo di lastra che verrà utilizzato con questo metodo di stampa. Per questo studio, utilizzare una piastra piatta 384-well non trattata.
- Selezionare la casella di testo accanto a Numero di lastre e digitare il numero di lastre di esempio che verranno utilizzate nel protocollo di stampa. Per questo studio, utilizzare 1 piastra.
- Selezionare una configurazione pin da utilizzare con questo metodo. Per questo studio, utilizzare una configurazione a 8 pin.
- Assicurarsi che gli scostamenti di origine siano le distanze (nelle direzioni X e Y) tra l'origine della diapositiva (calibrata nella sezione amministrativa) e la posizione in cui i pin di stampa inizieranno a stampare sulle diapositive.
- Utilizzando la scheda Struttura serie, definire le dimensioni e la forma delle serie (spaziatura dei punti e numero di punti per sottomatrice). Per questo studio, stampate 324 punti (180 m di diametro con spaziatura di 300 m) su diapositive da 16 pad in formato 18x18 utilizzando 8 pin.
- Selezionare i parametri per il modo in cui i perni di stampa raccolgono e distribuiscono i campioni. Per questo studio, ogni pin aspira 250 nL di soluzione antigene e stampa 1 nL su ciascuno dei 40 punti (totale di 20 diapositive per tutti gli 8 pin)
- Configurare il protocollo di pulizia dei pin e il protocollo di blotting. Per questo studio, ogni perno stampa l'antigene, viene immerso in sterile ddH2O nel contenitore di lavaggio sonicato, e poi aspira l'antigene successivo.
- Definire la sequenza di stampa dei blocchi campione sulle diapositive. Il software di array costruirà un file di indice di griglia con annotato (.gal) per descrivere la disposizione degli antigeni all'interno di ogni microarray.
NOTA: La riga superiore viene utilizzata per i fiduciali (con fluorescenza a tutte le lunghezze d'onda utilizzate nell'imaging, ad esempio, mix di Qdot 585 nm streptavidin e Qdot 800 nm streptavidin conjugate in questo studio) per orientare le griglie durante l'imaging. Per le lunghe tirature di stampa, gli antigeni nella lastra di origine possono essere periodicamente ri-sospesi con il pipetting su e giù e quindi centrifugati, o è possibile preparare e utilizzare una nuova piastra di origine.
- Programmare il software di stampa (ad esempio, Gridder) con la configurazione della lastra di origine e i parametri di stampa.
- Posizionare i vetrini microarray non sondati in una scatola anti-luce e conservarli in un armadietto a temperatura ambiente per uno stoccaggio a lungo termine.
NOTA: a questo punto il protocollo può essere sospeso a tempo indeterminato.
-
Eseguire il controllo qualità (richiede tag poli-istidina)
- Collegare la diapositiva alle camere di sondaggio e reidratarsi con il buffer di blocco come descritto nel passaggio 2.1.1-2.1.2 e illustrato nella Figura 2.
- Diluire il mouse monoclonale poli-Il suo anticorpo 1:100 in filtrato 1x buffer di blocco.
NOTA: Se gli antigeni proteici non purificati (ad es. espressi in sistema di trascrizione e traduzione in vitro) vengono utilizzati direttamente per stampare microarray, aggiungere componenti del sistema di espressione delle proteine (ad esempio, E. coli lysato) in un rapporto di 1:10 buffer di blocco utilizzato per la diluizione del siero al fine di bloccare eventuali anticorpi diretti contro questi componenti. - Aggiungete 100 l di poli-il suo anticorpo diluito ad ogni camera scorrevole contenente cuscinetto d'array dopo l'aspirazione e incubate per 2 h a temperatura ambiente o pernottando a 4 gradi centigradi su shaker. Lavare 3x con buffer T-TBS come descritto nel passaggio 2.2.1. Diluire l'anticorpo secondario IgG di capra biotina-coniugata anti-topo 1:200 nel buffer di blocco, aggiungere 100 L per bene dopo l'aspirazione e incubare per 1 h a temperatura ambiente su shaker. Lavare 3x con buffer T-TBS.
- Diluire il Qdot 585 nm streptavidin a 4 nM nel buffer di blocco, aggiungere 100 l per bene dopo l'aspirazione e incubare per 1 h a temperatura ambiente sullo shaker. Lavare 3x con buffer T-TBS e quindi una volta con il buffer TBS (senza Tween).
- Dissimulare e quantificare le diapositive come descritto nel passaggio 2.2.5 – 3.1.2.
NOTA: gli antigeni proteici devono contenere i suoi10 tag per utilizzare questo protocollo di controllo qualità. In alternativa, se è incluso un tag diverso, il controllo qualità può essere eseguito con anticorpo o ligando per tale tag.
2. Sonda sera per anticorpi influenzali che utilizzano microarray
-
Incubare sera su microarray per il legame degli anticorpi
- Collegare diapositive microarray alle camere utilizzando clip e posizionare nei fotogrammi come illustrato nella Figura 3.
NOTA: Evitare sempre di toccare il pad microarray con mani e strumenti. Assicurarsi che le diapositive siano orientate con il lato del pad verso l'alto e la piccola tacca nell'angolo in alto a destra. - Reidratare i vetrini microarray con 100 gradi l per pozzo di buffer di blocco 1x filtrato e diluire il siero 1:100 in 100 l di buffer di blocco (può utilizzare piastre non trattate o tubi da 2 mL). Incubare entrambi i vetrini microarray reidratati in telai coperti e diluito sera separatamente per 30 minuti a temperatura ambiente su shaker orbitale a 100-250 rpm. Eseguire tutti i successivi passaggi di incubazione in modo simile sullo shaker.
NOTA: Sera deve essere equipaggiata e congelata a -80 gradi centigradi per lo stoccaggio a lungo termine per ridurre al minimo i cicli di congelamento-scongelamento e deve essere vortice per mescolare e centrifugare per rimuovere il particolato prima dell'uso. Osservare attentamente le camere di scorrimento durante e dopo questo passaggio per rilevare eventuali perdite che richiedono il rimontaggio delle camere di scorrimento. - Utilizzando punte pipette collegate a una linea a vuoto con fiaschetta di raccolta secondaria, aspirare con attenzione buffer di blocco da angolo di ogni camera senza toccare i pad. Eseguire tutti i passaggi di aspirazione successivi in modo simile. Aggiungere sera diluita a pastiglie rapidamente dopo aspirazione al fine di non lasciare asciugare le pastiglie.
- Mettere i telai coperti in vassoi all'interno di un contenitore secondario circondato da asciugamani di carta umidi e sigillati per evitare l'evaporazione. Incubare pernottamento a 4 gradi centigradi su scossoni a dondolo (in alternativa, può incubare per 2 h a temperatura ambiente su shaker orbitale a 100-250 rpm).
- Collegare diapositive microarray alle camere utilizzando clip e posizionare nei fotogrammi come illustrato nella Figura 3.
-
Anticorpi di siero legati all'etichetta con anticorpi secondari quantistici-punto-punto
- Aspirati sera da camere accuratamente come descritto sopra, aggiungere 100 L per pozzetto di tampone T-TBS (20 mM Tris-HCl, 150 mM NaCl, 0.05% Tween-20 in ddH2O regolato a pH 7.5 e filtrato, può essere ottenuto a gitare commercialmente), e incubare per 5 min orbitale a orbitr a 100-250 giri/min Ripetere questo passaggio di lavaggio per un totale di 3x (tutti i passaggi di lavaggio successivi vengono eseguiti in modo simile).
- Preparare la miscela di anticorpi secondari diluiti a 1 M nel buffer di blocco e mescolare accuratamente con la pipettatura prima e durante l'uso per mantenere l'omogeneità.
NOTA: per mantenere la riproducibilità degli analisi, lo stesso lotto di ogni anticorpo secondario deve essere utilizzato per tutti gli esperimenti di sondaggio per i quali è previsto il confronto quantitativo dei dati tra gli esperimenti. La concentrazione specifica dell'anticorpo secondario può essere necessario variare a seconda dell'affinità; seguire i protocolli del produttore quando disponibili. - Tamponi aspirati dalle camere dopo il lavaggio finale, aggiungere 100 L per pozzo di miscela di anticorpi secondario e incubare per 2 h a temperatura ambiente sullo shaker.
- Lavaggio della miscela di anticorpi secondari aspirati 3x con tampone T-TBS, quindi lavare una volta con buffer TBS (senza Tween).
- Dissemble microarray diapositive da camere con attenzione per evitare di toccare le pastiglie, risciacquare delicatamente con ddH filtrato2O, e asciugare mettendo in tubi da 50 mL e centrifugando a 500 x g per 10 min.
- Mettere i vetrini sondati in una scatola a prova di luce e conservarli in un armadietto a temperatura ambiente per lo stoccaggio a lungo termine.
NOTA: a questo punto il protocollo può essere sospeso per un massimo di 1 settimana.
3. Quantificare il legame degli anticorpi agli antigeni all'interno del microarray
-
Visualizzare i vetrini dei microarray e quantificare l'intensità della fluorescenza spot per misurare l'associazione degli anticorpi
- Acquisire immagini di diapositive microarray utilizzando l'imager portatile con il software integrato.
- Nella scheda Configura Imager, selezionare la configurazione diapositiva corretta. Per questo studio, utilizzare diapositive a 16 pad.
- Nella scheda Controllo immagine, selezionare il canale fluorescente appropriato e regolare il guadagno, il tempo di esposizione e il tempo di acquisizione in base alla reattività della sera per ottenere immagini ottimali. Per questo studio, i canali fluorescenti per IgA e IgG erano rispettivamente di 585 nm e 800 nm, e le impostazioni di imaging erano guadagni di 50, tempo di esposizione di 500 ms e tempo di acquisizione di 1 s.
- Fare clic su Acquisisci per avviare il processo di acquisizione dell'immagine.
NOTA: I micro-diapositive microarray possono essere ri-immagine a più impostazioni senza degradazione del segnale, purché siano memorizzati scuri e asciutti. Altri sistemi di imaging possono essere utilizzati se compatibili con i vetrini.
- Rileva i punti dell'array utilizzando griglie orientate in base ai marcatori fiduciari e misura l'intensità dei punti come mediana dell'intensità dei pixel meno lo sfondo misurato intorno alle macchie. Eseguire questo algoritmo di quantificazione in batch utilizzando il software imager incorporato, che utilizza il file .gal costruito nel passaggio 1.2.2 per collegare le intensità dei punti ai singoli antigeni di ogni microarray.
- Nel pannello Info file, caricate il file .gal selezionandolo dalla relativa cartella sul computer e specificate la cartella in cui salvare i file di output dell'analisi nella sezione Opzioni analisi.
- Nella scheda Controllo immagine, aprire una delle immagini acquisite da quantificare e selezionare il pulsante Auto nell'angolo in alto a destra.
- Nella sezione Analisi serie nell'angolo inferiore destro, creare un modello fiduciario come indicato dal software.
- Fare clic su Analisi batch, selezionare la cartella contenente le immagini da quantificare e selezionare il modello fiduciario creato nel passaggio precedente. Il software analizza ogni immagine e quantifica l'intensità del punto.
NOTA: Questo passaggio genererà un file .csv contenente intensità spot quantificando anticorpi all'interno di ogni campione di siero che si legano a ogni singolo antigene sul microarray che può essere successivamente manipolato nel software di manipolazione o analisi del foglio di calcolo.
- Analizzare i dati grezzi per confrontare il legame degli anticorpi tra antigeni e campioni di siero. Per questo studio, gli anticorpi IgA e IgG misurati come intensità di spot fluorescenti da 585 nm e 800 nm sono stati confrontati tra tutti gli antigeni tra 2 esecuzioni indipendenti dell'esperimento utilizzando diapositive diverse in giorni diversi, e l'analisi della correlazione è stata eseguita per misurare la riproducibilità del saggio.
NOTA: per le proteine non purificate stampate come miscela di espressione, l'analisi dei dati deve iniziare con la sottrazione di sfondo di un controllo del DNA assenza.
- Acquisire immagini di diapositive microarray utilizzando l'imager portatile con il software integrato.
Risultati
Come dimostrazione del protocollo, sera di base sono stati studiati da 16 individui all'interno di uno studio di coorte prospettico di studenti universitari seguiti per l'infezione influenzale sul microarray antigene influenzale. Per dimostrare la riproducibilità del saggio, questi campioni sono stati sondati due volte, su diversi vetrini e giorni diversi.
Per questo studio, gli antigeni influenzali purificati contenenti i suoi10 tag sono stati ottenuti da fornitori commerciali (vedi Tabella dei materiali) e collaboratori. Questi antigeni includono 251 antigeni HA totali, con 63 domini di testa globulare (HA1) e 186 proteine a lunghezza intera (HA0), tra cui 96 proteine HA0 monomerico e 90 proteine HA0 trimerized contenenti trimerizzazione fusa ("foldon") dominio23. Un elenco completo degli antigeni e dei controlli utilizzati in questo studio è incluso come file supplementare. Per questo studio, gli anticorpi secondari utilizzati sono stati gli anticorpi anti-umani di capra utilizzati per l'emissione di punti quantici a 800 nm (GAH-IgG-Q800) e l'IgA anti-umano di capra coniugato all'emissione di punti quantici a 585 nm (GAH-IgA-Q585) per il rilevamento multiplo di anticorpi IgG e IgA come illustrato nella Figura 3. Il legame degli anticorpi IgG e IgA a diversi sottotipi e forme molecolari di antigeni influenzali HA sono stati confrontati per sera ottenuti dalla coorte clinica descritta sopra.
La mappa termica risultante è illustrata nella Figura 4 con rappresentazione grafica nella figura 5. In queste cifre, solo i sottotipi clinicamente rilevanti con alta rappresentazione sulla matrice sono etichettati per risparmiare spazio, con il "" che indica tutti i sottotipi rimanenti di numero superiore e sottotipi minori o meno ben rappresentati inclusi come ordinati (ad esempio, H2 in tra H1 E H3). Un elenco completo dei ceppi e dei sottotipi nell'array è incluso come File supplementare. Questi dati dimostrano che gli anticorpi verso il gruppo capo dell'HA sono specifici di sottotipi con un'elevata quantità prevista di anticorpi per ceppi clinicamente prevalenti (H1N1, H3N2 e B) e bassa quantità di anticorpi per altri ceppi. Tuttavia, gli anticorpi per l'intera HA, che include il dominio del gambo, sono più reattivi incrociati tra i sottotipi, e questo effetto sembra essere aumentato quando l'intera HA viene trimerizzata. Questo risultato non è inaspettato, in quanto l'intera HA include la regione staminale, che è altamente conservata tra sottotipi. Pertanto, gli anticorpi contro le molecole HA intere provenienti da sottotipi non clinici (ad esempio, H5 e H7) rappresentano probabilmente anticorpi anti-stem originariamente suscitati contro sottotipi clinici (ad esempio, H1 e H3) che reagiscono tra le regioni staminali degli altri sottotipi HA sull'array. Questo punto illustra l'importanza di includere sia la testa HA che l'HA a intere molecole sull'array per distinguere tra anticorpi alla testa e regioni staminali che mostrano diversi profili di reattività.
Il saggio dimostra una buona riproducibilità tra le corse di sondaggio. La seconda tiratura mostra anticorpi IgG leggermente inferiori in tutti i ceppi, anche se il modello tra i ceppi è coerente. Questa lieve diminuzione trasversale è probabilmente dovuta alla variabilità batch-to-batch nell'anticorpo secondario, che è stato modificato tra le esecuzioni per IgG ma non per IgA. Pertanto, come indicato nel protocollo, si raccomanda di utilizzare lo stesso lotto di ogni anticorpo secondario per qualsiasi esperimento tra cui è previsto il confronto quantitativo. Se sono necessari diversi lotti di anticorpi a causa di un numero elevato di campioni da testare, è consigliabile includere campioni condivisi tra esecuzioni sperimentali con diversi lotti di anticorpi per consentire il confronto quantitativo con la correzione.
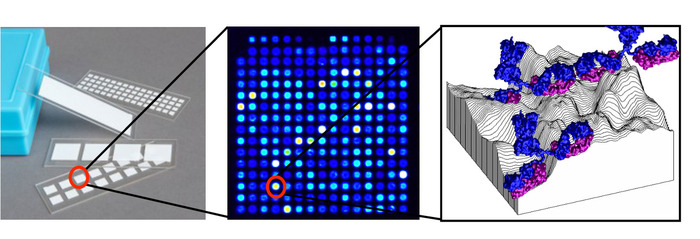
Figura 1: Schematica del microarray proteico. Ogni diapositiva contiene più pastiglie ciascuna con un'unica matrice, costituita da centinaia di antigeni stampati su macchie disposte in una griglia, con ogni punto contenente un adsorbito antigene sulla topografia tridimensionale della superficie di nitrocellulosa a cui anticorpi dal siero sono legati. Fare clic qui per visualizzare una versione più grande di questa figura.
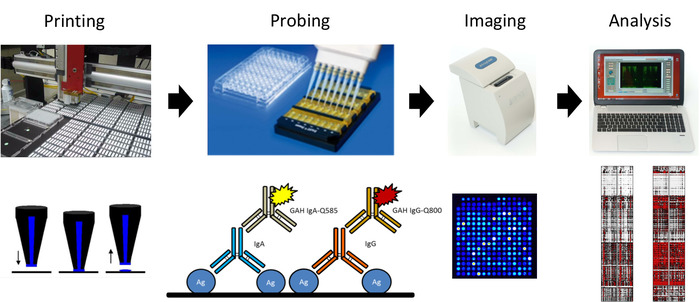
Figura 2: Schema ticodella stampa di microarray antigene influenzale e protocollo di sondaggio. Da sinistra a destra, il microarray viene stampato utilizzando diapositive rivestite di nitrocellulosa, che vengono utilizzati per sondare gli anticorpi IgG e IgA utilizzando anticorpi secondari quantistici-punto-punto, con diapositive imageizzate utilizzando un imager portatile, e risultati analizzati per generare una mappa termica. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Procedura per l'attaccamento della camera di sonda al vetrino microarray. Da A a F, la camera di sonda viene posizionata sopra la diapositiva con l'orientamento corretto, attaccata alla diapositiva utilizzando clip orizzontali sui lati e posizionata nel vassoio di sonda. Fare clic qui per visualizzare una versione più grande di questa figura.
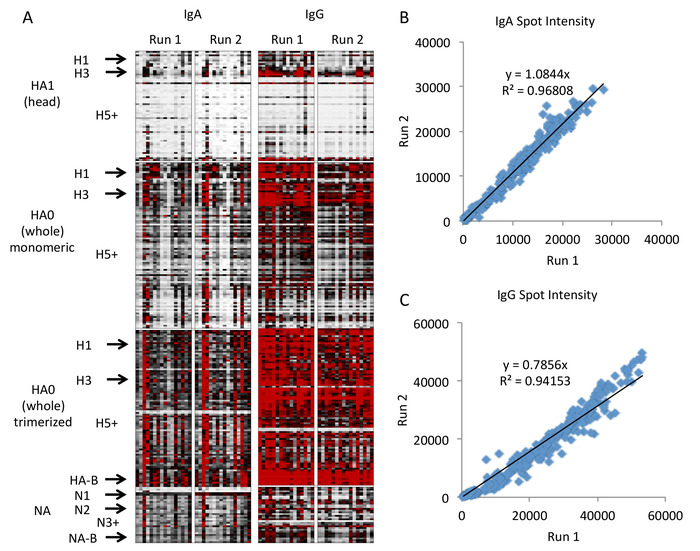
Figura 4: Risultati rappresentativi del microarray di antigene influenzale. Le mappe termiche rappresentano le risposte dell'anticorpo specifiche dell'antigene, con ogni riga che rappresenta un singolo antigene organizzato per molecola, sottotipo e ceppo, e ogni colonna che rappresenta una serie di sondaggi di un singolo campione, disposta per isotipo e corsa dell'anticorpo (A, bianco : 0, nero e 20000, rosso - 40000 intensità di fluorescenza). I sottotipi di antigene, compresi tutti i sottotipi di emagglutina da 1 a 18 e tutti i sottotipi di neuraminideda da 1 a 10, sono disposti verticalmente ed etichettati a sinistra. Un confronto dell'intensità della fluorescenza tra due piste dimostra una buona riproducibilità per regressione lineare per IgA (B) e IgG(C). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Pane di anticorpi del siero misurato su microarray antigene influenzale. Il siero IgA (A) e IgG (B) sono raggruppati da forme molecolari e sottotipi HA e NA per dimostrare un'elevata specificità degli anticorpi del gruppo di teste HA per i sottotipi clinici e un'elevata reattività incrociata di interi anticorpi HA e l'inclusione della regione del gambo. Fare clic qui per visualizzare una versione più grande di questa figura.
File supplementare: Elenco degli antigeni sul microarray antigene influenzale. Il contenuto è mostrato per tutti i 324 punti dell'array, inclusi spazi vuoti, fiduciali, controlli (IgG umano e IgA e Anti-umano IgG e IgA a 0,1 mg/mL e 0,3 mg/mL), e antigeni con informazioni su fonte, forma molecolare, sottotipo e ceppo. Abbreviazioni sono le seguenti: per la fonte, Sino - Sino Biological Inc., FKL - Florian Krammer Laboratory; per la forma molecolare, HA1 - testa HA, HA0 - HA intero, HA2 - gambo HA, NP - nucleoproteina. Per gli antigeni provenienti da Sino Biological Inc., vengono visualizzati i numeri di catalogo; per gli antigeni provenienti dal Krammer Laboratory, sono elencati gli ID antigene. Fare clic qui per scaricare questo file.
Discussione
Il protocollo di microarray dell'antigene influenzale qui descritto è adattabile a qualsiasi progetto che richieda l'analisi delle risposte degli anticorpi a molti antigeni. La piattaforma di microarray può essere utilizzata con qualsiasi set desiderato di antigeni proteici espressi in qualsiasi sistema in grado di raggiungere 0,1 mg/mL o una resa superiore con o senza purificazione, come descritto in precedenza12. Se gli antigeni proteici non purificati (ad esempio, espressi nel sistema di trascrizione e traduzione in vitro) vengono utilizzati direttamente per stampare microarray, i componenti del sistema di espressione proteica (ad esempio, E. coli lysato) devono essere aggiunti 1:10 al buffer di blocco utilizzato per diluizione di sera e anticorpi di controllo della qualità al fine di bloccare eventuali anticorpi diretti contro questi componenti. Le configurazioni delle diapositive sono disponibili con un numero inferiore di pastiglie più grandi su ogni diapositiva per contenere un numero maggiore di antigeni per array. Per questo studio, abbiamo utilizzato una stampante microarray GeneMachines OmniGrid 100 che utilizza perni che contattano direttamente la superficie della nitrocellulosa per depositare punti sull'array. Anche se questa stampante microarray non è più disponibile in commercio, altre stampanti microarray disponibili in commercio possono essere utilizzate in questo protocollo, ma possono richiedere pin personalizzati (sia a contatto che senza contatto) e software e devono avere una risoluzione spot sufficiente e compatibilità con diapositive e imager. A seconda della reattività di sera, è possibile utilizzare diluizioni da 1:50 a 1:400. Il buffer senza siero può essere utilizzato come controllo negativo, mentre gli anticorpi monoclonali noti per legarsi all'antigene possono essere usati come controllo positivo.
Gli utenti devono essere a conoscenza di alcuni problemi di risoluzione dei problemi. Lo scopo del controllo di qualità utilizzando anticorpi al suo tag è quello di verificare la presenza di eventuali antigeni per i quali la stampa non ha avuto successo. Tutti i punti che danno costantemente un segnale basso nel controllo di qualità di più array probabilmente rappresentano un antigene insufficiente stampato. I motivi possibili includono l'aggregazione o la precipitazione o l'antigene nella lastra di origine o uno scarso contatto tra il perno di stampa e lo scivolo microarray a causa dello spessore variabile del pad di nitrocellulosa. A questo punto, non eseguiamo la normalizzazione di analisi basata sui risultati quantitativi del controllo di qualità, dato che il legame rilevato degli anticorpi può essere influenzato dalla disponibilità di questo tag, che dipende dalla conformazione tridimensionale specifica per ogni antigene.
Il microarray antigene influenzale offre diversi vantaggi rispetto ai metodi tradizionali come ELISA ed è complementare a saggi funzionali come HAI e MN. Il microarray proteico a 16 pad è una tecnologia di risparmio di campioni con capacità di misurare simultaneamente anticorpi di isotipi multipli contro circa 300 antigeni provenienti da 1 l di siero. Il numero di antigeni può essere aumentato a migliaia diminuendo il numero di pastiglie per diapositive. L'analisi multiplex attina consente inoltre di risparmiare tempo e risorse di consumo, dato che centinaia di sera possono essere sondati per anticorpi in 2 giorni, e tutti i materiali diversi da vetrini e reagenti sono riutilizzabili in modo da non generare grandi quantità di rifiuti di plastica.
Mentre le stampanti microarray potrebbero non essere ampiamente distribuite, i vetrini microarray possono essere stampati in una posizione centralizzata e quindi trasportati all'utente finale per il sondaggio. L'unica attrezzatura necessaria per il sondaggio è l'imager portatile e a basso costo. L'obiettivo della diffusione di questo protocollo è quello di rendere più diffuso l'utilizzo di questa tecnica.
I principali limiti del microarray antigene influenzale sono l'incapacità di caratterizzare la funzione e la cinetica degli anticorpi rilevati. Il microarray sta rilevando un insieme policlonale di anticorpi leganti per ogni antigene. Questi anticorpi possono o non possono essere funzionali nella neutralizzazione del virus nei saggi HAI e MN. Tuttavia, i saggi HAI e MN richiedono una coltura di virus vivo con la necessità di strutture specializzate con armadi di biosicurezza di alto livello per testare gli anticorpi contro i sottotipi di influenza aviaria, mentre il microarray proteico non coinvolge il virus vivo componenti in modo che possano essere utilizzati in qualsiasi laboratorio di base. Per quanto riguarda la cinetica legante, una singola diluizione di siero sondato con un array contenente una singola concentrazione di ogni antigene produce un singolo punto dati, che rappresenta un composto di quantità e affinità riassunte su tutti gli anticorpi che si legano all'antigene . Per risolvere completamente la cinetica legante antigene-anticorpo, sono necessarie concentrazioni multiple di antigeni e/o diluizioni seriali di sera.
Nonostante queste limitazioni, il microarray dell'antigene influenzale è uno strumento utile per caratterizzare l'ampiezza degli anticorpi influenzali in tutto il paesaggio antigenico che possono integrare i saggi funzionali che sono più limitati in termini di produttività e disponibilità.
Divulgazioni
Gli autori non hanno divulgazioni.
Riconoscimenti
Gli autori desiderano riconoscere il professor Don Milton (Institute of Applied Public Health, University of Maryland, College Park, MD, USA) per aver raccolto la sera umana nell'ambito del protocollo IRB dell'Università del Maryland #313842 finanziata dal DARPA N66001-18-2-4015 P00001. Gli autori vorrebbero anche riconoscere il prof. S. Khan è parzialmente supportato dal National Center for Research Resources e dal National Center for Advancing Translational Sciences, National Institutes of Health, attraverso la sovvenzione KL2 TR001416. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali della NIH.
Materiali
| Name | Company | Catalog Number | Comments |
| 16-pad nitrocellulose-coated glass slides | Grace Bio Labs | 305016 | |
| 1x GVS FAST blocking buffer | Fischer Scientific | 10485356 | |
| ArrayCam portable imager | Grace Bio Labs | 400S | Other imaging devices can be used to visualize slides if capable of achieving the resolution of the microarray spots and the excitation and emission wavelengths of the quantum dots. |
| Biotin-conjugated goat anti-mouse-IgG antibody | Thermo Fischer | 31800 | |
| HiBase 384-well plate | Greiner Bio-One | T-3037-11 | |
| Microarray pins | ArrayIt | GMP2 | Each different microarray printer may require its own custom microarray pins. |
| Mouse monoclonal poly-His antibody | Sigma-Aldrich | H1029 | |
| OmniGrid 100 microarray printer | GeneMachines | The version of the microarray printer used in this work is no longer commercially available, but the updated similar equipment is the OmniGrid Accent microarray printer from Digilab (Hopkinton, MA), and the same protocol can be carried out with most commercially available microarray printers. | |
| ProPlate slide chambers | Grace Bio Labs | 246890 | |
| ProPlate slide clips | Grace Bio Labs | 204838 | |
| ProPlate slide frames | Grace Bio Labs | 246879 | |
| Quantum dot 585 nm conjugated goat anti-human-IgA antibody | Grace Bio Labs | 110620 | |
| Quantum dot 585 nm streptavidin conjugate | Thermo Fischer | Q10111MP | |
| Quantum dot 800 nm conjugated goat anti-human-IgG antibody | Grace Bio Labs | 110610 |
Riferimenti
- Murray, C. J., et al. Disability-adjusted life years (DALYs) for 291 diseases and injuries in 21 regions, 1990-2010: a systematic analysis for the Global Burden of Disease Study. Lancet. 380 (9859), 2197-2223 (2010).
- Renegar, K. B., Crouse, D., Floyd, R. A., Krueger, J. Progression of influenza viral infection through the murine respiratory tract: the protective role of sleep deprivation. Sleep. 23 (7), 859-863 (2000).
- Li, L., et al. Heterogeneity in Estimates of the Impact of Influenza on Population Mortality: A Systematic Review. American Journal of Epidemiology. 187 (2), 378-388 (2018).
- Fauci, A. S. Seasonal and pandemic influenza preparedness: science and countermeasures. Journal of Infectious Diseases. 194 Suppl 2, S73-S76 (2006).
- Duggal, A., Pinto, R., Rubenfeld, G., Fowler, R. A. Global Variability in Reported Mortality for Critical Illness during the 2009-10 Influenza A(H1N1) Pandemic: A Systematic Review and Meta-Regression to Guide Reporting of Outcomes during Disease Outbreaks. PLoS One. 11 (5), 2009-2010 (2016).
- Demicheli, V., Jefferson, T., Ferroni, E., Rivetti, A., Di Pietrantonj, C. Vaccines for preventing influenza in healthy adults. Cochrane Database Systematic Reviews. 2, CD001269 (2018).
- Erbelding, E. J., et al. A Universal Influenza Vaccine: The Strategic Plan for the National Institute of Allergy and Infectious Diseases. Journal of Infectious Diseases. , (2018).
- Xie, H., et al. H3N2 Mismatch of 2014-15 Northern Hemisphere Influenza Vaccines and Head-to-head Comparison between Human and Ferret Antisera derived Antigenic Maps. Scientific Reports. 5, 15279 (2015).
- Tricco, A. C., et al. Comparing influenza vaccine efficacy against mismatched and matched strains: a systematic review and meta-analysis. BMC Medicine. 11, 153 (2013).
- Katz, J. M., Webster, R. G. Efficacy of inactivated influenza A virus (H3N2) vaccines grown in mammalian cells or embryonated eggs. Journal of Infectious Disease. 160 (2), 191-198 (1989).
- Zost, S. J., et al. Contemporary H3N2 influenza viruses have a glycosylation site that alters binding of antibodies elicited by egg-adapted vaccine strains. Proceedings of the National Academy of Science. 114 (47), 12578-12583 (2017).
- Davies, D. H., et al. Profiling the humoral immune response to infection by using proteome microarrays: high-throughput vaccine and diagnostic antigen discovery. Proceedings of the National Academy of Science U. S. A. 102 (3), 547-552 (2005).
- Liang, L., Felgner, P. L. A systems biology approach for diagnostic and vaccine antigen discovery in tropical infectious diseases. Current Opinion in Infectious Diseases. 28 (5), 438-445 (2015).
- Jain, A., et al. Evaluation of quantum dot immunofluorescence and a digital CMOS imaging system as an alternative to conventional organic fluorescence dyes and laser scanning for quantifying protein microarrays. Proteomics. 16 (8), 1271-1279 (2016).
- Desbien, A. L., et al. Development of a high density hemagglutinin protein microarray to determine the breadth of influenza antibody responses. Biotechniques. 54 (6), 345-348 (2013).
- Mace, C. R., et al. Label-free, arrayed sensing of immune response to influenza antigens. Talanta. 83 (3), 1000-1005 (2011).
- Koopmans, M., et al. Profiling of humoral immune responses to influenza viruses by using protein microarray. Clinical and Microbiology Infections. 18 (8), 797-807 (2012).
- Bucukovski, J., Latorre-Margalef, N., Stallknecht, D. E., Miller, B. L. A Multiplex Label-Free Approach to Avian Influenza Surveillance and Serology. PLoS One. 10 (8), e0134484 (2015).
- Meade, P., Latorre-Margalef, N., Stallknecht, D. E., Krammer, F. Development of an influenza virus protein microarray to measure the humoral response to influenza virus infection in mallards. Emerging Microbes and Infection. 6 (12), e110 (2017).
- Vigil, A., Davies, D. H., Felgner, P. L. Defining the humoral immune response to infectious agents using high-density protein microarrays. Future Microbiology. 5 (2), 241-251 (2010).
- Nakajima, R., et al. Protein Microarray Analysis of the Specificity and Cross-Reactivity of Influenza Virus Hemagglutinin-Specific Antibodies. mSphere. 3 (6), (2018).
- Van Hoeven, N., et al. A Formulated TLR7/8 Agonist is a Flexible, Highly Potent and Effective Adjuvant for Pandemic Influenza Vaccines. Scientific Reports. 7, 46426 (2017).
- Krammer, F., et al. A carboxy-terminal trimerization domain stabilizes conformational epitopes on the stalk domain of soluble recombinant hemagglutinin substrates. PLoS One. 7 (8), e43603 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon