È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Cancro ipossia stampato in cellule 3D su chip per ricapitolare la progressione patologica del cancro solido
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'ipossia è un segno distintivo del microambiente tumorale e svolge un ruolo cruciale nella progressione del cancro. Questo articolo descrive il processo di fabbricazione di un cancro ipossico su chip basato sulla tecnologia di stampa cellulare 3D per ricapitolare una patologia del cancro correlata all'ipossia.
Abstract
Il microambiente tumorale ha un impatto significativo sulla progressione della malattia. In particolare, l'ipossia è il principale motore della sopravvivenza, dell'invasione e della chemoresistance del cancro. Sebbene siano stati sviluppati diversi modelli in vitro per studiare la patologia del cancro legata all'ipossia, la complessa interazione del microambiente tumorale osservato in vivo non è stata ancora riprodotta a causa della mancanza di un preciso controllo spaziale. Invece, sono stati proposti approcci di biofabbricazione 3D per creare sistemi microfisiologici per una migliore emulazione dell'ecologia del cancro e un'accurata valutazione del trattamento antitumorali. Qui, proponiamo un approccio di stampa cellulare 3D per fabbricare un cancro ipossico su un chip. I componenti ipossia-inducenti nel chip sono stati determinati sulla base di una simulazione al computer della distribuzione dell'ossigeno. Gli anelli concentrici dello stroma tumorale sono stati stampati utilizzando bioinchiostri contenenti cellule di glioblastoma e cellule endoteliali per ricapitolare un tipo di cancro solido. Il chip risultante ha realizzato ipossia centrale e malignità aggravata nel cancro con la formazione di marcatori fisiopatologici rappresentativi. Nel complesso, l'approccio proposto per la creazione di un sistema microfisiologico solido-cancero-mimetico dovrebbe colmare il divario tra modelli in vivo e in vitro per la ricerca sul cancro.
Introduzione
Il microambiente oncologico è un fattore critico che guida la progressione del cancro. Più componenti, inclusi segnali biochimici, biofisici e cellulari, determinano le caratteristiche patologiche del cancro. Tra questi, l'ipossia è fortemente associata alla sopravvivenza, alla proliferazione e all'invasionedel cancro 1. A causa della crescita e della divisione illimitate di cellule tumorali, i nutrienti e l'ossigeno sono continuamente esauriti e viene generato un gradiente ipossia. In condizioni di basso ossigeno, le cellule attivano la cascata molecolare associata al fattore di trascrizione ipossia-inducibile (HIF). Questo processo induce un nucleo necrotico, innesca cambiamenti metabolici e avvia l'iperplasia e la metastasi dei vasisanguigni 2,3. Successivamente, l'ipossia nelle cellule tumorali causa la distruzione dei tessuti normali vicini. Inoltre, l'ipossia è fortemente associata alla resistenza terapeutica dei tumori solidi in modi multifattoriali. L'ipossia può ostacolare gravemente la radioterapia, in quanto la radiosensibilità è limitata a causa della speciereattiva dell'ossigeno 1,4. Inoltre, diminuisce i livelli di pH dei microambientati tumorali, il che diminuisce l'accumulo difarmaci 1. Pertanto, la riproduzione di caratteristiche patologiche legate all'ipossia in vitro è una strategia promettente per i risultati scientifici e precli clinici.
La modellazione di un microambiente specifico del cancro è essenziale per comprendere lo sviluppo del cancro ed esplorare trattamenti appropriati. Sebbene i modelli animali siano stati ampiamente utilizzati a causa della loro forte rilevanza fisiologica, esistono problemi relativi alle differenze di specie e ai problemietici 5. Inoltre, sebbene i modelli 2D e 3D convenzionali consentano la manipolazione e l'imaging in tempo reale delle cellule tumorali per un'analisi approfondita, la loro complessità architettonica e cellulare non può essere completamente riepilogata. Ad esempio, i modelli di sferoidi tumorali sono stati ampiamente utilizzati, poiché l'aggregazione delle cellule tumorali in uno sferoide può generare naturalmente ipossia nel nucleo. Inoltre, un gran numero di sferoidi cellulari di dimensioni uniformi sono stati prodotti utilizzando sistemi multi-pozzo a base di plastica o silicone6,7. Tuttavia, la minore flessibilità per quanto riguarda la cattura dell'esatta struttura eterogenea dei tessuti cancerosi con piattaforme convenzionali ha richiesto la creazione di una tecnologia avanzata di biofabbricazione per costruire una piattaforma altamente biomimetica per migliorare la ricerca sulcancro 8.
I sistemi microfisiologici 3D (MPS) sono strumenti utili per ricapitolare la complessa geometria e progressione patologica delle cellule tumorali9. Poiché le cellule tumorali percepisce il gradiente biochimico dei fattori di crescita e delle chemiochine e l'eterogeneità meccanica riprodotta sul sistema, importanti caratteristiche dello sviluppo del cancro possono essere studiate in vitro. Ad esempio, la vitalità del cancro, la malignità metastatica e la resistenza ai farmaci a seconda delle diverse concentrazioni di ossigeno sono state studiate utilizzando MPS10,11. Nonostante i recenti progressi, la generazione di condizioni ipossiche di modelli in vitro si basa su complesse procedure di fabbricazione, incluso il collegamento con pompe di gas fisiche. Pertanto, sono necessari metodi semplici e flessibili per costruire microambientati specifici per il cancro.
La tecnologia di stampa cellulare 3D ha attirato notevole attenzione grazie al suo preciso controllo della disposizione spaziale dei biomateriali per ricapitolare architetture biologiche native12. In particolare, questa tecnologia supera i limiti esistenti dei modelli di ipossia 3D grazie alla sua elevata controllabilità e fattibilità per costruire le caratteristiche spaziali del microambiente tumorali. La stampa 3D facilita anche la produzione computer-aided attraverso un processo strato per strato, fornendo così una costruzione rapida, accurata e riproducibile di geometrie complesse per imitare le architetture tissutali reali. Oltre ai vantaggi delle strategie di produzione esistenti per gli MPS 3D, le caratteristiche fisiopatiche della progressione del cancro possono essere riprodotte modellando i componenti biochimici, cellulari e biofisici13,14.
Nel presente documento, presentiamo una strategia di stampa cellulare 3D per un cancro ipossico su chip per ricapitolare l'eterogeneità di un cancro solido (Figura 1)15. I parametri di fabbricazione sono stati determinati attraverso una simulazione computazionale della formazione centrale di ipossia nel sistema. Gli anelli concentrici dello stroma tumorale sono stati stampati utilizzando bioinchiostri di collagene contenenti cellule di glioblastoma e cellule endoteliali per emulare la fisiopatologia del glioblastoma, un tipo di cancro solido. La formazione di un gradiente radiale di ossigeno aggravava la malignità del cancro, indicando una maggiore aggressività. Inoltre, indichiamo prospettive future per le applicazioni del chip a modelli preclinici specifici del paziente. L'approccio proposto per la creazione di un sistema microfisiologico solido-cancero-mimetico dovrebbe colmare il divario tra modelli di cancro in vivo e in vitro.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Simulazione al computer della formazione di gradienti di ossigeno
-
Generazione di un modello di geometria 3D per la stampa ipossica cancro su chip
- Eseguire un software CAD 3D.
- Tracciate il modello geometrico del cancro ipossico su chip. Fate clic su Sketch e selezionate il piano desiderato per disegnare la geometria. Fare riferimento al disegno (Figura 2A) per la scala di dettaglio di ciascuna parte.
- Impostate lo spessore della geometria facendo clic su Boss/Base feature-protrusione (Feature-Protrusion Boss/Base). Immettete lo spessore desiderato (fare riferimento alla Figura 2A) nella casella vuota e selezionate l'icona di controllo verde per formare la geometria 3D.
NOTA: La dimensione del cancro su chip è definita in base ai volumi desiderati di media e idrogel. Nel presente esperimento, i volumi desiderati di mezzi e idrogel erano rispettivamente di circa 1.500 μL e 500 μL, sulla base delle precedenti esperienze pratiche per la risoluzione del biostampatore a base di estrusione. - Salvate il file di geometria come formato di file CAD 3D (prt o stl).
-
Determinazione della densità cellulare per l'induzione del nucleo ipossia
- Eseguire un programma di simulazione della diffusione fisica.
- Fate clic su LiveLink e selezionate il programma CAD utilizzato. Fare clic su Sincronizza per importare la geometria del cancer-on-a-chip ipossia nel programma di simulazione. Poiché lo spazio interno della camera sarà riempito con un mezzo di coltura in un ambiente sperimentale reale, l'ossigeno si diffonderà attraverso lo spazio interno della camera e il costrutto cellulare, che sarà composto da idrogel carichi di cellule.
NOTA: Fare riferimento allo studio precedente per i dettagli sui parametri fisici15. - Definire la geometria 3D importata come volume di controllo dello spazio in cui l'ossigeno si diffonde e le celle consumano ossigeno (Figura 2B).
- Eseguire un'analisi computer per l'analisi della diffusione del gas seguendo una guida dell'utente e metodiprecedentemente stabiliti 16,17.
- Dai risultati dell'analisi computerizzati, esportare i dati stimati sulla concentrazione di ossigeno sulla sezione trasversale A-A' in ogni momento seguendo la guida dell'utente. L'equazione di governo si basa sulla prima legge di Fick, come espresso in Eq. (1) (Figura 2C).
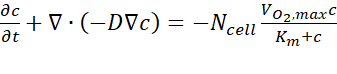
dove c è la concentrazione, D è il coefficiente di diffusione dell'ossigeno, N cella è la densità delle cellule, è la velocità massima di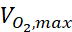 assorbimento dell'ossigeno, e Km è la costante di Michaelis-Menten. Le costanti sono state applicate come descritto in una pubblicazione precedente15.
assorbimento dell'ossigeno, e Km è la costante di Michaelis-Menten. Le costanti sono state applicate come descritto in una pubblicazione precedente15.
NOTA: Ogni punto di tempo indica un punto di passaggio per osservare il cambiamento della diffusione dell'ossigeno nel tempo. - Valutare se il livello minimo di ossigeno raggiunge una soglia di ipossia e ripetere il processo di analisi del computer con un incremento o decremento della densità cellulare.
NOTA: Definire che il gradiente di ipossia si forma nel costrutto se il livello di ossigeno dell'80% nell'area dell'idrogel è inferiore a 0,02 mM dopo 24 ore. - Confermare il numero di cellule necessarie per generare il gradiente di ossigeno che induce ipossia nella regione centrale dalla prima legge di Fick nel passaggio 1.2.5 e i risultati della simulazione dal passaggio 1.2.6.
NOTA: In questo protocollo, il numero di cella era 2 × 106 celle/ogni costrutto.
2. Coltura cellulare delle cellule tumorali e delle cellule stromali
-
Preparazione di mezzi di coltura cellulare per evitare stress fisiologico
- Per le cellule U-87 MG (linea cellulare immortalata di glioblastoma umano), posizionare 12 mL del mezzo Aquila modificato di Dulbecco ad alto glucosio contenente il 10% di siero bovino fetale, 100 U/mL di penicillina e 100 μg/mL di streptomicina in un pallone da coltura cellulare T-75 in un incubatore umidificato di 37 °C, 5% di CO2 per 30 minuti per ridurre al minimo gli effetti termici e alcalini del mezzo sulle cellule.
NOTA: Il glioblastoma è stato scelto come tipo di cancro solido perché ha caratteristiche aggressive in un ambiente ipossico. Altri vari tipi di tumori possono essere applicati a questo modello. - Per le cellule endoteliali venei ombelicali umane (HUVEC), posizionare 12 ml di mezzo di crescita cellulare endoteliale in un pallone da coltura cellulare T-75 in un incubatore umidificato di CO2 al 5% a 37 °C per 30 minuti.
NOTA: gli HUVEC sono stati scelti perché è una delle linee cellulari endoteliali più rappresentative. Vari tipi di cellule stromali possono anche essere applicati a questo modello.
- Per le cellule U-87 MG (linea cellulare immortalata di glioblastoma umano), posizionare 12 mL del mezzo Aquila modificato di Dulbecco ad alto glucosio contenente il 10% di siero bovino fetale, 100 U/mL di penicillina e 100 μg/mL di streptomicina in un pallone da coltura cellulare T-75 in un incubatore umidificato di 37 °C, 5% di CO2 per 30 minuti per ridurre al minimo gli effetti termici e alcalini del mezzo sulle cellule.
-
Rapido scongelamento delle cellule tumorali crioconservate e delle cellule stromali e loro mantenimento
- Spostare i crioviali contenenti 5 x 105 cellule U-87 MG e HUVEC dal contenitore di azoto liquido a un armadio a flusso laminare. Allentare immediatamente e spirare il cappuccio per rilasciare la pressione interna.
- Posizionare delicatamente le cellule crioconservate in un bagno d'acqua a 37 °C per 2 minuti, tenendo il tappo fuori dall'acqua. Risciacquare le fiale con il 70% di etanolo sotto flusso laminare per prevenire la contaminazione.
- Trasferire le cellule scongelate nei contenitori contenenti il supporto di coltura cellulare preparato descritto al passaggio 2.1 e posizionare i contenitori contenenti cellule in un incubatore umidificato di CO2 al 5% a 37 °C per il recupero cellulare.
- Aggiornare il supporto di coltura cellulare ogni 2 giorni e mantenere la crescita cellulare.
- Dopo 24 ore di scongelamento, sostituire il supporto di coltura cellulare per evitare la citotossicità del solfossido di dimetile (DMSO), che è stato utilizzato per il congelamento cellulare. Utilizzare huvec, che ha subito meno di 6 passaggi.
3. Preparazione della soluzione pre-gel di collagene
-
Solubilizzazione della spugna di collagene con 0,1 N acido cloridrico (HCl)
- Preparare una soluzione di 0,1 N HCl e filtrarla con un filtro siringa da 0,2 μm.
- Per 3 mL di una soluzione pre-gel di collagene neutralizzata all'1% (w/v), preparare spugne di collagene tagliate in 5 x 5 mm2 pezzi e del peso di 30 mg.
- Trasferire i pezzi di collagene tagliati in una fiala sterile in vetro da 10 mL.
NOTA: Preparare 1,5 volte il volume dell'idrogel di collagene richiesto, considerando la perdita dell'idrogel a causa della caratteristica appiccicosa della soluzione di collagene. - Aggiungere 2,4 mL di 0,1 N HCl nel flaconcino di vetro contenente collagene e incubarlo sul rocker a 15 giri/min e 4 °C per 3 giorni.
NOTA: Il volume della soluzione di 0,1 N HCl era di quattro quinti del volume finale dell'idrogel di collagene richiesto. In questo caso, sono stati preparati 3 mL di collagene. - Dopo la digestione, setacciare le particelle di collagene non digerite utilizzando un colino cellulare da 40 μm. Conservare la soluzione acida di collagene a 4 °C e utilizzarla entro 7 giorni.
-
regolazione del pH per una soluzione pre-gel di collagene neutralizzata all'1%
- Centrifugare la soluzione acida di collagene a 1224 x g per 5 min a 4 °C.
- Aggiungere 30 μL di soluzione rosso fenolo come indicatore di pH ad una concentrazione finale dell'1% (v/v) e 300 μL di tamponamento salino (PBS) tamponato con fosfato 10x a una concentrazione finale del 10% (v/v) nella soluzione pre-gel di collagene.
- Neutralizzare il pH a 7 con 1 N idrossido di sodio (NaOH), verificando il cambiamento di colore.
NOTA: In base alla formula, le talpe H+ = molarità H+ x volume H+ = talpe OH-= molarità OH- x volume OH-, aggiungono 240 μL di NaOH. - Aggiungere acqua distillata per ottenere un volume totale di 3 mL.
- Dopo la regolazione del pH, conservare la soluzione pre-gel di collagene neutralizzata all'1% (w/v) a 4 °C e utilizzarla entro 3 giorni.
NOTA: Per precontrollare la gelazione della soluzione pre-gel di collagene neutralizzata, fare goccioline di collagene da 50 μL su un piccolo piatto utilizzando una pipetta di spostamento positiva e incubarle in un incubatore di 37 °C per 1 h. Fare riferimento ai seguenti tre metodi per verificare il collegamento incrociato delle goccioline di collagene. - Controlla se il colore del collagene è cambiato in bianco opaco dal colore trasparente.
- Inclinare il contenitore e verificare se il collagene è aderto al fondo del contenitore.
- Versare 1x PBS sulle goccioline e verificare se il costrutto di collagene non è rotto nella soluzione.
4.3D stampa di barriera permeabile al gas
-
Stampa 3D di uno stampo in poli (acetato di etilene-vinile) (PEVA) sacrificale
- Generare la geometria 3D dello stampo PEVA sacrificale definito nel passaggio 1 utilizzando un software CAD 3D (Figura 3A).
NOTA: la geometria 3D e la scala dettagliata del modello, incluse le dimensioni, le unità e i tipi di linea, sono state visualizzate nella figura 2A. - Convertire il file CAD 3D in un formato di file STL facendo clic su File | Tipo di file di salvataggio come STL. Inoltre, clicca su Opzione | Modulo di output come ASCII per la generazione di codice G.
- Clicca su File | Aprite il file STL e selezionate il file STL salvato per importare il file STL generato. Fare clic sul modello Slice dello scambiatore STL-CAD per generare automaticamente il codice G dello stampo PEVA sacrificale ( Figura3B, C).
NOTA: Il percorso di stampa viene generato con la connessione di punti intersecati tra la figura fondamentale del file STL e il piano di affezione (cioè il livello). Fondamentalmente, la figura fondamentale di un frammento in un file STL è un triangolo che contiene le coordinate 3D. Dopo aver ottenuto i punti intersecati tra il triangolo e il livello, viene generato un codice G per la stampa collegando ogni punto senza un tracciato sovrapposto su un livello18. Qualsiasi algoritmo di generazione di codice G a bordo può essere utilizzato per generare percorsi di stampa per la fabbricazione del chip. - Preparare un adesivo sterile e uno scivolo istologico idrofilo.
NOTA: Il vetro scorrevole idrofilo è fondamentale per il legame permanente del polidimetilsiloxano (PDMS) sul vetro e l'adesione dei costrutti di collagene che incapsulano le cellule tumorali e le cellule stromali. - Stampare lo stampo PEVA sacrificale sullo scivolo con un ugello di precisione da 50 G a una pressione pneumatica di 500 kPa a 110 °C.
NOTA: la larghezza della linea è influenzata dalla velocità di avanzamento, dall'indicatore dell'ugello e dalla temperatura del materiale. L'ugello da 50 G è stato utilizzato e una velocità di avanzamento di 400 è stata applicata per generare una larghezza della linea di 500 μm per la parete sacrificale. Il calibro dell'ugello, la pressione pneumatica e la velocità di avanzamento sono definiti con risultatipratici 19. La parete sacrificale deve essere sufficientemente spessa per contenere la soluzione PDMS, che è la fase di fabbricazione successiva.
- Generare la geometria 3D dello stampo PEVA sacrificale definito nel passaggio 1 utilizzando un software CAD 3D (Figura 3A).
-
Fusione della barriera polidimetilsiloxano (PDMS)
- Mescolare 6 mL PDMS elastomero di base e 0,6 mL di polimerizzazione omogeneamente oltre 5 minuti in un serbatoio di plastica. Questo può fabbricare 6 cancero-on-chips ipossici, considerando la perdita dovuta alla caratteristica appiccicosa del PDMS.
- Caricare la soluzione PDMS miscelata in una siringa monouso da 10 ml e montare la testa della siringa con una punta di erogazione affusolata in plastica da 20 G.
- Riempire lo stampo PEVA sacrificale con la soluzione PDMS miscelata nella siringa. Il PDMS miscelato riempirà lo stampo PEVA sacrificale con una superficie convessa. L'altezza della barriera PDMS sarà superiore a quella dello stampo PEVA.
- Curare la barriera PDMS in forno a 40 °C per oltre 36 ore per evitare la fusione di PEVA. Non aumentare la temperatura a oltre 88 °C, che è la temperatura di fusione di PEVA.
- Staccare lo stampo PEVA sacrificale con un paio di pinzette di precisione e sterilizzare la barriera permeabile al gas a 120 °C in autoclave.
5. Preparazione di bio-inchiostri di collagene incapsulati in cellule
-
Distacco delle cellule tumorali preparate e delle cellule stromali
NOTA: Considerando la vitalità delle celle, l'intero processo di stampa deve essere completato il prima possibile dopo aver staccato le celle.- Lavare il cancro e le cellule stromali con 10 mL di 1x PBS utilizzando una pipetta sierologica; trattare con 2 mL dello 0,25% di acido tripsidendiamminatotraacetico (EDTA) utilizzando una pipetta e incubarli per 3 minuti a 37 °C.
- Neutralizzare le cellule tripsinziate con 3 mL di mezzi di coltura cellulare; raccogliere le sospensioni delle cellule in tubi conici da 15 ml e centrifugare a 516 x g per 5 min a 20 °C.
- Aspirare lentamente il supernatante; rimescolare il pellet cellulare in mezzi di coltura cellulare da 5 mL e contare il numero di cellule che utilizzano un emocitometro.
- Trasferire 5 x 106 celle di ciascun tipo di cella in nuovi tubi conici da 15 ml e centrifugarle a 516 x g per 5 minuti a 20 °C.
- Aspirare il supernatante e posizionarlo sul ghiaccio bagnato.
-
Miscelazione di ogni tipo di cellula con la soluzione pre-gel di collagene neutralizzata all'1%
NOTA: Per evitare la solidificazione termica della soluzione pre-gel di collagene neutralizzata all'1%, questo processo deve essere eseguito su ghiaccio umido.- Resuspend ogni tipo di pellet cellulare raccolto nel passaggio 5.1.4 con 20 μL di mezzi di coltura cellulare ciascuno.
- Aggiungere 1 mL della soluzione pre-gel di collagene neutralizzata all'1% in ciascuna delle sospensioni cellulari rimorsipendate e mescolarle in modo omogeneo utilizzando una pipetta di spostamento positiva. La concentrazione finale di ogni tipo di cella sarà di 5 x 106 celle/mL.
- Trasferire i bioinchiostri di collagene incapsulati in cellule in siringhe monouso da 3 ml utilizzando una pipetta monouso positiva e conservare le siringhe a 4 °C fino alla stampa cellulare 3D.
6.3D stampa cellulare di anelli concentrici cancro-stroma
-
Stampa cellulare 3D di bioinchiostri di collagene che incapsulano cellule tumorali e cellule stromali
- Generare la geometria 3D degli anelli concentrici cancro-stroma definiti nel passaggio 1.2 utilizzando un software CAD 3D.
NOTA: Le dimensioni degli anelli concentrici dello stroma tumorale sono definite tramite parametri simulati. Le quote finali dei parametri di quota sono indicate nella figura 3A. - Convertire il file CAD 3D in un formato di file STL e generare un codice G degli anelli concentrici cancer-stroma utilizzando uno scambiatore STL-CAD.
NOTA: fare riferimento alla nota del passaggio 4.1.2 per l'algoritmo di generazione del codice G. - Caricare i bioinchiostri di collagene incapsulati in cellule contenuti in siringhe monouso da 3 ml sulla testa della stampante 3D e impostare la temperatura della testa e della piastra a 15 °C.
NOTA: Se la temperatura della testa e della piastra della stampante raggiunge oltre i 37 °C, il bioinvenk viene collegato e non stampa più. - Caricare il percorso di stampa generato sul software di controllo della stampante 3D.
- Cliccando sul pulsante Start, stampare i bioinchiostri di collagene che incapsulano le cellule tumorali e le cellule stromali sulla barriera permeabile al gas seguendo il codice G caricato con un ago di plastica da 18 G a pressione pneumatica di circa 20 kPa a 15 °C.
- Al termine di ogni operazione di stampa, posizionare manualmente una copertura in vetro sterilizzata da 22 mm x 50 mm sopra la barriera permeabile al gas per generare il gradiente ipossia.
NOTA: Confrontare due gruppi a seconda della presenza di copertura in vetro (GR+) e assenza (GR-) di quello per verificare la generazione del gradiente ipossico. - Dopo aver generato tre cancerosi ipossici su chip, trasferire i chip in un'incubatrice a 37 °C per 1 h per collegare attraverso i bioinchiostri di collagene.
- Generare la geometria 3D degli anelli concentrici cancro-stroma definiti nel passaggio 1.2 utilizzando un software CAD 3D.
-
Completamento del processo di fabbricazione e mantenimento del tumore ipossico su chip
- Dopo aver completato tutti i processi di stampa cellulare 3D dell'ipossico cancero-on-a-chip, strofinare delicatamente gli occhiali di copertura sopra le barriere permeabili al gas con il raschietto cellulare per un incollaggio stretto(Figura 4A,B).
NOTA: Il vetro di copertura e la barriera permeabile al gas vengono assemblati tramite incollaggio idrofobico senza colle chimiche, semplicemente raschiando la parte incollata tra il vetro di copertura e la barriera PDMS. - Introdurre 1,5 mL di mezzo di crescita cellulare endoteliale in ogni chip. Per evitare il distacco del costrutto tumorale, introdurre il mezzo di coltura cellulare da un lato del chip. Inclinare il chip per consentire al supporto di coltura cellulare di fluire utilizzando una pipetta.
- Aggiornare i supporti di coltura cellulare ogni giorno per una settimana. Utilizzare una pipetta per aspirare il mezzo di coltura cellulare; non utilizzare una pompa di pressione.
- Dopo aver completato tutti i processi di stampa cellulare 3D dell'ipossico cancero-on-a-chip, strofinare delicatamente gli occhiali di copertura sopra le barriere permeabili al gas con il raschietto cellulare per un incollaggio stretto(Figura 4A,B).
7. Valutazione della fattibilità delle celle di post-stampa
-
Preparazione di campioni e trattamento con calceina AM ed soluzione EthD-1
- Caldo 1x PBS in un bagno d'acqua a 37 °C.
- Preparare la soluzione di dosaggio aggiungendo 0,75 μL di calceina acetoximetil (calceina AM) e 3 μL di omodimero di etidio (EthD-1) a 1,5 mL PBS prerifabbrato.
- Aspirare con cura tutti i supporti dal chip utilizzando una pipetta.
- Lavare il costrutto del cancro con PBS prebellici. Riempire 1,5 mL PBS nel chip utilizzando una pipetta e lasciarlo riposare per 10 minuti a temperatura ambiente. Per evitare la deformazione del costrutto tumorale, introdurre 1x PBS da un lato dei chip e inclinare i trucioli per consentire il flusso di 1x PBS.
- Aspirare il PBS dal chip; trattare la soluzione di dosaggio da 1,5 mL e incubare il truciolo a 37 °C per 20 minuti utilizzando un foglio per proteggere dalla luce. Utilizzare una pipetta per aspirare 1x PBS; non utilizzare una pompa di aspirazione.
-
Imaging della vitalità cellulare utilizzando un microscopio a fluorescenza
- Visualizzare e acquisire le celle etichettate utilizzando un microscopio a fluorescenza (Figura 4C).
NOTA: Calcein AM contrassegna le cellule vive con fluorescenza verde (lunghezza d'onda ~488 nm). EthD-1 rappresenta il segnale dei globuli morti con fluorescenza rossa (lunghezza d'onda ~594 nm). - Contare il numero di celle vive e morte utilizzando il software di imaging, un programma di elaborazione delle immagini open source e calcolare la fattibilità con i numeri .
- Visualizzare e acquisire le celle etichettate utilizzando un microscopio a fluorescenza (Figura 4C).
8. Immunofluorescenza per convalidare la formazione di ipossia centrale e il suo effetto sulla malignità del cancro
-
Fissazione, permeabilizzazione e blocco del costrutto del cancro
- Preparare 1x PBS, 4% paraformaldeide (PFA), 0,1% (v/v) Tritone X-100 e 2% (con v) albumina di siero bovino (BSA) a temperatura ambiente.
- Aspirare con cura tutti i supporti dal chip utilizzando una pipetta e risciacquare il chip tre volte con 1x PBS. Per evitare la deformazione del costrutto tumorale, introdurre 1x PBS da un lato dei chip e inclinare i trucioli per consentire il flusso di 1x PBS. Tra ogni fase di lavaggio, lasciare riposare il truciolo con 1x PBS per 5 minuti per rimuovere le soluzioni residue.
NOTA: 1x PBS è stato aspirato utilizzando una pipetta, non una pompa a pressione. - Aggiungere 500 μL di PFA al 4% di PFA al costrutto di cancro sul chip utilizzando una pipetta; lasciarlo per 15 minuti e lavare tre volte con 1 PBS per fissare le cellule nel costrutto del cancro.
- Trattare il costrutto di cancro con 500 μL dello 0,1% di Tritone X-100 utilizzando una pipetta a temperatura ambiente per 5 minuti e lavare tre volte con 1x PBS per solubilizzare e permeabilizzare la membrana cellulare.
- Trattare il costrutto di cancro con 500 μL di BSA al 2% utilizzando una pipetta a temperatura ambiente per 1 h per bloccare gli epitopi reattivi.
NOTA: Coprire il truciolo con pellicola di paraffina per evitare l'evaporazione. - Dopo 1 h, lavare il chip tre volte con 1x PBS.
-
Trattamento con anticorpo primario, anticorpo secondario e DAPI e imaging della struttura utilizzando un microscopio confocale.
- Preparare gli anticorpi di controllo dell'isotipo e il cocktail di anticorpi primari diluire gli anticorpi in 1x PBS ad ogni concentrazione di lavoro desiderata.
NOTA: I dettagli specifici degli anticorpi sono elencati nella tabella dei materiali. Devono essere utilizzate le stesse concentrazioni di lavoro degli anticorpi di controllo dell'isotipo degli anticorpi primari. - Aspirare con cura tutto il 1x PBS dal chip utilizzando una pipetta e trattare il chip con soluzione di anticorpi primari da 200 μL a 4 °C durante la notte. Coprire i trucioli con pellicola di paraffina per evitare l'evaporazione.
- Aspirare la soluzione anticorpale primaria e lavare il chip tre volte con 1x PBS.
- Diluire gli anticorpi secondari e il DAPI in 1x PBS alla concentrazione di lavoro desiderata.
NOTA: In questo caso viene utilizzato un anticorpo secondario coniugato a fluorescenza verde con un rapporto di 1:200. DAPI è stato utilizzato con un rapporto di 1:1000. - Aspirare con cura tutto il 1x PBS dal chip utilizzando una pipetta e trattare il chip con soluzione anticorpo-DAPI secondaria da 200 μL a 4 °C per 3 ore. Coprire il truciolo con pellicola di paraffina per evitare l'evaporazione e quindi avvolgerlo con un foglio di alluminio per evitare il fotobleaching.
- Aspirare la soluzione anticorpo-DAPI secondaria e lavare il chip tre volte con 1x PBS.
- Dopo aver terminato la fase di colorazione, trasferire il costrutto di cancro su un piatto confocale afferrando delicatamente con le forcep.
- Visualizzare e acquisire le celle etichettate utilizzando un microscopio confocale (Figura 5).
NOTA: La lunghezza d'onda del microscopio confocale è stata regolata, a seconda del tipo di marcatori fluorescenti. I dettagli specifici degli anticorpi sono elencati nella tabella dei materiali. Per rilevare in modo efficiente la posizione della cella, sarebbe meglio osservare i nuclei macchiati DAPI del costrutto all'inizio. Le lunghezze d'onda di eccitazione/emissione di rilevamento dei segnali fluorescenti erano 358/461 nm (DAPI, Blue), 494/517 nm (Green) e 590/617 nm (Rosso). Gli ingrandimenti erano 4x, 10x e 20x, regolati dal più basso al più alto.
- Preparare gli anticorpi di controllo dell'isotipo e il cocktail di anticorpi primari diluire gli anticorpi in 1x PBS ad ogni concentrazione di lavoro desiderata.
9. Analisi statistica
-
Conteggio delle celle con programma di elaborazione delle immagini
- Eseguire un programma di elaborazione delle immagini per contare il numero di celle vive e morte.
- Aprire i file di immagine fluorescenti. Clicca su File | Aprire e importare le immagini TIFF.
- Convertire le immagini in immagini in scala di grigi a 16 bit. Clicca su Immagine | Digitare | Scala di grigi a 16 bit.
- Regolare la soglia cliccando su Immagine | Regola | Soglia e quindi selezionare il colore delle celle in nero.
- Tagliare le celle unite facendo clic su Elabora | Binario | Spartiacque per un conteggio preciso delle celle.
- Contare il numero di celle cliccando su Analizza e quindi su Analizza particelle tre volte; calcolare la media e presentare i dati come la media ± errore standard.
NOTA: I marcatori di immunofluorescenza sono stati analizzati confrontando l'intensità della fluorescenza.
Access restricted. Please log in or start a trial to view this content.
Risultati
L'ipossico cancer-on-a-chip è stato sviluppato utilizzando la tecnologia di stampa cellulare 3D aiutata dal computer per ricapitolare l'ipossia e la patologia correlata al cancro (Figura 1). Il trasporto e il consumo di ossigeno sono stati simulati utilizzando il modello di geometria 3D. Il chip è stato progettato sotto forma di anelli concentrici per imitare la diffusione e l'esaurimento radiale dell'ossigeno nei tessuti tumorali (Figu...
Access restricted. Please log in or start a trial to view this content.
Discussione
In questo studio, descriviamo il processo di fabbricazione di un cancro ipossico su chip basato sulla tecnologia di stampa cellulare 3D. La formazione del gradiente ipossia nel chip progettato è stata prevista attraverso simulazioni al computer. L'ambiente in grado di indurre un gradiente ipossia eterogeneo è stato riprodotto attraverso una semplice strategia che combina la barriera permeabile al gas stampata in 3D e la copertura in vetro. Le caratteristiche patologiche correlate all'ipossia del glioblastoma, tra cui l...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno rivelazioni.
Riconoscimenti
Questa ricerca è stata sostenuta dalla National Research Foundation of Korea (NRF) finanziata dal Ministero dell'Istruzione (n. 2020R1A6A1A03047902 e NRF-2018H1A2A1062091) e dal governo coreano (MSIT) (n. NRF-2019R1C1C1009606 e NRF-2019R1A3A3005437).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Cells | |||
| Human umbilical vein endothelial cells | Promocell | C-12200 | |
| U-87 MG cells | ATCC | ATCC HTB-14 | |
| Disposable | |||
| 0.2 μm syringe filter | Sartorius | 16534-K | |
| 10 mL disposable syringe | Jung Rim | 10ml 21G32 | |
| 10 mL glass vial | Hubena | A0039 | |
| 10 mL Serological pipette tip | SPL lifescience | 91010 | |
| 15 mL conical tube | SPL lifescience | 50015 | |
| 18G plastic needle | Musashi engineering | PN-18G-B | |
| 20G plastic tapered dispense tip | Musashi engineering | TPND-20G-U | |
| 22x50 glass cover | MARIENFIELD | 0101142 | |
| 25 mL Serological pipette tip | SPL lifescience | 90125 | |
| 3 mL disposable syringes | HENKE-JET | 4020-X00V0 | |
| 40 µm cell strainer | Falcon | 352360 | |
| 5 mL Serological pipette tip | SPL lifescience | 91005 | |
| 50 mL conical tube | SPL lifescience | 50050 | |
| 50 mL Serological pipette tip | SPL lifescience | 90150 | |
| 50N precision nozzle | Musashi engineering | HN-0.5ND | |
| Aluminum foil | SINKWANG | ||
| Capillary tips | Gilson | CP1000 | |
| Cell-scrapper | SPL lifescience | 90030 | |
| Confocal dish | SPL lifescience | 200350 | |
| Parafilm | Bemis | PM996 | |
| Pre-coated histology slide | MATSUNAMI | MAS-11 | |
| Reservoir | SPL lifescience | 23050 | |
| T-75 cell culture flask | SPL lifescience | 70075 | |
| Equipment | |||
| 3DX printer | T&R Biofab | ||
| Autoclave | JEIOTECH | AC-12 | |
| Centrifuger | Cyrozen | 1580MGR | |
| Confocal laser microscopy | Olympus Life Science | FV 1000 | |
| Fluorescence microscope | FISHER SCEINTIFIC | O221S366 | |
| Forcep | Korea Ace Scientific | HC.203-30 | |
| Hand tally counter | KTRIO | ||
| Hemocytometer | MARIENFIELD | 0650030 | |
| Incubator | Panasonic | MCO-170AIC | |
| Laminar flow cabinet | DAECHUNG SCIENCE | CB-BMMS C-001 | |
| Metal syringe | IWASHITA engineering | SUS BARREL 10CC | |
| Operating Scissors | Hirose | HC.13-122 | |
| Oven | JEIOTECH | OF-12, H070023 | |
| Positive displacement pipette | GILSON | NJ05652 | |
| Refrigerator | SAMSUNG | CRFD-1141 | |
| Voltex Mixer | DAIHAN scientific | VM-10 | |
| Water bath | DAIHAN SCIENTIFIC | WB-11 | |
| Water purifier | WASSER LAB | DI-GR | |
| Materials | |||
| 0.25 % Trypsin-EDTA | Gibco | 25200-072 | |
| 10x PBS | Intron | IBS-BP007a | |
| 4% Paraformaldehyde | Biosesang | ||
| 70% Ethanol | Daejung | 4018-4410 | |
| Anti-CD31 antibody | Abcam | ab28364 | |
| Anti-HIF-1 alpha antibody | Abcam | ab16066 | |
| Anti-SHMT2/SHMT antibody | Abcam | ab88664 | |
| Anti-SOX2 antibody | Abcam | ab75485 | |
| Bovine Serum Albumin | Thermo scientific | J10857-22 | |
| Collagen from porcine skin | Dalim tissen | PC-001-1g | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Thermofisher | D1306 | |
| Endothelial Cell Growth Medium-2 | Promocell | C22011 | |
| Fetal bovine serum | Gibco | 12483-020 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Theromofisher | A-11001 | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 594 | Theromofisher | A-11012 | |
| High-glucose Dulbecco’s Modified Eagle Medium(DMEM) | Hyclone | SH30243-0 | |
| Hydrochloric acid | Sigma-Aldrich | 311413-100ML | |
| Live/dead assay kit | Invitrogen | L3224 | |
| Mouse IgG1, kappa monoclonal [15-6E10A7] - Isotype Control | Abcam | ab170190 | |
| Penicillin/streptomycin | Gibco | 15140-122 | |
| Phenol red solution | Sigma-Aldrich | P0290-100ML | |
| Poly(ethylene-vinyl acetate) | Poly science | 06108-500 | |
| Polydimethylsiloxane | Dowhitech | sylgard 184 | |
| Rabbit IgG, polyclonal - Isotype Control | Abcam | ab37415 | |
| Sodium hydroxide solution | Samchun | S0610 | |
| Triton X-100 | Biosesang | TRI020-500-50 | |
| Trypan Blue | Sigma-Aldrich | T8154 | |
| Software | |||
| COMSOL Multiphysics 3.5a | COMSOL AB | ||
| IMS beamer | in-house software | ||
| SolidWorks Package | Dassault Systems SolidWorks Corporation |
Riferimenti
- Jing, X., et al. Role of hypoxia in cancer therapy by regulating the tumor microenvironment. Molecular Cancer. 18 (1), 157(2019).
- Al Tameemi, W., Dale, T. P., Al-Jumaily, R. M. K., Forsyth, N. R. Hypoxia-modified cancer cell metabolism. Frontiers in Cell and Developmental Biology. 7, 4(2019).
- Petrova, V., Annicchiarico-Petruzzelli, M., Melino, G., Amelio, I. The hypoxic tumour microenvironment. Oncogenesis. 7 (1), 1-13 (2018).
- Hockel, M., Vaupel, P. Tumor hypoxia: definitions and current clinical, biologic, and molecular aspects. Journal of the National Cancer Institute. 93 (4), 266-276 (2001).
- Kim, H., Lin, Q., Glazer, P. M., Yun, Z. The hypoxic tumor microenvironment in vivo selects the cancer stem cell fate of breast cancer cells. Breast Cancer Research. 20 (1), 16(2018).
- Jeong, G. S., Lee, J., Yoon, J., Chung, S., Lee, S. -H. Viscoelastic lithography for fabricating self-organizing soft micro-honeycomb structures with ultra-high aspect ratios. Nature Communications. 7 (1), 1-9 (2016).
- Razian, G., Yu, Y., Ungrin, M. Production of large numbers of size-controlled tumor spheroids using microwell plates. Journal of Visualized Experiments:JoVE. (81), e50665(2013).
- Nunes, A. S., Barros, A. S., Costa, E. C., Moreira, A. F., Correia, I. J. 3D tumor spheroids as in vitro models to mimic in vivo human solid tumors resistance to therapeutic drugs. Biotechnology and Bioengineering. 116 (1), 206-226 (2019).
- Wan, L., Neumann, C., LeDuc, P. Tumor-on-a-chip for integrating a 3D tumor microenvironment: chemical and mechanical factors. Lab on a Chip. 20 (5), 873-888 (2020).
- Nam, H., Funamoto, K., Jeon, J. S. Cancer cell migration and cancer drug screening in oxygen tension gradient chip. Biomicrofluidics. 14 (4), 044107(2020).
- Palacio-Castañeda, V., Kooijman, L., Venzac, B., Verdurmen, W. P., Le Gac, S. Metabolic switching of tumor cells under hypoxic conditions in a tumor-on-a-chip model. Micromachines. 11 (4), 382(2020).
- Ronaldson-Bouchard, K., Vunjak-Novakovic, G. Organs-on-a-chip: a fast track for engineered human tissues in drug development. Cell Stem Cell. 22 (3), 310-324 (2018).
- Mi, S., Du, Z., Xu, Y., Sun, W. The crossing and integration between microfluidic technology and 3D printing for organ-on-chips. Journal of Materials Chemistry B. 6 (39), 6191-6206 (2018).
- Yi, H. -G., Lee, H., Cho, D. -W. 3D printing of organs-on-chips. Bioengineering. 4 (1), 10(2017).
- Yi, H. -G., et al. A bioprinted human-glioblastoma-on-a-chip for the identification of patient-specific responses to chemoradiotherapy. Nature Biomedical Engineering. 3 (7), 509-519 (2019).
- Kang, T. -Y., Hong, J. M., Jung, J. W., Yoo, J. J., Cho, D. -W. Design and assessment of a microfluidic network system for oxygen transport in engineered tissue. Langmuir. 29 (2), 701-709 (2013).
- Woo Jung, J., et al. Evaluation of the effective diffusivity of a freeform fabricated scaffold using computational simulation. Journal of Biomechanical Engineering. 135 (8), (2013).
- Brown, A. C., De Beer, D. Development of a stereolithography (STL) slicing and G-code generation algorithm for an entry level 3-D printer. 2013 Africon (IEEE). , 1-5 (2013).
- Shim, J. -H., Lee, J. -S., Kim, J. Y., Cho, D. -W. Bioprinting of a mechanically enhanced three-dimensional dual cell-laden construct for osteochondral tissue engineering using a multi-head tissue/organ building system. Journal of Micromechanics and Microengineering. 22 (8), 085014(2012).
- Gillispie, G., et al. Assessment methodologies for extrusion-based bioink printability. Biofabrication. 12 (2), 022003(2020).
- Kim, B. S., Das, S., Jang, J., Cho, D. -W. Decellularized extracellular matrix-based bioinks for engineering tissue-and organ-specific microenvironments. Chemical Reviews. 120 (19), 10608-10661 (2020).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon