È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un sistema di coltura automatizzato da utilizzare nei test preclinici di terapie dirette all'ospite per la tubercolosi
In questo articolo
Riepilogo
La quantificazione rapida ed efficiente della crescita intracellulare di M. tuberculosis è fondamentale per perseguire terapie migliori contro la tubercolosi (TB). Questo protocollo descrive un test di rilevamento colorimetrico basato sul brodo utilizzando un sistema di coltura liquida automatizzato per quantificare la crescita di Mtb nei macrofagi trattati con terapie candidate dirette dall'ospite.
Abstract
Mycobacterium tuberculosis (Mtb), l'agente eziologico della tubercolosi (TB), è stato il killer di malattie infettive più significativo a livello globale fino all'avvento di COVID-19. Mtb si è evoluto per persistere nel suo ambiente intracellulare, eludere le difese dell'ospite e ha sviluppato resistenza a molti farmaci antitubercolari. Un approccio per risolvere la resistenza è identificare i farmaci approvati esistenti che aumenteranno la risposta immunitaria dell'ospite alla Mtb. Questi farmaci potrebbero quindi essere riproposti come terapie aggiuntive dirette dall'ospite (HDT) per ridurre i tempi di trattamento e aiutare a superare la resistenza agli antibiotici.
La quantificazione della crescita intracellulare di Mtb nei macrofagi è un aspetto cruciale della valutazione del potenziale HDT. Il gold standard per misurare la crescita della Mtb è il conteggio delle unità formanti colonie (CFU) su piastre di agar. Questo è un test lento e laborioso che non si presta a uno screening rapido dei farmaci. In questo protocollo, un sistema di coltura automatizzato basato sul brodo, che è più comunemente usato per rilevare Mtb nei campioni clinici, è stato adattato per lo screening preclinico delle terapie dirette all'ospite. È stata valutata la capacità del sistema di analisi della coltura liquida di studiare la crescita intracellulare di Mtb nei macrofagi trattati con HDT. Gli HDT testati per la loro capacità di inibire la crescita di Mtb erano acido retinoico all-trans (AtRA), sia in soluzione che incapsulati in microparticelle di poli(acido lattico-co-glicolico) (PLGA) e la combinazione di interferone-gamma e linezolid. I vantaggi di questa tecnica automatizzata basata su colture liquide rispetto al metodo CFU includono semplicità di configurazione, preparazione meno laboriosa e tempi di risultati più rapidi (5-12 giorni rispetto ai 21 giorni o più per le piastre di agar).
Introduzione
Il Mycobacterium tuberculosis (Mtb), l'agente eziologico della tubercolosi, è stato il killer di malattie infettive più significativo a livello globale nel 20191. Per eludere le difese dell'ospite, Mtb sovverte l'attività micobattericida delle cellule immunitarie innate come i macrofagi e le cellule dendritiche (DC), permettendogli di persistere intracellulare e replicarsi2. La mancanza di un vaccino efficace per prevenire la tubercolosi polmonare adulta e la crescente comparsa di ceppi resistenti ai farmaci evidenziano l'urgente necessità di nuove terapie.
Le terapie aggiuntive dirette dall'ospite (HDT) potrebbero ridurre i tempi di trattamento e aiutare a superare la resistenza3. La valutazione preclinica dei candidati HDT in vitro per determinare l'attività micobattericida all'interno dei macrofagi si basa spesso sulla quantificazione della crescita di Mtb da parte di unità formanti colonie (CFU) su piastre di agar solido. Questo è un test lento e laborioso che non si presta a uno screening rapido dei farmaci. I sistemi di rilevamento microbico automatizzati basati sul brodo disponibili in commercio sono più comunemente utilizzati nei laboratori di microbiologia clinica per il rilevamento e i test di sensibilità ai farmaci di Mtb e altre specie micobatteriche nei campioni clinici4. Questi strumenti misurano la crescita indirettamente in base all'attività metabolica batterica che porta a cambiamenti fisici nei terreni di coltura (variazione dei livelli di CO 2 o O2 o pressione) monitorati nel tempo5. La lettura è il tempo di positività (TPP), che ha precedentemente dimostrato di correlare con CFU Mtb in campioni di espettorato di pazienti affetti da tubercolosi in risposta al trattamento 6,7 e in lisati di polmone murino infetto e milza8. Inoltre, sono stati utilizzati sistemi di rilevamento di colture liquide per misurare l'effetto delle terapie convenzionali dirette da patogeni sulla crescita di micobatteri in coltura axenica e macrofagi in coltura 9,10. Lo strumento è stato utilizzato anche per studiare la capacità innata delle cellule dendritiche e dei macrofagi alveolari di controllare la crescita intracellulare di Mtb11,12. Questo protocollo sperimentale dimostra che un sistema diagnostico di coltura liquida può essere adattato per eseguire lo screening preclinico dell'HDT per la tubercolosi nei macrofagi in coltura. Rispetto al censimento delle CFU, il vantaggio principale di questa tecnica è che riduce considerevolmente il lavoro sperimentale e il tempo necessario per quantificare la crescita/sopravvivenza micobatterica intracellulare. Questa tecnica si basa sull'accesso a uno strumento di coltura automatizzato che può essere utilizzato per valutare la sopravvivenza micobatterica intracellulare nelle cellule immunitarie trattate con una vasta gamma di reagenti farmacologici mirati alle funzioni cellulari per aumentare l'immunità dell'ospite.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Gli esperimenti delineati in questo protocollo sono stati condotti utilizzando il ceppo H37Ra attenuato di Mtb, che può essere gestito in un laboratorio di livello di contenimento 2. Tutte le manipolazioni di micobatteri vivi sono state effettuate in un armadio di sicurezza biologica di classe II (BSC). Le procedure sperimentali sono state progettate per ridurre al minimo la generazione di aerosol. La coltura cellulare eucariotica (cellule THP-1) è stata effettuata anche in una BSC di Classe II. Il laboratorio ha effettuato una valutazione del rischio e ha assicurato che tutte le procedure fossero eseguite in linea con le normative istituzionali e nazionali sulla sicurezza biologica. La linea cellulare monocitica umana THP-1 è stata utilizzata per eseguire il metodo come descritto (fase 1). Le cellule sono differenziate in macrofagi dopo stimolazione con forbolo 12-miristato 13-acetato (PMA) prima dell'infezione da micobatteri.
1. Coltura cellulare
- Propagare lo stock di semi di H37Ra alla fase logaritmica nel brodo Middlebrook 7H9 (MB) integrato con arricchimento di albumina-destrosio-catalasi (ADC) (10%) e polisorbato allo 0,05% 80. Conservare le scorte di H37Ra in 1 mL di aliquote in un congelatore a -80 °C per un massimo di 1 anno.
- Scongelare un flaconcino da 1 mL di Mtb-H37Ra e trasferirlo in un matraccio T25 con un tappo filtrante contenente 9 mL di brodo integrato MB circa 1 settimana prima dell'esperimento pianificato. Incubare a 37 °C per 5-7 giorni in incubatrice statica.
- Coltivare cellule THP-1 in RPMI-1640 integrate con siero fetale di vitello non ucciso dal calore (RPMI completo (c) RPMI) in un pallone T75 in un incubatore umidificato di CO 2 al 5% di CO2/37 °C e sottocoltura due volte alla settimana per mantenere una densità inferiore a 1 x 106 cellule / ml.
- Differenziare le cellule THP-1 in macrofagi 3 giorni prima dell'infezione pipettando delicatamente le cellule più volte usando una pipetta sierologica in palloni T75 per disperdere eventuali grumi e metterli in un tubo conico da 50 ml.
- Centrifugare le celle a 300 x g per 10 minuti a temperatura ambiente, decantare il surnatante e risospendere delicatamente il pellet in 2 ml di RPMI. Eseguire il conteggio delle cellule per stimare le cellule/ml.
- Semina 2 mL di macrofagi THP-1 in piastre di coltura tissutale a 12 pozzetti ad una densità di 100.000 cellule/ml in cRPMI con 100 ng/mL PMA per 72 ore. Rimuovere il mezzo contenente PMA dalle cellule e ricostituire con cRPMI fresco prima dell'infezione da Mtb.
- Impostare le singole piastre per ogni punto temporale richiesto.
- Cellule seme alla stessa densità (100.000 cellule / ml) in vetrini a 2 pozzetti per determinare la molteplicità dell'infezione (MOI).
- Mettere in un incubatore umidificato al 5% di CO2 per 3 giorni a 37 °C.
2. Quantificazione dell'assorbimento di Mtb
- Determinazione dell'assorbimento di Mtb da parte dei macrofagi (MOI)
- Installare l'armadio di sicurezza biologica di classe II (BSC) il giorno dell'infezione e lavorare su due strati di carta velina per rilevare eventuali fuoriuscite. Impostare i contenitori per gli scarti dei rifiuti secondo le normative locali.
- Prelevare 6-8 mL di coltura micobatterica dal matraccio T25 e trasferirlo in un tubo di polipropilene da 15 ml.
NOTA: tubi di volume più piccolo possono essere utilizzati per esperimenti più piccoli. - Centrifugare il tubo in una centrifuga da banco a temperatura ambiente per 10 minuti a 2890 x g.
- Rimuovere con cautela il tubo dalla centrifuga e trasferirlo nell'armadio di sicurezza biologica. Attendere 1 minuto per consentire ai batteri di depositarsi.
- Versare il surnatante nel contenitore di scarto del disinfettante, ricapitolare il tubo e risospendere i batteri nel mezzo rimanente toccando il lato del tubo. Attendere 1 minuto per consentire ai batteri di depositarsi.
- Aggiungere 2 mL di cRPMI preriscaldato, mescolare delicatamente e trasferire in un tubo conico da 50 mL.
- Risospendere i micobatteri con molta attenzione utilizzando un ago da 25 G e una siringa da 5 ml. Per risospendere, aspirare la sospensione di micobatteri nella siringa ed espellere molto delicatamente lungo la parete laterale del tubo per ridurre al minimo la produzione di aerosol. Ripeti 6-8 volte.
NOTA: Prestare la massima attenzione in quanto si tratta di una coltura ad alta densità di micobatteri. Per evitare il rischio di lesioni da puntura d'ago, utilizzare aghi smussati ove possibile e siringhe Luer lock. - Smaltire l'ago e la siringa in un contenitore per oggetti taglienti nella BSC.
- Trasferire la sospensione in un tubo di microfughe da 2 ml (con tappo a vite) e centrifugare a temperatura ambiente per 3 minuti a 100 x g per pellettare eventuali grumi rimanenti. Riportare il tubo nell'armadio di sicurezza e attendere 1 minuto per consentire ai batteri di depositarsi.
- Trasferire i primi 1-1,5 ml del surnatante in una nuova provetta. Gettare il tubo originale nel cestino dei rifiuti contenente il disinfettante. Mescolare bene e aggiungere varie quantità di sospensione micobatterica (ad es. 5, 25, 50, 150 μL) ai vetrini a 2 pozzetti e incubare per 3 ore in un incubatore di CO2 a 37 °C.
- Colorazione per batteri acidi veloci (AFB)
NOTA: Dopo 3 ore di incubazione, i macrofagi vengono lavati e fissati con paraformaldeide per inattivare i micobatteri. I vetrini vengono quindi colorati utilizzando un kit Auramine O modificato (vedi Tabella dei materiali) per stimare i micobatteri fagocitati per cellula. A causa della loro parete cellulare cerosa, i micobatteri trattengono il colorante Auramine dopo un lavaggio acido-alcolico. I nuclei dei macrofagi vengono quindi controcolorati con Hoechst. Questo metodo consente di contare il numero di batteri fagocitati per cellula e viene utilizzato per determinare la molteplicità di infezione (MOI) dei macrofagi.- Rimuovere il mezzo dal vetrino della camera di vetro dopo aver pipettato su e giù tre volte per rimuovere i batteri che non sono stati fagocitati.
- Lavare una volta con 2 ml di PBS a temperatura ambiente.
- Conservare scorte di paraformaldeide al 4%, disciolta in PBS in aliquote a -20 °C per un massimo di 6 mesi. Scongelare un'aliquota di paraformaldeide al 4% immediatamente prima dell'uso. Diluire al 2% di paraformaldeide con PBS e aggiungere 2 ml per pozzetto.
- Incubare per 10 minuti a temperatura ambiente. La slitta della camera di vetro può essere rimossa dall'armadio di sicurezza in questa fase per la colorazione.
- Lavare il vetrino sotto un getto delicato di acqua di rubinetto.
- Erogare una quantità sufficiente di Auramina sul vetrino per coprire le cellule utilizzando una pipetta di trasferimento di plastica e incubare per 1 minuto a temperatura ambiente al buio (coprire con un foglio di alluminio).
- Lavare il colorante in eccesso dal vetrino con acqua di rubinetto e aggiungere il decoloratore / quencher per 1 minuto al buio.
- Lavare via l'eccesso con acqua di rubinetto e incubare per 15 minuti a temperatura ambiente con Hoechst 33342 (10 μg/mL in PBS) al buio.
- Lavare via la macchia Hoechst con acqua di rubinetto, rimuovere le camere, drenare l'acqua in eccesso dal vetrino, aggiungere una goccia di antisbiadimento e copricopertina e asciugare all'aria.
- Esaminare il vetrino al microscopio fluorescente utilizzando l'obiettivo olio 100x. I micobatteri diventano verdi fluorescenti sotto il filtro FITC. I nuclei diventano blu fluorescente sotto il filtro DAPI (Figura 1C).
- Determinare il MOI contando il numero di micobatteri fagocitati per cellula e la percentuale di cellule infette.
- Calcolare il volume di sospensione micobatterica necessaria per ottenere il MOI richiesto in base alla superficie di un pozzetto nella piastra; Ad esempio, la superficie dei vetrini della camera di vetro utilizzati in questo esperimento è di 4 cm2. Un basso MOI (circa 1-2 bacilli / cellula) è auspicabile per esperimenti condotti per diversi giorni (ad esempio, 5 giorni).
- Infezione dei macrofagi
- Mescolare bene la sospensione di micobatteri e aggiungere la quantità necessaria alle cellule su piastre a 12 pozzetti una volta determinato il volume richiesto per ottenere il MOI desiderato.
- Incubare a 37 °C per 3 ore per consentire la fagocitazione dei micobatteri.
- Rimuovere i batteri extracellulari lavando i pozzetti con RPMI caldo o HBSS più volte.
- Lisare i macrofagi in un pozzetto (campione di 3 ore) per determinare il tempo percentuale di positività (TTP) dell'inoculo iniziale (campione di 3 ore) come descritto nel passaggio 3 di seguito.
- Aggiungere cRPMI fresco e le dosi di farmaco necessarie o il controllo del veicolo ai pozzetti rimanenti, incubare le piastre nell'incubatore di CO2 a 37 °C per il tempo necessario (a seconda del disegno sperimentale, ma di solito a intervalli diversi tra 1 e 8 giorni).
3. Raccolta di campioni per il sistema di rilevamento della coltura liquida
NOTA: Il giorno dell'infezione, i micobatteri extracellulari vengono rimossi mediante lavaggio e i micobatteri intracellulari vengono raccolti mediante lisi di un pozzetto di macrofagi (campione di 3 ore) per determinare la quantità iniziale fagocitata come controllo di base per l'infezione. In tempi successivi sia il substrato, il lisato cellulare e i lavaggi vengono combinati per misurare la crescita micobatterica totale. La crescita extracellulare e intracellulare può anche essere valutata separatamente, se lo si desidera.
- Raccolta di 3 ore di campione per determinare TTP
- Lavare via i micobatteri extracellulari da tutti i pozzetti dopo le prime 3 ore di infezione come descritto al punto 2.3.3. Aggiungere 1 mL di fluido fresco al pozzetto di controllo di 3 ore per equalizzare il volume di lisato con quelli dei punti temporali successivi.
NOTA: vedere il punto 3.2.7 se i micobatteri extracellulari devono essere esclusi dall'analisi.
- Lavare via i micobatteri extracellulari da tutti i pozzetti dopo le prime 3 ore di infezione come descritto al punto 2.3.3. Aggiungere 1 mL di fluido fresco al pozzetto di controllo di 3 ore per equalizzare il volume di lisato con quelli dei punti temporali successivi.
- Raccolta di campioni
- Riscaldare il brodo MB e le bottiglie di coltura degli strumenti per portarle a temperatura ambiente.
- Trasferire il mezzo dalla piastra a 12 pozzetti ai corrispondenti tubi conici marcati.
- Aggiungere 500 μL di tampone di lisi sterile (0,1% Triton x-100 in PBS filtrato attraverso un filtro sterile da 0,2 μm) a ciascun pozzetto per 10 minuti.
- Raschiare delicatamente le cellule dal pozzetto con un raschietto sterile e combinare con il mezzo nel tubo conico appropriato.
- Lavare il pozzetto con 0,5 ml di PBS sterile e trasferire nel tubo appropriato.
- Passare delicatamente ogni campione attraverso un ago e una siringa (25 G) 6-8 volte per rompere i grumi. Diluire i campioni 1:10 in brodo MB; Campione da 100 μL + 900 μL MB di terreno.
- Nei tempi/giorni richiesti (di solito da 1 a 8 giorni), prelevare i campioni rimanenti seguendo i passaggi 3.2.1-3.2.6 di cui sopra.
NOTA: I ricercatori potrebbero preferire escludere i micobatteri extracellulari dalla loro analisi, nel qual caso, nel punto 3.2.2 sopra, il mezzo da ciascun pozzetto viene scartato e i macrofagi vengono lavati più volte prima di aggiungere tampone di lisi.
- Inoculazione e caricamento di bottiglie per colture di strumenti
NOTA: I dettagli dello strumento di coltura liquida e dei relativi materiali di consumo sono forniti nella tabella dei materiali.- Sterilizzare il tappo di gomma del flacone per la coltura dello strumento con carta velina imbevuta di alcool al 70% e lasciarlo asciugare all'aria.
NOTA: questo passaggio deve essere eseguito nel BSC. - Preparare i flaconi trasferendo una quantità sufficiente di integratori nutrizionali per tutti i campioni in un tubo conico (0,5 ml/bottiglia). Utilizzare un ago e una siringa per iniettare 0,5 ml di integratore alimentare nel flacone di coltura dello strumento.
- Pipettare 500 μL del campione diluito (1:10) in una provetta con fondo a V da 1 mL.
- Utilizzare un ago e una siringa per iniettare i 500 μL di campione nel flacone di coltura dello strumento assegnato.
- Sterilizzare il tappo di gomma del flacone per la coltura dello strumento con carta velina imbevuta di alcool al 70% e lasciarlo asciugare all'aria. Pulire le bottiglie con carta velina imbevuta di alcool al 70% prima di essere rimosse dalla BSC.
- Trasportare con cura le bottiglie dall'armadio di biosicurezza allo strumento per il caricamento.
- Premere il pulsante di caricamento sul sistema di rilevamento microbico automatizzato.
- Scansiona i codici a barre sui flaconi per la coltura degli strumenti e posizionali nell'incubatore del sistema di rilevamento a 37 °C per un massimo di 42 giorni. Leggi e registra il tempo impiegato per raggiungere la positività dallo schermo dello strumento.
NOTA: Il codice a barre consente allo strumento di identificare la bottiglia e collegare le letture di riflettanza con una particolare bottiglia. - Calcolare il tempo percentuale di positività (TTP) confrontando il TTP dell'inoculo intracellulare iniziale (Giorno 0) con quello dei macrofagi coltivati per i tempi indicati. Una variazione positiva della percentuale TTP significa crescita micobatterica13.
Ad esempio, per il giorno 3:
Variazione percentuale nel tempo di positività =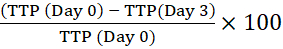
- Sterilizzare il tappo di gomma del flacone per la coltura dello strumento con carta velina imbevuta di alcool al 70% e lasciarlo asciugare all'aria.
Access restricted. Please log in or start a trial to view this content.
Risultati
Lo strumento automatizzato di coltura liquida utilizzato in questo studio monitora i livelli di CO2 ogni 10 minuti. Un cambiamento di colore nel sensore nella parte inferiore della bottiglia dello strumento viene misurato colorimetricamente ed espresso come unità di riflettanza. Il software dello strumento applica quindi algoritmi di rilevamento per calcolare il tempo di positività (TTP), cioè il numero di giorni dall'inoculazione fino a quando le colture vengono contrassegnate come positive (
Access restricted. Please log in or start a trial to view this content.
Discussione
Gli autori hanno utilizzato il metodo di coltura liquida descritto in questo protocollo per monitorare la crescita di Mtb in macrofagi derivati da monociti e macrofagi alveolari e cellule THP-1 differenziate con PMA11,16,17. Questa tecnica può anche essere modificata per l'uso con cellule non aderenti12. Più recentemente, lo strumento è stato utilizzato anche in studi preclinici per valutare l'acido re...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato finanziato dalla Science Foundation Ireland (SFI 08/RFP/BMT1689), dall'Health Research Board in Irlanda [HRA-POR/2012/4 e HRA-POR-2015-1145] e dal Royal City of Dublin Hospital Trust.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| IX51 Fluorescent Microscope | Olympus, Japan | N/A | AFB detection and imaging |
| 2 mL microtube, flat bottom, screw cap, sterile | Sarstedt, North Carolina, USA | 72.694.006 | Mtb infection of macropahges |
| 5 mL syringe, Luer lock | BD Biosciences, San Jose, CA, USA | SZR-150-031K | Mtb infection of macropahges/CFU |
| 50 mL tube, sterile | Sarstedt, North Carolina, USA | 62.547.254 | Mtb infection of macropahges |
| all-trans-Retinoic Acid (ATRA) ≥98% (HPLC) | Sigma Aldrich, Missouri, USA | R2625 | Host directed therapy candidate |
| BacT/ALERT 3D Microbial Detection System | Biomerieux ( Hampshire, UK) | 247001 | Broth-based colormetric detection system |
| BACT/ALERT MP BACT/ALERT MP Nutrient Supplement | Biomerieux ( Hampshire, UK) | 414997 | Broth-based colormetric detection assay |
| BACT/ALERT MP culture bottles | Biomerieux ( Hampshire, UK) | 419744 | Broth-based colormetric detection assay |
| BD BBL Middlebrook ADC Enrichment, 20 mL | BD Biosciences, San Jose, CA, USA | M0553 | Mycobacterium liquid culture |
| BD BBL Middlebrook OADC Enrichment, 20mL | BD Biosciences, San Jose, CA, USA | M0678 | Colony Forming Units |
| Cell scraper, 25 cm | Sarstedt, North Carolina, USA | 83.1830 | Harvest of lmacrophage lysates |
| Corning Syringe Filter, 0.2 µm | Corning Incorporated, Germany | 431219 | Sterilization of lysis buffer |
| Cover glass (borosilicate), 24 x 50 mm, #1.5 thickness | VWR International Limited | 631 - 0147 | |
| Cycloheximide, from microbial | Sigma Aldrich, Missouri, USA | C7698 | Colony Forming Units |
| Dako Fluorescent Mounting Medium | Agilent Technologies Ireland Limited | S3023 | Antifade mounting medium |
| Dulbecco’s Phosphate Buffered Saline | Sigma Aldrich, Missouri, USA | D8537 | Mtb infection of macropahges |
| Fetal Bovine Serum, Gibco | Thermo Fisher, Massachusetts, USA | 10270106 | Macrophage cell culture |
| Glycerol, Difco | BD Biosciences, San Jose, CA, USA | 228220 | Colony Forming Units |
| Hoescht 33342 (bisBenzimide H 33342 trihydrochloride) | Sigma Aldrich, Missouri, USA | B2261 | Nuclear stain |
| IFNγ, recombinant human | R&D Systems Inc, Minnesota, USA | 285-IF | Host directed therapy candidate |
| Labtek 2-well chamber slide, sterile, Nunc | Thermo Fisher, Massachusetts, USA | TKT-210-150R | Mtb infection of macropahges |
| L-Asparagine, anhydrous | Sigma Aldrich, Missouri, USA | A4159 | Colony Forming Units |
| Linezolid | Sigma Aldrich, Missouri, USA | PZ0014 | Antibiotic |
| Microlance Hypodermic Needle 25 G | BD Biosciences, San Jose, CA, USA | 300400 | Mtb infection of macropahges/CFU |
| Middlebrook 7H10 Agar Base | BD Biosciences, San Jose, CA, USA | M0303 | Colony Forming Units |
| Middlebrook 7H9 Broth Base | BD Biosciences, San Jose, CA, USA | M0178 | Mycobacterium liquid culture |
| Modified Auramine O Stain and Decolourizer | Scientific Device Laboratory, IL, USA | 345-250 | AFB stain |
| Paraformaldehyde | Sigma Aldrich, Missouri, USA | 158127 | Mtb infection of macropahges |
| Petri dishes, 92 x 16mm (20/bag) | Sarstedt, North Carolina, USA | 82.1473.001 | Colony Forming Units |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma Aldrich, Missouri, USA | P8139 | Macrophage cell culture |
| Polysorbate 80, Difco | BD Biosciences, San Jose, CA, USA | 231181 | Mycobacterium liquid culture |
| RPMI-1640, Gibco | Thermo Fisher, Massachusetts, USA | 52400025 | Macrophage cell culture |
| Sterile Cell Spreader, L-Shaped | Fisherbrand, Thermo Fisher, MA, USA | RB-44103 | Colony Forming Units |
| T25 TC flask, angled neck, filter cap, sterile, Nunc | Thermo Fisher, Massachusetts, USA | 156367 | Mycobacterium liquid culture |
| THP-1 cell line | ATCC, Virginia, USA | ATCC TIB-202 | Macrophage cell culture |
Riferimenti
- WHO. Global Tuberculosis Report. World Health Organization. , Geneva. (2020).
- Russell, D. G. Mycobacterium tuberculosis and the intimate discourse of a chronic infection. Immunological Reviews. 240 (1), 252-268 (2011).
- Young, C., Walzl, G., Du Plessis, N. Therapeutic host-directed strategies to improve outcome in tuberculosis. Mucosal Immunology. 13 (2), 190-204 (2020).
- Angeby, K. A., et al. Evaluation of the BacT/ALERT 3D system for recovery and drug susceptibility testing of Mycobacterium tuberculosis. Clinical Microbiology and Infection. 9 (11), 1148-1152 (2003).
- Asmar, S., Drancourt, M. Rapid culture-based diagnosis of pulmonary tuberculosis in developed and developing countries. Frontiers in Microbiology. 6, 1184(2015).
- Diacon, A. H., et al. Time to liquid culture positivity can substitute for colony counting on agar plates in early bactericidal activity studies of antituberculosis agents. Clinical Microbiology and Infection. 18 (7), 711-717 (2012).
- Diacon, A. H., et al. Time to positivity in liquid culture predicts colony forming unit counts of Mycobacterium tuberculosis in sputum specimens. Tuberculosis. 94 (2), Edinburgh. 148-151 (2014).
- O'Sullivan, D. M., et al. Evaluation of liquid culture for quantitation of Mycobacterium tuberculosis in murine models. Vaccine. 25 (49), 8203-8205 (2007).
- Heinrichs, M., et al. Mycobacterium tuberculosis Strains H37ra and H37rv have equivalent minimum inhibitory concentrations to most antituberculosis drugs. International Journal of Mycobacteriology. 7 (2), 156-161 (2018).
- Sorrentino, F., et al. Development of an intracellular screen for new compounds able to inhibit mycobacterium tuberculosis growth in human macrophages. Antimicrobial Agents and Chemotherapy. 60 (1), 640-645 (2016).
- O'Leary, S. M., et al. Cigarette smoking impairs human pulmonary immunity to Mycobacterium tuberculosis. American Journal of Respiratory and Critical Care Medicine. 190 (12), 1430-1436 (2014).
- Ryan, R. C., O'Sullivan, M. P., Keane, J. Mycobacterium tuberculosis infection induces non-apoptotic cell death of human dendritic cells. BMC Microbiology. 11, 237(2011).
- Keane, J., Remold, H. G., Kornfeld, H. Virulent mycobacterium tuberculosis strains evade apoptosis of infected alveolar macrophages. The Journal of Immunology. 164 (4), 2016-2020 (2000).
- Gao, X. F., Yang, Z. W., Li, J. Adjunctive therapy with interferon-gamma for the treatment of pulmonary tuberculosis: a systematic review. International Journal of Infectious Diseases. 15 (9), 594-600 (2011).
- Thorpe, T. C., et al. BacT/Alert: an automated colorimetric microbial detection system. Journal of Clinical Microbiology. 28 (7), 1608-1612 (1990).
- Gleeson, L. E., et al. Cutting edge: Mycobacterium tuberculosis induces aerobic glycolysis in human alveolar macrophages that is required for control of intracellular bacillary replication. The Journal of Immunology. 196 (6), 2444-2449 (2016).
- O'Connor, G., et al. Inhalable poly(lactic-co-glycolic acid) (PLGA) microparticles encapsulating all-trans-Retinoic acid (ATRA) as a host-directed, adjunctive treatment for Mycobacterium tuberculosis infection. European Journal of Pharmaceutics and Biopharmaceutics. 134, 153-165 (2019).
- O'Connor, G., et al. Sharpening nature's tools for efficient tuberculosis control: A review of the potential role and development of host-directed therapies and strategies for targeted respiratory delivery. Advanced Drug Delivery Reviews. 102, 33-54 (2016).
- Rodriguez, D. C., Ocampo, M., Salazar, L. M., Patarroyo, M. A. Quantifying intracellular Mycobacterium tuberculosis: An essential issue for in vitro assays. Microbiologyopen. 7 (2), 00588(2018).
- Ortalo-Magne, A., et al. Molecular composition of the outermost capsular material of the tubercle bacillus. Microbiology. 141, Reading. Pt 7 1609-1620 (1995).
- Stokes, R. W., et al. The glycan-rich outer layer of the cell wall of Mycobacterium tuberculosis acts as an antiphagocytic capsule limiting the association of the bacterium with macrophages. Infection and Immunity. 72 (10), 5676-5686 (2004).
- O'Sullivan, M. P., O'Leary, S., Kelly, D. M., Keane, J. A caspase-independent pathway mediates macrophage cell death in response to Mycobacterium tuberculosis infection. Infection and Immunity. 75 (4), 1984-1993 (2007).
- Cheon, S. H., et al. Bactericidal activity in whole blood as a potential surrogate marker of immunity after vaccination against tuberculosis. Clinical and Diagnostic Laboratory Immunology. 9 (4), 901-907 (2002).
- Nathan, C., Barry, C. E. TB drug development: immunology at the table. Immunological Reviews. 264 (1), 308-318 (2015).
- Bowness, R., et al. The relationship between Mycobacterium tuberculosis MGIT time to positivity and cfu in sputum samples demonstrates changing bacterial phenotypes potentially reflecting the impact of chemotherapy on critical sub-populations. Journal of Antimicrobial Chemotherapy. 70 (2), 448-455 (2015).
- Basu Roy, R., et al. An auto-luminescent fluorescent BCG whole blood assay to enable evaluation of paediatric mycobacterial responses using minimal blood volumes. Frontiers in Pediatrics. 7, 151(2019).
- Christophe, T., et al. High content screening identifies decaprenyl-phosphoribose 2' epimerase as a target for intracellular antimycobacterial inhibitors. PLoS Pathogens. 5 (10), 1000645(2009).
- Franzblau, S. G., et al. Comprehensive analysis of methods used for the evaluation of compounds against Mycobacterium tuberculosis. Tuberculosis. 92 (6), Edinburgh. 453-488 (2012).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon